ASPRV1作为生物标志物在喉鳞癌诊疗中的应用的制作方法
本发明属于生物医药领域,涉及asprv1作为生物标志物在喉鳞癌诊疗中的应用。
背景技术:
喉癌是耳鼻咽喉头颈外科最常见的恶性肿瘤之一,占全身癌症的5.7%-7.6%。喉癌中96%~98%为鳞状细胞癌,其他病理类型的癌如:腺癌,肉瘤较少见。喉癌好发于四十到六十岁,男性病人居多。近年来经济不断发展,生活环境的恶化,人民生活方式的改变,喉癌的发病率在逐步上升。手术切除肿瘤是喉癌的首选治疗,加上放射治疗、化疗及基因靶向治疗等的配合,喉癌病人的5年生存率及喉功能的恢复有了较大的提高,但是晚期喉癌的患者治疗效果不理想,一旦伴有颈部淋巴结转移,其5年存活率为50%,肿瘤向远处转移,其5年生存率仅为20%左右。近几十年里,喉癌的诊疗理念发生了很大的变化,诊疗方案的选择取决于疾病所处的阶段。目前喉癌的发病原因考虑与不良的生活习惯(吸烟、饮酒),成人型喉乳头状瘤病毒,长期接触放射线,某些微量元素的缺乏等有关,其病因仍不明确。因此,如何从分子水平阐明喉鳞癌的发生发展机制,寻求理想的分子标志物对喉癌病人进行早期诊断,对其治疗的预后效果非常重要。
随着生物学技术的发展,高通量技术的出现为疾病发病机理的研究提供了更加全面和快速的分析手段,采用高通量测序技术结合生物信息学分析,寻找与疾病发生发展相关的重要基因,并结合定向的生物学实验,为实现疾病精准化治疗提供了新途径。
技术实现要素:
为了弥补现有技术的不足,本发明的目的在于提供一种与喉鳞癌发生发展相关的生物标志物,通过检测样本中生物标志物的表达水平,可以判断患者是否患有喉鳞癌或者患喉鳞癌的风险的大小,通过靶向于生物标志物,改变生物标志物的表达水平或活性,可以实现喉鳞癌患者的精准靶向治疗。
为了实现上述目的,本发明采用如下技术方案:
本发明提供了检测asprv1的试剂在制备诊断喉鳞癌的产品中的应用。
进一步,所述检测asprv1的试剂包括:通过测序技术、核酸杂交技术、核酸扩增技术或免疫测定的方法检测样本中asprv1基因的表达水平用试剂。
进一步,所述试剂选自:
特异性识别asprv1基因的探针;或
特异性扩增asprv1基因的引物;或
特异性结合asprv1编码的蛋白的抗体或配体。
进一步,所述试剂至少包括一对特异性扩增asprv1的引物,在本发明的具体实施例中,所述特异性扩增asprv1基因的引物序列如seqidno.3~4所示。
本发明中,可以采用任何方法检测asprv1的表达水平。
本发明提供了一种诊断喉鳞癌的产品,所述产品包括检测样品中asprv1表达水平的制剂、芯片或试剂盒,其中芯片包括基因芯片、蛋白芯片;所述基因芯片包括固相载体以及固定在固相载体的寡核苷酸探针,所述寡核苷酸探针包括用于检测asprv1基因转录水平的针对asprv1基因的寡核苷酸探针;所述蛋白芯片包括固相载体以及固定在固相载体的针对asprv1蛋白的特异性抗体;试剂盒包括基因检测试剂盒、蛋白检测试剂盒。
本发明所述的产品可用于检测包括asprv1在内的多个基因或基因表达产物的表达水平(与喉鳞癌相关的多个基因或其表达产物),将喉鳞癌的多个标志物同时进行检测,可大大提高喉鳞癌诊断的准确率。
本发明提供了asprv1在筛选预防或治疗喉鳞癌的候选化合物中的应用。
进一步,所述筛选候选化合物的步骤如下:
测试组中,在培养体系中添加测试化合物,并观察所述测试组的细胞中asprv1的表达量和/或活性;在对照组中,在相同的培养体系中不添加测试化合物,并观察对照组的所述细胞中asprv1的表达量和/或活性;
其中,如果测试组中细胞的asprv1的表达量和/或活性低于对照组,就表明该测试化合物是对asprv1的表达和/或活性有抑制作用的治疗癌症的候选化合物。
在本发明中,所述的步骤还包括:对获得的候选化合物进行进一步的细胞实验和/或动物试验,以从候选化合物中进一步选择和确定对于预防、缓解或治疗喉鳞癌有用的物质。
在本发明中,筛选预防或治疗喉鳞癌的候选化合物的体系不限于细胞体系,还包括细胞体系、亚细胞体系、溶液体系、组织体系、器官体系或动物体系等,所述体系不局限于上述形式,只要所述体系可以检测测试化合物可以降低asprv1的表达和/活性即可。
所述候选化合物包括但不限于:针对asprv1基因或其编码的蛋白或其上游或下游基因或蛋白设计的干扰分子、核酸抑制物、结合分子(如抗体或配体)、小分子化合物等。
本发明提供了asprv1功能性表达的抑制剂在制备预防或治疗喉鳞癌的药物组合物中的应用。
进一步,所述asprv1功能性表达的抑制剂选自:核酸抑制物,蛋白抑制剂,蛋白水解酶,蛋白结合分子,优选的所述抑制剂为核酸抑制物sirna。
在本发明中,所述asprv1的抑制剂还可用于抑制喉鳞癌细胞的侵袭和增殖。
在本发明的具体实施例中,所述sirna的序列如seqidno.11~12所示。
本发明提供了一种治疗喉鳞癌的药物组合物,所述药物组合物包括asprv1功能性表达的抑制剂。
所述抑制剂选自:以asprv1或其转录本为靶序列、且能够抑制asprv1基因表达或基因转录的干扰分子,包括:shrna(小发夹rna)、小干扰rna(sirna)、dsrna、微小rna、反义核酸,或能表达或形成所述shrna、小干扰rna、dsrna、微小rna、反义核酸的构建物;或特异性与asprv1编码的蛋白结合的结合分子(如能够抑制asprv1蛋白活性的抗体或配体)。
进一步,所述药物组合物还包括药学上可接受的载体。所述载体包括(但不限于)稀释剂、赋形剂、粘合剂、湿润剂、吸收促进剂、表面活性剂、致湿剂、吸附载体、润滑剂、缓冲剂、稳定剂、抑菌剂、等渗剂、螯合剂、ph控制剂。
附图说明
图1是利用qpcr检测asprv1基因在喉鳞癌组织中的表达情况图;
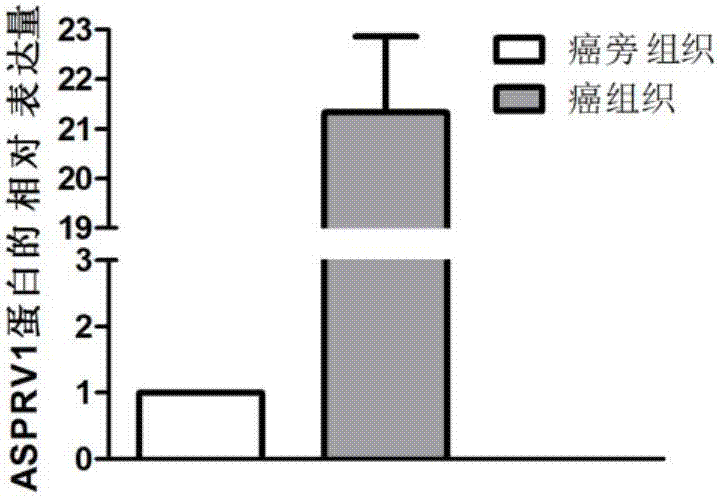
图2是利用westernblot检测asprv1蛋白在喉鳞癌组织中的表达情况图;
图3是利用qpcr检测sirna转染对喉鳞癌细胞中asprv1基因表达的影响图;
图4是利用westernblot检测sirna转染对喉鳞癌细胞中asprv1蛋白表达的影响图;
图5是用mtt法检测asprv1基因对喉鳞癌细胞增殖的影响图;
图6是用流式细胞仪检测asprv1基因对喉鳞癌细胞凋亡的影响图;
图7是利用细胞划痕实验检测asprv1对喉鳞癌细胞迁移的影响图;
图8是利用transwell小室检测asprv1对喉鳞癌细胞侵袭的影像图。
具体的实施方式
本发明经过广泛而深入的研究,通过高通量测序方法,检测喉鳞癌标本中基因在肿瘤组织和癌旁组织的表达,发现其中具有明显表达差异的基因,探讨其与喉鳞癌的发生之间的关系,从而为喉鳞癌的早期检测及靶向治疗寻找更好的途径和方法。通过筛选,本发明首次发现了喉鳞癌中asprv1显著性上调。实验证明,通过降低asprv1的表达水平,能够有效地抑制喉鳞癌细胞的生长和侵袭,提示检测asprv1基因的表达水平可成为喉鳞癌早期诊断的辅助诊断指标之一,干扰asprv1基因表达可成为预防或治疗喉鳞癌或喉鳞癌转移的新途径。
标志物
标志物(单独使用或与其他定性术语组合例如喉鳞癌标志物、喉鳞癌特异性标志物、对照标志物、外源标志物、内源标志物)指可测量、可计算或可以其他方式获得的,与任何分子或分子组合相关,可用作生物和/或化学状态的指示物的参数。在本发明中,“标志物”指与一个或多个生物分子相关的参数(即“生物标志物”)例如天然或人工合成产生的核酸(即个体基因,以及编码和非编码dna和rna)和蛋白(例如肽、多肽)。本发明中的“标志物”还包括指可通过考虑来自两个或多个不同标志物的表达数据计算或以其他方式获得的单个参数。
喉鳞癌标志物指可以用作(单独或与其他标志物组合)受试者中喉鳞癌指示物的特定类型的标志物,在本发明的具体实施方案中,喉鳞癌标志物可用于提供(单独或与其他标志物组合)受试者中喉鳞癌临床评估的标志物。
asprv1基因
asprv1是位于人2号染色体短臂1区3带上,本发明中的asprv1包括野生型、突变型或其片段。一种代表性的asprv1基因seqidno.1和seqidno.2所示的核苷酸序列和氨基酸序列。本发明的人asprv1核苷酸全长序列或其片段通常可以用pcr扩增法、重组法或人工合成的方法获得。
检测技术(方法))
本发明的基因使用本领域普通技术人员已知的多种检测技术进行检测,这些技术包括但不限于:核酸测序、核酸杂交、核酸扩增技术、免疫检测技术。
核酸测序技术的示例性非限制性实例包括但不限于链终止子(sanger)测序和染料终止子测序。本领域的普通技术人员将认识到,由于rna在细胞中不太稳定并且在实验中更易受到核酸酶攻击,因此在测序前通常将rna逆转录成dna。
核酸测序技术的另一示例性非限制性实例包括下一代测序(深度测序/高通量测序),高通量测序技术是一种基于单分子簇的边合成边测序技术,基于专有的可逆终止化学反应原理。测序时将基因组的dna的随机片段附着到光学透明的玻璃表面,这些dna片段经过延伸和桥式扩增后,在玻璃表面形成数以亿计的簇,每个簇是具有数千份相同模板的单分子簇,然后利用带荧光基团的四种特殊脱氧核糖核苷酸,通过可逆性的边合成边测序技术对待测的模板dna进行测序。
核酸杂交技术的示例性非限制性实例包括但不限于原位杂交(ish)、微阵列和southern或northern印迹。原位杂交(ish)是一种使用标记的互补dna或rna链作为探针以定位组织一部分或切片(原位)或者如果组织足够小则为整个组织(全组织包埋ish)中的特异性dna或rna序列的杂交。dnaish可用于确定染色体的结构。rnaish用于测量和定位组织切片或全组织包埋内的mrna和其他转录本(例如,ncrna)。通常对样本细胞和组织进行处理以原位固定靶转录本,并增加探针的进入。探针在高温下与靶序列杂交,然后将多余的探针洗掉。分别使用放射自显影、荧光显微术或免疫组织化学,对组织中用放射、荧光或抗原标记的碱基标记的探针进行定位和定量。ish也可使用两种或更多种通过放射性或其他非放射性标记物标记的探针,以同时检测两种或更多种转录本。
本发明可在检测前或与检测同时地对核酸(例如,ncrna)进行扩增。核酸扩增技术的示例性非限制性实例包括但不限于:聚合酶链式反应(pcr)、逆转录聚合酶链式反应(rt-pcr)、转录介导的扩增(tma)、连接酶链式反应(lcr)、链置换扩增(sda)和基于核酸序列的扩增(nasba)。本领域的普通技术人员将认识到,某些扩增技术(例如,pcr)需要在扩增前将rna逆转录成dna(例如,rt-pcr),而其他扩增技术则直接扩增rna(例如,tma和nasba)。
通常称为pcr的聚合酶链式反应使用变性、引物对与相反链的退火以及引物延伸的多个循环,以指数方式增加靶核酸序列的拷贝数;tma的转录介导的扩增(在基本上恒定的温度、离子强度和ph的条件下自身催化地合成靶核酸序列的多个拷贝,其中靶序列的多个rna拷贝自身催化地生成另外的拷贝;lcr的连接酶链式反应使用与靶核酸的相邻区域杂交的两组互补dna寡核苷酸;其他扩增方法包括例如:通常称为nasba的基于核酸序列的扩增;使用rna复制酶(通常称为qβ复制酶)扩增探针分子本身的扩增;基于转录的扩增方法;以及自我维持的序列扩增。
本发明中非扩增或扩增的核酸可通过任何常规的手段检测。
芯片、试剂盒
在本发明中,“芯片”、“微阵列”、“阵列”可以等同替代,包括但不限于:dna微阵列(例如,cdna微阵列和寡核苷酸微阵列)、蛋白质微阵列、组织微阵列、转染或细胞微阵列、化学化合物微阵列和抗体微阵列。通常称为基因芯片、dna芯片或生物芯片的dna微阵列是微观dna点的集合,这些点连接到固体表面(例如,玻璃、塑料或硅芯片)上,形成用于对数千种基因同时进行表达谱分析或表达水平监测的阵列。固定的dna片段称为探针,其数千个可用于单个dna微阵列中。微阵列可用于通过比较疾病和正常细胞中的基因表达而识别疾病基因或转录本(例如,ncrna)。微阵列可使用多种技术加以制造,包括但不限于:用细尖针印刷到载玻片上、使用预制的掩模进行光刻、使用动态微镜器件进行光刻、喷墨印刷或微电极阵列上的电化学方法。
本发明中的试剂盒可用于检测asprv1的表达,优选的,所述的试剂盒包括检测有效量的检测asprv1基因的试剂,选自下组的一种或多种物质:容器、使用说明书、阳性对照物、阴性对照物、缓冲剂、助剂或溶剂。例如用于混悬或固定细胞的溶液,可检测的标签或标记,使核酸易于杂交的溶液,用于裂解细胞的溶液,或用于核酸纯化的溶液。
本发明的试剂盒中还可附有试剂盒的使用说明书,其中记载了如何采用试剂盒进行检测,和如何利用检测结果对肿瘤发展进行判断、对治疗方案进行选择。
采用本发明的试剂盒,可通过选自下组的各种方法(包括但不限于)检测asprv1:实时定量反转录pcr、生物芯片检测法、dna印迹法、或rna印迹法或原位杂交法。本领域普通技术人员可根据实际条件和需要对检测方式进行调整和改变。
抑制剂和药物组合物
基于发明人的发现,本发明提供了一种asprv1的抑制剂的用途,用于制备抑制喉鳞癌的药物组合物。如本文所用,所述的asprv1的抑制剂包括但不限于抑制剂、拮抗剂、阻滞剂、阻断剂、核酸抑制物等。
所述的asprv1基因或蛋白的抑制剂是指任何可降低asprv1蛋白的活性、降低asprv1基因或蛋白的稳定性、下调asprv1蛋白的表达、减少asprv1蛋白有效作用时间、或抑制asprv1基因的转录和翻译的物质,这些物质均可用于本发明,作为对于下调asprv1有用的物质,从而可用于预防或治疗喉鳞癌。例如,所述的抑制剂是:核酸抑制物,蛋白抑制剂,抗体,配体,蛋白水解酶,蛋白结合分子,只要其能够在蛋白或基因水平上下调asprv1蛋白或其编码基因的表达。
作为本发明的一种选择方式,所述的asprv1的抑制剂是一种特异性与asprv1结合的抗体。所述特异性抗体包括单克隆抗体、多克隆抗体;本发明不仅包括完整的抗体分子,也包括抗体的任何片段或修饰,例如,嵌合抗体、scfv、fab、f(ab’)2、fv等。只要所述片段能够保留与asprv1蛋白的结合能力即可。用于蛋白质水平的抗体的制备时本领域技术人员公知的,并且本发明可以使用任何方法来制备所述抗体
作为本发明的一种优选方式,所述asprv1的抑制剂是一种asprv1特异性的小干扰rna分子。如本文所用,所述的“小干扰rna”是指一种短片段双链rna分子,能够以同源互补序列的mrna为靶目标降解特定的mrna,这个过程就是rna干扰(rnainterference)过程。小干扰rna可以制备成双链核酸的形式,它含有一个正义链和一个反义链,这两条链仅在杂交的条件下形成双链。一个双链rna复合物可以由相互分离的正义链和反义链来制备。因此,举例来讲,互补的正义链和反义链是化学合成的,其后可通过退火杂交,产生合成的双链rna复合物。
在筛选有效的sirna序列时,本发明人通过大量的比对分析,从而找出最佳的有效片段。本发明人设计合成了多种sirna序列,并将它们分别通过转染试剂转染喉鳞癌细胞系进行验证,选出干扰效果最佳的sirna,它们分别具有seqidno.11、seqidno.12所示的序列,进一步地在细胞水平实验,结果证明对于细胞试验而言抑制效率非常高。
本发明的核酸抑制物如sirna可以化学合成,也可以通过一个重组核酸结构里的表达盒转录成单链rna之后进行制备。sirna等核酸抑制物,可通过采用适当的转染试剂被输送到细胞内,或还可采用本领域已知的多种技术被输送到细胞内。
作为本发明的一种可选方式,所述的asprv1的抑制剂也可以是一种“小发夹rna(smallhairpinrna,shrna)”,其是能够形成发夹结构的非编码小rna分子,小发夹rna能够通过rna干扰途径来抑制基因的表达。如上述,shrna可以由双链dna模板来表达。双链dna模板被插进一个载体,例如质粒或病毒载体,然后在体外或体内连接到一个启动子进行表达。shrna在真核细胞内dicer酶的作用下,可被切割成小干扰rna分子,从而进入rnai途径。“shrna表达载体”是指一些本领域常规用于构建shrna结构的质粒,通常该质粒上存在“间隔序列”以及位于“间隔序列”两边的多克隆位点或供替换序列,从而人们可以将shrna(或类似物)相应的dna序列通过正向和反向的方式插入多克隆位点或替换其上的供替换序列,该dna序列转录后的rna可形成shrna(shorthairpin)结构。所述的“shrna表达载体”目前已经完全可以通过商购的途径购买获得,例如一些病毒载体。
本发明还提供了一种药物组合物,它含有有效量的所述的asprv1的抑制剂,以及药学上可接受的载体。所述的组合物可用于抑制喉鳞癌。任何前述的asprv1的抑制剂均可用于组合物的制备。所述载体包括(但不限于)稀释剂、赋形剂、粘合剂、崩解剂、吸收促进剂、表面活性剂、致湿剂、吸附载体、润滑剂、缓冲剂、稳定剂、抑菌剂、等渗剂、螯合剂、ph控制剂。
其中,稀释剂如乳糖、氯化钠、葡萄糖、尿素、淀粉、水等;赋形剂如乳糖、氯化钠、葡萄糖、尿素、淀粉、水等;粘合剂如单糖浆、葡萄糖溶液、淀粉溶液、纤维素衍生物、藻酸盐、明胶和聚乙烯吡咯烷酮;崩解剂如干淀粉、海藻酸钠、海带多糖粉末、琼脂粉末、碳酸钙和碳酸氢钠;吸收促进剂季铵化合物、十二烷基硫酸钠等;表面活性剂如聚氧化乙烯山梨聚糖脂肪酸酯、十二烷基硫酸钠、硬脂酸单甘油酯、十六烷醇等;致湿剂如甘油、淀粉等;吸附载体如淀粉、乳糖、斑脱土、硅胶、高岭土和皂粘土等;润滑剂如滑石粉、硬脂酸钙和镁、聚乙二醇、硼酸粉末等;缓冲剂可以包括硼酸、磷酸、乙酸、柠檬酸、谷氨酸及相应的盐(它们的碱金属或碱性稀土金属盐,例如钠盐、钾盐、钙盐及镁盐);稳定剂包括人类血清蛋白、l-氨基酸、糖及纤维素衍生物;抑菌剂包括但不限于有效浓度(例如<1%w/v)的苄醇、苯酚、间甲酚、氯丁醇、对羟基苯甲酸甲酯和/或对羟基苯甲酸丙酯;等渗剂包括氯化钾、氯化钠、糖及甘油;螯合剂包括乙二胺四乙酸钠及柠檬酸;
如本文所用,所述“有效量”是指可对人和/或动物产生功能或活性的且可被人和/或动物所接受的量。抑制剂的有效量可随给药的模式和待治疗的疾病的严重程度等而变化。优选的有效量的选择可以由本领域普通技术人员根据各种因素来确定(例如通过临床试验)。所述的因素包括但不限于:所述的asprv1基因的抑制剂的药代动力学参数例如生物利用率、代谢、半衰期等;患者所要治疗的疾病的严重程度、患者的体重、患者的免疫状况、给药的途径等。
本发明所述药物组合物可口服给药、非胃肠道给药、通过吸入喷雾给药、局部给药、直肠给药、鼻给药、颊给药、阴道给药或通过植入的贮药装置给药。优选口服给药或注射给药。本发明药物组合物可含有任何常用的无毒可药用载体、辅料或赋形剂。
本发明的药物组合物还可以与其他治疗喉鳞癌的药物联用,其他治疗性化合物可以与主要的活性成分同时给药,甚至在同一组合物中同时给药。还可以以单独的组合物或与主要的活性成分不同的剂量形式单独给予其它治疗性化合物。
优选的,可采用基因治疗的手段进行。比如,可直接将asprv1的抑制剂通过诸如注射等方法给药于受试者;或者,可通过一定的途径将携带asprv1的抑制剂的表达单位(比如表达载体或病毒等,或sirna或shrna)递送到靶点上,并使之表达活性的asprv1抑制剂,具体情况需视所述的抑制剂的类型而定,这些均是本领域技术人员所熟知的。
本发明中,术语“样本”以其最广泛的含义使用。意欲包括任何源自活或死亡的人的组织或材料,其可包括本发明的标志物。在本发明的具体实施例中,样品可以是肿瘤或肺肿瘤组织,并且可包括例如含有来自其的与喉组织相关的细胞或标志物的任何组织或材料。
下面结合附图和实施例对本发明作进一步详细的说明。以下实施例仅用于说明本发明而不用于限制本发明的范围。实施例中未注明具体条件的实验方法,通常按照常规条件,例如sambrook等人,分子克隆:实验室手册(newyork:coldspringharborlaboratorypress,1989)中所述的条件,或按照制造厂商所建议的条件。
实施例1筛选与喉鳞癌相关的基因标志物
1、样品收集
各收集6例喉鳞癌组织及对应的正常黏膜组织样本,组织样本的取得获得患者的知情同意,并且取得均通过组织伦理委员会的同意。
2、rna样品的制备
1)加入液氮研磨组织至粉末,加入1mltrizol(invitrogen)溶液,吹打混匀,使组织充分裂解,静置5min;
2)12000rpm4℃离心5min,将上清液转移至1.5mlrnasefreeep管中;
3)加入200μl氯仿,剧烈振荡混匀30s,使水相和有机相充分接触,室温静置15min;
4)4℃环境中12000g离15min,溶液离心为三层,rna在上层水相,移至另一个新的rnasefreeep管;
5)加入0.5ml异丙醇,轻柔充分混匀,室温静置10min;
6)4℃下,12000g离心10min,加入与rnaisoplus等体积的75%乙醇沉淀rna,7500g4℃离心5min,去上清;
7)用75%乙醇洗涤两次,超净台风干;加入30μldepc水溶解沉淀。
8)rna样品的质量分析
利用nanodrop2000对所提取的rna浓度和纯度进行检测,琼脂糖凝胶电泳检测rna完整性,agilent2100测定rin值。浓度≥200ng/μl,od260/280介于1.8~2.2之间。
3、去除rrna
使用ribo-zero试剂盒除去总rna中的核糖体rna。
4、构建cdna文库
利用利用illumina的truseqrnasampleprepkit进行cdna文库的构建,具体操作按说明书进行。
5、上机测序
使用hiseq4000测序平台对cdna文库进行测序,具体操作按说明书进行。
6、高通量转录组测序数据分析
对测序结果进行生物信息学分析,利用tophatv1.3.1进行rna-seq读段定位,通过cufflinksv1.0.3将rna-seq片段数目进行标准化计算转录本的相对丰度,利用cuffdiff检测差异表达,当p值<0.001,|log2(fold_change)normalized|>2时,认为mrna显著差异表达。
7、结果
rna-seq结果显示,在喉鳞癌患者中差异表达的基因有665个,上调的450个,下调的215个,其中基因asprv1在喉鳞癌组织中的表达量显著高于正常癌旁黏膜组织中的表达量。
实施例2qpcr测序验证asprv1基因的差异表达
1、对asprv1基因差异表达进行大样本qpcr验证。按照实施例1中的样本收集方式选择喉鳞癌患者正常癌旁粘膜组织和喉鳞癌组织各50例。
2、rna提取具体步骤如实施例1所述。
3、逆转录
3、逆转录:采用fastquantcdna第一链合成试剂盒(货号:kr106)进行mrna反转录。具体步骤如下:
(1)加入5×gdnabuffer2.0μl,总rna1μg,加rnasefreeddh2o使总体积至10μl,水浴锅中42℃加热3min;
(2)构建20μl反应体系,10×fastrtbuffer2.0μl,rtenzymemix1.0μl,fq-rtprimermix2.0μl,rnasefreeddh2o5.0μl混合后加入(1)中的混合液中混匀;
(3)水浴锅中42℃加热15min,95℃加热3min,-20℃储存备用。
4、qpcr扩增
(1)引物设计
根据genebank中asprv1基因和管家基因gapdh基因的编码序列设计qpcr扩增引物,由博迈德公司合成。
asprv1基因的引物序列:
正向引物序列为5’-aaccattgggaccatatc-3’(seqidno.3);
反向引物序列为5’-actgagcctattgtagac-3’(seqidno.4)。
gapdh基因的引物序列:
正向引物序列为5’-ggagcgagatccctccaaaat-3’(seqidno.5);
反向引物序列为5’-ggctgttgtcatacttctcatgg-3’(seqidno.6)。
(2)pcr反应体系:正向引物和反向引物各0.6μl,2×superrealpremixplus10μl,dna模板2μl,ddh2o7.4μl,50×roxreferencedye△2μl,灭菌蒸馏水4.8μl。
(3)pcr反应条件:95℃15min,(95℃10s,55℃30s,72℃32s)×40个循环,95℃15s,60℃60s,95℃15s。在abi7300型荧光定量pcr仪上进行pcr反应,通过融解曲线分析和电泳确定目的条带,δδct法进行相对定量。
5、统计学方法
实验都是按照重复3次来完成的,结果数据都是以平均值±标准差的方式来表示,采用spss18.0统计软件来进行统计分析的,癌与癌旁组织的配对比较采用t检验,认为当p<0.05时具有统计学意义。
6、结果
结果如图1所示,与喉鳞癌正常癌旁粘膜组织相比,asprv1在喉鳞癌组织中表达上调,差异具有统计学意义(p<0.05),同高通量测序结果一致。
实施例3蛋白免疫印记实验检测asprv1蛋白的差异表达
1、组织总蛋白的提取
用剪刀剪碎组织后将其放入置于冰内的玻璃匀浆器内,ripa裂解液和pmsf以100:1的比例混匀,按照每20mg组织标本加入100μl裂解液的比例加入相应量的ripa裂解液,玻璃匀浆器碾碎组织直至其充分裂解,将裂解后的液体吸至ep管中,4℃下14000rpm离心5min,收集上清。
2、总蛋白浓度测定
按照bca蛋白浓度测定试剂盒的说明书进行蛋白浓度的测定。
3、sds-page电泳
按照sds-page凝胶配制试剂盒的说明书配制8%的分离胶和5%的浓缩胶并进行电泳。
4、western检测
1)电转移
将pvdf膜放入甲醇溶液中激活5min,放入转膜缓冲液中平衡20min。取出page胶放入转膜缓冲液中,切下相应的page胶,按照由下到上依次为滤纸、pvdf膜、page胶、滤纸的顺序放到半干的转膜仪中,恒压25v转膜1.5h;
2)免疫杂交
取出pvdf膜,pbs冲洗后置于5%bsa溶液中室温下摇动封闭2h,将pvdf膜放入杂交袋中,加入一抗过夜,用tbst缓冲液洗涤pvdf膜,再加入相应的二抗,室温下孵育2h,tbst缓冲液洗涤。
3)dab显色
pvdf膜稍干后滴加新鲜配制的dab显色液,pvdf膜显色后扫描记录。以β-actin作为内参照,采用quantityone凝胶成像分析系统对条带进行半定量灰度分析,实验重复3次,结果取平均灰度值;
5、统计学分析
采用spss18.0统计软件来进行统计分析,结果数据都是以平均值±标准差的方式来表示的,采用繁多样本均数的方差检验(anova)进行分析,认为当p<0.05时具有统计学意义。
6、结果
结果如图2所示,asprv1蛋白在喉鳞癌组织中的表达水平显著高于正常癌旁粘膜组织。
实施例4asprv1基因的沉默
1、细胞培养
人喉鳞癌细胞株hep2,以含10%胎牛血清和1%p/s的rpmi1640培养基在37℃、5%co2、相对湿度为90%的培养箱中培养。2-3天换液1次,细胞生长良好,呈单层贴壁生长。使用0.25%含edta的胰蛋白酶常规消化传代。
2、转染
1)转染前细胞的处理
转染前一天,6孔培养板上种3~5×105个细胞/孔,在无抗生素培养基中培养一天,转染时细胞密度为30~50%,于转染前换成无血清培养基。
2)sirna的设计
阴性对照sirna序列(sirna-nc):
正义链为5’-uucuccgaacgugucacgu-3’(seqidno.7)
反义链为5’-acgugacacguucggagaa-3’(seqidno.8)
sirna1:
正义链为5’-ucauugaugacuucaaagcug-3’(seqidno.9)
反义链为5’-gcuuugaagucaucaaugacc-3’(seqidno.10)
sirna2:
正义链为5’-acucuuucaggaaccuuagcu-3’(seqidno.11)
反义链为5’-cuaagguuccugaaagagucc-3’(seqidno.12)
sirna3:
正义链为5’-cuaagguuccugaaagagucc-3’(seqidno.13)
反义链为5’-ccugaaagggaagaaguuucg-3’(seqidno.14)
将实验分为三组:对照组(hep2)、阴性对照组(sirna-nc)和实验组(sirna1、sirna2、sirna3),其中阴性对照组sirna与asprv1基因的序列无同源性。
3)转染
a.取浓度为50pmol的sirna3μl加入47μl的无血清培养基,轻轻混匀,室温孵育5min;
b.取1μllipofectamine2000加入49μl无血清培养基。轻轻混匀,室温孵育5min;
c.将上述两种混合物混合(总体积100μl),轻轻混匀,室温下孵育25min,以使复合体形成;
d.在6孔板中每孔加入100μl的复合物及适量培养基,轻轻混匀;
e.孵育48~96h后观察基因的沉默效应。
5、qpcr检测asprv1基因的转录水平
5.1细胞总rna的提取
采用qiagen的细胞rna提取试剂盒对细胞中的rna进行提取,实验操作按照说明书进行。
5.2逆转录步骤同实施例2。
5.3qpcr扩增步骤同实施例2。
6、统计学方法
实验都是按照重复3次来完成的,结果数据都是以平均值±标准差的方式来表示,采用spss18.0统计软件来进行统计分析的,asprv1基因实验组与对照组之间的差异采用t检验,认为当p<0.05时具有统计学意义。
7、结果
结果如图3显示,与非转染组与转染sirna-nc组相比,实验组sirna2表达水平降低最为显著,差异具有统计学意义(p<0.05)。
实施例5westernblot检测转染sirna对asprv1蛋白表达的影响
1、细胞培养和转染
步骤同实施例4
2、细胞总蛋白的提取
收集处于对数期的不同处理组的细胞,用预冷的pbs洗涤细胞。将ripa细胞裂解液和pmsf以100:1的比例混匀,向细胞中加入上述裂解液150μl,冰上放置30min,使用细胞刮刀将裂解的细胞刮下来,使用移液器将裂解后的液体吸至ep管中,4℃下14000rpm离心5min。小心收集离心后的上清。
3、总蛋白浓度测定
按照bca蛋白浓度测定试剂盒的说明书进行蛋白浓度的测定。
4、sds-page电泳
按照sds-page凝胶配制试剂盒的说明书配制8%的分离胶和5%的浓缩胶并进行电泳。
5、western检测
步骤详见实施例3。
6、统计学分析
采用spss18.0统计软件来进行统计分析,结果数据都是以平均值±标准差的方式来表示的,采用繁多样本均数的方差检验(anova)进行分析,认为当p<0.05时具有统计学意义。
7、结果
结果如图4所示,与对照组相比,转染sirna2组的细胞中的asprv1蛋白表达量显著下调。
实施例6asprv1基因对喉鳞癌细胞增殖的影响
采用mtt实验检测asprv1基因对喉鳞癌细胞增殖能力影响。
1、取生长状况良好的细胞,常规消化成单细胞悬液后,计数细胞,将细胞稀释成合适浓度的细胞悬液;
2、于96孔培养板中,将稀释后的不同处理组细胞每孔接种2000细胞,至少设3个平行孔,37℃、5%co2培养24h;
3、于接种后1、2、3、4、5天每天取出3孔细胞以mtt法检测其490nm的od值,进行计数,计算平均值;
4、检测前弃去上清液,培养液洗3次,每孔加入mtt无血清培养基溶液(0.2mg/ml)100μl,37℃培养箱中继续培养4h;
5、终止培养,小心吸弃上清液,每孔加入150μldmso,震荡10min,使结晶物充分溶解,在酶标仪上用波长为490nm测定光密度(od)值,以时间为横轴,光密度值为纵轴绘制细胞生长曲线。
6、统计学分析
实验都是按照重复3次来完成的,采用spss18.0统计软件来进行统计分析,两者之间的差异采用t检验,认为当p<0.05时具有统计学意义。
7、结果
结果如图5所示,与对照相比,实验组在转染sirna2后,细胞的增殖明显受到了抑制,差异具有统计学意义(p<0.05)说明asprv1具有促进细胞增殖的作用。
实施例7asprv1基因对喉鳞癌细胞凋亡的影响
使用流式细胞仪检测asprv1基因对细胞凋亡的影响。
1、细胞培养步骤同实施例3。
2、细胞转染步骤同实施例3。
3、步骤
1)将处于对数生长期的不同处理组的细胞经胰酶消化后吹打成细胞悬液并计数。取106量的细胞悬液,1000rpm离心5min;
2)弃上清,加入195μlannexinv-fitc结合液轻轻重悬细胞;
3)加入5μl的annexinv-fitc,轻柔混匀,室温下避光孵育10min;
4)1000rpm离心5min,弃上清,加入190μl的annexinv-fitc结合液轻轻重悬细胞;
5)加入10μl碘化丙啶(pi)染色液,轻柔混匀,冰浴避光放置,进行流式细胞仪检测细胞凋亡情况,所有实验均重复3次,结果取平均值。
4、统计学方法
采用spss18.0统计软件来进行统计分析,结果数据都是以平均值±标准差的方式来表示的,采用繁多样本均数的方差检验(anova)进行分析,认为当p<0.05时具有统计学意义。
5、结果:
结果如图6所示,实验组与对照组相比,细胞的凋亡率无明显变化(p<0.05)。
实施例8细胞划痕实验
1、向6孔板中每孔加入1ml50μg/ml的纤连蛋白,并置于4℃冰箱中过夜;
2、弃去剩余的纤连蛋白溶液,用无血清培养基清洗,将处于对数生长期的不同处理组细胞经胰酶消化重悬后,接种于铺有纤连蛋白的6孔板中,每组细胞设2个复孔,每孔5×105个细胞;
3、置入37℃、5%co2培养箱中培养过夜;
4、待细胞长至约90%融合时,用10μl的tip头划出一条无细胞的细痕,pbs溶液洗去脱落的细胞,加入无血清培养基继续培养;
5、分别于划痕后0h、48h观察细胞划痕处的愈合情况并拍照。实验重复3次,结果取平均值。
6、统计学方法
采用spss18.0统计软件来进行统计分析,结果数据都是以平均值±标准差的方式来表示的,采用繁多样本均数的方差检验(anova)进行分析,认为当p<0.05时具有统计学意义。
7、结果
结果如图7所示,转染sirna2组的细胞相比对照组而言,细胞体外划痕后的迁移距离明显降低,而对照组之间无显著差异,说明asprv1过表达可以促进喉癌细胞的迁移。
实施例9细胞侵袭实验
1、transwell小室制备
将50mg/l的matrigel胶用4℃预冷的无血清培养基以1:8的比例稀释、混匀,包被transwell小室的底部膜的上室面,4℃风干。取60μl~80μl稀释的matrigel胶(3.9μg/μl)置于孔径为8μm的transwell上室的聚碳酸脂膜上,使膜上的所有的微孔均被matrigel覆盖,置于37℃30min使matrigel聚合成凝胶。
2、配置细胞悬液
将处于对数生长期的不同处理组的细胞经胰酶消化,无血清培养基重悬后,调整细胞浓度为5×104个/ml。
3、细胞接种
在transwell上室加入2ml的细胞悬液,下室加入1ml的含10%胎牛血清的完全培养基,放置于配套的6孔板上,37℃、5%co2条件下培养20-24h;取出transwell小室,棉签擦尽上室面的matrigel胶和未穿透膜的细胞。
4、染色
细胞培养结束后,取出transwell小室,棉签擦尽上室面的matrigel胶和未穿透膜的细胞,下室面用95%酒精固定15min后,苏木素染色2min,倒置显微镜下随机取5个高倍镜视野观察、计数并拍照。计数小室下室面的细胞数即为穿透matrigel胶的细胞数,取平均数作为实验结果,并以该细胞数代表肿瘤细胞的侵袭力,实验重复3次,每组细胞设3个复孔。
5、统计学方法
采用spss18.0统计软件来进行统计分析,结果数据都是以平均值±标准差的方式来表示的,采用繁多样本均数的方差检验(anova)进行分析,认为当p<0.05时具有统计学意义。
6、结果
结果如图8所示,与对照组相比,实验组的细胞穿过transwell小室聚碳酸酯膜的细胞数明显减少,而对照组之间无明显差异,结果说明asprv1过表达可以促进喉鳞癌的侵袭。
上述实施例的说明只是用于理解本发明的方法及其核心思想。应当指出,对于本领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以对本发明进行若干改进和修饰,这些改进和修饰也将落入本发明权利要求的保护范围内。
sequencelisting
<110>北京泱深生物信息技术有限公司
<120>asprv1作为生物标志物在喉鳞癌诊疗中的应用
<160>14
<170>patentinversion3.5
<210>1
<211>1032
<212>dna
<213>人源
<400>1
atggggagcccaggggccagcctaggcatcaaaaaggctctgcagagtgaacaggccaca60
gcactgcctgcctctgccccagcagtcagccagccgaccgcgcctgctccctcctgcttg120
cccaaggccgggcaagtcatccccactctgcttcgagaggccccgttttccagcgtgatt180
gcgccgacactgctctgtgggtttctcttcttggcgtgggttgctgctgaggttccagag240
gagagcagcaggatggccgggagcggagccaggagtgaggaaggccgccggcagcatgcc300
ttcgtcccggaaccttttgatggggccaatgtcgtcccaaacctctggctgcacagcttt360
gaagtcatcaatgacctcaaccattgggaccatatcaccaagctaaggttcctgaaagag420
tccctcagaggagaggccctgggtgtctacaataggctcagtccccaggaccagggagac480
tatgggactgtgaaagaggccctcctgaaggcctttggggtccctggggctgcccccagc540
cacctgcccaaagagatcgtctttgccaacagcatgggtaagggctactatctcaagggg600
aagattggcaaagtgcccgtgaggttcctggtggactctggggcccaggtctctgtggtc660
cacccaaacttgtgggaggaggtcactgatggcgatctggacaccctgcagccctttgag720
aatgtggtaaaggtggccaatggtgctgaaatgaagatcctgggtgtctgggatacagcg780
gtgtccctaggcaagctgaagctgaaggcacagttcctagtggccaatgcgagtgccgag840
gaagccatcattggcactgatgtgctccaggaccacaatgctatcctggactttgagcac900
cgcacatgcaccctgaaagggaagaagtttcgccttctgcctgtgggagggtccctggaa960
gatgagtttgacctggagctcatagaggaggacccctcctcagaagaagggcggcaggag1020
ctatcccactga1032
<210>2
<211>343
<212>prt
<213>人源
<400>2
metglyserproglyalaserleuglyilelyslysalaleuglnser
151015
gluglnalathralaleuproalaseralaproalavalserglnpro
202530
thralaproalaprosercysleuprolysalaglyglnvalilepro
354045
thrleuleuargglualapropheserservalilealaprothrleu
505560
leucysglypheleupheleualatrpvalalaalagluvalproglu
65707580
gluserserargmetalaglyserglyalaargserglugluglyarg
859095
argglnhisalaphevalproglupropheaspglyalaasnvalval
100105110
proasnleutrpleuhisserphegluvalileasnaspleuasnhis
115120125
trpasphisilethrlysleuargpheleulysgluserleuarggly
130135140
glualaleuglyvaltyrasnargleuserproglnaspglnglyasp
145150155160
tyrglythrvallysglualaleuleulysalapheglyvalprogly
165170175
alaalaproserhisleuprolysgluilevalphealaasnsermet
180185190
glylysglytyrtyrleulysglylysileglylysvalprovalarg
195200205
pheleuvalaspserglyalaglnvalservalvalhisproasnleu
210215220
trpglugluvalthraspglyaspleuaspthrleuglnpropheglu
225230235240
asnvalvallysvalalaasnglyalaglumetlysileleuglyval
245250255
trpaspthralavalserleuglylysleulysleulysalaglnphe
260265270
leuvalalaasnalaseralagluglualaileileglythraspval
275280285
leuglnasphisasnalaileleuaspphegluhisargthrcysthr
290295300
leulysglylyslyspheargleuleuprovalglyglyserleuglu
305310315320
aspglupheaspleugluleuileglugluaspprosersergluglu
325330335
glyargglngluleuserhis
340
<210>3
<211>18
<212>dna
<213>人工合成
<400>3
aaccattgggaccatatc18
<210>4
<211>18
<212>dna
<213>人工合成
<400>4
actgagcctattgtagac18
<210>5
<211>21
<212>dna
<213>人工合成
<400>5
ggagcgagatccctccaaaat21
<210>6
<211>23
<212>dna
<213>人工合成
<400>6
ggctgttgtcatacttctcatgg23
<210>7
<211>19
<212>rna
<213>人工合成
<400>7
uucuccgaacgugucacgu19
<210>8
<211>19
<212>rna
<213>人工合成
<400>8
acgugacacguucggagaa19
<210>9
<211>21
<212>rna
<213>人工合成
<400>9
ucauugaugacuucaaagcug21
<210>10
<211>21
<212>rna
<213>人工合成
<400>10
gcuuugaagucaucaaugacc21
<210>11
<211>21
<212>rna
<213>人工合成
<400>11
acucuuucaggaaccuuagcu21
<210>12
<211>21
<212>rna
<213>人工合成
<400>12
cuaagguuccugaaagagucc21
<210>13
<211>21
<212>rna
<213>人工合成
<400>13
cuaagguuccugaaagagucc21
<210>14
<211>21
<212>rna
<213>人工合成
<400>14
ccugaaagggaagaaguuucg21
- 还没有人留言评论。精彩留言会获得点赞!