一种鉴定黄瓜品种真实性的方法及其专用SNP引物组合与流程
本发明属于生物技术领域,具体涉及一种鉴定黄瓜品种真实性的方法及其专用snp引物组合。
背景技术:
黄瓜是我国重要的蔬菜作物,栽培面积已达139.6万公顷,占全国蔬菜栽培面积的6.2%。通过国家和各省审定的品种逐年增多。依据《非主要农作物品种登记办法》,2017年4月农业部公布了第一批非主要农作物品种登记目录,黄瓜位列其中。目前,全国通过鉴定、认定、登记的品种达1000多个,但是市场上的黄瓜品种数量远远超过这一数字。由于黄瓜育种企业较小且分散,各省市之间品种审定相互不协调,黄瓜的品种管理未能有效跟进,存在很多假冒伪劣品种,其中很多为衍生、派生近似品种。加之良种生产基地管理混乱,盗育、套购品种现象猖獗,同名异种现象严重,种子质量事故时有发生。根据《非主要农作物品种登记指南》的要求,品种特性说明和品种dus测试报告中涉及的有关性状如有明确关联基因,可以直接提交dna检测结果。因此,建立一套基于dna指纹有效进行黄瓜品种资源真实性和纯度鉴定的标准显得尤为重要。
近年来,snp作为第三代分子标记,以其数量多、分布广、遗传稳定等优点受到广泛重视。随着高通量测序技术的发展和测序成本的不断降低,大规模黄瓜重测序成为可能。基于分析黄瓜变异组信息,可以挖掘更加稳定高效的snp位点。采用等位基因竞争性特异pcr法,开发其特异性引物,最终获得样本在snp位点的基因型。
目前,我国黄瓜品种鉴定dna分子检测主要依据ssr分子标记法,但是ssr引物的研发没有参考黄瓜基因组变异组信息,存在不真实变异情况;ssr引物筛选所用的品种数量较少,且不能代表我国当前市场的销售品种;另外受限于ssr检测方式极易导致不真实、假阳性、假阴性结果;不能适应自动化、高通量、大规模的要求。与ssr标记相比,snp具有多个方面的优势:变异清晰稳定容易检测,真实性准确性高;每个作物均有数以百万计snp可供选择;适宜于高通量、低成本、自动化快速检测,可减少人为误差;snp分型无需对照品种,以准确的碱基呈现结果。
技术实现要素:
本发明的目的是鉴定黄瓜品种。
本发明首先保护snp位点组合,可包括黄瓜基因组的32个snp位点;所述32个snp位点如下:cusnp01位点为1号染色体上的第1976800位核苷酸;cusnp02位点为1号染色体上的第8433330位核苷酸;cusnp03位点为1号染色体上的第13502481位核苷酸;cusnp04位点为1号染色体上的第17508118位核苷酸;cusnp05位点为1号染色体上的第25296763位核苷酸;cusnp06位点为2号染色体上的第1200640位核苷酸;cusnp07位点为2号染色体上的第2563528位核苷酸;cusnp08位点为2号染色体上的第5891488位核苷酸;cusnp09位点为2号染色体上的第6045153位核苷酸;cusnp10位点为2号染色体上的第11947823位核苷酸;cusnp11位点为3号染色体上的第40726位核苷酸;cusnp12位点为3号染色体上的第1001975位核苷酸;cusnp13位点为3号染色体上的第10713052位核苷酸;cusnp14位点为3号染色体上的第14161884位核苷酸;cusnp15位点为3号染色体上的第18678235位核苷酸;cusnp16位点为3号染色体上的第27913661位核苷酸;cusnp17位点为4号染色体上的第16975775位核苷酸;cusnp18位点为4号染色体上的第21068185位核苷酸;cusnp19位点为5号染色体上的第2133319位核苷酸;cusnp20位点为5号染色体上的第4054461位核苷酸;cusnp21位点为5号染色体上的第8103814位核苷酸;cusnp22位点为5号染色体上的第9582207位核苷酸;cusnp23位点为5号染色体上的第17245080位核苷酸;cusnp24位点为6号染色体上的第195377位核苷酸;cusnp25位点为6号染色体上的第2397556位核苷酸;cusnp26位点为6号染色体上的第18079846位核苷酸;cusnp27位点为6号染色体上的第20929474位核苷酸;cusnp28位点为6号染色体上的第24439780位核苷酸;cusnp29位点为7号染色体上的第2085399位核苷酸;cusnp30位点为7号染色体上的第4866176位核苷酸;cusnp31位点为7号染色体上的第6739101位核苷酸;cusnp32位点为7号染色体上的第15415870位核苷酸。
所述snp位点组合具体可由上述32个snp位点组成。
本发明还保护snp引物组合,可包括用于扩增所述cusnp01的引物组1、用于扩增所述cusnp02的引物组2、用于扩增所述cusnp03的引物组3、用于扩增所述cusnp04的引物组4、用于扩增所述cusnp05的引物组5、用于扩增所述cusnp06的引物组6、用于扩增所述cusnp07的引物组7、用于扩增所述cusnp08的引物组8、用于扩增所述cusnp09的引物组9、用于扩增所述cusnp010的引物组10、用于扩增所述cusnp11的引物组11、用于扩增所述cusnp12的引物组12、用于扩增所述cusnp13的引物组13、用于扩增所述cusnp14的引物组14、用于扩增所述cusnp15的引物组15、用于扩增所述cusnp16的引物组16、用于扩增所述cusnp17的引物组17、用于扩增所述cusnp18的引物组18、用于扩增所述cusnp19的引物组19、用于扩增所述cusnp20的引物组20、用于扩增所述cusnp21的引物组21、用于扩增所述cusnp22的引物组22、用于扩增所述cusnp23的引物组23、用于扩增所述cusnp24的引物组24、用于扩增所述cusnp25的引物组25、用于扩增所述cusnp26的引物组26、用于扩增所述cusnp27的引物组27、用于扩增所述cusnp28的引物组28、用于扩增所述cusnp29的引物组29、用于扩增所述cusnp30的引物组30、用于扩增所述cusnp31的引物组31和用于扩增所述cusnp32的引物组32。
所述snp引物组合中,所述引物组1可由序列1所示的正向引物01f1、序列2所示的正向引物01f2和序列3所示的反向引物01r组成。所述引物组2可由序列4所示的正向引物02f1、序列5所示的正向引物02f2和序列6所示的反向引物02r组成。所述引物组3可由序列7所示的正向引物03f1、序列8所示的正向引物03f2和序列9所示的反向引物03r组成。所述引物组4可由序列10所示的正向引物04f1、序列11所示的正向引物04f2和序列12所示的反向引物04r组成。所述引物组5可由序列13所示的正向引物05f1、序列14所示的正向引物05f2和序列15所示的反向引物05r组成。所述引物组6可由序列16所示的正向引物06f1、序列17所示的正向引物06f2和序列18所示的反向引物06r组成。所述引物组7可由序列19所示的正向引物07f1、序列20所示的正向引物07f2和序列21所示的反向引物07r组成。所述引物组8可由序列22所示的正向引物08f1、序列23所示的正向引物08f2和序列24所示的反向引物08r组成。所述引物组9可由序列25所示的正向引物09f1、序列26所示的正向引物09f2和序列27所示的反向引物09r组成。所述引物组10可由序列28所示的正向引物10f1、序列29所示的正向引物10f2和序列30所示的反向引物10r组成。所述引物组11可由序列31所示的正向引物11f1、序列32所示的正向引物11f2和序列33所示的反向引物11r组成。所述引物组12可由序列34所示的正向引物12f1、序列35所示的正向引物12f2和序列36所示的反向引物12r组成。所述引物组13可由序列37所示的正向引物13f1、序列38所示的正向引物13f2和序列39所示的反向引物13r组成。所述引物组14可由序列40所示的正向引物14f1、序列41所示的正向引物14f2和序列42所示的反向引物14r组成。所述引物组15可由序列43所示的正向引物15f1、序列44所示的正向引物15f2和序列45所示的反向引物15r组成。所述引物组16可由序列46所示的正向引物16f1、序列47所示的正向引物16f2和序列48所示的反向引物16r组成。所述引物组17可由序列49所示的正向引物17f1、序列50所示的正向引物17f2和序列51所示的反向引物17r组成。所述引物组18可由序列52所示的正向引物18f1、序列53所示的正向引物18f2和序列54所示的反向引物18r组成。所述引物组19可由序列55所示的正向引物19f1、序列56所示的正向引物19f2和序列57所示的反向引物19r组成。所述引物组20可由序列58所示的正向引物20f1、序列59所示的正向引物20f2和序列60所示的反向引物20r组成。所述引物组21可由序列61所示的正向引物21f1、序列62所示的正向引物21f2和序列63所示的反向引物21r组成。所述引物组22可由序列64所示的正向引物22f1、序列65所示的正向引物22f2和序列66所示的反向引物22r组成。所述引物组23可由序列67所示的正向引物23f1、序列68所示的正向引物23f2和序列69所示的反向引物23r组成。所述引物组24可由序列70所示的正向引物24f1、序列71所示的正向引物24f2和序列72所示的反向引物24r组成。所述引物组25可由序列73所示的正向引物25f1、序列74所示的正向引物25f2和序列75所示的反向引物25r组成。所述引物组26可由序列76所示的正向引物26f1、序列77所示的正向引物26f2和序列78所示的反向引物26r组成。所述引物组27可由序列79所示的正向引物27f1、序列80所示的正向引物27f2和序列81所示的反向引物27r组成。所述引物组28可由序列82所示的正向引物28f1、序列83所示的正向引物28f2和序列84所示的反向引物28r组成。所述引物组29可由序列85所示的正向引物29f1、序列86所示的正向引物29f2和序列87所示的反向引物29r组成。所述引物组30可由序列88所示的正向引物30f1、序列89所示的正向引物30f2和序列90所示的反向引物30r组成。所述引物组31可由序列91所示的正向引物31f1、序列92所示的正向引物31f2和序列93所示的反向引物31r组成。所述引物组32可由序列94所示的正向引物32f1、序列95所示的正向引物32f2和序列96所示的反向引物32r组成。
所述snp引物组合中,所述引物组1可由序列1自5’末端起第22至46位所示的正向引物01f1、序列2自5’末端起第22至46位所示的正向引物01f2和序列3所示的反向引物01r组成。所述引物组2可由序列4自5’末端起第22至46位所示的正向引物02f1、序列5自5’末端起第22至43位所示的正向引物02f2和序列6所示的反向引物02r组成。所述引物组3可由序列7自5’末端起第22至48位所示的正向引物03f1、序列8自5’末端起第22至50位所示的正向引物03f2和序列9所示的反向引物03r组成。所述引物组4可由序列10自5’末端起第22至52位所示的正向引物04f1、序列11自5’末端起第22至51位所示的正向引物04f2和序列12所示的反向引物04r组成。所述引物组5可由序列13自5’末端起第22至47位所示的正向引物05f1、序列14自5’末端起第22至47位所示的正向引物05f2和序列15所示的反向引物05r组成。所述引物组6可由序列16自5’末端起第22至51位所示的正向引物06f1、序列17自5’末端起第22至51位所示的正向引物06f2和序列18所示的反向引物06r组成。所述引物组7可由序列19自5’末端起第22至46位所示的正向引物07f1、序列20自5’末端起第22至48位所示的正向引物07f2和序列21所示的反向引物07r组成。所述引物组8可由序列22自5’末端起第22至48位所示的正向引物08f1、序列23自5’末端起第22至48位所示的正向引物08f2和序列24所示的反向引物08r组成。所述引物组9可由序列25自5’末端起第22至48位所示的正向引物09f1、序列26自5’末端起第22至47位所示的正向引物09f2和序列27所示的反向引物09r组成。所述引物组10可由序列28自5’末端起第22至51位所示的正向引物10f1、序列29自5’末端起第22至51位所示的正向引物10f2和序列30所示的反向引物10r组成。所述引物组11可由序列31自5’末端起第22至46位所示的正向引物11f1、序列32自5’末端起第22至45位所示的正向引物11f2和序列33所示的反向引物11r组成。所述引物组12可由序列34自5’末端起第22至46位所示的正向引物12f1、序列35自5’末端起第22至47位所示的正向引物12f2和序列36所示的反向引物12r组成。所述引物组13可由序列37自5’末端起第22至43位所示的正向引物13f1、序列38自5’末端起第22至44位所示的正向引物13f2和序列39所示的反向引物13r组成。所述引物组14可由序列40自5’末端起第22至49位所示的正向引物14f1、序列41自5’末端起第22至49位所示的正向引物14f2和序列42所示的反向引物14r组成。所述引物组15可由序列43自5’末端起第22至48位所示的正向引物15f1、序列44自5’末端起第22至49位所示的正向引物15f2和序列45所示的反向引物15r组成。所述引物组16可由序列46自5’末端起第22至46位所示的正向引物16f1、序列47自5’末端起第22至48位所示的正向引物16f2和序列48所示的反向引物16r组成。所述引物组17可由序列49自5’末端起第22至51位所示的正向引物17f1、序列50自5’末端起第22至52位所示的正向引物17f2和序列51所示的反向引物17r组成。所述引物组18可由序列52自5’末端起第22至46位所示的正向引物18f1、序列53自5’末端起第22至44位所示的正向引物18f2和序列54所示的反向引物18r组成。所述引物组19可由序列55自5’末端起第22至48位所示的正向引物19f1、序列56自5’末端起第22至49位所示的正向引物19f2和序列57所示的反向引物19r组成。所述引物组20可由序列58自5’末端起第22至48位所示的正向引物20f1、序列59自5’末端起第22至47位所示的正向引物20f2和序列60所示的反向引物20r组成。所述引物组21可由序列61自5’末端起第22至43位所示的正向引物21f1、序列62自5’末端起第22至42位所示的正向引物21f2和序列63所示的反向引物21r组成。所述引物组22可由序列64自5’末端起第22至47位所示的正向引物22f1、序列65自5’末端起第22至46位所示的正向引物22f2和序列66所示的反向引物22r组成。所述引物组23可由序列67自5’末端起第22至46位所示的正向引物23f1、序列68自5’末端起第22至46位所示的正向引物23f2和序列69所示的反向引物23r组成。所述引物组24可由序列70自5’末端起第22至43位所示的正向引物24f1、序列71自5’末端起第22至44位所示的正向引物24f2和序列72所示的反向引物24r组成。所述引物组25可由序列73自5’末端起第22至51位所示的正向引物25f1、序列74自5’末端起第22至51位所示的正向引物25f2和序列75所示的反向引物25r组成。所述引物组26可由序列76自5’末端起第22至43位所示的正向引物26f1、序列77自5’末端起第22至42位所示的正向引物26f2和序列78所示的反向引物26r组成。所述引物组27可由序列79自5’末端起第22至49位所示的正向引物27f1、序列80自5’末端起第22至48位所示的正向引物27f2和序列81所示的反向引物27r组成。所述引物组28可由序列82自5’末端起第22至54位所示的正向引物28f1、序列83自5’末端起第22至54位所示的正向引物28f2和序列84所示的反向引物28r组成。所述引物组29可由序列85自5’末端起第22至46位所示的正向引物29f1、序列86自5’末端起第22至45位所示的正向引物29f2和序列87所示的反向引物29r组成。所述引物组30可由序列88自5’末端起第22至52位所示的正向引物30f1、序列89自5’末端起第22至52位所示的正向引物30f2和序列90所示的反向引物30r组成。所述引物组31可由序列91自5’末端起第22至54位所示的正向引物31f1、序列92自5’末端起第22至53位所示的正向引物31f2和序列93所示的反向引物31r组成。所述引物组32可由序列94自5’末端起第22至56位所示的正向引物32f1、序列95自5’末端起第22至56位所示的正向引物32f2和序列96所示的反向引物32r组成。
上述任一所述引物组中,名称中含有“f1”的引物、名称中含有“f2”的引物和名称中含有“r”的引物的摩尔比具体可为2:2:5。
上述任一所述snp引物组合具体可由所述引物组1、所述引物组2、所述引物组3、所述引物组4、所述引物组5、所述引物组6、所述引物组7、所述引物组8、所述引物组9、所述引物组10、所述引物组11、所述引物组12、所述引物组13、所述引物组14、所述引物组15、所述引物组16、所述引物组17、所述引物组18、所述引物组19、所述引物组20、所述引物组21、所述引物组22、所述引物组23、所述引物组24、所述引物组25、所述引物组26、所述引物组27、所述引物组28、所述引物组29、所述引物组30、所述引物组31和所述引物组32组成。
上文中,序列表中序列1自5′末端起第1至21位所示的核苷酸序列为荧光标签序列(即fam荧光标签序列),荧光信号具体为蓝色。序列表中序列2自5′末端起第1至21位所示的核苷酸序列也为荧光标签序列(即hex荧光标签序列),荧光信号具体为红色。
含有上述任一所述snp引物组合的试剂盒也属于本发明的保护范围。
所述试剂盒的制备方法也属于本发明的保护范围。所述试剂盒的制备方法包括将上述任一所述引物组中的各条引物分别单独包装的步骤。
所述试剂盒的应用也属于本发明的保护范围。所述试剂盒的应用可为x3)或x4):x3)鉴定黄瓜品种;x4)鉴别黄瓜品种真伪性。
上述任一所述snp位点组合或上述任一所述snp引物组合的应用,可为x1)至x4)中的任一种:x1)制备用于鉴定黄瓜品种的试剂盒;x2)制备用于鉴别黄瓜品种真伪性的试剂盒;x3)鉴定黄瓜品种;x4)鉴别黄瓜品种真伪性。
本发明还保护一种鉴定待测黄瓜属于241个黄瓜品种中哪个品种的方法,可包括如下步骤:分别检测待测黄瓜和241个黄瓜品种基于所述32个snp位点的基因型,然后进行如下判断:如果待测黄瓜基于所述32个snp位点的基因型和241个黄瓜品种中某品种基于所述32个snp位点的基因型完全一致,则待测黄瓜与该黄瓜品种属于同一个品种;如果待测黄瓜基于所述32个snp位点的基因型和241个黄瓜品种中各个品种基于所述32个snp位点的基因型均不一致,则待测黄瓜与241个黄瓜品种的品种均不相同。
上述方法中,所述“检测待测黄瓜和241个黄瓜品种基于所述32个snp位点的基因型”的步骤可如下:
(1)分别以待测黄瓜的基因组dna和241个黄瓜品种的基因组dna为模板,分别采用上述任一所述snp引物组合中引物组的进行pcr扩增,得到pcr扩增产物;
(2)完成步骤(1)后,采用仪器检测pcr扩增产物的荧光信号,根据荧光信号的颜色获得待测黄瓜和241个黄瓜品种基于所述32个snp位点的基因型。
上述方法中,所述“检测检测待测黄瓜和241个黄瓜品种基于所述32个snp位点的基因型”的步骤可如下:
(1)分别以待测黄瓜的基因组dna和241个黄瓜品种的基因组dna为模板,分别采用上述任一所述snp引物组合中引物组的进行pcr扩增,得到pcr扩增产物;
(2)取步骤(1)得到的pcr扩增产物,测序;
(3)根据步骤(2)得到的测序结果,获得待测黄瓜和241个黄瓜品种基于所述32个snp位点的基因型。
本发明还保护一种鉴定待测黄瓜属于241个黄瓜品种中哪个品种的方法,可包括如下步骤:
(1)以待测黄瓜的基因组dna为模板,分别采用上述任一所述snp引物组合中引物组的进行pcr扩增,得到pcr扩增产物;以标准黄瓜品种群中的各个黄瓜品种的基因组dna为模板,分别采用上述任一所述snp引物组合中引物组的进行pcr扩增,得到pcr扩增产物;所述标准黄瓜品种群由241个黄瓜品种组成;
(2)将待测黄瓜得到的各个pcr扩增产物与每个标准黄瓜品种对应的pcr扩增产物进行聚类分析,聚类分析中待测黄瓜与哪个标准黄瓜品种为同一类,该待测黄瓜与该标准黄瓜品种即属于同一个品种。
上述任一所述的方法中,241个黄瓜品种可为翠绿、吉杂16号、中农20号、鲁黄瓜9号、中农29号、戴安娜、中研迷8f1、贝莎、中研23f1、金胚99-3f1、中研17f1、金胚99-2f1、金胚99f1、金胚99-1f1、金胚98f1、金胚98-1f1、中研19f1、银胚88f1、128一代杂种、中农6号、中农10号、北农佳秀、京研106、超越黄瓜f1、宁星全能春秋f1、亚美特、amata765、金山老黄瓜、京研迷你9号、京研迷你3号、京研迷你5号、京研迷你4号、绿精灵4号、华研丽人水果黄瓜、京研腌渍宝、白贵妃水果黄瓜、北京白玉三、春美、水果型101、义王水果型黄瓜、唐山翠宝f1、精品白叶三、唐春100、胡瓜、奥光、满冠、全能春秋f1、京研绿玲珑2号、京研旱宝5号、优亮王中王、京研夏美、京研优胜、京丰298、津优35号、京研秋美、京研春美、绿博十一号、绿博七号、北京403、绿秀、北京204、新研四号、津研四号、密刺王黄瓜、摘不败露地王、龙泉王-龙泉绿冠、xsbn4、xsbn6、xsbn8、xsbn11、xsbn16、xsbn1、xsbn2、xsbn3、xsbn5、xsbn7、xsbn9、xsbn10、xsbn12、xsbn13、xsbn14、xsbn15、xsbn17、xsbn18、xsbn19、津优401号、津优108号、vlaspik、宁运3号、中农19号、春玉、春秋王3号、p-2-5-1-1、春秋王2号、壮瓜、申绿72、cucumber1404、申绿64、i1an、京研翠玉迷你2号、13ac230、碧玉、绿翠、京研迷你1号、亮靓、东农804、宁佳3号、优加全雌09、何旱、玉女、津优12号、津春4号、沪杂6号、宝杂2号、选四、津优2号、冬丽、津优36号、mc2065、中农15号、日本北星、中农26号、津优38号、津优31号、博美11号、博美74号、津春三号、津绿3号、吉杂九号、田骄八号、硕丰八号、四季丰、翠龙、春王9801、绿衣天使、航遗1号、鲁黄瓜三号、津优10号、博美6913、乾德117、津春3号、津优315号、津优308、p02、万农三号、9930、白马王子、本地王黄瓜、德瑞特723、海诺一号、军科十八号、碧绿节成、欧洲一号、强果节结80、京a008、博美8号、京ak42、美丰瓜王、京ak18、德尔ld-1、德瑞特f16、德瑞特y8、鼎好油亮重茬王、黑油亮999、津正a207、绿宝珠8号、德瑞特727、德瑞特7876、德瑞特、德尔ld-3、油绿王子、京研118、汇赢15、花青大吊瓜、迷你3号、津旺203、翠美水果黄瓜、绿翠宝、硕九、田一、奇剑1号、绿优188、春喜f1、强旱节结、兴研四号、兴研二号、黑油亮507、奇剑2号、新津春四号、津优409、谷雨美利新星、83-ms702、京研迷你2号、戴多星、绿精灵2号、cu003、维吉尾亚、cu001、斯托克9006、cu002、绿精灵5号、绿精灵3号、无刺水果短黄瓜3966、88-ms701、玛莎黄瓜腌渍型、吉杂迷你、白精灵2号、君优66黄瓜、德瑞特黄瓜136-7、京白玉超白叶三、特选吉杂四号、京研绿玲珑、翠冠亮星、田骄7号华南型、瑞光二号、伯仲绿优黄瓜、精选叶三、谷雨佳丽、谷雨旱黄瓜、同52、德瑞特黄瓜l14-2、德瑞特黄瓜gz1601、德瑞特黄瓜l14-5、德瑞特黄瓜、韩国黄瓜2-06-97-164、盛丰908、德瑞特15-10密刺黄瓜、津冬科润99密刺黄瓜、中农106号、中农27号、津优4号、津优11号、津杂2号、中农16号、津优38、pi197088、h9、wi2757、truelemon、13642014.11和z2982013.11。
上述任一所述的方法中,“分别采用上述任一所述snp引物组合中引物组的进行pcr扩增”的反应程序具体可为:94℃预变性,15min;94℃变性20s,61℃-55℃(选用touchdown程序,每循环降低0.6℃),1min,扩增10个循环;94℃变性20s,55℃复性&延伸1min,继续扩增26个循环。若pcr扩增结束后荧光信号弱,影响数据分析,可以加循环(94℃变性20s,55℃复性及延伸1min,5个循环),直至结果满意为止。
本发明提供的snp引物组合可用于对黄瓜品种在种子或幼苗期进行早期鉴定,保证品种的真实性,切实保护生产者和育种家的权益,并为黄瓜种质资源和新品种保护提供技术支持。本发明提供的方法既可以对未知黄瓜品种进行鉴定,也可以对已知品种的真实性进行鉴定。本发明提供的方法具有高通量、准确、低成本、操作简单、节约人力、物力等有点,具有十分广阔的应用前景。
附图说明
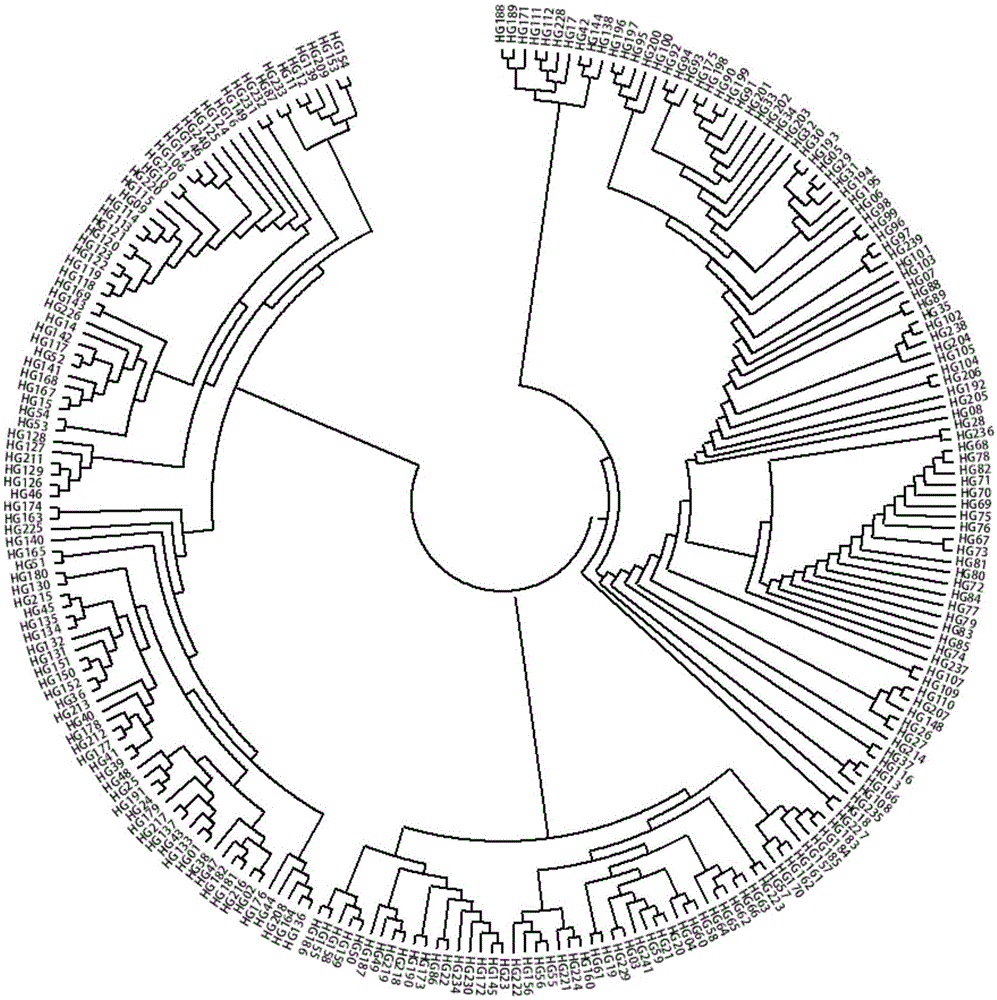
图1为建立在32个引物组上的241个供试黄瓜品种的聚类图。
图2为snp标记个数(即snp位点个数)与区分241个供试黄瓜品种的关系图。
图3为32个引物组在部分供试黄瓜品种中的snp分型效果。
具体实施方式
以下的实施例便于更好地理解本发明,但并不限定本发明。
下述实施例中的实验方法,如无特殊说明,均为常规方法。
下述实施例中所用的试验材料,如无特殊说明,均为自常规生化试剂商店购买得到的。
以下实施例中的定量试验,均设置三次重复实验,结果取平均值。
实施例1、用于鉴定黄瓜品种真实性的snp引物组合的获得
一、32个snp位点的发现
本发明基于49份黄瓜代表资源的重测序数据,获得32个snp位点。这49份黄瓜资源类型丰富,涵盖华北型(7份)、印度型(18份)、日本型(3份)、华南型(2份)、欧洲水果型(5份)、美洲加工型(4份)、西双版纳型(5份)以及中间型(5份),基本包括了黄瓜主要的生态类型与农艺性状,尽可能多地体现了种质代表性,具有较高的遗传多样性。
具体的,snp位点的筛选标准如下:在全基因组范围内选择位置均匀、多态性好、杂合度小、maf>0.3、pca聚类效果好、区分度高且两翼50bp序列保守(无indel,无ssr,无其它snp)的snp位点。
32个snp位点的基本信息详见表1中第1至4列。其中snp位点在染色体上的位置是基于黄瓜9930参考基因组序列比对确定的,所述黄瓜9930参考基因组序列的版本号为v2(下载地址为:http://cucurbitgenomics.org/organism/2)。
表1.32个snp位点的基本信息
二、用于鉴定黄瓜品种真实性的snp引物组合的获得
基于步骤一发现的32个snp位点,本发明的发明人开发了具有较高的多态性信息量(即pic值,见表1中第5列)的用于鉴定黄瓜品种真实性的snp引物组合。
snp引物组合由32个引物组组成,每个引物组的名称见表2中第2列。每个引物组由3条引物序列组成,用于扩增一个snp位点。32个引物组中各个引物的核苷酸序列如表2中第4列所示。
表2
注:单下划线为fam荧光标签序列,双下划线为hex荧光标签序列。
实施例2、实施例开发的snp引物组合的有效性检验
本实施例中的241个供试黄瓜品种的基本信息见表3。241个供试黄瓜品种均为常见的优良品种或部分国外引进品种。
表3.241个供试黄瓜品种的基本信息
1、供试黄瓜品种的基因组dna的获得
采用ctab法分别提取241个供试黄瓜品种的叶片(混取30粒种子的真叶)的基因组dna,得到供试黄瓜品种的基因组dna。
供试黄瓜品种的基因组dna的质量和浓度均须满足pcr要求,达标标准为:琼脂糖电泳显示dna条带单一,没有明显弥散;紫外分光光度计nanodrop2000(thermo)检测a260/a280比值在1.8左右,a260/a230比值大于1.8;供试黄瓜品种的基因组dna的浓度在10-30ng/μl。
2、分别以241个供试黄瓜品种的基因组dna为模板,分别采用32个引物组进行pcr扩增,得到pcr扩增产物。各个pcr反应体系中,名称中含有“f1”的引物、名称中含有“f2”的引物和名称中含有“r”的引物的浓度比为2:2:5。
反应程序为:94℃预变性,15min;94℃变性20s,61℃-55℃(选用touchdown程序,每循环降低0.6℃),1min,扩增10个循环;94℃变性20s,55℃复性&延伸1min,继续扩增26个循环。
3、完成步骤2后,待pcr扩增产物温度降至40℃以下时通过酶标仪的fam、hex光束扫描读取荧光值(fam荧光标签序列在激发光485nm,发射光520nm波长下观察读值,hex荧光标签序列在激发光528nm,发射光560nm波长下观察读值),根据荧光信号颜色判断241个供试黄瓜品种基于每个snp位点的基因型。具体的判断原则如下:如果某供试黄瓜品种基于某snp位点显示蓝色荧光信号,则该供试黄瓜品种基于该snp位点的基因型为“扩增该snp位点且名称中含有“f1”的引物的3’末端第1个碱基的互补碱基”纯合型;如果某供试黄瓜品种基于某snp位点显示红色荧光信号,则该供试黄瓜品种基于该snp位点的基因型为“扩增该snp位点且名称中含有“f2”的引物的3’末端第1个碱基的互补碱基”纯合型;如果某供试黄瓜品种基于某snp位点显示绿色荧光信号,则该供试黄瓜品种基于该snp位点的基因型为杂合型,一个碱基为“扩增该snp位点且名称中含有“f1”的引物的3’末端第1个碱基的互补碱基”,另一个碱基“扩增该snp位点且名称中含有“f2”的引物的3’末端第1个碱基的互补碱基”。
需要说明的是,若pcr扩增结束后荧光信号弱,影响数据分析,可以加循环(94℃变性20s,55℃复性及延伸1min,5个循环),直至结果满意为止。
部分结果见图3。结果表明,各个引物组在供试黄瓜品种中可以得到很好的分型效果。
4、聚类分析
根据241个供试黄瓜品种基于32个snp位点的基因型,利用minimarker和mega7软件对241个供试黄瓜品种进行聚类分析。
建立在32个引物组上的241个供试黄瓜品种的聚类图如图1所示。结果表明,32个引物组可以完全区分表3中的241个供试黄瓜品种。由此可见,实施例1开发的snp引物组合可以应用于黄瓜品种dna指纹数据库构建和品种真实性鉴定。
5、效率评价
品种真实性鉴定可以采用序贯分析方式减少工作量。本发明的发明人比较了snp标记个数(即引物组数)与区分241个供试黄瓜品种区分率的关系。
实验结果表明(图2),32个引物组(即32个snp标记个数)在241个供试黄瓜品种中的区分率达到100%。
<110>北京市农林科学院
<120>一种鉴定黄瓜品种真实性的方法及其专用snp引物组合
<160>96
<170>patentinversion3.5
<210>1
<211>46
<212>dna
<213>人工序列
<220>
<223>
<400>1
gaaggtgaccaagttcatgctttatcaaacaactcgacggcttcga46
<210>2
<211>46
<212>dna
<213>人工序列
<220>
<223>
<400>2
gaaggtcggagtcaacggattttatcaaacaactcgacggcttcgt46
<210>3
<211>30
<212>dna
<213>人工序列
<220>
<223>
<400>3
gattgcactttatgggaaatgtgggttttt30
<210>4
<211>46
<212>dna
<213>人工序列
<220>
<223>
<400>4
gaaggtgaccaagttcatgctttaaatggtcttctggctgccttca46
<210>5
<211>43
<212>dna
<213>人工序列
<220>
<223>
<400>5
gaaggtcggagtcaacggattaatggtcttctggctgccttcg43
<210>6
<211>27
<212>dna
<213>人工序列
<220>
<223>
<400>6
gctacaagttcgaatggaggtgcaaaa27
<210>7
<211>48
<212>dna
<213>人工序列
<220>
<223>
<400>7
gaaggtgaccaagttcatgcttttcattttgttcccttttggcttcag48
<210>8
<211>50
<212>dna
<213>人工序列
<220>
<223>
<400>8
gaaggtcggagtcaacggattcatttcattttgttcccttttggcttcaa50
<210>9
<211>32
<212>dna
<213>人工序列
<220>
<223>
<400>9
gaaaaatagtctaccacaattttgaaaccgaa32
<210>10
<211>52
<212>dna
<213>人工序列
<220>
<223>
<400>10
gaaggtgaccaagttcatgctgaatgaaagtttaatcaaggatgcaactatt52
<210>11
<211>51
<212>dna
<213>人工序列
<220>
<223>
<400>11
gaaggtcggagtcaacggattaatgaaagtttaatcaaggatgcaactatg51
<210>12
<211>31
<212>dna
<213>人工序列
<220>
<223>
<400>12
agattttgtgccaatagagcaaagtcatata31
<210>13
<211>47
<212>dna
<213>人工序列
<220>
<223>
<400>13
gaaggtgaccaagttcatgctgttgatgaccaatgaccatttcgtag47
<210>14
<211>47
<212>dna
<213>人工序列
<220>
<223>
<400>14
gaaggtcggagtcaacggattgttgatgaccaatgaccatttcgtac47
<210>15
<211>31
<212>dna
<213>人工序列
<220>
<223>
<400>15
cgacattaaccccaatcttggtaaataatta31
<210>16
<211>51
<212>dna
<213>人工序列
<220>
<223>
<400>16
gaaggtgaccaagttcatgctgttcaatgcttggatctttactttcattaa51
<210>17
<211>51
<212>dna
<213>人工序列
<220>
<223>
<400>17
gaaggtcggagtcaacggattgttcaatgcttggatctttactttcattat51
<210>18
<211>28
<212>dna
<213>人工序列
<220>
<223>
<400>18
gtttcaagcctattcgcatcatcttctt28
<210>19
<211>46
<212>dna
<213>人工序列
<220>
<223>
<400>19
gaaggtgaccaagttcatgctctcaagcgttttgctaaatgcatgc46
<210>20
<211>48
<212>dna
<213>人工序列
<220>
<223>
<400>20
gaaggtcggagtcaacggattaactcaagcgttttgctaaatgcatgt48
<210>21
<211>30
<212>dna
<213>人工序列
<220>
<223>
<400>21
gttcaaaaaaggaagactcagtcaagtcaa30
<210>22
<211>48
<212>dna
<213>人工序列
<220>
<223>
<400>22
gaaggtgaccaagttcatgctacctgtgttgtttgcatctagcataaa48
<210>23
<211>48
<212>dna
<213>人工序列
<220>
<223>
<400>23
gaaggtcggagtcaacggattacctgtgttgtttgcatctagcataat48
<210>24
<211>27
<212>dna
<213>人工序列
<220>
<223>
<400>24
cgccatcttttccgtaagcgatgttat27
<210>25
<211>48
<212>dna
<213>人工序列
<220>
<223>
<400>25
gaaggtgaccaagttcatgctcttgtctgttggacttccacaattttt48
<210>26
<211>47
<212>dna
<213>人工序列
<220>
<223>
<400>26
gaaggtcggagtcaacggattttgtctgttggacttccacaattttg47
<210>27
<211>25
<212>dna
<213>人工序列
<220>
<223>
<400>27
aaaccaacggtcactctcaggcaaa25
<210>28
<211>51
<212>dna
<213>人工序列
<220>
<223>
<400>28
gaaggtgaccaagttcatgctaaagtgattgttaatatcgattgcttgttg51
<210>29
<211>51
<212>dna
<213>人工序列
<220>
<223>
<400>29
gaaggtcggagtcaacggattaaagtgattgttaatatcgattgcttgttc51
<210>30
<211>28
<212>dna
<213>人工序列
<220>
<223>
<400>30
atccagaaaagcatcccacaattacgta28
<210>31
<211>46
<212>dna
<213>人工序列
<220>
<223>
<400>31
gaaggtgaccaagttcatgctgtctctgcttgcataagactggtta46
<210>32
<211>45
<212>dna
<213>人工序列
<220>
<223>
<400>32
gaaggtcggagtcaacggatttctctgcttgcataagactggttg45
<210>33
<211>31
<212>dna
<213>人工序列
<400>33
tacaccaggtttcagtaataacattaaccat31
<210>34
<211>46
<212>dna
<213>人工序列
<400>34
gaaggtgaccaagttcatgctactaggtttggaactttggagtgtc46
<210>35
<211>47
<212>dna
<213>人工序列
<400>35
gaaggtcggagtcaacggattgactaggtttggaactttggagtgtt47
<210>36
<211>34
<212>dna
<213>人工序列
<400>36
caacgtataattttagcatgttagaacttagaaa34
<210>37
<211>43
<212>dna
<213>人工序列
<400>37
gaaggtgaccaagttcatgctgtcatcaagagtctcagctgac43
<210>38
<211>44
<212>dna
<213>人工序列
<400>38
gaaggtcggagtcaacggattcgtcatcaagagtctcagctgaa44
<210>39
<211>22
<212>dna
<213>人工序列
<400>39
tgtcgttgctggccacccagaa22
<210>40
<211>49
<212>dna
<213>人工序列
<400>40
gaaggtgaccaagttcatgctgaatcgaaaacttctgtgtgcatcaaat49
<210>41
<211>49
<212>dna
<213>人工序列
<400>41
gaaggtcggagtcaacggattgaatcgaaaacttctgtgtgcatcaaaa49
<210>42
<211>31
<212>dna
<213>人工序列
<400>42
tgcaggtaacttagttcaattgtagagaatt31
<210>43
<211>48
<212>dna
<213>人工序列
<400>43
gaaggtgaccaagttcatgcttttgcctaagtgaacacatacttagtg48
<210>44
<211>49
<212>dna
<213>人工序列
<400>44
gaaggtcggagtcaacggattgtttgcctaagtgaacacatacttagtt49
<210>45
<211>28
<212>dna
<213>人工序列
<400>45
gtgcacataaaaaggtatggcaagtcta28
<210>46
<211>46
<212>dna
<213>人工序列
<400>46
gaaggtgaccaagttcatgcttgctgtctgaagagaaaatgcatgc46
<210>47
<211>48
<212>dna
<213>人工序列
<400>47
gaaggtcggagtcaacggatttttgctgtctgaagagaaaatgcatgt48
<210>48
<211>34
<212>dna
<213>人工序列
<400>48
ctatttcatttgtatggaaagacagatatttcat34
<210>49
<211>51
<212>dna
<213>人工序列
<400>49
gaaggtgaccaagttcatgctataaatataaggctagactagtagcaaaag51
<210>50
<211>52
<212>dna
<213>人工序列
<400>50
gaaggtcggagtcaacggattgataaatataaggctagactagtagcaaaaa52
<210>51
<211>31
<212>dna
<213>人工序列
<400>51
gaagaagtctatgttttccctctatctaaaa31
<210>52
<211>46
<212>dna
<213>人工序列
<400>52
gaaggtgaccaagttcatgctttcggtcagtgctttaatccccttt46
<210>53
<211>44
<212>dna
<213>人工序列
<400>53
gaaggtcggagtcaacggattcggtcagtgctttaatccccttc44
<210>54
<211>25
<212>dna
<213>人工序列
<400>54
cagcagattctgcaatggcaacgtt25
<210>55
<211>48
<212>dna
<213>人工序列
<400>55
gaaggtgaccaagttcatgctagactagcaaagagtaatatggttgac48
<210>56
<211>49
<212>dna
<213>人工序列
<400>56
gaaggtcggagtcaacggattgagactagcaaagagtaatatggttgat49
<210>57
<211>31
<212>dna
<213>人工序列
<400>57
tacatccgttatggaattttgagttacacta31
<210>58
<211>48
<212>dna
<213>人工序列
<400>58
gaaggtgaccaagttcatgctcggcataatatgaatcaacctggtaaa48
<210>59
<211>47
<212>dna
<213>人工序列
<400>59
gaaggtcggagtcaacggattggcataatatgaatcaacctggtaag47
<210>60
<211>24
<212>dna
<213>人工序列
<400>60
gaccaccaaggcctatcactccaa24
<210>61
<211>43
<212>dna
<213>人工序列
<400>61
gaaggtgaccaagttcatgctgggatgtaatagtcggagccct43
<210>62
<211>42
<212>dna
<213>人工序列
<400>62
gaaggtcggagtcaacggattggatgtaatagtcggagcccc42
<210>63
<211>29
<212>dna
<213>人工序列
<400>63
gaatatacaaaagccaagcccaccaaaaa29
<210>64
<211>47
<212>dna
<213>人工序列
<400>64
gaaggtgaccaagttcatgctcaacgagttgttaatgctttggagct47
<210>65
<211>46
<212>dna
<213>人工序列
<400>65
gaaggtcggagtcaacggattaacgagttgttaatgctttggagcc46
<210>66
<211>25
<212>dna
<213>人工序列
<400>66
actgacactccaggagcaatctcaa25
<210>67
<211>46
<212>dna
<213>人工序列
<400>67
gaaggtgaccaagttcatgctattcgagattactccaagttgtccc46
<210>68
<211>46
<212>dna
<213>人工序列
<400>68
gaaggtcggagtcaacggattattcgagattactccaagttgtccg46
<210>69
<211>31
<212>dna
<213>人工序列
<400>69
aagtcccaagccataaaaaaagaaaggaaat31
<210>70
<211>43
<212>dna
<213>人工序列
<400>70
gaaggtgaccaagttcatgctctgaggtcgttatggtgaccac43
<210>71
<211>44
<212>dna
<213>人工序列
<400>71
gaaggtcggagtcaacggattcctgaggtcgttatggtgaccaa44
<210>72
<211>31
<212>dna
<213>人工序列
<400>72
aaataaagaaacaaaagaatgggcgtagcaa31
<210>73
<211>51
<212>dna
<213>人工序列
<400>73
gaaggtgaccaagttcatgctaacaaacaaatggagctttaagtaaagtac51
<210>74
<211>52
<212>dna
<213>人工序列
<400>74
gaaggtcggagtcaacggattcaacaaacaaatggagctttaagtaaagtaa52
<210>75
<211>32
<212>dna
<213>人工序列
<400>75
gccttttgactcgtatgttttactttacttta32
<210>76
<211>43
<212>dna
<213>人工序列
<400>76
gaaggtgaccaagttcatgcttgaacagctgagggccattgca43
<210>77
<211>42
<212>dna
<213>人工序列
<400>77
gaaggtcggagtcaacggattgaacagctgagggccattgcg42
<210>78
<211>27
<212>dna
<213>人工序列
<400>78
cacaacgtcttcctcttggctagttat27
<210>79
<211>49
<212>dna
<213>人工序列
<400>79
gaaggtgaccaagttcatgctcatatacacacaatacatagctgttgga49
<210>80
<211>48
<212>dna
<213>人工序列
<400>80
gaaggtcggagtcaacggattatatacacacaatacatagctgttggg48
<210>81
<211>31
<212>dna
<213>人工序列
<400>81
ctctgccagaaacaaacaataacttttgtaa31
<210>82
<211>54
<212>dna
<213>人工序列
<400>82
gaaggtgaccaagttcatgctatgaggaagaaatgagcatatatttatttgttt54
<210>83
<211>54
<212>dna
<213>人工序列
<400>83
gaaggtcggagtcaacggattatgaggaagaaatgagcatatatttatttgtta54
<210>84
<211>30
<212>dna
<213>人工序列
<400>84
gattaggaggggtagaagtgattaatgaat30
<210>85
<211>46
<212>dna
<213>人工序列
<400>85
gaaggtgaccaagttcatgctgatgtgtgcagatgacgatgataga46
<210>86
<211>45
<212>dna
<213>人工序列
<400>86
gaaggtcggagtcaacggattatgtgtgcagatgacgatgatagg45
<210>87
<211>34
<212>dna
<213>人工序列
<400>87
acttagtttggaattattgataatttcctgcaat34
<210>88
<211>52
<212>dna
<213>人工序列
<400>88
gaaggtgaccaagttcatgctgtttattgggtttatttaaactaagacatcg52
<210>89
<211>52
<212>dna
<213>人工序列
<400>89
gaaggtcggagtcaacggattgtttattgggtttatttaaactaagacatcc52
<210>90
<211>31
<212>dna
<213>人工序列
<400>90
agagaagaagccaaatatgaatggaattctt31
<210>91
<211>54
<212>dna
<213>人工序列
<400>91
gaaggtgaccaagttcatgcttctttttctcttttcttttttctgacttcttta54
<210>92
<211>53
<212>dna
<213>人工序列
<400>92
gaaggtcggagtcaacggattctttttctcttttcttttttctgacttctttg53
<210>93
<211>30
<212>dna
<213>人工序列
<400>93
ctgaaacatgcgtacaactaaactaagcta30
<210>94
<211>56
<212>dna
<213>人工序列
<400>94
gaaggtgaccaagttcatgcttttgtttccaatgattagaagtatatttatatatc56
<210>95
<211>56
<212>dna
<213>人工序列
<400>95
gaaggtcggagtcaacggatttttgtttccaatgattagaagtatatttatatatg56
<210>96
<211>36
<212>dna
<213>人工序列
<400>96
ctttgttccaattttctaaattttgtttttccatta36
- 还没有人留言评论。精彩留言会获得点赞!