大肠杆菌全局调控因子的突变基因soxR及应用的制作方法
本发明属于大肠杆菌遗传工程及抗生素耐药性的研究领域。具体而言,涉及一种大肠杆菌全局调控因子的突变基因soxr(g121p)及该基因编码的氨基酸序列及应用。
背景技术:
抗生素是能选择性地抑制某些生物生命活动的微生物次级代谢产物,及其半合成或全合成的衍生物;广泛应用于医疗行业以及畜牧、渔业等农业部门,为人类社会发展做出了重要贡献。然而,抗生素的过度使用以及环境中存在的抗生素所造成的选择压力加剧了细菌耐药性的形成以及多重药物耐药性菌株的出现,给医疗行业带来了前所未有的挑战。另外,日益严峻的抗生素环境污染问题也越来越多的引起了人们的重视。
微生物能够感应外界环境的变化,通过调控因子调控相关基因的表达来调节细胞的一系列生理活动。soxr属于merr蛋白家族(汞抗性操纵子调节因子家族),通过调控soxs的表达来调节下游一系列基因的表达。已有研究表明soxr的突变对于微生物的抗生素耐药性有重要影响,koutsolioutsou等研究发现soxr蛋白第128位丝氨酸残基删除、第20位精氨酸突变为组氨酸菌株表现出抗生素耐药表型(koutsolioutsou,pena-llopis,&demple.2005)。tugce等的研究也发现,在高浓度的选择压力下soxr基因编码蛋白发生的氨基酸突变a146v及g143d会导致大肠杆菌对氯霉素产生耐药性(oz,guvenek,yildiz,karaboga,tamer,&mumcuyan,etal.2014)。鉴于全局调控因子在微生物生理代谢活动中所产生的多方面影响及其在抗生素耐药性方面的重要作用,本发明公开了采用全局调控因子工程所鉴定的细菌抗生素耐药性相关基因以及构建的抗生素高耐药性菌株,为细菌耐药性机制研究提供重要的应用支持。
目前,研究大都以肠球菌、铜绿假单胞菌和金黄色葡萄球菌等多药耐药性菌株作为模式生物来研究抗生素多药耐药性,尚未有大肠杆菌全局调控因子的突变基因soxr(g121p)在抗生素多药耐药性研究领域的报道。
技术实现要素:
本发明的目的是克服现有技术的不足,提供一种大肠杆菌全局调控因子的突变基因soxr。
本发明的第二个目的是提供一种大肠杆菌全局调控因子的突变基因soxr所编码的蛋白质。
本发明的第三个目的是提供包含有大肠杆菌全局调控因子的突变基因soxr的工程菌。
本发明的第四个目的是提供所述工程菌在细菌耐药性遗传机制研究的应用。
本发明的技术方案概述如下:
大肠杆菌全局调控因子的突变基因soxr(g121p),所述突变基因的核苷酸序列是seqidno.1所示;
大肠杆菌全局调控因子的突变基因soxr(g121p)所编码的蛋白质,所述蛋白质的氨基酸序列是seqidno.2所示;
包含有所述大肠杆菌全局调控因子的突变基因soxr(g121p)的工程菌。
上述工程菌在细菌耐受性遗传机制研究的应用。
本发明所构建的包含大肠杆菌全局调控因子的突变基因soxr(g121p)的工程菌生物安全,遗传背景清晰,包含的大肠杆菌全局调控因子的突变基因soxr(g121p)在细菌耐药性遗传机制研究方面具有重要应用。
附图说明
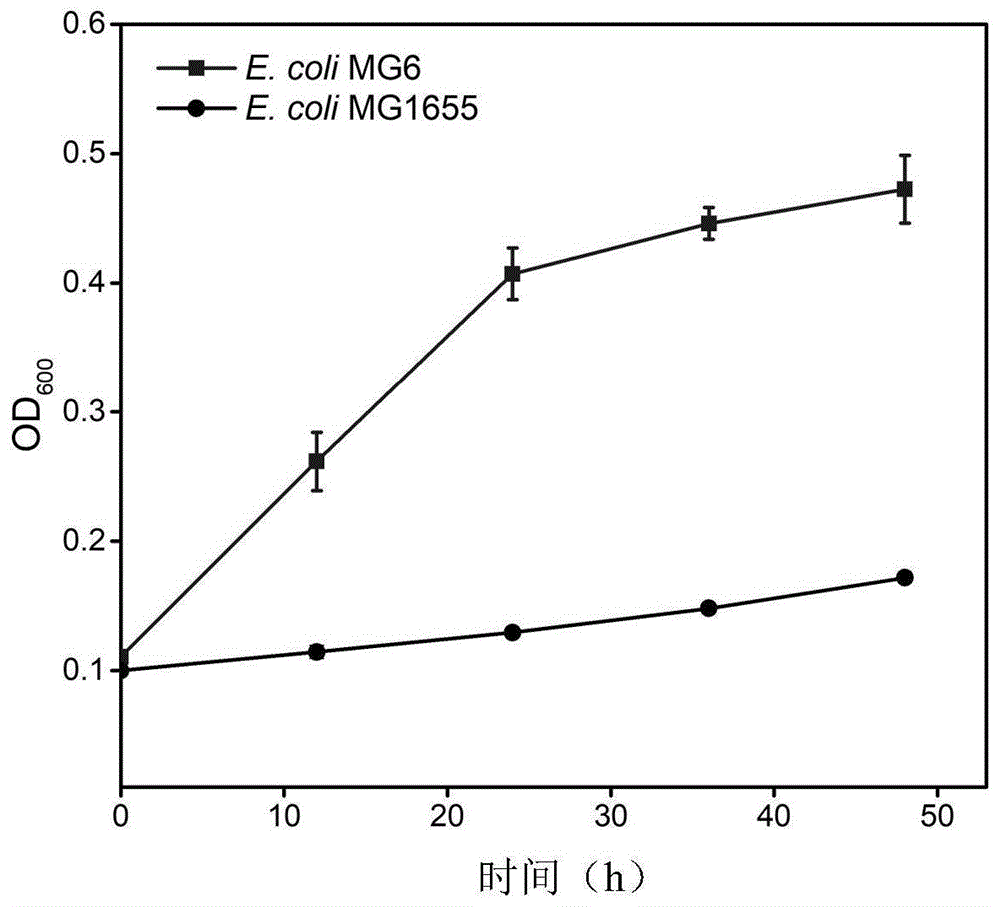
图1为包含大肠杆菌全局调控因子的突变基因soxr的工程菌(简称工程菌株e.colimg6)对多西环素耐药性验证。
图2为工程菌株e.colimg6对甲砜霉素耐药性验证。
图3为工程菌株e.colimg6对阿奇霉素耐药性验证。
具体实施方式
下面结合实施例对本发明做进一步说明,下述实施例是为了使本领域的技术人员能够更好地理解本发明,但对本发明不作任何限制。
本发明所用到的原始菌株e.colimg1655来源为atcc保藏的典型菌株(
本发明所用到的甲砜霉素从上海源叶生物科技有限公司(http://www.shyuanye.com)购买,所用限制性内切酶、dna连接酶等分子生物学试剂从thermo公司购买(http://www.thermoscientificbio.com/fermentas)。所用其他生化试剂从生工生物工程(上海)股份有限公司购买(http://www.sangon.com/)。
实施例1
全局调控因子突变文库的设计和高通量合成
基于crispr可追踪基因工程(crisprenabledtrackablegenomeengineering,create)系统,包括三个质粒:阿拉伯糖诱导cas9蛋白表达的质粒x2-cas9、热激诱导表达λ-red重组系统的温度敏感型质粒psim5、grna和供体dna表达的全局调控因子突变文库质粒(garst,bassalo,pines,lynch,halweg-edwards,&liu,etal)。全局调控因子突变文库质粒盒子序列总共200bp,包括17bp子文库引物序列、100bp同源臂、4bp间隔序列、35bpgrna启动子序列、grna序列与cas9蛋白结合的序列。通过将混合的文库质粒转化进入大肠杆菌,实现一次转化下的多个细胞的突变。
本发明中所用到的全局调控因子突变文库包含大肠杆菌23个全局调控因子涉及的34340个突变,是根据调控因子数据库rugulondb和ecocyc的分析选择了23个全局调控因子,然后从数据库rugulondb,ncbi和ecocyc中提取最可能对靶位点(即dna结合位点,活性位点,二聚化位点和预测位点)的调节因子具有功能性影响的位点作为调控因子饱和突变位点,利用全局调控因子突变文库自动化设计工具bioverse(http://www.thebioverse.org/)设计突变文库,设计的文库序列经由美国安捷伦公司(agilenttechnologies)合成。通过公司合成的全局调控因子突变文库片段,首先经过20轮pcr延伸反应,然后延伸产物利用6%的聚丙烯酰胺凝胶电泳(page)胶分离和纯化。纯化后的产物作为模板,利用设计的引物进行子文库的扩增,扩增产物进一步经过纯化,最后利用gibson组装方法连接到全局调控因子突变文库载体质粒,获得5个质粒形式的不同子文库,这五个文库分别命名为g1,g2,g3,g4和g5,每个文库的信息详见表1。
表1文库的分类
实施例2
基于全局调控因子突变文库构建大肠杆菌突变体库
(1)采用电转法将x2-cas9质粒导入到菌株e.colimg1655操作流程如下:
将-80℃冻存的野生型e.colimg1655在lb固体培养基上培养至长出单菌落,挑取单菌落接种到装有5mllb液体培养基的试管中,在37℃、220rpm培养12h。以1%的接种量接种到装有100mllb液体培养基的500ml三角瓶中,在37℃、220rpm培养大约2h,至od600为0.35。将三角瓶取出置冰上冷却10min,然后将菌液转移至预冷的100ml离心管中,4℃离心收集菌体,再用预冷的无菌水和10%甘油各洗两次,然后用1ml预冷的10%甘油重悬菌体,最后分装到预冷的1.5ml无菌ep管中,每管100μl,得到制备好的mg1655感受态。向mg1655感受态中加入3μlx2-cas9质粒,混匀后加入电击杯中,使用micropulser(bio-rad公司)电穿孔仪在1.8kv条件下电击4.5ms,电击后迅速加入800μllb液体培养基,然后在37℃、220rpm复苏培养3h。复苏培养后,取100μl菌液涂布在含有终浓度为40μg/ml卡那霉素的lb平板上,37℃培养至长出单菌落,用菌落pcr验证,得到x2-cas9质粒正确导入的菌株e.colimg1。
(2)采用电转法将质粒psim5导入到菌株e.colimg1操作流程如下:
将菌株e.colimg1在含有终浓度为40μg/ml卡那霉素lb固体培养基上培养至长出单菌落,挑取单菌落接种到装有终浓度为0.4%阿拉伯糖、40μg/ml卡那霉素的5mllb液体培养基的试管中,在37℃、220rpm培养12h。之后以1%的接种量接种到装有终浓度为0.4%阿拉伯糖、40μg/ml卡那霉素的100mllb液体培养基的500ml三角瓶中,在37℃条件下培养大约2h,直至od600为0.35。将三角瓶取出置冰上冷却10min,然后将菌液转移至预冷的100ml离心管中,4℃离心收集菌体,再用预冷的无菌水和10%甘油各洗两次,然后用1ml预冷的10%甘油重悬菌体,最后分装到预冷的1.5ml无菌ep管中,每管100μl,得到制备好的e.colimg1感受态。向e.colimg1感受态中加入3μl预冷的psim5质粒,混匀后加入电击杯中,使用电穿孔仪在1.8kv条件下电击4.5ms,电击后迅速加入800μllb液体培养基,然后在30℃、220rpm复苏培养3h。复苏培养后,取100μl菌液涂布在含有终浓度为40μg/ml卡那霉素和30μg/ml氯霉素的lb平板上,30℃培养至长出单菌落,用菌落pcr验证,得到psim5质粒正确导入e.colimg1的菌株e.colimg2。
(3)采用电转法将全局调控因子突变文库质粒(g1、g2、g3、g4、g5)、空白对照ntg质粒、估计重组效率的galk-off质粒(garst,bassalo,pines,lynch,halweg-edwards,&liu,etal)分别导入到菌株e.colimg2获得不同转化子操作流程如下:
将e.colimg2在含有终浓度为40μg/ml卡那霉素和30μg/ml氯霉素的lb固体培养基上培养至长出单菌落,挑取单菌落接种到装有终浓度为0.4%阿拉伯糖、40μg/ml卡那霉素、30μg/ml氯霉素的5mllb液体培养基的试管中,在30℃、220rpm培养12h。之后以1%的接种量接种到装有终浓度为0.4%阿拉伯糖、40μg/ml卡那霉素、30μg/ml氯霉素的100mllb液体培养基的500ml三角瓶中,在30℃、220rpm培养大约2h,直至od600为0.35。然后在42℃水浴条件下诱导15min,之后将三角瓶取出置冰上冷却10min,然后将菌液转移至预冷的100ml离心管中,4℃离心收集菌体,再用预冷的无菌水和10%甘油各洗两次,然后用1ml预冷的10%甘油重悬菌体,最后分装到预冷的1.5ml无菌ep管中,每管300μl,得到制备好的e.colimg2感受态。向e.colimg2感受态中分别加入3μl透析后的全局调控因子突变文库质粒(g1、g2、g3、g4、g5)、空白对照ntg质粒、估计重组效率的galk-off质粒,混匀后加入电击杯中,使用电穿孔仪在2.5kv条件下电击约6.0ms,电击后迅速加入1mllb液体培养基,将菌液转移至含4mllb液体培养基的试管中,在30℃、220rpm复苏培养3h。复苏培养后,含全局调控因子突变文库质粒或ntg质粒的菌液取10μl涂布到含有终浓度为100μg/ml奇霉素的lb培养基上,含galk-off质粒的菌液取10μl涂布到含有终浓度为1%半乳糖和100μg/ml奇霉素的麦康凯琼脂培养基上,30℃培养至长出单菌落。涂板后,将试管中剩余菌液取1ml转移至装有50mllb液体培养基的250ml三角瓶中扩大培养,培养12h后保菌,得到含ntg质粒的对照菌株记为e.colimg3(对照菌株),含galk-off质粒的菌株记为e.colimg4(估计重组效率),及全局调控因子突变文库重组菌群(突变体库)。
lb液体培养基配方为:10g/l蛋白胨,5g/l酵母提取物,10g/lnacl,调节ph至7.0。0.1mpa压力下灭菌20min。
lb固体培养基配方为:在lb液体培养基中加入琼脂粉(终浓度15g/l),0.1mpa压力下灭菌20min。
含1%半乳糖的康凯琼脂培养基配方为:蛋白胨17g/l,胨3g/l,胆盐1.5g/l,氯化钠5g/l,琼脂糖13.5g/l,中性红0.03g/l,结晶紫0.001g/l,1%半乳糖,0.1mpa压力下灭菌30min。
本发明中的菌株代号如e.colimg1、e.colimg3等是为了方便描述,但不应理解为对本发明的限定。
实施例3
多西环素耐药性筛选及适应度分析
(1)在多西环素胁迫条件下的耐药性筛选
将e.colimg3、e.colimg4和全局调控因子突变文库重组菌群在装有终浓度为100μg/ml奇霉素和5mllb液体培养基的试管中培养12h,然后将整管菌液接种到装有终浓度为100μg/ml奇霉素和50mllb液体培养基的250ml三角瓶中,在37℃、220rpm培养至od600为4.5。以初始接种量od600为0.1接入到装有终浓度为10μg/ml多西环素的lb液体培养基中,在37℃、220培养至优势菌群表现出明显生长优势,逐渐提高多西环素的浓度到15μg/ml再到25μg/ml,经过3次从低浓度到高浓度多西环素的转接及富集,可以筛选出正向突变富集文库菌群g1和g5。
(2)全局调控因子突变的适应度分析
对筛选得到的正向突变文库菌群g1和g5提取混合质粒,之后进行illumina测序(详细的测序过程可参考公开发表文献3)得到富集数据,然后进行不同突变的适应度分数计算,计算公式如下:
实施例4
耐受多西环素突变菌株的重构
采用crispr基因组编辑系统将突变基因soxr(g121p)引入到e.colimg1655。具体过程是通过goldengate组装构建出grna质粒、通过pcr及pcr融合构建出外源双链dna片段,然后借助crispr基因组编辑系统,将grna质粒和外源片段共转化进入感受态中,引入突变。
(1)grna质粒pgrs的构建的操作流程如下:
grna质粒的构建是通过goldengate反应完成的。利用引物pgrs-f/pgrs-l(seqidno.3/seqidno.4),以质粒pgrb为模板(li,lin,huang,&yan,etal),进行pcr扩增获得片段长度为2142bp的质粒骨架。将pcr得到的片段进行纯化,之后用核酸蛋白定量仪(bio-rad公司)定量dna片段的浓度,按照goldengate克隆反应体系的要求配制反应体系,置于pcr仪中进行扩增,再进行转化和培养,挑取单克隆进行菌落pcr验证。用引物pgrs-test-f/pgrs-test-l(seqidno.5/seqidno.6)对构建完成的质粒进行pcr验证,扩增的pcr片段长度为365bp,将pcr片段送金维智公司测序,得到正确的质粒,该质粒命名为pgrs。操作流程中使用的引物序列详见表2。
(2)外源供体dna片段构建的操作流程如下:
利用121p-f1/121p-l1(seqidno.7/seqidno.8)和121p-f2/121p-l2(seqidno.9/seqidno.10)两对引物,以e.colimg1655基因组为模板,使用fastpfu高保真dna聚合酶扩增分别得到大小为400bp和687bp的上下游同源臂121p*-f和121p*-b。121p*-f和121p*-b的pcr片段经切胶回收后,利用引物121p-f1/121p-l2(seqidno.7/seqidno.10),以121p*-f和121p*-b为模板,融合pcr得到突变soxr(g121p)所需外源片段,长度1070bp。
表2菌株构建所用引物序列
(3)crispr基因组编辑系统实现soxr(g121p)突变操作流程如下:
第一步:将ptkredcas9质粒(lin,xu,li,&wang,etal)导入到野生型e.colimg1655中,该菌株记为e.colimg5,此处需说明ptkredcas9质粒的抗性是奇霉素,电转后涂布奇霉素抗性的lb平板,其余步骤参考实施案例1(2)。
第二步:将质粒pgrs和外源供体dna片段共转化进e.colimg5。将e.colimg5在含有终浓度为100μg/ml奇霉素的lb固体培养基上培养至长出单菌落,挑取单菌落接种到装有终浓度为100μg/ml奇霉素和5mllb液体培养基的试管中,在30℃、220rpm培养12h。之后以1%的接种量接种到装有2mmiptg、100μg/ml奇霉素及100mllb液体培养基的500ml三角瓶中,在30℃、220rpm培养2.5h,直至od600长至0.35。然后将三角瓶取出置冰上冷却10min,之后将菌液转移至预冷的100ml离心管中,4℃离心收集菌体,再用预冷的无菌水和10%甘油各洗两次,然后用1ml预冷的10%甘油重悬菌体,最后分装到预冷的1.5ml无菌ep管中,每管100μl,得到制备好的e.colimg5感受态。向e.colimg5感受态中加入pgrs质粒和外源供体dna片段,混匀后加入电击杯中,使用电穿孔仪在1.8kv条件下电击约4.5ms,电击后迅速加入800μllb培养基,在30℃、220rpm复苏培养3h。复苏培养后,取100μl菌液涂布在装有终浓度为100μg/ml奇霉素和100μg/ml氨苄青霉素的lb平板上,30℃培养至长出单菌落。挑取单菌落pcr验证质粒是否导入,得到正确的突变菌株,之后借助grna质粒消除系统消除grna质粒。正确的克隆在含有4%阿拉伯糖的lb液体培养基中培养10h后在含有100μg/ml奇霉素lb平板上划线,培养后长出的菌落对点在含有终浓度为100μg/ml氨苄青霉素的lb平板上,不具有氨苄青霉素抗性的菌株说明其grna质粒消除,将其在lb培养基中37℃条件下培养,消除温度敏感型质粒ptkredcas9。最终,得到经测序后的仅含有目的突变的重构菌株,引入突变基因soxr(g121p)对应的菌株命名为e.colimg6。
goldengate克隆反应体系为:10xt4dnaligase缓冲液1μl、thermofastdigestbsai0.5μl、cutsmart1μl、t4dna连接酶0.5μl、dna模板2.5μl、加双蒸水补齐至总体积为10μl。
fastpfudna聚合酶扩增体系为:5xtransstartfastpfudnapolymerase缓冲液10μl、dntp5μl、dna模板2-10μl、引物各2μl、transstartfastpfudna聚合酶1μl、加双蒸水补齐至总体积为50μl。
实施例5
工程菌株e.colimg6对于不同抗生素的耐药性验证
(1)工程菌株e.colimg6对多西环素耐药性验证的操作流程如下:
分别将菌株e.colimg1655和e.colimg6接入装有终浓度为25μg/ml多西环素的lb液体培养基中,250ml锥形瓶装液量为50ml,在37℃、220rpm培养48h。
耐药性验证结果见图1。从耐药性验证结果可以看出,相对于野生型e.colimg1655,包含有大肠杆菌全局调控因子的突变基因soxr(g121p)的工程菌(e.colimg6)显著提高了菌株对多西环素的耐药性。
(2)工程菌株e.colimg6对甲砜霉素耐药性的验证操作流程如下:
分别将菌株e.colimg1655和e.colimg6接入装有终浓度为100μg/ml甲砜霉素的lb液体培养基中,250ml锥形瓶装液量为50ml,在37℃、220rpm培养12h。
耐药性验证结果见图2。从耐药性验证结果可以看出,相对于野生型e.colimg1655,包含有大肠杆菌全局调控因子的突变基因soxr(g121p)的工程菌(e.colimg6)显著提高了菌株对甲砜霉素的耐药性。
(3)工程菌株e.colimg6对阿奇霉素耐药性的验证操作流程如下:
分别将菌株e.colimg1655和e.colimg6接入装有终浓度为120μg/ml阿奇霉素的lb液体培养基中,250ml锥形瓶装液量为50ml,在37℃、220rpm培养12h。
耐药性验证结果见图3。从耐药性验证结果可以看出,相对于野生型e.colimg1655,包含有大肠杆菌全局调控因子的突变基因soxr(g121p)的工程菌(e.colimg6)显著提高了菌株对阿奇霉素的耐药性。
接种方式为:lb平板活化菌株,37℃过夜培养,挑取单菌落接种到含5mllb液体培养基的试管,培养12h后,以初始od600为0.1的接种量接种到lb液体培养基。
实施例6
大肠杆菌全局调控因子的突变基因soxr(g121p)引入后细胞的转录调控变化
(1)总rna的提取采用全式金生物公司的easypurernakit。具体操作步骤如下:
将菌株e.colimg1655和e.colimg6在37℃、220rpm条件下培养长至对数中期。将离心机预冷至4℃,收集1.5ml菌体,13000rpm离心2min。离心后,用枪头去除全部培养基上清,再用100μl含有溶菌酶的te缓冲液彻底重悬菌体。之后加入350μlbb4(用前需加入β-巯基乙醇),漩涡震荡混匀,室温孵育5分钟。用无rna酶的针管反复吹吸几次,使溶液均质化,室温条件下12000rpm离心2min,吸上清于rnase-free的离心管中,向上清液中加入250μl无水乙醇,混匀后转移至离心柱中,12000rpm离心30s,弃掉流出液。之后向离心柱中加入500μlcb4,室温12000rpm离心30s,弃掉流出液。再加入500μlwb4,室温12000rpm离心30s,弃掉流出液,并重复一次。之后室温12000rpm离心2min,为了去除残留的乙醇,在室温静置数分钟彻底晾干离心柱,然后将离心柱转移至一个新的1.5mlrnase-free离心管中,向离心柱中央加入30-100μlrnase-freewater,室温静置1min,室温12000rpm离心2min,洗脱rna,置于-80℃保存。
(2)cdna的合成采取全式金生物公司的one-stepgdnaremovalandcdnasynthesis试剂盒。具体操作步骤如下:
将已提取的rna及试剂randomprimer、2×tsreactionmix、rnase-freewater置于冰上。在pcr管中加入10μl2×tsreactionmix,1μlrna样品,1μlgdnaremover,1μlrandomprimer,6μlrnase-freewater,1μlenzymemix,混匀后,25℃孵育10min,50℃孵育15min,之后在85℃加热5s失活酶活性,最后将所得的cdna于-80℃保存。
(3)实时荧光定量pcr采取全式金生物公司的topgreenqpcr试剂盒。具体操作步骤如下:
qpcr的体系与反应条件见表3。体系在冰上配制完成后,封好封口膜,在1000×g,4℃离心1min,置于实时荧光定量pcr仪中(cycler480,roche),反应程序设置见表4。rt-pcr所用引物见表2。
表3rt-pcr反应体系
表4rt-pcr反应程序
大肠杆菌全局调控因子突变基因soxr(g121p)对所调控的多西环素耐药性相关基因表达水平的影响见表5,表中将对照菌株e.colimg1655的表达水平看作1。结果表明,大肠杆菌全局调控因子突变基因soxr(g121p)在一定程度上改写了细胞的代谢网络,调控多个基因表达水平的改变,如大肠杆菌的外排蛋白相关基因acrz、acrb、acra的转录水平分别上调2.07、3.02和5.43倍,说明soxr基因的突变使菌株向胞外运输多西环素的作用增强,从而从而使大肠杆菌对多西环素的耐药性增强。细胞膜通透性相关基因ompn在抗生素进入时形成渗透屏障,其转录水平上调5.35倍,细胞膜通透性相关基因ompf编码外膜孔蛋白,形成抗生素进入的通道,其转录水平下调0.27倍,说明soxr基因的突变使细胞通透性降低,限制了多西环素进入细胞,从而提高菌株对多西环素的耐药性。
表5rt-pcr分析突变基因soxr(g121p)对所调控基因表达水平的影响
参考文献:
1.koutsolioutsoua,pena-llopiss,dempleb.constitutivesoxrmutationscontributetomultiple-antibioticresistanceinclinicalescherichiacoliisolates[j].antimicrobialagentsandchemotherapy,2005,49(7):2746-2752.
2.ozt,guveneka,yildizs,etal.strengthofselectionpressureisanimportantparametercontributingtothecomplexityofantibioticresistanceevolution[j].molecularbiologyandevolution,2014,31(9):2387-2401.
3.garstad,bassalomc,pinesg,etal.genome-widemappingofmutationsatsingle-nucleotideresolutionforprotein,metabolicandgenomeengineering[j].naturebiotechnology,2016,35(1):48-55.
4.liy,linz,huangc,etal.metabolicengineeringofescherichiacoliusingcrispr-cas9meditatedgenomeediting[j].metabolicengineering,2015,31:13-21.
5.linz,xuz,liy,etal.metabolicengineeringofescherichiacolifortheproductionofriboflavin[j].microbialcellfactories,2014,13(1).
序列表
<110>天津大学
<120>大肠杆菌全局调控因子的突变基因soxr及应用
<160>30
<170>siposequencelisting1.0
<210>1
<211>465
<212>dna
<213>人工序列(artificialsequence)
<400>1
atggaaaagaaattaccccgcattaaagcgctgctaacccccggcgaagtggcgaaacgc60
agcggtgtggcggtatcggcgctgcatttctatgaaagtaaagggttgattaccagtatc120
cgtaacagcggcaatcagcggcgatataaacgtgatgtgttgcgatatgttgcaattatc180
aaaattgctcagcgtattggcattccgctggcgaccattggtgaagcgtttggcgtgttg240
cccgaagggcatacgttaagtgcgaaagagtggaaacagctttcgtcccaatggcgagaa300
gagttggatcggcgcattcataccttagtggcgctgcgtgacgaactggacggatgtatt360
ccgtgtggctgcctttcgcgcagtgattgcccgttgcgtaacccgggcgaccgcttagga420
gaagaaggtaccggcgcacgcttgctggaagatgaacaaaactaa465
<210>2
<211>154
<212>prt
<213>人工序列(artificialsequence)
<400>2
metglulyslysleuproargilelysalaleuleuthrproglyglu
151015
valalalysargserglyvalalavalseralaleuhisphetyrglu
202530
serlysglyleuilethrserileargasnserglyasnglnargarg
354045
tyrlysargaspvalleuargtyrvalalaileilelysilealagln
505560
argileglyileproleualathrileglyglualapheglyvalleu
65707580
progluglyhisthrleuseralalysglutrplysglnleuserser
859095
glntrpargglugluleuaspargargilehisthrleuvalalaleu
100105110
argaspgluleuaspglycysileprocysglycysleuserargser
115120125
aspcysproleuargasnproglyaspargleuglyglugluglythr
130135140
glyalaargleuleugluaspgluglnasn
145150
<210>3
<211>55
<212>dna
<213>人工序列(artificialsequence)
<400>3
cacaccaggtctcactggacggatgtatgttttagagctagaaatagcaagttaa55
<210>4
<211>50
<212>dna
<213>人工序列(artificialsequence)
<400>4
cacaccaggtctcaccagttcgtcaactagtattatacctaggactgagc50
<210>5
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>5
catttatcagggttattgtctc22
<210>6
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>6
cgtattaccgcctttgag18
<210>7
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>7
tcatctgttggggagtataa20
<210>8
<211>42
<212>dna
<213>人工序列(artificialsequence)
<400>8
ggcagccacacgggatgcatccgtccagttcgtcacgcagcg42
<210>9
<211>42
<212>dna
<213>人工序列(artificialsequence)
<400>9
ggatgcatcccgtgtggctgcctttcgcgcagtgattgcccg42
<210>10
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>10
tgatggcgtagacataagaa20
<210>11
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>11
cgagcatattgaccagccgcttaacat27
<210>12
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>12
ctgctgcgagacataacccaggt23
<210>13
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>13
ctggatattaccgcggcacagtt23
<210>14
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>14
cacatattgccgcgccgccggt22
<210>15
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>15
catgaaaccactttcatccgcaat24
<210>16
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>16
tagcgtgttgattataatagggcac25
<210>17
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>17
ctgtgcttagcggttagaatagt23
<210>18
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>18
cgactaagcgggcattcaggga22
<210>19
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>19
cacctctccgattagcggtcgcat24
<210>20
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>20
cgggaacttaatgccgtcactggt24
<210>21
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>21
gcttcttcggctggtttaaccgcat25
<210>22
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>22
ctgtgtacgttcctgcgttgcacct25
<210>23
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>23
caatggcgtcgcgacttatcgtaat25
<210>24
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>24
cattggtgcggtcagaagaggtgtat26
<210>25
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>25
cgatgacttcttcgttggtcgtgt24
<210>26
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>26
caggttggtacggtcagctgcaccat26
<210>27
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>27
ctgatcaccaaactggaccagct23
<210>28
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>28
caccagccatgcccagccggaac23
<210>29
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>29
cttgcaggatgccacgccgttaacgct27
<210>30
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>30
ggtaacaaacggtgcacaggtaat24
- 还没有人留言评论。精彩留言会获得点赞!