人类ZNT8抗体
相关申请的交叉引用
本申请要求2018年8月16日提交的美国临时专利申请第62/719,016号的权益,其内容通过引用整体并入本文。
政府利益声明
本发明是在美国国立卫生研究院(thenationalinstitutesofhealth)授予的批准号gm065137的资助下在政府支持下完成的。政府对本发明享有一定的权利。
本发明涉及免疫学和自身免疫学领域。更具体地说,本发明提供涉及胰腺锌转运体znt8的抗体的产生和使用的方法和组合物。
通过引用合并电子提交的材料
本申请包含序列表。该序列表已经由efs-web以名称为“p15404-02_st25.txt”的ascii文本文件通过电子方式提交。序列表的大小为69547字节,创建于2019年8月15日。该序列表通过引用整体并入本文。
背景技术:
人类基因组编码大量锌转运蛋白(zinctransporter),该锌转运蛋白参与调节细胞锌稳态和信号传导(1)。在所有锌转运蛋白中,锌转运蛋白8(znt8)在其组织特异性表达方面是独特的(2)。小鼠组织的微阵列分析(3)和人胰岛的单细胞转录组分析(4)表明,scl30a8转录主要限于胰岛的内分泌细胞。slc30a8的转录谱具有与众所周知的β细胞标记物(例如pdx1和nkx6.1)相似的胰岛特异性程度(4)。单细胞rnaseq显示,这些β细胞标记物也具有向非β内分泌细胞的“溢出(spill-over)”转录,但“溢出”并未延伸至外分泌细胞或导管细胞(4)。在胰岛β细胞中,znt8主要在胰岛素分泌颗粒中表达(2,5),其中需要znt8以六聚体胰岛素的形式进行颗粒状锌富集和锌胰岛素晶体封装(6,7)。胰岛素颗粒内的锌含量超过10mm(8)。高的锌水平与胰岛素合成、储存、胰岛素分泌的调节和葡萄糖刺激的胰岛素分泌(gsis)后的肝胰岛素清除有关(9-11)。
胰岛β细胞是人体内提供胰岛素的唯一来源。胰岛素生产不足是2型糖尿病(type-2diabete,t2d)的主要致病因素(12)。常见的t2d是一种复杂的多基因疾病,其与150多个t2d风险基因相关(13)。到目前为止,scl30a8是唯一已知的具有保护性功能丧失(loss-of-function,lof)突变的基因(14)。对跨多个人群队列的~150000个人进行基因分型显示,lof突变携带者患t2d的风险降低了65%(15)。slc30a8在t2d易感性遗传环境中的独特位置使znt8成为有吸引力的治疗靶点(16)。值得注意的是,scl30a8(rs13266634)中的错义单核苷酸多态性与对t2d的易感性增加有关(17-19)。与较低风险的w325变体相比,较高风险的r325变体的锌转运活性是极度活跃的(20),这与来自携带r325变体的供体的人胰岛中较高的锌水平相对应(21)。此外,尽管znt8ko小鼠模型的gsis表型是多样化的(6,7,23,24),高脂饮食后过表达r325变体的转基因小鼠胰岛锌水平增加以及葡萄糖耐量降低(22)。将来自znt8生物化学、动物模型和人类遗传学的新证据合并在一起似乎显示出了将功能获得性r235变异体与t2d风险增加联系起来的因果关系,这支持了将znt8抑制作为潜在的抗糖尿病策略的情况(16、25)。
可以使用两种替代方法来下调β细胞中的znt8活性:抑制锌转运或降低znt8表达。驱动锌转运的机制从细菌到人类都是保守的(26,27)。在细菌同源物yiip(28)中,锌转运通过锌与胞质c末端结构域(c-terminaldomain,ctd)结合易受变构调节的影响(29)。同样,可以使用mab将变构性靶向用于znt8抑制以将znt8保持在固定的构象,但仍缺乏原理证明变构抑制剂。此外,由于缺乏用于在异源背景下追踪znt8细胞水平的znt8特异性试剂,因此尚未研究β细胞中znt8表达的功能调节。发现具有特异性和高亲和力的mab将允许测试变构抑制假说,同时开发量化znt8细胞水平的测定法。
技术实现要素:
本发明至少部分地基于特异性结合胰腺锌转运蛋白znt8的单克隆抗体的开发。编码znt8的基因slc30a8是2型糖尿病的易感基因。降低znt8转运活性或下调其细胞表达被认为是一种抗糖尿病的策略,该策略模仿了slc30a8单倍体不足对人的保护作用。然而,尚无用于抑制znt8和测量znt8细胞水平的研究工具。如本文所述,本发明开发了适用于解决这些未满足的需求的单克隆znt8抗体。mab对人类znt8表现出次纳摩尔亲和力,并且对具有不同种间反应性的同源锌转运蛋白表现出选择性和表位识别。抗原结合片段(fab)使znt8免受解折叠并抑制在蛋白脂质体中的znt8介导的锌转运。负染色电子显微镜显示znt8单体与两种不同fab的1:1:1的化学计量比的三元结合复合物。两个mab与单个znt8蛋白的共结合使单个的抗znt8特异性倍增,从而能够通过均相时间分辨荧光(homogeneoustimeresolvedfluorescence,htrf)对znt8细胞水平进行定量。本文所述的mab可用作人类znt8的变构抑制剂和高度特异性生物传感器。
在特定实施方式中,本发明提供了与znt8特异性结合的分离的抗体或其抗体结合片段,其中该抗体或抗体结合片段包含重链互补决定区(complementaritydeterminingregion,cdr)1、2和3,其中该重链cdr1包含seqidno:3、13、23、33、43和53中的一个所示的氨基酸序列,或在两个或更少的氨基酸位置具有置换的seqidno:3、13、23、33、43和53中的一个所示的氨基酸序列,该重链cdr2包含seqidno:4、14、24、34、44和54中的一个所示的氨基酸,或在两个或几个氨基酸位置具有置换的seqidno:4、14、24、34、44和54中的一个所示的氨基酸,以及该重链cdr3包含seqidno:5、15、25、35、45和55中的一个所示的氨基酸序列,或在两个或更少的氨基酸位置具有置换的seqidno:5、15、25、35、45和55中的一个所示的氨基酸序列。
在其他实施方式中,该分离的抗体或抗原结合片段还包含轻链cdr1、cdr2和cdr3,其中该轻链cdr1包含seqidno:8、18、28、38、48和58中的一个所示的氨基酸序列,或在两个或更少的氨基酸位置具有置换的seqidno:8、18、28、38、48和58中的一个所示的氨基酸序列,该轻链cdr2包含seqidno:9、19、29、39、49和59中的一个所示的氨基酸序列,或在两个或更少的氨基酸位置具有置换的seqidno:9、19、29、39、49和59中的一个所示的氨基酸序列,以及该轻链cdr3包含seqidno:10、20、30、40、50和60中的一个所示的氨基酸序列,或在两个或更少的氨基酸位置具有置换的seqidno:10、20、30、40、50和60中的一个所示的氨基酸序列。
本发明还提供与znt8特异性结合的分离的抗体或其抗原结合片段,其中该抗体或其抗原结合片段包含重链可变区(heavychainvariableregion,vh),该重链可变区包含cdr1、cdr2和cdr3,该cdr1、cdr2和cdr3由以下所示的氨基酸序列组成:(a)分别地seqidno:3-5;(b)分别地seqidno:13-15;(c)分别地seqidno:23-25;(d)分别地seqidno:33-35;(e)分别地seqidno:43-45;以及(f)分别地seqidno:53-55。
在替代地实施方式中,与znt8特异性结合的分离的抗体或其抗原结合片段包含轻链可变区(lightchainvariableregion,vl),该轻链可变区包含cdr1、cdr2和cdr3,该cdr1、cdr2和cdr3由以下所示的氨基酸序列组成:(a)分别地seqidno:8-10;(b)分别地seqidno:18-20;(c)分别地seqidno:28-30;(d)分别地seqidno:38-40;(e)分别地seqidno:48-50;以及(f)分别地seqidno:58-60。
在特定实施方式中,与znt8特异性结合的分离的抗体或其抗原结合片段包括(a)vh以及(b)vl,该vh包含cdr1、cdr2和cdr,该cdr1、cdr2和cdr由以下所示的氨基酸序列组成:(i)分别地seqidno:3-5;(ii)分别地seqidno:13-15;(iii)分别地seqidno:23-25;(iv)分别地seqidno:33-35;(v)分别地seqidno:43-45;或(vi)分别地seqidno:53-55,该vl包含cdr1、cdr2和cdr3,该cdr1、cdr2和cdr3由以下所示的氨基酸序列组成:(vii)分别地seqidno:8-10;(viii)分别地seqidno:18-20;(ix)分别地seqidno:28-30;(x)分别地seqidno:38-40;(xi)分别地seqidno:48-50;或(xii)分别地seqidno:58-60。
在某些实施方式中,分离的抗体或抗原结合片段是znt8活性的拮抗剂。本发明还提供编码本文所述的抗znt8抗体或其抗原结合片段的分离的核酸分子。本发明还提供载体,该载体包含本文所述的核酸分子。在特定实施方式中,宿主细胞包含本文所述的载体。宿主细胞可以是原核或真核细胞。
在另一个方面,本发明提供用于产生抗znt8抗体或其抗原结合片段的方法。在一个实施方式中,用于产生抗znt8抗体或其抗原结合片段的方法包括以下步骤:(a)在适于宿主细胞表达znt8抗体或其抗原结合片段的条件下培养本文所述的宿主细胞;(b)回收该znt8抗体或其抗原结合片段。宿主细胞可以是原核或真核细胞。
在另一个方面,本发明提供组合物,该组合物包含本文所述的抗znt8抗体或其抗原结合片段以及合适的药物载体。该组合物可以配制用于静脉内给药、肌内给药、口服给药、皮下给药、腹膜内给药、鞘内给药或肌内给药。
在又一个方面,本发明提供用于治疗znt8介导的疾病或病症的方法,给疾病或病症包括但不限于糖尿病。在一个特定实施方式中,治疗哺乳动物的糖尿病的方法包括以下步骤:将治疗有效量的本文所述的与znt8特异性结合的抗体或其抗原结合片段给药至该哺乳动物。
在特定实施方式中,与znt8特异性结合的分离的抗体或其抗原结合片段,其中该抗体或其抗原结合片段包括:(a)包含具有分别在seqidno:3-5中示出的氨基酸序列的cdr1、cdr2和cdr3的vh;和包含具有分别在seqidno:8-10中示出的氨基酸序列的cdr1、cdr2和cdr3的vh;或(b)包含具有分别在seqidno:13-15中示出的氨基酸序列的cdr1、cdr2和cdr3的vh;和包含具有分别在seqidno:18-20中示出的氨基酸序列的cdr1、cdr2和cdr3的vh;或(c)包含具有分别在seqidno:23-25中示出的氨基酸序列的cdr1、cdr2和cdr3的vh;和包含具有分别在seqidno:28-30中示出的氨基酸序列的cdr1、cdr2和cdr3的vh;或(d)包含具有分别在seqidno:33-35中示出的氨基酸序列的cdr1、cdr2和cdr3的vh;和包含具有分别在seqidno:38-40中示出的氨基酸序列的cdr1、cdr2和cdr3的vh;或(e)包含具有分别在seqidno:43-45中示出的氨基酸序列的cdr1、cdr2和cdr3的vh;和包含具有分别在seqidno:48-50中示出的氨基酸序列的cdr1、cdr2和cdr3的vh;或(f)包含具有分别在seqidno:53-55中示出的氨基酸序列的cdr1、cdr2和cdr3的vh;和包含具有分别在seqidno:58-60中示出的氨基酸序列的cdr1、cdr2和cdr3的vh。在某些实施方式中,该抗原结合片段选自由以下组成的组:scfv、sc(fv)2、fab、f(ab)2和双抗体(diabody)。
本发明还提供了分离的抗体或其抗原结合片段,其特异性结合包含vh的znt8,该vh包含seqidno:2、12、22、32、42和52中所示的氨基酸序列之一。在替代实施方式中,与znt8特异性结合的分离的抗体或其抗原结合片段包含vl,该vl包含seqidno:7、17、27、37、47和57中所示的氨基酸序列之一。
在其他实施方式中,与znt8特异性结合的分离的抗体或其抗原结合片段包括:(a)包含seqidno:2所示的氨基酸序列的vh,和包含seqidno:7所示的氨基酸序列的vl;(b)包含seqidno:12所示的氨基酸序列的vh,和包含seqidno:17所示的氨基酸序列的vl;(c)包含seqidno:22所示的氨基酸序列的vh,和包含seqidno:27所示的氨基酸序列的vl;(d)包含seqidno:32所示的氨基酸序列的vh,和包含seqidno:37所示的氨基酸序列的vl;(e)包含seqidno:42所示的氨基酸序列的vh,和包含seqidno:47所示的氨基酸序列的vl;或(f)包含seqidno:52所示的氨基酸序列的vh,和包含seqidno:57所示的氨基酸序列的vl。
附图说明
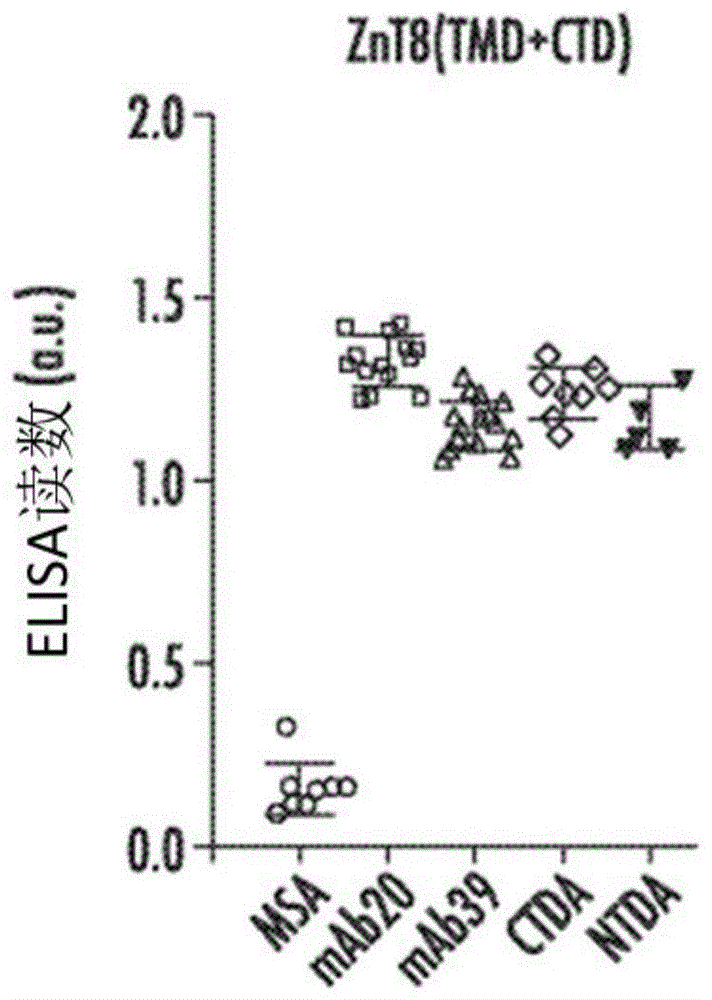
图1a-图1b.针对通过被动包被固定在蛋白脂质体中的znt8(tmd+ctd)绘制mab结合位点(图1a)elisa。通过hrp缀合的二抗来检测结合的抗体。对测定进行校准,ctda和ntda作为阳性对照并且msa作为阴性对照,z’因子>0.5。误差棒表示标准偏差。(图1b)通过c末端聚组氨酸标签对固定在ni-nta板上的单体ctd抗原进行的elisa。ctda和ntda分别用作阳性对照和阴性对照(z>0.5);
图2.以各种组合和结合化学计量将znt8与mab20、fab20和/或mab39复合得到的znt8抗体复合物的尺寸排阻hplc色谱图。解析了8个不同的峰,标记为1至8。表1总结了相应的复合物分子量和预测的复合物组成。对mab20/fab20和mab39进行荧光标记,并在多通道荧光检测器的绿色(显示为蓝色迹线)或红色(红色迹线)通道进行检测。灰色迹线是通过紫外线吸收检测到的未结合的znt8;
图3a-图3f.znt8-fab复合物的em分析。(图3a)负染色的二元复合物znt8-(fab20)2的代表性原始颗粒。比例尺为
图4a-图4c.mab结合稳定了znt8折叠(图4a),mab与折叠或变性的znt8结合。将洗剂胶束中的znt8-his在复制中固定在ni-nta板上,没有sds变性。如所示的,比较mab20和mab39对变性(三角形)和未变性znt8(圆形)的elisa。ctda和msa分别用作阳性对照和阴性对照。误差棒是标准偏差。(图4b)mab与洗剂胶束中的折叠的znt8-gfp结合。将mab20或mab39与znt8-gfp混合,然后通过尺寸排阻hplc和gfp荧光检测对结合混合物进行分析。注意,mab20将未结合的znt8-gfp峰(黑色)向左移动到mab20-znt8-gfp位置(红色),而mab39不会引起峰移位(品红色)。(图4c)对znt8解折叠的保护。将洗剂胶束中的znt8等分试样与fab20(红色)、fab39(品红色)或fab20+fab39(棕色)一起孵育。游离znt8用作对照(黑色)。在所示的指定的热变性时间后,通过超速离心去除蛋白质聚集体,并通过mab20免疫印迹法(上图)检测上清液中znt8的残留量。误差棒是使用imagej进行的一项代表性实验的6次独立的光密度测量的标准误差;
图5a-图5b.响应于znt8蛋白脂质体与囊外锌缓冲液的快速混合,通过fab结合抑制锌转运(图5a)的fluozin-3荧光迹线。如箭头所示,缓冲液中的游离锌浓度从1.6μm增加到116μm。(图5b)所示的由znt8或与fab20、fab39或fab20+fab39结合的znt8介导的锌转运的稳态动力学。实线是用最小二乘法拟合成的米氏方程(michaelis–mentenequation)。表2总结了km值和vmax值;
图6a-图6c.mab结合亲和力。(图6a)如所示的,用mab20或mab39染色的固定的和皂苷透化的hek293细胞的免疫荧光染色的代表性共聚焦显微镜图像。znt8(+)或znt8(-)表示没有znt8过表达的hek293细胞。红色:mab20或mab39染色,蓝色:dapi,比例尺:5μm。(图6b)流式细胞仪计数事件的直方图。箭头指示由于mab系列稀释而导致的直方图的左移。(图6c)如所示的,mab与没有znt8过表达的hek293细胞结合。从(图6b)获得连续稀释的mab20或mab39标记的免疫荧光强度。实线是结合数据到单一组分结合过程的最小二乘拟合;
图7a-图7d.mab结合特异性(图7a),mab与固定在ni-nta板上的znt8-his结合。通过hrp缀合的二抗检测到结合的mab20(红色)或mab39(品红色)。实线是结合数据点到单一组分结合过程的最小二乘拟合。误差棒是标准误差(n=8)。(图7b)如所示的,通过mab20或mab39来检测免疫印迹上的sds变性的小鼠znt8-his(标记为m)和人类znt8-his(标记为h)。用抗his标签(anti-his-tag)抗体来探测复制的免疫印迹,显示在每个泳道上上样了大致相同量的hek293粗裂解物。(图7c)通过mab20或mab38免疫印迹对endoc-βh细胞中的内源性人类znt8进行检测。将等量的endoc-βh裂解物(5μg总蛋白)上样至每个泳道。(图7d)如所示的具有znt同源物或znt同源物过表达的hek裂解物的免疫印迹。如所示的,在每个泳道上放置大约等量的总裂解物蛋白(5μg),并用mab20、mab39或抗falg抗体进行探测;
图8.htrf对人类znt8、小鼠znt8和人类znt3的响应。
具体实施方式
应当理解,本发明不限于本文描述的特定方法和成分等,因为它们可以变化。也应当理解,本文所使用的术语仅用于描述特定实施方式的目的,并不旨在限制本发明的范围。必须注意的是,除非上下文另有明确说明,否则如本文和所附权利要求书中所使用的单数形式的“一”、“一个”和“该”包括复数形式。因此,例如,提及“蛋白质”是提及一种或多种蛋白质,并且包括本领域技术人员已知的其等同物等等。
除非另有定义,否则本文使用的所有技术和科学术语均具有如本发明所属的领域中的普通技术人员通常所理解的相同含义。尽管与本文描述的那些方法或材料相似或等同的任何方法和材料都可以用于本发明的实践或测试中,但对特定的方法、装置和材料进行了描述。
本文引用的所有出版物均通过引用并入本文,包括所有期刊文章、书籍、手册、公开的专利申请和已发布的专利。另外,提供了在说明书、实施例和所附权利要求中使用的某些术语和短语的含义。所述定义本质上并不意味是限制性的,而是用于提供对本发明某些方面的更清楚的理解。
i.定义
术语“抗体”意指通过免疫球蛋白分子的可变区内的至少一个抗原识别位点来识别并特异性结合靶(例如蛋白质(例如,znt8,其亚基或受体复合物)、多肽、肽、碳水化合物、多核苷酸、脂质或前述组合)的免疫球蛋白分子。典型的抗体包含通过二硫键互连的至少两条重链(heavychain,hc)和两条轻链(lightchain,lc)。每条重链由“重链可变区(heavychainvariableregion)”或“重链可变结构域(heavychainvariabledomain)”(本文缩写为vh)和重链恒定区(heavychainconstantregion)组成。重链恒定区由三个结构域ch1、ch2和ch3组成。每条轻链由“轻链可变区(lightchainvariableregion)”或“轻链可变结构域(lightchainvariabledomain)”(本文缩写为vl)和轻链恒定区(lightchainconstantregion)组成。轻链恒定区由一个结构域ci组成。vh区和vl区可以进一步细分为高度可变区,称为互补决定区(complementaritydeterminingregion,cdr),其间散布着更为保守的区,称为框架区(frameworkregion,fr)。每个vh区和vl区由三个cdr和四个fr组成,从氨基端到羧基端按以下顺序排列:fri、cdri、fr2、cdr2、fr3、cdr3、fr4。重链和轻链的可变区包含与抗原相互作用的结合结构域。如本文所用,术语“抗体”涵盖:完整的多克隆抗体、完整的单克隆抗体、抗体片段(例如fab、fab'、f(ab')2、fd、facb和fv片段)、单链fv(scfv)、微抗体(例如,sc(fv)2、双抗体)、多特异性抗体(例如由至少两种完整的抗体产生的双特异性抗体)、嵌合抗体、人源化抗体、人抗体、包含抗体的抗原决定部分的融合蛋白、以及任何其他包含抗原识别位点的修饰的免疫球蛋白分子,只要抗体表现出所需的生物活性即可。因此,术语“抗体”包括整个抗体及其任何抗原结合片段或单链。抗体可以是裸露的或与其他分子(例如毒素、放射性同位素、小分子药物、多肽等)缀合。
术语“分离的抗体”是指已经从其天然环境的成分中鉴定、分离和/或回收的抗体。其天然环境的污染成分是会干扰抗体诊断或治疗用途的物质,可能包括酶、激素和其他蛋白质溶质或非蛋白质溶质。在一些实施方式中,将抗体纯化(1)至大于抗体的95wt%,例如,通过lowry方法测定的,以及包括大于99wt%,(2)至足以通过使用旋杯式序列分析仪获得n-末端或内部氨基酸序列的至少15个残基的程度,或(3)达到在还原或非还原条件下使用考马斯蓝(coomassieblue)或银染通过sds-page获得的均一性。分离的抗体包括重组细胞内的原位抗体,因为不会存在抗体的天然环境的至少一种成分。然而,通常分离的抗体将通过至少一个纯化步骤来制备。
术语“人源化”免疫球蛋白是指包含人框架区和来自非人类(通常是小鼠或大鼠)免疫球蛋白的一个或多个cdr的免疫球蛋白。提供cdr的非人免疫球蛋白称为“供体”,以及提供框架的人免疫球蛋白称为“受体”。恒定区不必存在,但如果其存在,则它们必须与人免疫球蛋白恒定区基本相同,即至少约85%-90%、优选约95%或更高相同。因此,人源化免疫球蛋白(可能不包括cdr)的所有部分与天然人免疫球蛋白序列的相应部分基本相同。“人源化抗体”是包含人源化轻链和人源化重链免疫球蛋白的抗体。例如,人源化抗体将不涵盖如上定义的典型的嵌合抗体,例如,因为嵌合抗体的整个可变区是非人的。
术语“抗原结合片段”是指完整的抗体的一部分,并且是指完整的抗体的抗原决定可变区。在本领域中已知,抗体的抗原结合功能可以通过全长抗体的片段来进行。抗原结合抗体片段的实例包括但不限于:fab、fab'、f(ab')2、facb、fd和fv片段、线性抗体、单链抗体和由抗体片段形成的多特异性抗体。在某些情况下,可以通过蛋白水解消化完整的抗体或整个抗体来制备抗体片段。例如,可以通过用酶(例如,木瓜蛋白酶、胃蛋白酶或血纤维蛋白溶酶)对整个抗体进行处理来获得抗体片段。对整个抗体进行木瓜蛋白酶消化产生f(ab)2或fab片段;对整个抗体进行胃蛋白酶消化得到f(ab')2或fab';对整个抗体进行血纤维蛋白溶酶消化得到facb片段。
术语“fab”是指基本上等同于通过用木瓜蛋白酶消化免疫球蛋白(通常为igg)而获得的抗体片段。fab片段的重链区段是fd段。这样的片段可以:通过对完整的抗体进行片段化而酶促产生或化学产生、从编码部分抗体序列的基因重组产生,或者其可以全部或部分合成产生。术语“f(ab’)2”是指基本上等同于通过在ph4.0-4.5下用胃蛋白酶消化免疫球蛋白(通常为igg)而获得的片段的抗体片段。这样的片段可以:通过对完整的抗体进行片段化而酶促产生或化学产生、从编码部分抗体序列的基因重组产生,或者其可以全部或部分合成产生。术语“fv”是指由通过非共价相互作用保持在一起的一个nh和一个n结构域组成的抗体片段。
术语“znt8抗体”、“抗znt8抗体”、“抗znt8”、“与znt8结合的抗体”及其任何语法变形是指能够以足够的亲和力与znt8特异性结合使得该抗体可用作靶向znt8的治疗剂或诊断试剂的抗体。如通过例如放射免疫测定(radioimmunoassay,ria)、biacoretm(使用重组znt8作为分析物以及抗体作为配体,或相反)或本领域已知的其他结合测定法测定的,本文公开的抗znt8抗体与不相关的非znt8蛋白的结合程度小于该抗体与znt8的结合的约10%。在某些实施方式中,与znt8结合的抗体的解离常数(dissociationconstant,kd)<1μm、<100nm、<50nm、<10nm或<1nm。
两条多肽(或多核苷酸)序列之间的术语“%同一性”是指考虑到为了最佳比对两个序列而必须引入的添加或缺失(即空位),序列在比对窗口中共享的相同匹配位置的数量。匹配位置是在靶序列和参考序列中都存在相同核苷酸或氨基酸的任何位置。由于缺口不是核苷酸或氨基酸,因此不对靶序列中出现的缺口进行计数。同样,由于对靶序列的核苷酸或氨基酸而不是参考序列的核苷酸或氨基酸进行了计数,因此不对参考序列中的缺口进行计数。序列同一性百分比通过以下计算:确定两个序列中出现相同氨基酸残基或核酸碱基的位置数以得到匹配位置数,将匹配位置数除以比对窗口中的位置总数,然后将结果乘以100,得到序列同一性百分比。序列的比对和两个序列之间百分比序列同一性的确定可以使用在线和下载都易于使用的软件来完成。合适的软件程序可从各种来源获得,并用于蛋白质和核苷酸序列的比对。一种确定百分比序列同一性的合适程序是bl2seq,它是程序blast套件的一部分,该程序可从美国政府的国家生物技术信息中心(centerforbiotechnologyinformation)blast网站获得。bl2seq使用blastn或blastp算法在两个序列之间进行比对。blastn用于比对核酸序列,而blastp用于比对氨基酸序列。其他合适的程序是例如needle、stretcher、water或matcher,它们是生物信息学程序emboss套件的一部分,并且也可在www.ebi.ac.uk/tools/psa上从欧洲生物信息学研究所(europeanbioinformaticsinstitute,ebi)获得。在某些实施方式中,将第一氨基酸序列与第二序列氨基酸的同一性百分比“x”计算为100×(y/z),其中y是在第一序列和第二序列的比对(如通过目视检查或特定的序列比对程序进行比对的)中被评分为相同匹配的氨基酸残基的数目,z为第二序列中的残基总数。如果第一序列的长度大于第二序列的长度,则第一序列与第二序列的百分比同一性将高于第二序列与第一序列的百分比同一性。本领域技术人员将理解,用于计算百分比序列同一性的序列比对的产生不限于由一级序列数据驱动的二元序列-序列比对。序列比对可以源自多个序列比对。一种生成多个序列比对的合适的程序是clustalw2(clustalx是移植到windows环境的clustalw2程序的一个版本)。另一个合适的程序是muscle。clustalw2和muscle可选地从例如欧洲生物信息学研究所(ebi)获得。
术语“治疗剂”是指在治疗疾病或病症中使用的任何生物制剂或化学制剂。治疗剂包括任何合适的生物活性化合物、生物衍生的成分(例如细胞、肽、抗体和多核苷酸),以及放射化学治疗剂(例如放射性同位素)。在一些实施方式中,治疗剂包括化学治疗剂或止痛剂。
如本文所用,术语“......的治疗(treatment)”、“对......进行治疗(treating)”、“治疗(treat)”等是指获得所需的药理作用和/或生理作用。该术语还用于给药“治疗有效量”的试剂,例如抗znt8抗体。就完全或部分预防特殊结果:疾病或其症状而言,该作用可以是预防性的,和/或就部分或完全治愈疾病和/或可归因于该疾病的不利影响而言,该作用可以是治疗性的。如本文所用的“......的治疗”涵盖对对象、特别是人的疾病的任何治疗,并且包括:(a)预防该疾病在可能易患该疾病但尚未被诊断患有该疾病的对象中发生;(b)抑制疾病,即阻止其发展;以及(c)缓解疾病,例如,导致疾病的消退,例如,以完全或部分消除疾病的症状。在特定的实施方式中,该术语用于预防或治疗任何znt8介导的疾病的情况,包括糖尿病。
ii.抗znt8抗体
本公开的抗体或其抗原结合片段特异性结合znt8。在特定的实施方式中,这些抗体或抗原结合片段与人znt8特异性结合。如本文所用,“特异性结合”意指抗体或抗原结合片段比其他蛋白质优先结合znt8(例如,人znt8、小鼠znt8)。在某些情况下,本公开的抗znt8抗体对znt8的亲和力高于对其他蛋白质的亲和力。与znt8特异性结合的抗znt8抗体对人znt8的结合亲和力可以小于或等于1×10-7m、小于或等于2×10-7m、小于或等于3×10-7m、小于或等于4×10-7m、小于或等于5×10-7m、小于或等于6×10-7m、小于或等于7×10-7m、小于或等于8×10-7m、小于或等于9×10-7m、小于或等于1×10-8m、小于或等于2×10-8m、小于小于或等于3×10-8m、小于或等于4×10-8m、小于或等于5×10-8m、小于或等于6×10-8m、小于或等于等于7×10-8m、小于或等于8×10-8m、小于或等于9×10-8m、小于或等于1×10-9m、小于或等于2×10-9m、小于或等于3×10-9m、小于或等于4×10-9m、小于或等于5×10-9m、小于或等于6×10-9m、小于或等于7×10-9m、小于或等于8×10-9m、小于或等于9×10-9m、小于或等于1×10-10m、小于或等于2×10-10m、小于或等于3×10-10m、小于或等于4×10-10m、小于或等于5×10-10m、小于或等于6×10-10m、小于或等于7×10-10m、小于或等于8×10-10m、小于或等于9×10-10m、小于或等于1×10-11m、小于或等于2×10-11m、小于或等于3×10-11m、小于或等于4×10-11m、小于或等于5×10-11m、小于或等于6×10-11m、小于或等于7×10-11m、小于或等于8×10-11m、小于或等于9×10-11m、小于或等于1×10-12m、小于或等于2×10-12m、小于或等于等于3×10-12m、小于或等于4×10-12m、小于或等于5×10-12m、小于或等于6×10-12m、小于或等于7×10-12m、小于或等于8×10-12m或小于或等于9×10-12m。测量抗体结合亲和力的方法是本领域众所周知的,其包括表面等离子体共振(surfaceplasmonresonance,spr)(morton和myszka“kineticanalysisofmacromolecularinteractionsusingsurfaceplasmonresonancebiosensors”methodsinenzymology(1998)295,268-294)、生物膜层干涉技术(bio-layerinterferometry)(abdicheetal“determiningkineticsandaffinitiesofproteininteractionsusingaparallelreal-timelabel-freebiosensor,theoctet”analyticalbiochemistry(2008)377,209-217)、动能排斥试验(kineticexclusionassay)(kinexa)(darlingandbrault“kineticexclusionassaytechnology:characterizationofmolecularinteractions”assayanddrugdevtech(2004)2,647-657)、等温量热法(isothermalcalorimetry)(pierceetal“isothermaltitrationcalorimetryofprotein-proteininteractions”methods(1999)19,213-221)以及分析超速离心(analyticalultracentrifugation)(lebowitzetal“modemanalyticalultracentrifugationinproteinscience:atutorialreview”proteinscience(2002),11:2067-2079)。
a.抗体片段
本公开涵盖本文所述的保留与znt8(例如人类znt8)特异性结合的能力的抗体片段或结构域。抗体片段包括例如fab、fab’、f(ab’)2、facb和fv。这些片段可以是人源化的或完全是人类的。可以通过对完整的抗体进行蛋白水解消化来制备抗体片段。例如,可以通过用酶(例如,木瓜蛋白酶、胃蛋白酶或血纤维蛋白溶酶)对整个抗体进行处理来获得抗体片段。对整个抗体进行木瓜蛋白酶消化产生f(ab)2或fab片段;对整个抗体进行胃蛋白酶消化得到f(ab')2或fab';对整个抗体进行血纤维蛋白溶酶消化得到facb片段。
或者,可以重组产生抗体片段。例如,可以构建编码目标抗体片段的核酸,将其引入表达载体中,并在合适的宿主细胞中表达。参见,例如,co,m.s.etal.,jimmunol.,152:2968-2976(1994);better,m.andhorwitz,a.h.,methodsinenzymology,178:476-496(1989);pluckthun,aandskerra,a,methodsinenzymology,178:476-496(1989);lamoyi,e.,methodsinenzymology,121:652-663(1989);rousseaux,j.etal.,methodsinenzymology,(1989)121:663-669(1989);以及bird,re.etal.,tibtech,9:132-137(1991))。抗体片段可以在大肠杆菌(e.coli)中表达并从大肠杆菌中分泌出来,因此使得轻松地生产大量的这些片段。可以从抗体噬菌体文库中分离抗体片段。或者,可以从大肠杆菌中直接回收fab’-sh片段,并对其进行化学偶联以形成f(ab)2片段(carteretal.,bio/technology,10:163-167(1992))。根据另一种方法,可以直接从重组宿主细胞培养物中分离f(ab’)2片段。在美国专利第5,869,046号中描述了包含补救受体结合表位残基的具有增加的体内半衰期的fab和f(ab’)22片段。
b.微抗体(minibody)
还涵盖本文描述的抗体的微抗体。抗znt8抗体的微抗体包括双抗体、单链(scfv)和单链(fv)2(sc(fv)2)。
“双抗体”是通过基因融合构建的二价微抗体(参见,例如,holliger,p.etal.,proc.natl.acad.sci.us.a.,90:6444-6448(1993);ep404,097;wo93/11161)。双抗体是由两条多肽链组成的二聚体。双抗体的每个多肽链的vl和vh结构域通过接头结合。构成接头的氨基酸残基的数目可以在2至12个残基之间(例如3-10个残基或5个或大约5个残基)。双抗体中多肽的接头通常太短,以至于vl和vh不能彼此结合。因此,在同一多肽链中编码的vl和vh不能形成单链可变区片段,而是形成具有不同的单链可变区片段的二聚体。结果是,双抗体具有两个抗原结合位点。
scfv是通过将vh和vl与接头连接而获得的单链多肽抗体(参见,例如,hustonetal.,proc.natl.acad.sci.us.a.,85:5879-5883(1988);和pluckthun,“thepharmacologyofmonoclonalantibodies”vol.113,edresenburgandmoore,springerverlag,newyork,pp.269-315,(1994)。每个可变结构域(或其一部分)衍生自相同或不同的抗体。单链fv分子优选包含插入在vh结构域和vl结构域之间的scfv接头。示例性scfv分子在本领域中是已知的,并且例如在:美国专利第5,892,019号;hoetal,gene,77:51(1989);birdetal.,science,242:423(1988);pantolianoetal,biochemistry,30:10117(1991);milenicetal,cancerresearch,51:6363(1991);takkinenetal,proteinengineering,4:837(1991)中有所描述。
如本文所用,术语“scfv接头”是指插入在scfv的vl和vh结构域之间的部分。scfv接头优选将scfv分子维持在抗原结合构象。在一个实施方式中,scfv接头包含scfv接头肽或由scfv接头肽组成。在某些实施方式中,scfv接头肽包含gly-ser接头肽或由gly-ser接头肽组成。在其他实施方式中,scfv接头包含二硫键。
vh和vl的要连接的顺序没有特别限制,并且他们可以以任何顺序排列。排列的示例包括:[vh]接头[vl];或[vl]接头[vh]。scfv中的h链v区和l链v区可以衍生自本文所述的任何抗znt8抗体或其抗原结合片段。
sc(fv)2是其中两个vh和两个vl通过接头连接形成了单链的微抗体(hudson,etal.,jimmunol.methods,(1999)231:177-189(1999))。例如,可以通过将scfv与接头连接来制备sc(fv)2。本发明的sc(fv)2包括抗体,优选地其中两个vh和两个vl按以下顺序排列:从单链多肽的n末端开始,vh、vl、vh和vl([vh]接头[vl]接头[vh]接头[vl]);然而,两个vh和两个vl的顺序不限于上述排列,并且它们可以以任何顺序排列。下面列出了排列示例:
[vl]接头[vh]接头[vh]接头[vl]
[vh]接头[vl]接头[vl]接头[vh]
[vh]接头[vh]接头[vl]接头[vl]
[vl]接头[vl]接头[vh]接头[vh]
[vl]接头[vh]接头[vl]接头[vh]
通常,当四个抗体可变区连接时,需要三个接头;使用的接头可以是相同的或不同的。连接微抗体的vh和vl区的接头没有特别限制。在一些实施方式中,接头是肽接头。包含约3至25个残基(例如,5个、6个、7个、8个、9个、10个、11个、12个、13个、14个、15个、16个、17个、18个)的任何的任意单链肽都可以用作接头。
在其他实施方式中,接头是合成化合物接头(化学交联剂)。市场上可得的交联剂的例子包括:n-羟基琥珀酰亚胺(nhs)、双琥珀酰亚胺辛二酸酯(dss)、双(磺基琥珀酰亚胺基)辛二酸酯(bs3)、二硫代双(琥珀酰亚胺丙酸酯)(dsp)、二硫代双(磺基琥珀酰亚胺丙酸酯)(dtssp)、乙二醇双(琥珀酰亚胺基琥珀酸酯)(egs)、乙二醇双(磺基琥珀酰亚胺基琥珀酸酯)(磺基-egs)、双琥珀酰亚胺酒石酸酯(dst)、二磺基琥珀酰亚胺酒石酸酯(磺基-dst)、双[2-(琥珀酰亚胺氧羰基氧)乙基]砜(bsocoes)以及双[2-(磺基琥珀酰亚胺氧羰基氧)乙基]砜(磺基-bsocoes)。
抗体片段或微抗体中vh或vl的氨基酸序列可包括修饰,例如置换、缺失,添加和/或插入。例如,修饰可以在本文所述的抗znt8抗体的一个或多个cdr中。在某些实施方式中,修饰涉及抗znt8微抗体的vh的1、2或3个cdr和/或vl结构域的1、2或3个cdr中的1、2或3个氨基酸置换。进行这种取代以改善抗znt8微抗体的结合活性和/或功能活性。在其他实施方式中,可以删除或添加抗znt8抗体或其抗原结合片段的六个cdr中的一个或多个的1、2或3个氨基酸,只要当vh和vl相关联时具有znt8结合和/或功能活性即可。
c.vhh
vhh也称为纳米抗体,其源自骆驼(camel)和美洲驼(llama)中的重链抗体(缺少轻链)的抗原结合可变重链区(antigen-bindingvariableheavychainregion,vhh)。本公开涵盖特异性结合znt8的vhh。
d.新抗原受体可变结构域(variabledomainofnewantigenreceptor,vnar)
vnar是新抗原受体(ignar)的可变域。ignars以共价连接的重链同型二聚体形式存在于鲨鱼的血清中。它以可溶性和受体结合形式存在,由可变结构域(vnar)和不同数量的恒定结构域组成。vnar由cdr1和cdr3组成,并且代替具有hv2和hv4结构域的cdr2(参见,例如,barelleandporter,antibodies,4:240-258(2015))。本公开涵盖特异性结合znt8的vnar。
e.恒定区
本公开的抗体可以是整个抗体或单链fc(scfc),并且可以包含本领域已知的任何恒定区。轻链恒定区可以是例如κ型或λ型轻链恒定区,例如人κ或人λ轻链恒定区。重链恒定区可以是例如α型、δ型、ε型、γ型或μ型重链恒定区,例如人α型、人δ型、人ε型、人γ型或人μ型重链恒定区。在某些情况下,抗znt8抗体是iga抗体、igd抗体、ige抗体、igg1抗体、igg2抗体、igg3抗体、igg4抗体或igm抗体。
在一个实施方式中,轻链或重链恒定区是天然存在的恒定区的片段、衍生物、变体或突变蛋白。在一些实施方式中,本文所述的抗znt8抗体的可变重链与包含ch1结构域和铰链区的重链恒定区连接。在一些实施方式中,可变重链与包含ch2结构域的重链恒定区连接。在一些实施方式中,可变重链与包含ch3结构域的重链恒定区连接。在一些实施方式中,可变重链与包含ch2和ch3结构域的重链恒定区连接。在一些实施方式中,可变重链与包含铰链区、ch2和ch3结构域的重链恒定区连接。ch1、铰链区、ch2和/或ch3可以来自igg抗体(例如,igg1、igg4)。在某些实施方式中,本文所述的抗znt8抗体的可变重链与重链恒定区连接,该重链恒定区包含来自igg4的chi结构域、铰链区和ch2结构域以及来自igg1的ch3结构域。在某些实施方式中,这样的嵌合抗体可以在重链恒定区中包含一个或多个增加嵌合抗体的稳定性的另外的突变。在某些实施方式中,重链恒定区包括修饰抗体性质的置换。
在某些实施方式中,本公开的抗znt8抗体是igg同种型抗体。在一些实施方式中,抗体是igg1。在另一实施方式中,抗体是igg2。在又一实施方式中,抗体是igg4。在一些情况下,igg4抗体具有一种或多种减少或阻止其采用功能性单价形式的突变。例如,可以使igg4的铰链区突变以使其氨基酸序列与人igg1的铰链区相同(人igg4铰链中的丝氨酸突变为脯氨酸)。在一些实施方式中,抗体具有嵌合重链恒定区(例如,具有igg4的ch1区、铰链区和ch2区以及igg1的ch3区)。
f.双特异性抗体
在某些实施方式中,本公开的抗znt8抗体是双特异性抗体。双特异性抗体是对至少两个不同表位具有结合特异性的抗体。示例性的双特异性抗体可以结合znt8蛋白的两个不同的表位。其他这样的抗体可以将znt8结合位点与另一种蛋白质的结合位点结合。可以将双特异性抗体制备为全长抗体或其低分子量形式(例如,f(ab’)2双特异性抗体、sc(fv)2双特异性抗体、双抗体双特异性抗体)。
全长双特异性抗体的传统生产是基于两个免疫球蛋白重链-轻链对的共表达,其中两条链具有不同的特异性(millsteinetal.,nature,305:537-539(1983))。在不同的方法中,将具有所需结合特异性的抗体可变结构域与免疫球蛋白恒定结构域序列融合。将编码免疫球蛋白重链融合物和(如果需要)免疫球蛋白轻链的dna插入单独的表达载体中,并共转染到合适的宿主细胞中。这为调节三个多肽片段的比例方面提供了更大的灵活性。然而,当至少两条多肽链以相等的比率表达导致高产量时,可以将两条或全部三条多肽链的编码序列插入单个表达载体中。
根据美国专利第5,731,168号中描述的另一种方法,可以对一对抗体分子之间的界面进行工程改造,以使从重组细胞培养物中回收的异二聚体的百分比最大化。优选的界面包括ch3结构域的至少一部分。在该方法中,将来自第一抗体分子的界面的一个或多个小氨基酸侧链替换为较大的侧链(例如,酪氨酸或色氨酸)。通过用较小的氨基酸侧链(例如,丙氨酸或苏氨酸)替换大的氨基酸侧链,在第二抗体分子的界面上产生了与大的侧链相同或相似大小的补偿“腔(cavity)”。这提供了为相对于其他不需要的最终产物(例如同二聚体)提高异二聚体的产量提供了机制。
双特异性抗体包括交联或“异源缀合(heteroconjugate)”抗体。例如,异源缀合物中的一种抗体可以与抗生物素蛋白偶联,另一种可以与生物素偶联。异源缀合抗体可以使用任何方便的交联方法来制备。
“双抗体”技术为制备双特异性抗体片段提供了可选择的机制。片段包含通过接头连接至vl的vh,该接头太短以至于不允许同一链上两个结构域之间配对。因此,一个片段的vh结构域和vl结构域被迫与另一片段的互补的vl和vh结构域配对,从而形成两个抗原结合位点。
g.缀合抗体
本文公开的抗体或抗原结合片段可以与各种分子缀合,包括:大分子物质,例如聚合物(例如,聚乙二醇(peg)、用peg修饰的聚乙烯亚胺(pei-peg)、聚谷氨酸(pga)(n-(2-羟丙基)甲基丙烯酰胺(hpma)共聚物);人血清白蛋白或其片段;放射性物质(例如,90y、131i);荧光物质;发光物质;半抗原;酶;金属螯合物;和药物。
在某些实施方式中,抗znt8抗体或其抗原结合片段的部分被修饰,这改善了其在循环(例如,在血液、血清或其他组织中)中的稳定性和/或滞留性,例如改善了至少1.5倍、2倍、5倍、10倍、15倍、20倍、25倍、30倍、40倍或50倍。例如,抗znt8抗体或其抗原结合片段可以与聚合物相关联(例如缀合),该聚合物例如基本上非抗原性的聚合物,例如聚烯化氧或聚环氧乙烷。合适的聚合物在重量上将差异很大。可以使用数均分子量为约200道尔顿至约35000道尔顿(或约1000道尔顿至约15000道尔顿,以及2000道尔顿至约12500道尔顿)的聚合物。例如,抗znt8抗体或其抗原结合片段可以与水溶性聚合物(例如亲水性聚乙烯聚合物,例如聚乙烯醇或聚乙烯吡咯烷酮)缀合。这样的聚合物的实例包括:聚烯化氧均聚物(例如聚乙二醇(peg)或聚丙二醇)、聚氧乙烯化的多元醇、其共聚物和其嵌段共聚物,前提是保持嵌段共聚物的水溶性。其他有用的聚合物包括:聚氧化烯,例如聚氧化乙烯、聚氧化丙烯、以及聚氧化乙烯和聚氧化丙烯的嵌段共聚物;聚甲基丙烯酸酯;卡波姆(carbomer)以及支化或未支化多糖。
可以通过对本文所述的对抗体或其较低分子量形式进行化学修饰来制备上述缀合的抗体或片段。用于修饰抗体的方法是本领域众所周知的。
ii.抗体的表征
本文所述抗体的znt8结合特性可以通过任何标准方法来测量,例如以下方法中的一种或多种方法:
目标蛋白质(抗znt8抗体或其功能片段)与靶(例如,znt8)的结合相互作用可以使用
可以使用表面等离子体共振(spr)来分析目标蛋白(抗znt8抗体或其功能片段)与靶(例如znt8)的结合相互作用。spr或生物分子相互作用分析(biomolecularinteractionanalysis,bia)实时检测生物特异性相互作用,而无需标记任何相互作用物。
bia芯片的结合表面的质量变化(指示结合事件)导致表面附近的光的折射率发生变化(表面等离子体共振(spr)的光学现象)。折射率的变化产生可检测的信号,测量该信号作为生物分子之间实时反应的指示。例如,在美国专利第5,641,640号;raether(1988)surfaceplasmonsspringerverlag;sjolanderandurbaniczky(1991)anal.chem63:2338-2345;szaboetal.(1995)curr.opin.struct.biol.5:699-705以及on-lineresourcesprovidebybiacoreinternationalab(uppsala,sweden)中描述了使用spr的方法。来自spr的信息可用于提供对生物分子与靶结合的平衡解离常数(kd)以及动力学参数(包括kon和koff)进行精确和定量测量。
也可以使用biacore色谱技术通过评估不同的抗znt8抗体或其功能片段彼此竞争结合人类znt8的能力来直接绘制表位(pharmaciabiatechnologyhandbook,“epitopemapping”,section6.3.2,(may1994);也可以参见johneetal.(1993)j.immunol.methods,160:191-198)。
当使用酶免疫测定法时,将包含抗体的样品,例如,产生抗体的细胞的培养物上清液或纯化的抗体,添加至抗原包被的板中。添加用酶(例如碱性磷酸酶)标记的第二抗体,将板孵育,洗涤后,添加酶底物(例如对硝基苯基磷酸酯),并测量吸光度以评估抗原结合活性。
用于评估抗体的其他通用指南,例如蛋白质印迹(westernblots)和免疫沉淀法,可以在antibodies:alaboratorymanual,ed.byharlowandlane,coldspringharborpress(1988)中找到。
iii.亲和力成熟
在一个实施方式中,例如通过诱变修饰抗znt8抗体或其抗原结合片段,以提供一池经修饰的抗体。然后对修饰的抗体进行评估以鉴定一种或多种具有改变的功能特性(例如,改善的结合、改善的稳定性、降低的抗原性或增加的体内稳定性)的抗体。在一个实施方式中,使用显示文库技术来选择或筛选修饰的抗体库。然后例如通过使用更高的严格性或更有竞争性的结合和洗涤条件,从第二文库中鉴定出更高的亲和力的抗体。也可以使用其他筛选技术。影响亲和力成熟的方法包括:随机诱变(例如,fukudaetal.,nucleicacidsres.,34:el27(2006);定向诱变(例如,rajpaletal.,proc.natl.acad.sci.usa,102:8466-71(2005);改组方法(shufflingapproach)(例如,jermutusetal.,proc.natl.acad.sci.usa,98:75-80(2001);以及在二氧化硅中的方法(insilicaapproache;例如,lippowetal.,nat.biotechnol.,25:1171-6(2005)。
在一些实施方式中,诱变靶向已知的或可能在结合界面处的区。如果,例如,所鉴定的结合蛋白是抗体,则诱变可以指向本文所述的重链或轻链的cdr区。此外,诱变可以指向靠近或邻近cdr的框架区,例如,特别是在cdr连接的10、5或3个氨基酸内的框架区。在抗体的情况下,诱变也可以限于一个或几个cdr,例如以进行逐步改进。
在一个实施方式中,诱变用于使抗体与一种或多种种系序列更相似。一种示例性种系方法可以包括:鉴定与分离的抗体的序列相似(例如,在特定数据库中最相似)的一种或多种种系序列。然后,可以在分离的抗体中进行突变(在氨基酸水平上),既可以是增量突变,也可以是组合突变,或两者兼有。例如,制备包含编码一些或所有可能的种系突变的序列的核酸文库。然后评估突变的抗体,例如以鉴定相对于分离的抗体具有一个或多个另外的种系残基并且仍然有用(例如具有功能活性)的抗体。在一个实施方式中,将尽可能多的种系残基引入分离的抗体中。
在一个实施方式中,诱变用于置换一个或多个种系残基或将其插入cdr区。例如,种系cdr残基可以来自与被修饰的可变区相似(例如,最相似)的种系序列。诱变后,可以评估抗体的活性(例如结合或其他功能活性),以确定种系残基或残基是否耐受。可以在框架区中进行类似的诱变。
可以以不同的方式选择种系序列。例如,如果种系序列满足选择性或相似性的预定标准,例如,相对于供体非人抗体具有至少一定百分比的同一性,例如至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、97%、98%、99%或99.5%的同一性,则可以选择该种系序列。可以使用至少2、3、5或10个种系序列进行选择。在cdr1和cdr2的情况下,鉴定相似的种系序列可以包括选择一个这样的序列。在cdr3的情况下,鉴定相似的种系序列可以包括选择一个这样的序列,但是可以包括使用分别有助于氨基末端部分和羧基末端部分的两个种系序列。在其他实施方式中,使用多于一个或两个的种系序列,例如以形成共有序列。
两个序列之间的“序列同一性”的计算如下。为了最佳比对目的,对序列进行比对(例如,为了最佳比对,可以在第一和第二氨基酸或核酸序列中的一个或这两个中引入空位,而为了比对目的,可以忽略非同源序列)。使用gcg软件包中的gap程序,根据最佳得分确定最佳比对,其中blossum62评分矩阵的空位罚分为12,空位延伸罚分为4,以及移码空位罚分为5。然后比较相应氨基酸位置或核苷酸位置处的氨基酸残基或核苷酸。当第一序列中的位置被与第二序列中的相应位置处的相同的氨基酸残基或核苷酸占据时,则分子在该位置处是相同的。两个序列之间的百分比同一性是序列共享的相同位置数的函数。
在其他实施方式中,可以对抗体进行修饰以具有改变的糖基化模式(即,从原始或天然糖基化模式改变)。如本文所用,“改变的”意指缺失一个或多个碳水化合物部分,和/或添加一个或多个糖基化位点至原始抗体。可以通过改变氨基酸序列以包含糖基化位点共有序列来实现将糖基化位点添加到目前公开的抗体中;这种技术在本领域中是众所周知的。增加在抗体上的碳水化合物部分的数目的另一种方法是通过糖苷与抗体氨基酸残基的化学或酶促偶联。这些方法在例如wo87/05330以及aplinandwriston(1981)crccrit.rev.biochem.,22:259-306中有所描述。去除抗体上存在的任何碳水化合物部分可以如本领域中所描述的那样通过化学或酶促完成(hakimuddinetal.(1987)arch.biochem.biophys.,259:52;edgeetal.(198l)anal.biochem.,118:131;以及thotakuraetal.(1987)meth.enzymol.,138:350)。参见例如美国专利第5,869,046号的修饰,该修饰通过提供补救受体结合表位来增加体内半衰期。
在一个实施方式中,抗znt8抗体具有与本文所述的那些不同的一个或多个cdr序列(例如,chothia、增强的chothia或kabatcdr)。在一个实施方式中,抗znt8抗体具有一个或多个cdr序列,其包括氨基酸变化,例如如果cdr的长度为5-7个氨基酸,则置换1、2、3或4个氨基酸,或者如果cdr的长度为8个氨基酸或更多的氨基酸,则置换在cdr序列中的1、2、3、4或5个氨基酸。被置换的氨基酸可以具有相似的电荷、疏水性或立体化学特征。在一些实施方式中,氨基酸置换是保守置换。“保守氨基酸置换”是氨基酸残基被具有带有相似电荷的侧链的氨基酸残基取代的氨基酸置换。具有带有相似电荷的侧链的氨基酸残基的家族已经在本领域中定义。这些家族包括具有以下侧链的氨基酸:碱性侧链(例如,赖氨酸、精氨酸、组氨酸)、酸性侧链(例如,天冬氨酸、谷氨酸)、不带电荷的极性侧链(例如,甘氨酸、天冬酰胺、谷氨酰胺、丝氨酸、苏氨酸、酪氨酸、半胱氨酸)、非极性侧链(例如,丙氨酸、缬氨酸、亮氨酸、异亮氨酸、脯氨酸、苯丙氨酸、甲硫氨酸、色氨酸)、β支化侧链(例如,苏氨酸、缬氨酸、异亮氨酸)和芳香族侧链(例如,酪氨酸、苯丙氨酸、色氨酸、组氨酸)。在其他实施方式中,氨基酸置换是非保守置换。可以筛选含有置换的cdr的抗体或其抗体片段以鉴定目标抗体。
与cdr不同的是,在不会不利地影响抗体的结合特性的情况下,可以对结构框架区(fr)进行更实质性的改变。fr的变化包括但不限于:使非人类衍生的框架人源化或工程改造对抗原接触或对稳定结合位点很重要的某些框架残基,例如,改变恒定区的类别或亚类、改变特定的可能会改变效应子功能(例如fc受体结合)的氨基酸残基(lundetal.,jimmun.,147:26s7-62(1991);morganetal.,immunology,86:319-24(199s)),或改变衍生出恒定区的物种。
iv.产生抗znt8抗体的方法
本公开的抗znt8抗体(或抗体的抗原结合结构域或其功能片段)可以在细菌或真核细胞中产生。为了产生目标多肽,构建了编码该多肽的多核苷酸,将其引入表达载体中,然后在合适的宿主细胞中表达。使用标准分子生物学技术来制备重组表达载体,转染至宿主细胞,选择转化体,培养宿主细胞并回收抗体。
如果抗体要在细菌细胞(例如大肠杆菌)中表达,则表达载体应具有允许载体在细菌细胞中扩增的特征。另外,当使用大肠杆菌(例如jm109、dh5a、hb101或xli-blue)作为宿主时,载体必须具有可以允许在大肠杆菌中有效表达的启动子,例如,lacz启动子(wardetal.,341:544-546(1989))、arab启动子(betteretal.,science,240:1041-1043(1988))或t7启动子。这种载体的例子包括,例如,m13系列载体、puc系列载体、pbr322、pbluescript、pcr-script、pgex-5x-1(pharmacia)、“qiaexpress系统”(qiagen)、pegfp和pet(当使用该表达载体,宿主优选是表达t7rna聚合酶的bl21)。表达载体可包含用于抗体分泌的信号序列。为了在大肠杆菌的周质中产生,可以使用pelb信号序列(leietal.,j.bacteriol.,169:4379(1987))作为用于抗体分泌的信号序列。对于细菌表达,可以使用氯化钙方法或电穿孔方法将表达载体引入细菌细胞。
如果抗体要在动物细胞例如cho、cos、293、293t和nih3t3细胞中表达,则表达载体包括在这些细胞中表达所必需的启动子,例如,sv40启动子(mulliganetal.,nature,277:108(1979))、mmlv-ltr启动子、ef1a启动子(mizushimaetal.,nucleicacidsres.,18:5322(1990))或cmv启动子。除了编码免疫球蛋白或其结构域的核酸序列,重组表达载体还可以携带其他序列,例如调节载体在宿主细胞中复制的序列(例如复制起点)和选择标记基因。选择标记基因有助于选择已引入载体的宿主细胞(参见,例如,美国专利第4,399,216、4,634,665和5,179,017号)。例如,通常,选择标记基因在已引入载体的宿主细胞上赋予对药物(例如g418、潮霉素或甲氨蝶呤)的抗性。具有选择标记的载体的实例包括pmam、pdr2、pbk-rsv、pbk-cmv、poprsv和pop13。
在一个实施方式中,抗体在哺乳动物细胞中产生。用于表达多肽的示例性哺乳动物宿主细胞包括:中国仓鼠卵巢(chinesehamsterovary,cho细胞)(包括dhfr-cho细胞,如在urlaubandchasin(1980)proc.natl.acad.sci.usa77:4216-4220中描述的,与dhfr选择标记一起使用,例如,如在kaufmanandsharp(1982)mol.biol.159:601621中所描述的)、人类胚胎肾293细胞(例如,293、293e,293t)、cos细胞、nih3t3细胞、淋巴细胞细胞系(例如,nso骨髓瘤细胞和sp2细胞,以及来自转基因动物(例如,转基因哺乳动物)的细胞。例如,该细胞是乳腺上皮细胞。
可以从宿主细胞的内部或外部(例如培养基)分离本公开的抗体,并将其纯化为基本上纯的和均质的抗体。通常可以将用于多肽的分离和纯化的方法用于本文所述的抗体的分离和纯化中,并且不限于任何特定方法。可以通过适当地选择和合并(例如,柱层析、过滤、超滤、盐析、溶剂沉淀、溶剂提取、蒸馏、免疫沉淀、sds-聚丙烯酰胺凝胶电泳、等电聚焦、透析和重结晶)来分离和纯化抗体。色谱法包括例如亲和色谱法、离子交换色谱法、疏水色谱法、凝胶过滤、反相色谱法和吸附色谱法(strategiesforproteinpurificationandcharacterization:alaboratorycoursemanual.eddanielr.marshaketal.,coldspringharborlaboratorypress,1996)。可以使用液相色谱法(例如hplc和fplc)进行色层分析。用于亲和色谱法的柱包括蛋白a柱和蛋白g柱。使用蛋白a柱的柱示例包括hyperd、poros和sepharoseff(gehealthcarebiosciences)。本公开还包括使用这些纯化方法进行高度纯化的抗体。
本公开还提供了编码本文公开的抗znt8抗体或其抗原结合分子的核酸分子或成组的核酸分子。在一个实施方式中,本发明包括编码多肽链的核酸分子,该核酸分子包含如本文所述的抗znt8抗体或其抗原结合分子的轻链。在一个实施方式中,本发明包括编码多肽链的核酸分子,该核酸分子包含如本文所述的抗znt8抗体或其抗原结合分子的重链。
还提供了包含这种核酸分子或成组的核酸分子或其补体的载体或成组的载体,以及包含该载体的宿主细胞。
本公开还提供了用于产生本文公开的znt8或其抗原结合分子或嵌合分子的方法,这种方法包括培养本文公开的宿主细胞并从培养基中回收抗体、其抗原结合分子或嵌合分子。
有多种方法可用于重组产生本文公开的znt8抗体或其抗原结合分子或本文公开的嵌合分子。应当理解,由于密码的简并性,多种核酸序列将编码多肽的氨基酸序列。所需的多核苷酸可通过重新固相dna合成或通过对较早制备的多核苷酸进行pcr诱变来产生。
为了重组生产,将编码多肽(例如,本文公开的znt8抗体或其抗原结合分子,或本文公开的任何嵌合分子)的多核苷酸序列插入合适的表达载体,即包含插入的编码序列的转录和翻译所必需的元件的载体,或在rna病毒载体的情况下,包含复制和翻译所必需的元件的载体。
将编码多肽(例如,本文公开的znt8抗体或其抗原结合分子,或本文公开的任何嵌合分子)的核酸以适当的阅读框插入载体。然后将表达载体转染到合适的靶细胞中,该靶细胞将表达该多肽。本领域已知的转染技术包括但不限于磷酸钙沉淀(wigleretal.1978,cell14:725)和电穿孔(neumannetal.1982,emboj.1:841)。可以利用多种宿主表达载体系统在真核细胞中表达本文所述的多肽(例如,本文公开的znt8抗体或其抗原结合分子,或本文公开的任何嵌合分子)。在一个实施方式中,真核细胞是动物细胞,包括哺乳动物细胞(例如,293细胞、perc6、cho、bhk、cos、hela细胞)。当多肽在真核细胞中表达时,编码多肽(例如,本文公开的znt8抗体或其抗原结合分子,或本文公开的任何嵌合分子)的dna也可以编码将允许多肽分泌的信号序列。本领域技术人员将理解,当翻译多肽时,信号序列被细胞切割以形成成熟的嵌合分子。各种信号序列在本领域中是已知的并且是技术人员所熟悉的。或者,在不包括信号序列的情况下,可以通过裂解细胞来回收多肽(例如,本文公开的znt8抗体或其抗原结合分子,或本文公开的任何嵌合分子)。
v.药物组合物
本公开还提供了包含以下中的一种或多种的药物组合物:(i)本文公开的znt8抗体或其抗原结合分子;(ii)编码如本文公开的znt8抗体或抗原结合分子的核酸分子或核酸分子组;或(iii)本文公开的载体或成组的载体,以及药学上可接受的载体。
可将本文所述的抗znt8抗体或其片段配制成用于给药至对象(例如治疗本文所述的病症)的药物组合物。通常,药物组合物包含药学上可接受的载体。如本文所用,“药学上可接受的载体”包括生理上相容的任何和所有溶剂、分散介质、包衣、抗细菌剂和抗真菌剂、等渗剂和吸收延迟剂等。该组合物可以包含药学上可接受的盐,例如,酸加成盐或碱加成盐(参见例如,berge,s.m.,etal.(1977)j.pharm.sci.66:1-19)。
药物制剂是已经确定好的技术,并且进一步在,例如,gennaro(ed.),remington:thescienceandpracticeofpharmacy,20thed.,lippincott,williams&wilkins(2000)(isbn:0683306472);anseletal.,pharmaceuticaldosageformsanddrugdeliverysystems,7thed.,lippincottwilliams&wilkinspublishers(1999)(isbn:0683305727);以及kibbe(ed.),handbookofpharmaceuticalexcipientsamericanpharmaceuticalassociation,3rded.(2000)(isbn:091733096x)中描述。
药物组合物可以是多种形式。这些包括例如,液体、半固体和固体剂型,例如,液体溶液(例如可注射和可输注溶液)、分散剂或悬浮剂、片剂、药丸剂、粉末剂、脂质体和栓剂。优选的形式可取决于给药的预期模式和治疗应用。通常,本文所述试剂的组合物为可注射或可输注溶液的形式。
在一个实施方式中,本文所述的抗体与赋形剂材料一起配制,所述赋形剂材料例如为柠檬酸钠、七水合磷酸氢二钠、磷酸二氢钠、
药物组合物还可包含在配制时减少抗体聚集的试剂。聚集减少剂的实例包括一种或多种选自由以下组成的组的氨基酸:蛋氨酸、精氨酸、赖氨酸、天冬氨酸、甘氨酸和谷氨酸。这些氨基酸可以添加到制剂中至浓度为约0.5mm至约145mm(例如,0.5mm、1mm、2mm、5mm、10mm、25mm、50mm、100mm)。药物组合物还可包含糖(例如,蔗糖、海藻糖、甘露醇、山梨醇或木糖醇)和/或张力调节剂(例如。氯化钠、甘露醇或山梨醇)和/或表面活性剂(例如,聚山梨酯20或聚山梨酯80)。
该组合物可配制成溶液、微乳液、分散体、脂质体或其他适合于以高浓度稳定存储的有序结构。根据需要,可通过将所需量的本文所述的试剂与一种或以上列举的成分的组合掺入适当的溶剂中,然后过滤灭菌来制备无菌可注射的溶液。
通常,分散体是通过将本文所述的试剂掺入含有基本分散介质和以上列举的所需的其他成分的无菌溶媒中制备的。在用于制备无菌可注射溶液的无菌粉末的情况下,优选的制备方法是真空干燥和冷冻干燥,这两种方法从其先前的无菌过滤溶液中得到本文所述的试剂和任何其他期望的成分的粉末。例如,通过使用诸如磷脂的包衣,通过维持所需的粒径(在分散的情况下)和通过使用表面活性剂,可以维持溶液的适当流动性。通过在组合物中包括延迟吸收的试剂,例如单硬脂酸盐和明胶,可以实现可注射组合物的延长吸收。
在某些实施方式中,抗体可以与将保护化合物免于快速释放的运载体(例如控释制剂,包括植入物和微囊递送系统)一起制备。可以使用生物可降解的、生物相容性聚合物,如乙烯醋酸乙烯酯、聚酸酐、聚乙醇酸、胶原蛋白、聚原酸酯和聚乳酸。用于制备这种制剂的许多方法已获得专利或是普遍已知的。参见,例如,sustainedandcontrolledreleasedrugdeliverysystems,j.r.robinson,ed.,marceldekker,inc.,newyork(1978)。
在一个实施方式中,药物制剂包含与药学上可接受的载体一起配制的浓度为约0.005mg/ml至500mg/ml(例如,0.005mg/ml、0.01mg/ml、0.05mg/ml、0.1mg/ml、0.5mg/ml、1mg/ml、5mg/ml、10mg/ml、25mg/ml、30mg/ml、35mg/ml、40mg/ml、45mg/ml、50mg/ml、55mg/ml、60mg/ml、65mg/ml、70mg/ml、75mg/ml、80mg/ml、85mg/ml、90mg/ml、95mg/ml、100mg/ml、125mg/ml、150mg/ml、175mg/ml、200mg/ml、250mg/ml、300mg/ml、350mg/ml、400mg/ml、450mg/ml、500mg/ml)的抗体。在一些实施方式中,抗体在无菌蒸馏水或磷酸盐缓冲盐水中配制。药物制剂的ph可以为5.5至7.5(例如,5.5、5.6、5.7、5.8、5.9、6.0、6.1、6.26.3、6.46.5、6.66.7、6.8、6.97.0、7.1、7.3、7.4、7.5)。
药物组合物可以包含“治疗有效量”的本文所述的试剂。可以基于所给药的试剂的效果或(如果使用多于一种试剂)试剂的组合效果来确定这样的有效量。试剂的治疗有效量还可以根据因素而变化,例如个体的疾病状态、年龄、性别和体重以及化合物在个体中引起期望的反应的能力,例如改善至少一种病症参数或改善病症的至少一种症状。治疗有效量也是这样的量:其中治疗有益效果超过了组合物的任何毒性或有害作用。
a.给药
可以通过多种方法将本公开的抗体或其抗原结合片段或编码本公开的抗体或其抗原结合片段的核酸给药至对象,例如有所需要的对象,例如人或动物对象。对于许多应用,给药途径是以下途径之一:静脉内注射或肠胃外、输注(iv)、皮下注射(sc)、腹膜内(ip)或肌内注射、肿瘤内(it)。也可以使用其他肠胃外给药方式。这种方式的例子包括:动脉内、鞘内、囊内、眶内、心内、真皮内、经气管、皮下、关节内、囊下、蛛网膜下、脊柱内、以及硬膜外和胸骨内注射。
在一个实施方式中,本发明抗体的给药途径是肠胃外的。本文所用的术语“肠胃外的”包括静脉内、动脉内、腹膜内、肌内、皮下、直肠或阴道给药。肠胃外给药的静脉内形式是优选的。尽管所有这些给药形式都明确地认为是在本发明的范围内,但是给药形式将是注射用溶液,特别是用于静脉内或动脉内注射或滴注的溶液。通常,合适的注射用药物组合物可包含缓冲液(例如,乙酸盐、磷酸盐或柠檬酸盐缓冲液)、表面活性剂(例如,聚山梨酯)、可选地稳定剂(例如人白蛋白)等。然而,在其他的与本文的教导相兼容的方法中,可以将多肽直接递送至不良细胞群体的位点,从而增加患病组织对治疗剂的暴露。
用于肠胃外给药的制剂包括无菌水溶液或非水溶液、悬浮剂和乳剂。非水溶剂的实例是丙二醇、聚乙二醇、植物油(例如橄榄油)和可注射有机酯(例如油酸乙酯)。水性运载体包括水、醇/水溶液、乳剂或悬浮剂,包括盐水和缓冲介质。
药学上可接受的载体包括但不限于0.01m-0.1m以及优选为0.05m的磷酸盐缓冲液或0.8%盐水。其他常见的肠胃外溶媒包括磷酸钠溶液、林格氏右旋糖(ringer’sdextrose)、右旋糖和氯化钠、乳酸林格氏液(lactatedringer’s)或不挥发性油。静脉内溶媒包括液体和营养补充剂、电解质补充剂(例如基于林格氏右旋糖的补充剂)等。也可以存在防腐剂和其他添加剂,例如,抗微生物剂、抗氧化剂、螯合剂和惰性气体等。
更特别地,适用于注射使用的药物组合物包括无菌水溶液(在水溶性的情况下)或分散体以及用于临时制备无菌可注射的溶液或分散体的无菌粉末。在这种情况下,该组合物必须是无菌的,并且应该到易注射性存在的程度的流动。它在制造和储存条件下应该是稳定的,并且将优选地被保存,抵抗诸如细菌和真菌的微生物的污染作用。载体可以是溶剂或分散介质,其包含例如,水、乙醇、多元醇(例如,甘油、丙二醇和液体聚乙二醇等)及其合适的混合物。例如,通过使用诸如磷脂的包衣,通过维持所需的粒径(在分散的情况下)和通过使用表面活性剂,可以维持适当流动性。
可以通过各种抗细菌剂和抗真菌剂来实现防止微生物作用,例如对羟基苯甲酸酯、氯代丁醇、苯酚、抗坏血酸、硫柳汞等。在许多情况下,优选的将是在组合物中包含等渗剂,例如,糖、多元醇(例如甘露醇、山梨醇)或氯化钠。通过在组合物中包括延迟吸收的试剂,例如单硬脂酸铝和明胶,可以带来可注射组合物的延长吸收。
在任何情况下,根据需要,可通过将所需量的活性化合物(例如,多肽本身或与其他活性剂组合)与一种或以上列举的成分的组合掺入适当的溶剂中,然后过滤灭菌来制备无菌可注射的溶液。
通常,分散剂是通过将活性化合物掺入含有基本分散介质和上述所列其它所需成分的无菌溶媒中制备的。在用于制备无菌可注射的溶液的无菌粉末的情况下,优选的制备方法是真空干燥和冷冻干燥,这两种方法从其先前的无菌过滤溶液中得到活性成分和任何其他期望的成分的粉末。根据本领域已知的方法,对注射用制剂进行加工,填充到诸如安瓿瓶、袋、瓶、注射器或小瓶的容器中,并在无菌条件下密封。此外,可以包装这些制剂并以试剂盒的形式出售。这种制品将优选具有标签或包装说明书,其指示相关的组合物可用于治疗患有或易患凝血障碍的对象。
用于治疗病症的本公开的组合物的有效剂量取决于许多不同因素而变化,所述因素包括:给药方式、靶位点、患者的生理状态、患者是人还是动物、所施用的其他药物以及治疗是预防性的还是治疗性的。通常,患者是人类,但是也可以治疗非人类哺乳动物,包括转基因哺乳动物。可以使用本领域技术人员已知的常规方法来滴定治疗剂量以优化安全性和功效。
抗znt8抗体或其片段的给药途径和/或方式也可以针对个体情况进行调整,例如通过监测对象。
抗体或其片段可以以固定剂量或以mg/kg剂量给药。还可以选择剂量以减少或避免产生针对抗znt8抗体或其片段的抗体。调整剂量方案以提供所需的反应,例如治疗反应或组合治疗效果。通常,可以使用一定剂量的抗体或其片段(和可选地第二种试剂),以向对象提供生物可利用量的试剂。例如,可以给药的剂量范围为0.1mg/kg-100mg/kg、0.5mg/kg-100mg/kg、1mg/kg-100mg/kg、0.5mg/kg-20mg/kg、0.1mg/kg-10mg/kg或1mg/kg-10mg/kg。也可以使用其他剂量。在某些实施方式中,将约1mg/kg至约30mg/kg的剂量的抗体或其片段给药至需要用抗体或其片段进行治疗的对象。在一些实施方式中,将1mg/kg、2mg/kg、4mg/kg、5mg/kg、7mg/kg10mg/kg、12mg/kg、15mg/kg、20mg/kg、25mg/kg、28mg/kg、30mg/kg、35mg/kg、40mg/kg或50mg/kg的剂量的抗体或其片段给药至需要用抗znt8抗体或其片段进行治疗的对象。在一个具体的实施方式中,以1mg/kg至3mg/kg的剂量皮下给药抗体或其片段。在另一个实施方式中,以4mg/kg至30mg/kg的剂量静脉内给药抗体或其片段。
组合物可以包含约1mg/ml至100mg/ml、或约10mg/ml至100mg/ml、或约50mg/ml至250mg/ml、或约100mg/ml至150mg/ml、或约100至250mg/ml的抗体或其片段。
如本文所用,单位剂型或“固定剂量”是指适合作为待治疗对象的单位剂量的物理上离散的单位;每个单元包含预定量的抗体或其片段,所述抗体或其片段经计算与所需的药物运载体结合并可选与其他试剂结合以产生所需的治疗效果。可以给予单次剂量或多次剂量。可选地或另外地,可以通过连续输注来给药抗体或其片段。
抗体或其片段的剂量可以,例如,在足以涵盖:至少2剂、3剂、5剂、10剂或更多剂,例如每天一次或两次,或每周约一到四次,或优选地每周、两周(每两周)、每三周、每月,例如,约1至12周,优选地2至8周,更优选地约3至7周,甚至更优选约4周、5周或6周的时间段(一个疗程)中以周期性间隔给药。可能影响有效地治疗对象所需的剂量和时间的因素包括,例如,疾病或病症的阶段或严重程度、制剂、递送途径、先前的治疗、对象的总体健康和/或年龄,以及存在其他疾病。此外,用治疗有效量的化合物对对象进行治疗可以包括单一治疗,或者优选地,可以包括一系列治疗。
如果对象有发生本文所述病症的风险,则可以在病症全面发作之前给药抗体或其片段,例如,作为预防措施。这种预防性治疗的持续时间可以是抗体或其片段的单剂量,或者该治疗可以继续(例如,多剂量)。例如,可以用该抗体或其片段对处于患该病症的风险中或易患有该病症的对象治疗数天、数周、数月甚至数年,以防止该病症的发生或爆发。
在某些实施方式中,以约1mg/ml至约500mg/ml(例如,1mg/ml、2mg/ml、3mg/ml、4mg/ml、5mg/ml、10mg/ml、15mg/ml、20mg/ml、25mg/ml、30mg/ml、35mg/ml、40mg/ml、45mg/ml、50mg/ml、55mg/ml、60mg/ml、65mg/ml、70mg/ml、75mg/ml、80mg/ml、85mg/ml、90mg/ml、95mg/ml、100mg/ml、125mg/ml、150mg/ml、175mg/ml、200mg/ml、225mg/ml、250mg/ml、275mg/ml、300mg/ml、325mg/ml、350mg/ml、400mg/ml、450mg/ml)的浓度皮下给药抗体或其片段。在一个实施方式中,以50mg/ml的浓度皮下给药抗znt8抗体或其片段。在另一个实施方式中,以约1mg/ml至约500mg/ml的浓度静脉内给药抗体或其片段。在一个实施方式中,以50mg/ml的浓度静脉内给药抗体或其片段。
在上述范围内的中间剂量也意图在本发明的范围内。可以每天、隔天、每周或根据通过经验分析确定的任何其他时间表向对象给药这样的剂量。一种示例性的治疗方法需要在例如至少六个月的延长时间内以多种剂量给药。在一些方法中,可以同时给药两种或更多种多肽,在这种情况下,所给药的每种多肽的剂量都在指定的范围内。
本发明的多肽可以在多个时机给药。单一剂量之间的间隔可以是每天、每周、每月或每年。如通过测量患者中修饰的多肽或抗原的血液水平所指示的,间隔也可以是不规律的。或者,多肽可以作为缓释制剂给药,在这种情况下,需要较少的给药频率。剂量和频率取决于患者中多肽的半衰期。
给药的剂量和频率可以根据治疗是预防性的还是治疗性的而变化。在预防性应用中,将含有本发明的多肽或其混合物的组合物给药至尚未处于疾病状态的患者,以增强患者的抵抗力或使疾病影响最小化。将这种量定义为“预防有效剂量”。在很长一段时间内,以相对不频繁的间隔给药相对低的剂量。一些患者终生持续接受治疗。
vi.用于治疗的装置和试剂盒
可以在试剂盒中提供抗znt8抗体或其片段。在一个实施方式中,试剂盒包括:(a)容器,该容器包含组合物,该组合物包含如本文所述的抗znt8抗体或其片段;和可选地(b)信息材料。信息材料可以是与本文描述的方法和/或试剂用于治疗益处的用途有关的描述性、指导性、行销性或其他材料。
在一个实施方式中,试剂盒还包括用于治疗本文所述的病症的第二试剂。例如,试剂盒包括:第一容器,该第一容器包含含有抗znt8抗体或其片段的组合物;和第二容器,该第二容器包含第二试剂。
试剂盒的信息材料不限于其形式。在一个实施方式中,信息材料可以包括有关化合物的生产、化合物的分子量、浓度、有效期、批次或生产地点信息等的信息。在一个实施方式中,信息材料涉及,例如以合适的剂量、剂型或给药方式(例如本文所述的剂量、剂型或给药方式)给药抗znt8抗体或其片段的方法,以治疗已患有本文所述疾病或处于患有本文所述疾病的风险中的对象。可以以多种格式提供信息,包括印刷文本、计算机可读材料、视频记录或音频记录,或者提供例如互联网上的实质性材料的链接或地址的信息。
除了抗znt8抗体或其片段外,试剂盒中的组合物还可以包含其他成分,例如溶剂或缓冲液、稳定剂或防腐剂。抗znt8抗体或其片段可以以任何形式提供,例如液体、干燥或冻干形式,优选地基本上是纯的和/或无菌的。当试剂以液体溶液提供时,该液体溶液优选为水溶液。在某些实施方式中,抗znt8抗体或其片段在液体溶液中的浓度为约25mg/ml至约250mg/ml(例如,40mg/ml、50mg/ml、60mg/ml、75mg/ml、85mg/ml、100mg/ml、125mg/ml、150mg/ml和200mg/ml)。当抗znt8抗体或其片段作为冻干产物提供时,抗znt8抗体或其片段为约75mg/小瓶至约200mg/小瓶(例如,100mg/小瓶、108.5mg/小瓶、125mg/小瓶、150mg/小瓶)。冻干粉末通常通过添加合适的溶剂来复原。试剂盒中可以可选地提供溶剂,例如无菌水或缓冲液(例如,pbs)。
试剂盒可包括一个或多个用于包含试剂的一种或多种组合物的容器。在一些实施方式中,试剂盒包含用于组合物和信息材料的分开的容器、分隔物或隔室。例如,该组合物可以包含在瓶、小瓶或注射器中,而信息材料可以包含在塑料套或小包中。在其他实施方式中,试剂盒的分开的元件包含在单个不分开的容器中。例如,该组合物包含在以标签形式附有信息材料的瓶、小瓶或注射器中。在一些实施方式中,试剂盒包括多个(例如,一包)单独的容器,每个容器包含试剂的一种或多种单位剂型(例如,本文所述的剂型)。所述容器可以包括组合单位剂量,例如包含抗znt8抗体或其片段和第二试剂的单位,例如以期望的比例。例如,该试剂盒包括多个注射器、安瓿、箔袋、泡罩包装或医疗器械,例如,每个包含单个组合单位剂量。试剂盒的容器可以是气密的、防水的(例如,对水分或蒸发的变化不渗透的)和/或不透光的。
试剂盒可选地包括适用于给药组合物的装置,例如,注射器或其他合适的递送装置。该装置可以预先装有一种或两种试剂,也可以是空的,但适合装载。
无需进一步阐述,相信使用前面的描述,本领域技术人员可以最大程度地利用本发明。以下实施例仅是说明性的,并且不以任何方式限制本公开的其余内容。
实施例
提出以下实施例以向本领域普通技术人员提供如何制备和评价本文所述和要求保护的化合物、组合物、制品、装置和/或方法的完整公开内容和描述,并且该实施例仅是示例性的,而并非旨在对发明人认为是其发明的范围进行限制。已经做出努力以确保关于数字(例如,数量、温度等)的准确性,但是本文应当考虑一些误差和偏差。除非另有说明,否则份数是重量份,温度是摄氏度或处于环境温度,并且压力为大气压或接近大气压。反应条件有多种变化和组合,例如组分浓度、所需溶剂、溶剂混合物、温度、压力和其他反应范围和条件,这些可用于优化从所述方法获得的产物纯度和产率。将仅需要合理且常规的实验来优化这种工艺条件。
如下所述,本发明人通过针对天然形式的纯化的人类znt8进行多层筛选鉴定了两个mab(mab20、mab39)。详细的mab表征显示出独特的结合特性和特异性,其具有稳定化znt8结构折叠的共同能力,因此通过减慢锌的转换率来抑制其转运活性。这些发现证明了mab作为人类znt8的变构抑制剂的实用性。本发明人还鉴定了znt8单体与两种不同的fab的稳定的三元复合物。znt8介导的两个mab的邻近关系允许基于一对供体和与mab结合的共价连接的受体荧光团之间的fret信号进行znt8定量。在该测定中,我们使用了笼状镧系元素供体(铽穴状化合物(terbiumcryptate)),其特征是荧光发射半衰期长。在短暂的背景荧光衰减之后,可以使用延迟荧光测量来获得衍生自三元复合物的fret信号。因此,该测定可以以均相形式进行,而无需将结合复合物与未结合的mab分开(30)。这种简单的添加和读取htrf测定(add-and-readhtrfassay)突显了mab作为znt8特异性生物传感器的另一种实用性。
材料和方法
生成杂交瘤。通过每周一次腹膜内注射在复原的蛋白脂质体中的人类znt8抗原(r325变体)对四对的雄性/雌性纯合slc30a8-/-小鼠进行免疫。对小鼠进行的所有实验均得到约翰霍普金斯大学医学院的机构动物护理和使用委员会(institutionalanimalcareandusecommitteeofjohnshopkinsuniversityschoolofmedicine)的批准。先前已经描述了通过crispr/cas9znt8敲除进行血清抗体滴定和抗znt8特异性的验证(36)。最终免疫加强后,分离脾脏。通过1分钟内将1ml的50%peg1500(w/v;roche)逐滴加入细胞混合物中,将脾细胞与永生化的骨髓瘤细胞系sp2/0-ag14(crl-1581;atcc)融合,脾细胞与骨髓瘤的比例为4:1(37)。使细胞在温和混合下再停留在peg溶液中一分钟,然后在4-5分钟内逐步添加20ml无血清dmem。将细胞剥离,然后重悬于15mldmem中,该dmem中添加了10%fbs以及杂交瘤融合和克隆补充剂(hybridomafusionandcloningsupplement,hfcs)和次黄嘌呤、氨基蝶呤和胸苷(hypoxanthine,aminopterinandthymidine,hat)。融合八天后,去除死的sp2/0细胞,并将杂交瘤细胞以每孔~5个细胞的密度接种在96孔板中,并保持在hat选择下。
筛选杂交瘤。开发了多重elisa,以评估杂交瘤上清液。第一次筛选是通过被动包被(200ngznt8/孔)使用在复原的蛋白脂质体中的天然人类znt8抗原。用5%bsa的测定缓冲液(20mmhepes、100mmnacl,ph7.0)预封闭包被的板,然后向每个孔中加入20μl杂交瘤上清液,在rt孵育2hr,然后用测定缓冲液加1%bsa洗涤3次。通过1:3000稀释度的hrp缀合的山羊抗小鼠二抗(invitrogen,目录号62-6500)的测定缓冲液来检测结合的抗体,然后进行abts比色反应(lifetechnologies),并在酶标仪(moleculardevice)上进行415nm信号读取。第二次筛选通过与ni-nta板(100ngctd/孔)亲和结合,使用天然ctd-his单体。elisa程序与上述相同。第三次筛选在洗剂胶束中使用了天然znt8。抗原结合在两层抗体之间:捕获抗体是固定在蛋白a/g板(800ng/孔)上的mab20。仅结合至非重叠表位的检测抗体给出了阳性elisa读数。使用纯化的msa和市售ntda(proteintech,目录号16169-i-ap)和ctda(amgen)作为阴性和阳性对照,以校准elisa动态范围和灵敏度。
验证mab。通过流式细胞仪单细胞分选建立单克隆杂交瘤细胞系,以确保克隆纯度。克隆扩增后,分泌的mab通过蛋白a/g亲和层析(thermoscientific,目录号20423)进行纯化,分亚型(thermoscientific,目录号37503),然后通过木瓜蛋白酶消化(thermoscientific,目录号44985)制备fab片段。对纯化的mab或fab进行了以下测试:(i)在尺寸排阻hplc上与纯化的znt8或znt8-gfp形成结合复合物;(ii)对hek293细胞(不含znt8表达)进行免疫荧光染色,然后进行共聚焦显微镜成像;(iii)对大细胞群进行免疫荧光染色,随后进行流式细胞仪定量;以及(iv)通过em负染色和单颗粒分析形成fab-znt8复合物。
荧光尺寸排阻hplc。将纯化的mab20/fab20或mab39与alexafluor488或alexafluor555羧酸琥珀酰亚胺酯在pbs缓冲液中以1:20摩尔比在rt下混合60min,然后通过100mmtris-hcl淬灭终止标记反应。对荧光标记的mab或fab进行hplc纯化,以除去痕量的游离荧光染料,将其浓缩至1mg/ml,以便在4℃下在pbs缓冲液中储存。将纯化的人类znt8的洗剂胶束与mab20/fab20或mab30孵育2小时,以258000×g离心30min,以使交联的znt8聚集体沉淀,然后注入用缓冲液(添加0.02%ddm)预先平衡的tskgelg3000swxl尺寸排阻hplc柱(tosohbiosciences)。或者,通过gfp-荧光尺寸-hplc(gfp-fluorescencesizing-hplc)来纯化稳定表达人类znt8的ins-1e细胞的洗剂粗提物中的znt8-gfp。接下来,将znt8-gfp的峰级分与mab20或mab39在4℃下孵育3hr,离心去除交联的聚集体,然后如上所述注入tsk柱中。使用具有以下激发/发射对的多通道荧光检测器来监测洗脱图:alexafluor488、alexafluor555和gfp荧光分别用480/520nm、485/555nm、480/520nm。单独的znt8峰由与荧光检测器相连的uv检测器监测。
负染色电子显微镜。如先前所述对人类znt8进行纯化(20)。fab20或fab20+fab39以10:1的摩尔比添加到znt8中。通过尺寸排阻hplc再次纯化结合复合物,并将其浓缩至1mg/ml-2mg/ml。将蛋白质样品稀释至20μg/ml,将3μl稀释样品的等份试样涂在覆盖有连续薄碳膜的辉光放电em栅格上,并用2%乙酸铀酰水溶液染色1.5min。将栅格加载到在120kv高压下运行的tecnaispirit电子显微镜上。使用gatanoriusccd相机以低剂量模式
免疫荧光染色和细胞成像。如前所述,使稳定表达人类znt8变体的flp-in、t-rex-hek293细胞在盖玻片上生长并诱导,汇合度为50%(20)。将细胞固定(r&dsystems,目录号fc004)、透化(r&dsystems,目录号fc005)。用来自1mg/mlmab储备液的1:3000稀释度的mab20或mab39对hek293细胞进行染色,而用来自同一批mab20和mab39以1:1000稀释度对endoc-βh细胞进行染色。按照相同步骤,将无znt8表达的亲本flp-in、t-rex-hek293细胞用作阴性对照。如上所述(20),将与alexafluor-594(thermoscientific,目录号a11005,稀释度1:1000)和dapi缀合的山羊抗小鼠二抗在带63x油镜的zeisslsm700倒置共聚焦显微镜上进行荧光成像。
流式细胞仪分析。如前所述(43),将没有znt8过表达的hek293细胞固定、透化并以1x106/ml的密度重新悬浮于pbs加5%bsa中。将100μl细胞悬液的等分试样在rt下用3倍系列稀释的mab20或mab39染色2小时,然后用5%bsa与以1:1000稀释的alexafluor647-缀合的驴抗小鼠二抗(thermofisherscientific,目录号a31571)进行染色。立即通过流式细胞仪分析标记的细胞。在前向散射和侧向散射中进行门控的细胞显示出单独的群,其以~3000个细胞计数事件的直方图表示。使用流式细胞仪分析软件summit4.3(beckmancoulter)获得平均免疫荧光强度和标准误差。如上所述对作为znt8-ko替代物的endoc-βh细胞和hek293细胞进行固定和透化,并用1:1000稀释度的mab20和mab39进行染色,然后如上所述进行第二抗体染色。使用相同的流式细胞仪方案来分析标记的endoc-βh细胞和hek293细胞。
免疫印迹。对过表达znt8的endoc-βh细胞或hek293细胞的sds溶解的细胞裂解液进行sds-page并用1:3000的mab20或mab39进行探测,然后用1:3000抗小鼠hrp缀合的二抗(gehealthcare,目录号na931v)进行探测。对凝胶复制品进行考马斯染色,显示每个泳道上样的蛋白质相等(100μg)。为了比较mab20或mab39与不同znt同源物的反应性,我们使用了慢病毒介导的过表达znt1、znt2、znt3、znt4、znt7、znt8、znt10(每个都在c末端标记有flag标签(origene,目录号ly500001、lc412033、lc423751、lc401170、ly415673、lc428544、lc406320和lc412886))的商品化hek293裂解物。将大约等量的总蛋白(5μg)上样至每个泳道,然后如上所述用mab20、mab39或抗flag抗体(origene,目录号ta50011-100)进行探测。znt同源物的表达水平随最丰富表达的znt1而变化。
停流动力学。如前所述(20),对人类znt8(r325变体)进行纯化并将其复原为蛋白脂质比为1:50的具有确定的脂质组成dopc:dopg:dope(2:1:1)的蛋白脂质体(wt/wt)。将预先形成的蛋白脂质体与200μm的fluozin-3(不含100μgfab)在0.2ml分析缓冲液中混合,进行三个循环的冻融,然后进行10秒超声处理以完成染料和fab的封装。通过用3×25ml分析缓冲液洗涤囊来去除囊外fluozin-3,然后添加额外的100μgfab以确保fab结合饱和。估计的fab与znt8的摩尔比为2.8:1。如前所述(20),通过在sfm-3000停流装置(bio-logic)上快速混合蛋白脂质体和测定缓冲液触发znt8介导的向蛋白脂质体的锌转运。根据maxchelator(maxchelator.stanford.edu),将数量越来越多的znso4添加到1mm柠檬酸钠的测定缓冲液中,该缓冲液缓冲6μm到116μm的游离锌浓度。所有动力学迹线是9次连续记录的累积平均值。收集脂质体迹线作为基线,并从蛋白脂质体迹线中减去基线以得到净荧光变化δf。通过将δf标准化为包含2mmznso4和2%β-og的测定缓冲液引起的最大脂质体反应,从而获得δf/δfmax。通过初始荧光上升的准线性相中数据点(t<10s)的线性回归获得锌流入的初始速率。通过对v=vmaxm/(m+km)限定的双曲线的初始转运速率的最小二乘拟合来分析浓度依赖性数据,其中m表示游离锌离子浓度,vmax是转运速率接近准稳态时的最大转运速率,而km是米氏常数(michaelis-mentenconstant)。实验数据的拟合使用数据分析软件sigmaplot(spssinc.)进行。
htrf测定。按照制造商的规程(cisbiobioassays,bedford,ma)将纯化的mab20和mab39分别用铽穴状化合物和d2标记。缀合物中分子和染料之间的分子比对于mab20为xx,对于mab39为xx。标记的mab在pbs(?)中进行hplc纯化,浓缩至1mg/ml,储存在4℃。收获悬浮培养中瞬时表达人类znt8、人类znt3或小鼠znt8的293-f细胞。通过将细胞沉淀在20mmhepes、100mmnacl(ph7.0)和蛋白酶混合物中用1%ddm增溶,并在16000g下离心15分钟以除去碎片,来制备洗剂粗提物。收集上清液用于znt8检测。沉淀用2%ddm的pbs溶液溶解,粗提物中清除了碎片,用于znt8检测。通过添加1:1比例的标记的mab20、mab39以及适当稀释度的293-f细胞或endoc-βh细胞的洗剂提取物至总体积为20μl,在384孔板(greinerbio-one,目录号784075)上进行htrf分析。在室温下与mab结合2小时后,使用酶标仪(moleculardevices)测量荧光发射,该酶标仪配备有用于在340nm下进行40μs延迟的荧光团激发的闪光灯,用于铽穴状化合物荧光读数的620nm滤光片,以及用于d2荧光检测的665nm滤光片。根据665nm(d2)和620nm(铽穴状化合物)测量的荧光的比率,数据下降,使用620nm的信号作为内标。比例测量针对孔间差异和来自结合混合物中潜在干扰成分的信号猝灭进行了校正。测量背景信号并用于计算相对znt8读数δf,以进行批间比较:δf=(比率样品-比率背景)/比率背景.基于293-f细胞瞬时表达的znt8产量来估算测定中的实际znt8浓度,并通过线性回归(r2=0.99)将其与htrf读数相关联以得出标准曲线。lod值和loq值是通过在标准曲线上测量数据点的内插获得的。如前所述(44),通过比较从在单个实验中的endoc-βh、hek293和ins-1e细胞中收集的洗剂粗提物的htrf读数的多次重复,计算出htrf测定的z'因子值。
结果
鉴定不同的抗znt8mab。人类znt8(b型)是两个35.1kda启动子的同型二聚体,每个启动子由23.8kda跨膜结构域(transmembranedomain,tmd)后接着11.3kdactd组成。下文将znt8同型二聚体称为znt8。用蛋白脂质体中的纯化的人类znt8免疫znt8-ko小鼠产生了血清抗体,与分别针对n末端结构域和c末端结构域(ntd和ctd)的线性肽序列的两种市售抗体的效价(31)相比,该血清抗体展现出抗体效价提高了~100倍。分离免疫小鼠的脾细胞,并将其与骨髓瘤细胞融合以建立永生化的杂交瘤。通过酶联免疫吸附测定(enzymelinkedimmunosorbentassay,elisa)进行的初步筛选鉴定了抗znt8抗体,该抗znt8抗体对蛋白脂质体中的全长znt8抗原和单体ctd抗原(aa275-369)表现出不同的反应性。对这些杂交瘤细胞进行单细胞分选和克隆扩增,然后对两种分泌的单克隆抗体(mab20和mab39)进行纯化用于进一步表征。使用市售抗ntd抗体和抗ctd抗体(ntda和ctda)或来自未免疫znt8-ko小鼠的纯化的小鼠血清抗体(mouseserumantibody,msa)作为阳性或阴性对照。mab20、mab39、ctda和ntda均产生针对znt8蛋白脂质体抗原的阳性读数(图1a)。相反,只有mab20和阳性对照ctda识别出单体ctd抗原。mab39和阴性对照ntda在用ctd探测的elisa中给出阴性结果(图1b),这表明mab20识别出的表位与mab39识别出的不同。
稳定的抗体znt8复合物。通过分析尺寸排阻hplc检查稳定的mab-znt8复合物或fab-znt8复合物的形成。超过znt8的mab倾向于形成交联的蛋白质聚集体。通过调节mab:znt8的化学计量,我们检测到如图2中的1-8所标记的一系列八个不同的单分散峰。mab20和fab20用绿色荧光团标记,而mab39用红色荧光团标记。在荧光检测器的分开的通道中对与znt8复合的mab20/fab20以及mab39进行监测(图2)。计算每个峰的表观分子量(mw),并将预测的与mw一致的复合物组成汇总在表1中。对于二元结合,fab20形成单个(fab20)2-znt8复合物(峰3),mab20主要形成mab20-znt8复合物(峰2)。相比之下,mab39主要形成(mab39)2-znt8复合物(峰1)和与峰2对齐的mab39-znt8次要复合物。对于znt8、mab20/fab20和mab39之间的三元结合,绿色和红色通道均记录了相似的(mab39)2-znt8-mab20复合物(峰5、峰7)和mab39-znt8-(fab20)2复合物(峰6、峰8),表明mab20/fab20和mab39独立结合到单个znt8(峰4)。
表1.二元复合物峰和三元复合物峰的汇总
单粒子电子显微镜(em)分析。为了进一步表征抗体结合,我们对二元(fab20)2-znt8和三元(fab20)2-znt8-(fab39)2复合物进行了纯化,并通过负染色em直接观察fab结合。二元复合物的原始图像(图3a)和相应的2d无参考平均值显示了一个完整复合物中的两个fab,每个fab均具有清晰可见的在可变区和恒定区之间的特征性fab凹槽(图3b)。因此,将2重对称应用于单粒子3d重建,从而产生了低分辨率(~3nm)的3d图。该3d包迹拟合有两个fab分子(pdbid1m71)和一个znt8同源物yiip(pdbid3h90)的晶体结构。znt8包迹大于晶体结构,这可能是由于跨膜区周围的洗剂胶束(图3e)所致。对于三元复合物,原始图像显示了多个fab分子与单个znt8二聚体的结合(图3c)。代表性2d平均值还显示了多个fab结合(图3d)。3d重建显示了ctd表面上的三个离散突起(图3f)。与在znt8-(fab20)2中观察到的一样,两个外部突起较小且处于相似但略低的位置,因此归属于一对fab20分子(图3f)。中间突起较大并且可以由跨越znt8二聚体界面的二重轴的两个fab分子装配,因此归属于两个fab39分子(图3f)。尽管图谱分辨率不足以区分三元复合物中的不同类型的fab,但是3d模型的同源物结构对接表明,mab39结合需要ctd二聚化。这一发现得到了ctd-elisa中针对单体ctd抗原的阴性mab39读数的支持(图1b)。通过比较,每个fab20与3d模型中的单独的ctd单体结合(图3e-图3f),这与ctd-elisa中的阳性mab20读数一致。
折叠依赖性。为了比较mab与天然znt8和变性znt8结合的差异,我们使用2%sds在ni-nta板上解折叠固定化的znt8-his,然后将变性elisa读数与未变性的复制品进行比较。mab20的天然读数是变性读数的约两倍(图4a),这表明与解折叠的znt8相比,对折叠构象具有中等选择性。作为对照,使ctda不加选择地与天然znt8和变性znt8进行反应。出乎意料的是,尽管mab39与二聚体界面结合且抗ctd阴性(图1b),但对于固定在ni-nta板上的变性znt8而言,却是elisa阳性(图4a)。一种可能性是mab39表位靠近c末端,其中来自znt8二聚体的一对his标签可能通过ni-nta结合稳定了局部二聚体相互作用。为了验证这一假设,我们在c末端添加了一个更大的绿色荧光蛋白(greenfluorescenceprotein,gfp),并检查了其与mab结合的潜在空间位阻。尺寸排阻hplc显示出,如对mab20-znt8-gfp二元复合物所预测的,mab20使znt8-gfp峰向左移动(图4b)。相反,c末端gfp完全阻断了mab39-znt8-gfp二元复合物的形成。gfp与mab39结合之间的空间冲突将mab39结合位点定位在c末端。该结果进一步证实了mab39对接至跨越二聚体界面的远端ctd表面(图3f)。
增强znt8的稳定性。mab与构象表位的结合有望使蛋白质折叠稳定。解折叠的znt8可以通过超速离心沉淀为蛋白质聚集体。为了评估抗体结合对znt8稳定性的影响,我们在具有fab或不具有fab的条件下孵育了洗剂增溶的znt8,然后将蛋白质样品在37℃下暴露于热变性,并在超速离心后监测上清液中折叠的znt8。定量免疫印迹显示出znt8不稳定,并且在热暴露一小时后开始沉淀(图4c)。fab20或fab39结合将znt8沉淀的发生延迟到2小时,并且通过同时结合fab20和fab39来进一步增强这种保护作用。在3小时时,仅12%的初始znt8保留在上清液中,而超过70%的(fab20)2-znt8-(fab39)2仍然是可溶的。
抑制锌转运。fab20和fab39的稳定化作用表明fab结合将znt8保持在更稳定的构象。我们进一步检查了构象限制对znt8介导的锌转运动力学的影响。将纯化的人类znt8复原于封装有锌荧光指示剂fluozin-3的蛋白脂质体中,然后在停流装置上将其暴露于一系列囊外锌浓度(图5a)。将固定的1mm柠檬酸盐添加到含增加的锌浓度的测定缓冲液中,得到的游离锌浓度为1.6μm至116μm。这样确保了游离锌的持续供应以满足稳态条件。基于密封的且洗剂穿孔的蛋白脂质体之间mab免疫荧光染色的差异,估计复原的znt8的跨膜方向暴露了50%-75%的ctd在蛋白脂质体的囊外侧。fab20或fab39通过短暂的超声处理被加载到蛋白脂质体上,以平衡囊内fab和囊外fab。重新溶解的、fab处理的蛋白脂质体的尺寸hplc证实了fab结合的完全占有。蛋白脂质体与锌缓冲液的快速混合触发了在最初的10秒钟内fluozin-3荧光的准线性增加(图5a)。计算荧光上升的速率,并对其进行米氏方程拟合(图5b)。表2中总结了稳态动力学的vmax值和km值,显示出与未结合的蛋白脂质体对照相比,fab20或fab39使vmax适度降低(30%-41%)。个体fab结合的抑制作用是累加的,当znt8与fab20和fab39双重结合时,vmax降低57%。fab抑制的作用大小类似于低风险的w325变体的作用大小,为高风险的r325变体的~50%(20)。没有观察到km值的显著差异,表明fab通过降低其转运速率抑制了znt8。
表2.稳态锌转运动力学参数
结合亲和力。我们检查了mab与稳定表达人类znt8的hek293细胞的结合。使用mab20和mab39进行的免疫荧光染色的共聚焦显微镜成像显示出强的细胞内信号(图6a)。在不具有znt8过表达的亲代hek293细胞中没有检测到mab染色。接下来,我们使用流式细胞仪将成像分析扩展至大细胞群体,以分析连续稀释的标记有mab20或mab39的稳定转染细胞或亲代细胞。对于每个mab浓度,记录约3000个细胞计数事件以形成荧光强度直方图(图6b)。mab稀释逐渐将峰向左移动,从而产生了对浓度依赖性mab标记的定量测度。mab20和mab39显示出相似的结合曲线,亲和力分别为0.8nm±0.1nm和0.7nm±0.1nm(图6c)。
结合特异性。我们首先通过比较mab与人类znt8和小鼠znt8的结合来检查跨物种特异性,二者均在hek293细胞中过表达,然后用洗剂(ddm)溶解,并通过c末端多组氨酸标签固定在ni-nta板上。对mab20进行的mab滴定显示出人与小鼠之间的强烈差异,其使小鼠结合曲线向右移动,表观结合亲和力的差异为566.8倍(2.5nm±0.2nm与1422.0nm±72.6nm)(图7a)。相比之下,mab39和人类znt8的结合与其和小鼠znt8的结合相似,仅有微不足道的2.0倍差异(17.6nm±1.7nm与35.8nm±3.0nm)(图7a)。这些结果表明,mab20对人类znt8具有特异性,而mab39识别人类znt8和小鼠znt8。值得注意的是,与通过流式细胞仪确定的亲和力相比,znt8固定化的表位掩盖作用减弱了两种mab的表观结合亲和力,并且这种减弱作用对于mab39更为明显,这可能是由于mab39表位更接近c末端his标签。接下来,我们比较了mab对变性znt8的免疫反应性。hek293转染子的免疫印迹裂解物显示,sds-page上的znt8变性显著降低了mab39对人类znt8和小鼠znt8的免疫反应性。另一方面,mab20仅检测人类znt8,但不检测小鼠znt8(图7b)。在重复的印迹上通过抗his标签抗体检测到相似的水平的人类znt8和小鼠znt8,以证实相似的蛋白质负载量(图7b)。mab39免疫反应性的降低归因于sds-page上znt8二聚体缔合的丧失。尽管对于变性的znt8检测,mab39比mab20弱得多,但在印迹上,mab39的znt8反应性似乎与针对his标记的znt8的商品化抗体的znt8反应性相当(图7b)。下一步,我们使用mab探测endoc-βh(一种衍生自人胰腺β细胞的胰岛素瘤细胞系)中的内源性znt8表达(32)。用mab20对endoc-βh裂解物进行的免疫印迹在可忽略的背景上显示了一条单一的znt8蛋白条带。mab39既未检测到znt8,也未检测到非特异性内源蛋白(图7c)。最后,我们使用过表达znt1、znt2、znt3、znt4、znt7、znt8或znt10(每个标记有c末端flag八肽)的hek293细胞的裂解物检查了mab20和mab39对znt同源物的特异性。在每个泳道中具有相似的样品上样量时,两个mab都仅检测到znt8(图7f-图7g),这证明了相对于其他znt同源物,两个mab对人类znt8具有极好的选择性。
定量细胞znt8水平。mab20-mab39对与单个znt8蛋白的共结合使得可以通过测量衍生自紧邻的标记荧光供体和受体的fret信号来定量znt8水平。用长发光的铽穴状化合物标记mab20为作为能量供体,而用d2标记mab39作为近红外能量受体。将固定量的供体和受体以1:1的比例添加到过表达人类znt8、小鼠znt8或人类znt3的hek293细胞的裂解物中。在一系列裂解物稀释液上进行htrf校准,显示出线性htrf响应直至人类znt8浓度为50ng/ml,然后浓度由于结合饱和而水平弯曲(图8)。即使在500ng/ml的高znt浓度下,小鼠znt8或人类znt3(与人类znt8最接近的同源物)也显示出微不足道的htrf响应或无htrf响应。因此,将小鼠znt8和人类znt3读数作为htrf背景读数。在高于背景的3×sd处,人类znt8的检出限(limitofdetection,lod)为0.51ng/ml,而在高于背景的10×sd处,定量限(limitofquantification,loq)为1.71ng/ml(图8,插图)。loq的方差系数(cv)为9.8%。
讨论
znt8的与众不同的生化特性对开发znt8特异性试剂提出了重大挑战。迄今为止,所有抗znt8抗体均是针对衍生自znt8蛋白序列的线性表位产生的,因此,对于胰腺β细胞中内源性znt8的免疫检测缺乏足够的特异性。在本工作中,我们表征了针对复原的蛋白脂质体中全长人类znt8的两种抗znt8mab。两种mab均识别具有次纳摩尔结合亲和力的折叠znt8,并且能够保护znt8免受解折叠并部分抑制znt8转运活性。尽管mab39的反应性大大降低,但折叠依赖性并不阻止mab在sds-page上识别变性的znt8。两种mab均对人类znt8具有高度特异性,没有显示出对免疫印迹上的一组人类znt同源物的可检测到的反应性。通过将两个znt8特异性mab与单个znt8蛋白同时结合,进一步增强了人类znt8的特异性免疫检测。因为mab20不识别小鼠znt8,所以mab20-mab39对没有产生可检测到的小鼠znt8的fret读数(图7b)。单独的mab39与小鼠znt8的结合(图7a)未能引起高于测定的lod的fret响应,表明htrf检测的特异性是两个单独的mab特异性相乘的产物。
fab结合的抑制作用反映了人类znt8的第325位上的多态性r到w置换的作用。r325和w325变体之间的稳态动力学的早期比较显示出w325将vmax降低了~50%,而对km无明显影响(20)。基于人类znt8的同源模建,r325w多态性变体位于c末端附近的远端二聚体界面(20)。巧合的是,通过我们的em分析,fab39似乎与相同区域结合。多态性ctd变体的功能表征表明,ctd-w325比ctd-r325具有更高的二聚化亲和力(33),而去除锌与ctd的结合则显示出触发了铰链状运动,从而产生了两个张开的ctd(29、34)。w325变体和fab39结合都导致二聚体相互作用的稳定化,从而导致部分抑制了znt8转运活性。因此,fab39结合可以模拟w325变体通过作用于相同的变构途径的抑制作用。我们的结果证明了mab作为变构抑制剂的实用性。
新兴数据表明,胰腺β细胞中的人类znt8活性可能与发展t2d的风险成反比(15、20、22)。目前,尚不清楚人类znt8中lof突变与较低的t2d风险之间的因果关系下的分子机制。对这种天然保护作用的机制理解可以促进向新型抗糖尿病疗法的转化。在一般人群中,风险r325的等位基因频率约为72%(35),但是其在人胰岛中的表达水平尚未量化,并且与t2d风险相关。htrf测定对人类znt8具有高度特异性,在临床研究环境中准确且易于执行。该测定可以适于测量健康和患病的胰岛中人类znt8的丰度,以将胰岛znt8表达谱与t2d相关的临床指标相关联。预期r325表达的改变会加剧或减缓锌转运的过度活跃。将mab作为一种特异性znt8生物传感器应用于临床研究中可以产生有关znt8病理生理学的新见解。
参考文献
1.kambe,t.,hashimoto,a.,andfujimoto,s.(2014)currentunderstandingofzipandzntzinctransportersinhumanhealthanddiseases.cellmollifesci71,3281-3295
2.chimienti,f.,devergnas,s.,favier,a.,andseve,m.(2004)identificationandcloningofabeta-cell-specificzinctransporter,znt-8,localizedintoinsulinsecretorygranules.diabetes53,2330-2337
3.lemaire,k.,chimienti,f.,andschuit,f.(2012)zinctransportersandtheirroleinthepancreaticbeta-cell.jdiabetesinvestig3,202-211
4.segerstolpe,a.,palasantza,a.,eliasson,p.,andersson,e.m.,andreasson,a.c.,sun,x.,picelli,s.,sabirsh,a.,clausen,m.,bjursell,m.k.,smith,d.m.,kasper,m.,ammala,c.,andsandberg,r.(2016)single-celltranscriptomeprofilingofhumanpancreaticisletsinhealthandtype2diabetes.cellmetab24,593-607
5.chimienti,f.,devergnas,s.,pattou,f.,schuit,f.,garcia-cuenca,r.,vandewalle,b.,kerr-conte,j.,vanlommel,l.,grunwald,d.,favier,a.,andseve,m.(2006)invivoexpressionandfunctionalcharacterizationofthezinctransporterznt8inglucose-inducedinsulinsecretion.jcellsci.119,4199-4206.
6.lemaire,k.,ravier,m.a.,schraenen,a.,creemers,j.w.,vandeplas,r.,granvik,m.,vanlommel,l.,waelkens,e.,chimienti,f.,rutter,g.a.,gilon,p.,in’tveld,p.a.,andschuit,f.c.(2009)insulincrystallizationdependsonzinctransporterznt8expression,butisnotrequiredfornormalglucosehomeostasisinmice.procnatlacadsciusa106,14872-14877
7.nicolson,t.j.,bellomo,e.a.,wijesekara,n.,loder,m.k.,baldwin,j.m.,gyulkhandanyan,a.v.,koshkin,v.,tarasov,a.i.,carzaniga,r.,kronenberger,k.,taneja,t.k.,dasilvaxavier,g.,libert,s.,froguel,p.,scharfmann,r.,stetsyuk,v.,ravassard,p.,parker,h.,gribble,f.m.,reimann,f.,sladek,r.,hughes,s.j.,johnson,p.r.,masseboeuf,m.,burcelin,r.,baldwin,s.a.,liu,m.,lara-lemus,r.,arvan,p.,schuit,f.c.,wheeler,m.b.,chimienti,f.,andrutter,g.a.(2009)insulinstorageandglucosehomeostasisinmicenullforthegranulezinctransporterznt8andstudiesofthetype2diabetes-associatedvariants.diabetes58,2070-2083
8.foster,m.c.,leapman,r.d.,li,m.x.,andatwater,i.(1993)elementalcompositionofsecretorygranulesinpancreaticisletsoflangerhans.biophysj64,525-532
9.dunn,m.f.(2005)zinc-ligandinteractionsmodulateassemblyandstabilityoftheinsulinhexamer--areview.biometals18,295-303
10.rutter,g.a.(2010)thinkzinc:newrolesforzincinthecontrolofinsulinsecretion.islets2,49-50
11.tamaki,m.,fujitani,y.,hara,a.,uchida,t.,tamura,y.,takeno,k.,kawaguchi,m.,watanabe,t.,ogihara,t.,fukunaka,a.,shimizu,t.,mita,t.,kanazawa,a.,imaizumi,m.o.,abe,t.,kiyonari,h.,hojyo,s.,fukada,t.,kawauchi,t.,nagamatsu,s.,hirano,t.,kawamori,r.,andwatada,h.(2013)thediabetes-susceptiblegeneslc30a8/znt8regulateshepaticinsulinclearance.jclininvest123,4513-4524
12.ashcroft,f.m.,androrsman,p.(2012)diabetesmellitusandthebetacell:thelasttenyears.cell148,1160-1171
13.prasad,r.b.,andgroop,l.(2015)geneticsoftype2diabetes-pitfallsandpossibilities.genes(basel)6,87-123
14.bonnefond,a.,andfroguel,p.(2015)rareandcommongeneticeventsintype2diabetes:whatshouldbiologistsknow?cellmetab21,357-368
15.flannick,j.,thorleifsson,g.,beer,n.l.,jacobs,s.b.,grarup,n.,burtt,n.p.,mahajan,a.,fuchsberger,c.,atzmon,g.,benediktsson,r.,blangero,j.,bowden,d.w.,brandslund,i.,brosnan,j.,burslem,f.,chambers,j.,cho,y.s.,christensen,c.,douglas,d.a.,duggirala,r.,dymek,z.,farjoun,y.,fennell,t.,fontanillas,p.,forsen,t.,gabriel,s.,glaser,b.,gudbjartsson,d.f.,hanis,c.,hansen,t.,hreidarsson,a.b.,hveem,k.,ingelsson,e.,isomaa,b.,johansson,s.,jorgensen,t.,jorgensen,m.e.,kathiresan,s.,kong,a.,kooner,j.,kravic,j.,laakso,m.,lee,j.y.,lind,l.,lindgren,c.m.,linneberg,a.,masson,g.,meitinger,t.,mohlke,k.l.,molven,a.,morris,a.p.,potluri,s.,rauramaa,r.,ribel-madsen,r.,richard,a.m.,rolph,t.,salomaa,v.,segre,a.v.,skarstrand,h.,steinthorsdottir,v.,stringham,h.m.,sulem,p.,tai,e.s.,teo,y.y.,teslovich,t.,thorsteinsdottir,u.,trimmer,j.k.,tuomi,t.,tuomilehto,j.,vaziri-sani,f.,voight,b.f.,wilson,j.g.,boehnke,m.,mccarthy,m.i.,njolstad,p.r.,pedersen,o.,groop,l.,cox,d.r.,stefansson,k.,andaltshuler,d.(2014)loss-of-functionmutationsinslc30a8protectagainsttype2diabetes.natgenet46,357-363
16.pearson,e.(2014)zinctransportanddiabetesrisk.natgenet46,323-324
17.sladek,r.,rocheleau,g.,rung,j.,dina,c.,shen,l.,serre,d.,boutin,p.,vincent,d.,belisle,a.,hadjadj,s.,balkau,b.,heude,b.,charpentier,g.,hudson,t.j.,montpetit,a.,pshezhetsky,a.v.,prentki,m.,posner,b.i.,balding,d.j.,meyre,d.,polychronakos,c.,andfroguel,p.(2007)agenome-wideassociationstudyidentifiesnovelrisklocifortype2diabetes.nature.445,881-885.
18.scott,l.j.,mohlke,k.l.,bonnycastle,l.l.,willer,c.j.,li,y.,duren,w.l.,erdos,m.r.,stringham,h.m.,chines,p.s.,jackson,a.u.,prokunina-olsson,l.,ding,c.j.,swift,a.j.,andetal.(2007)agenome-wideassociationstudyoftype2diabetesinfinnsdetectsmultiplesusceptibilityvariants.science.316,1341-1345.
19.zeggini,e.,weedon,m.n.,lindgren,c.m.,frayling,t.m.,elliott,k.s.,lango,h.,timpson,n.j.,perry,j.r.,rayner,n.w.,freathy,r.m.,barrett,j.c.,andetal.(2007)replicationofgenome-wideassociationsignalsinuksamplesrevealsrisklocifortype2diabetes.science.316,1336-1341.
20.merriman,c.,huang,q.,rutter,g.a.,andfu,d.(2016)lipid-tunedzinctransportactivityofhumanznt8proteincorrelateswithriskfortype-2diabetes.jbiolchem291,26950-26957
21.wong,w.p.,allen,n.b.,meyers,m.s.,link,e.o.,zhang,x.,macrenaris,k.w.,andelmuayed,m.(2017)exploringtheassociationbetweendemographics,slc30a8genotype,andhumanisletcontentofzinc,cadmium,copper,iron,manganeseandnickel.scirep7,473
22.li,l.,bai,s.,andsheline,c.t.(2017)hznt8(slc30a8)transgenicmicethatoverexpressther325wpolymorphhavereducedisletzn2+andproinsulinlevels,increasedglucosetoleranceafterahigh-fatdiet,andalteredlevelsofpancreaticzincbindingproteins.diabetes66,551-559
23.pound,l.d.,sarkar,s.a.,benninger,r.k.,wang,y.,suwanichkul,a.,shadoan,m.k.,printz,r.l.,oeser,j.k.,lee,c.e.,piston,d.w.,mcguinness,o.p.,hutton,j.c.,powell,d.r.,ando’brien,r.m.(2009)deletionofthemouseslc30a8geneencodingzinctransporter-8resultsinimpairedinsulinsecretion.biochemj421,371-376
24.pound,l.d.,sarkar,s.a.,ustione,a.,dadi,p.k.,shadoan,m.k.,lee,c.e.,walters,j.a.,shiota,m.,mcguinness,o.p.,jacobson,d.a.,piston,d.w.,hutton,j.c.,powell,d.r.,ando’brien,r.m.(2012)thephysiologicaleffectsofdeletingthemouseslc30a8geneencodingzinctransporter-8areinfluencedbygenderandgeneticbackground.plosone7,e40972
25.friend,s.h.,andschadt,e.e.(2014)translationalgenomics.cluesfromtheresilient.science344,970-972
26.ohana,e.,hoch,e.,keasar,c.,kambe,t.,yifrach,o.,hershfinkel,m.,andsekler,i.(2009)identificationofthezn2+bindingsiteandmodeofoperationofamammalianzn2+transporter.jbiolchem284,17677-17686
27.hoch,e.,lin,w.,chai,j.,hershfinkel,m.,fu,d.,andsekler,i.(2012)histidinepairingatthemetaltransportsiteofmammalianznttransporterscontrolszn2+overcd2+selectivity.procnatlacadsciusa109,7202-7207
28.lu,m.,andfu,d.(2007)structureofthezinctransporteryiip.science.317,1746-1748.29.lu,m.,chai,j.,andfu,d.(2009)structuralbasisforautoregulationofthezinctransporteryiip.natstructmolbiol16,1063-1067
30.degorce,f.,card,a.,soh,s.,trinquet,e.,knapik,g.p.,andxie,b.(2009)htrf:atechnologytailoredfordrugdiscovery-areviewoftheoreticalaspectsandrecentapplications.currchemgenomics3,22-32
31.merriman,c.(2018)asubclassofserumanti-znt8antibodiesdirectedtothesurfaceoflivepancreaticbeta-cells.293,579-587
32.ravassard,p.,hazhouz,y.,pechberty,s.,bricout-neveu,e.,armanet,m.,czernichow,p.,andscharfmann,r.(2011)ageneticallyengineeredhumanpancreaticbetacelllineexhibitingglucose-inducibleinsulinsecretion.jclininvest121,3589-3597
33.parsons,d.s.,hogstrand,c.,andmaret,w.(2018)thec-terminalcytosolicdomainofthehumanzinctransporterznt8anditsdiabetesriskvariant.febsj285,1237-1250
34.cherezov,v.,hofer,n.,szebenyi,d.m.,kolaj,o.,wall,j.g.,gillilan,r.,srinivasan,v.,jaroniec,c.p.,andcaffrey,m.(2008)insightsintothemodeofactionofaputativezinctransporterczrbinthermusthermophilus.structure.16,1378-1388.
35.boesgaard,t.w.,zilinskaite,j.,vanttinen,m.,laakso,m.,jansson,p.a.,hammarstedt,a.,smith,u.,stefan,n.,fritsche,a.,haring,h.,hribal,m.,sesti,g.,zobel,d.p.,pedersen,o.,hansen,t.,andconsortium,e.(2008)thecommonslc30a8arg325trpvariantisassociatedwithreducedfirst-phaseinsulinreleasein846non-diabeticoffspringoftype2diabetespatients--theeugene2study.diabetologia51,816-820
36.merriman,c.,huang,q.,gu,w.,yu,l.,andfu,d.(2018)asubclassofserumanti-znt8antibodiesdirectedtothesurfaceoflivepancreaticbeta-cells.jbiolchem293,579-587
37.lim,h.h.,fang,y.,andwilliams,c.(2011)high-efficiencyscreeningofmonoclonalantibodiesformembraneproteincrystallography.plosone6,e24653
38.rohou,a.,andgrigorieff,n.(2015)ctffind4:fastandaccuratedefocusestimationfromelectronmicrographs.jstructbiol192,216-221
39.scheres,s.h.(2012)relion:implementationofabayesianapproachtocryo-emstructuredetermination.jstructbiol180,519-530
40.punjani,a.,rubinstein,j.l.,fleet,d.j.,andbrubaker,m.a.(2017)cryosparc:algorithmsforrapidunsupervisedcryo-emstructuredetermination.natmethods14,290-296
41.vyas,n.k.,vyas,m.n.,chervenak,m.c.,johnson,m.a.,pinto,b.m.,bundle,d.r.,andquiocho,f.a.(2002)molecularrecognitionofoligosaccharideepitopesbyamonoclonalfabspecificforshigellaflexneriylipopolysaccharide:x-raystructuresandthermodynamics.biochemistry41,13575-13586
42.pettersen,e.f.,goddard,t.d.,huang,c.c.,couch,g.s.,greenblatt,d.m.,meng,e.c.,andferrin,t.e.(2004)ucsfchimera--avisualizationsystemforexploratoryresearchandanalysis.jcomputchem25,1605-1612
43.huang,q.,merriman,c.,zhang,h.,andfu,d.(2017)couplingofinsulinsecretionanddisplayofagranule-residentzinctransporterznt8onthesurfaceofpancreaticbetacells.jbiolchem292,4034-4043
44.zhang,j.h.,chung,t.d.,andoldenburg,k.r.(1999)asimplestatisticalparameterforuseinevaluationandvalidationofhighthroughputscreeningassays.jbiomolscreen4,67-73
表3.序列表说明
序列表
<110>约翰霍普金斯大学
<120>人类znt8抗体
<130>p15820-02
<150>62/719,016
<151>2018-08-16
<160>62
<170>patentinversion3.5
<210>1
<211>1404
<212>dna
<213>artificialsequence
<220>
<223>mab12heavychain
<400>1
atggacatgagggtgcccgctcagctcctggggctcctgctgctgtggctgagaggtgcg60
cgctgtcagatccagttggtgcagtctggacctgagctgaagaagcctggagagacagtc120
aagatctcctgcaaggcttctgggtataccttcacaaactatccaatgcactgggtgaag180
cagactccaggaaagggtttaaagtggatgggctggataaacacctactctggagtgcca240
acatatggagatgacttcaagggacggtttgccttctctttggaaacctctgccagtact300
gcatatttgcagatcaacaacctcaaaaatgaagacatggctacatatttctgtgcaaga360
tcgaacccctatgattacttgtatgctatggactcctggggtcaaggaacctcagtcacc420
gtctctagtgccaaaacgacacccccatctgtctacccactggcccctggatctgctgcc480
caaactaactccatggtgaccctgggatgcctggtcaagggctatttccctgagccagtg540
acagtgacctggaactctggatccctgtccagcggtgtgcacaccttcccagctgtcctg600
cagtctgacctctacactctgagcagctcagtgactgtcccctccagcacctggcccagc660
gagaccgtcacctgcaacgttgcccacccggccagcagcaccaaggtggacaagaaaatt720
gtgcccagggattgtggttgtaagccttgcatatgtacagtcccagaagtatcatctgtc780
ttcatcttccccccaaagcccaaggatgtgctcaccattactctgactcctaaggtcacg840
tgtgttgtggtagacatcagcaaggatgatcccgaggtccagttcagctggtttgtagat900
gatgtggaggtgcacacagctcagacgcaaccccgggaggagcagttcaacagcactttc960
cgctcagtcagtgaacttcccatcatgcaccaggactggctcaatggcaaggagttcaaa1020
tgcagggtcaacagtgcagctttccctgcccccatcgagaaaaccatctccaaaaccaaa1080
ggcagaccgaaggctccacaggtgtacaccattccacctcccaaggagcagatggccaag1140
gataaagtcagtctgacctgcatgataacagacttcttccctgaagacattactgtggag1200
tggcagtggaatgggcagccagcggagaactacaagaacactcagcccatcatggacaca1260
gatggctcttacttcgtctacagcaagctcaatgtgcagaagagcaactgggaggcagga1320
aatactttcacctgctctgtgttacatgagggcctgcacaaccaccatactgagaagagc1380
ctctcccactctcctggtaaatga1404
<210>2
<211>467
<212>prt
<213>artificialsequence
<220>
<223>mab12heavychain
<400>2
metaspmetargvalproalaglnleuleuglyleuleuleuleutrp
151015
leuargglyalaargcysglnileglnleuvalglnserglyproglu
202530
leulyslysproglygluthrvallysilesercyslysalasergly
354045
tyrthrphethrasntyrpromethistrpvallysglnthrprogly
505560
lysglyleulystrpmetglytrpileasnthrtyrserglyvalpro
65707580
thrtyrglyaspaspphelysglyargphealapheserleugluthr
859095
seralaserthralatyrleuglnileasnasnleulysasngluasp
100105110
metalathrtyrphecysalaargserasnprotyrasptyrleutyr
115120125
alametaspsertrpglyglnglythrservalthrvalserserala
130135140
lysthrthrproproservaltyrproleualaproglyseralaala
145150155160
glnthrasnsermetvalthrleuglycysleuvallysglytyrphe
165170175
progluprovalthrvalthrtrpasnserglyserleusersergly
180185190
valhisthrpheproalavalleuglnseraspleutyrthrleuser
195200205
serservalthrvalproserserthrtrpprosergluthrvalthr
210215220
cysasnvalalahisproalaserserthrlysvalasplyslysile
225230235240
valproargaspcysglycyslysprocysilecysthrvalproglu
245250255
valserservalpheilepheproprolysprolysaspvalleuthr
260265270
ilethrleuthrprolysvalthrcysvalvalvalaspileserlys
275280285
aspaspprogluvalglnphesertrpphevalaspaspvalgluval
290295300
histhralaglnthrglnproargglugluglnpheasnserthrphe
305310315320
argservalsergluleuproilemethisglnasptrpleuasngly
325330335
lysgluphelyscysargvalasnseralaalapheproalaproile
340345350
glulysthrileserlysthrlysglyargprolysalaproglnval
355360365
tyrthrileproproprolysgluglnmetalalysasplysvalser
370375380
leuthrcysmetilethraspphepheprogluaspilethrvalglu
385390395400
trpglntrpasnglyglnproalagluasntyrlysasnthrglnpro
405410415
ilemetaspthraspglysertyrphevaltyrserlysleuasnval
420425430
glnlysserasntrpglualaglyasnthrphethrcysservalleu
435440445
hisgluglyleuhisasnhishisthrglulysserleuserhisser
450455460
proglylys
465
<210>3
<211>7
<212>prt
<213>artificialsequence
<220>
<223>mab12heavychaincdr1
<400>3
glytyrthrphethrasntyr
15
<210>4
<211>5
<212>prt
<213>artificialsequence
<220>
<223>mab12heavychaincdr2
<400>4
thrtyrserglyval
15
<210>5
<211>12
<212>prt
<213>artificialsequence
<220>
<223>mabheavychaincdr3
<400>5
serasnprotyrasptyrleutyralametaspser
1510
<210>6
<211>726
<212>dna
<213>artificialsequence
<220>
<223>mab12lightchain
<400>6
atggacatgagggtgcccgctcagctcctggggctcctgctgctgtggctgagaggtgcg60
cgctgtgatgttgtgatgacccaaactccactctccctgcctgtcagtcttggagatcaa120
gcctccatctcctgcagatctagtcagagccttgtacacatcaatggaaacacctatata180
cattggtacctgcagaagccaggccagtctccaaagctcctgatctacaaagtttccaac240
cgattttctggggtcccagacaggttcagtggcagtggatcagggacagattttacactc300
aagatcagaagagtggaggctgaggatctgggagtttatttctgctctcaaaatacacat360
gttccattcacattcggctcggggacaaagttggaaataaaacgtgcagatgctgcgcca420
actgtatccatcttcccaccatctagcgagcagttaacatctggaggtgcctcagtcgtg480
tgcttcttgaacaacttctaccccaaagacatcaatgtcaagtggaagattgatggcagt540
gaacgacaaaatggcgtcctgaacagttggactgatcaggacagcaaagacagcacctac600
agcatgagcagcaccctcacgttgaccaaggacgagtatgaacgacataacagctatacc660
tgtgaggccactcacaagacatcaacttcacccattgtcaagagcttcaacaggaatgag720
tgttag726
<210>7
<211>241
<212>prt
<213>artificialsequence
<220>
<223>mab12lightchain
<400>7
metaspmetargvalproalaglnleuleuglyleuleuleuleutrp
151015
leuargglyalaargcysaspvalvalmetthrglnthrproleuser
202530
leuprovalserleuglyaspglnalaserilesercysargserser
354045
glnserleuvalhisileasnglyasnthrtyrilehistrptyrleu
505560
glnlysproglyglnserprolysleuleuiletyrlysvalserasn
65707580
argpheserglyvalproaspargpheserglyserglyserglythr
859095
aspphethrleulysileargargvalglualagluaspleuglyval
100105110
tyrphecysserglnasnthrhisvalprophethrpheglysergly
115120125
thrlysleugluilelysargalaaspalaalaprothrvalserile
130135140
pheproprosersergluglnleuthrserglyglyalaservalval
145150155160
cyspheleuasnasnphetyrprolysaspileasnvallystrplys
165170175
ileaspglysergluargglnasnglyvalleuasnsertrpthrasp
180185190
glnaspserlysaspserthrtyrsermetserserthrleuthrleu
195200205
thrlysaspglutyrgluarghisasnsertyrthrcysglualathr
210215220
hislysthrserthrserproilevallysserpheasnargasnglu
225230235240
cys
<210>8
<211>16
<212>prt
<213>artificialsequence
<220>
<223>mab12lightchaincdr1
<400>8
argserserglnserleuvalhisileasnglyasnthrtyrilehis
151015
<210>9
<211>7
<212>prt
<213>artificialsequence
<220>
<223>mab12lightchaincdr2
<400>9
lysvalserasnargpheser
15
<210>10
<211>9
<212>prt
<213>artificialsequence
<220>
<223>mab12lightchaincdr3
<400>10
serglnasnthrhisvalprophethr
15
<210>11
<211>1401
<212>dna
<213>artificialsequence
<220>
<223>mab16heavychain
<400>11
atggacatgagggtgcccgctcagctcctggggctcctgctgctgtggctgagaggtgcg60
cgctgtcagatccagttggtgcagtctggacctgagctgaagaagcctggagagacagtc120
acgatctcctgcaaggcttctggatataccttcacacactatccagtgcactgggtgaag180
caggctccaggaaagggtttacagtggatgggctggataaacacctactctggagtgcca240
acatatgcagatgccttcaagaaacgttttgccttctctttggaaacctctgccagcact300
gcatatttgcagatcaacaacctcaaaagtgaggacatggctacatatttctgtgcaaga360
tcgagggtctatgatgggtactattttgactactggggccaaggcaccactctcaccgtc420
tctagtgccaaaacgacacccccatctgtctacccactggcccctggatctgctgcccaa480
actaactccatggtgaccctgggatgcctggtcaagggctatttccctgagccagtgaca540
gtgacctggaactctggatccctgtccagcggtgtgcacaccttcccagctgtcctgcag600
tctgacctctacactctgagcagctcagtgactgtcccctccagcacctggcccagcgag660
accgtcacctgcaacgttgcccacccggccagcagcaccaaggtggacaagaaaattgtg720
cccagggattgtggttgtaagccttgcatatgtacagtcccagaagtatcatctgtcttc780
atcttccccccaaagcccaaggatgtgctcaccattactctgactcctaaggtcacgtgt840
gttgtggtagacatcagcaaggatgatcccgaggtccagttcagctggtttgtagatgat900
gtggaggtgcacacagctcagacgcaaccccgggaggagcagttcaacagcactttccgc960
tcagtcagtgaacttcccatcatgcaccaggactggctcaatggcaaggagttcaaatgc1020
agggtcaacagtgcagctttccctgcccccatcgagaaaaccatctccaaaaccaaaggc1080
agaccgaaggctccacaggtgtacaccattccacctcccaaggagcagatggccaaggat1140
aaagtcagtctgacctgcatgataacagacttcttccctgaagacattactgtggagtgg1200
cagtggaatgggcagccagcggagaactacaagaacactcagcccatcatggacacagat1260
ggctcttacttcgtctacagcaagctcaatgtgcagaagagcaactgggaggcaggaaat1320
actttcacctgctctgtgttacatgagggcctgcacaaccaccatactgagaagagcctc1380
tcccactctcctggtaaatga1401
<210>12
<211>466
<212>prt
<213>artificialsequence
<220>
<223>mab16heavychain
<400>12
metaspmetargvalproalaglnleuleuglyleuleuleuleutrp
151015
leuargglyalaargcysglnileglnleuvalglnserglyproglu
202530
leulyslysproglygluthrvalthrilesercyslysalasergly
354045
tyrthrphethrhistyrprovalhistrpvallysglnalaprogly
505560
lysglyleuglntrpmetglytrpileasnthrtyrserglyvalpro
65707580
thrtyralaaspalaphelyslysargphealapheserleugluthr
859095
seralaserthralatyrleuglnileasnasnleulyssergluasp
100105110
metalathrtyrphecysalaargserargvaltyraspglytyrtyr
115120125
pheasptyrtrpglyglnglythrthrleuthrvalserseralalys
130135140
thrthrproproservaltyrproleualaproglyseralaalagln
145150155160
thrasnsermetvalthrleuglycysleuvallysglytyrphepro
165170175
gluprovalthrvalthrtrpasnserglyserleuserserglyval
180185190
histhrpheproalavalleuglnseraspleutyrthrleuserser
195200205
servalthrvalproserserthrtrpprosergluthrvalthrcys
210215220
asnvalalahisproalaserserthrlysvalasplyslysileval
225230235240
proargaspcysglycyslysprocysilecysthrvalprogluval
245250255
serservalpheilepheproprolysprolysaspvalleuthrile
260265270
thrleuthrprolysvalthrcysvalvalvalaspileserlysasp
275280285
aspprogluvalglnphesertrpphevalaspaspvalgluvalhis
290295300
thralaglnthrglnproargglugluglnpheasnserthrphearg
305310315320
servalsergluleuproilemethisglnasptrpleuasnglylys
325330335
gluphelyscysargvalasnseralaalapheproalaproileglu
340345350
lysthrileserlysthrlysglyargprolysalaproglnvaltyr
355360365
thrileproproprolysgluglnmetalalysasplysvalserleu
370375380
thrcysmetilethraspphepheprogluaspilethrvalglutrp
385390395400
glntrpasnglyglnproalagluasntyrlysasnthrglnproile
405410415
metaspthraspglysertyrphevaltyrserlysleuasnvalgln
420425430
lysserasntrpglualaglyasnthrphethrcysservalleuhis
435440445
gluglyleuhisasnhishisthrglulysserleuserhisserpro
450455460
glylys
465
<210>13
<211>7
<212>prt
<213>artificialsequence
<220>
<223>mab16heavychaincdr1
<400>13
glytyrthrphethrhistyr
15
<210>14
<211>6
<212>prt
<213>artificialsequence
<220>
<223>mab16heavychaincdr2
<400>14
asnthrtyrserglyval
15
<210>15
<211>11
<212>prt
<213>artificialsequence
<220>
<223>mab16heavychaincdr3
<400>15
serargvaltyraspglytyrtyrpheasptyr
1510
<210>16
<211>726
<212>dna
<213>artificialsequence
<220>
<223>mab16lightchain
<400>16
atggacatgagggtgcccgctcagctcctggggctcctgctgctgtggctgagaggtgcg60
cgctgtgatgttgtgatgacccaaactccactctccctgcctgtcagtcttggagatcaa120
gcctccatctcttgcagatctagtcagagccttgtacacagtaatggaaagacctattta180
cattggtacctgcagaagccaggccagtctccaaacctcctgatctacaaagtttccaac240
cgattttctggggtcccagacaggttcagtggcagtggatcagggacagatttcacactc300
aagatcagcagagtggaggctgaggatctgggagtttatttctgctctcaacttacacat360
gttccgtggacgttcggtggaggcaccaagctggaaatcaaacgtgcagatgctgcgcca420
actgtatccatcttcccaccatctagcgagcagttaacatctggaggtgcctcagtcgtg480
tgcttcttgaacaacttctaccccaaagacatcaatgtcaagtggaagattgatggcagt540
gaacgacaaaatggcgtcctgaacagttggactgatcaggacagcaaagacagcacctac600
agcatgagcagcaccctcacgttgaccaaggacgagtatgaacgacataacagctatacc660
tgtgaggccactcacaagacatcaacttcacccattgtcaagagcttcaacaggaatgag720
tgttag726
<210>17
<211>241
<212>prt
<213>artificialsequence
<220>
<223>mab16lightchain
<400>17
metaspmetargvalproalaglnleuleuglyleuleuleuleutrp
151015
leuargglyalaargcysaspvalvalmetthrglnthrproleuser
202530
leuprovalserleuglyaspglnalaserilesercysargserser
354045
glnserleuvalhisserasnglylysthrtyrleuhistrptyrleu
505560
glnlysproglyglnserproasnleuleuiletyrlysvalserasn
65707580
argpheserglyvalproaspargpheserglyserglyserglythr
859095
aspphethrleulysileserargvalglualagluaspleuglyval
100105110
tyrphecysserglnleuthrhisvalprotrpthrpheglyglygly
115120125
thrlysleugluilelysargalaaspalaalaprothrvalserile
130135140
pheproprosersergluglnleuthrserglyglyalaservalval
145150155160
cyspheleuasnasnphetyrprolysaspileasnvallystrplys
165170175
ileaspglysergluargglnasnglyvalleuasnsertrpthrasp
180185190
glnaspserlysaspserthrtyrsermetserserthrleuthrleu
195200205
thrlysaspglutyrgluarghisasnsertyrthrcysglualathr
210215220
hislysthrserthrserproilevallysserpheasnargasnglu
225230235240
cys
<210>18
<211>16
<212>prt
<213>artificialsequence
<220>
<223>mab16lightchaincdr1
<400>18
argserserglnserleuvalhisserasnglylysthrtyrleuhis
151015
<210>19
<211>7
<212>prt
<213>artificialsequence
<220>
<223>mab16lightchaincdr2
<400>19
lysvalserasnargpheser
15
<210>20
<211>8
<212>prt
<213>artificialsequence
<220>
<223>mab16lightchaincdr3
<400>20
serglnleuthrhisvalprotrp
15
<210>21
<211>1407
<212>dna
<213>artificialsequence
<220>
<223>mab17heavychain
<400>21
atggacatgagggtgcccgctcagctcctggggctcctgctgctgtggctgagaggtgcg60
cgctgtcagatccagttggtgcagtctggacctgagctgaagaagcctggagagacagtc120
aagatctcctgcaaggcttctgggtataccttcacaaactatccaatgcactggttgaag180
caggctccaggaaagggtttaaagtggatgggctggataaacacctactctggagtgcca240
acatatgcagatgacttcaagggacggtttgccttctctttggaaacctctgccagcact300
gcatatttgcagatcaacaacctcaaaaatgaggacatggctacatatttctgtacaaaa360
tcgcgcattactacgatggggggttatgctatggactgctggggtcaaggaacctcagtc420
accgtctctagtgccaaaacgacacccccatctgtctacccactggcccctggatctgct480
gcccaaactaactccatggtgaccctgggatgcctggtcaagggctatttccctgagcca540
gtgacagtgacctggaactctggatccctgtccagcggtgtgcacaccttcccagctgtc600
ctgcagtctgacctctacactctgagcagctcagtgactgtcccctccagcacctggccc660
agcgagaccgtcacctgcaacgttgcccacccggccagcagcaccaaggtggacaagaaa720
attgtgcccagggattgtggttgtaagccttgcatatgtacagtcccagaagtatcatct780
gtcttcatcttccccccaaagcccaaggatgtgctcaccattactctgactcctaaggtc840
acgtgtgttgtggtagacatcagcaaggatgatcccgaggtccagttcagctggtttgta900
gatgatgtggaggtgcacacagctcagacgcaaccccgggaggagcagttcaacagcact960
ttccgctcagtcagtgaacttcccatcatgcaccaggactggctcaatggcaaggagttc1020
aaatgcagggtcaacagtgcagctttccctgcccccatcgagaaaaccatctccaaaacc1080
aaaggcagaccgaaggctccacaggtgtacaccattccacctcccaaggagcagatggcc1140
aaggataaagtcagtctgacctgcatgataacagacttcttccctgaagacattactgtg1200
gagtggcagtggaatgggcagccagcggagaactacaagaacactcagcccatcatggac1260
acagatggctcttacttcgtctacagcaagctcaatgtgcagaagagcaactgggaggca1320
ggaaatactttcacctgctctgtgttacatgagggcctgcacaaccaccatactgagaag1380
agcctctcccactctcctggtaaatga1407
<210>22
<211>468
<212>prt
<213>artificialsequence
<220>
<223>mab17heavychain
<400>22
metaspmetargvalproalaglnleuleuglyleuleuleuleutrp
151015
leuargglyalaargcysglnileglnleuvalglnserglyproglu
202530
leulyslysproglygluthrvallysilesercyslysalasergly
354045
tyrthrphethrasntyrpromethistrpleulysglnalaprogly
505560
lysglyleulystrpmetglytrpileasnthrtyrserglyvalpro
65707580
thrtyralaaspaspphelysglyargphealapheserleugluthr
859095
seralaserthralatyrleuglnileasnasnleulysasngluasp
100105110
metalathrtyrphecysthrlysserargilethrthrmetglygly
115120125
tyralametaspcystrpglyglnglythrservalthrvalserser
130135140
alalysthrthrproproservaltyrproleualaproglyserala
145150155160
alaglnthrasnsermetvalthrleuglycysleuvallysglytyr
165170175
pheprogluprovalthrvalthrtrpasnserglyserleuserser
180185190
glyvalhisthrpheproalavalleuglnseraspleutyrthrleu
195200205
serserservalthrvalproserserthrtrpprosergluthrval
210215220
thrcysasnvalalahisproalaserserthrlysvalasplyslys
225230235240
ilevalproargaspcysglycyslysprocysilecysthrvalpro
245250255
gluvalserservalpheilepheproprolysprolysaspvalleu
260265270
thrilethrleuthrprolysvalthrcysvalvalvalaspileser
275280285
lysaspaspprogluvalglnphesertrpphevalaspaspvalglu
290295300
valhisthralaglnthrglnproargglugluglnpheasnserthr
305310315320
pheargservalsergluleuproilemethisglnasptrpleuasn
325330335
glylysgluphelyscysargvalasnseralaalapheproalapro
340345350
ileglulysthrileserlysthrlysglyargprolysalaprogln
355360365
valtyrthrileproproprolysgluglnmetalalysasplysval
370375380
serleuthrcysmetilethraspphepheprogluaspilethrval
385390395400
glutrpglntrpasnglyglnproalagluasntyrlysasnthrgln
405410415
proilemetaspthraspglysertyrphevaltyrserlysleuasn
420425430
valglnlysserasntrpglualaglyasnthrphethrcysserval
435440445
leuhisgluglyleuhisasnhishisthrglulysserleuserhis
450455460
serproglylys
465
<210>23
<211>7
<212>prt
<213>artificialsequence
<220>
<223>mab17heavychaincdr1
<400>23
glytyrthrphethrasntyr
15
<210>24
<211>6
<212>prt
<213>artificialsequence
<220>
<223>mab17heavychaincdr2
<400>24
asnthrtyrserglyval
15
<210>25
<211>13
<212>prt
<213>artificialsequence
<220>
<223>mab17heavychaincdr3
<400>25
serargilethrthrmetglyglytyralametaspcys
1510
<210>26
<211>726
<212>dna
<213>artificialsequence
<220>
<223>mab17lightchain
<400>26
atggacatgagggtgcccgctcagctcctggggctcctgctgctgtggctgagaggtgcg60
cgctgtgaagttgtgatgacccaaactccactctccctgcctgtcagtcttggagatcaa120
gcctccatctcttgcagatctagtcagagccttctacacagtaatggaaacacctattta180
cattggtacctgcagaggccaggccagtctccaaacctcctgatctccaaagtttccaac240
cgattttctggggtcccagacaggttcagtggcagtggatcagggacagatttcacactc300
aagatcagcagagtggaggctgaggatctgggagtttatttctgctctcaaaatacacat360
gttccgtggacgttcggtggaggcaccaagctggaaatcaaacgtgcagatgctgcgcca420
actgtatccatcttcccaccatctagcgagcagttaacatctggaggtgcctcagtcgtg480
tgcttcttgaacaacttctaccccaaagacatcaatgtcaagtggaagattgatggcagt540
gaacgacaaaatggcgtcctgaacagttggactgatcaggacagcaaagacagcacctac600
agcatgagcagcaccctcacgttgaccaaggacgagtatgaacgacataacagctatacc660
tgtgaggccactcacaagacatcaacttcacccattgtcaagagcttcaacaggaatgag720
tgttag726
<210>27
<211>241
<212>prt
<213>artificialsequence
<220>
<223>mab17lightchain
<400>27
metaspmetargvalproalaglnleuleuglyleuleuleuleutrp
151015
leuargglyalaargcysgluvalvalmetthrglnthrproleuser
202530
leuprovalserleuglyaspglnalaserilesercysargserser
354045
glnserleuleuhisserasnglyasnthrtyrleuhistrptyrleu
505560
glnargproglyglnserproasnleuleuileserlysvalserasn
65707580
argpheserglyvalproaspargpheserglyserglyserglythr
859095
aspphethrleulysileserargvalglualagluaspleuglyval
100105110
tyrphecysserglnasnthrhisvalprotrpthrpheglyglygly
115120125
thrlysleugluilelysargalaaspalaalaprothrvalserile
130135140
pheproprosersergluglnleuthrserglyglyalaservalval
145150155160
cyspheleuasnasnphetyrprolysaspileasnvallystrplys
165170175
ileaspglysergluargglnasnglyvalleuasnsertrpthrasp
180185190
glnaspserlysaspserthrtyrsermetserserthrleuthrleu
195200205
thrlysaspglutyrgluarghisasnsertyrthrcysglualathr
210215220
hislysthrserthrserproilevallysserpheasnargasnglu
225230235240
cys
<210>28
<211>16
<212>prt
<213>artificialsequence
<220>
<223>mab17lightchaincdr1
<400>28
argserserglnserleuleuhisserasnglyasnthrtyrleuhis
151015
<210>29
<211>7
<212>prt
<213>artificialsequence
<220>
<223>mab17lightchaincdr2
<400>29
lysvalserasnargpheser
15
<210>30
<211>9
<212>prt
<213>artificialsequence
<220>
<223>mab17lightchaincdr3
<400>30
serglnasnthrhisvalprotrpthr
15
<210>31
<211>1407
<212>dna
<213>artificialsequence
<220>
<223>mab20heavychain
<400>31
atggacatgagggtgcccgctcagctcctggggctcctgctgctgtggctgagaggtgcg60
cgctgtcagatccagttggtgcagtctggacctgagctgaagaagcctggagagacagtc120
aagatctcctgcaaggcttctgggtatatcttcacaaactatccaatgcactgggtgaag180
caggctccaggaaagggtttaaagtggatgggctggataaacacctactctggagtgcca240
acacatgcagatgacttcaagggacggtttgccttctctttggaaacctctgccaacagt300
gcatttttgcagatcaacaacctcaaaaatgaggacacggctacatatttctgtacaaga360
tcgcgcattactccgacggggggctatgctatggactactggggtcaaggaacctcagtc420
accgtctctagtgccaaaacgacacccccatctgtctacccactggcccctggatctgct480
gcccaaactaactccatggtgaccctgggatgcctggtcaagggctatttccctgagcca540
gtgacagtgacctggaactctggatccctgtccagcggtgtgcacaccttcccagctgtc600
ctgcagtctgacctctacactctgagcagctcagtgactgtcccctccagcacctggccc660
agcgagaccgtcacctgcaacgttgcccacccggccagcagcaccaaggtggacaagaaa720
attgtgcccagggattgtggttgtaagccttgcatatgtacagtcccagaagtatcatct780
gtcttcatcttccccccaaagcccaaggatgtgctcaccattactctgactcctaaggtc840
acgtgtgttgtggtagacatcagcaaggatgatcccgaggtccagttcagctggtttgta900
gatgatgtggaggtgcacacagctcagacgcaaccccgggaggagcagttcaacagcact960
ttccgctcagtcagtgaacttcccatcatgcaccaggactggctcaatggcaaggagttc1020
aaatgcagggtcaacagtgcagctttccctgcccccatcgagaaaaccatctccaaaacc1080
aaaggcagaccgaaggctccacaggtgtacaccattccacctcccaaggagcagatggcc1140
aaggataaagtcagtctgacctgcatgataacagacttcttccctgaagacattactgtg1200
gagtggcagtggaatgggcagccagcggagaactacaagaacactcagcccatcatggac1260
acagatggctcttacttcgtctacagcaagctcaatgtgcagaagagcaactgggaggca1320
ggaaatactttcacctgctctgtgttacatgagggcctgcacaaccaccatactgagaag1380
agcctctcccactctcctggtaaatga1407
<210>32
<211>468
<212>prt
<213>artificialsequence
<220>
<223>mab20heavychain
<400>32
metaspmetargvalproalaglnleuleuglyleuleuleuleutrp
151015
leuargglyalaargcysglnileglnleuvalglnserglyproglu
202530
leulyslysproglygluthrvallysilesercyslysalasergly
354045
tyrilephethrasntyrpromethistrpvallysglnalaprogly
505560
lysglyleulystrpmetglytrpileasnthrtyrserglyvalpro
65707580
thrhisalaaspaspphelysglyargphealapheserleugluthr
859095
seralaasnseralapheleuglnileasnasnleulysasngluasp
100105110
thralathrtyrphecysthrargserargilethrprothrglygly
115120125
tyralametasptyrtrpglyglnglythrservalthrvalserser
130135140
alalysthrthrproproservaltyrproleualaproglyserala
145150155160
alaglnthrasnsermetvalthrleuglycysleuvallysglytyr
165170175
pheprogluprovalthrvalthrtrpasnserglyserleuserser
180185190
glyvalhisthrpheproalavalleuglnseraspleutyrthrleu
195200205
serserservalthrvalproserserthrtrpprosergluthrval
210215220
thrcysasnvalalahisproalaserserthrlysvalasplyslys
225230235240
ilevalproargaspcysglycyslysprocysilecysthrvalpro
245250255
gluvalserservalpheilepheproprolysprolysaspvalleu
260265270
thrilethrleuthrprolysvalthrcysvalvalvalaspileser
275280285
lysaspaspprogluvalglnphesertrpphevalaspaspvalglu
290295300
valhisthralaglnthrglnproargglugluglnpheasnserthr
305310315320
pheargservalsergluleuproilemethisglnasptrpleuasn
325330335
glylysgluphelyscysargvalasnseralaalapheproalapro
340345350
ileglulysthrileserlysthrlysglyargprolysalaprogln
355360365
valtyrthrileproproprolysgluglnmetalalysasplysval
370375380
serleuthrcysmetilethraspphepheprogluaspilethrval
385390395400
glutrpglntrpasnglyglnproalagluasntyrlysasnthrgln
405410415
proilemetaspthraspglysertyrphevaltyrserlysleuasn
420425430
valglnlysserasntrpglualaglyasnthrphethrcysserval
435440445
leuhisgluglyleuhisasnhishisthrglulysserleuserhis
450455460
serproglylys
465
<210>33
<211>7
<212>prt
<213>artificialsequence
<220>
<223>mab20heavychaincdr1
<400>33
glytyrilephethrasntyr
15
<210>34
<211>6
<212>prt
<213>artificialsequence
<220>
<223>mab20heavychaincdr2
<400>34
asnthrtyrserglyval
15
<210>35
<211>13
<212>prt
<213>artificialsequence
<220>
<223>mab20heavychaincdr3
<400>35
serargilethrprothrglyglytyralametasptyr
1510
<210>36
<211>726
<212>dna
<213>artificialsequence
<220>
<223>mab20lightchain
<400>36
atggacatgagggtgcccgctcagctcctggggctcctgctgctgtggctgagaggtgcg60
cgctgtgatgttgtgatgacccaaactccactctccctgcctgtcagtcttggagatcaa120
gcctccatctcttgcagatctagtcagagccttgtacacagtaatggaaacacctattta180
cattggtacctgcagaagccaggccagtctccaaacctcctgatctccaaagtttccaac240
cgattttctggggtcccagaaaggttcagtggcagtggatcagggacagatttcacactc300
aagatcagcagagtggaggctgaggatctgggagtttatttctgctctcaaaatacacat360
gttccgtggacgttcggtggaggcaccaagctggaaatcaaacgtgcagatgctgcgcca420
actgtatccatcttcccaccatctagcgagcagttaacatctggaggtgcctcagtcgtg480
tgcttcttgaacaacttctaccccaaagacatcaatgtcaagtggaagattgatggcagt540
gaacgacaaaatggcgtcctgaacagttggactgatcaggacagcaaagacagcacctac600
agcatgagcagcaccctcacgttgaccaaggacgagtatgaacgacataacagctatacc660
tgtgaggccactcacaagacatcaacttcacccattgtcaagagcttcaacaggaatgag720
tgttag726
<210>37
<211>241
<212>prt
<213>artificialsequence
<220>
<223>mab20lightchain
<400>37
metaspmetargvalproalaglnleuleuglyleuleuleuleutrp
151015
leuargglyalaargcysaspvalvalmetthrglnthrproleuser
202530
leuprovalserleuglyaspglnalaserilesercysargserser
354045
glnserleuvalhisserasnglyasnthrtyrleuhistrptyrleu
505560
glnlysproglyglnserproasnleuleuileserlysvalserasn
65707580
argpheserglyvalprogluargpheserglyserglyserglythr
859095
aspphethrleulysileserargvalglualagluaspleuglyval
100105110
tyrphecysserglnasnthrhisvalprotrpthrpheglyglygly
115120125
thrlysleugluilelysargalaaspalaalaprothrvalserile
130135140
pheproprosersergluglnleuthrserglyglyalaservalval
145150155160
cyspheleuasnasnphetyrprolysaspileasnvallystrplys
165170175
ileaspglysergluargglnasnglyvalleuasnsertrpthrasp
180185190
glnaspserlysaspserthrtyrsermetserserthrleuthrleu
195200205
thrlysaspglutyrgluarghisasnsertyrthrcysglualathr
210215220
hislysthrserthrserproilevallysserpheasnargasnglu
225230235240
cys
<210>38
<211>16
<212>prt
<213>artificialsequence
<220>
<223>mab20lightchaincdr1
<400>38
argserserglnserleuvalhisserasnglyasnthrtyrleuhis
151015
<210>39
<211>7
<212>prt
<213>artificialsequence
<220>
<223>mab20lightchaincdr2
<400>39
lysvalserasnargpheser
15
<210>40
<211>9
<212>prt
<213>artificialsequence
<220>
<223>mab40lightchaincdr3
<400>40
serglnasnthrhisvalprotrpthr
15
<210>41
<211>1407
<212>dna
<213>artificialsequence
<220>
<223>mab28heavychain
<400>41
atggacatgagggtgcccgctcagctcctggggctcctgctgctgtggctgagaggtgcg60
cgctgtcagatccagttggtgcagtctggacctgagctgaagaagcctggagagacagtc120
aagatctcctgcaaggcttctgggtataccttcacaaactatccaatgcactgggtaaag180
caggctccaggaaaggatttaaagtggatgggctggataaacacctactctggaatgtca240
acatatgcagatgacttcaagggacggtttgccttctctttggaaacctctgccagcact300
gcgtatttgcagatcaacaacctcaaaaatgaggacatggctacatatttctgtgcaaga360
tcgcgcattacaacgatggggggctatgctatggactactggggtcaaggagcctcagtc420
accgtctctagtgccaaaacgacacccccatctgtctacccactggcccctggatctgct480
gcccaaactaactccatggtgaccctgggatgcctggtcaagggctatttccctgagcca540
gtgacagtgacctggaactctggatccctgtccagcggtgtgcacaccttcccagctgtc600
ctgcagtctgacctctacactctgagcagctcagtgactgtcccctccagcacctggccc660
agcgagaccgtcacctgcaacgttgcccacccggccagcagcaccaaggtggacaagaaa720
attgtgcccagggattgtggttgtaagccttgcatatgtacagtcccagaagtatcatct780
gtcttcatcttccccccaaagcccaaggatgtgctcaccattactctgactcctaaggtc840
acgtgtgttgtggtagacatcagcaaggatgatcccgaggtccagttcagctggtttgta900
gatgatgtggaggtgcacacagctcagacgcaaccccgggaggagcagttcaacagcact960
ttccgctcagtcagtgaacttcccatcatgcaccaggactggctcaatggcaaggagttc1020
aaatgcagggtcaacagtgcagctttccctgcccccatcgagaaaaccatctccaaaacc1080
aaaggcagaccgaaggctccacaggtgtacaccattccacctcccaaggagcagatggcc1140
aaggataaagtcagtctgacctgcatgataacagacttcttccctgaagacattactgtg1200
gagtggcagtggaatgggcagccagcggagaactacaagaacactcagcccatcatggac1260
acagatggctcttacttcgtctacagcaagctcaatgtgcagaagagcaactgggaggca1320
ggaaatactttcacctgctctgtgttacatgagggcctgcacaaccaccatactgagaag1380
agcctctcccactctcctggtaaatga1407
<210>42
<211>468
<212>prt
<213>artificialsequence
<220>
<223>mab28heavychain
<400>42
metaspmetargvalproalaglnleuleuglyleuleuleuleutrp
151015
leuargglyalaargcysglnileglnleuvalglnserglyproglu
202530
leulyslysproglygluthrvallysilesercyslysalasergly
354045
tyrthrphethrasntyrpromethistrpvallysglnalaprogly
505560
lysaspleulystrpmetglytrpileasnthrtyrserglymetser
65707580
thrtyralaaspaspphelysglyargphealapheserleugluthr
859095
seralaserthralatyrleuglnileasnasnleulysasngluasp
100105110
metalathrtyrphecysalaargserargilethrthrmetglygly
115120125
tyralametasptyrtrpglyglnglyalaservalthrvalserser
130135140
alalysthrthrproproservaltyrproleualaproglyserala
145150155160
alaglnthrasnsermetvalthrleuglycysleuvallysglytyr
165170175
pheprogluprovalthrvalthrtrpasnserglyserleuserser
180185190
glyvalhisthrpheproalavalleuglnseraspleutyrthrleu
195200205
serserservalthrvalproserserthrtrpprosergluthrval
210215220
thrcysasnvalalahisproalaserserthrlysvalasplyslys
225230235240
ilevalproargaspcysglycyslysprocysilecysthrvalpro
245250255
gluvalserservalpheilepheproprolysprolysaspvalleu
260265270
thrilethrleuthrprolysvalthrcysvalvalvalaspileser
275280285
lysaspaspprogluvalglnphesertrpphevalaspaspvalglu
290295300
valhisthralaglnthrglnproargglugluglnpheasnserthr
305310315320
pheargservalsergluleuproilemethisglnasptrpleuasn
325330335
glylysgluphelyscysargvalasnseralaalapheproalapro
340345350
ileglulysthrileserlysthrlysglyargprolysalaprogln
355360365
valtyrthrileproproprolysgluglnmetalalysasplysval
370375380
serleuthrcysmetilethraspphepheprogluaspilethrval
385390395400
glutrpglntrpasnglyglnproalagluasntyrlysasnthrgln
405410415
proilemetaspthraspglysertyrphevaltyrserlysleuasn
420425430
valglnlysserasntrpglualaglyasnthrphethrcysserval
435440445
leuhisgluglyleuhisasnhishisthrglulysserleuserhis
450455460
serproglylys
465
<210>43
<211>7
<212>prt
<213>artificialsequence
<220>
<223>mab28heavychaincdr1
<400>43
glytyrthrphethrasntyr
15
<210>44
<211>6
<212>prt
<213>artificialsequence
<220>
<223>mab28heavychaincdr2
<400>44
asnthrtyrserglymet
15
<210>45
<211>13
<212>prt
<213>artificialsequence
<220>
<223>mab28heavychaincdr3
<400>45
serargilethrthrmetglyglytyralametasptyr
1510
<210>46
<211>726
<212>dna
<213>artificialsequence
<220>
<223>mab28lightchain
<400>46
atggacatgagggtgcccgctcagctcctggggctcctgctgctgtggctgagaggtgcg60
cgctgtgaagttgtgatgacccaaactccactctccctgcctgtcagtcttggagatcaa120
gcctccatctcttgcagatctagtcagagccttgtacacagtaatggaaacacctattta180
cattggtacctgcagaagccaggccagtctccaaagctcctgatctccaaagtttccaac240
cgattttctggggtcccagacaggttcagtggcagtggatcagggacagatttcacactc300
aagatcatcagagtggaggctgaggatctgggagtttatttctgctctcaaaatacacat360
gttccgtggacgttcggtggaggcaccaagctggaaatcaaacgtgcagatgctgcgcca420
actgtatccatcttcccaccatctagcgagcagttaacatctggaggtgcctcagtcgtg480
tgcttcttgaacaacttctaccccaaagacatcaatgtcaagtggaagattgatggcagt540
gaacgacaaaatggcgtcctgaacagttggactgatcaggacagcaaagacagcacctac600
agcatgagcagcaccctcacgttgaccaaggacgagtatgaacgacataacagctatacc660
tgtgaggccactcacaagacatcaacttcacccattgtcaagagcttcaacaggaatgag720
tgttag726
<210>47
<211>241
<212>prt
<213>artificialsequence
<220>
<223>mab28lightchain
<400>47
metaspmetargvalproalaglnleuleuglyleuleuleuleutrp
151015
leuargglyalaargcysgluvalvalmetthrglnthrproleuser
202530
leuprovalserleuglyaspglnalaserilesercysargserser
354045
glnserleuvalhisserasnglyasnthrtyrleuhistrptyrleu
505560
glnlysproglyglnserprolysleuleuileserlysvalserasn
65707580
argpheserglyvalproaspargpheserglyserglyserglythr
859095
aspphethrleulysileileargvalglualagluaspleuglyval
100105110
tyrphecysserglnasnthrhisvalprotrpthrpheglyglygly
115120125
thrlysleugluilelysargalaaspalaalaprothrvalserile
130135140
pheproprosersergluglnleuthrserglyglyalaservalval
145150155160
cyspheleuasnasnphetyrprolysaspileasnvallystrplys
165170175
ileaspglysergluargglnasnglyvalleuasnsertrpthrasp
180185190
glnaspserlysaspserthrtyrsermetserserthrleuthrleu
195200205
thrlysaspglutyrgluarghisasnsertyrthrcysglualathr
210215220
hislysthrserthrserproilevallysserpheasnargasnglu
225230235240
cys
<210>48
<211>16
<212>prt
<213>artificialsequence
<220>
<223>mab28lightchaincdr1
<400>48
argserserglnserleuvalhisserasnglyasnthrtyrleuhis
151015
<210>49
<211>7
<212>prt
<213>artificialsequence
<220>
<223>mab28lightchaincdr2
<400>49
lysvalserasnargpheser
15
<210>50
<211>9
<212>prt
<213>artificialsequence
<220>
<223>mab28lightchaincdr3
<400>50
serglnasnthrhisvalprotrpthr
15
<210>51
<211>1389
<212>dna
<213>artificialsequence
<220>
<223>mab39heavychain
<400>51
atgggatggagctgtatcatgttctttttggtagcaacagctacagatgtccactcccag60
gtccaactgcagcagcctggggctgaactggtgaagcctggggcttcagtgaagctgtcc120
tgcaaggcttctggctactccttcaccacctactggatgcactgggtgaagcagaggcct180
ggacaaggcctagagtgggttggagatattaatcctaggaacggtcgtactaactacaat240
gagaagtccaagagcaaggccacactgactgtagacatatcatccagcacagtatacatg300
caagtcagcagcctgacatctgaggactctgcggtctattactgtgcaatatggtcgggt360
gctatggactactggggtccaggaacctcagtcaccgtctcctcagccaaaacaacagcc420
ccatcggtctatccactggcccctgtgtgtggagatacaactggctcctcggtgactcta480
ggatgcctggtcaagggttatttccctgagccagtgaccttgacctggaactctggatcc540
ctgtccagtgatgtgcacaccttcccagctctcctgcagtctggcctctacaccctcagc600
agctcagtgactgtaaccacctggcccagccagaccatcacctgcaatgtggcccacccg660
gcaagcagcaccaaagtggacaagaaaattgagcccagagggtccccaacacataaaccc720
tgtcctccatgcccagctcctaacctcttgggtggaccatccgtcttcatcttccctcca780
aagatcaaggatgtactcatgatctccctgagccccatggtcacgtgtgtggtggtggat840
gtgagcgaggatgacccagatgtccatgtcagctggttcgtgaacaacgtggaagtacac900
acagctcagacacaaacccatagagaggattacaacagtactatccgggtggtcagtgcc960
ctccccatccagcaccaggactggatgagtggcaaggagttcaaatgcaaggtcaacaac1020
aaagccctcccagcgcccatcgagagaaccatctcaaaacccaaagggccagtaagagct1080
ccacaggtatatgtcttgcctccaccagaagaagagatgactaagaaacaggtcactctg1140
acctgcatgatcacagacttcatgcctgaagacatttacgtggagtggaccaacaacggg1200
caaacagagctaaactacaagaacactgaaccagtcctggactctgatggttcttacttc1260
atgtacagcaagctgagagtggaaaagaagaactgggtggaaagaaatagctactcctgt1320
tcagtggtccacgagggtctgcacaatcaccacacgactaagagcttctcccggtctccg1380
ggtaaatga1389
<210>52
<211>462
<212>prt
<213>artificialsequence
<220>
<223>mab39heavychain
<400>52
metglytrpsercysilemetphepheleuvalalathralathrasp
151015
valhisserglnvalglnleuglnglnproglyalagluleuvallys
202530
proglyalaservallysleusercyslysalaserglytyrserphe
354045
thrthrtyrtrpmethistrpvallysglnargproglyglnglyleu
505560
glutrpvalglyaspileasnproargasnglyargthrasntyrasn
65707580
glulysserlysserlysalathrleuthrvalaspileserserser
859095
thrvaltyrmetglnvalserserleuthrsergluaspseralaval
100105110
tyrtyrcysalailetrpserglyalametasptyrtrpglyprogly
115120125
thrservalthrvalserseralalysthrthralaproservaltyr
130135140
proleualaprovalcysglyaspthrthrglyserservalthrleu
145150155160
glycysleuvallysglytyrpheprogluprovalthrleuthrtrp
165170175
asnserglyserleuserseraspvalhisthrpheproalaleuleu
180185190
glnserglyleutyrthrleuserserservalthrvalthrthrtrp
195200205
proserglnthrilethrcysasnvalalahisproalaserserthr
210215220
lysvalasplyslysilegluproargglyserprothrhislyspro
225230235240
cysproprocysproalaproasnleuleuglyglyproservalphe
245250255
ilepheproprolysilelysaspvalleumetileserleuserpro
260265270
metvalthrcysvalvalvalaspvalsergluaspaspproaspval
275280285
hisvalsertrpphevalasnasnvalgluvalhisthralaglnthr
290295300
glnthrhisarggluasptyrasnserthrileargvalvalserala
305310315320
leuproileglnhisglnasptrpmetserglylysgluphelyscys
325330335
lysvalasnasnlysalaleuproalaproilegluargthrileser
340345350
lysprolysglyprovalargalaproglnvaltyrvalleupropro
355360365
proglugluglumetthrlyslysglnvalthrleuthrcysmetile
370375380
thraspphemetprogluaspiletyrvalglutrpthrasnasngly
385390395400
glnthrgluleuasntyrlysasnthrgluprovalleuaspserasp
405410415
glysertyrphemettyrserlysleuargvalglulyslysasntrp
420425430
valgluargasnsertyrsercysservalvalhisgluglyleuhis
435440445
asnhishisthrthrlysserpheserargserproglylys
450455460
<210>53
<211>7
<212>prt
<213>artificialsequence
<220>
<223>mab39heavychaincdr1
<400>53
glytyrserphethrthrtyr
15
<210>54
<211>6
<212>prt
<213>artificialsequence
<220>
<223>mab39heavychaincdr2
<400>54
asnproargasnglyarg
15
<210>55
<211>7
<212>prt
<213>artificialsequence
<220>
<223>mab39heavychaincdr3
<400>55
trpserglyalametasptyr
15
<210>56
<211>717
<212>dna
<213>artificialsequence
<220>
<223>mab39lightchain
<400>56
atgaagttgcctgttaggctgttggtgctgatgttctggattcctgcttccagcagtgat60
gttgtgatgacccaaactccactctccctgcctgtcagtcttggagatcaaccctccatc120
tcttgcaaatctagtcagagccttgtacacaataatggaaacacctatttacattggtac180
ctgcagaagccaggccagtctccaaagctcctgatctacaaagtttccaaccgattttct240
ggggtcccagacaggttcagtggcagtggatcagggacagatttcacactcaagatcagc300
agagtggaggctgaggatctgggagtttatttctgctctcaaactacacatgttcctccg360
acgttcggtggaggcaccaagctggaaatcaaacgggctgatgctgcaccaactgtatcc420
atcttcccaccatccagtgagcagttaacatctggaggtgcctcagtcgtgtgcttcttg480
aacaacttctaccccaaagacatcaatgtcaagtggaagattgatggcagtgaacgacaa540
aatggcgtcctgaacagttggactgatcaggacagcaaagacagcacctacagcatgagc600
agcaccctcacgttgaccaaggacgagtatgaacgacataacagctatacctgtgaggcc660
actcacaagacatcaacttcacccattgtcaagagcttcaacaggggagagtgttga717
<210>57
<211>238
<212>prt
<213>artificialsequence
<220>
<223>mab39lightchain
<400>57
metlysleuprovalargleuleuvalleumetphetrpileproala
151015
serserseraspvalvalmetthrglnthrproleuserleuproval
202530
serleuglyaspglnproserilesercyslysserserglnserleu
354045
valhisasnasnglyasnthrtyrleuhistrptyrleuglnlyspro
505560
glyglnserprolysleuleuiletyrlysvalserasnargpheser
65707580
glyvalproaspargpheserglyserglyserglythraspphethr
859095
leulysileserargvalglualagluaspleuglyvaltyrphecys
100105110
serglnthrthrhisvalproprothrpheglyglyglythrlysleu
115120125
gluilelysargalaaspalaalaprothrvalserilephepropro
130135140
sersergluglnleuthrserglyglyalaservalvalcyspheleu
145150155160
asnasnphetyrprolysaspileasnvallystrplysileaspgly
165170175
sergluargglnasnglyvalleuasnsertrpthraspglnaspser
180185190
lysaspserthrtyrsermetserserthrleuthrleuthrlysasp
195200205
glutyrgluarghisasnsertyrthrcysglualathrhislysthr
210215220
serthrserproilevallysserpheasnargglyglucys
225230235
<210>58
<211>16
<212>prt
<213>artificialsequence
<220>
<223>mab39lightchaincdr1
<400>58
lysserserglnserleuvalhisasnasnglyasnthrtyrleuhis
151015
<210>59
<211>7
<212>prt
<213>artificialsequence
<220>
<223>mab39lightchaincdr2
<400>59
lysvalserasnargpheser
15
<210>60
<211>9
<212>prt
<213>artificialsequence
<220>
<223>mab39lightchaincdr3
<400>60
serglnthrthrhisvalproprothr
15
<210>61
<211>1389
<212>dna
<213>artificialsequence
<220>
<223>mab39heavychainv2(t1375a-->s459t)
<400>61
atgggatggagctgtatcatgttctttttggtagcaacagctacagatgtccactcccag60
gtccaactgcagcagcctggggctgaactggtgaagcctggggcttcagtgaagctgtcc120
tgcaaggcttctggctactccttcaccacctactggatgcactgggtgaagcagaggcct180
ggacaaggcctagagtgggttggagatattaatcctaggaacggtcgtactaactacaat240
gagaagtccaagagcaaggccacactgactgtagacatatcatccagcacagtatacatg300
caagtcagcagcctgacatctgaggactctgcggtctattactgtgcaatatggtcgggt360
gctatggactactggggtccaggaacctcagtcaccgtctcctcagccaaaacaacagcc420
ccatcggtctatccactggcccctgtgtgtggagatacaactggctcctcggtgactcta480
ggatgcctggtcaagggttatttccctgagccagtgaccttgacctggaactctggatcc540
ctgtccagtgatgtgcacaccttcccagctctcctgcagtctggcctctacaccctcagc600
agctcagtgactgtaaccacctggcccagccagaccatcacctgcaatgtggcccacccg660
gcaagcagcaccaaagtggacaagaaaattgagcccagagggtccccaacacataaaccc720
tgtcctccatgcccagctcctaacctcttgggtggaccatccgtcttcatcttccctcca780
aagatcaaggatgtactcatgatctccctgagccccatggtcacgtgtgtggtggtggat840
gtgagcgaggatgacccagatgtccatgtcagctggttcgtgaacaacgtggaagtacac900
acagctcagacacaaacccatagagaggattacaacagtactatccgggtggtcagtgcc960
ctccccatccagcaccaggactggatgagtggcaaggagttcaaatgcaaggtcaacaac1020
aaagccctcccagcgcccatcgagagaaccatctcaaaacccaaagggccagtaagagct1080
ccacaggtatatgtcttgcctccaccagaagaagagatgactaagaaacaggtcactctg1140
acctgcatgatcacagacttcatgcctgaagacatttacgtggagtggaccaacaacggg1200
caaacagagctaaactacaagaacactgaaccagtcctggactctgatggttcttacttc1260
atgtacagcaagctgagagtggaaaagaagaactgggtggaaagaaatagctactcctgt1320
tcagtggtccacgagggtctgcacaatcaccacacgactaagagcttctcccggactccg1380
ggtaaatga1389
<210>62
<211>462
<212>prt
<213>artificialsequence
<220>
<223>mab39heavychain(s459t)
<400>62
metglytrpsercysilemetphepheleuvalalathralathrasp
151015
valhisserglnvalglnleuglnglnproglyalagluleuvallys
202530
proglyalaservallysleusercyslysalaserglytyrserphe
354045
thrthrtyrtrpmethistrpvallysglnargproglyglnglyleu
505560
glutrpvalglyaspileasnproargasnglyargthrasntyrasn
65707580
glulysserlysserlysalathrleuthrvalaspileserserser
859095
thrvaltyrmetglnvalserserleuthrsergluaspseralaval
100105110
tyrtyrcysalailetrpserglyalametasptyrtrpglyprogly
115120125
thrservalthrvalserseralalysthrthralaproservaltyr
130135140
proleualaprovalcysglyaspthrthrglyserservalthrleu
145150155160
glycysleuvallysglytyrpheprogluprovalthrleuthrtrp
165170175
asnserglyserleuserseraspvalhisthrpheproalaleuleu
180185190
glnserglyleutyrthrleuserserservalthrvalthrthrtrp
195200205
proserglnthrilethrcysasnvalalahisproalaserserthr
210215220
lysvalasplyslysilegluproargglyserprothrhislyspro
225230235240
cysproprocysproalaproasnleuleuglyglyproservalphe
245250255
ilepheproprolysilelysaspvalleumetileserleuserpro
260265270
metvalthrcysvalvalvalaspvalsergluaspaspproaspval
275280285
hisvalsertrpphevalasnasnvalgluvalhisthralaglnthr
290295300
glnthrhisarggluasptyrasnserthrileargvalvalserala
305310315320
leuproileglnhisglnasptrpmetserglylysgluphelyscys
325330335
lysvalasnasnlysalaleuproalaproilegluargthrileser
340345350
lysprolysglyprovalargalaproglnvaltyrvalleupropro
355360365
proglugluglumetthrlyslysglnvalthrleuthrcysmetile
370375380
thraspphemetprogluaspiletyrvalglutrpthrasnasngly
385390395400
glnthrgluleuasntyrlysasnthrgluprovalleuaspserasp
405410415
glysertyrphemettyrserlysleuargvalglulyslysasntrp
420425430
valgluargasnsertyrsercysservalvalhisgluglyleuhis
435440445
asnhishisthrthrlysserpheserargthrproglylys
450455460
- 还没有人留言评论。精彩留言会获得点赞!