番茄SlOST1基因及应用
本发明属于生物技术领域,具体涉及番茄slost1基因及应用。
背景技术:
番茄由于具有低热量、富含抗氧化的番茄红素、多种矿物质和维生素等优势,深受人们喜爱;根据世界粮农组织统计,全球番茄总产值每年接近1千亿美元,因此被称为“世界第一大蔬菜作物”。
snrk2(snf1-relatedproteinkinase2)家族是一类具有典型的丝氨酸/苏氨酸蛋白激酶保守结构域的蛋白激酶,在植物感知和应答外界环境胁迫信号以及植物激素脱落酸(abscisicacid,aba)信号转导过程中起着重要的调控作用。拟南芥基因组中有10个snrk2家族成员,但仅iii类snrk2成员的激酶活性可被aba激活,且参与渗透胁迫应答。openstomata1(ost1)是调节植物气孔运动和逆境胁迫响应的一个关键蛋白激酶。近年来,研究发现ost1不仅在aba信号转导和非生物胁迫响应中起到重要作用,它还能够参与植物生长发育的调控。然而在番茄中slost1调节植物发育和抗逆的功能仍然未知。因此解析番茄中slost1调控植株生长发育和耐旱的生物学功能,能为挖掘番茄生长发育和耐旱基因提供重要理论依据。
技术实现要素:
本发明的目的在于提供番茄(solanumlycopersicum)slost1基因以及其应用,主要是其调节植株生长发育和干旱应答的功能解析。
为实现上述目的,本发明采取如下技术方案:
发明通过生物信息学分析和鉴定了番茄中的slost1基因,slost1基因全长编码框核苷酸序列长度为1089bp,其核苷酸序列如seqidno.1所示。番茄slost1基因编码的蛋白,由362个氨基酸组成,分子量大小约为41.1kd,其氨基酸序列如seqidno:2所示。
上述番茄slost1基因编码的蛋白还可以包括将seqidn0:2氨基酸序列经过一个或多个((如1-30个;较佳地1-20个;更佳地1-10个;如5个,3个))氨基酸残基的取代、缺失或添加而形成的,且具有(1)蛋白功能的由(1)衍生的蛋白;或与(1)限定的蛋白序列有80%((较佳地90%以上,如95%,98%,99%或更高))以上同源性且具有(1)蛋白功能的由(1)衍生的蛋白。
也就是说本发明所保护的基因的功能,不仅包括上述番茄slost1基因,还包括与seqidno:1具有较高同源性(如同源性高于40%;较佳地高于50%;较佳地高于60%;更佳地高于70%;更佳地高于80%;更佳地高于90%;更佳地高于95%;更佳地高于98%)的同源基因。
此外本发明还构建番茄slost1基因编辑载体,含有上述基因的表达载体、基因编辑遗传材料以及含有所述载体的宿主细胞也落入本发明的保护范围之内。
本发明还将所述的基因编辑载体利用农杆菌转化法导入到野生型番茄。获得slost1功能缺失的基因编辑番茄植物。
本发明的最主要的目的是公开了番茄中slost1基因的应用,对slost1基因进行了克隆和鉴定,并通过构建基因敲除系,分析slost1基因在番茄生长发育和耐旱中的生物学功能。
本发明还公开了番茄slost1基因在调控植株生长发育和干旱胁迫反应中的应用,通过实验得出:
(1)番茄slost1基因编辑植物具有矮化表型;
(2)番茄slost1基因编辑植物具有晚花表型;
(3)番茄slost1基因编辑植物具有产量降低表型
(4)番茄slost1基因编辑植物对干旱敏感;
上述应用通过正常培养条件和模拟干旱实验得出结论。
通过基因敲除株系和野生株系的比较,得出该基因主要有以下方面的应用:
番茄slost1基因在提高番茄产量和/或提高抗旱性和/或提早番茄开花时间中的应用。抑制番茄slost1基因的功能在矮化番茄植株和/或延迟番茄植株开花时间中的应用。其中,矮化表现包括茎高、叶片长度和宽度均有明显降低。
本发明中,对于适用于本发明的植物没有特别的限制,只要其适合进行基因的转化操作,如各种农作物、花卉植物、或林业植物等。所述的植物比如可以是(不限于):双子叶植物、单子叶植物或裸子植物。
作为一种实施方式,所述的“植物”包括但不限于:茄科的番茄,凡是具有该基因或者与之同源的基因均适用。尤其适用于需要矮化的植物,比如蔷薇科苹果树、樱桃树;在实际的应用过程中,对于需要提高产量和/或提高抗旱性和/或提早开花时间的植物,均可以通过转基因的方式培育转入该基因的株系。
本发明中所说的“植物”包括整株植物,其亲本和子代植株以及植物的不同部位,包括种子、果实、芽、茎、叶、根(包括块茎)、花、组织和器官,在这些不同的部分均有我们目的基因或者核酸。这里所提及的“植物”也包括植物细胞、悬浮培养物、愈伤组织、胚、分生组织区、配子体、孢子体、花粉和小孢子,同样,其中每种前述对象包含目的基因/核酸。
本发明包括任何植物细胞,或任何由其中的方法获得或可获得的植物,以及所有的植物部分及其繁殖体。本专利也包含由任何前述方法所获得的转染细胞、组织、器官或完整植物。唯一的要求是子代表现出相同的的基因型或表型特征,使用本专利中的方法获得的子代特性相同。
本发明还扩展到如上所述的植物的可收获的部分,但不限于种子、叶、果实、花、茎、根、根茎、块茎和球茎。同时进一步涉及植株收获后的其他衍生物,如干燥颗粒或粉末、油、脂肪和脂肪酸、淀粉或蛋白质。本发明还涉及由相关植物获得的食品或食品添加剂。
本发明具有如下优点:
(1)本发明通过生物信息分析获得番茄slost1基因,并克隆得到番茄slost1基因。
(2)发明人利用基因编辑技术将slost1从番茄mt中敲除进行功能验证,有利于从分子机制上阐明slost1在发育和植物干旱反应中的作用,为番茄分子育种提供理论基础和基因资源。
(3)slost1基因缺失可导致番茄矮化,晚花,且产量降低。这也体现了slost1在植物生长发育过程中扮演着关键作用。
(4)通过转基因技术,为今后获得高产量的株系,提高植物抗旱性,以及使得植株提早开花具有十分重要的意义。
(5)对于一些需要矮化的植物如果树和观赏植物(减少不必要的养分消耗,以便充分利用光能,地力,提早结果、提高产量或增加观赏效果),可以通过敲除slost1同源基因的方式;对于在特殊情况下,需要推迟植物开花时间的情况,也可以采用敲除slost1同源基因的方式,因此该基因为植物育种提供一种新的途径。
附图说明
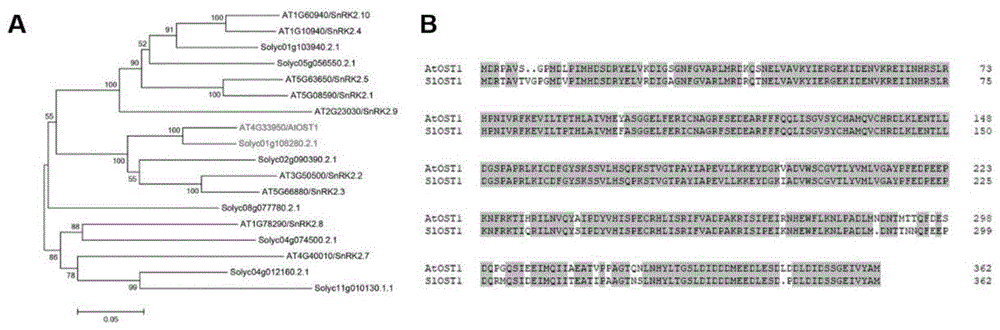
图1是拟南芥snrk2.6s和番茄snrk2.6s家族成员序列分析;
图中,(a)图是拟南芥snrk2.6s和番茄snrk2.6s家族的系统发育树;(b)图是拟南芥和番茄ost1的蛋白序列比对分析。
图2是番茄slost1基因敲除靶点sgrna的设计示意图。
图3是slost1基因敲除转基因番茄阳性苗鉴定;
图中,(a)图是t1代阳性苗的slost1基因pcr鉴定及cas9基因的pcr鉴定电泳图;(b)图是纯合slost1基因敲除株系的基因编辑类型。
图4是野生型番茄株系(wt)和slost1基因编辑番茄的发育表型及统计。
图5是野生型番茄株系(wt)和slost1基因编辑番茄的开花表型。
图中,(a)图是短日照条件下的开花情况及统计;(b)图是长日照条件下的开花情况及统计。
图6是野生型番茄株系(wt)和slost1基因编辑番茄的果实表型及统计。
图7是野生型番茄株系(wt)和slost1基因编辑番茄的干旱表型;
图中,(a)图是野生型番茄株系(wt)和slost1基因编辑番茄的干旱生长表型;(b)图是野生型番茄株系(wt)和slost1基因编辑番茄的干旱胁迫后的水分含量统计;(c)图是野生型番茄株系(wt)和slost1基因编辑番茄的叶片失水率。
具体实施方式
下面将通过具体实施例对本发明进行详细的描述。提供这些实施例是为了能够更透彻地理解本发明,并且能够将本发明的范围完整的传达给本领域的技术人员。
若未特别指明,实施例中所用技术手段为本领域技术人员所熟知的常规手段。下述实施例中的试验方法,如无特别说明,均为常规方法。如无特殊说明,所采用的试剂及材料,均可以通过商业途径获得。
除非另行定义,文中所使用的所有专业与科学用语与本领域熟练人员所熟悉的意义相同。此外,任何与所记载内容相似或均等的方法及材料皆可应用于本发明中。文中所述的较佳实施方法与材料仅作示范之用。
除非另有说明,本发明的实施将使用本领域技术人员显而易见的植物学常规技术、微生物、组织培养、分子生物学、化学、生物化学、dna重组及生物信息学技术。这些技术均在已经公开的文献中进行了充分解释,另外,本发明所采用的dna提取、系统发育树的构建、基因编辑方法、基因编辑载体的构建、基因编辑植物获得等方法,除了下述实施例采用的方法外,采用现有文献中已经公开的方法均能实现。
此处使用的“核酸”、“核酸序列”、“核苷酸”、“核酸分子”或“多聚核苷酸”术语意思是指包括分离的dna分子(例如,cdna或者基因组dna),rna分子(例如,信使rna),自然类型,突变类型,合成的dna或rna分子,核苷酸类似物组成的dna或rna分子,单链或是双链结构。这些核酸或多聚核苷酸包括基因编码序列、反义序列及非编码区的调控序列,但不仅限于此。这些术语包括一个基因。“基因”或“基因序列”广泛用来指一有功能的dna核酸序列。因此,基因可能包括基因组序列中的内含子和外显子,和/或包括cdna中的编码序列,和/或包括cdna及其调控序列。在特殊实施方案中,例如有关分离的核酸序列,优先默认其为cdna。
另外,为了对本发明技术方案更直观的理解,对于本发明涉及到的一些专业术语解释如下:
“基因编辑”,geneediting,是一种新兴的精确对生物体基因组特定靶序列进行修饰的基因功能技术。
“基因敲除”,geneknockout,是通过同源重组将外源基因定点整合入靶细胞基因组上某一确定的位点,以达到定点修饰改造染色体上某一基因的目的的一种技术。
“突变体”,mutant,是指发生突变的个体,具有与野生型不同的表型的特点。
“表达载体”,expressionvectors,是指在克隆载体基本骨架的基础上增加表达元件(如启动子、rbs、终止子等),使目的基因能够表达的载体。
“农杆菌介导转化法”,agrobacterium-mediatedtransformation,指将目的基因插入到经过改造的t-dna区,借助农杆菌的感染实现外源基因向植物细胞的转移与整合,然后通过细胞和组织培养技术,再生出转基因植株的技术。
本发明中所述的基因编辑的番茄植物,基因功能缺失番茄植株以及基因编辑突变体表达的意思一致。
实施例
发明人利用生物信息学技术在番茄基因组基础上得到slost1基因序列。通过分子克隆克隆了slost1基因的编码框序列。另外利用crispr/cas9基因编辑系统在野生型番茄中对slost1基因进行了编辑,获得了其功能缺失突变体,并对其基因编辑植株进行功能验证。
1.基因来源及分离:
基于茄科基因组公布的番茄全基因组测序(sl3.0),利用拟南芥的snrk2s的蛋白序列进行blast。在番茄中总共鉴定出8个同源基因。利用mafft在线工具(https://mafft.cbrc.jp/alignment/server/)进行序列比对,并手动进行矫正。利用mega7软件构建系统发育树(邻接树,nj)(图1a)。系统发育树结果显示,番茄中atost1的同源基因为solyc01g108280其蛋白序列高度相似(图1b),将其命名为slost1。slost1基因全长编码框核苷酸序列长度为1089bp,由362个氨基酸组成,分子量大小约为41.1kd。利用primerpremier5软件设计序列特异性引物:
f:5′-atggatcggacggcagtgacagtag-3′(seqidno:3);
r:5′-cattgcatagacaatctctccactg-3′(seqidno:4)。
野生型番茄幼苗总rna的提取按照trizol提取试剂(takara)说明进行,第一链cdna合成按照反转录试剂盒primerscripttmii1ststrandcdnasynthesiskit(takara)说明进行。以番茄的cdna为模板,使用primerstarmax高保真酶扩增(takara),退火温度为55℃。
2.番茄slost1基因的功能鉴定
为了研究slost1基因在番茄发育和抗旱中的作用,通过构建基因编辑植株来研究其功能。
2.1基因编辑植物材料的构建
2.1.1基因编辑载体的构建
使用crispr/cas9基因编辑系统,通过在线预测网站(http://www.genome.arizona.edu/crispr/crisprsearch.html),筛选番茄slost1基因的敲除靶点。针对slost1的基因组序列,在靠近起始编码区的外显子上设计两个sgrna(图2),按照载体phse401构建说明(http://www.biomedcentral.com/1471-2229/14/327)构建slost1的cas9基因编辑载体。
随后使用冻融法转化农杆菌,将0.1-1μg质粒加入50μl农杆菌感受态gv3101,冰上放置30min。随后使用液氮速冻1min,37℃热击3min,冰上放置2min,并加入无抗lb培养液28℃,250r/min振荡培养2h。取100μl菌液涂布至含有利福平和卡纳抗性的lb培养基,28℃静置培养48h。通过菌落pcr鉴定,即可获得阳性克隆。
2.1.2基因编辑植物材料的转化
挑选成熟野生型番茄种子使用10%的次氯酸钠消毒后播种到1/2ms培养基上,25℃光照培养箱培养6-8天。随后,选取幼苗子叶和下胚轴,切割至适当大小,放入共培养培养基预培养1天。
将上述所获得的阳性克隆,摇至od600=0.8-1.0,5000rpm/min离心15分钟后,使用重悬液稀释到od600=0.2。在50ml离心管中,旋转侵染已切割的幼苗15-25分钟。侵染结束后,蒸馏水清洗5-10遍,晾干后共培养培养基25度暗培养2天。
暗培养2天后,将已切割的幼苗移至脱分化培养基上25℃光照培养箱培养20天左右,再移至再分化培养基直至长出幼芽。
从外植体切下幼芽,用镊子转移至生根培养基,生根后,即可获得t1代slost1的基因敲除番茄植株。
2.2基因编辑植物材料的阳性鉴定
根据番茄slost1基因组序列,在sgrna靶位点附近设计特异性的pcr引物:
f:5’-caactaccaactgtactcaatcctgt-3’:
r:5’-gaccagtatcgaacgactatcttga-3’。
电泳检测发现很多t1代植物出现两个条带或者明显变小的条带(图3a),结合cas9引物扩增的结果进行分析,表明对slost1的基因编辑是成功的。经过对t2代转基因材料的slost1基因的突变测序分析,我们获得了两个纯合的slost1的基因敲除植株#1和#2,突变类型分别是22个碱基的缺失和220个碱基的大片段缺失(图3b)。
2.3番茄slost1基因编辑突变体表型分析
在获得纯合的slost1的基因编辑植株后,首先分析了它们生长发育表型,如图4所示,与野生型番茄(wt)相比,slost1基因编辑植物明显矮小,叶片长度和宽度均有明显降低,而茎高尤为明显,从野生型植株的7.1cm分别降至4.2cm及2.2cm。
slost1基因敲除系表现出晚花表型,如图5所示。在短日照条件下,野生型开放5朵花仅需40.4天,而slost1基因编辑植物分别需要44.4天,46.4天。在长日照条件下,野生型开放5朵花需39.2天,而slost1基因编辑植物则需要41.8天和42.6天。同时,slost1基因编辑植物表现出产量下降,据统计,在正常培养条件下生长50天后,野生型植株存在约17.8个果实,而slost1基因编辑植物分别存在3.7,3.2个果实;且slost1基因编辑植物果重也低于野生型。
除此之外,还有明显干旱胁迫反应的表型。如图7所示slost1基因编辑番茄与野生型番茄相比,在干旱胁迫后萎焉程度更加严重(图7a)。在复水后,野生型番茄的叶片大多可以吸水恢复,但是slost1基因编辑番茄的叶片大多不能恢复;我们测量了野生型和slost1基因编辑番茄在水分正常条件和干旱处理后的叶片相对含水量变化,数据显示在正常条件下,野生型和基因编辑植物的相对含水量相当,但是在干旱处理后,基因编辑植物的相对含水量明显低于野生型植物(图7b);叶片离体失水速率进一步表明基因编辑植物的叶片失水速率比野生型显著加快(图7c)。总之,以上数据表明番茄slost1基因在番茄植物生长发育和干旱胁迫应答起着正调控的作用。
以上所述之实施例,只是本发明的较佳实施例而已,仅仅用以解释本发明,并非限制本发明实施范围,对于本技术领域的技术人员来说,当然可根据本说明书中所公开的技术内容,通过置换或改变的方式轻易做出其它的实施方式,故凡在本发明的原理上所作的变化和改进等,均应包括于本发明申请专利范围内。
序列表
<110>河南大学
<120>番茄slost1基因及应用
<130>2021
<160>4
<170>siposequencelisting1.0
<210>1
<211>1089
<212>dna
<213>solanumlycopersicum
<400>1
atggatcggacggcagtgacagtaggaccaggtatggacgtaccgatcatgcacgatagc60
gatagatatgaacttgtacgggatattggtgctgggaattttggtgttgcaaggcttatg120
agagataggcagactaatgaacttgttgctgttaagtacatcgagagaggtgagaagatt180
gatgaaaatgttaagagagaaatcatcaaccatagatcattgaggcatcctaacatagtc240
agatttaaagaggtcatactcacaccaactcatttggctattgtgatggaatttgcatct300
ggaggggagctgtttgagcgcatatgtaatgctggtcgttttagcgaggatgaggcacgg360
tttttcttccaacaactcatatcaggggtcagttattgtcatgctatgcaagtgtgccat420
agagacttgaaattagagaatacattactggatggaagtcctgcaccaaggttaaagatt480
tgtgattttggatattctaagtcctcggtgttgcattcacaacctaagtcaactgttggt540
acacctgcatatattgctccagaggtgttattgaagaaagaatatgacggaaagattgca600
gatgtctggtcttgtggagtgactttgtatgtcatgctggtgggagcatacccttttgaa660
gacccagaggaacccaaaaattttcggaagacaatacagcgaatcttgaacgtacaatat720
tcgattcctgattatgtacatatctctcctgaatgtcgtcatctaatatcaaggattttt780
gttgcagatcctgcaaagaggatatcaatccctgagatcaagaaccatgagtggttcttg840
aagaaccttcctgcagatctcatggataatactacaaacaaccagtttgaggagccagat900
caacgtatgcagagcattgacgaaatcatgcagataataactgaggccaccattcctgct960
gctgggaccaacagccttaatcattacctcactggaagcttggacattgacgatgacatg1020
gaagaagatttggagagtgatcctgacctcgatatcgatagcagtggagagattgtctat1080
gcaatgtaa1089
<210>2
<211>362
<212>prt
<213>solanumlycopersicum
<400>2
metaspargthralavalthrvalglyproglymetaspvalproile
151015
methisaspseraspargtyrgluleuvalargaspileglyalagly
202530
asnpheglyvalalaargleumetargaspargglnthrasngluleu
354045
valalavallystyrilegluargglyglulysileaspgluasnval
505560
lysarggluileileasnhisargserleuarghisproasnileval
65707580
argphelysgluvalileleuthrprothrhisleualailevalmet
859095
gluphealaserglyglygluleuphegluargilecysasnalagly
100105110
argphesergluaspglualaargphephepheglnglnleuileser
115120125
glyvalsertyrcyshisalametglnvalcyshisargaspleulys
130135140
leugluasnthrleuleuaspglyserproalaproargleulysile
145150155160
cysasppheglytyrserlysserservalleuhisserglnprolys
165170175
serthrvalglythrproalatyrilealaprogluvalleuleulys
180185190
lysglutyraspglylysilealaaspvaltrpsercysglyvalthr
195200205
leutyrvalmetleuvalglyalatyrprophegluaspprogluglu
210215220
prolysasnphearglysthrileglnargileleuasnvalglntyr
225230235240
serileproasptyrvalhisileserproglucysarghisleuile
245250255
serargilephevalalaaspproalalysargileserileproglu
260265270
ilelysasnhisglutrppheleulysasnleuproalaaspleumet
275280285
aspasnthrthrasnasnglnpheglugluproaspglnargmetgln
290295300
serileaspgluilemetglnileilethrglualathrileproala
305310315320
alaglythrasnserleuasnhistyrleuthrglyserleuaspile
325330335
aspaspaspmetglugluaspleugluseraspproaspleuaspile
340345350
aspserserglygluilevaltyralamet
355360
<210>3
<211>25
<212>dna
<213>solanumlycopersicum
<400>3
atggatcggacggcagtgacagtag25
<210>4
<211>25
<212>dna
<213>solanumlycopersicum
<400>4
cattgcatagacaatctctccactg25
- 还没有人留言评论。精彩留言会获得点赞!