一种利用细胞器基因组的同源模块进行系统发生分析的方法与流程
本发明涉及一种利用细胞器基因组的同源模块进行系统发生分析的方法,属于系统进化分析领域。
背景技术:
细胞器在生物体内主要的功能是负责光合作用和能量的代谢过程,细胞器基因组因多拷贝、母系遗传、突变速率高、基因数目多、易测序等特点,逐步走入系统发生分析的视野。在物种的起源进化分析中,细胞器基因组的信息不仅可以帮助我们认识系统进化关系还可以明确其物种起源问题。而在利用细胞器基因构建系统演化关系的过程中,准确的多序列比对是相关系统演化关系能够被正确推断的基石。在生物信息学高速发展的今天,许多准确、快速、符合生物学理论的比对工具被开发出来;得益于这些工具,使得生物学研究得以在更深层次更高层面进行开展。但是随着高通量测序技术的发展,测序数据爆炸式增长,对多序列比对提出了更高的要求;如何对进行大数据的快速多序列比对,得到更优更准确的结果,是多序列比对软件开发的重要目标。
在基于细胞器的系统演化关系中,目前普遍的做法是挑取多个物种的共有基因进行建树,需要将每一组直系同源基因进行比对,随后联合在一起,但随着系统发生分析研究物种的增多,使得需要进行手动比对的基因数目则呈几何倍数增长,这将大大拖慢分析进程,整个过程显得费时费力。
鉴于以上种种弊端,我们探索出一种基于细胞器基因组的快速比对流程,使得细胞器基因组系统发生分析过程变得快速、容易,加快分析进程。单基因数据的系统发育发析已是司空见惯,但由于单基因序列所包含的信息有限,不足以解决研究者感兴趣所有分类单元间的系统发育关系,例如许多研究表明不同的基因具有不同的进化速率,例如叶绿体基因rbcl的进化速率是核基因18srdna的1.4倍左右。在这种情况下,通过联合不同基因的数据集,可以增加系统发育信号数量,从而增强对系统发育分类单元的解析力,也就说多基因串联在一起可以消除单个基因的进化速率的差异,利用多基因甚至是整个基因来构建系统进化树的准确性要比单基因或者多个基因的可靠性高。因此,在进化关系的研究中,随着物种基因组信息(核基因组和细胞器基因组)的完善,对物种的系统衍化关系会有更加深入的了解。
目前,随着测序技术的飞速发展,涵盖各个门类物种的细胞器基因组逐渐被完善,因此基于细胞器基因组构建的系统演化关系解决了不少系统分类学的难题。然而与之对应的结果是随着数据量的加大(包括物种和基因数据),导致工作量越来越大,例如多基因联合建树,需要手动将多个物种每一组直系同源基因单独进行进行比对,随后将多个基因联合在一起(叶绿体基因组基因往往高达100个以上),这将大大拖慢分析进程,整个过程显得费时费力。
技术实现要素:
为了克服现有技术中系统发生分析过程中的处理过程繁琐,处理时间长的技术不足,本发明提供一种利用细胞器基因组同源模块的进行系统发生分析的方法,其主要包括以下个技术流程:(1)利用mauve提取不同物种细胞器基因组的同源模块;(2)同源模块序列的修剪和串联;(3)共线性模块数据集最适模型的检测;(4)系统演化树的构建。
本发明所述的利用细胞器基因组的同源模块进行系统发生分析的方法,其具体包括如下步骤:
1)利用mauve提取不同物种细胞器基因组的同源模块:在genbank中下载需要进行系统演化关系的物种的细胞器基因组,将下载好的细胞器基因组构建成一个本地数据库;使用mauvealigner中导入本地数据库中全部物种的细胞器基因组,利用progressivemauve检测不同物种细胞器基因组间的结构变异,根据比对结果划分出共线性模块;对于划分出的共线性模块进行统计,将共线性模块利用脚本从比对结果序列中全部抽取出来;
2)同源模块序列的修剪和串联:通过gblocks使用保守的序列修剪策略对共线性模块进行序列修剪,将未提取到系统发生信息的序列模块舍弃;将修剪后的共线性模块合并得到比对序列,并报告每个模块在最终合并序列上的分布;
3)共线性模块数据集最适模型的检测:基于共线性模块构建的比对序列,按照每个共线性模块在最终合并序列的分布进行序列切割和模型选择,并确定最优核酸替代模型和序列切割策略;
4)系统演化树的构建:通过快速比对的共线性模块的串联序列,利用mrbayes构建多个物种的系统发生关系。
本发明提供了一种基于细胞器基因组的快速比对流程,既能包含完整的蛋白编码基因也包含非编码区域,同时也节省了大量的时间成本,同时也具有较高的可靠性和准确性。具体地,本发明所述的系统发生分析方法可以有效地解决:
1、细胞器全基因组的比对速度的问题;
2、细胞器基因组的信息(包括编码区和非编码区)的全面覆盖信息;
3、快速解决不同分类单元中保守区域的比对问题;
4、快速明确不同分类单元中保守基因种类的问题。
总之,通过这种基于细胞器基因组的快速比对流程的方法,可以快速实现不同分类单元的的同源模块的比对,更加全面地涵盖细胞器基因组信息,更加准确地推断和还原物种的系统演化关系。
附图说明
图1为本发明同源模块的实验流程图。
图2为同源模块比对的可视化结果图。
图3为藻类线粒体系统进化树图,其中左图为快速比对流程选取序列构建的系统进化树,右图为共有基因的序列构建的系统进化树。
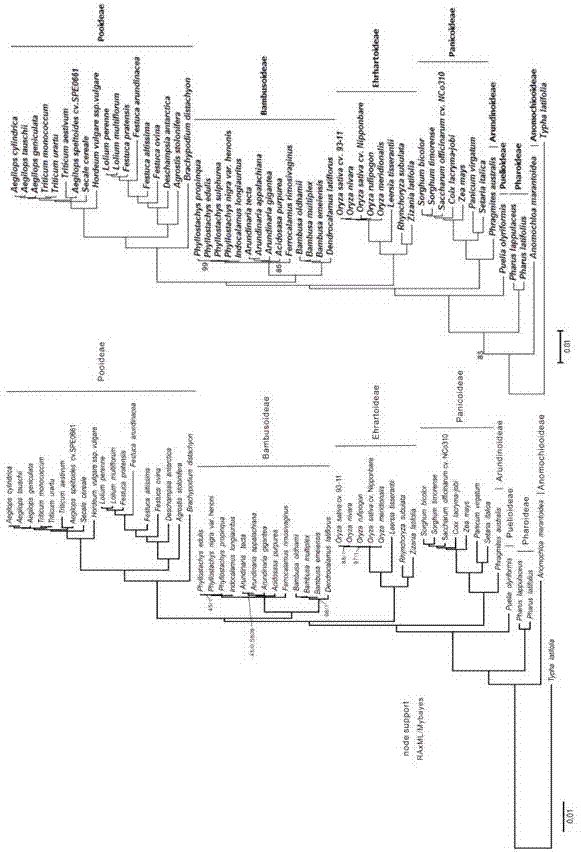
图4为植物叶绿体系统进化树图,左图为快速比对流程选取序列构建的系统进化树,右图为共有基因的序列构建的系统进化树。
图5为啮齿类动物线粒体系统进化树图,左图为快速比对流程选取序列构建的系统进化树,右图为共有基因的序列构建的系统进化树。
具体实施方式
以下通过具体实施例进一步描述本发明,但本领域技术人员应能知晓,所述实施例并不以任何方式限定本发明专利保护的范围。
实施例本发明利用细胞器基因组的同源模块进行系统发生分析的方法
本发明细胞器基因组系统发生分析中同源模块的快速构建步骤的主要包括以下个技术流程:(1)利用mauve提取不同物种细胞器基因组的同源模块;(2)同源模块序列的修剪和串联;(3)共线性模块数据集最适模型的检测;(4)系统演化树的构建。图1为本发明同源模块的实验流程图。本发明所述的利用细胞器基因组的同源模块进行系统发生分析的方法,其具体包括如下步骤:
1.利用mauve提取不同物种细胞器基因组的同源模块
a)在genbank中下载需要进行系统演化关系的物种的细胞器基因组,比对数据格式支持fasta、gb、gbk、fas等主流核酸序列格式;
b)将下载好的细胞器基因组构建成一个本地的数据库;
c)使用mauvealigner中导入多物种的细胞器基因组,利用progressivemauve检测不同细胞器基因组间的结构变异,并根据比对结果划分出共线性模块。这一步可解决序列结构变异无法直接比对的问题,同时也就避免了单基因比对这一重复和冗余过程;
d)对于划分出的共线性模块进行统计,将共线性模块利用脚本从比对结果序列中全部抽取出来。
2.同源模块序列的修剪和串联
a)接下来进行共线性模块序列的修剪,即提取有系统发生信息的位点:序列修剪采用的是gblocks,使用最保守的序列修剪策略。序列修剪后,抛弃没有提取到系统发生信息的序列模块;将修剪好的序列模块进行合并,并报告每个模块在最终合并序列上的分布。
3.共线性模块数据集最适模型的检测
基于共线性模块构建的比对序列,其最小序列单位为一个共线性模块,我们可以对每个共线性模块,按照它在最终序列的分布,进行序列切割和模型选择,以选出最优核酸替代模型和序列切割策略。
4.系统演化树的构建
以上通过快速比对的同源模块的串联序列,利用mrbayes构建多个物种的系统发生关系。
本发明快速比对流程的系统进化分析准确度验证。
目前并不存在为构建细胞器基因组比对序列构建的软件。本流程的构建将整个细胞器基因组系统发生分析的时间缩短到十几分钟到几小时。为了验证我们构建流程的准确性和广适性。我们将已发表文章中利用蛋白编码序列构建的系统进化树(目前普遍的做法)和本发明的快速比对后的序列构建的系统进化树进行准确性验证。藻类细胞器基因组系统演化关系的验证,验证结果如图3所示。植物细胞器基因组系统演化关系的验证,验证结果如图4所示。动物细胞器基因组系统演化关系的验证,验证结果如图5所示。
由以上验证实验可知,整个快速比对的流程提取的序列在构建系统进化树时可以准确的进行物种的系统演化关系的推断,而且适用范围广泛,极大的节约时间成本。
- 还没有人留言评论。精彩留言会获得点赞!