靶向RNA的化学小分子药物计算机筛选方法与流程
本发明涉及一种药物计算机筛选方法,尤其涉及一种靶向rna的化学小分子药物计算机筛选方法。
背景技术
基因(dna)是遗传物质的存储者,它负责指导建造蛋白,蛋白被认为是最终执行具体生物学功能的分子,而rna则被认为是连接dna和蛋白的中间分子。因此,传统上,人们更多的集中在研究蛋白(包括蛋白编码dna)上,对rna研究不多,重视不够。传统的药物研发亦是以靶向蛋白为主,如drugbank数据库中收录的95%以上的药物其作用靶点是蛋白,但是,绝大多数蛋白并不具备成靶性(drugaabletarget),截止到目前能够成靶的蛋白只有400个左右,因此靶向其他种类分子的药物研发对于疾病防治已是当务之急。近年,随着人类基因组计划和encode(dna元件百科全书)计划的实施,人们吃惊的发现人体中能够编码蛋白的dna只占全部dna的2%左右,其余98%的dna大部分能转录成rna,但并不翻译成蛋白,因此叫做非编码rna(noncodingrna,ncrna)。随着rna-seq等高通量技术的飞速发展,目前已发现大量非编码rna,如人体内已发现mirna(微小rna,microrna)近4万个,长非编码rna(longnoncodingrna,lncrna)近10万个。研究表明这些rna分子具有重要的生物功能,和疾病关系密切,即使信使rna其功能也不限于沟通dna和蛋白,而是在rna层面有各种各样的重要功能,人们开始意识到rna正在成为疾病干预的潜在关键靶点,靶向rna的药物研发正在引起广泛的关注。
具有成药潜能的一大类靶向rna的分子是rna或dna(为和“rna靶点”区分,在此称为核酸),如小干扰rna(smallinterferingrna,sirna)、反义寡核苷酸(antisenseoligonucleotide,aso)、mirna、核酸适体(aptamer)等等。比如,罗氏(roche)公司的针对人肝脏特异mirnamir-122的用于治疗丙肝的核酸药物mirvirasen已开始了2期临床试验。然而,核酸药物天然有一些缺点,如脱靶效应(off-target)、外源大分子易引起机体免疫反应、稳定性差和进细胞困难等。这些缺点尤其是后两点,严重阻碍了核酸的成药性。例如,sirna进入血液循环后,短短几分钟就会被降解掉,稳定性极差,这是核酸成药的主要障碍之一。此外,细胞经过数亿年的进化,为了抵御外部不良物质的侵害,进化出了双层脂质的细胞膜,阻碍了外源性核酸进入细胞,从而难以调控靶rna,这是核酸成药的另一主要障碍。因此,除了继续深入研究基于核酸的rna靶向药物,国际科学界亦开始把眼光投向其他可能的靶向rna的策略,其中化学小分子开始展露头角。药物研发中的化学小分子指分子量小于900道尔顿的有机分子。
化学小分子稳定性好、容易进入细胞,极大克服了核酸药物的上述缺陷,并且历史上小分子在靶向rna方面曾取得过成功,如链霉素(streptomycin)和四环素(tetracycline)就以细菌的rna为靶。但目前严重阻碍本领域发展的一大瓶颈是靶向rna的化学小分子筛选计算方法不足。国际上包括申请人课题组在该领域已有尝试。如基于mirna转录组或mrna转录组的mirna-环境因子(绝大多数为化学小分子)生物信息学数据库和预测平台mirenvironment,小分子与mirna关联数据库sm2mir以及预测算法,但这些方法本质上预测的都是mirna和小分子“功能”上的关联性,并不是真正的靶向mirna的药物预测。kuntz实验室虽然尝试将“蛋白-小分子”对接软件“dock6.0”应用于“rna-小分子”对接,但是该方法亦存在如下重大缺陷:1)它依赖于rna三级结构,但是大多数rna三级结构未知,并且rna三级结构与蛋白三级结构不同,前者刚性更差,柔性更强;2)dock6是设计用于“蛋白-小分子”对接,rna理化性质、力场参数和蛋白相去甚远,因此dock6并不能套用于rna。最近disney实验室首先用生物学手段确定了一些带有发卡环(hairpin)和脊(bulge)的小rna片段结合的化学小分子,然后利用这种相互作用数据,他们设计了预测算法inforna,但这种算法只适用于小的rna片断,不适合大的rna分子,而后者数量更多,更复杂,作用机制和小rna亦不同。另外,因为inforna数据、程序和服务器均没有公开,其准确性如何亦不清楚。综合以上分析可知,当前的这些初步尝试,均存在不足,靶向rna药物筛选问题依然任重道远,需要更新的计算方法补充进来。
根据以上分析可知,直接靶向分子药物的筛选,分子的空间结构以及力场似乎不可或缺,目前已知空间结构的rna分子则少之又少,rna力场亦不清楚,这似乎是一对难以调和的矛盾。
技术实现要素:
本发明的目的在于针对上述已有技术的不足,提供一靶向rna的化学小分子药物计算机筛选方法,该方法利用rna序列来源信息和化学小分子理化性质构建随机森林模型,能够更便捷有效地帮助筛选靶向rna的化学小分子。本发明的化学小分子指分子量小于900道尔顿的有机分子。
本发明的目的是通过以下技术方案实现的:
一种靶向rna的化学小分子药物计算机筛选方法,包含以下步骤:(1)收集和整理数据集,(2)挖掘用于训练预测方法的特征,(3)创建预测方法与模型,(4)验证预测方法与模型。
优选的,所述步骤收集和整理数据集,步骤包括:
(a)从pdb(proteindatabank)数据库检索并获取仅由rna和小分子组成的结构,并从这些结构当中提取相应的信息,包括rna与小分子相互作用的情况和rna与小分子相互作用的具体位置,作为训练数据集;
(b)从smmrna(smallmoleculemodulatorsofrna)数据库以及文献报道中搜集pdb数据库之外的rna与小分子相互作用的数据,作为测试数据集。
优选的,所述的挖掘用于训练预测方法的特征,包括:
(a)提取rna包括序列、结构,功能的相关特征;
(b)计算小分子的理化性质,包括氢键受体数量(numberofhydrogenbondacceptors,hba)、氢键供体数量(numberofhydrogenbonddonors,hbd)、辛醇/水分布系数(octanol/waterpartitioncoefficient,logp)、摩尔折射率(molarrefractivity,mr)、分子量(molecularweight,mw)、拓扑极性表面面积(topologicalpolarsurfacearea,tpsa)。
优选的,所述的相关特征包括:核苷酸类别、功能位点、核苷酸距离和nds(nucleotidedistancesum)曲线、核苷酸频率和配对状态。
所述的创建预测方法与模型,步骤包括:采用均衡随机森林(balancedrandomforest,brf)模型创建rna-化学小分子相互作用预测的计算方法。
由于小分子通常只是结合于rna的局部区域,首先将rna转换为片段,不过由此产生的片段里面小分子结合的片段(正样本)要远少于未结合的片段(负样本),因此采用均衡随机森林(balancedrandomforest,brf)模型创建rna-化学小分子相互作用预测的计算方法。
优选的,采用均衡随机森林模型创建rna-化学小分子相互作用预测的计算方法,步骤包括:将训练数据集中的负样本分成多份以缩小每一份负样本与正样本的数量差距,并分别与正样本匹配进行模型训练,并汇总这些模型的输出结果。
所述的验证预测方法与模型,包括:对经过步骤(3)得到的模型进行性能评测。
优选的,所述的性能评测包括:使用训练数据集进行交叉验证,和/或使用测试数据集进行独立验证。
优选的,所述的性能评测包括:选择5个阳性预测结果和5个阴性预测结果进行生物学验证。
本发明还采用了如下方案,靶向rna的化学小分子药物计算机筛选方法在高通量筛选平台的应用。
本发明还采用了如下方案,靶向rna的化学小分子药物计算机筛选方法在以rna为靶向化合物计算机筛选的应用。
本发明还采用了如下方案,靶向rna的化学小分子药物计算机筛选方法在pdb数据库的应用。
本发明还采用了如下方案,所述的靶向rna的化学小分子药物计算机筛选方法在以下领域的应用:在高通量筛选平台中的应用;在以rna为靶向化合物计算机筛选的应用;和/或在pdb数据库的应用;和/或在smmrna数据库的应用;和/或在基于mirna的环境因子研发平台mirenvironment的应用;和/或在靶向药物筛选中的应用;和/或在重大疾病防治中的应用。
本发明还采用了如下方案,靶向rna的化学小分子药物计算机筛选方法在靶向药物帅选中的应用。应用本方法,我们预测得到了靶向lncshgl的化学小分子山奈酚(kaempferol)和槲皮素(quercetin)。
本发明还采用了如下方案,靶向rna的化学小分子药物计算机筛选方法在在重大疾病防治中的应用。我们前期发现一个新的lncrna,lncshgl,在肝脏糖脂代谢中起关键作用,在脂肪肝、糖尿病等代谢性疾病干预的新的药物靶点。利用本方法,我们预测到山奈酚(kaempferol)和槲皮素(quercetin)结合lncshgl,这两个化学小分子,是脂肪肝和糖尿病的潜在防治药物。
本发明的有益效果为:
本发明针对rna这一新兴疾病干预靶点的化学小分子药物筛选这一重要问题,由于到目前rna空间结构数据少、结构柔性、力场未知等局限,在分析rna序列特征和小分子理化性质的基础上,创建基于机器学习(本发明用随机森林法)的靶向rna的化学小分子筛选的计算方法。本发明可为靶向rna的化学小分子的计算机筛选;基于rna的重大疾病防治提供新的解决策略。
本发明为靶向rna的药物筛选提供新思路、新策略和新方法。
附图说明:
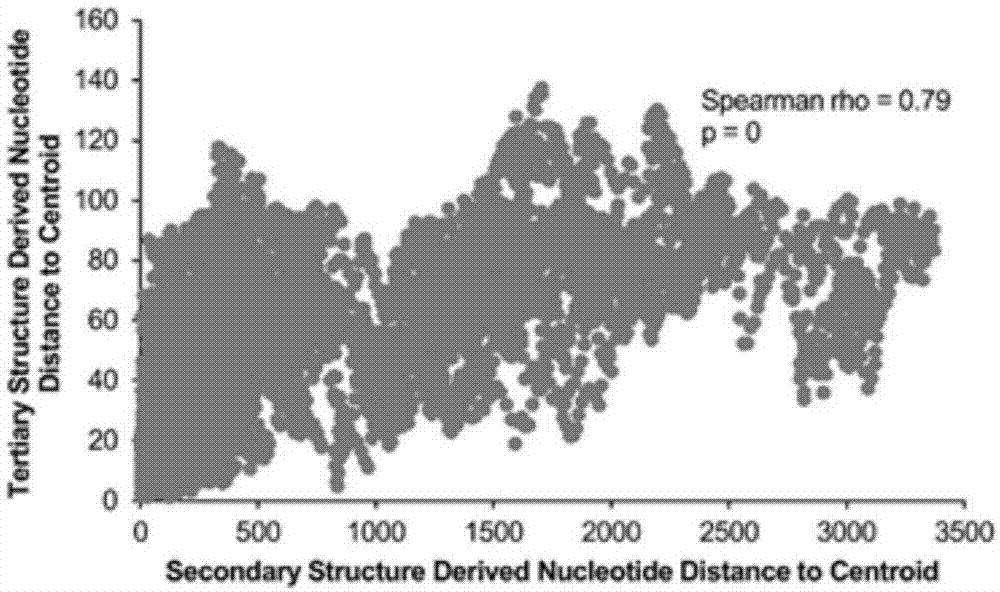
图1.由rna序列(先用序列预测二级结构,再计算距离)计算的核苷酸距离与空间结构计算的核苷酸距离高度相关;
图2.ak098656在血管平滑肌细胞表达具有高特异性;
图3.转入ak098656基因后,大鼠的收缩压(a)和舒张压(b)均显著升高;
图4.所创建的计算方法交叉验证的结果(a)和在独立smmrna和文献来源的独立数据集上的测试结果(b)。
具体实施方式
以下实施例和实验例用于说明本发明,但不用来限制本发明的范围。下面结合具体实施例和实验例对本发明作进一步说明。
实施例1:
1、收集和整理rna-化学小分子相互作用数据
1)训练数据集
检索pdb数据库中仅由rna链和小分子组成的结构,下载的pdb结构数据经过清理后作为训练数据集的来源。如果pdb结构当中含有的小分子全部为金属离子或者是结构生物学研究中常用的缓冲液中的溶剂分子,或者所含的rna链长度不超过20,则不予保留。接下来,从保留下来的pdb结构中提取rna-小分子相互作用的信息。因为4.0埃(angstrom)大约是最弱的氢键和最强的范德华力(vanderwaalsforce)的转折点,因此采取4.0埃作为判断小分子和rna相互作用的阈值。如果核苷酸与小分子的原子之间的最近距离小于4.0埃,则认为两者存在相互作用。由于到作为训练数据集来源的pdb结构中鲜有无相互作用的rna-小分子对,文中先将所有的pdb结构中涉及的小分子整理出来计算它们的理化性质之间的欧氏距离,接着,根据每个pdb结构中与所含rna链相互作用的一个或多个小分子,将剩余的小分子根据与结构中所含各个小分子之间理化性质的欧氏距离分别进行排序,为了尽量降低生成假阴性rna-小分子对的可能性,选取理化性质的欧氏距离排序在第80到第90分位数之间的小分子的交集用于人为地产生“无相互作用”的rna-小分子相互作用对。
2)独立测试数据集
从文献手动收集rna-小分子相互作用以及可能存在的无相互作用的rna-小分子对作为测试数据集,从smmrna数据库获取未包括在pdb数据库的新的rna小分子相互作用数据。
2、计算rna相关特征及小分子理化性质
一方面,本文从序列、结构和功能等多个角度提取rna相关的特征,具体地来说,对于每个核苷酸,依次分别提取以下特征:
(1)该核苷酸本身种类(a、u、c、g和n);
(2)是否与另外的核苷酸形成配对;
(3)是否为申请人前期提出的rsite2算法所预测的功能位点;
(4)该核苷酸在二级结构中标准化的几何距离打分nnds值:
nnds=∑dist(nti-ntj)/∑dist(ntcentroid-ntj)
其中nti,ntj,ntcentroid分别是待测核苷酸,rna中任意核苷酸以及rna中心的坐标向量,计算核苷酸距离时采用欧氏距离。
随后,由于对rna进行片段化处理,上述特征中的(1)-(3)会被置入相应片段的向量,而(4)被转换为平均值赋给对应片段,片段与小分子是否相互作用根据位于片段中心位置的核苷酸是否与小分子相互作用确定,超出rna序列两端的片段中的缺失值默认分别使用(1)n(2)否(3)否(4)第一个或最后一个核苷酸的标准化nds值进行填充。此外,还在各个片段范围内统计各个核苷酸的频率以及核苷酸三联体的频率。用于判断核苷酸配对状态的rna二级结构产生自多个途径,包括利用rnapdbee(http://rnapdbee.cs.put.poznan.pl/)从pdb结构中提取,根据相关文献报道手动注释以及使用rnafold对rna序列进行预测。
另一方面,化学小分子结构文件包括从pdb数据库中直接获取的结构数据格式(structuredataformat,sdf)文件和从ncbi的pubchem数据库(https://pubchem.ncbi.nlm.nih.gov/)中检索得到的简化分子线性输入规范(simplifiedmolecularinputlineentryspecification,smiles)格式文件。随后,运用openbabel软件包根据上面所得化学小分子结构文件计算出其理化性质,包括氢键受体数量hba、氢键供体数量hda、辛醇/水分布系数mw、摩尔折射率mr、以及拓扑极性表面面积tpsa等。这些指标可以直接计数获得或者通过已知的小分子片段的理化性质整合而成。比如对于含有n种片段的小分子,可以查询每种片段的tpsa,并根据片段数目加权求和算得:
3、创建rna-化学小分子相互作用预测方法
1)计算rna-化学小分子相互作用倾向性分数
由于到rna只是局部和化学小分子相互作用,申请人提出将rna片段化处理的思路。因此,输入模型的rna相关特征是基于rna序列片段得到的,模型直接对rna序列片段是否与化学小分子相互作用进行预测,还需要将片段水平的预测结果进一步整合到rna分子水平,对rna分子与化学小分子相互作用的倾向性做出整体的评估。为此,先找出rna序列中被预测可能和化学小分子存在相互作用的片段,计算出这些片段包括自身及其左右至多各5个邻近片段当中被预测为可能与化学小分子相互作用的片段所占的比例,然后根据该比例对这些片段进行排序,取其中的至多前5个,计算出它们的比例平均值和片段中心序列距离平均值的比值,可以推知,该比值越高意味着化学小分子可能作用的rna序列片段在rna分子上分布得越密集,在此将此相互作用倾向性分数记作drip(drug-rnainteractionpredictor)分数。
2)创建rna-化学小分子相互作用预测模型
由于数据集中与小分子无相互作用的片段要远远多于有相互作用的片段,采用将负样本分成多份分别与正样本匹配的均衡随机森林(balancedrandomforest,brf)模型,另外在尽可能缩小每一份中负样本与正样本的数量差距的同时为避免过度增加模型复杂度,限制负样本最多被分为10份。随机森林模型使用r包randomforest构建。
随机森林是一种系宗分类模型(ensembleclassifier),它实际上由若干决策树集合而成,一棵决策树训练自一部分样本,其中从根节点到叶子节点之间的路径指出了不同特征的取值条件θ(xi)应该如何按照权重w组合,以实现对选出的这一部分样本的分类。最终,随机森林模型通过整合一系列决策树,实现对分类向量y的预测:
鉴于模型中整合的特征以及构建流程中可调整的参数较多,采用分步的方式予以优化。首先,因为对rna进行了片段化处理,测试不同rna序列片段长度对模型性能的影响;调整好rna序列片段长度之后,进行特征的筛选。在训练好的随机森林模型中,单个特征的重要性打分以基尼重要性(giniimportance,gi)表示,它将每个树中对特征的分割(split)方式κ造成的分类优度以基尼不纯性i(κ)表示,然后汇总所有树t中的基尼不纯性,以得到特征总体的重要性打分:
测试了不同特征组合对模型性能的影响,这些特征组合中包括有保留全部特征,分别剔除每一组rna相关特征,以及将小分子理化性质用分子量进行标准化然后保留或剔除分子量;选好特征组合之后,紧接着对数据集中的正负片段比例进行调整,由于每个小分子对应的正负片段比例各不相同,可能导致模型预测结果产生偏倚,通过操作未与小分子相互作用的负片段将小分子对应负片段与正片段的比例控制到同一水平,从10比1开始一直翻倍至640比1为止,对于小分子对应负片段的数量不足的情况,缺口由从现有的负片段当中随机采样并随机突变其中一个核苷酸所产生的伪负片段填补,人为制造的伪负片段除序列外其余特征与原负片段的保持一致,而对于小分子对应负片段的数量有余的情况,使用cd-hit工具在负片段内部以及负片段与正片段之间将这些rna序列片段进行聚类,然后依据聚类结果优先保留与正片段相似的负片段同时减少负片段内部的冗余,尽量保证保留下的负片段的代表性;随后,在控制好小分子对应的正负片段比例的条件下,再一次比较不同的rna序列长度对模型性能的影响;最后,设置随机森林模型中分类树的数量为从100开始每次递增100直至1000,对分类树的个数进行比较选择。
4、验证所创建的rna-化学小分子相互作用预测方法
在训练数据集上进行5折交叉验证,主要采用敏感性(sensitivity),特异性(specificity)和马修相关系数(matthewscorrelationcoefficient,mcc)评价预测表现,这些评价指标的定义如下:
由于这些评价指标依赖于特定分类器阈值,为全面评估预测器,我们还将绘制roc曲线,并采用曲线下面积auc值进行评价。
在独立测试数据集上运行所创建方法,评估其准确性。
从drugbank数据库(https://www.drugbank.ca/)下载所有的药物小分子结构数据,将优化过程中设置了不同参数的模型应用于在drugbank药物库中筛选可能同ak098656相互作用的小分子。选择阳性预测和阴性预测各5个进行进一步的生物学验证。因为ge公司的biacore生物分子间相互作用分析仪具有适用样品类型广泛(包括化学小分子和rna)、分子无需标记、实时、具有超高灵敏度(可监测到微弱、瞬时的分子相互作用)等优点,用ge公司的biacore分析仪验证预测的阳性和阴性结果。
实施例2:
1、rna-化学小分子相互作用数据的收集和整理
一套可靠的已获证实的rna-化学小分子相互作用数据是创建靶向rna化学小分子筛选计算方法的基础。为此,申请人从pdb数据库下载相关数据,并分析整理,作为训练数据集。另外,为验证所提出的预测方法,从smmrna(smallmoleculemodulatorsofrna)数据库获取新的不包含在pdb内的rna-化学小分子相互作用对,从已发表的文献中人工检索新的已获实验证实的rna-化学小分子相互作用对,将smmrna和文献检索结果共同作为独立测试数据集。
2、rna相关特征及小分子理化性质的计算
基于训练数据集的rna-化学小分子相互作用数据,从序列、结构和功能等多个角度提取rna相关的特征,如核苷酸类别、功能位点、核苷酸距离和(nucleotidedistancesum,nds)曲线、核苷酸频率和配对状态等;从化学小分子结构数据中提取结构文件,计算理化性质,包括氢键受体数量(numberofhydrogenbondacceptors,hba)、氢键供体数量(numberofhydrogenbonddonors,hbd)、辛醇/水分布系数(octanol/waterpartitioncoefficient,logp)、摩尔折射率(molarrefractivity,mr)、分子量(molecularweight,mw)、拓扑极性表面面积(topologicalpolarsurfacearea,tpsa)等。
3、rna-化学小分子相互作用预测方法的创建
由于数据集中与化学小分子无相互作用的rna片段要远远多于有相互作用的片段,采用将负样本分成多份分别与正样本匹配的均衡随机森林(balancedrandomforest,brf)模型创建rna-化学小分子相互作用预测的计算方法。另外,鉴于模型中整合的特征以及构建流程中可调整的参数较多,采用分步的方式予以优化。
4、rna-化学小分子相互作用预测方法的验证
为验证所创建rna-化学小分子相互作用预测方法的准确性,在训练数据集上进行5折交叉验证,随机森林模型的预测性能采用auc值进行评价。然后在独立测试数据集上运行,亦采用auc值进行评价。最后,将该方法应用于申请人前期发现的血管平滑肌特异的lncrna-ak098656,选择5个阳性预测结果和5个阴性预测结果进行生物学验证。
实施例3:
针对靶向rna的化学小分子药物筛选的计算方法研究,申请人已创建基于mirna的环境因子(大部分为化学小分子)研发平台mirenvironment(cui*etal.bioinformatics2011)。小分子干预靶蛋白一般是干预蛋白上的功能位点,因此确定rna的功能位点是干预靶rna的重要基础。申请人相继提出了rsite、rsite2(cui*etal.scientificreports2015,2016)、sramp(cui*etal.nucleicacidsres2016,m6a甲基化位点预测)和ppus(cui*etal.bioinformatics2015,假尿嘧啶化位点预测)等rna功能位点预测方法。申请人公开了通过rna序列与空间结构获得的功能位点具有显著一致性和相关性(图1),这表明rna序列蕴含着rna空间结构信息,这进一步提示,在rna空间结构数据极度缺乏以及rna力场未知的情况下,利用rna序列特征有可能达到预测与其相互作用的化学小分子的目的。
实施例4:
验证了一个血管平滑肌特异lncrna-ak098656(图2),在高血压病人血液中显著升高,且给大鼠转入ak098656基因后,其血压明显升高(jinletal.hypertension2018,71(2):262-272),表明ak098656高血压防治的潜在新rna靶点(图3)。
实施例5:
申请人已从pdb数据库收集整理了300多对rna-化学小分子相互作用。从smmrna数据库和文献中获取100多对rna-化学小分子相互作用对。经分析,初步发现一些rna序列特征与化学小分子相互作用有关,如三联体频率、rsite2位点等,亦发现一些小分子的理化性质与rna相互作用有关,如辛醇/水分布系数、拓扑极性表面面积等。基于随机森林初步构建了预测方法drip,5折交叉验证结果表明,auc达到0.818,在smmrna和文献来源的独立测试数据集上auc达到0.829(图4),表明所创建的方法在预测rna-化学小分子相互作用上具有了一定的准确率。
- 还没有人留言评论。精彩留言会获得点赞!