基于改进哈里斯鹰算法的DNA存储编码优化方法与流程
本发明涉及元启发式算法和dna存储中使用的组合约束条件,具体来说是使用非线性控制参数策略和随机反向学习策略来改进哈里斯鹰算法,然后将其应用于dna存储中的编码设计领域,构建最优约束编码集合。
背景技术:
dna分子的高密度、大容量和长期稳定性使其成为一种新兴的存储介质,特别适合大数据集的长期存储。baum首次提出构建dna存储的模型,为dna存储技术的研究奠定了基础。此后dna存储技术得到不断的发展和成熟。在编码方面,dna编码基于atcg四个碱基分子,计算机上的原始文件的二进制码元会经过转码映射到具体的编码模型上。常见的编码模型有church使用的二进制模型(a、c=0;t、g=1),goldman使用的三进制模型(huffmancode)和四进制模型(c-1、t-2、a-3、g-4)。对于二进制编码模型而言,每个碱基表示为每个二进制位(0或1)转换为另一个二进制要进行多次重复变换,可表示为,c=x、t=xx、a=xxx、g=xxxx。例如,10101→cccc,100101→ctcct。所以在解码时容易出现一个问题,c可以解码成0或1就导致了很多错误,因此二进制模型没有得到广泛的应用。由于三进制、四进制编码具有更高的编码效率和低错误率,所以得到了广泛使用。另外,组合约束条件是保证存储中序列鲁棒性的必要条件,构建足够多的约束编码序列对dna存储具有至关重要的意义。dna存储除了在存储容量、存储密度或是在长期保存等方面的优势外,最近伴随着dna合成技术和测序技术的飞速发展,dna存储在将来一定是一个很有竞争力的存储解决方案。
技术实现要素:
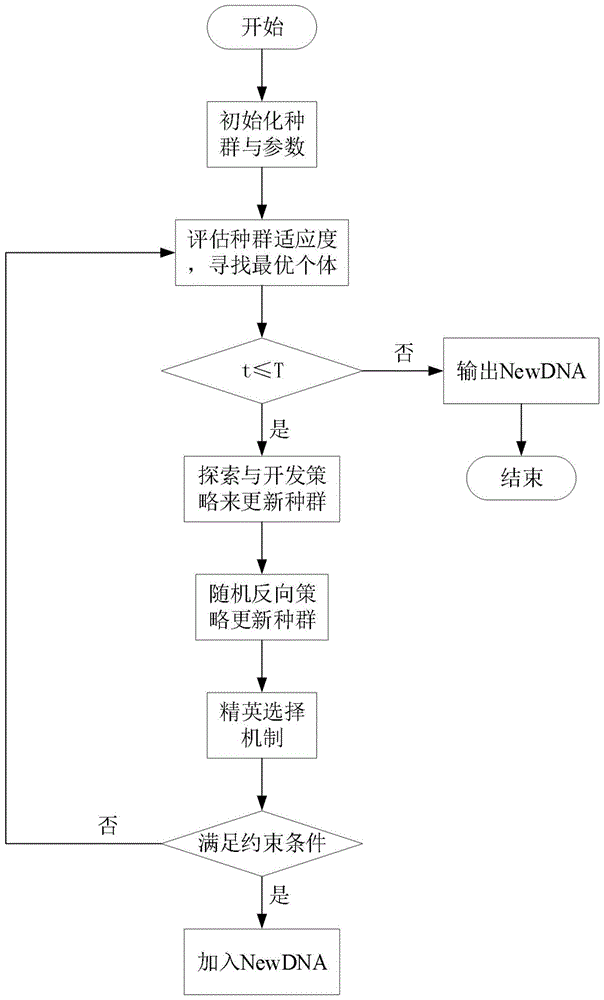
本申请提出了基于改进哈里斯鹰算法的dna存储编码优化方法,该方法首先用哈里斯鹰算法探索与开发中的不同策略对初始种群进行更新;在过程中使用提出的非线性参数控制策略来平衡探索与开发的平稳过渡;接着,通过随机反向学习策略对得到的解集进行扩展,并用精英选择机制挑选最优集合;最后,判断更新出的集合是否满足约束条件,符合约束的加入备选解集合;该方法可以搜索出数量较优的编码序列。
为实现上述目的,本申请的技术方案为:基于改进哈里斯鹰算法的dna存储编码优化方法,具体步骤如下:
步骤1:随机生成数量为n的哈里斯鹰种群,初始化约束参数:最大迭代次数t;
步骤2:计算每个个体适应度,并根据适应度对初始哈里斯鹰种群进行排序,选出最优适应度个体当作猎物xrabbit,更新初始能量e0、跳跃强度j,采用非线性控制策略来更新逃逸能量e;参数更新如下:
j=2*(1-rand())(1)
e0=2*rand()-1(2)
其中,其中bfin和bini分别代表控制参数的最终值2和初始值0,t代表当前迭代次数;
步骤3:当|e|≥1时进入探索阶段,此时采用了两种等概率的不同探索方式来更新鹰的种群位置;
步骤4:当|e|<1时进入开发阶段,此时哈里斯鹰派对于探索阶段检测到的猎物进行追逐和“惊奇突袭”,其采用等概率的四种不同开发方式进行种群更新;
步骤5:对更新出的种群进行随机反向学习,并根据适应度排序,避免陷入局部最优解;公式如下:
其中lj与uj分别为搜索范围的上界和下界;
步骤6:对比反向学习前的个体,采用精英选择机制挑选出n个最优个体;
y代表反向学习前的个体,z代表发现学习后的个体,f(y)代表反向学习前的个体适应度值,f(z)代表反向学习后的个体适应度值;
步骤7:挑选出适应度最优的个体来更新猎物xrabbit;
步骤8:判断更新的种群和初始种群是否满足约束条件,若满足即加入新的集合;
步骤9:判断是否达到最大迭代次数,若是进行步骤10,否则返回步骤2;
步骤10:对结果进行统计,输出不同长度满足约束的序列集合的最大数量。
进一步的,计算个体适应度采用如下方式:
i代表第几条序列,m代表序列的条数,hammingdistant(xi)代表第i条序列与其他不同序列间的汉明距离值。
进一步的,步骤3中两种不同的探索方式为:1)当q≥0.5时,鹰会随机选取一个高大的栖息地作为自己的位置,以便检测到猎物;2)当q<0.5时,鹰会根据其他成员和猎物的位置来更新自己位置,以便捕食时与其他成员更靠近,协助团队合作。
进一步的,步骤4中四种不同开发方式为:
1)软攻击:猎物拥有足够多的逃逸能量,但是随机跳跃没能使它逃脱,鹰派轻轻的环绕猎物使其疲惫,然后进行突袭;
2)硬攻击:很低的逃逸能量使猎物失去活力感到疲惫,因此鹰派无需花费太大力气就将猎物包围进行突袭;
3)渐进式快速潜水的软围攻:此阶段猎物具有足够多的能量,如果下一个位置比当前位置好,鹰派继续保持软围攻的策略,则用公式(5)更新鹰的位置;否则,加入莱维飞行来模拟猎物越级逃逸运动,鹰派在环绕猎物时会快速下潜根据猎物欺骗性逃逸运动不断调整自己飞行方向与位置,从而提高开发能力;
4)渐进式快速潜水的硬围攻:此阶段猎物具有较低的能量无法逃逸,在突袭之前鹰派采取硬围攻策略;猎物依然采用莱维飞行来模拟越级逃逸运动,鹰派试图通过逃逸的猎物来减小其与团体平均位置的距离进行突袭。
进一步的,非线性控制参数策略应用于参数e1,
其中,bfin和bini分别代表控制参数的最终值2和初始值0,t代表当前迭代次数,t代表最大迭代次数。
本发明由于采用以上技术方案,能够取得如下的技术效果:
1、使用哈里斯鹰算法的不同策略更新种群并进行适应度计算,同时引入非线性控制参数策略来维持更新过程中探索与开发的平稳过渡;
2、随机反向学习策略克服哈里斯鹰算法后期容易陷入局部最优的缺点,加快收敛速度,使得算法去寻找更有希望的区域从而达到全局最优;
3、本发明提出的使用非线性控制参数策略和随机反向学习策略改进后的哈里斯鹰算法在dna序列优化中能够快速搜索出数量较优的dna编码序列。
附图说明
图1为本发明的实现流程图。
具体实施方式
下面将结合本发明实施例中的附图,对本发明技术方案进行清楚、完整的描述,可以理解的是,所描述的实例仅仅是本发明的一部分实例,而不是全部的实施例。基于本发明的实施例,本领域的技术人员在没有做出创造性劳动的前提下所获得的所有其他实施例,都属于本发明的保护范围。
本发明中用到的的组合约束条件有全不连续性约束、汉明距离约束、gc含量约束。适应度函数为汉明距离之和,其他两项作为约束条件。全不连续性约束表示在一个dna序列中相同的碱基在相邻时不能连续出现。汉明距离指成对的等长dna序列x,y中,序列x与序列y中相同位置元素不同的数量。gc含量约束表示在dna序列集合中任意一个序列中鸟嘌呤(g)和胞嘧啶(c)的数量占整个序列碱基数量的百分比,一般固定在50%。
详细步骤如下所示:
步骤1:随机生成数量为n的哈里斯鹰种群,初始化约束参数:最大迭代次数t;
步骤2:计算每个个体适应度(汉明距离之和),并根据适应度对初始哈里斯鹰种群进行排序,(适应度越大序列越优),选出最优适应度个体当作猎物xrabbit,更新初始能量e0、跳跃强度j,采用提出的非线性控制策略来更新逃逸能量e;参数更新如下:
j=2*(1-rand())(1)
e0=2*rand()-1(2)
步骤3:当|e|≥1时进入探索阶段。此时采用了两种等概率的不同的探索方式来进行更新鹰的种群位置,探索方式如下:
(1)当q≥0.5时,鹰会随机选取一个高大的栖息地作为自己的位置,以便检测到猎物;(2)当q<0.5时,鹰会根据其他成员和猎物的位置来更新自己位置,以便捕食时与其他成员更靠近,协助团队合作。
步骤4:当|e|<1时进入开发阶段。此时哈里斯鹰派对于探索阶段检测到的猎物进行追逐和“惊奇突袭”。狡猾的猎物会进行欺骗性逃逸,针对于此鹰派会表现出不同追逐策略,主要包括等概率的四种不同开发方式进行种群更新。详细开发策略如下:
1)软攻击
猎物拥有足够多的逃逸能量,但是随机跳跃没能使它逃脱。鹰派轻轻的环绕猎物使其疲惫,然后进行突袭。
2)硬攻击
很低的逃逸能量使猎物失去活力感到疲惫,因此鹰派无需花费太大力气就将猎物包围进行突袭。
3)渐进式快速潜水的软围攻
此阶段|e|≥0.5且r<0.5,猎物具有足够多的能量,如果下一个位置比当前位置好,鹰派继续保持软围攻的策略,则用公式(5)更新鹰的位置。否则,加入莱维飞行来模拟猎物越级逃逸运动,团队在环绕猎物时会快速下潜根据猎物欺骗性逃逸运动不断调整自己飞行方向与位置,从而提高开发能力。
4)渐进式快速潜水的硬围攻
此阶段猎物具有较低的能量无法逃逸,在突袭之前鹰派采取硬围攻策略。猎物依然采用莱维飞行来模拟越级逃逸运动,不同的是鹰派通过逃逸的猎物来减小其与团体平均位置的距离进行突袭。
步骤5:对更新出的种群进行随机反向学习策略,并根据适应度排序,避免陷入局部最优解;随机反向学习公式如下:
步骤6:对比反向学习前的个体,采用精英选择机制挑选出n个最优个体;
步骤7:挑选出适应度最优的个体来更新猎物xrabbit;
步骤8:判断更新的种群和初始种群是否满足约束条件,若满足即加入新的集合;
步骤9:判断是否达到最大迭代次数,若是进行步骤10,否则返回步骤2;
步骤10:对结果进行统计,输出不同长度满足约束的序列集合的最大数量。
实施例1
本发明的实施例是在以本发明技术方案为前提下进行实施的,给出了详细的实施方式和具体的操作过程,但本发明的保护范围不限于下述实施例。实例中dna编码长度n为8,汉明距离约束为d≥5,全不连续性约束、gc含量约束如上所述。
步骤1:对种群进行随机初始化生成1000条长度为8的dna编码序列。初始化算法所需要的相关参数,如初始能量e0、跳跃强度j,最大迭代次数t。
步骤2:通过matlab进行仿真实验对初始种群进行gc含量和全不连续约束筛选得到37条序列,然后对筛选完的37条序列进行两两判别是否都符合汉明距离约束,不满足则删除,最后得到满足所有约束条件的11条序列作为newdna。
步骤3:将步骤2用得到的11条8维的dna序列计算适应度函数(采用汉明距离之和作为适应度函数,值越大序列间相似性越低,序列越优),找出最优个体,然后用哈里斯鹰算法进行更新,此过程中采用非线性控制参数e1来更新逃逸能量e,可以平衡探索与开发过程的平稳过渡。具体参数e1的计算公式如下:
其中bfin和bini分别代表控制参数的最终值2和初始值0,t代表当前迭代次数,t代表最大迭代次数。
(2)适应度函数f(x)的公式如下所示,其中i代表第几条序列,m=32,每条序列维度为8。
步骤4:当|e|≥1时进入探索阶段,此时采用了两种等概率不同的探索方式来进行更新鹰的种群位置;当|e|<1时进入开发阶段。在开发阶段,哈里斯鹰派对于探索阶段检测到的猎物进行追逐和“惊奇突袭”。狡猾的猎物会进行欺骗性逃逸,针对于此鹰派会表现出不同追逐策略,包括等概率的四种不同开发方式进行种群更新。
步骤5:对更新出的种群进行随机反向学习策略,并根据适应度排序,避免陷入局部最优解。策略如下:
其中,rand()指[0,1]之间的随机数,lj与uj为搜索范围的上、下界。
步骤6:对比反向学习前的个体,采用精英选择机制挑选出32个最优个体;
步骤7:从中挑选出适应度最优的个体来更新猎物xrabbit;
步骤8:判断更新后的种群是否满足约束条件,若满足即加入初始宇宙集合newdna;
步骤9:判断是否达到最大迭代次数t(可以为500)代,若是进行步骤10,否则返回步骤3;
步骤10:对结果进行统计,输出满足约束条件序列的最大数量;
本发明提出基于非线性控制参数策略和随机反向学习策略的哈里斯鹰算法的dna存储编码优化方法,用哈里斯鹰算法对初始种群进行搜索。通过gc和全不连续性约束筛选出符合要求的dna序列,以这些序列为基础根据哈里斯鹰算法进行不断的更新,更新过程中用非线性控制参数策略来平衡探索与开发的平稳过渡,并结合随机反向学习策略寻找全局最有希望的区域,最终将得到的最大dna序列编码集合作为输出结果。本发明在intel(r)cpu3.6ghz、6.0gb内存、windows10运行环境下,借助matlab2018a对该算法进行仿真实验,实验结果表明本实例的方法结果优于其他算法的实验结果。
表1为初始dna序列
表2为n=8,d≥5时最优newdna序列集合
以上所述仅是本发明的优选实施方式,并不用于限制本发明,应当指出,对于本技术领域的普通技术人员,在不脱离本发明技术原理的前提下,还可以做出若干改进和变型,这些改进和变型也应视为本发明的保护范围。
- 还没有人留言评论。精彩留言会获得点赞!