基于GSA优化神经网络的生物活细胞表面力学特性评估方法
基于gsa优化神经网络的生物活细胞表面力学特性评估方法
技术领域
1.本发明涉及微纳操纵技术和细胞生物力学领域,特别涉及基于gsa优化神经网络表征生物细胞力学特性评估方法。
背景技术:
2.生物细胞力学的研究对于理解各种基本的细胞行为,如运动、分化和增殖等至关重要,并在组织工程、细胞生物学和癌症治疗领域引起了极大的关注
1.。近年来,国内外的专家学者对生物力学进行了大量的实验研究,尤其是对单个细胞的力学研究尤为关注。研究表明,细胞的力学性质在细胞的迁移、分裂、分化、粘附与纳米颗粒内吞等生理活动中扮演着重要的角色,对单个细胞的力学特性研究将帮助人类更清楚地了解疾病(包括癌症)的发病机理,并为疾病的早期筛查和诊断等提供理论依据及新的研究方法
2.。因此,细胞力学性质的精确测量对于认知生命活动的本质和维护人类生命健康有着重要的意义。
3.但是,研究者们基于不同线性粘弹性模型所测量出的细胞本构方程仍旧难以统一,往往需要大量的粘弹性参数才可拟合出自己实验中的测量数据,却又不能预测出他人实验中同种细胞不同加载方式下的力学响应。在力学发展过程中,研究者往往通过提出理想化的力学模型开展理论分析。这些理想模型一般具有较为规则的几何形状、边界条件和基于显式表达式描述的材料响应函数,将真实的复杂力学问题抽象为简单的数学模型,进而寻求问题的解析解,计算力学方法的出现使求解具有更复杂的几何模型和边界条件的问题成为可能
3.。这方面神经网络具有其优越性:首先,神经网络的监督学习可以依据已知样例的迭代学习,快速获得输入输出间映射关系,这种映射关系以连接权值与阈值形式表现,类似大脑神经元,可以描述各种非线性关系
4.;其次,神经网络具有泛化能力,即神经网络对未在训练(学习)过程中遇到的数据仍可以得到合理的输出。
4.但实际应用中bp网络差曲面存在很多局部最小点,在某些初值的条件下算法的结果会陷入局部最小且bp网络训练学习的收敛速度很慢。考虑到sa具有摆脱局部最优解的能力,可是收敛速度较慢,而ga由于处理数据方式简单,可以较快收敛,所以结合两者优点提出一种基于gsa来改进神经网络建立的力学模型得法,使之更具有普遍适用性。
5.[1]dingyue,xuguang-kui,wanggang-feng.onthedeterminationofelasticmoduliofcellsbyafmbasedindentation.[j].scientificreports,2017,7{5}:
[0006]
[2]卜洋.基于原子力显微镜的细胞粘弹性力学性质测量[d].兰州大学,2020.
[0007]
[3]belytschkot,liuwk,moranb,etal.nonlinearfiniteelementsforcontinuaandstructures.hoboken:johnwiley&sons,2013.
[0008]
[4]haykins.神经网络与机器学习[m].3版.北京:机械工业出版社,2016:1-22.
技术实现要素:
[0009]
本发明技术解决问题:克服现有技术的不足,提供一种基于gsa优化神经网络表
征生物细胞力学特性评估方法,具有拟合度高的特点。针对不同类型细胞 可以根据其力学参数建立适用的力学模型,帮助研究人员更快速更准确的研究细 胞力学特点。
[0010]
本发明解决技术问题所采用的技术方案如下:一种基于gsa优化神经网络 表征生物细胞力学特性评估方法:生理状态下采集生物活细胞力学特性,细胞粘 弹性测试,制作细胞力学参数样本集,gsa神经网络优化算法,细胞表面力学 特征预测,细胞表面力学建模,细胞力学参量聚类分析,细胞力学评估模型;具 体步骤如下:
[0011]
步骤1:生理状态下采集生物活细胞力学特性,在生理条件下用原子力显微 力学测试系统进行细胞力学性能实验,通过原子力探针对细胞表面高低起伏形态 进行逼近-保持-撤回过程的力学测试,记录细胞力学参数数据存储与电子表格中 并且以室温、液相、探针振幅统一来保证实验的可复制性以及细胞的状态良好;
[0012]
步骤2:细胞粘弹性测试,进行细胞粘弹性测试,考虑细胞粘弹性以自建粘 弹性模型获得细胞力学特征参数描述,通过hertz或sneddon模型来获得细胞每 点的弹性模量。在粘弹性测试中先对细胞参数进行初筛,保证其误差在可接受范 围中;
[0013]
步骤3:制作细胞力学参数样本集,通过力学测试所获得的力学数据筛选力 学特征参数及其取值范围,等分设定各参数的水平数并且去掉极值,根据析因设 计法构建计算方案,使用有限元计算模拟后生成训练样本。且训练样本按70%、 15%、15%的比例分为训练集、验证集、测试集。
[0014]
步骤4:gsa神经网络优化算法,首先在数据分析软件中进行整体的编程与 建模,根据析因设计得到的样本集构造神经网络的整体结构。再根据网络预设输 入输出节点个数以及连接方式初始化ga遗传算法,采用参数化编码将样本集中 的接触点深度、接触点应力、细胞整体高度、细胞局部弹性模量、粘弹系数以及 松弛时间构成六维结构体作为初始种群。种群最大迭代次数为100,种群数量为 50,交叉及变异概率分别为0.1、0.3。其次初始化模拟退火的的初始温度及降温 系数,分别为t0=100,alpha=0.8。
[0015]
模拟退火的循环中进行遗传迭代。首先对初始训练后的网络权值进行精英模 式遗传迭代,若子代群体的最佳个体适应值小于当前群体最佳个体的适应值,则 将当前群体最佳个体或者适应度大于子代最佳个体适应值的多个个体直接复制 到子代,随机替代最差的下一代群体中的相应数量的个体。在每一轮迭代结束后, 确定当前温度,并且更改新的适应度函数,重复循环,直到周期性降温达到终止 温度,得到最优网络权值,并基于此权值预测输入输出关系。
[0016]
步骤5:细胞表面力学特征预测,以建立好的神经网络模型,进行数值计算 得到新的力-位移曲线特征数据,并将其与力学实验测量的力-位移曲线数据的差 值作为优化线索,其误差公式为其中f1、f2为模拟 特征应力值,fr、fe分别为实际测量中进针及曲线压入点及峰值点应力值,通 过参数寻优算法得到最优参数,即为该细胞当前区域内的表征值;在此基础上对 同种类型细胞不同时间多次多区域采集,获得不同条件下力学参数,并通过神经 网络算法提取细胞力学特征;
[0017]
步骤6:细胞表面力学建模,将获得的细胞不同区域内的表征力学参数重新 带入到原自建模型中,获得当前细胞的力学模型,并对比传统模型、神经网络以 及gsa优化网络之间的误差率;
[0018]
步骤7:细胞力学参量聚类分析,根据数值模拟确定细胞弹性模量及粘附力 上下限,将两种参量及其对应细胞高度位置输入bp神经网络中进行聚类分析, 并根据网格化位置标记其力学参量的分布;
[0019]
步骤8:细胞力学评估模型,用于不同类型细胞建立其力学分布特征重新训 练神经网络,获得其力学模型。
[0020]
进一步地,所述生理状态下采集生物活细胞力学特性,对细胞表面进行纳米 压痕粘弹性测试实验,获取细胞表面粘弹性的力-位移曲线和力-时间曲线,压痕 深度、保持时间、接触应力、细胞高度等力学参数。
[0021]
进一步地,所述细胞粘弹性测试,hertz或sneddon等弹性模型,hertz模型 适用于针尖为微球形原子力探针,sneddon模型适用于针尖为锥型原子力探针, 分别记录细胞表面逼近-保持-撤回过程的力学参数;而自建粘弹性模型描述细胞 高度、粘附功与接触应力弹性模量所建立的定量关系。
[0022]
进一步地,所述细胞力学参数样本集,筛选确定输入的力学特征参数为接触 点的接触应力、达到压痕峰值的接触应力、细胞高度、压痕深度,而输出的力学 参量为弹性模量、粘附力。
[0023]
进一步地,所述细胞力学参数样本集,所采用的的实验数据试验方法为析因 设计法,可以研究单个因素多个水平的效应,也可以研究因素之间是否有交互作 用,同时找到最佳组合,使得后续神经网络模型映射关系以及优化后的参数更准 确且对不同细胞区域更有针对性。
[0024]
进一步地,所述神经网络算法,采用模拟退火算法和遗传算法相结合对神经 网络模型优化,从数据集中自学习特征进行分类,适应度函数为整体算法的执行流程为首先初始化bp神经网络,根据输入 输出集维数确定bp神经网络的输入节点及输出节点数量,根据公式 遍历获得隐含层节点数,其中l为隐含层节点数,m=输入层节点 数,n=输出层节点数,
ɑ
为小于10正整数;确定网络整体结构后,根据网络层数、 每层神经元数、各层互连方式进行参数化编码初始化遗传算法,种群规模预设为 50,最大迭代次数为100次;再预设模拟退火算法的初始温度t0=100,退火速 度为0.8,终止温度t1=0.0001;全部遗传迭代过程在退火模拟的循环中进行, 在初始训练后由网络输入输出误差构成个体适应度再通过基于精英选择模型的 迭代方式获得最优子代,再通过退火循环这个过程,在周期性降温中得到网络最 优权值。
[0025]
进一步地,所述细胞表面力学特征预测,对不同细胞区域进行纳米压痕时以 8
×
8网格来划分细胞表面依次进行压痕,以保证收集的力学参量能够进行区域 化聚类并方便定位。
[0026]
进一步地,所述细胞表面力学特征预测,将对不同种细胞的力学特性进行表 征,并使用传统方法,对比神经网络以及改进的神经网络误差率以证明算法的鲁 棒性。
[0027]
进一步地,所述细胞表面力学参量聚类分析,进行聚类分析的细胞表面力学 特征包括细胞弹性模量、粘附力,并根据数值模拟的样本集划定范围。
[0028]
另外,根据本发明实例中提供的一种基于gsa优化神经网络表征生物细胞力 学特
性评估方法,所述细胞粘弹性测试中的hertz模型为 sneddon模型为,其中f是施加在探针上的法向压力,r为 微球型探针针尖半径,δ是压痕深度,θ是圆椎体的半角,e是样本的弹性模量, ν是样本泊松比(一般视为0.5);
[0029]
另外,根据本发明实例中提供的一种基于gsa优化神经网络表征生物细胞力 学特性评估方法,所述细胞粘弹性测试中的自建粘弹性模型为 其中e为有效弹性模量,f为接触应力,h 为压痕深度,
ɑ
为探针探针半角,δγ为粘附功。
[0030]
另外,根据本发明实例中提供的一种基于gsa优化神经网络表征生物细胞力 学特性评估方法,所述制作细胞力学参数样本集中的细胞细胞力学参数数据获取 方式具体为原子力探针在细胞表面不同条件下某个位置点进行z方向逼近-保持
‑ꢀ
撤回过程测试,其中原子力探针单次测试从恒高的位置逼近细胞,在细胞表面产 生压痕直至探针悬臂梁加载力达到设定值,探针保持停留一定时间,探针从细胞 表面撤回至初始位置。
[0031]
与传统方法相比,本发明具有以下优点:
[0032]
本发明的一种基于gsa优化神经网络表征生物细胞力学特性评估方法,相比 于传统的细胞力学建模方式,拟合度更高,可以为多细胞的力学模型建立提供针 对性的手段,有助于更快速更准确的研究细胞力学特点。并且解决了以往人工神 经网络收敛速度慢和过拟合的问题。通过不同种类细胞不同区域力学数据的神经 网络聚类分析,解决细胞力学特性中表面力学参量无法聚类分析的难点。通过模 拟退火与遗传算法结合方式对神经网络的网络权值进行优化,解决权值优化过程 中容易陷入局部最小和收敛速度的问题。通过该优化神经网络进行建模,使得细 胞力学评估结果具有良好的泛化能力,符合生物细胞力学规律。
附图说明
[0033]
图1为基于gsa优化神经网络表征生物细胞力学特性评估方法示意图;
[0034]
其中1原子力显微镜力学测试系统,2细胞粘弹性测试,3细胞力学参数样 本集,4神经网络算法,5细胞表面力学特征,6细胞力学建模,7表征待测细胞, 8细胞力学模型;
[0035]
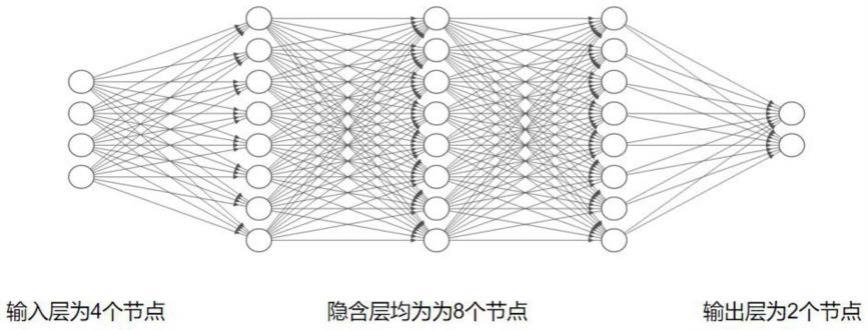
图2为本发明所用bp神经网络拓扑图;
[0036]
图3为本发明方法对肝癌细胞(smcc-7721)原子力显微镜力-位移曲线对 比图;
[0037]
图4为本发明方法对人体乳腺癌细胞原子力显微镜力-位移曲线对比图;图5为不同细胞应用传统模型、神经网络模型以及gsa优化神经网络模型的平均误差 率对比。
具体实施方式
[0038]
下面结合附图对本发明进行进一步说明。
[0039]
如图1所示,一种基于gsa优化神经网络表征生物细胞力学特性评估方法内 容包括:生理状态下采集生物活细胞力学特性1,细胞粘弹性测试2,制作细胞 力学参数样本集
3,gsa神经网络优化算法4,细胞表面力学特征预测5,细胞 表面力学建模6,细胞力学参量聚类分析7,细胞力学评估模型8。通过原子力 探针纳米压痕1,进行生理条件下用原子力显微力学测试系统进行细胞力学性能 实验,记录细胞力学参数数据记录在电子表格中;利用细胞粘弹性测试技术2, 结合hertz或sneddon等弹性模型以及自建粘弹性模型,获得原子力探针在细胞 表面逼近-保持-撤回过程的力学参数数据,建立细胞力学参数样本集3;基于gsa 神经网络优化算法4,在数据分析中进行建模,通过细胞力学参数样本集进行训 练和测试,并进行细胞力学参数的神经网络模型优化;以细胞力学参数的数据作 为输入,预测最优细胞表面力学参量5;将获得的细胞不同区域内的表征力学参 数重新带入到原自制模型中,获得当前细胞的力学模型6;将数据模拟得到的细 胞弹性模量及粘附力、细胞高度通过bp神经网络进行聚类分析,得到网格化的 细胞表面力学特征分布7;针对不同类型的细胞根据其力学分布特征及最优参量 通过有限元进行建模,获得其力学模型8。
[0040]
如图2所示图,神经网络的拓扑结构根据力-位移关系设定,根据输入数据 类型有4种设置输入层节点为4,神经网络隐含层节点的选取无绝对理论依据, 一般依照训练表现与经验,不断调整获取最佳值。经调试隐含层层数为3层、每 层节点为8个,输出层节点为1。
[0041]
如图3所示图,为通过本发明方法拟合后的肝癌细胞原子力显微镜力-位移 曲线对比图。通过原子力显微镜对细胞进行纳米压痕,并将所得数据经sneddon 接触模型处理得到相关力学参数输入进网络中,经网络训练反演参数,得到对应 粘弹性系数,经公式拟合得到图3对比图,该细胞力学数据经神经网络训练后, 其力位移曲线拟合度较为贴合实际测量值,并优于传统的力学建模方法。
[0042]
如图4所示图,为通过本发明拟合后的人体乳腺癌细胞原子力显微镜力-位 移曲线对比图。通过原子力显微镜对细胞进行纳米压痕,并将所得数据经 sneddon接触模型处理得到相关力学参数输入进网络中,经网络训练反演参数, 得到对应粘弹性系数,经公式拟合得到图4对比图,该细胞力学数据经神经网络 训练后,其力位移曲线拟合度较为贴合实际测量值,并优于传统的力学建模方法。
[0043]
如图5所示图,对不同细胞表面的力学表征过程进行gsa优化的神经网络 模型分析,获得细胞表面力学特征的平均相对误差要比神经网络的平均相对误差 小,并且明显优于传统模型。
[0044]
本发明的一种基于gsa优化神经网络表征生物细胞力学特性评估方法建模 流程,通过原子力显微镜粘弹性测试实验进行细胞表面力-位移曲线和力-时间曲 线,获得粘附力、压痕深度、保持时间、杨氏模量等力学参数;通过原子力显微 镜力学测试系统,进行生理条件下细胞表面粘弹性测试;采集不同条件下的细胞 表面力学参数,建立数据集,并分析数据;将细胞力学参数的数据集引入神经网 络(如图2所示图)进行训练,采用gsa算法对神经网络模型进行优化;细胞 力学参数的数据作为输入,以细胞粘弹性参数作为输出,通过神经网络预测得到 输入输出关系,进行细胞力学建模;通过对同种细胞不同时间多次多区域采集获 得网格标记的细胞表面力学参数,以bp神经网络聚类分析获得其分布特征;针 对不同类型的细胞根据其力学分布特征及最优参量通过有限元进行建模,获得其 力学模型。
[0045]
实施例1:
[0046]
如图3所示图,为通过本发明方法拟合后的肝癌细胞原子力显微镜力-位移 曲线
对比图。通过原子力显微镜以四棱锥探针对肝癌细胞smcc-7721进行纳米 压痕,肝癌细胞最大压痕深度为0.6微米,最大应力为4.2nn,并将所得数据经 sneddon接触模型处理得到相关力学参数后确定弹性模量范围0.9-3.2kp,经数据 分析数值模拟后得到参数后输入进网络中,经gsa优化神经网络训练反演参数, 得到对应粘弹性系数,经公式拟合得到图3对比图,该细胞力学数据经神经网络 训练后,其力位移曲线拟合度较为贴合实际测量值,并优于传统的力学建模方法, 误差约为6%。
[0047]
实施实例2:
[0048]
如图4所示图,为通过本发明拟合后的人体乳腺癌细胞原子力显微镜力-位 移曲线对比图。通过原子力显微镜以四棱锥探针对人体乳腺癌细胞mcf-7进行 纳米压痕,人体乳腺癌细胞最大压痕深度为0.8微米,最大应力为8.6nn,并将 所得数据经sneddon接触模型处理得到相关力学参数后确定弹性模量范围 1.7-4.5kp,经数据分析数值模拟后得到参数后输入进网络中,经gsa优化神经 网络训练反演参数,得到对应粘弹性系数,经公式拟合得到图4对比图,该细胞 力学数据经神经网络训练后,其力位移曲线拟合度较为贴合实际测量值,并优于 传统的力学建模方法,误差约为6.5%。
[0049]
如图5所示图,为对不同种类癌细胞以不同方式进行力学建模后的误差率对 比,分别为传统赫兹模型,bp神经网络模型以及优化神经网络模型。
[0050]
本发明的一种基于gsa优化神经网络表征生物细胞力学特性评估方法,相比 于传统的细胞力学建模方式,拟合度更高,可以为多细胞的力学模型建立提供针 对性的手段。
[0051]
以上对本发明提供的一种基于gsa优化神经网络表征生物细胞力学特性评 估方法进行了详细介绍,提供以上实例仅仅是为了帮助理解本发明的方法,而并 非要限制本发明的范围。本发明的范围由所附权利要求限定,不脱离本发明的精 神和原理而做出的各种等同替换和修改,均应涵盖在本发明的范围之内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1