一种基因调控网络的降维建模方法及系统
本发明涉及基因调控网络的降维建模方法及系统,属于基因调控网络构建技术、涉及数字医疗及人工智能等领域。
背景技术:
1、基因调控网络是一种用于发现基因与基因、基因与蛋白质或蛋白质与蛋白质等生物信息物质之间的相互作用关系的工具。基因调控网络的构建从输入数据开始,通常由基因表达数据集组成,在输入数据集选定之后,可以通过数据预处理以提高建模效果。然后,将预处理后的数据作为计算推理算法的输入,得到最终的网络。其中计算推理算法中效果较突出的计算推理算法为微分方程模型,该方法使用连续变量代替离散变量,能够构建更精确的模型,并使基因调控的动态建模成为可能。然而微分方程模型无法构建大规模的基因调控网络,并且在某些情况下,由于计算的复杂性,模型参数的值估计会变得十分困难。
2、文献号为cn114155913a的现有技术公开了一种基于高阶动态贝叶斯的基因调控网络构建方法,其通过利用时序基因表达数据去构建基于时间变量的高阶动态贝叶斯基因调控网络,在不同的时间延迟下为目标基因寻找父节点,以提高网络结构构建的准确率。由于高阶网络结构学习的搜索空间非常大,导致结构学习的时间复杂度特别高,为了加快学习速度,提出了在结构学习之前进行潜在调控因子筛选的办法,首先筛选出与目标基因关联度较高的基因作为目标基因的潜在调控因子集,删除与目标基因关联程度较低的基因,以达到缩小搜索空间的目的,可以加快后面进行网络结构学习的速度。但对于,如何有效解决建模过程中精度与计算量之间的矛盾,在保证建模精度的条件下实现了计算量的指数级降低,没有提及。
3、文献号为cn116246713a的现有技术公开了一种基于基因调控网络的单细胞肿瘤微环境数据分析方法,其包括以下步骤:获取若干个细胞的原始数据值,并进行预处理和特征基因识别;基于获得的特征基因之间的调控关系,构建degree基因表征矩阵,并识别细胞亚群;对所述细胞亚群进行细胞熵的分析、差异degree基因的识别以及基因功能富集的分析。该现有技术通过scile算法以每一基因在每一细胞中基因调控网络的重要程度评估细胞整体的干性熵值,相比于传统基于表达量的评估方法,避免因高表达低调控基因对于细胞干性的影响。在这个应用场景下,对于基因调控网络如何进行降维也没给出技术手段。
4、目前最常用的基因调控网络推理算法为贝叶斯模型,该模型利用概率的贝叶斯定理,结合概率和图论对基因调控网络的性质进行定性建模,该模型没有微分方程模型建模精度高,然而微分方程模型因为计算量过大,无法应用于大基因组的基因调控网络构建。因此,亟需提出一种基因调控网络的降维建模方法,以有效解决建模过程中精度与计算量之间的矛盾,在保证建模精度的条件下实现了计算量的指数级降低。
技术实现思路
1、本发明要解决的技术问题是:
2、本发明为了有效解决建模过程中精度与计算量之间的矛盾,在保证建模精度的条件下实现计算量的指数级降低,为了解决大基因组建模过程所需计算量过大导致无法建模等问题,进而提出了一种基因调控网络的降维建模方法及系统。
3、本发明为解决上述技术问题所采用的技术方案为:
4、本发明所述的一种基因调控网络的降维建模方法,包含以下步骤:
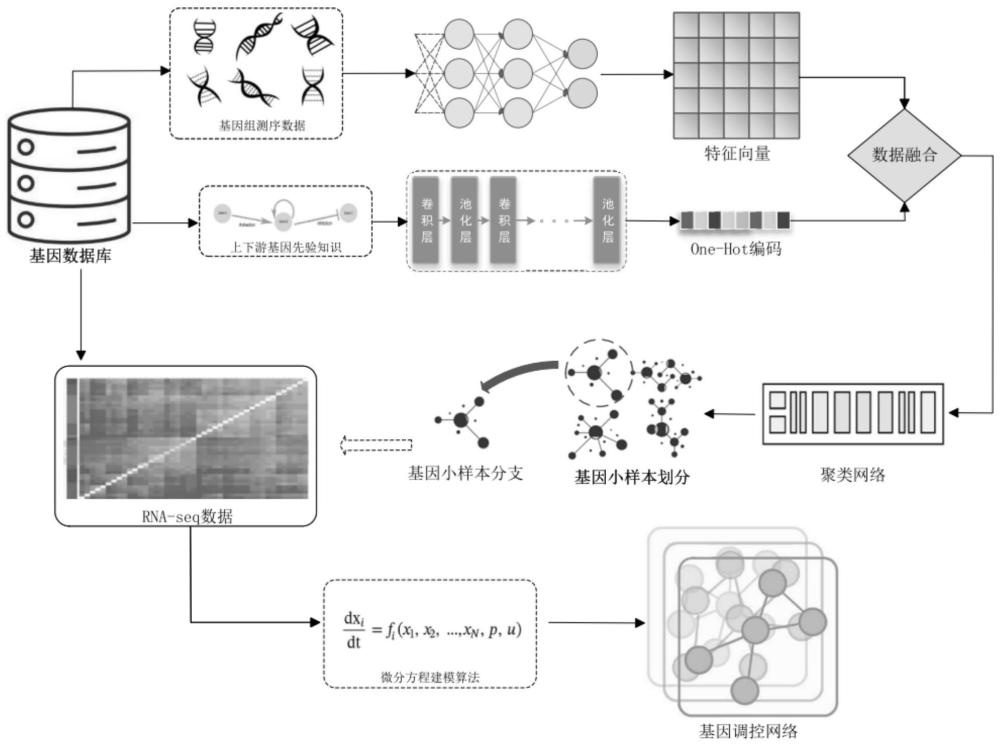
5、s1、将基因组测序数据与上下游基因先验知识进行数据融合得到融合数据;
6、s2、对基因组融合数据进行基因组小样本划分:通过无监督深度学习网络对基因组融合数据进行聚类,输出各基因所属的基因样本分支序列,各基划分到不同基因小样本分支;
7、s3、根据基因组小样本分支中的元素从基因数据库中提取对应的rna-seq数据集合;
8、s4、构建各分支基因调控网络的微分方程模型;
9、s5、将各分支基因调控网络进行汇总得到完整基因调控网络。
10、进一步地:
11、s1、将基因组测序数据gi与上下游基因先验知识pij进行数据融合得到融合数据ci;
12、s2、对基因组融合数据进行基因组小样本划分:通过无监督深度学习网络对基因组融合数据ci进行聚类,输出各基因所属的基因样本分支序列di,各基因根据公式2划分到不同基因小样本分支dk;
13、m(c)=d,d=1,2,…,n (1)
14、式中m代表无监督深度学习网络;n代表基因组小样板分支的个数;
15、dk={ci|m(ci)=k},k=1,2,…,m (2)
16、式中m表示基因小样本分支的个数;
17、s3、根据基因组小样本分支dk中的元素从基因数据库中提取对应的rna-seq数据集合rk;
18、s4构建各基因调控网络分支gk的微分方程模型:通过公式3构建基因调控网络分支gk,其中系数aj大于零表示基因gi受基因gj正调控,aj等于零表示基因gi不受基因gj调控,aj小于零表示基因gi受基因gj负调控;
19、
20、式中ri表示基因gi对应的rna-seq数据,nk表示基因组小样本分支dk中的基因数,aj为基因gj对基因gi的调控因数,b为常数;
21、s5、将基因调控网络分支进行汇总得到基因调控网络g。
22、g={g1,g2,…,gk} (4)
23、步骤s1所述的数据融合方法包含如下步骤:
24、s11、通过特征提取网络提取基因组测序数据,得到特征向量fi:
25、f(gi)=fi (5)
26、其中f代表特征提取网络;
27、s12、将上下游基因先验知识通过编码网络进行编码;
28、s13、将步骤s11与步骤s12得到的基因组测序特征向量与先验知识编码进行数据融合,该数据融合方法将先验知识融合进基因组测序特征,可以有效提升基因关联信息,提升聚类精度。
29、本发明可使用图神经网络进行聚类,符合条件的网络包括daegc、sdcn或agc。进行聚类采用的网络为daegc、sdcn或agc或三者的融合;通过特征提取网络提取基因组测序数据,得到特征向量;将上下游基因先验知识通过编码网络进行编码;将得到的基因组测序特征向量与先验知识编码进行数据融合,该数据融合方法将先验知识融合进基因组测序特征以提升基因关联信息、聚类精度。
30、所述的基因调控网络的降维建模方法的应用,将所述的基因调控网络的降维建模方法用于大基因组建模。广义上的大型基因组(genome size)通常指基因组大小超过5gb,多集中在裸子植物、两栖动物、爬行动物类等。它们往往具有超高的重复序列,还兼具大量的杂合区域。如被子植物(开花植物)基因组大小相差达2400倍(1c=0.063–148.8gb),平均基因组大小为1c=5.7gb。
31、本发明具有以下有益技术效果:
32、本发明提出的一种基因调控网络的降维建模方法首先将基因组测序数据与基因上下游先验知识进行数据融合,用于提升基因聚类的精准性。为了解决大基因组建模过程所需计算量过大导致无法建模的问题,利用聚类网络对基因进行聚类,通过基因序列与先验知识对基因组进行预划分,将各基因划分到不同的小样本分支分别进行基因调控网络建模,以降低每次建模所需的计算量。然后构建各小样本分支的基因调控网络微分方程模型,最后对各分支调控网络进行整合,汇总成为该基因组基因调控网络。
33、本发明提出的一种基因调控网络的降维建模方法,其主要思想是通过聚类网络对基因组进行小样本分支划分,通过对不同分支分别建模,降低建模复杂度,减小建模过程计算量,提升建模鲁棒性。实验表明,我们的基因调控网络降维建模方法可以有效解决建模过程中精度与计算量之间的矛盾,在保证建模精度的条件下实现了计算量的指数级降低,有效解决了大基因组建模过程所需计算量过大导致无法建模等问题,完全适用于大基因组的基因调控网络构建,大大提高了基因调控网络的应用范围。
- 还没有人留言评论。精彩留言会获得点赞!