融合蛋白Slit2D2(C386S)-HSA及其在治疗纤维化疾病中的应用的制作方法
本发明涉及生物医药技术领域,具体涉及一种融合蛋白Slit2D2(C386S)-HSA及其在治疗和/或预防纤维化疾病中的应用。
背景技术:
纤维化(fibrosis)可发生于多种器官,主要病理改变为器官组织内纤维结缔组织增多,实质细胞减少,持续进展可致器官结构破坏和功能减退,乃至衰竭,严重威胁人类健康和生命。任何原因只要能引起组织细胞损伤,均可导致组织细胞发生变性、坏死和炎症反应,如果损伤很小,损伤细胞周边正常实质细胞将发生增生修复,这种修复可完全恢复正常的结构和功能。然而如果损伤较大或反复损伤超出了损伤周围实质细胞的再生能力时,间质纤维结缔组织(细胞外基质)将大量增生对缺损组织进行修复,即发生纤维化的病理改变。因此本质上纤维化是组织遭受损伤后的修复反应,以保护组织器官的相对完整性。增生的纤维结缔组织虽然修复了缺损,但却不具备原来器官实质细胞的结构和功能。如果这种修复反应过度、过强和失控时,就会引起器官的纤维化和导致器官的功能下降。由此可见,纤维化是指由于炎症导致器官实质细胞发生坏死,组织内细胞外基质异常增多和过度沉积的病理过程。轻者成为纤维化,重者引起组织结构破坏而发生器官硬化。在全世界范围内,组织纤维化是许多疾病致残、致死的主要原因,据美国有关统计资料证明,该国因各种疾病而致死的病人中,接近45%可以归于组织纤维增生疾病。纤维化疾病中包括肺纤维化、肝脏纤维化、肾脏纤维化等。
目前纤维化疾病的治疗药物非常少,以肺纤维化为例,目前美国FDA批准的药物只有由InterMune,Inc.公司研制生产的Esbriet(pirfenidone,吡非尼酮)和德国殷格翰公司研制生产的(尼达尼布,Nintedanib),但这两种药物目前患者获益并不理想。
神经迁移蛋白Slit是一种在进化上高度保守的分泌型细胞外基质糖蛋白,分子量约为200kD,对轴突生长和神经元迁移起导向作用,哺乳类动物中克隆到的Slit基因有三个,分别命名为Slit1、Slit2和Slit3,其结构由N端的胞外分泌信号肽,4个富含亮氨酸的重复序列(leucine-rich repeat,LRRs),也被命名为D1-D4结构域,多个EGF(epidermal growth factor)样的重复序列(在果蝇中是7个,在脊椎动物中是9个),一个laminin G样结构域和一个富含半胱氨酸的C端结构域组成,其中以Slit2最为重要。Robo蛋白家族是Slit的受体家族,是一种单通道的跨膜受体,Slit蛋白通过结合受体Robo发挥功能,LRRs是Slit蛋白和受体Robo的结合区域。目前已有文献表明,Slit2蛋白可能会抑制中性粒细胞的迁移。
研究发现Slit蛋白在血管生成、肿瘤细胞迁移、白细胞趋化等方面发挥重要作用,美国专利US8399404B2使用Slit蛋白和核酸用于治疗血小板凝血和其他相应障碍,并公开了一种使用slit蛋白包衣的血管装置和可表达Slit蛋白的细胞。专利WO2009105457公开了一种癌症诊断、研究和治疗的方法和组合物,涉及以Slit2蛋白作为肿瘤标记物有效用作诊断标记物和前列腺癌的临床目标。
Tole等通过研究发现Slit2蛋白通过抑制趋化因子引起的肌动蛋白刺端形成和细胞极化来抑制中性粒细胞的迁移(The axonal repellent,Slit2,inhibits directional migration of circulating neutrophils.J Leukoc Biol.2009,86(6):1403-15)。
Hohenester发现Slit与其受体Robo相结合是通过前者的D2域与后者的IG1的结合实现的(Structural insight into Slit-Robo signaling.Biochemical Society Transactions.2008,36:251-256),因此D2域对Slit蛋白发挥其生理活性非常重要。
中国专利CN201310150884.4利用基因重组技术制备了一种Slit2蛋白的D1-2序列的融合蛋白,该融合蛋白有助于LRR正确折叠,形成有活性的功能多肽,用于Slit2蛋白的研究和应用。
WO2014/194402A1专利报道Slit2分子可用于纤维化疾病的治疗;Pilling D等人对Slit2分子抑制纤维化发生的机制进行了探讨(Fibroblasts secrete Slit2to inhibit fibrocyte differentiation and fibrosis.PNAS.2014,111(51):18291-6)。
分子量小于20kD的蛋白类的药物在代谢过程中易被肾小球滤过,导致其体内半衰期较短,为了达到治疗效果,往往需要频繁或大剂量给药,给患者带来极大的不便。人血清白蛋白(HSA)是一个稳定的“惰性”蛋白,在正常生理状况下不易透过肾小球,在血清中的半衰期达14-21天,可以作为一种载体与血液中的其他因子,包括生物活性蛋白结合,从而保持或延长其他因子在体内的生物活性。将一些小分子肽或蛋白质药物与HSA进行融合是改进小分子肽或蛋白质药物半衰期的有效方法。与其他方法相比,构建长效白蛋白融合蛋白药物可以避免复杂的化学修饰和处理过程,因而具有容易操作和更好的经济性优势。
因此,为克服现有技术的不足,本发明提供一种治疗和/或预防纤维化疾病的融合蛋白及其应用。
技术实现要素:
本发明的一方面提供一种含有或由Slit2蛋白D2结构域(以下简称为Slit2D2)组成的多肽或蛋白,其中在Slit2蛋白D2结构域中对应于Slit2蛋白的第5个半胱氨酸被突变为其他氨基酸或被缺失。
优选的,所述Slit2蛋白是来源于哺乳动物的Slit2蛋白,优选为人的Slit2蛋白,且在Slit2蛋白D2结构域中对应于Slit2蛋白的第5个半胱氨酸位于Slit2蛋白的386位;
优选的,所述其他氨基酸是极性氨基酸,优选自Ser、Gln、Thr、Asn和Tyr的氨基酸残基;
更优选的,所述多肽或蛋白包含或由如SEQ ID NO:1所示的氨基酸序列组成;
优选的,所述的多肽或蛋白还包括经过一个或几个氨基酸残基的取代和/或缺失和/或添加且具有相同功能的由其衍生的氨基酸序列;优选的,所述一个或几个氨基酸残基的取代和/或缺失和/或添加为不超10个氨基酸残基的取代和/或缺失和/或添加;
本发明的另一方面提供编码上述多肽或蛋白的核苷酸。
优选的,所述编码核苷酸具有如SEQ ID NO:2所示的核苷酸序列或其简并序列。
本发明的另一方面提供一种包含上述多肽或蛋白的融合蛋白。
优选的,所述的融合蛋白还包括人血清白蛋白(HSA)。
在本发明的一个优选实施例中,所述融合蛋白为Slit2D2(C386S)-HSA,由上述多肽或蛋白和HSA连接得到,所述的连接为直接连接或通过连接肽连接。
所述的直接连接为所述的多肽或蛋白通过C末端与HSA蛋白的N末端直接连接,或多肽或蛋白通过其N末端与HSA蛋白的C末端直接连接,中间没有任何连接肽。
优选的,所述的融合蛋白包含或由如SEQ ID NO:3所示的氨基酸序列组成。
本发明的另一方面提供编码上述融合蛋白的核苷酸。
优选的,所述的编码核苷酸包含或由如SEQ ID NO:4所示的核苷酸序列组成。
本发明的另一方面提供一种包含上述多肽或蛋白的编码核苷酸、融合蛋白的编码核苷酸的载体。
本发明另一方面提供一种包含上述多肽或蛋白的编码核苷酸、融合蛋白的编码核苷酸、上述载体的宿主细胞。
优选的,所述的宿主细胞为包含上述载体的宿主细胞。
本发明另一方面提供一种上述融合蛋白的制备方法,包括培养上述宿主细胞及,任选地,分离和纯化的步骤。
优选的,上述制备方法包括如下步骤:
(1)重组载体的构建和验证;
(2)转化体的制备和发酵;
(3)融合蛋白的分离和纯化;
任选的,(4)融合蛋白的鉴定。
优选的,步骤(1)中所述的重组载体的构建包括利用T/A克隆为将融合蛋白的基因片段插入到表达载体中得到重组载体;
优选的,所述的融合蛋白的基因通过全基因合成得到;
优选的,所述的表达载体为pCDNA质粒;在本发明的一个优选实施例中,所述的表达载体为pCDNA3.4。
优选的,步骤(1)中所述的重组载体的验证包括将重组载体转化到宿主细胞1中,筛选阳性克隆,确认载体构建成功并保种;
优选的,所述的宿主细胞1为大肠杆菌,在本发明的一个优选实施例中,所述的宿主细胞1为大肠杆菌TOP10。
优选的,所述的步骤(2)包括提取经过步骤(1)验证的重组载体,转染宿主细胞2,发酵;
在本发明的一个优选实施例中,所述的宿主细胞2为ExpiCHO-STM细胞。
优选的,步骤(3)中所述的融合蛋白的分离和纯化包括亲和层析和/或离子交换层析。
本发明的另一方面提供一种药物组合物,包括上述多肽或蛋白和/或融合蛋白,任选地,以及药学上可接受的辅料。
本发明所述的药物组合物可以为片剂(包括糖衣片剂、膜包衣片剂、舌下片剂、口腔崩解片、口腔片剂等等)、丸剂、粉剂、颗粒剂、胶囊剂(包括软胶囊、微胶囊)、锭剂、糖浆剂、液体、乳剂、混悬剂、控制释放制剂(例如,瞬时释放制剂、缓释制剂、缓释微囊)、气雾剂、膜剂(例如,口服崩解膜剂、口腔粘膜-粘附膜剂)、注射剂(例如,皮下注射、静脉注射、肌内注射、腹膜内注射)、静脉滴注剂、透皮吸收制剂、软膏剂、洗剂、粘附制剂、栓剂(例如,直肠栓剂、阴道栓剂)、小药丸、鼻制剂、肺制剂(吸入剂)、眼睛滴剂等等、口服或胃肠外制剂(例如,静脉内、肌内、皮下、器官内、鼻内、皮内、滴注、脑内、直肠内等给药形式、给药至肿瘤的附近和直接给药至病变处)。优选的,所述的药物组合物为注射剂。
本发明所述的药学上可接受的辅料优选为药学上可接受的注射剂辅料,例如等渗的无菌盐溶液(磷酸二氢钠、磷酸氢二钠、氯化钠、氯化钾、氯化钙、氯化镁等,或上述盐的混合物),或干燥的例如是冷冻干燥的组合物,其适当地通过加入无菌水或生理盐水形成可注射溶质。
本发明另一方面提供一种上述多肽或蛋白、融合蛋白、药物组合物在制备预防和/或治疗纤维化疾病、脓毒症的药物中的用途。
优选的,所述的纤维化疾病为肺纤维化;
优选的,所述的脓毒症为重症脓毒症、脓毒症休克。
本发明中术语“预防”或“治疗”包括治疗性或预防性处理或措施,目标是预防或减慢靶向的病理性状况或病症。如果根据本发明的方法接收治疗量的本发明所述融合蛋白后,对象表现出可观察和/或可测量的特定疾病一个或多个体征和症状的减少或消失,则该对象被成功“预防”或“治疗”。
本发明提供的融合蛋白中,只保留了Slit2D2结构域,与^lit2分子相比分子量更小,组织渗透性更好,更易于药物开发;且在分析和实验的基础上,将^lit2D2结构域中氨基酸残基进行突变,与未突变蛋白相比,提高了融合蛋白的稳定性;上述融合蛋白为Slit2D2(C386S)与HSA蛋白相融合得到的,在提高药物的稳定性的同时延长了药物的体内代谢时间。本发明提供的融合蛋白在纤维化疾病,特别是肺纤维化,的预防和治疗方面比阳性对照药物效果更好,显示了良好的药物成药性。
附图说明
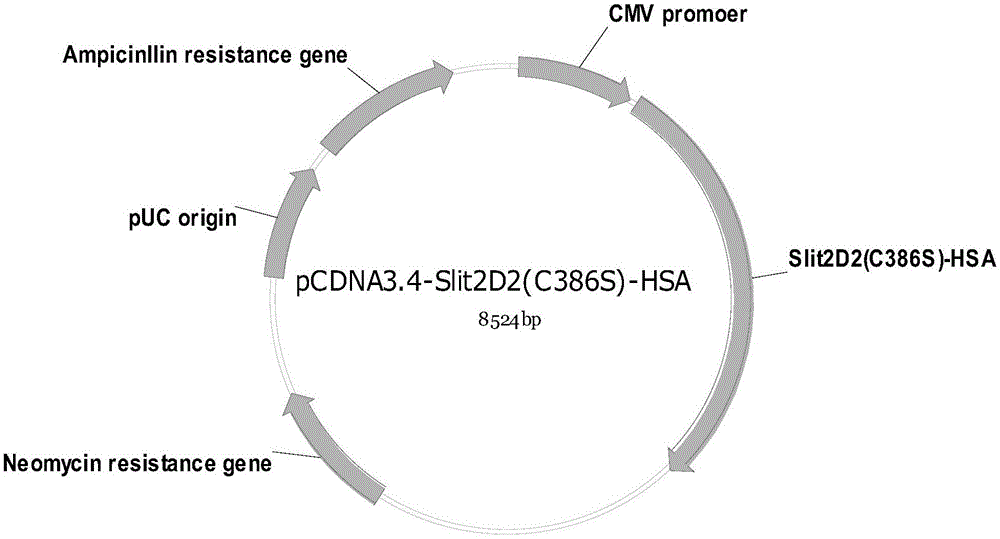
图1所示为本发明实施例1提供的重组载体pCDNA3.4-Slit2D2(C386S)-HSA图谱。
图2所示为本发明实施例1提供的Slit2D2(C386S)-HSA融合蛋白的SDS-PAGE蛋白电泳检测图;其中,A泳道:Slit2D2(C386S)-HSA变性蛋白;B泳道:Slit2D2(C386S)-HSA非变性蛋白。
图3所示为本发明实施例1提供的Slit2D2(C386S)-HSA融合蛋白的SEC-HPLC检测谱图。
图4所示为本发明实施例3提供的融合蛋白的稳定性检测结果。
图5所示为本发明实施例4提供的食蟹猴单次静脉注射给予2mg/kg的SLIT2D2(C386S)-HSA个体血浆药物浓度-时间曲线。
图6所示为本发明实施例4提供的食蟹猴单次静脉注射给予2mg/kg的SLIT2D2(C386S)-HSA雌雄均值血浆药物浓度-时间曲线。
图7所示为本发明实施例5提供的肺终末细支气管和伴行微小肺动脉损伤和炎症变化病理分析示意图。
图8所示为本发明实施例5提供的肺纤维化病理评分示意图(示意图中胶原纤维使用甲苯胺蓝染色)。
图9所示为本发明实施例5提供的实验期间动物的体重变化结果,其中,ZD018代表Slit2D2(C386S)-HSA。
图10所示为本发明实施例5提供的实验期间动物的体重变化百分比结果,其中,ZD018代表Slit2D2(C386S)-HSA。
图11所示为本发明实施例5提供的实验动物的左肺湿重变化结果。
图12所示为本发明实施例5提供的实验动物的左肺体积变化结果。
图13所示为本发明实施例5提供的实验动物的左肺灌注后的大体影像。
图14所示为本发明实施例5提供的实验动物的HE染色肺纤维化病灶区域。
图15所示为本发明实施例5提供的实验动物的肺纤维化病灶内细支气管和肺细小动脉损伤评分结果,One-way ANOVA:**p<0.001vs.group-1;***p<0.001vs.group-1;T-test:#p<0.05vs.group-2。
图16所示为本发明实施例5提供的实验动物的肺纤维化病灶边缘细支气管和肺细小动脉损伤评分结果,One-way ANOVA:**p<0.01vs.group-1;***p<0.001vs.group-1;T-test:#p<0.05vs.group-2。
图17所示为本发明实施例5提供的实验动物的Masson三色染色肺纤维化病灶区域。
图18所示为本发明实施例5提供的实验动物的肺纤维化病灶面积结果。
图19所示为本发明实施例5提供的实验动物的肺纤维化评分结果,T-test:*p<0.05vs.group-1;**p<0.01vs.group-1;***p<0.001vs.group-1。
图20所示为本发明实施例5提供的实验动物的肺纤维化评分百分比结果,Two-way ANOVA:***p<0.001vs.group-1;##p<0.01vs.group-2。
具体实施方式
下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有作出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
实施例1融合蛋白Slit2D2(C386S)-HSA的制备
根据已知的Slit2序列[GenBank:EAW92793.1]分析设计构建Slit2的第二个结构域Slit2D2,设计Slit2D2(C386S)如SEQ ID NO:1所示,进而设计Slit2D2(C386S)和Slit2D2(C386S)-HSA的编码序列分别如SEQ ID NO:2和SEQ ID NO:4所示。
通过全基因合成得到Slit2D2(C386S)-HSA的编码序列,利用T/A克隆将其插入到pCDNA3.4(品牌:Thermo,货号:A14697)表达载体,重组载体pCDNA3.4-Slit2D2(C386S)-HSA图谱如图1所示。将上述重组表达载体转化到大肠杆菌TOP10后转种到含有氨苄青霉素(AMP)的固体培养基中进行繁殖、筛选阳性克隆、通过测序确认载体构建成功及保种。
用无内毒素质粒提取试剂盒提取大肠杆菌TOP10中的重组质粒,用于转染ExpiCHO-STM细胞(Gibco Catalog No.A29127)细胞。培养ExpiCHO-STM细胞,当细胞密度达到4×106–6×106细胞/毫升时进行重组质粒的转染(转染试剂:ExpiFectamineTMCHO Transfection Kit,Gibco Catalog No.A29129),转染后培养10天,收集上清,高速离心,通过HSA亲和层析(层析填料:Thermo,货号191297050)和弱阴离子交换层析(品牌:天地人和,货号:DEAE Beads 6FF,SI005025)纯化Slit2D2(C386S)-HSA融合蛋白。
通过SDS-PAGE方法检测上述纯化得到的融合蛋白的分子量,并通过SEC-HPLC检测融合蛋白纯度。其中,SDS-PAGE结果和SEC-HPLC谱图分别如图2和3所示。由图2可知,已成功构建表达融合蛋白Slit2D2(C386S)-HSA的重组表达载体,并实现了在宿主细胞中表达和纯化融合蛋白Slit2D2(C386S)-HSA,纯度达到97.48%。
实施例2通过SPR测定融合蛋白对靶蛋白Robo1的亲和力
利用SPR(Surface Plasmon resonance BIAcore200)方法检测蛋白与Robo1蛋白之间的亲和常数,将Robo1(ORIGEN公司,货号:TP327713)蛋白结合在CM5芯片上,分析融合蛋白Slit2D2(C386S)-HSA(实施例1制备)、Slit2D2-HSA(参见专利申请PCT/CN2015/092079)和受体蛋白Robo1之间的相互作用。根据Canziani等人的方案(2004,Anal.Biochem.325:301-307)进行动力学测量。同时也用相同方法测定Slit2N蛋白(Slit2N为slit2蛋白N端的分子量约为120KDa的蛋白)与Robo1蛋白的亲和力。结果如表1所示。
表1 SPR测定融合蛋白与受体Robo1的亲和力的结果
实验结果表明:设计构建的融合蛋白Slit2D2-HSA和Slit2D2(C386S)-HSA与受体Robo1蛋白均具有较好的亲和能力,该能力与Slit2N蛋白具有相近的特性。
实施例3ELISA测定蛋白稳定性
1、试剂:
Neutroavidin-HRP稀释液;
包被缓冲液(Coating buffer):-0.16%Na2CO3;-0.3%NaHCO3;-pH9.8;
洗涤缓冲液(Washing buffer):含-0.1%Tween20的PBS;
封闭缓冲液(Blocking buffer):含-1%Goat Serum的洗涤缓冲液;
TMB:购自上海碧云天生物技术有限公司;
终止液(Stop solution):购自上海碧云天生物技术有限公司;
注:所有抗体都用封闭缓冲液稀释。
2、实验流程:
Robo1蛋白稀释为1μg/ml,100μl/well包板,4℃过夜。Washing buffer洗板3次。Blocking buffer(200μl/well)室温封闭2小时。Washing buffer洗板3次。分别加入待测样品(融合蛋白Slit2D2(C386S)-HSA(实施例1制备)、Slit2D2-HSA(参见专利申请PCT/CN2015/092079)100μl),室温孵育2小时。Washing buffer洗板3次。Anti-HSA-HRP抗体以1:10000稀释为1μg/ml,100μl/well包板,室温孵育1小时。Washing buffer洗板3次。Washing buffer洗板3次。每孔加入100μl TMB显色15分钟。每孔加入50μl Stop solution终止显色。读取450nm处的吸光值,实验结果如图4所示。
实验结果显示:Slit2D2(C386S)-HSA与Slit2D2-HSA的EC50值分别为10.56、214.6nM,二者接受处理后,Slit2D2(C386S)-HSA分子稳定性优于Slit2D2-HSA蛋白。
实施例4药代动力学检测
1.实验动物
1.1基本信息
品系和来源:食蟹猴,广西雄森灵长类实验动物养殖开发有限公司;
动物储备库:999M-014,
实验动物年龄:3.0-4.5岁;
试验开始前动物体重:2.75~3.00kg;
数量和性别:2雄,2雌。
1.2动物饲养
每只动物饲养在试验机构环境可调控的房间内,单笼饲养(不锈钢移动笼)。房间的温度和相对湿度每天记录两次。试验期间的温度和相对湿度在18~26℃和40~70%范围内。实际的温度和相对湿度记录保存在原始记录中。动物照明,明暗交替,每天大约各12小时。
实验猴配合饲料(生产批号1650230222和1650230527,失效日期分别为2016-05-21和2016-08-26,购于北京华阜康生物科技股份有限公司),试验期间动物自由采食。实验猴配合饲料由本试验机构委托的第三方PONY谱尼进行测试,对饲料中特定的微生物、重金属和农药残留进行检测。反渗透水通过水瓶不中断的供给各动物。申请人和委托的第三方定期对饮用水的pH、硬度、重金属和微生物进行检测,饲料和水的检测结果符合国家的相关规定。
2、试验所用主要计算机软件或计算机系统
表2试验所用主要计算机软件或计算机系统
3、试验方法
3.1试验设计
表3试验设计表
*:单次给药。
3.2给药
3.2.1给药方式
供试品通过静脉注射单次给药。
3.3检测指标
3.3.1观察
试验过程中所有动物在给药前、采血各个时间点,对动物进行观察;观察内容包括发病、损害、死亡率和供食供水情况。
3.4药代动力学及免疫原性研究
3.4.1动物数
所有动物,采集样本前动物无需禁食。
3.4.2生物样品采集
经眼眶采血,每个样品采集约2.0mL,置于含200μL 3.8%柠檬酸钠的Eppendorf管中,采集后放置冰上。
3.4.2.1采集时间
给药前(0h)、给药后3h、6h、12h、24h以及第4、6、8、11、14、17、19、22、26、29、36和43天各1次,共17个时间点。
3.4.3血浆样品处理
血液样本采集后离心分离血浆(离心条件:8000转/分钟,6分钟,2-8℃)。标签内容包括:美迪西普亚(上海)生物医药有限公司课题号、相关试验天数、动物编号、日期以及采集时间点。采集的血浆样本在分析前存放于≤-65℃冰箱内,分析后剩余血浆样本继续存放于≤-65℃冰箱,备用于后续处理。
3.4.4样品分析
生物样品分析方法及所有样品的分析由美迪西普亚(上海)生物医药有限公司分析实验室完成。样品分析的每一块板都必须含有标准曲线,构成标曲的点中至少3/4且不少于6个非零(不含锚定点)浓度点应该满足方法验证时的接收标准;每一块板也应该含有至少2套且每套包括至少3个不同浓度水平的质控样品(高浓度质控、中间浓度质控、低浓度质控),并要求占总数67%(2/3)以上且每个浓度水平的不低于50%的质控样品应该满足精密度在20%以内且准确度80-120%之间。
3.5动物处置
所有实验动物在试验结束后采集空白血浆,转入实验机构储备动物库。动物的处置将记录在试验记录上。
4、试验结果
4.1观察
试验过程中观察无异常发现。
4.2血浆药物浓度测定
食蟹猴静脉注射Slit2D2(C386S)-HSA血浆药物浓度测定结果见表4,药物浓度-时间曲线图见图5和6。
Slit2D2(C386S)-HSA组内不同性别动物间(除#101-Male外)血浆药物浓度-时间变化趋势基本一致或有一定的差异。
表4食蟹猴单次静脉注射给予2mg/kg的Slit2D2(C386S)-HSA后的血药浓度
NA:无/不适用;
BLQ:低于最低定量限;
LLOQ=1ng/mL。
4.3药代动力学参数
采用Phoenix药代动力学软件非房室模型计算药代动力学参数:AUC0-t、AUC0-∞、Cmax、t1/2、Tmax、C0、Cl、MRT0-∞和Vss。
静脉注射给药Slit2D2(C386S)-HSA药代动力学参数见表5。
Slit2D2(C386S)-HSA组雄性与雌性的t1/2比值为1.24,Cmax比值为1.11,AUC(0-∞)比值为1.41;组内不同性别动物间主要药物代谢动力学参数(t1/2、Cmax和AUC(0-∞))基本一致或有一定的差异,比值差异范围在1.11~1.41之内。
表5食蟹猴单次静脉注射给予2mg/kg的Slit2D2(C386S)-HSA的部分药动学参数
实施例5肺纤维化模型药效评估
1、实验材料
1.1实验动物
实验动物SD大鼠,SPF级,由北京维通利华实验动物有限公司提供,动物合格证号为11400700171426。
1.2造模剂
注射用盐酸博来霉素,购自日本化药株式会社;
规格:15mg/瓶;
批号:Y50512;
生产日期:2015年6月8日;
有效期至2017年6月7日。
1.3溶媒
生理盐水:安徽双鹤药业有限公司:产品批号:160502 8T;
甲基纤维素:Sigma,货号:M0512-100G,产品批号:079K0054V;
Tween 80:阿拉丁,货号:T104865-500ml,产品批号:K1519036;
DMSO:NA,货号:NA,产品批号:LE20Q62;
PEG400:Sigma,货号:NA,产品批号:MKBG7718V。
1.4供试品和对照品的配制
0.5%MC/0.2%Tween 80溶媒的配制:加热100mL的DDW至80-90℃,加入5g甲基纤维素搅拌均匀。移去热源,加入约400mL冰的DDW,并冰浴搅拌30分钟,然后转移溶液至1L的容量瓶中。恢复至室温后,加DDW至最终容积1L,搅拌直至获得澄清溶液。取2ml Tween 80加入1L的0.5%甲基纤维素溶液中溶解,涡旋后获得均匀溶液,4℃保存备用。
5.0mg/ml的PFD溶液的配制:称取1440mg的PFD粉末置于棕色配药瓶内,加入体积为288mL的0.5%MC/0.2%Tween 80溶液,超声水浴后获得均匀溶液。4℃保存用3天,之后重新配制。
Slit2D2(C386S)-HSA用PBS稀释配置;
阳性对照药:吡非呢酮(Pirfenidone),缩写PFD,用PBS稀释配置。
2、实验方法
2.1动物饲养
雄性SD大鼠,24只,由北京维通利华实验动物有限公司提供。动物饲养于南京百家汇医药研发平台动物中心SPF级屏障系统内,遵循国际标准温、湿、光控制系统。
2.2模型建立
动物采用异氟烷吸入麻醉;确认动物麻醉之后,消毒后剪开颈部皮肤,顿性分离肌肉暴露气管,沿气管环之间直接注入博来霉素(剂量3mg/kg,体积1.0mL/kg)。手术完毕后,将动物置于37℃电热毯保温至动物完全苏醒,确认能够自由采食和饮水后将动物返回饲养笼正常饲养。
2.3实验分组
本实验共分四组,即模型组(实验组-1,n=60)、PFD组(实验组-2,n=6)、Slit2D2(C386S)-HSA-1mg/kg(实验组-3,n=6)、Slit2D2(C386S)-HSA-5mg/kg(实验组-4,n=6),实验分组具体信息如表6所示。
表6实验分组
2.4试验给药
本实验阳性对照药PFD,大鼠灌胃给药,每天两次,造模后当天开始,连续给药14天。受试化合物Slit2D2(C386S)-HSA静脉注射给药,隔天一次,造模后当天开始,一共七次给药(如表6所示)。
2.5实验动物生理观察
实验动物生理观察:检测动物体重变化(每天给药前测量一次体重);监测试验周期内动物死亡率。
2.6试验终点
模型14天最后一次给药结束后24小时动物执行安乐死,确认动物死亡后,左肺肺内福尔马林灌注固定,测量灌注后左肺体积和重量,肺病理相关检测。
2.7肺脏病理学检测
大体病理检测:左肺等量福尔马林液灌注后采用微量天平分别称取/记录灌注后左肺湿重;采用微量量杯测量测量、记录灌注后左肺体积。
肺组织病理学检测:整肺脱水,石蜡块制作,肺石蜡切片,HE染色切片厚3μm,Masson Trichrome染色4μm切片。遵循病理标准染色SOP进行HE染色、Masson Trichrome染色,并通过Digital Pathscope切片扫描仪进行整片扫描。通过HE染色切片进行病灶周边终末细支气管和伴行微小肺动脉损伤和炎症变化病理分析和评分(如表7、8和图7所示)。通过Masson Trichrome染色切片进行病变面积计算,病变病理分析和评分(如表9和图8所示)。
表7终末细支气管损伤和炎症浸润病理评价指标
表8细小肺动脉损伤和炎症浸润病理评价指标
表9肺纤维化病理评价指标
2.8数据分析
使用graphpad prism软件,计算mean±sd或mean±sem,差异显著性检验使用t-test,one-way ANOVA和two-way ANOVA检验。p<0.05时认为两组之间具有显著性差异。
3、实验结果
3.1给药期间动物基本生理观察
给药期间所有实验动物无明显生理、行为异常变化。
3.2实验期间所有动物的体重变化
实验期间各组动物体重短时间内(5-7天内)均有轻度下降。随着试验进程所有动物体重均逐渐增加(结果如表10和图9、10)。各用药组动物体重恢复及增长与模型组基本一致。受试化合物组与模型组比较统计学上无显著性差异,证明药物的安全性。
表10动物体重变化(Mean±SEM)
3.3肺大体检测结果
动物左肺等量固定液灌注后的肺体积变化和湿重变化见表11。与模型组相比各给药组动物左肺无论是体积和湿重均缩小,但无显著性差异。各组病变肺组织的体积和湿重无显著差异。左肺肺纤维化两周后各实验组的肺部体积缩小,重量相应减轻,与模型组比较未见显著性差异(试验结果如图11、12和13所示)。
表11左肺重量和体积(Mean±SEM)
3.4左肺损伤病理评价
病变肺组织的组织学观察可见肺部组织界限较为清晰的显著性肺损伤(如图14所示),表现为细支气管、终末细支气管、肺泡管上皮细胞不同程度的增生,部分上皮乃至全层上皮杯状细胞化,管腔内可见不等量粘液组织。管壁不同程度的炎细胞浸润,部分管壁增厚,平滑肌增生以及管壁外模肉芽组织增生。病灶内肺泡组织不同程度损伤,表现为肺泡上皮脱落与再生,肺泡壁增厚,纤维化。肺泡腔内炎性渗出,炎细胞浸润。部分肺泡腔纤维化导致整体肺泡组织结构消失。各受试化合物的药效结果显示不同的受试化合物表现出不同的纤维化病灶内和病灶周边的细支气管以及伴行的细小肺动脉的损伤和炎症反应的疗效。对这两个区域进行损伤和炎症病理评分(如图15和16所示),结果显示纤维化病灶内以及病灶周边的终末细支气管和细小肺动脉的损伤经过受试化合物的治疗呈现不同程度的缓解。
3.5肺纤维化病理评价
肺组织学masson染色清晰可见左肺均匀一致的纤维化病灶以及病灶分布范围(如图17所示)。纤维化面积占左肺面积的计算结果显示模型组和各给药组之间病灶面积基本相同,提示本模型的稳定和均匀一致性(如图18所示),用于药效学评价该模型是可靠的。
采用Masson Trichrome染色对左肺肺纤维化病理改变和纤维化程度进行评分。组织学病变包括肺泡壁结果损伤,增厚,炎细胞浸润,胶原纤维沉积;肺泡腔内充满不均一的炎性渗出物,部分肺泡腔内纤维化团块。损伤严重部位肺组织正常结构完全消失,由纤维化和炎性肉芽肿组织取代。各受试化合物组显示不同的抑制纤维化的疗效。Ashcroft评分结果提示不同的受试化合物对肺纤维化的抑制、缓解疗效(如图19所示)。以肺纤维化评分的3分为界计算3分以下和4分以上的肺纤维化程度在整个纤维化评分中所占百分比,结果显示在模型组中小于等于3分以下的病灶百分比占整个病灶的20%左右。然而,经过药物治疗可见各不同受试化合物的药效反应(如图20所示)。
4、结论
BLM气管直接注射成功诱导左侧单侧肺纤维化。所有动物左肺BLM所致肺损伤面积均匀一致,占左肺切面的80%左右。各实验组之间无显著性差异,提示BLM诱导的左肺肺纤维化模型稳定。
阳性对照药(PFD)连续给药14天可见显著性抑制肺纤维化的进程。
隔天一次Slit2D2(C386S)-HSA静脉注射给药连续2周可见显著性抑制肺纤维化的进程,同时呈现出剂量依存性抑制肺纤维化进程的趋势。Slit2D2(C386S)-HSA 5mg/kg剂量组抑制肺纤维化的效果优于阳性化合物PFD。
以上所述仅为本发明的较佳实施例而已,并不用以限制本发明,凡在本发明的精神和原则之内,所作的任何修改、等同替换等,均应包含在本发明的保护范围之内。
SEQUENCE LISTING
<110> 李华顺
<120> 融合蛋白Slit2D2(C386S)-HSA及其在治疗纤维化疾病中的应用
<130> 1
<160> 4
<170> PatentIn version 3.3
<210> 1
<211> 209
<212> PRT
<213> 未知
<400> 1
Leu His Cys Pro Ala Ala Cys Thr Cys Ser Asn Asn Ile Val Asp Cys
1 5 10 15
Arg Gly Lys Gly Leu Thr Glu Ile Pro Thr Asn Leu Pro Glu Thr Ile
20 25 30
Thr Glu Ile Arg Leu Glu Gln Asn Thr Ile Lys Val Ile Pro Pro Gly
35 40 45
Ala Phe Ser Pro Tyr Lys Lys Leu Arg Arg Ile Asp Leu Ser Asn Asn
50 55 60
Gln Ile Ser Glu Leu Ala Pro Asp Ala Phe Gln Gly Leu Arg Ser Leu
65 70 75 80
Asn Ser Leu Val Leu Tyr Gly Asn Lys Ile Thr Glu Leu Pro Lys Ser
85 90 95
Leu Phe Glu Gly Leu Phe Ser Leu Gln Leu Leu Leu Leu Asn Ala Asn
100 105 110
Lys Ile Asn Ser Leu Arg Val Asp Ala Phe Gln Asp Leu His Asn Leu
115 120 125
Asn Leu Leu Ser Leu Tyr Asp Asn Lys Leu Gln Thr Ile Ala Lys Gly
130 135 140
Thr Phe Ser Pro Leu Arg Ala Ile Gln Thr Met His Leu Ala Gln Asn
145 150 155 160
Pro Phe Ile Cys Asp Cys His Leu Lys Trp Leu Ala Asp Tyr Leu His
165 170 175
Thr Asn Pro Ile Glu Thr Ser Gly Ala Arg Cys Thr Ser Pro Arg Arg
180 185 190
Leu Ala Asn Lys Arg Ile Gly Gln Ile Lys Ser Lys Lys Phe Arg Cys
195 200 205
Ser
<210> 2
<211> 627
<212> DNA
<213> 未知
<400> 2
ttgcactgcc ctgccgcctg tacctgtagc aacaatatcg tagactgtcg tgggaaaggt 60
ctcactgaga tccccacaaa tcttccagag accatcacag aaatacgttt ggaacagaac 120
acaatcaaag tcatccctcc tggagctttc tcaccatata aaaagcttag acgaattgac 180
ctgagcaata atcagatctc tgaacttgca ccagatgctt tccaaggact acgctctctg 240
aattcacttg tcctctatgg aaataaaatc acagaactcc ccaaaagttt atttgaagga 300
ctgttttcct tacagctcct attattgaat gccaacaaga taaacagtct tcgggtagat 360
gcttttcagg atctccacaa cttgaacctt ctctccctat atgacaacaa gcttcagacc 420
atcgccaagg ggaccttttc acctcttcgg gccattcaaa ctatgcattt ggcccagaac 480
ccctttattt gtgactgcca tctcaagtgg ctagcggatt atctccatac caacccgatt 540
gagaccagtg gtgcccgttg caccagcccc cgccgcctgg caaacaaaag aattggacag 600
atcaaaagca agaaattccg ttgttca 627
<210> 3
<211> 794
<212> PRT
<213> 未知
<400> 3
Leu His Cys Pro Ala Ala Cys Thr Cys Ser Asn Asn Ile Val Asp Cys
1 5 10 15
Arg Gly Lys Gly Leu Thr Glu Ile Pro Thr Asn Leu Pro Glu Thr Ile
20 25 30
Thr Glu Ile Arg Leu Glu Gln Asn Thr Ile Lys Val Ile Pro Pro Gly
35 40 45
Ala Phe Ser Pro Tyr Lys Lys Leu Arg Arg Ile Asp Leu Ser Asn Asn
50 55 60
Gln Ile Ser Glu Leu Ala Pro Asp Ala Phe Gln Gly Leu Arg Ser Leu
65 70 75 80
Asn Ser Leu Val Leu Tyr Gly Asn Lys Ile Thr Glu Leu Pro Lys Ser
85 90 95
Leu Phe Glu Gly Leu Phe Ser Leu Gln Leu Leu Leu Leu Asn Ala Asn
100 105 110
Lys Ile Asn Ser Leu Arg Val Asp Ala Phe Gln Asp Leu His Asn Leu
115 120 125
Asn Leu Leu Ser Leu Tyr Asp Asn Lys Leu Gln Thr Ile Ala Lys Gly
130 135 140
Thr Phe Ser Pro Leu Arg Ala Ile Gln Thr Met His Leu Ala Gln Asn
145 150 155 160
Pro Phe Ile Cys Asp Cys His Leu Lys Trp Leu Ala Asp Tyr Leu His
165 170 175
Thr Asn Pro Ile Glu Thr Ser Gly Ala Arg Cys Thr Ser Pro Arg Arg
180 185 190
Leu Ala Asn Lys Arg Ile Gly Gln Ile Lys Ser Lys Lys Phe Arg Cys
195 200 205
Ser Asp Ala His Lys Ser Glu Val Ala His Arg Phe Lys Asp Leu Gly
210 215 220
Glu Glu Asn Phe Lys Ala Leu Val Leu Ile Ala Phe Ala Gln Tyr Leu
225 230 235 240
Gln Gln Cys Pro Phe Glu Asp His Val Lys Leu Val Asn Glu Val Thr
245 250 255
Glu Phe Ala Lys Thr Cys Val Ala Asp Glu Ser Ala Glu Asn Cys Asp
260 265 270
Lys Ser Leu His Thr Leu Phe Gly Asp Lys Leu Cys Thr Val Ala Thr
275 280 285
Leu Arg Glu Thr Tyr Gly Glu Met Ala Asp Cys Cys Ala Lys Gln Glu
290 295 300
Pro Glu Arg Asn Glu Cys Phe Leu Gln His Lys Asp Asp Asn Pro Asn
305 310 315 320
Leu Pro Arg Leu Val Arg Pro Glu Val Asp Val Met Cys Thr Ala Phe
325 330 335
His Asp Asn Glu Glu Thr Phe Leu Lys Lys Tyr Leu Tyr Glu Ile Ala
340 345 350
Arg Arg His Pro Tyr Phe Tyr Ala Pro Glu Leu Leu Phe Phe Ala Lys
355 360 365
Arg Tyr Lys Ala Ala Phe Thr Glu Cys Cys Gln Ala Ala Asp Lys Ala
370 375 380
Ala Cys Leu Leu Pro Lys Leu Asp Glu Leu Arg Asp Glu Gly Lys Ala
385 390 395 400
Ser Ser Ala Lys Gln Arg Leu Lys Cys Ala Ser Leu Gln Lys Phe Gly
405 410 415
Glu Arg Ala Phe Lys Ala Trp Ala Val Ala Arg Leu Ser Gln Arg Phe
420 425 430
Pro Lys Ala Glu Phe Ala Glu Val Ser Lys Leu Val Thr Asp Leu Thr
435 440 445
Lys Val His Thr Glu Cys Cys His Gly Asp Leu Leu Glu Cys Ala Asp
450 455 460
Asp Arg Ala Asp Leu Ala Lys Tyr Ile Cys Glu Asn Gln Asp Ser Ile
465 470 475 480
Ser Ser Lys Leu Lys Glu Cys Cys Glu Lys Pro Leu Leu Glu Lys Ser
485 490 495
His Cys Ile Ala Glu Val Glu Asn Asp Glu Met Pro Ala Asp Leu Pro
500 505 510
Ser Leu Ala Ala Asp Phe Val Glu Ser Lys Asp Val Cys Lys Asn Tyr
515 520 525
Ala Glu Ala Lys Asp Val Phe Leu Gly Met Phe Leu Tyr Glu Tyr Ala
530 535 540
Arg Arg His Pro Asp Tyr Ser Val Val Leu Leu Leu Arg Leu Ala Lys
545 550 555 560
Thr Tyr Glu Thr Thr Leu Glu Lys Cys Cys Ala Ala Ala Asp Pro His
565 570 575
Glu Cys Tyr Ala Lys Val Phe Asp Glu Phe Lys Pro Leu Val Glu Glu
580 585 590
Pro Gln Asn Leu Ile Lys Gln Asn Cys Glu Leu Phe Glu Gln Leu Gly
595 600 605
Glu Tyr Lys Phe Gln Asn Ala Leu Leu Val Arg Tyr Thr Lys Lys Val
610 615 620
Pro Gln Val Ser Thr Pro Thr Leu Val Glu Val Ser Arg Asn Leu Gly
625 630 635 640
Lys Val Gly Ser Lys Cys Cys Lys His Pro Glu Ala Lys Arg Met Pro
645 650 655
Cys Ala Glu Asp Tyr Leu Ser Val Val Leu Asn Gln Leu Cys Val Leu
660 665 670
His Glu Lys Thr Pro Val Ser Asp Arg Val Thr Lys Cys Cys Thr Glu
675 680 685
Ser Leu Val Asn Arg Arg Pro Cys Phe Ser Ala Leu Glu Val Asp Glu
690 695 700
Thr Tyr Val Pro Lys Glu Phe Asn Ala Glu Thr Phe Thr Phe His Ala
705 710 715 720
Asp Ile Cys Thr Leu Ser Glu Lys Glu Arg Gln Ile Lys Lys Gln Thr
725 730 735
Ala Leu Val Glu Leu Val Lys His Lys Pro Lys Ala Thr Lys Glu Gln
740 745 750
Leu Lys Ala Val Met Asp Asp Phe Ala Ala Phe Val Glu Lys Cys Cys
755 760 765
Lys Ala Asp Asp Lys Glu Thr Cys Phe Ala Glu Glu Gly Lys Lys Leu
770 775 780
Val Ala Ala Ser Gln Ala Ala Leu Gly Leu
785 790
<210> 4
<211> 2382
<212> DNA
<213> 未知
<400> 4
ttgcactgcc ctgccgcctg tacctgtagc aacaatatcg tagactgtcg tgggaaaggt 60
ctcactgaga tccccacaaa tcttccagag accatcacag aaatacgttt ggaacagaac 120
acaatcaaag tcatccctcc tggagctttc tcaccatata aaaagcttag acgaattgac 180
ctgagcaata atcagatctc tgaacttgca ccagatgctt tccaaggact acgctctctg 240
aattcacttg tcctctatgg aaataaaatc acagaactcc ccaaaagttt atttgaagga 300
ctgttttcct tacagctcct attattgaat gccaacaaga taaacagtct tcgggtagat 360
gcttttcagg atctccacaa cttgaacctt ctctccctat atgacaacaa gcttcagacc 420
atcgccaagg ggaccttttc acctcttcgg gccattcaaa ctatgcattt ggcccagaac 480
ccctttattt gtgactgcca tctcaagtgg ctagcggatt atctccatac caacccgatt 540
gagaccagtg gtgcccgttg caccagcccc cgccgcctgg caaacaaaag aattggacag 600
atcaaaagca agaaattccg ttgttcagat gcacacaaga gtgaggttgc tcatcggttt 660
aaagatttgg gagaagaaaa tttcaaagcc ttggtgttga ttgcctttgc tcagtatctt 720
cagcagtgtc catttgaaga tcatgtaaaa ttagtgaatg aagtaactga atttgcaaaa 780
acatgtgttg ctgatgagtc agctgaaaat tgtgacaaat cacttcatac cctttttgga 840
gacaaattat gcacagttgc aactcttcgt gaaacctatg gtgaaatggc tgactgctgt 900
gcaaaacaag aacctgagag aaatgaatgc ttcttgcaac acaaagatga caacccaaac 960
ctcccccgat tggtgagacc agaggttgat gtgatgtgca ctgcttttca tgacaatgaa 1020
gagacatttt tgaaaaaata cttatatgaa attgccagaa gacatcctta cttttatgcc 1080
ccggaactcc ttttctttgc taaaaggtat aaagctgctt ttacagaatg ttgccaagct 1140
gctgataaag ctgcctgcct gttgccaaag ctcgatgaac ttcgggatga agggaaggct 1200
tcgtctgcca aacagagact caagtgtgcc agtctccaaa aatttggaga aagagctttc 1260
aaagcatggg cagtagctcg cctgagccag agatttccca aagctgagtt tgcagaagtt 1320
tccaagttag tgacagatct taccaaagtc cacacggaat gctgccatgg agatctgctt 1380
gaatgtgctg atgacagggc ggaccttgcc aagtatatct gtgaaaatca agattcgatc 1440
tccagtaaac tgaaggaatg ctgtgaaaaa cctctgttgg aaaaatccca ctgcattgcc 1500
gaagtggaaa atgatgagat gcctgctgac ttgccttcat tagctgctga ttttgttgaa 1560
agtaaggatg tttgcaaaaa ctatgctgag gcaaaggatg tcttcctggg catgtttttg 1620
tatgaatatg caagaaggca tcctgattac tctgtcgtgc tgctgctgag acttgccaag 1680
acatatgaaa ccactctaga gaagtgctgt gccgctgcag atcctcatga atgctatgcc 1740
aaagtgttcg atgaatttaa acctcttgtg gaagagcctc agaatttaat caaacaaaat 1800
tgtgagcttt ttgagcagct tggagagtac aaattccaga atgcgctatt agttcgttac 1860
accaagaaag taccccaagt gtcaactcca actcttgtag aggtctcaag aaacctagga 1920
aaagtgggca gcaaatgttg taaacatcct gaagcaaaaa gaatgccctg tgcagaagac 1980
tatctatccg tggtcctgaa ccagttatgt gtgttgcatg agaaaacgcc agtaagtgac 2040
agagtcacca aatgctgcac agaatccttg gtgaacaggc gaccatgctt ttcagctctg 2100
gaagtcgatg aaacatacgt tcccaaagag tttaatgctg aaacattcac cttccatgca 2160
gatatatgca cactttctga gaaggagaga caaatcaaga aacaaactgc acttgttgag 2220
ctcgtgaaac acaagcccaa ggcaacaaaa gagcaactga aagctgttat ggatgatttc 2280
gcagcttttg tagagaagtg ctgcaaggct gacgataagg agacctgctt tgccgaggag 2340
ggtaaaaaac ttgttgctgc aagtcaagct gccttaggct ta 2382
- 还没有人留言评论。精彩留言会获得点赞!