治疗血红蛋白病的材料和方法与流程
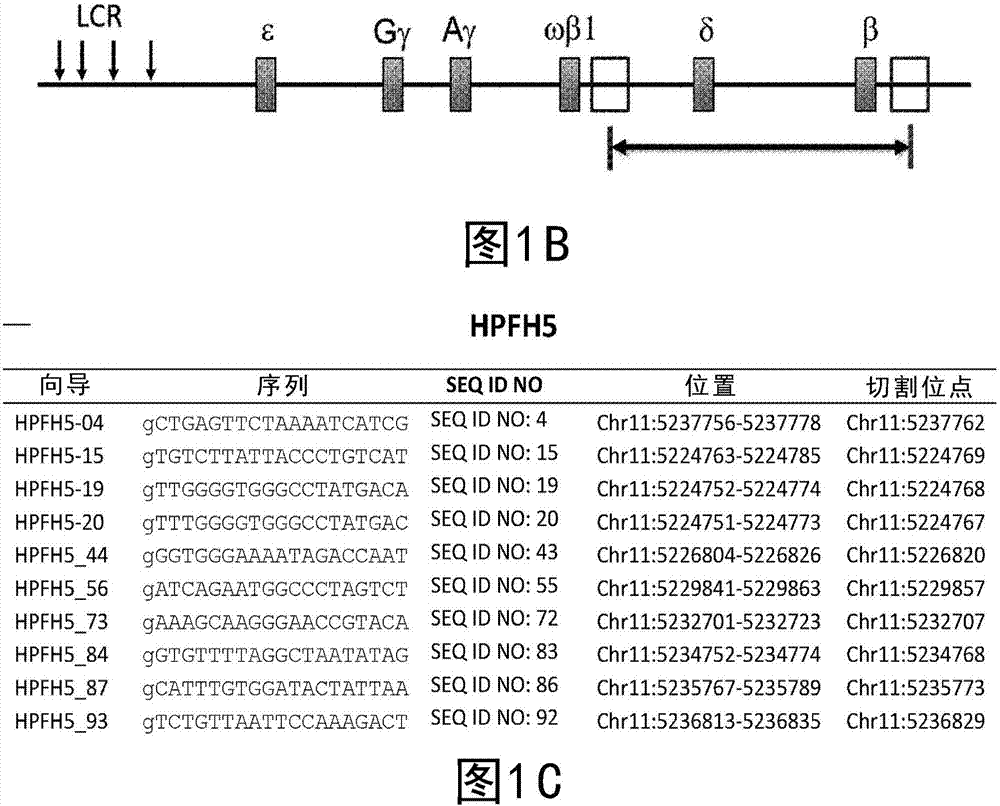
相关申请的交叉引用本申请要求提交于2015年2月23日的美国临时专利申请序列号62/119,754的权益,该美国临时专利申请全文以引用方式并入本文。序列表的引入序列表在此被提供为标题为“49064pct2_seqlisting.txt”的文本文件,该文本文件创建于2016年2月23日并且具有47,138字节的大小。此序列表的内容全文以引用方式并入本文。本申请提供用于治疗血红蛋白病的材料和方法。更特别地,本申请提供用于产生通过基因组编辑增加胎血红蛋白(hbf)的产生而经遗传修饰的祖细胞的方法,以及产生增加水平的hbf的经修饰的祖细胞(包括例如cd34+人造血干细胞(hhsc)),以及使用这种细胞治疗血红蛋白病(诸如镰状细胞性贫血和β-地中海贫血)的方法。
背景技术:
:血红蛋白病涵盖与遗传决定的血红蛋白结构或表达方面的变化相关的多种贫血。这些包括对血红蛋白链的分子结构的改变,诸如伴随镰状细胞性贫血发生的,以及其中一个或更多个链的合成减少或缺乏的变化,诸如在多种地中海贫血中所发生的。与β-球蛋白特别相关的疾病通常被称为β-血红蛋白病。例如,β-地中海贫血是由于β球蛋白基因表达的部分或完全缺陷导致血红蛋白a(hba)缺乏或不存在。hba是最常见的人血红蛋白四聚体并且由两个α-链和两个β链组成(α2β2)。β-地中海贫血是由于染色体11上的成体β-球蛋白基因(hbb)的突变,并以常染色体隐性方式遗传。β-地中海贫血(β-thalassemia或β-thal)分为通过症状严重程度区分的两种临床显著类型(这是症状管理、医学治疗和本申请的焦点):重型β-地中海贫血症(或β0,其中突变阻断β-球蛋白链的产生,从而导致也被称为“库利氏贫血(cooley'sanemia)”的严重病症),以及中间型β-地中海贫血(或β+,其中突变减少但不阻断β-球蛋白的产生的中间病症)。相比之下,轻型β-地中海贫血或轻度β-地中海贫血是指其中仅一个β-球蛋白等位基因中含有突变,使得可以通过从另一(即未突变)染色体11等位基因的表达产生β-球蛋白链的杂合情况。虽然这种个体是可能遗传给他们的孩子的β-地中海贫血突变等位基因的携带者,但是由于从未受影响的等位基因产生β-球蛋白,所以患有轻型β-地中海贫血的个体自身通常无症状或几乎无症状。重型地中海贫血的病征和症状通常出现在生命的头2年,此期间患有这种疾病的儿童可能会发展出危及生命的贫血。患有重型地中海贫血的儿童往往不能以预期的速率获得足够的体重或生长(发育不良),并可能发展出黄疸。受影响的个体也可能具有脾脏、肝脏和心脏肿大,并且这些个体的骨骼也可能畸形。许多患有重型地中海贫血的人具有需要频繁输血来补充他们的红血细胞供应的严重症状,其被称为输血依赖性地中海贫血。虽然输注已经成为许多患者的关键救生手段,但是输注是昂贵的并且经常与显著的副作用相关。除了别的以外,随着时间的推移,来自长期输血的含铁血红蛋白的施用趋向于导致体内铁的积累,这可能导致肝脏、心脏和内分泌问题。中间型地中海贫血比重型地中海贫血更轻度。中间型地中海贫血的病征和症状出现在儿童期早期或生命的稍后时期中。虽然症状不那么严重,但受影响的个体仍然具有轻度至中度贫血并且还可能遭受缓慢生长和骨骼畸形。镰状细胞病(scd)是一组影响全球数百万人的疾病。这种疾病在居住在非洲;地中海国家,诸如希腊、土耳其和意大利;阿拉伯半岛;印度;中美洲和南美洲中的西班牙语地区,以及加勒比的部分地区或其祖先来自于上述的人中最常见。然而,scd也是美国最常见的遗传性血液疾病。scd包括镰状细胞性贫血,以及镰状血红蛋白c疾病(hbsc),镰状β加地中海贫血(hbs/β+)和镰状β零地中海贫血(hbs/β0)。镰状细胞性贫血(sca)是最普遍存在的scd形式,是世界上最常见的严重单基因遗传病之一,每年大约有25万儿童生来患有scd。西非和中非地区的sca发病率最高,其中1%至2%的婴儿生来患有该疾病,并且多达25%的人是杂合携带者。sca点突变被认为已经通过选择性优势传播,因为杂合性提供抗儿童疟疾死亡的适度保护。在疟疾同样普遍存在的印度,据估计存在超过250万名sca杂合携带者和大约15万患有该疾病的纯合体。尽管北美和欧洲疟疾相对不存在,但是事实上北美和欧洲各自有大量具有受影响地区遗传来源的群体,这意味着北美和欧洲地区都有大量的杂合性sca携带者群体,以及由此造成的受影响的纯合个体。例如,美国疾病控制中心(cdc)估计,存在约90,000到100,000名美国人患有sca;而在西欧国家,特别是具有大量移民人口的那些国家发病率也很高,例如估计在法国为10,000人,而在英国则为12,000至15,000人。医疗保健系统的相关成本同样很大。在1989年至1993年进行的为期五年的美国研究中,cdc估计,每年scd导致大于75,000次住院,费用为约5亿美元。现鉴于医疗保健成本在二十年间稳步上升,预计系统范围成本将大幅增加。所有形式的scd均由β-球蛋白结构基因(hbb)中的突变引起。镰状细胞性贫血(sca)是由β-球蛋白基因的第六密码子的单个错义突变(hbb;a→t)导致的由缬氨酸取代谷氨酸(glu→val)而引起的常染色体隐性遗传病。与正常成体血红蛋白或hba(其为α2β2a)相比,突变型蛋白当掺入血红蛋白(hb)时会导致不稳定的血红蛋白hbs(其为α2β2s)。在脱氧时,hbs通过红细胞系中的一个四聚体的βs-6缬氨酸与相邻四聚体的β-85苯丙氨酸和β-88亮氨酸之间的疏水相互作用聚合形成hbss,这导致强直和血管闭塞[atweh,semin.hematol.38(4):367-73(2001)]。当hbs是血红蛋白的主要形式时,如在患有sca的个体中,他们的红细胞(rbc)倾向于变形成镰状或新月形。镰状rbc过早死亡,这可导致贫血。此外,镰状细胞比正常的rbc更不灵活并且倾向于卡在小血管中,从而引起血管闭塞事件。这种血管闭塞事件与组织缺血相关,导致急性和慢性疼痛,以及可影响体内任何器官(包括骨骼、肺、肝、肾、脑、眼睛和关节)的器官损伤。脾脏特别地受到梗塞,并且大多数患有scd的个体在幼儿期中功能上无脾,从而增加了他们患上某些类型细菌感染的风险。小血管阻塞也可引起被称为“危象”的急性发作性发热性疾病,该疾病伴有严重的疼痛和多器官功能障碍。在几十年的过程中,存在进行性器官疾病和过早死亡。患有scd的儿童可以通以新生儿筛查诊断出,但未诊断出的话则直到稍后当由于血红蛋白等位基因从胎血红蛋白(由hbg1(a-γ,也写作aγ)和hbg2(g-γ,也写作gγ)编码)“转换”成由hbb编码的成体β形式)而使胎血红蛋白(hbf)水平下降并且hbs水平升高时才会呈现出。从hbf到成体形式的β-球蛋白(即未受影响的儿童体内中的hba或患有sca的那些儿童体内中的hbs)的转换通常在出生前几个月开始,并且在约6月龄时完成。scd的临床效力直到hbf水平变成相对于hbs显著低时才明显,这通常在出生后两至三个月发生。scd通常首先表现为趾炎或“手足综合征”,这是一种可能伴有肿胀的与手和/或足疼痛相关的病症。此外,脾脏可能会变成充满血细胞,从而导致被称为“脾隔离症”的病症。与scd相关的溶血可能导致贫血、黄疸、胆石病,以及延迟生长。具有最高scd溶血率的个体还倾向于经受肺动脉高压、持续勃起症和腿溃疡。镰状细胞性贫血(纯合hbss)占美国镰状细胞病的60%至70%。其他形式的镰状细胞病是由于hbs与其他异常球蛋白β链变异体的共遗传而产生的,最常见的形式是镰状血红蛋白c病(hbsc)和两种类型的镰状β-地中海贫血(hbsβ+-地中海贫血和hbsβ°-地中海贫血)。β-地中海贫血分为β+-地中海贫血(其中产生降低水平的正常β-球蛋白链)和β°-地中海贫血(其中没有β-球蛋白链合成)。其他球蛋白β链变体如d-punjab、o-arab和e也在与hbs共同遗传时导致镰状细胞疾病。虽然scd管理的改善已经降低了由新生儿筛查跟踪的受影响儿童的死亡率,但对大多数患有scd的个体的治疗支持仍然是支持性的。目前的治疗旨在缓解症状和治疗并发症,诸如:因血管闭塞性危象引起的疼痛、感染、贫血、中风、持续勃起症、肺高压或慢性器官损伤。预防性疗法包括使用常规青霉素的感染预防、抗肺炎链球菌(streptococcuspneumoniae)和流感嗜血杆菌(haemophilusinfluenzae)的疫苗接种、以及对具有异常经颅多普勒超声的儿童进行定期输血,以预防中风和因输血铁超载造成的铁螯合。中风也被认为是儿童和青少年骨髓移植的指征,他们具有有同样的人白细胞抗原(hla)的兄弟姐妹。急性疼痛的有效治疗是sca管理中出现的最常见问题之一。因此,目前,显著改变疾病的自然病程的明确疗法(如定期输血或交换输血、长期羟基脲和hsc移植)是有限的。wo2014/085593涉及通过靶向bcl11a远端调节元件来治疗血红蛋白病的方法和组合物,所述bcl11a远端调节元件据称通过抑制γ-球蛋白诱导来充当胎血红蛋白表达的阶段特异性调节因子。因此,例如,wo2014/085593的权利要求1涉及一种用于产生具有降低的bcl11amrna或蛋白质表达的祖细胞的方法,该方法包括使分离的祖细胞与结合染色体2的位置60,716,189至60,728,612(根据ucsc基因组浏览器hg19人基因组集合)上的细胞基因组dna的试剂接触,从而降低bcl11a的mrna或蛋白质表达。出于这些和其他目标,长期以来基因治疗已经被提出作为血红蛋白病的潜在治愈选择(参见例如demontalembert,bmj,337:a1397(2008);sheth等人,britishjournalofhaematology,162:455-464(2013),及其中引用的参考文献)。然而,正如chandrakasan和malik在题为“genetherapyforhemoglobinopathies:thestateofthefieldandthefuture”的综述[hematoloncolclinnortham.28(2):199–216(2014)]中所最近总结的,血红蛋白病的基因治疗面临一些挑战。例如,逆转录病毒(rv)载体是用于临床试验中的第一载体,并且尽管具有长末端重复序列(ltr)的载体完整介导高水平的转基因表达从而导致临床改善,但是试验的成功很快被通过rvltr从细胞癌基因的转录激活插入肿瘤发生的安全性问题所破坏。x-scid中的淋巴增殖和白血病归因于lmo2癌基因的插入激活。在慢性肉芽肿病(cgd)的基因治疗试验中,在一些初步的成功之后,存在由病毒启动子的甲基化引起的转基因表达沉默,以及由于嗜亲性病毒整合位点1的插入激活而发展出的具有单体性7的脊髓发育不良。参见chandrakasan和malik,出处同上,以及其中所引用的参考文献。没有任何致病因素的hiv-1的生物工程导致了慢病毒(lv)载体的发展。初步研究已将lv载体确立为高效基因转移的可靠载体。bluebirdbio公司正在开发bb305作为潜在的治疗方法,其中自体cd34+造血干细胞(hsc)用慢病毒βat87q-球蛋白载体离体转导,目的是在患有重型β-地中海贫血的患者中插入完全功能化的人β-球蛋白基因。bluebird研究旨在以来自lg001研究的早期临床资料为基础,在lg001研究中已经将药物产品施用于患有重型β-地中海贫血的患者[cavazzana-calvo等人,nature,467:318-322(2010)]。还已经考虑了使用γ-球蛋白的基因治疗。然而,已知γ-球蛋白转录物在成年人中是高度沉默的,因此回避此的方法包括用β-球蛋白启动子和增强子驱动γ-球蛋白表达,如chandrakasan和malik所述,出处同上。然而,在基因治疗的情境下引入强启动子和增强子,特别是使用在基因组内的不可预测的位置处整合的载体(包括rv和lv载体))引起了安全性考虑,因为原癌基因的激活或其他有害事件可能因引入此类元件而被触发。在重症联合免疫缺陷疾病(scid)试验中,例如20例患者中有5例发生与其治疗相关的白血病[wu等人,frontmed.5(4):356–371(2011)]。总而言之,尽管有来自一直在努力解决血红蛋白病(诸如β-地中海贫血和镰状细胞病)的全球研究人员和医疗专业人士的数十年努力,并且尽管有基因治疗方法的希望,但仍然迫切需要开发用于这些和相关疾病(它们是最流行和致衰弱的遗传疾病之一)的安全有效的治疗方法。发明概述本文提供了通过基因组编辑增加人细胞中胎血红蛋白(hbf;其两条多肽链从γ-球蛋白基因表达,如下所述)水平的方法,其可用于治疗血红蛋白病诸如β-地中海贫血和镰状细胞病;并且提供了用于进行此种方法的组分、试剂盒和组合物、以及由它们产生的细胞,包括但不限于可施用于患有血红蛋白病的患者的自体cd34+人造血干细胞(hhsc)。在一个方面,本文提供了通过基因组编辑来增加人细胞中的hbf水平的方法,其使用dna核酸内切酶实现一对双链断裂(dsb),第一断裂位于人染色体11的δβ-球蛋白区域内的5'dsb位点处,第二断裂位于人染色体11的δβ-球蛋白区域内的3'dsb位点处,从而引起5'dsb位点与3'dsb位点之间的区域的dna缺失,这导致γ-球蛋白基因中的任一个或两者的表达增加,从而导致细胞中hbf的水平增加。在另一方面,本文提供了通过基因组编辑来增加人细胞中的hbf水平的方法,其使用dna核酸内切酶实现一对dsb,第一断裂位于人染色体11的δβ-球蛋白区域内的5'dsb位点处,第二断裂位于人染色体11的δβ-球蛋白区域内的3'dsb位点处,从而引起5'dsb位点与3'dsb位点之间的区域的倒位(inversion),这导致γ-球蛋白的表达增加,从而导致细胞中hbf的水平增加。在另一方面,本文提供了通过基因组编辑来增加人细胞中hbf水平的方法,其使用dna核酸内切酶实现定位于人染色体11的β-球蛋白区域内的一个或更多个位点处的dsb,从而引起该一个或更多个位点处的染色体dna缺失或插入,这导致γ-球蛋白的表达增加,从而增加细胞中hbf的水平。在例示这方面的一种类型的方法中,至少一个dsb位于人染色体11的γ-球蛋白调节区内。在例示这一方面的另一种类型的方法中,至少一个dsb位于人染色体11的δβ-球蛋白区域内。可使用的示例性dna核酸内切酶包括例如cas9核酸内切酶、锌指核酸酶、转录激活子样效应核酸酶(talen)、归巢核酸内切酶、dcas9-foki核酸酶或megatal核酸酶。可以通过多种方式将dna核酸内切酶引入细胞中,包括通过引入和/或表达编码dna核酸内切酶的一个或更多个多核苷酸,如在本领域中已知的并且如本文所进一步描述和说明的。在一些实施方案中,dna核酸内切酶和/或基因组编辑系统的其它组分(诸如cas9基因组编辑情况中的向导rna)是由被引入细胞中的rna编码的。在一些实施方案中,dna核酸内切酶是cas9核酸内切酶,并且所述方法包括向细胞中引入编码cas9的一个或更多个多核苷酸和两个向导rna,第一向导rna包含与5'dsb位点的区段互补的间隔序列,并且第二向导rna包含与3'dsb位点的区段互补的间隔序列。两个向导rna可以作为单分子向导rna(包括tracrrna和crisprrna)提供,或者其中的任一者或两者可以作为双分子向导rna提供,该双分子向导rna包含彼此不连接而是作为分开的分子的crisprrna和tracrrna。在另一些实施方案中,dna核酸内切酶是锌指核酸酶(zfn),并且该方法包括向细胞中引入编码靶向5'dsb位点的区段的第一对zfn和靶向3'dsb位点的区段的第二对zfn的一个或更多个多核苷酸。可替换地,可以使用talen或其他核酸内切酶。在一些实施方案中,待修饰的人细胞是分离的祖细胞,并且在一些用于治疗血红蛋白病的实施方案中,待修饰的人细胞是能够产生红系谱系(erythroidlineage)的细胞的造血祖细胞。分离的祖细胞也可以是经诱导多能干细胞。在多种实施方案中,一个或两个dsb位点靠近于与胎血红蛋白(hpfh)或δβ-地中海贫血corfu的遗传性持续相关的缺失,如本文进一步描述和说明的。(术语“靠近于”在本文中是指在特定参考点的5'端或3'端的限定区域内或附近的位置,在本文中特别地参照这些缺失使用,并且在下文进一步在标题为“靶序列选择”的部分中进一步详细描述,并通过本文提供的多种示例性实施方案进一步说明。)与hpfh和δβ-地中海贫血corfu相关的缺失都与hbf的增加有关,并且在本文中统称为hpfh缺失,在本文中描述和说明了这些缺失中的一些,并且其他缺失是本领域中已知的。因此,5'dsb位点可靠近于hpfh缺失的5'边界,3'dsb位点可靠近于hpfh缺失的3'边界,或者为两者,这会导致模仿天然存在的hpfh缺失的缺失。如本文所示的示例性缺失包括例如hpfh-4缺失、hpfh-5缺失、hpfh-kenya缺失、hpfh-black缺失、小缺失,长corfu缺失和短corfu缺失。还提供了具有共享一个或更多个区段的缺失实施方案,所述区段在hpfh中缺失并且与增加的hbf水平相关但不与天然存在的缺失相连(co-terminous)。在一些方面,如本文进一步描述的,缺失移除全部或部分δβ-球蛋白区域。在一些方面,如本文进一步描述的,缺失移除全部或部分β-球蛋白基因(hbb)。在镰状细胞病的情境中,破坏或消除β-球蛋白基因可以有效地减少或消除镰状细胞血红蛋白(hbs)的表达,这除了提高胎血红蛋白(hbf)的水平外,还可以对患有scd(如镰状细胞性贫血)的患者有显著附加益处。在一些实施方案中,所述方法涉及对来自患有scd的患者的细胞进行基因组编辑,其中hbf增加并且hbs减少。在β-地中海贫血的情境中,与β-球蛋白链缺乏相关的问题由于过量的不成对α-球蛋白链而加剧,所述不成对α-球蛋白链与红细胞(rbc)膜相互作用,从而导致对膜骨骼组分以及潜在地其他组分的氧化损伤,从而导致rbc存活期缩短、红细胞生成不良和贫血。在一些实施方案中,所述方法涉及对来自患有β-地中海贫血的患者的细胞进行基因组编辑,其中hbf增加并且不配对的α-球蛋白链的水平降低。还提供了通过前述方法修饰以增加其hbf水平的人细胞。在一些实施方案中,细胞来源于患有scd的患者,并且这种细胞中的hbs水平降低。在某些其他实施方案中,细胞来源于患有β-地中海贫血的患者,并且这种细胞中不配对的α-球蛋白链的水平降低。此类细胞可以是分离的祖细胞,例如能够产生红细胞系细胞的造血祖细胞。分离的祖细胞可以是诱导的多能干细胞。本文进一步提供了通过施用已经用前述方法修饰以增加其hbf水平的细胞来改善血红蛋白病的方法。示例性血红蛋白病包括但不限于镰状细胞病(包括镰状细胞性贫血)、血红蛋白c疾病、血红蛋白c性状(trait)、血红蛋白s/c疾病、血红蛋白d疾病、血红蛋白e疾病、地中海贫血、具有增加的氧亲和力的血红蛋白相关病症、具有降低的氧亲和力的血红蛋白相关病症、不稳定血红蛋白病和高铁血红蛋白症。本文提供了一种通过基因组编辑增加人细胞中胎血红蛋白(hbf)水平的方法,所述方法包括以下步骤:向人细胞中引入一种或更多种化脓性链球菌(s.pyogenes)cas9脱氧核糖核酸(dna)核酸内切酶和一种或两种核糖核酸(rna)向导序列,以实现一对双链断裂(dsb),第一dsb位于人染色体11的δβ-球蛋白区域内的5'dsb位点处,而第二dsb位于人染色体11的δβ-球蛋白区域内的3'dsb位点处,从而引起在5'dsb位点和3'dsb位点之间的染色体dna的缺失或倒位,这导致γ-球蛋白的表达增加,从而增加了细胞中的hbf水平。本文还提供了一种通过基因组编辑来编辑人细胞中人染色体11的δβ-球蛋白区域的方法,该方法包括以下步骤:向人细胞中引入一种或更多种化脓性链球菌cas9脱氧核糖核酸(dna)核酸内切酶和一种或两种核糖核酸(rna)向导,以实现一对双链断裂(dsb),第一dsb位于人染色体11的δβ-球蛋白区域内的5'dsb位点处,而第二dsb位于人染色体11的δβ-球蛋白区域内的3'dsb位点处,从而引起在5'dsb位点和3'dsb位点之间的染色体dna的缺失或倒位,这导致γ-球蛋白的表达增加,从而增加了细胞中的胎血红蛋白(hbf)的水平。本文提供了一种用于治疗患有血红蛋白病的患者的离体方法,该方法包括以下步骤:i)产生患者特异性的诱导多能干细胞(ipsc);ii)在ipsc的人染色体11的δβ-球蛋白区域内进行编辑;iii)将经基因组编辑的ipsc分化成造血祖细胞或白血细胞;以及iv)将造血祖细胞或白血细胞植入患者体内。在一些实施方案中,产生患者特异的诱导多能干细胞(ipsc)的步骤包括:a)从患者中分离体细胞;以及b)将一组多能性相关基因引入体细胞以诱导体细胞变成多能干细胞。在一些实施方案中,体细胞是成纤维细胞。在一些实施方案中,多能性相关基因的所述组是选自由oct4、sox2、klf4、lin28、nanog和cmyc组成的组的基因中的一种或更多种。在一些实施方案中,在ipsc的人染色体11的δβ-球蛋白区域内编辑的步骤包括:向ipsc中引入一种或更多种化脓性链球菌cas9脱氧核糖核酸(dna)核酸内切酶和一种或两种核糖核酸(rna)向导,以实现一对双链断裂(dsb),第一dsb位于人染色体11的δβ-球蛋白区域内的5'dsb位点处,而第二dsb位于人染色体11的δβ-球蛋白区域内的3'dsb位点处,从而导致5'dsb位点和3'dsb位点之间的染色体dna的缺失或倒位,这导致γ-球蛋白的表达增加,从而增加细胞中胎血红蛋白(hbf)的水平。在一些实施方案中,将基因组编辑的ipsc分化成造血祖细胞或白血细胞的步骤包括以下步骤中的一个或更多个以将基因组编辑的ipsc分化成造血祖细胞或白血细胞:用小分子的组合进行处理或递送主转录因子(mastertranscriptionfactor)。在一些实施方案中,将造血祖细胞或白血细胞植入患者体内的步骤包括通过局部注射、全身输注或其组合将造血祖细胞或白血细胞植入患者体内。本文提供了一种用于治疗患有血红蛋白病的患者的离体方法,该方法包括以下步骤:i)从患者分离白血细胞;ii)在白血细胞的人染色体11的δβ-球蛋白区域内编辑;以及iii)将基因组编辑的白血细胞植入患者体内。在一些实施方案中,从患者分离白血细胞的步骤包括:细胞差速离心、细胞培养、或它们的组合。在一些实施方案中,在白血细胞的人染色体11的δβ-球蛋白区域内编辑的步骤包括:向白血细胞中引入一种或更多种化脓性链球菌cas9脱氧核糖核酸(dna)核酸内切酶和一种或两种核糖核酸(rna)向导,以实现一对双链断裂(dsb),第一dsb位于人染色体11的δβ-球蛋白区域内的5'dsb位点处,而第二dsb位于人染色体11的δβ-球蛋白区域内的3'dsb位点处,从而导致5'dsb位点和3'dsb位点之间的染色体dna的缺失或倒位,这导致γ-球蛋白的表达增加,从而增加细胞中胎血红蛋白(hbf)的水平。在一些实施方案中,将基因组编辑的白血细胞植入患者体内的步骤包括通过局部注射、全身输注或其组合将基因组编辑的白血细胞植入患者体内。本文提供了一种用于治疗患有血红蛋白病的患者的离体方法,该方法包括以下步骤:i)从患者分离间充质干细胞;ii)在间充质干细胞的人染色体11的δβ-球蛋白区域内编辑;iii)将基因组编辑的间充质干细胞分化成造血祖细胞或白血细胞;以及iv)将造血祖细胞或白血细胞植入患者体内。在一些实施方案中,从患者的骨髓或外周血中分离间充质干细胞。在一些实施方案中,从患者中分离间充质干细胞的步骤包括:抽吸骨髓和使用percoll通过进行密度梯度离心来分离间充质细胞。在一些实施方案中,在间充质干细胞的人染色体11的δβ-球蛋白区域内编辑的步骤包括:向间充质干细胞中引入一种或更多种化脓性链球菌cas9脱氧核糖核酸(dna)核酸内切酶和一种或两种核糖核酸(rna)向导,以实现一对双链断裂(dsb),第一dsb位于人染色体11的δβ-球蛋白区域内的5'dsb位点处,而第二dsb位于人染色体11的δβ-球蛋白区域内的3'dsb位点处,从而导致5'dsb位点和3'dsb位点之间的染色体dna的缺失或倒位,这导致γ-球蛋白的表达增加,从而增加细胞中胎血红蛋白(hbf)的水平。在一些实施方案中,将基因组编辑的间充质干细胞分化成造血祖细胞或白血细胞的步骤包括以下步骤中的一个或更多个以将基因组编辑的间充质干细胞分化成造血祖细胞或白血细胞:用小分子的组合进行处理或递送主转录因子。在一些实施方案中,将造血祖细胞或白血细胞植入患者体内的步骤包括通过局部注射、全身输注或其组合将造血祖细胞或白血细胞植入患者体内。本文提供了一种用于治疗患有血红蛋白病的患者的离体方法,该方法包括以下步骤:i)从患者分离造血祖细胞;ii)在造血祖细胞的人染色体11的δβ-球蛋白区域内编辑;以及iii)将基因组编辑的造血祖细胞植入患者体内。在一些实施方案中,该方法还包括在分离步骤之前用粒细胞集落刺激因子(gcsf)处理患者。在一些实施方案中,该处理步骤与普乐沙福(plerixaflor)组合进行。在一些实施方案中,从患者分离造血祖细胞的步骤包括分离cd34+细胞。在一些实施方案中,在造血祖细胞的人染色体11的δβ-球蛋白区域内编辑的步骤包括:向造血祖细胞中引入一种或更多种化脓性链球菌cas9脱氧核糖核酸(dna)核酸内切酶和一种或两种核糖核酸(rna)向导,以实现一对双链断裂(dsb),第一dsb位于人染色体11的δβ-球蛋白区域内的5'dsb位点处,而第二dsb位于人染色体11的δβ-球蛋白区域内的3'dsb位点处,从而导致5'dsb位点和3'dsb位点之间的染色体dna的缺失或倒位,这导致γ-球蛋白的表达增加,从而增加细胞中胎血红蛋白(hbf)的水平。在一些实施方案中,将基因组编辑的造血祖细胞植入患者体内的步骤包括通过局部注射、全身输注或其组合将基因组编辑的造血祖细胞植入患者体内。本文提供了一种用于治疗患有血红蛋白病的患者的体内方法,该方法包括在患者的人染色体11的δβ-球蛋白区域内进行编辑的步骤。在一些实施方案中,在患者的人染色体11的δβ-球蛋白区域内编辑的步骤包括:向细胞中引入一种或更多种化脓性链球菌cas9脱氧核糖核酸(dna)核酸内切酶和一种或两种核糖核酸(rna)向导,以实现一对双链断裂(dsb),第一dsb位于人染色体11的δβ-球蛋白区域内的5'dsb位点处,而第二dsb位于人染色体11的δβ-球蛋白区域内的3'dsb位点处,从而导致5'dsb位点和3'dsb位点之间的染色体dna的缺失或倒位,这导致γ-球蛋白的表达增加,从而增加细胞中胎血红蛋白(hbf)的水平。在一些实施方案中,细胞是骨髓细胞、造血祖细胞或cd34+细胞。在上述方法的一些实施方案中,两个rna向导产生一对双链断裂(dsb),第一dsb在人染色体11的δβ-球蛋白区域中的5'dsb位点处,而第二dsb在人染色体11的δβ-球蛋白区域中的3'dsb位点处,从而导致5'dsb位点与3'dsb位点之间的染色体dna的缺失,其中第一rna向导包含与5'dsb位点的区段互补的间隔序列,而第二rna向导包含与3'dsb位点的区段互补的间隔序列。在所述方法的一些实施方案中,全部或部分的δβ-球蛋白基因缺失。在所述方法的一些实施方案中,5'dsb位点靠近于hpfh缺失的5'边界。在所述方法的一些实施方案中,5'dsb位点靠近于选自由以下项组成的组的hpfh缺失的5'边界:hpfh-4缺失、hpfh-5缺失、hpfh-kenya缺失、hpfh-black缺失、长corfu缺失和短corfu缺失。在所述方法的一些实施方案中,3'dsb位点靠近于hpfh缺失的3'边界。在所述方法的一些实施方案中,3'dsb位点靠近于选自由以下项组成的组的hpfh缺失的3'边界:hpfh-4缺失、hpfh-5缺失、hpfh-kenya缺失、hpfh-black缺失、长corfu缺失和短corfu缺失。在所述方法的一些实施方案中,5'dsb位点靠近于hpfh缺失的5'边界,而3'位点靠近于hpfh缺失的3'边界。在所述方法的一些实施方案中,5'dsb位点靠近于选自由以下项组成的组的hpfh缺失的5'边界,而3'位点靠近于选自由以下项组成的组的hpfh缺失的3'边界:hpfh-4缺失、hpfh-5缺失、hpfh-kenya缺失、hpfh-black缺失、长corfu缺失和短corfu缺失。在所述方法的一些实施方案中,缺失的3'边界靠近于chr11:5224779,而缺失的5'边界靠近于chr11:5237723。在所述方法的一些实施方案中,两个rna向导选自由seqidno:1至seqidno:103组成的组。在所述方法的一些实施方案中,两个rna向导是seqidno:4和seqidno:15。在所述方法的一些实施方案中,缺失的3'边界靠近于chr11:5234665,而缺失的5'边界靠近于chr11:5238138。在所述方法的一些实施方案中,两个rna向导选自由seqidno:104至seqidno:110组成的组。在所述方法的一些实施方案中,两个rna向导是seqidno:105和seqidno:109。在所述方法的一些实施方案中,缺失的3'边界靠近于chr11:5234655,而缺失的5'边界靠近于chr11:5238138。在所述方法的一些实施方案中,两个rna向导选自由seqidno:104至seqidno:110组成的组。在所述方法的一些实施方案中,两个rna向导是seqidno:105和seqidno:109。在所述方法的一些实施方案中,缺失的3'边界靠近于chr11:5233055,而缺失的5'边界靠近于chr11:5240389。在所述方法的一些实施方案中,两个rna向导选自由seqidno:111至seqidno:120组成的组。在所述方法的一些实施方案中,两个rna向导序列是seqidno:111和seqidno:118。在所述方法的一些实施方案中,缺失的3'边界靠近于chr11:5226631,而缺失的5'边界靠近于chr11:5249422。在所述方法的一些实施方案中,两个rna向导选自由seqidno:121至seqidno:137组成的组。在所述方法的一些实施方案中,两个rna向导是seqidno:122和seqidno:137。在所述方法的一些实施方案中,缺失的3'边界靠近于chr11:5196709,而缺失的5'边界靠近于chr11:5239223。在所述方法的一些实施方案中,缺失的3'边界靠近于chr11:5225700,而缺失的5'边界靠近于chr11:5236750。在所述方法的一些实施方案中,缺失的3'边界靠近于chr11:5255885,而缺失的5'边界靠近于chr11:5259368。在上述方法的一些实施方案中,一个rna向导产生一对双链断裂(dsb),第一dsb在人染色体11的δβ-球蛋白区域中的5'dsb位点处,而第二dsb在人染色体11的δβ-球蛋白区域中的3'dsb位点处,从而导致5'dsb位点与3'dsb位点之间的染色体dna的缺失,其中该rna向导包含与5'dsb位点的区段互补或与3'dsb位点的区段互补的间隔序列。在所述方法的一些实施方案中,全部或部分的δβ-球蛋白基因缺失。在所述方法的一些实施方案中,5'dsb位点靠近于hpfh缺失的5'边界。在所述方法的一些实施方案中,5'dsb位点靠近于选自由小缺失组成的组的hpfh缺失的5'边界。在所述方法的一些实施方案中,3'dsb位点靠近于hpfh缺失的3'边界。在所述方法的一些实施方案中,3'dsb位点靠近于选自由小缺失组成的组的hpfh缺失的3'边界。在所述方法的一些实施方案中,5'dsb位点靠近于hpfh缺失的5'边界,而3'位点靠近于hpfh缺失的3'边界。在所述方法的一些实施方案中,5'dsb位点靠近于选自由小缺失组成的组的hpfh缺失的5'边界,而3'位点靠近于选自由小缺失组成的组的hpfh缺失的3'边界。在所述方法的一些实施方案中,缺失的3'边界靠近于chr11:5249959,而缺失的5'边界靠近于chr11:5249971。在所述方法的一些实施方案中,rna向导选自由seqidno:138至seqidno:142组成的组。在所述方法的一些实施方案中,rna向导是seqidno:138。在所述方法的一些实施方案中,rna向导是seqidno:139。在所述方法的一些实施方案中,一个或更多个cas9dna核酸内切酶是天然存在的分子的同源物、重组物、其密码子优化或修饰形式。在所述方法的一些实施方案中,所述方法包括将编码一个或更多个cas9dna核酸内切酶的一个或更多个多核苷酸引入细胞中。在所述方法的一些实施方案中,所述方法包括将编码一个或更多个cas9dna核酸内切酶的一个或更多个核糖核酸(rna)引入细胞中。在所述方法的一些实施方案中,一个或两个rna向导是crisprrna和tracrrna(grna)、单分子向导rna(sgrna)、或两者的组合。在所述方法的一些实施方案中,一个或更多个cas9dna核酸内切酶是多肽。在所述方法的一些实施方案中,将一个或更多个cas9dna核酸内切酶与一个或更多个grna或一个或更多个sgrna预复合。在所述方法的一些实施方案中,将cas9和一个或两个rna向导电穿孔到细胞内。在所述方法的一些实施方案中,细胞来自患有β-血红蛋白病的患者,β-血红蛋白病为镰状细胞病或β-地中海贫血。在所述方法的一些实施方案中,β-血红蛋白病是镰状细胞性贫血,并且其中细胞中镰状细胞血红蛋白(hbs)的水平降低。在所述方法的一些实施方案中,β-血红蛋白病是β-地中海贫血,并且其中细胞中不配对的α血红蛋白链的水平降低。在所述方法的一些实施方案中,β-血红蛋白病选自由镰状细胞病、镰状细胞性状、血红蛋白c疾病、血红蛋白c性状、血红蛋白s/c疾病、血红蛋白d疾病、血红蛋白e疾病、地中海贫血、具有增加的氧亲和力的血红蛋白相关病症、具有降低的氧亲和力的血红蛋白相关病症、不稳定血红蛋白病和高铁血红蛋白症组成的组。本文提供了用于在来自患有血红蛋白病的患者的细胞中的人染色体11的δβ-球蛋白区域内编辑的一种或更多种rna向导,该一种或更多种rna向导包含选自由表1中的核酸序列组成的组的间隔序列。在一些实施方案中,一个或两个rna向导是crisprrna和tracrrna(grna)、单分子向导rna(sgrna)、或两者的组合。本文中描述和例示了多种其他方面。附图说明图1a至图1d示出了hpfh5缺失的crispr靶位点的基因组位置。图1a示出了与野生型β-球蛋白位点(上部图)相比,hpfh5缺失变体(下部图)的限制性酶切图谱,如由camaschella等人,,haematologica,75(补充卷5):26-30(1990)所定义的。图1b示出了具有用空心框突出显示的示例性hpfh5样5'和3'crispr靶位点的人β-球蛋白位点的示意图。12.9kb的缺失起始于δ基因的5'端的3kb处,并结束于β基因末端的3'端的1.7kb处(从β聚a信号下游690bp处),如由camaschella等人,出处同上所描述的。图1c示出用于在人β-球蛋白位点中产生hpfh5样缺失的说明性crispr向导rna靶位点的序列和基因组位置。图1d示出靶位点序列上的示例性向导rna靶位点的比对。上部图示出了5’crispr靶位点的示例,而下部图示出了3’crispr靶位点的示例。图2a至图2c示出靶向hpfh5的示例性单独向导rna(grna)的活性。通过使用t7核酸内切酶i(t7ei)测定来测定grna的活性。所有实验一式三份进行。图2a示出靶向hek293t和k-562细胞系两者中的hpfh5缺失的5’边界的grna的活性。图2b示出靶向hek293t和k-562细胞系两者中的hpfh5缺失的3’边界的grna的活性。图2c示出在k562细胞中由hpfh5-4向导rna靶向的单独位点处的crispr介导切割和nhej修复产生的示例性dna序列修饰。图3a至图3b示出了检测一起靶向所指示的基因组区域的5'和3'边界的向导rna对的基因组编辑的结局的结果。图3a示出用于检测13kb片段的倒位和缺失的pcr引物位置的示意图。图3b示出切割位点之间的基因组片段的倒位(上部图)或相应切割位点之间的基因组片段缺失(中间图)。矩阵示出在每个测试样本中使用的5’和3’向导rna配对(下部图)。图4示出获得的序列数据,示出使用向导rna的hpfh5-4和hpfh5-15对进行的缺失。pcr缺失产物是克隆的,并对10个克隆进行测序。新的连接在对应于序列的下划线部分中的第一a和t之间的位置的位置处产生。粗体字母表示插入的核苷酸碱基。点表示缺失的核苷酸碱基。图5示出使用成对grna产生的hpfh5缺失等位基因的定量。将靶向hpfh5缺失的5'和3'边界两者的grna的组合共转染到k562和hek293细胞中。使用dropletdigitalpcr测量两个切口之间所产生的缺失的频率。在每种情况下,5’grna是hfpf5-4,而3’grna配偶体变化。图6a至图6b示出了对前导向导rna的在靶和脱靶位点切割活性的比较。图6a示出了与向导rnahpfh5-4和hpfh5-15的在靶(on)位点相比,由生物信息学所预测的最高评分脱靶(ot)位点的序列比较。序列被示出为5’端至3’端,其中最3’端的三联体表示pam序列。粗体字母表示相对于在靶序列的偏差。图6b示出了如通过深度测序确定的在靶(hpfh5-4on;hpfh5-15on)和预测的脱靶位点处的基因组编辑频率。图7a至图7b示出了在hpfh-513kb缺失位点的整个长度中向导rna靶向位点的基因编辑效率。图7a示出了向导rna的靶位点和基因组位置。图7b示出了向导rna的基因组编辑效率。靶位点被分组为距hpfh-5缺失边界的5’边界的距离的一个kb增量。图8示出了hpfhcorfu3.5kb(上部图)和7.2kb(下部图)缺失的基因组位置的示意图。图9示出了基于人类基因组数据库版本hg38,用于hpfhcorfu缺失的向导rna的序列和基因组靶标。图10a至图10c示出了靶向hek293细胞中的hpfhcorfu位点的grna的crispr介导的基因组修饰效率。图10a示出了corfu3.5kb缺失(cs=corfu3.5kb缺失)。图10b示出了corfu7.5kb缺失(cl=corfu7.5kb缺失)。图10c示出了向导rna在缺失的5'和3'边界处的相对分布(cs=corfu3.5kb缺失,cl=corfu7.5kb缺失)。图11a至图11c示出了靶向k562细胞中的hpfhcorfu位点的grna的crispr介导的基因组修饰效率。图11a示出了corfu3.5kb缺失(cs=corfu3.5kb缺失)。图11b示出了corfu7.5kb缺失(cl=corfu7.5kb缺失)。图11c示出了向导rna在缺失的5'和3'边界处的相对分布(cs=corfu3.5kb缺失,cl=corfu7.5kb缺失)。图12a至图12b示出了针对一起靶向指定基因组区域的5'和3'边界的向导rna对检测基因组编辑事件的结果。图12a示出了pcr产物检测到corfu7.5kb和3.5kb区域的缺失(左图)和倒位(右图)。图12b示出了显示在所描绘的泳道中使用的5’和3’向导rna配对的矩阵。靶序列的位置示出在图11c中。图13a至图13c示出了hek293细胞中hpfhkenya缺失向导rna的位置和活性。图13a示出了β-球蛋白位点的示意图,其示出了向导rna的位置(左框,向导1-8)和3'的位置(右框,向导9-17)。图13b示出了靶向hpfhkenya缺失的每个边界的向导rna的序列和基因组位置。图13c示出了如通过t7e1测定确定的向导rna的基因组修饰活性。注意:在一些凝胶泳道中,存在高水平的背景条带,其贡献于所测量的插入缺失频率。穿过数据的白线表示与此背景相关联的信号的估计水平。图14a至图14d示出了用于hpfh-sd13bp缺失的向导rna的位置。图14a示出人γ-球蛋白位点的野生型和13bp缺失变体的序列比对。crispr的潜在pam位点被圈出。图14b示出了向导rna的位置(箭头)。还示出了13bp缺失序列的位置以及预测为介导导致13bp缺失的微同源驱动的nhej事件的两个重复序列的位置。图14c示出了设计用于产生hpfh-sd缺失的向导rna的序列和基因组位置。图14d示出hbg1和hbg2基因的序列比对,示出了保守靶区域(虚线框)以及由两个基因中的靶位点处的切割引起的潜在约5kb的缺失(下部图)。图15a至图15c示出了对hek293细胞中的hpfh-sd靶位点处的dna修复事件的分析。图15a示出了用不同向导rna切割后对检测到的dna修复事件的序列分析。针对每个向导rna定量缺失(-vex轴)和插入(+vex轴)事件的频率。图15b示出了对向导sd2的修复结局的分布的总结,表示期望的13bp缺失以9.3%的频率发生。图15c示出除了13bp缺失之外检测到的nhej介导的dna修复事件的序列。下划线示出重复序列。还示出了13bp缺失的位置。图16a至图16c示出了与hpfh相关的β-球蛋白位点的其他缺失和非缺失修饰。图16a示出了示出hpfh-4缺失位置的示意图。图16b示出了示出hpfhblack缺失位置的示意图。图16c示出了gγ-175(t至c)突变区域中的基因组序列。化脓性链球菌cas9的潜在pam位点被圈出。核苷酸t175以粗体显示。图17a至图17c示出了针对基础水平的γ-球蛋白转录物的多供体筛选。图17a示出了针对多种供体的α-球蛋白归一化的γ/α-球蛋白转录物的图。图17b示出了针对多种供体的18srrna归一化的α-球蛋白转录物的图。图17c示出了针对多种供体的18srrna归一化的γ-球蛋白转录物的图。cb是对照。图18a至图18c示出了对球蛋白转录水平的向导的验证。图18a示出了针对多种供体的18srrna归一化的γ-球蛋白转录水平的图。图18b示出了针对多种供体的18srrna归一化的α-球蛋白转录水平的图。图18c示出了针对多种供体的18srrna归一化的β-球蛋白转录水平的图。corfu-大是cf-l;corfu-小是cf-s;kenya是kenya;小缺失是sd;未编辑的是未经编辑的对照。图19是针对多种供体的α-球蛋白归一化的γ/α-球蛋白转录水平的图。corfu-大是cf-l;corfu-小是cf-s;kenya是kenya;小缺失是sd;未编辑的是未经编辑的对照。图20示出了用于γ-球蛋白基因的去抑制的推荐策略。图21a至图21c示出,hpfh-5重新激活cd34+细胞中的γ-球蛋白位点。图21a示出了针对多种供体的18srrna归一化的α-球蛋白转录物的图。图21b示出了针对多种供体的18srrna归一化的γ-球蛋白转录物的图。图21c示出了针对多种供体的α-球蛋白归一化的γ/α-球蛋白转录物的图。corfu-大是cf-l;hpfh5是hpfh5;pb是未经编辑的外周血对照。图22a至图22b示出了向导rna的设计工作流。图22a示出了从对潜在脱靶位点的生物信息学筛选、在293t细胞中筛选、在k-562细胞中的验证,到对向导rna进行排名的向导rna设计工作流的图示。图22b示出了来自表1的具有优异插入缺失频率的向导。图23a至图23d示出了hpfh缺失的最佳向导rna。图23a示出了具有用于hpfh5缺失的最佳缺失频率的向导rna的图。图23b示出了具有用于corfu小缺失和corfu大缺失的最佳缺失频率的向导rna的图。图23c示出了具有用于kenya缺失的最佳缺失频率的向导rna的图。图23d示出了具有用于小缺失的最佳插入缺失频率的向导rna的图。图24示出了嵌合实验(tilingexperiment)中表1中的多种hpfh5向导的缺失频率的图。图25a至图25c示出了影响缺失频率的因素。图25a是示出缺失频率作为缺失大小的函数的曲线图。图25b是示出缺失频率作为平均对nhej水平的函数的曲线图。图25c是示出缺失频率作为pam取向的结果的曲线图。图25d是图25c中的pam取向的图示。图26是示出向导rna对缺失频率的影响的图。图27a至图27d示出了向导rna对缺失频率的剂量曲线。图27a示出hpfh5向导rna(hpfh5-5和hpfh5-15)对缺失频率的剂量曲线。图27b示出corfu大向导rna(cl01和cl08)对缺失频率的剂量曲线。图27c示出corfu小向导rna(cs02和cl08)对缺失频率的剂量曲线。图27d示出kenya向导rna(k2和k17)对缺失频率的剂量曲线。图28是示出如通过深度测序确定的在靶(hpfh5-4on;hpfh5-15on)和预测的脱靶位点处的基因组编辑频率的图。多种在靶和脱靶序列与图6a中相同。图29是示出在k562细胞中进行的对小缺失向导rnahpfhsd_02的测试的图。具体地,示出了向导rnahpfhsc_02的插入缺失频率。图30a至图30b显示了在k562细胞中的测试结果。图30a是示出corfu大缺失向导rnacl01和cl08的单独经修饰等位基因频率的图。图30b是示出corfu大缺失向导rnacl01和cl08组合的缺失频率的图。图31a至图31c示出了k562细胞中dna、rna和蛋白质之间在插入缺失频率和缺失频率方面的差异。图31a是示出每个cl01和cl08向导rna针对dna、rna和蛋白质每个的插入缺失频率的图。图31b是示出corful大7.2kb缺失针对dna、rna和蛋白质每个的缺失频率的图。图31c是示出corfu大7.2kb缺失针对dna、rna和蛋白质每个的大缺失与nhej的比率的图。图32是示出在cd34+细胞中测试的图。具体地,示出了如通过tide分析测量的向导rnacl01、cl08、k2和sd2的插入缺失频率。图33是示出在cd34+细胞中测试的图。具体地,示出了corfu缺失和kenya缺失在24小时的hpfh缺失频率。图34a至图34c示出了hbf表达细胞。图34a是示出hsc扩增和终末分化成红细胞的图。图34b是示出在使用hpfhcl01和hpfhcl08、hpfhk2和hpfhk17、hpfhsd02和对照进行基因编辑后,为bypa+和cd71+的细胞的百分比的图。图34c是示出在使用hpfhcl01和hpfhcl08、hpfhk2和hpfhk17、hpfhsd02和对照进行基因编辑后,为hbf+的细胞的百分比的图。发明详述血红蛋白病胎血红蛋白(hbf)是两个成体α-球蛋白多肽和两个胎儿β样γ-球蛋白多肽的四聚体。γ-球蛋白基因(hbg1和hbg2)通常在胎儿肝、脾和骨髓中表达。两个γ链与两个α链的四聚体一起构成hbf。在妊娠期间,复制的γ-球蛋白基因构成了从β-球蛋白位点转录的主要基因。出生后,γ-球蛋白变成逐渐被成体β-球蛋白替代,这一过程被称为“胎儿转换”。这种从主要产生hbf(α2γ2)到产生成体血红蛋白或hba(α2β2)的发育转换开始于妊娠约28至34周时,并且在出生之后不久继续,此时hba变为主要的。此转换主要是因γ-球蛋白基因的转录减少和β-球蛋白基因的转录增加而产生的。平均来说,正常成年人的血液只含有总血红蛋白的约2%为hbf形式,尽管健康成年人体内的残留hbf水平差异超过20倍(atweh,semin.hematol.38(4):367-73(2001))。两种类型的γ链在残基136处不同,其中在g-γ-产物(hbg2)中存在甘氨酸,而在a-γ-产物(hbg1)中存在丙氨酸。hbg1血红蛋白基因(aγ或a-γ[智人(homosapiens)(人)]基因id:3047)在2014年4月16号更新(wwwdotncbidotnlmdotnihdotgov/gene/3047)。如本文所用,术语“血红蛋白病”是指个体的任何血红蛋白的结构、功能或表达方面的任何缺陷,并且包括由任何突变引起的血红蛋白的初级、二级、三级或四级结构的缺陷,所述突变诸如β-球蛋白基因的编码区中的缺失突变或替代突变,或导致产生的血红蛋白的量相较于正常或标准条件减少的基因的启动子或增强子的突变或缺失。该术语还包括由外部因素如疾病、化学治疗、毒素、毒物等引起的血红蛋白(无论是正常还是异常的)的量或有效性的任何降低。本文考虑的β-血红蛋白病包括但不限于镰状细胞病(scd,也称为镰状细胞性贫血或sca)、镰状细胞性状、血红蛋白c疾病、血红蛋白c性状、血红蛋白s/c疾病、血红蛋白d疾病、血红蛋白e疾病、地中海贫血、具有增加的氧亲和力的血红蛋白、具有降低的氧亲和力的血红蛋白、不稳定血红蛋白病和高铁血红蛋白血症。通过增加胎血红蛋白(α2γ2;hbf)的水平来解决β-血红蛋白病的可行性由观察到具有共遗传的纯合β-地中海贫血和遗传性胎儿血红蛋白持续症(hpfh)的个体的轻度表型所支持,以及由不合成成体血红蛋白但在体内存在增加浓度的hbf的情况下观察到减少的输血需求的那些纯合β-地中海贫血患者所支持。另外的支持来自观察到,具有β链异常的成体患者的某些群体具有高于正常水平的hbf,并且已经观察到具有比具有正常成体水平的hbf的患者更轻度的疾病临床病程。例如,一组表达20-30%hbf(为占总血红蛋白的百分比)的沙特阿拉伯镰状细胞贫血患者仅有轻度的疾病临床表现[pembrey等人,br.j.haematol.40:415-429(1978)]。现在认为,β-血红蛋白病,诸如镰状细胞性贫血和β-地中海贫血,通过增加hbf产生来改善的。[在jane和cunningham,br.j.haematol.102:415-422(1998)和bunn,n.engl.j.med.328:129-131(1993)中评论]。人β-球蛋白位点由位于染色体11的短区域上的五个β样基因和一个假β基因构成(约45kb),负责产生血红蛋白的β链。所有这些基因的表达由单位点控制区域(lcr)控制,并且基因在整个发育中差异地表达。如图1b所示,β-球蛋白簇中的lcr和基因的顺序如下:5'–[lcr]–ε(ε,hbe1)–gγ(g-γ,hbg1)–aγ(a-γ,hbg2)–[ψβ(ψ-β假基因)]–δ(δ,hbd)–β(β,hbb)-3'。五个β样基因的布置反映了它们的表达在发育期间的时间分化,其中早期胚胎阶段版本hbe(由ε基因编码)定位为最接近lcr;之后是胎儿版本hbf(由γ基因编码);δ版本,其在出生前不久开始并在成体中以低水平表达为hba-2(构成正常成体中成体血红蛋白的约3%);以及最后是β基因,其编码主要的成体版hba-1(构成正常成年人中的hba的剩余97%)。β样基因的表达在胚胎红细胞生成中由许多转录因子调节,包括与成体发育完全红细胞中的hba上调相关的klf1和与胎血红蛋白的表达相关的klf2。bcl11a被klf1激活,并且同样已知参与从胎儿到成体血红蛋白的转换。bcl11a表达的下调或其活性的破坏或与转录调节位点的结合已经是来自多个小组的用以增加hbf的水平的长期努力的焦点。参见例如us8383604、us2014085593、us20140093913、以及其中所引用的参考文献。人β-球蛋白位点内的某些天然存在的遗传突变与γ-球蛋白基因表达的去抑制和hpfh的临床表现相关。这样的突变的范围从与多种形式的非缺失hfpf相关的单碱基取代到在某些形式的缺失hpfh的情况下跨几十kb的缺失。在由奥古斯塔市,ga,usa的镰状细胞性贫血基金会(thesicklecellanemiafoundation)出版的由titushjhuisman、mariannefhcarver和erolbaysal所著的asyllabusofthalassemiamutations(1997)及其中所引用的参考文献中描述了多种天然存在的hpfh,包括缺失和非缺失类型二者。已经基于来自被发现在本文中称为“δβ-球蛋白区域”的区域中具有缺失的个体和家族的研究报道了许多不同形式的缺失hpfh,所述区域从ψ-β假基因延伸通过δ、β,以及较大hpfh等位基因(诸如hpfh-1)缺失的β下游区域,如在本领域中所述。在hpfh的一些情况下,几乎所有产生的血红蛋白是hbf。然而,在大多数情况下,hbf的范围为总血红蛋白的约15-30%,这取决于hpfh的类型以及个体之间的变化。缺失破坏或消除β-球蛋白基因和这种缺失在治疗scd中的优势在如本文所描述和说明的一些实施方案中,除了增加γ-球蛋白基因产物hbf的表达之外,β-球蛋白基因产物的表达通过与基因组编辑程序相关的β-球蛋白基因的破坏或消除而被显著降低或消除。这在基因组编辑使用dna核酸内切酶实现一对dsb时发生,第一dsb在人染色体11的δβ-球蛋白区域内的5'dsb位点处,而第二dsb在其3'dsb位点处,从而导致在5'dsb位点和3'dsb位点之间的染色体dna的缺失,这导致γ-球蛋白的表达增加,该缺失还移除全部或部分β-球蛋白基因(hbb),从而导致伴随的β-球蛋白基因产物表达减少或β-球蛋白基因产物的消除,继而导致以下的组合:(i)增加细胞中的hbf水平,以及(ii)减少或消除来自染色体11上的至少一个hbb等位基因的β-球蛋白基因产物的表达。增加的hbf和减少或消除的β-球蛋白基因表达的组合效应在改善血红蛋白病如scd的情况下具有特别的附加优点,在所述血红蛋白病中变体β-球蛋白等位基因的产物(即hbs)对表达其的细胞是有害的,导致细胞过早死亡(以及与hbs相关的其他负面影响)。因此,不仅镰状rbc对患者造成如上文和在本领域中所论述的多重问题,而且镰状rbc还相对于正常rbc具有显著降低的寿命。hbs和镰状rbc的存在还导致许多其他负面影响,如在本文和本领域中所述。在其中β-球蛋白基因如本文所述被有效破坏或消除的实施方案的情况下,甚至“敲低”(减少)或“敲除”(消除)表达hbs的β-球蛋白等位基因中的仅一个,例如通过成功编辑纯合scd患者(其具有两个缺陷β-球蛋白等位基因,染色体11的每个拷贝上各具有一个)中的两个基因拷贝中的仅一个,也能够具有非常显著的益处。特别地,将hbf水平增加至约20%的范围被认为基本上消除镰状化。然而,作为在显著范围内的相对连续或增量因子(通常称为“定量性状”),甚至更低水平的hbf也可以具有如本文和在本领域中所述的显著有益效果。因此,在这些实施方案中,虽然scd患者具有两个缺陷型β-球蛋白等位基因,但是通过使用针对这些实施方案所描述和说明的方法进行基因组编辑增加hbf(其本身有助于减少scd的功效)以及减少hbs(其本身以定量方式导致许多有害功效)的组合能够带来一起减轻疾病的一或多种症状的组合效应。在一些情况下,基因组编辑程序可以有效地改变等位基因的两个拷贝。这样的双等位基因编辑可以在一些情况下被筛选或选择,但是即使不选择其也可以自然地发生,尽管与单等位或单个等位基因命中相比频率较低,这是因为相同的靶位点通常存在于成对染色体的每个成员上。然而,由于如上所述的技术原因,如本文所述和说明的其中只有一个β-球蛋白等位基因被破坏或消除的实施方案——除了增加hbf的水平外——预期会在改善与scd相关的一种或更多种症状或病症方面具有显著的积极效应。使用在此类实施方案中反映的基因组编辑类型产生这些显著的“顺式型”(在相同等位基因上)效应的能力可比取决于“反式型”效应的方法(诸如涉及敲除或敲低或反式作用因子如阻遏物的那些)更有利。特别地,如上所述,其中β-球蛋白基因被有效破坏或消除的实施方案中的基因组编辑可以通过成功地编辑两个等位基因中的一个来显著减轻hbs的效应。在反式作用阻遏物如γ-球蛋白基因表达的阻遏物的情况下,敲低或敲除阻遏物基因的一个拷贝可能不足够,因为来自该基因的另一拷贝的阻遏物的表达仍然能够减少γ-球蛋白基因表达,从而限制了可实现的hbf水平。在β-地中海贫血的情况下增加hbf的影响如上所述,β-地中海贫血是由于β球蛋白基因表达的部分或完全缺陷,从而导致血红蛋白a(hba)缺乏或不存在。由于没有hbs的产生,β-地中海贫血患者中的rbc不表现出与scd相关的镰状化以及相关问题。然而,由于在β-地中海贫血的情况下缺乏hba,所以发生不同类型的rbc“毒性”和过早的细胞死亡。特别地,β地中海贫血中的过量未配对α球蛋白(α-球蛋白)链与红细胞(rbc)膜相互作用,从而导致对膜骨架组分和潜在的其他组分的氧化损伤。这种相互作用导致刚性的、机械不稳定的膜,其引起增加的凋亡(即,程序性细胞死亡)和缩短的rbc存活,特点是低效的红细胞生成和贫血。增加此类患者的rbc中的hbf水平能够显著改善β-地中海贫血的一种或更多种症状,因为通过增加γ-球蛋白基因表达产生的β链可以与先前未配对的α链配对以产生hbf,这不仅产生功能性血红蛋白四聚体,而且还伴随地降低非配对的α-球蛋白链的水平,非配对的α-球蛋白链是由于过早的rbc细胞死亡造成的β-地中海贫血症状的成因。某些经编辑细胞的正向选择性优势关于在本发明的一些实施方案中提供的前述优点,特别是关于可以由不仅增加hbf的水平而且还降低hbs的水平的基因组编辑介导的镰状细胞rbc的rbc存活方面的优点,以及关于不仅增加hbf水平而且还降低未配对的α链的水平的β-地中海贫血rbc的rbc存活的优点,由本文所描述和所说明的这种基因组编辑技术修饰的细胞将具有相对于患病rbc群体的选择性优势,可以例如通过以下方式将其引入:离体基因编辑患者自身的hsc或红细胞祖细胞,然后将这种细胞重新引入患者体内,在患者体内所述重新引入的细胞一般必须成功地保持或“移植”,以便实现足够和持久的有益效果。作为前述选择性优点的结果,引入如本文所述编辑的甚至适度数量的适当干细胞预期将随时间推移产生改善的细胞,所述改善的细胞相较于它们最初引入患者后时占总rbc群体的显著更高分数。例如,通过使用最初占相应细胞的少至几百分比的经成功基因编辑的干细胞(即,相较于携带未编辑的血红蛋白病相关等位基因的驻留细胞群体相比),经基因编辑的细胞能够变成占细胞的大部分,这是由于通过使用如本文进一步所述的基因编辑技术而赋予它们的选择性存活优势。反映这种正向选择植入的最终数目将通常取决于原驻患病细胞在给定患者中表现出缩短寿命的程度,以及由经基因编辑的细胞表现出的相对存活优势而变化。然而,如上所述,与scd和β-地中海贫血相关的患病细胞具有显著缩短的寿命(由于分别存在hbs和未配对的α链),并且一些实施方案不仅增加hbf的水平而且还降低hbs的水平(与scd相关)或降低未配对的α链的水平(与β-地中海贫血相关),因此相对存活益处以及伴随它们的增加的移植预计将是显著的。corfu和corfu样缺失虽然在希腊儿童中首次发现的corfu染色体等位基因导致δβ-地中海贫血,但是其与hpfh的多种缺失形式共享一些重要特征,特别是hbf水平升高,因此与corfu相关的缺失包括在如本文所述的缺失hpfh形式中。然而,corfu在hbf水平和β-球蛋白表达方面不同于缺失hpfh形式。极高水平的hbf与corfu相关,在第一个确定的儿童的情况中接近总血红蛋白的100%——这是特别令人惊讶的,因为corfu杂合体(在第一种情况下的儿童的父母)被发现只有正常非常低水平的hbf(总血红蛋白的1-2%)——这种情况被血液学家称为“corfu悖论”。推定的解释是发现corfu染色体等位基因在β-球蛋白基因的ivs-i的位置5(“ivs-i-5”)中含有剪接位点突变并且具有较低水平的β-球蛋白基因转录物。已经报道,观察到的高水平的hbf通过增强的mrna成熟和/或γ-球蛋白转录物的稳定化来促进后转录,其显然与降低水平的β-球蛋白mrna相关;参见例如,chakalova,l.等人,blood105:2154-2160(2005)。由于corfu染色体等位基因含有大缺失和ivs-i-5突变两者,并且据信与后者相关的降低水平的β-球蛋白mrna独立地贡献于产生异常高水平的hbf,所以ivs-i-5“corfu相关的β-球蛋白突变”可以单独使用或与如本文所述的其他基因编辑改变组合使用,以增加用于减轻血红蛋白病的hbf水平。靶序列选择为了通过基因编辑减轻血红蛋白病,如本文所述,期望达到相对于在天然情况下观察到的那些处于高端的hbf水平来实现疾病的相对改善,但这并非必须的。特别地,虽然最初已经假设相对高水平的hbf对于观察到减轻效应是必需的,尤其是对于某些并发症,但是研究已经表明,即使小的hbf增量增加也可以对死亡率具有有益的影响。参见例如powars等人,blood63(4):921-926(1984);platt等人,nengljmed330(23):1639-1644(1994);以及akinsheye等人,blood118:19-27(2011)。在镰状细胞病的背景下甚至低水平的hbf的有益效果的一个原因是,即使小的hbf增量增加也已经显示具有一些有益效果,并且小于9%的hbf水平(相对于总血红蛋白,hb)表现为与显著降低的死亡率相关;参见例如platt等人,出处同上。较高水平的hbf与附加的临床益处以及发病率和死亡率的进一步降低相关,如在scd与某些天然存在的hpfh等位基因和/或corfu地中海贫血等位基因共遗传的情况下观察到的,其中20至30%范围的hbf水平已经非常显著地与scd表型的几乎完全正常化相关联。被考虑用于增加hbf表达以减轻如本文所述的血红蛋白病的δβ-球蛋白区域内的遗传修饰导致至少约5%、至少约9%、至少约14%、至少约20%、至少约25%、或高于30%的hbf(相对于受试者中的总hb)。如本文进一步描述和说明的,考虑用于将hbf表达增加至此类水平的δβ-球蛋白区域内的示例性遗传修饰包括但不限于以下缺失及其变型,在所述变型中缺失的大小被减少(例如,通过将下面指定的缺失的5'边界进一步移向下面指定的缺失的3'边界或将所述缺失的3'边界进一步朝向5'边界移动)或增加(通过在相反方向上移动任一边界)。本公开还具体考虑通过增加hbf表达的以下缺失边界中的两个的其他组合进行的缺失。a.基于人基因组装配的grch38/hg38版本在区域chr11:5224779-5237723内的染色体11中的缺失,其中该缺失的3'边界靠近于(如下定义)chr11:5224779并且该缺失的5'边界靠近于chr11:5237723;b.基于人基因组装配的grch38/hg38版本在区域chr11:5234665-5238138内的染色体11中的缺失,其中该缺失的3'边界靠近于chr11:5234665并且该缺失的5'边界靠近于chr11:5238138;c.基于人基因组装配的grch38/hg38版本在区域chr11:5233055-5240389内的染色体11中的缺失,其中该缺失的3'边界靠近于chr11:5233055并且该缺失的5'边界靠近于chr11:5240389;d.基于人基因组装配的grch38/hg38版本在区域chr11:5226631-5249422内的染色体11中的缺失,其中该缺失的3'边界靠近于chr11:5226631并且该缺失的5'边界靠近于chr11:5249422;e.基于人基因组装配的grch38/hg38版本在区域chr11:5249959-5249971内的染色体11中的缺失,其中该缺失的3'边界在或靠近于chr11:5249959并且该缺失的5'边界在或靠近于chr11:5249971;f.基于人基因组装配的grch38/hg38版本在区域chr11:5196709-5239223内的染色体11中的缺失,其中该缺失的3'边界靠近于chr11:5196709并且该缺失的5'边界靠近于chr11:5239223;g.基于人基因组装配的grch38/hg38版本在区域chr11:5225700-5236750内的染色体11中的缺失,其中该缺失的3'边界靠近于chr11:5225700并且该缺失的5'边界靠近于chr11:5236750;h.基于人基因组装配的grch38/hg38版本在区域chr11:5234655-5238138内的染色体11中的缺失,其中该缺失的3'边界靠近于chr11:5234655并且该缺失的5'边界靠近于chr11:5238138;i.基于人基因组装配的grch37/hg19版本在区域chr11:5255885-5259368内的染色体11中的缺失,其中该缺失的3'边界靠近于chr11:5255885并且该缺失的5'边界靠近于chr11:5259368。在另一方面,本文提供了通过基因组编辑来增加人细胞中胎血红蛋白(hbf)水平的方法,其使用dna核酸内切酶实现位于人染色体11的β-球蛋白区域内的一个或更多个位点处的双链断裂(dsb),其中至少一个dsb位于人染色体11的γ-球蛋白调节区内,该dsb位于一个γ-球蛋白基因(hbg1或hbg2)的起点上游小于2kb、小于1kb、小于0.5kb、或小于0.25kb的区域内,导致染色体dna在所述一个或更多个位点处的缺失或插入,从而导致γ-球蛋白的表达增加,从而增加细胞中的hbf水平。在例示这一方面的另一种类型的方法中,至少一个dsb位于人染色体11的δβ-球蛋白区域内。在γ-球蛋白调节区中的染色体11中的说明性修饰包括产生单碱基取代,诸如在gγ基因中的-175(t至c)、-202(c至g)和-114(c至t);以及在aγ基因中的-196(c至t)、-175(t至c)、-117(g至a)。δβ-球蛋白区域内的说明性修饰包括在上面提到的hpfh缺失位点内或附近的缺失和插入;以及人染色体11的δ-球蛋白调节区域内的缺失,其位于δ-球蛋白基因(hbd)的起点上游的小于3kb、小于2kb、小于1kb、小于0.5kb的区域内;以及在人染色体11的β-球蛋白调节区域内的缺失,其位于β-球蛋白基因(hbb)的起点上游的小于3kb、小于2kb、以及小于1kb、或小于0.5kb的区域内。鉴于与多种形式的hpfh相关的缺失相对较大的变化,加上甚至低水平的hbf也可以提供显著水平的血红蛋白病减轻(如上所述)的事实,以及根据多种研究了解到表现为存在可以有助于抑制hbf的多个位点和多种类型的控制,应当理解上面提及的缺失的多种变型(包括但不限于更大以及更小的缺失)预期会导致在所考虑范围内的hbf水平,如上所述。此类变型包括在5'和/或3'方向比天然存在的hpfh缺失更大,或者在任一方向上更小的缺失。因此,对于hpfh样缺失,“靠近”意指与期望的缺失边界(也在本文中称为终点)相关的dsb位点可以在距所述参考位点小于约3kb的区域内。在一些实施方案中,dsb位点更接近并在2kb内、在1kb内、在0.5kb内或在0.1kb内。在诸如组e中鉴定的小缺失的情况下,期望的终点位于或“邻近于”参考位点,这意图表示终点在距参考位点100bp内、50bp内、25bp内、或小于约10bp至5bp。一组实施方案包括“δ-区域”内的缺失(其包括ψβ1假基因和δ基因hbd之间的基因间序列的下游一半,以及δ中的近端序列下游序列)。δ-近端区域表现为包含与γ-球蛋白的抑制相关的许多元件。本文进一步描述和例示的7.2kb“大corfu”δβ地中海贫血缺失落入δ区域内,缺失δ基因的约1kb以及上游的6kb,并且与hbf水平的显著增加相关。本文进一步描述和说明的3.5“小corfu”缺失同样具有δ区域中的缺失,并且也与增加的hbf水平相关。δ区域在hpfh的所有主要形式中也被缺失。关于在3'方向上进一步下游的区域,其将与本文所述的较大缺失相关,hpfh-1至hpfh-5都具有缺失的δ和β基因。除了可有助于有效抑制γ-球蛋白的δ-区域中的调节元件,δ和β启动子的活性也可以通过竞争γ-球蛋白表达所需的转录因子来间接地促进抑制。许多hpfh类型还具有进一步向下游延伸的甚至更大的缺失,并且这些另外的下游区域也可以并入如本文所描述和说明的缺失中,因为已知它们与hbf的实质性增加相关,远高于已知减轻如上所述的血红蛋白病的hbf的范围。对于患有血红蛋白病的患者来说,复制或模拟具有hpfh的个体中天然存在的缺失的方面的一个优点是已经已知这种缺失是安全的并且与血红蛋白病的减轻相关。然而,在缺失hpfh中,还清楚的是较小的缺失如hpfh-5对于产生hbf的实质性增加是有效的。包含较小缺失的其他实施方案被预期提供实质性增加,并且如上所述,即使适度水平的hbf增加也具有有益效果。因此预期,本文所描述和说明的缺失的许多变化将有效减轻血红蛋白病。优选地,5'边界和/或3'边界的位置相对于特定参考位点的的偏移被用来促进或增强基因编辑的特定应用,其部分地取决于被选择用于编辑的核酸内切酶体系,如本文所进一步描述和说明的。在这种靶序列选择的第一方面,许多核酸内切酶体系具有引导对潜在切割靶位点的初始选择的规则或标准,诸如在crisprii型核酸内切酶的情况下需要pam序列基序在与dna切割位点相邻的特定位置。在靶序列选择或优化的另一方面,相对于在靶(on-target)活性的频率来评估靶序列和基因编辑核酸内切酶的特定组合的“脱靶(off-target)”活性的频率(即,在除了所选的靶序列之外的位点处发生的dsb的频率)。在一些情况下,在所期望位点处被正确编辑的细胞可相对于其他细胞具有选择性优势。选择性优势的说明性但非限制性示例包括属性的获得,诸如增强的复制速率、持久性、对某些条件的抗性,在引入患者体内后在体内成功植入或持久的速率增强,以及与维持与或增加此类细胞的数量或活力相关的其他属性。在其他情况下,已经在所期望位点处被正确编辑的细胞可以通过用于鉴定、分选或以其他方式选择已被正确编辑的细胞的一种或更多种筛选方法来阳性选择。选择性优势和定向选择方法二者都可以利用与校正相关的表型。无论任何选择性优势是否适用于特定情况或任何定向选择是否将应用于特定情况,靶序列选择还可以通过考虑脱靶频率来引导,以便增强应用的有效性和/或减少在除期望的靶之外的位点处发生不期望的改变的可能性。如本文和在本领域中进一步描述和说明的,脱靶活性的发生受许多因素的影响,包括靶位点和多种脱靶位点之间的相似性和不相似性,以及所使用的特定核酸内切酶。在许多情况下,生物信息学工具可用于帮助预测脱靶活性,并且经常此类工具还可以用于鉴定具有脱靶活性的最可能的位点,所述位点然后可以在实验设置中评价以评估脱靶相对于在靶活性的相对频率,从而允许选择具有较高相对在靶活性的序列。本文提供了这种技术的说明性示例,其他的是本领域已知的。靶序列选择的另一方面涉及同源重组事件。众所周知,共享同源区域的序列可以充当导致间插序列缺失的同源重组事件的焦点。这种重组事件发生在染色体和其他dna序列的正常复制过程中,以及当dna序列被合成的其他时间,诸如对在正常周期期间定期发生但也可能因为多种事件的发生(例如uv光和dna断裂的其他诱导物)或某些试剂的存在(如多种化学诱导剂)而增强的双链断裂(dsb)进行修复的情况下。许多此类诱导因素导致dsb在基因组中不加选择地发生,并且dsb在正常细胞中经常被诱导和修复。在修复期间,可以以全保真度重建原始序列,然而在一些情况下,会在dsb位点处引入小插入或缺失(称为“插入缺失(indels)”)。还可以在特定位置特异性诱导dsb,如本文所述的核酸内切酶体系的情况下,其可用于在选定的染色体位置引起定向或优先基因修饰事件。在dna修复(以及复制)的情况下,同源序列经受重组的趋势可以在许多情况下被利用,并且是基因编辑系统如crispr的一种应用的基础,其中同源定向修复(hdr)用于将通过使用“供体”多核苷酸提供的目的序列插入期望的染色体位置。特定序列之间的同源性区域可以是可包含少至10个碱基对或更少的“微同源性”的小区域,也可以用于产生所期望的缺失。例如,在本文例示的所谓“小缺失”的情况下,在表现出与附近序列的微同源性的位点处引入单个dsb。在这种dsb的正常修复过程中,高频发生的结果是由于dsb和伴随的细胞修复过程促进的重组而使间插序列缺失。在如图14b所示的γ-球蛋白基因的上游区域中的这种小缺失的情况下,缺失的结果是通过破坏基因沉默序列来明显地增加hbf的水平。然而,在一些情况下,选择同源区域内的靶序列也可以产生包括基因融合(当缺失在编码区中时)在内的更大缺失,其根据具体情况,可以是或可以不是所期望的。例如,如图14d所示,存在于两个密切相关的γ-球蛋白基因hbg1和hbg2之间的同源性可能产生通过多个同源的远端位点之间的同源重组产生的大缺失。本文提供的示例进一步说明了对用于产生设计用于诱导导致人细胞中hbf水平增加的缺失的dsb的多种靶区域的选择,以及对在这样的区域内设计用以相对于在靶事件最小化脱靶事件的特异性靶序列的选择。人细胞为了减轻血红蛋白病,如本文所描述和说明,基因编辑的主要靶标将是人细胞,人细胞在使用所述技术修饰后,可以在患有血红蛋白病(例如β-地中海贫血或镰状细胞病)的患者中产生具有增加的hbf水平的红细胞(rbc)。如本文和在本领域中所述,患有血红蛋白病(例如β-地中海贫血或镰状细胞病)的患者中的hbf水平的即使相对适度和增量增加也可以有益于改善症状和/或存活。在一些实施方案中,所实现的hbf的水平将趋近于在hpfh患者中观察到的那些水平,所述水平在不同患者和hpfh类型中不同,但是在大量病例中导致hbf在总血红蛋白的10-30%的范围内(相对于典型成人中的1-2%)。然而,研究已经表明,较低水平的hbf仍然可以具有被认为是降低各组scd患者的总体死亡率预期的足够显著的效果;参见例如,platt等人,nengljmed.330(23):1639-1644(1994)。并且甚至对症状的适度改善也可以对患者具有有益效果。例如,减少输血需要、减少一种或更多种血红蛋白病症状的发病率或严重性,或者由于治疗或手术的程度或频率降低而导致副作用减少都可以是有意义的并且对患者有益的。因此,在一些实施方案中,hbf的增加可以为在hpfh患者中观测到的hbf水平的约80%、60%、40%或20%的范围内。本文提供了关于可以实现的hbf水平的进一步考虑,包括详细描述和示例,如由本文所引用和/或在领域中公布的参考文献所补充的。通过对祖细胞如红系祖细胞(例如来源于需要治疗的患者并且因此已经与所述患者完全匹配的自体祖细胞)进行如本文所述的基因编辑,可以产生这样的细胞,所述细胞可以被安全地重新引入患者体内并有效地产生循环rbc的群体,该循环rbc的群体将有效地减轻与患者疾病相关的一种或更多种临床症状。虽然存在显著数量的具有升高的hbf水平的rbc是有益的,但是在一些实施方案中超过四分之一的循环红细胞(rbc)将具有显著升高的hbf水平,在一些实施方案中至少一半的循环rbc将具有显著升高的hbf水平,并且在一些实施方案中至少80%的循环rbc将具有显著升高的hbf水平,以有效预防临床红细胞镰状化。祖细胞(在本文中也称为干细胞)例如红细胞或造血祖细胞能够增殖并产生更多的祖细胞,这些祖细胞继而具有产生大量母细胞的能力,所述母细胞能够继而产生分化的或可分化的子细胞。子细胞本身可以被诱导增殖并产生随后分化成一种或更多种成熟细胞类型的子代,同时还保留一个或更多个具有亲本发育潜能的细胞。术语“干细胞”是指在特定情况下具有能力或潜力分化为更专门或分化的表型的细胞,并且所述细胞在某些情况下保留增殖而基本不分化的能力。在一个实施方案中,术语祖细胞或干细胞是指后代(子代)通过分化,例如通过获得完全个体特征(如在胚胎细胞和组织的进行性多样化中发生的)而通常在不同方向上专门化的广义母细胞。细胞分化是通常通过多次细胞分裂发生的复杂过程。分化的细胞可以源自本身源于多能细胞的多能细胞等等。虽然这些多能细胞中的每一种可以被认为是干细胞,但是每种可产生的细胞类型的范围可以有很大差异。一些分化的细胞还具有产生具有更大发育潜力的细胞的能力。这种能力可以是天然的,或者可以在用多种因子处理时人工地诱导。在许多生物学实例中,干细胞也是“多能的”,因为它们可以产生多于一种不同细胞类型的子代,但是这不是“干性”所必需的。自我更新是干细胞的另一个重要方面,如在本文档中所使用的。理论上,自我更新可以通过两种主要机制中的任一种发生。干细胞可以不对称地分裂,其中一种子代保持干性状态,另一种子代表达有一些不同的其他特定功能和表型。或者,群体中的一些干细胞可以对称地分裂成两个干,由此在群体中保持一些干细胞作为整体,而群体中的其他细胞仅产生分化的子代。通常,“祖细胞”具有更原始的细胞表型(即,比完全分化的细胞位于发育路径或进程的更早步骤)。通常,祖细胞也具有显著或非常高的增殖潜力。祖细胞可以产生多种不同的分化细胞类型或单个分化的细胞类型,这取决于发育路径以及细胞发育和分化的环境。在细胞个体发育的情境中,形容词“分化的”或“分化中的”是相对术语。“分化细胞”是相较于与其比较的细胞,已经更进一步沿着发育路径进展的细胞。因此,干细胞可以分化为谱系限制性前体细胞(例如造血祖细胞),该谱系限制性前体细胞能够继而进一步沿着所述路径分化为其他类型的前体细胞(例如红细胞前体),然后分化成末期分化细胞如红细胞,所述末期分化细胞在某些组织类型中具有特有作用,并且可以保留或可以不保留进一步增殖的能力。本文使用的术语“造血祖细胞”是指产生包括红系细胞(红细胞或红血细胞(rbc)、骨髓(单核细胞和巨噬细胞、嗜中性粒细胞、嗜碱性粒细胞、嗜酸性粒细胞、巨核细胞/血小板、以及树突细胞)和淋巴样细胞(t细胞、b细胞、nk细胞)在内的的所有血细胞类型的干细胞谱系的细胞。“红系(erythroid)谱系(lineage)细胞”表示所接触的细胞是经历红细胞生成使得在最终分化时其形成红细胞或红血细胞的细胞。此类细胞来源于骨髓造血祖细胞。在暴露于造血微环境中的特定生长因子和其他组分时,造血祖细胞能够通过一系列中间分化细胞类型(红系谱系的所有中间体)成熟为rbc。因此,本文使用的术语“红系谱系”的细胞包括造血祖细胞、红血胚细胞(rubriblast)、前红细胞(prorubricyte)、成红细胞(erythroblast)、晚幼红细胞(metarubricyte)、网织红细胞(reticulocyte)和红细胞。在一些实施方案中,造血祖细胞具有造血祖细胞特征的以下细胞表面标记特征中的至少一种:cd34+、cd59+、thyl/cd90+、cd381o/-和c-kit/cdl17+。在一些实施方案中,造血祖细胞是cd34+。在一些实施方案中,造血祖细胞是在患者已经用粒细胞集落刺激因子(任选地与普乐沙福组合)处理后从患者获得的外周血干细胞。在说明性实施方案中,使用细胞选择系统(miltenyibiotec公司)富集cd34+细胞。在一些实施方案中,在基因组编辑之前,在无血清培养基(例如,cellgrowscgm培养基,cellgenix)中用细胞因子(例如scf、rhtpo、rhflt3)微弱刺激cd34+细胞。在一些实施方案中,考虑添加sr1和dmpge2和/或其他因子以改善长期植入。在一些实施方案中,红细胞谱系的造血祖细胞具有红细胞谱系的细胞表面标记特征:例如cd71和terl19。经诱导多能干细胞在一些实施方案中,本文所述的遗传工程改造的人细胞来源于经诱导多能干细胞(ipsc)。使用ipsc的优点是细胞可以来源于将要施用祖细胞的相同受试者。也就是说,体细胞可以从受试者获得,重编程为诱导多能干细胞,然后重分化为要施用于受试者的造血祖细胞(例如,自体细胞)。由于祖细胞基本上来源于自体来源,与使用来自另一受试者或另一组受试者的细胞相比,移植排斥或过敏反应的风险降低。在一些实施方案中,造血祖细胞来源于非自体来源。此外,ipsc的使用取消了对从胚胎来源获得的细胞的需要。因此,在一个实施方案中,在所公开的方法中使用的干细胞不是胚胎干细胞。虽然分化在生理环境下通常是不可逆的,但是最近已经开发了若干种方法来将体细胞重编程为ipsc。示例性方法是本领域技术人员已知的并且在下文中简要描述。如本文所用,术语“重编程”是指改变或逆转分化细胞(例如体细胞)的分化状态的过程。换句话说,重编程是指将细胞分化向后驱动为更未分化或更原始类型的细胞的过程。应当注意,将许多原代细胞置于培养物中可导致完全分化的特征的一些损失。因此,简单地培养包括在术语分化细胞中的此类细胞不会使这些细胞变成非分化细胞(例如未分化细胞)或多能细胞。分化细胞向多能性的转变需要重编程刺激,该重编程刺激超过导致培养物中分化特征的部分丧失的刺激。相对于原代细胞亲本,重编程细胞还具有具延长的传代而不丧失生长潜力的能力特征,其通常具有在培养物中仅有限分裂次数的能力。要重编程的细胞可以在重编程之前部分分化或终末分化。在一些实施方案中,重编程涵盖分化细胞(例如体细胞)的分化状态向多能状态或多潜能状态的完全逆转。在一些实施方案中,重编程涵盖分化细胞(例如体细胞)的分化状态向未分化细胞(例如胚胎样细胞)的完全或部分逆转。重编程可导致细胞对特定基因的表达,所述特定基因的表达进一步有助于重编程。在本文所述的一些实施方案中,分化细胞(例如体细胞)的重编程导致分化的细胞呈现未分化状态(例如,是未分化细胞)。所得细胞被称为“重编程细胞”或“诱导多能干细胞(ipsc或ips细胞)”。重编程可以涉及在细胞分化期间发生的核酸修饰(例如甲基化)、染色质浓缩、表观遗传改变、基因组印迹等的至少一些可遗传模式的改变,例如逆转。重编程不同于简单地维持已经是多能的细胞的现有未分化状态或保持已经是多能细胞(例如造血干细胞)的细胞的现有小于完全分化状态。重编程也不同于促进已经是多能或多潜能的细胞的自我更新或增殖,尽管本文所述的组合物和方法在一些实施方案中也可以用于此类目的。用于从体细胞产生多能干细胞的具体途径或方法对于所要求保护的发明不是关键的。因此,将体细胞重编程为多能表型的任何方法将适用于本文所述的方法。已经描述了使用转录因子的限定组合产生多能细胞的重编程方法。通过oct4、sox2、klf4和c-myc的直接转导可以将小鼠体细胞转化为具有扩大的发育潜能的es细胞样细胞;参见例如,takahashi和yamanaka,cell126(4):663–76(2006)。ipsc类似于es细胞,因为它们恢复多能性相关的转录电路和许多表观遗传景观。此外,小鼠ipsc满足多能性的所有标准测定:具体地,体外分化成三个胚层的细胞类型、形成畸胎瘤、有助于嵌合体、种系传递[参见例如maherali和hochedlinger,cellstemcell.3(6):595-605(2008)]、以及四倍体互补。可以使用类似的转导方法获得人ipsc,并且转录因子trio、oct4、sox2和nanog已经被确立为控制多能性的转录因子的核心集合;参见例如,budniatzky和gepstein,stemcellstranslmed.3(4):448-57(2014);barrett等人,stemcellstransmed3:1-6sctm.2014-0121(2014);focosi等人,bloodcancerjournal4:e211(2014);以及其中引用的参考文献。.ipsc的产生可以通过历史上使用病毒载体将编码干细胞相关基因的核酸序列引入成体体细胞来实现。ipsc可以产生自或来源于终末分化的体细胞、以及成体干细胞或体干细胞。也就是说,非多能祖细胞能够通过重编程而变得多能或多潜能。在这种情况下,可能不需要包括如重编程终末分化细胞所需的那样多的重编程因子。此外,重编程可以通过非病毒地引入重编程因子诱导,例如通过引入蛋白质自身、或通过引入编码重编程因子的核酸,或通过引入在翻译后产生重编程因子的信使rna(参见例如,warren等人,cellstemcell,7(5):618-30(2010)。可以通过引入编码干细胞相关基因的核酸的组合来实现重编程,所述干细胞相关基因包括例如oct-4(也称为oct-3/4或pouf51)、soxl、sox2、sox3、sox15、sox18、nanog、klfl、klf2、klf4、klf5、nr5a2、c-myc、1-myc、n-myc、rem2、tert和lin28。在一个实施方案中,使用本文所述的方法和组合物进行重编程可以进一步包括将oct-3/4、sox家族的成员、klf家族的成员、以及myc家族的成员中的一个或更多个引入体细胞。在一个实施方案中,本文所述的方法和组合物进一步包括引入oct4、sox2、nanog、c-myc和klf4各者中的一个或更多个以用于重编程。如上所述,用于重编程的确切方法对于本文所述的方法和组合物不一定是关键的。然而,在从重编程细胞分化的细胞将用于例如人类治疗的情况下,在一个实施方案中,重编程不受改变基因组的方法的影响。因此,在此类实施方案中,例如在不使用病毒或质粒载体的情况下实现了重编程。来源于起始细胞群的重编程的效率(即,重编程细胞的数目)可以通过添加多种小分子来增强,如由shi等人,cell-stemcell2:525-528(2008);huangfu等人,naturebiotechnology26(7):795-797(2008)和marson等人,cell-stemcell3:132-135(2008)所示。因此,增强诱导多能干细胞产生的效率或速率的试剂或试剂组合可用于产生患者特异性或疾病特异性ipsc。增强重编程效率的试剂的一些非限制性示例包括可溶性wnt、wnt条件型培养基、bix-01294(g9a组蛋白甲基转移酶)、pd0325901(mek抑制剂)、dna甲基转移酶抑制剂,组蛋白脱乙酰酶(hdac)抑制剂、丙戊酸、5'-氮杂胞苷、地塞米松、辛二酰苯胺、异羟肟酸(saha)、维生素c和曲古抑菌素(tsa)等。重编程增强剂的其他非限制性示例包括:辛二酰苯胺异羟肟酸(saha(例如mk0683,伏立诺他)和其他异羟肟酸)、bml-210、depudecin(例如(-)-depudecin)、hc毒素、nullscript(4-(l,3-二氧代-lh,3h-苯并[de]异喹啉-2-基)-n-羟基丁酰胺)、苯基丁酸酯(例如苯基丁酸钠)和丙戊酸((vpa)和其他短链脂肪酸)、scriptaid、苏拉明钠、曲古抑菌素(tsa)、apha化合物8、apicidin、丁酸钠、丁酸新戊酰氧基甲基酯(pivanex,an-9)、trapoxinb、chlamydocin、缩酚酸肽(depsipeptide)(也称为fr901228或fk228)、苯甲酰胺(例如,ci-994(例如n-乙酰基地那林(n-acetyldinaline))和ms-27-275)、mgcd0103、nvp-laq-824、cbha(间羧基肉桂酸双羟基酰胺(m-carboxycinnaminicacidbishydroxamicacid))、jnj16241199、突巴新(tubacin)、a-161906、proxamide、oxamflatin、3-cl-ucha(例如,6-(3-氯苯基脲基)己酸异羟肟酸)、aoe(2-氨基-8-氧代-9,10-环氧癸酸)、chap31和chap50。其他重编程增强剂包括例如hdac的显性阴性形式(例如,催化失活形式)、hdac的sirna抑制剂、以及特异性结合hdacs的抗体。此类抑制剂是可获得的,例如来自biomolinternational、fukasawa、merckbiosciences、novartis、gloucesterpharmaceuticals、titanpharmaceuticals、methylgene和sigmaaldrich。为了证实对多能干细胞进行诱导以用于本文所述的方法,可以测试分离的克隆中干细胞标记物的表达。来源于体细胞的细胞中的此类表达将细胞鉴定为诱导多能干细胞。从包括ssea3、ssea4、cd9、nanog、fbxl5、ecatl、esgl、eras、gdf3、fgf4、cripto、daxl、zpf296、slc2a3、rexl、utfl和natl的非限制性组中选择干细胞标记物。在一个实施方案中,表达oct4或nanog的细胞被鉴定为多能性的。用于检测这些标记物的表达的方法可以包括例如检测经编码的多肽的存在的rt-pcr和免疫学方法,例如免疫印迹法(westernblot)或流式细胞术分析。在一些实施方案中,检测不仅仅涉及rt-pcr,而且还包括对蛋白质标记物的检测。可以通过rt-pcr或蛋白质检测方法(例如免疫细胞化学)最好地鉴定细胞内标记,而细胞表面标记物容易地通过例如免疫细胞化学鉴定。分离的细胞的多能干细胞特性可以通过测试来确认,所述测试评估ipsc分化为三个胚层中的每一个的细胞的能力。作为一个示例,裸鼠中的畸胎瘤形成可用于评估分离的克隆的多能性特征。将细胞引入裸鼠体内,并对由细胞产生的肿瘤进行组织学和/或免疫组织化学法。例如,包含来自所有三个胚层的细胞的肿瘤的生长进一步表明细胞是多能干细胞。产生患者特异性ipsc本公开的离体方法的一个步骤可以涉及产生一种或更多种患者特异性ips细胞或患者特异性ips细胞系。在本领域中存在用于产生患者特异性ips细胞的许多确立方法,如在takahashi和yamanaka2006;takahashi、tanabe等人,2007中所述。例如,产生步骤可以包括:a)从患者分离体细胞,例如皮肤细胞或成纤维细胞;以及b)将多能性相关基因的一集合引入体细胞中以诱导细胞变成多能干细胞。多能性相关基因的集合可以是选自由oct4、sox2、klf4、lin28、nanog和cmyc组成的组的基因中的一种或更多种。进行患者骨髓的活组织检查或抽吸活组织检查或抽出物是取自身体的组织或流体的样品。存在许多不同种类的活组织检查或抽出物。几乎所有活组织检查或抽出物都涉及使用锋利的工具来取出少量的组织。如果活组织检查将在皮肤或其他敏感区域上,则可以首先施加麻醉药。可以根据本领域中的任何已知方法进行活组织检查或抽吸。例如,在骨髓抽出物中,使用大针进入盆骨以收集骨髓。分离白血细胞可以根据本领域中已知的任何方法来分离白血细胞。例如,可以通过离心和细胞培养从液体样品中分离白血细胞。分离间充质干细胞间充质干细胞可以根据本领域中已知的任何方法,例如从患者的骨髓或外周血分离。例如,可以将骨髓抽出物收集到含有肝素的注射器中。可以洗涤细胞并在percoll上离心。可以在含有10%胎牛血清(fbs)的达尔伯克改良伊格尔培养基(dmem)(低葡萄糖)中培养细胞(pittingermf、mackayam、becksc等人,science1999;284:143-147)。用gcsf处理患者可以任选地根据本领域中已知的任何方法用粒细胞集落刺激因子(gcsf)处理患者。gcsf可以与普乐沙福联合施用。从患者分离造血祖细胞可以通过本领域中已知的任何方法从患者分离造血祖细胞。可以使用细胞选择系统(miltenyibiotec)来富集cd34+细胞。在基因组编辑之前,可以在无血清培养基(例如,cellgrowscgm培养基,cellgenix)中用细胞因子(例如scf、rhtpo、rhflt3)微弱刺激cd34+细胞。基因组编辑基因组编辑通常是指优选以精确或预定的方式修饰基因组的核苷酸序列的过程。本文所述的基因组编辑方法的示例包括使用定点核酸酶在基因组中的精确靶位置处切割dna,从而在基因组内的特定位置产生双链或单链dna断裂的方法。这种断裂可以并且通常通过天然的、内源性的细胞过程(例如同源定向修复(hdr)和非同源末端连接(nhej))来修复,如在cox等人,naturemedicine21(2),121-31(2015)中所最近评论的。nhej直接连接由双链断裂产生的dna末端,有时具有可能破坏或增强基因表达的核苷酸序列的损失或添加。hdr利用同源序列或供体序列作为模板,以用于在断裂点处插入确定的dna序列。同源序列可以在内源基因组中,例如姐妹染色单体。或者,供体可以是具有与经核酸酶切割的位点有高同源性的区域但也可以含有附加的序列或序列变化(包括可以并入经切割的靶位点的缺失)的外源核酸,例如质粒、单链寡核苷酸、双链体寡核苷酸或病毒。第三种修复机制是微同源性介导的末端连接(mmej),也称为“替代nhej”,其中遗传结局与nhej的相似处在于小缺失和插入可以发生在切割位点处。mmej利用位于dna断裂位点侧翼的几个碱基对的同源序列来驱动更有利的dna末端连接修复结局,并且最近的报道已经进一步阐明了此过程的分子机制;参见例如cho和greenberg,nature518,174-76(2015);kent等人,naturestructuralandmolecularbiology,adv.onlinedoi:10.1038/nsmb.2961(2015);mateos-gomez等人,nature518,254-57(2015);ceccaldi等人,nature528,258-62(2015)。在一些情况下,也许可以基于对dna断裂位点处的潜在微同源性的分析来预测可能的修复结局。这些基因组编辑机制中的每一个可以用于产生期望的基因组改变。基因组编辑过程中的第一步骤是通常在靶位点中产生尽可能接近预期突变位点的一个或两个dna断裂。这可以通过使用靶向核酸内切酶实现,如本文所描述和示出的。已经设计了若干种不同类型的核酸酶用于基因组编辑。这些核酸酶包括锌指核酸酶、转录激活因子样效应子(tale)核酸酶、crispr/cas核酸酶、归巢核酸内切酶(也称为大范围核酸酶)和其他核酸酶;参见例如,hafez和hausner,genome55,553-69(2012);carroll,ann.rev.biochem.83,409-39(2014);gupta和musunuru,j.clin.invest.124,4154-61(2014);以及cox等人,出处同上。这些核酸酶的不同之处主要在于它们结合dna并产生靶向dna双链(或单链)断裂(dsb)的方式。在产生dsb后,nhej或hdr的基本上相同的天然细胞dna修复机制被指派以实现期望的遗传修饰。因此,设想使用这些核酸酶中的任一者的基因组编辑技术可以用于实现本文所述的遗传和治疗结局。锌指核酸酶锌指核酸酶(zfn)是由与ii型核酸内切酶foki的催化结构域连接的工程化锌指dna结合结构域组成的模块化蛋白。由于foki仅用作二聚体,所以必须工程化一对zfn以结合在相对dna链上的同源靶“半位点”序列,并且在它们之间具有精确的间隔以使催化活性的foki二聚体形成。当本身没有序列特异性的foki结构域二聚化时,作为基因组编辑中的起始步骤,在zfn半位点之间产生dna双链断裂。每个zfn的dna结合结构域通常由具有丰富的cys2-his2架构的3-6个锌指组成,其中每个指状结构主要识别靶dna序列的一条链上的核苷酸三联体,尽管与第四个核苷酸的交叉链相互作用也可能是重要的。与dna进行关键接触的位置中的指状结构的氨基酸的改变改变了给定指状结构的序列特异性。因此,四指锌指蛋白将选择性地识别12bp的靶序列,其中靶序列是来源于各个指状结构的三联体偏好的复合物,尽管三联体偏好可能受到相邻指状结构的不同程度影响。zfn的一个重要方面是,它们可以通过修改个别指状结构而容易地重新靶向到几乎任何基因组位址,尽管需要相当多的专业知识来做到这一点。在zfn的大多数应用中,使用具有4-6个指状结构的蛋白质,来分别识别12-18bp。因此,一对zfn通常将识别24-36bp的组合靶序列,不包括半位点之间的5-7bp间隔区。此长度的靶序列可能在人基因组中是独特的,假设在设计过程中排除重复序列或基因同源物。然而,zfn蛋白-dna相互作用在其特异性方面不是绝对的,因此确实发生脱靶结合和切割事件,作为两种zfn之间的异源二聚体,或作为所述zfn中的一种或另一种的同源二聚体。后一种的可能性已经通过工程化foki结构域的二聚化界面以产生“加”和“减”变体(也称为专性异源二聚体变体)而有效地消除,所述变体仅可以彼此二聚化而不与其自身二聚化。强制专性异源二聚体阻止了同源二聚体的形成。这已经大大增强了zfn以及采用这些foki变体的任何其他核酸酶的特异性。已经在本领域中描述了多种基于zfn的系统,其修饰被经常报告,并且许多参考文献描述了用于向导zfn的设计的规则和参数;参见例如,segal等人,procnatlacadsciusa96(6):2758-63(1999);dreierb等人,jmolbiol.303(4):489-502(2000);liuq等人,jbiolchem.277(6):3850-6(2002);dreier等人,jbiolchem280(42):35588-97(2005);以及dreier等人,jbiolchem.276(31):29466-78(2001)。转录激活因子样效应子核酸酶(talen)talen代表模块化核酸酶的另一种形式,其中与zfn一样,工程化的dna结合结构域与foki核酸酶结构域连接,并且一对talen串联操作以实现靶向dna切割。与zfn的主要区别是dna结合结构域的性质和相关靶dna序列识别性质。talendna结合结构域来源于最初在植物细菌病原体黄单胞菌属中描述的tale蛋白。tale由33-35个氨基酸重复序列的串联阵列组成,其中每个重复序列识别靶dna序列中的单个碱基对,所述靶dna序列的长度通常至多20bp,从而提供至多40bp的总靶序列长度。每个重复序列的核苷酸特异性由重复可变二残基(rvd)确定,所述rvd在位置12和13处仅包括两个氨基酸。碱基鸟嘌呤、腺嘌呤、胞嘧啶和胸腺嘧啶分别主要通过四个rvdasn-asn、asn-ile、his-asp和asn-gly识别。这构成比锌指简单的多的识别码,并且因此代表超过后者的用于核酸酶设计的优点。然而,与zfn一样,talen的蛋白质-dna相互作用在其特异性方面不是绝对的,并且talen还已经受益于使用foki结构域的专性异源二聚体变体来减少脱靶活性。已经产生了在其催化功能方面被去活化的附加foki结构域变体。如果talen或zfn对的一半含有失活的foki结构域,则将在靶位点处将仅发生单链dna切割(切口),而不是dsb。结局与使用crispr/cas9“切口酶”突变体相当,其中cas9切割结构域中的一个已失活。dna切口可用于驱动hdr进行基因组编辑,但比使用dsb的效率低。主要的益处是脱靶切口被快速和准确地修复,不同于易发生nhej介导的错修复的dsb。已经在本领域中描述了多种基于talen的系统,并且经常报告对其的修饰;参见例如,boch,science326(5959):1509-12(2009);mak等人,science335(6069):716-9(2012);以及moscou等人,science326(5959):1501(2009)。基于“金门("goldengate)”平台的talen的使用已经由多个组描述;参见例如,cermak等人,nucleicacidsres.39(12):e82(2011);li等人,nucleicacidsres.39(14):6315-25(2011);weber等人,plosone.6(2):e16765(2011);wang等人,jgenetgenomics41(6):339-47,电子版,2014年5月17日(2014);以及cermakt等人,methodsmolbiol.1239:133-59(2015)。归巢核酸内切酶归巢内切核酸酶(he)是具有长识别序列(14-44个碱基对)的序列特异性核酸内切酶,并且以高特异性切割dna-通常在基因组中的独特位点处。存在根据其结构分类的至少六个已知的he家族,包括来自广泛的宿主(包括真核生物、原生生物、细菌、古细菌、蓝细菌和噬菌体)的laglidadg(seqidno:192)、giy-yig、his-cis盒、h-n-h、pd-(d/e)xk以及vsr样。与zfn和talen一样,作为基因组编辑中的初始步骤,he可用于在靶位点处产生dsb。此外,一些天然和工程化的he仅切割dna的单链,从而充当位点特异性切口酶。he的大靶序列和所提供的特异性已经使得它们成为产生位点特异性dsb的有吸引力的候选者。已经在本领域中描述了多种基于he的系统,并且经常报道其修饰;参见例如由steentoft等人,glycobiology24(8):663-80(2014);belfort和bonocora,methodsmolbiol.1123:1-26(2014);hafez和hausner,genome55(8):553-69(2012);以及其中所引用的参考文献所进行的评论。megatal/tev-mtalen/megatev作为杂交核酸酶的进一步示例,megatal平台和tev-mtalen平台利用taledna结合结构域与催化活性he的融合,利用tale的可调节dna结合和特异性以及he的切割序列特异性;参见例如boissel等人,nar42:2591-2601(2014);kleinstiver等人,g34:1155-65(2014);以及boissel和scharenberg,methodsmol.biol.1239:171-96(2015)。在进一步的变型中,megatev架构是大范围核酸酶(mega)与来源于giy-yig归巢核酸内切酶i-tevi(tev)的核酸酶结构域的融合。两个活性位点定位为在dna底物上间隔约30bp,并产生具有不相容的粘性末端的两个dsb;参见例如,wolfs等人,nar42,8816-29(2014)。预期现有的基于核酸酶的方法的其它组合将进化并且可用于实现本文所述的靶向基因组修饰。dcas9-foki及其他核酸酶结合上述核酸酶平台的结构和功能特性提供了能够潜在地克服一些固有缺陷的进一步基因组编辑方法。例如,crispr基因组编辑系统通常使用单个cas9核酸内切酶来产生dsb。靶向的特异性由向导rna中的20个核苷酸长的序列驱动,该20个核苷酸长的序列经历与靶dna的沃森-克里克碱基配对(在来自化脓性链球菌的cas9的情况下,在相邻nag或nggpam序列中加上另外2个碱基)。此类序列足够长以在人类基因组中是独特的,然而,rna/dna相互作用的特异性不是绝对的,具有有时特别是在靶序列的5’半部分中接纳的显著混杂性,从而有效地减少驱动特异性的碱基数。对于此的一个解决方案是完全去活化cas9催化功能——仅保留rna向导的dna结合功能——并且替代地将foki结构域融合到失活的cas9;参见例如,tsai等人,naturebiotech32:569-76(2014);以及guilinger等人,naturebiotech.32:577-82(2014)。由于foki必须二聚化以变得具有催化活性,所以需要两个向导rna来系联紧密接近的两个cas9-foki融合体以形成二聚体并切割dna。这基本上使组合靶位点中的碱基数目加倍,从而增加了基于crispr的系统的靶向的严格性。作为进一步的示例,taledna结合结构域与催化活性he(例如i-tevi)的融合利用tale的可调节dna结合和特异性以及i-tevi的切割序列特异性,以期望可以进一步减少脱靶切割。crispr/cas核酸内切酶体系crispr(成簇规律间隔短回文重复序列)基因组位点可以存在于许多原核生物(例如,细菌和古细菌)的基因组中。在原核生物中,crispr位点编码充当一种类型的免疫系统的产物,以帮助保护原核生物抵抗外来侵入物如病毒和噬菌体。crispr位点功能有三个阶段:将新序列整合到位点、crisprrna(crrna)的生物发生、以及外来侵略核酸的沉默。已经鉴定了四种类型的crispr体系(例如i型、ii型、iii型、u型)。crispr位点包括许多称为“重复序列”的短重复序列。重复序列可以形成发夹结构和/或包含非结构化的单链序列。重复序列通常存在于簇中并且经常在各物种之间不同。重复序列与称为“间隔区”的独特间插序列间隔开,从而产生重复序列-间隔区-重复序列的位点架构。间隔区与已知的外来侵入物序列相同或具有高同源性。间隔重复序列单元编码crisprrna(crrna),crrna被加工成间隔重复序列单元的成熟形式。crrna包含参与靶向靶核酸的“种子”或间隔序列(为在原核生物中天然存在的形式,所述间隔序列靶向外源侵入核酸)。间隔序列位于crrna的5'或3'末端。crispr位点还包括编码crispr相关(cas)基因的多核苷酸序列。cas基因编码参与原核生物中的crrna功能的生物发生和干扰阶段的核酸内切酶。一些cas基因包含同源的二级和/或三级结构。ii型crispr系统在自然界中ii型crispr系统中的crrna生物发生需要反式激活crisprrna(tracrrna)。tracrrna由内源rnaseiii修饰,然后与pre-crrna阵列中的crrna重复序列杂交。募集内源性rnaseiii以切割pre-crrna。经切割的crrna经受核糖核酸外切酶修剪以产生成熟的crrna形式(例如,5'端修剪)。tracrrna保持与crrna杂交,并且tracrrna和crrna与定点多肽(例如cas9)缔合。crrna-tracrrna-cas9复合物的crrna将复合物引导至crrna可以与其杂交的靶核酸。crrna与靶核酸的杂交激活cas9以进行靶向核酸切割ii型crispr系统中的靶核酸被称为前间隔序列相邻基序(pam)。在自然界中,pam对于促进定点多肽(例如cas9)与靶核酸的结合是必需的。ii型系统(也称为nmeni或cass4)进一步细分为ii-a型(cass4)和ii-b型(cass4a)。jinek等人,science,337(6096):816-821(2012)显示crispr/cas9系统可用于rna可编程基因组编辑,并且wo2013/176772提供了关于crispr/cas核酸内切酶系统用于位点特异性基因编辑的许多示例和应用。cas基因/多肽和前间隔序列相邻基序示例性crisprcas多肽包括fonfara等人,nucleicacidsresearch,42:2577-2590(2014)的图1中的cas9多肽。因为cas基因被探索,所以crispr-cas基因命名系统已经经历了广泛的重写。fonfara,出处同上中的图5提供了来自多种物种的cas9多肽的pam序列。定点多肽本公开中的定点多肽是在基因组编辑用以切割dna的核酸酶。在本文的crispr/cas系统的情境中,定点多肽可以与向导rna结合,该接合继而指定多肽所针对的靶dna中的位点。在本文的crispr/cas系统的实施方案中,定点多肽是核酸内切酶。在一些实施方案中,定点多肽包含多个核酸切割(即核酸酶)结构域。两个或更多个核酸切割结构域可以通过接头序列连接在一起。在一些实施方案中,接头序列包含柔性接头序列。接头序列包括1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、30、35、40个或更多个氨基酸长。天然存在的野生型cas9酶包含两个核酸酶结构域,即hnh核酸酶结构域和ruvc结构域。在本文中,“cas9”是指天然存在的cas9和重组的cas9两者。本文设想的cas9酶包含hnh或hnh样核酸酶结构域和/或ruvc或ruvc样核酸酶结构域。hnh或hnh样结构域包含mcra样折叠部。hnh或hnh样结构域包含两个反向平行的β-链和一个α-螺旋。hnh或hnh样结构域包含金属结合位点(例如二价阳离子结合位点)。hnh或hnh样结构域可以切割靶核酸的一条链(例如,crrna靶向链的互补链)。ruvc或ruvc样结构域包含rnaseh或rnaseh样折叠。ruvc/rnaseh结构域参与基于核酸的功能(包括作用于rna和dna)的不同集合。rnaseh结构域包含由多个α-螺旋包围的5个β-链。ruvc/rnaseh或ruvc/rnaseh样结构域包含金属结合位点(例如二价阳离子结合位点)。ruvc/rnaseh或ruvc/rnaseh样结构域可以切割靶核酸的一条链(例如双链靶dna的非互补链)。定点多肽可以在核酸(例如基因组dna)中引入双链断裂或单链断裂。双链断裂可以刺激细胞的内源性dna修复途径(例如,同源依赖性修复(hdr)和非同源末端连接(nhej)或替代的非同源末端连接(a-nhej)或微同源介导的末端连接(mmej)。nhej可以修复被切割的靶核酸,而不需要同源模板。这有时可导致在切割位点的靶核酸中的小缺失或插入(插入缺失),并且可导致对基因表达的破坏或改变。hdr可以在同源修复模板或供体可用时发生。同源供体模板包含与靶核酸切割位点侧翼的序列同源的序列。姐妹染色单体通常被细胞用作修复模板。然而,为了基因组编辑的目的,修复模板通常作为外源核酸供应,例如质粒、双链体寡核苷酸、单链寡核苷酸或病毒核酸。使用外源供体模板,通常在具有同源性的侧翼区之间引入附加核酸序列(例如转基因)或修饰(例如单碱基改变或缺失),因此附加的或改变的核酸序列也变成被引入到靶位点中。mmej导致的与nhej类似的遗传结局在于小缺失和插入可以发生在切割位点处。mmej利用在切割位点侧翼的具有几个碱基对的同源序列来驱动有利的末端连接dna修复结局。在一些情况下,可以基于对核酸酶靶区域中的潜在微同源性的分析来预测可能的修复结局。因此,在一些情况下,使用同源重组将外源多核苷酸序列插入靶核酸切割位点。外源多核苷酸序列在本文中被称为供体多核苷酸。在一些实施方案中,将供体多核苷酸,供体多核苷酸的一部分,供体多核苷酸拷贝或供体多核苷酸拷贝的一部分插入靶核酸切割位点。在一些实施方案中,供体多核苷酸是外源多核苷酸序列,即在靶核酸切割位点处不天然存在的序列。由于nhej和/或hdr产生的对靶dna的修饰可导致例如突变、缺失、改变、整合、基因校正、基因置换、基因标记、转基因插入、核苷酸缺失、基因破坏、易位和/或基因突变。缺失基因组dna和将非天然核酸整合到基因组dna中的过程是基因组编辑的示例。在一些实施方案中,定点多肽包含与野生型示范性定点多肽[例如,来自化脓性链球菌的cas9,us2014/0068797序列idno.8或sapranauskas等人,nucleicacidsres,39(21):9275-9282(2011)]具有至少10%、至少15%、至少20%、至少30%、至少40%、至少50%、至少60%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少99%或100%的氨基酸序列同一性的氨基酸序列,以及多种其他定点多肽)。在一些实施方案中,定点多肽包含与野生型示范性定点多肽(例如,来自化脓性链球菌的cas9,出处同上)的核酸酶结构域具有至少10%、至少15%、至少20%、至少30%、至少40%、至少50%、至少60%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少99%或100%的氨基酸序列同一性的氨基酸序列。在一些实施方案中,定点多肽包含在10个连续氨基酸上与野生型定点多肽(例如,来自化脓性链球菌的cas9,出处同上)至少70%、75%、80%、85%、90%、95%、97%、99%或100%的同一性。在一些实施方案中,定点多肽包含至多:在10个连续氨基酸上与野生型定点多肽(例如,来自化脓性链球菌的cas9,出处同上)70%、75%、80%、85%、90%、95%、97%、99%或100%的同一性。在一些实施方案中,定点多肽包含至少:在定点多肽的hnh核酸酶结构域中的10个连续氨基酸上与野生型定点多肽(例如,来自化脓性链球菌的cas9,出处同上)70%、75%、80%、85%、90%、95%、97%、99%或100%的同一性。在一些实施方案中,定点多肽包含至多:在定点多肽的hnh核酸酶结构域中的10个连续氨基酸上与野生型定点多肽(例如,来自化脓性链球菌的cas9,出处同上)70%、75%、80%、85%、90%、95%、97%、99%或100%的同一性。在一些实施方案中,定点多肽包含至少:在定点多肽的ruvc核酸酶结构域中的10个连续氨基酸上与野生型定点多肽(例如,来自化脓性链球菌的cas9,出处同上)70%、75%、80%、85%、90%、95%、97%、99%或100%的同一性。在一些实施方案中,定点多肽包含至多:在定点多肽的ruvc核酸酶结构域中的10个连续氨基酸上与野生型定点多肽(例如,来自化脓性链球菌的cas9,出处同上)70%、75%、80%、85%、90%、95%、97%、99%或100%的同一性。在一些实施方案中,定点多肽包含野生型示范性定点多肽的修饰形式。野生型示范性定点多肽的修饰形式包含降低定点多肽的核酸切割活性的突变。在一些实施方案中,野生型示范性定点多肽的修饰形式具有野生型示范性定点多肽(例如,来自化脓性链球菌的cas9,出处同上)的核酸切割活性的小于90%、小于80%、小于70%、小于60%、小于50%、小于40%、小于30%、小于20%、小于10%、小于5%或小于1%。定点多肽的修饰形式可以没有实质的核酸切割活性。当定点多肽是没有实质的核酸切割活性的修饰形式时,其在本文中被称为“无酶活性的”。在一些实施方案中,定点多肽的修饰形式包括使得可以在靶核酸上诱导单链断裂(ssb)的突变(例如,通过切割双链靶核酸的糖-磷酸骨架中的仅一个)。在一些实施方案中,突变导致野生型定点多肽(例如,来自化脓性链球菌的cas9,出处同上)的多个核酸切割结构域中的一个或更多个核酸切割结构域的核酸切割活性的小于90%、小于80%、小于70%、小于60%、小于50%、小于40%、小于30%、小于20%、小于10%、小于5%或小于1%。在一些实施方案中,突变导致保留切割靶核酸的互补链的能力但降低切割靶核酸的非互补链的能力的多个核酸切割结构域中的一个或更多个。在一些实施方案中,突变导致保留切割靶核酸的非互补链的能力但降低切割靶核酸的互补链的能力的多个核酸切割结构域中的一个或更多个。例如,野生型示范性化脓性链球菌cas9多肽中的残基(诸如asp10、his840、asn854和asn856)经突变以使多个核酸切割结构域(例如核酸酶结构域)中的一个或更多个失活。在一些实施方案中,待突变的残基对应于野生型示范性化脓性链球菌cas9多肽中的残基asp10、his840、asn854和asn856(例如,如通过序列和/或结构比对确定的)。突变的非限制性示例可以包括d10a、h840a、n854a或n856a。本领域的技术人员将认识到除丙氨酸取代之外的突变是合适的。在一些实施方案中,d10a突变与h840a、n854a或n856a突变中的一个或更多个组合以产生基本上没有dna切割活性的定点多肽。在一些实施方案中,h840a突变与d10a、n854a或n856a突变中的一个或更多个组合以产生基本上没有dna切割活性的定点多肽。在一些实施方案中,n854a突变与h840a、d10a或n856a突变中的一个或更多个组合以产生基本上没有dna切割活性的定点多肽。在一些实施方案中,n856a突变与h840a、n854a或d10a突变中的一个或更多个组合以产生基本上没有dna切割活性的定点多肽。包含一个基本上失活的核酸酶结构域的定点多肽在本文中称为切口酶。cas9的切口酶变体可用于增加crispr介导的基因组编辑的特异性。野生型cas9通常由设计为与靶序列中指定的约20nt序列(例如内源基因组位点)杂交的单个向导rna引导。然而,在向导rna和靶位点之间可以容忍若干个错配,从而有效地减少了靶位点中所需同源性的长度,例如低至13nt的同源性并且由此导致crispr/cas9复合物在靶基因组中的其他地方结合并进行双链核酸切割——也称为脱靶切割——的潜能升高。由于cas9的切口酶变体各自仅切割一条链,为了产生双链断裂,需要一对切口酶紧密接近并在靶核酸的相对链上结合,从而产生一对切口,该对切口等效于双链断裂。这要求两个单独的向导rna——每个切口酶一个——必须紧密接近并在靶核酸的相对链上结合。这种要求实质上使双链断裂发生所需的最小同源长度加倍,从而降低了双链切割事件将在基因组中其他地方发生的可能性,其中两个向导rna位点——如果它们存在的话——不太可能足够接近彼此来使双链断裂形成。如本领域中所述,切口酶也可用于促进hdr对nhej。hdr可用于通过使用有效介导所需变化的特异性供体序列来将所选择的变化引入基因组中的靶位点中。关于用于基因编辑中的多种crispr-cas系统的描述可见于例如wo2013/176772中和naturebiotechnology32,347–355(2014),以及其中所引用的参考文献中。所考虑的突变包括取代、添加和缺失,或其任何组合。在一些实施方案中,突变将突变的氨基酸转化为丙氨酸。在一些实施方案中,突变将突变的氨基酸转化为另一种氨基酸(例如甘氨酸、丝氨酸、苏氨酸、半胱氨酸、缬氨酸、亮氨酸、异亮氨酸、甲硫氨酸、脯氨酸、苯丙氨酸、酪氨酸、色氨酸、天冬氨酸、谷氨酸、天冬酰胺、谷氨酰胺、组氨酸、赖氨酸或精氨酸)。在一些实施方案中,突变将突变的氨基酸转化为非天然氨基酸(例如硒代甲硫氨酸)。在一些实施方案中,突变将突变的氨基酸转化为氨基酸模拟物(例如磷酸模拟物)。在一些实施方案中,突变是保守突变。例如,突变可以将突变的氨基酸转化为类似于突变氨基酸的大小、形状、电荷、极性、构象和/或旋转异构体的氨基酸(例如半胱氨酸/丝氨酸突变、赖氨酸/天冬酰胺突变、组氨酸/苯丙氨酸突变)。在一些实施方案中,突变导致阅读框移位和/或产生提前的终止密码子。在一些实施方案中,突变引起对基因或位点的调控区的改变,所述改变影响一个或更多个基因的表达。在一些实施方案中,定点多肽(例如变体、突变的、无酶活性的和/或条件性地无酶活性的定点多肽)靶向核酸。在一些实施方案中,定点多肽(例如变体、突变的、无酶活性的和/或条件性地无酶活性的核糖核酸内切酶)可以靶向rna。在一些实施方案中,定点多肽包含一个或更多个非天然序列(例如,定点多肽是融合蛋白)。在一些实施方案中,定点多肽包含与来自细菌(例如化脓性链球菌)的cas9具有至少15%的氨基酸同一性的氨基酸序列、核酸结合结构域以及两个核酸切割结构域(即,hnh结构域和ruvc结构域)。在一些实施方案中,定点多肽包含与来自细菌(例如化脓性链球菌)的cas9具有至少15%的氨基酸同一性的氨基酸序列和两个核酸切割结构域(即,hnh结构域和ruvc结构域)。在一些实施方案中,定点多肽包含与来自细菌(例如化脓性链球菌)的cas9具有至少15%的氨基酸同一性的氨基酸序列和两个核酸切割结构域,其中所述核酸切割结构域中的一个或两个具有与来自细菌(例如化脓性链球菌)的cas9的核酸酶结构域至少50%的氨基酸同一性。在一些实施方案中,定点多肽包含与来自细菌(例如化脓性链球菌)的cas9具有至少15%的氨基酸同一性的氨基酸序列、两个核酸切割结构域(即,hnh结构域和ruvc结构域)、以及非天然序列(例如,核定位信号)或将定点多肽连接到非天然序列的接头序列。在一些实施方案中,定点多肽包含与来自细菌(例如化脓性链球菌)的cas9具有至少15%的氨基酸同一性的氨基酸序列、两个核酸切割结构域(即,hnh结构域和ruvc结构域),其中定点多肽包含在所述核酸切割结构域的一个或两个中使核酸酶结构域的切割活性降低至少50%的突变。在一些实施方案中,定点多肽包含与来自细菌(例如,化脓性链球菌)的cas9具有至少15%的氨基酸同一性的氨基酸序列和两个核酸切割结构域(即,hnh结构域和ruvc结构域),其中核酸酶结构域中的一个包含天冬氨酸10的突变,和/或其中核酸酶结构域中的一个包含组氨酸840的突变,并且其中所述突变使一个或更多个核酸酶结构域的切割活性降低至少50%。核酸靶向核酸本公开提供了核酸靶向核酸,该核酸靶向核酸可以将相关多肽(例如,定点多肽)的活性导向靶核酸内的特定靶序列。在一些实施方案中,核酸靶向核酸是rna。核酸靶向rna在本文中称为“向导rna”。向导rna包含与所关注的靶核酸序列、crispr重复序列和tracrrna序列杂交的至少一间隔序列。在向导rna中,crispr重复序列和tracrrna序列彼此杂交以形成双链体。双链体结合定点多肽,使得向导rna和定点多肽形成复合物。核酸靶向核酸通过其与定点多肽的缔合来提供对所述复合物的靶特异性。因此,核酸靶向核酸向导定点多肽的活性。示例性向导rna包括在表1中显示有向导rna的基因组靶序列、其靶序列的基因组位置和相关cas9切割位点的向导rna,其中靶序列和基因组位置基于grch38/hg38人基因组组件。如本领域的普通技术人员所理解的,每个向导rna被设计为包括与其基因组靶序列互补的间隔序列。例如,图1至图6中的每个间隔序列可以被放入单个rna嵌合体或crrna(连同相应的tracrrna一起)中。参见jinek等人,science,337,816-821(2012)和deltcheva等人,nature,471,602-607(2011)。表1在一些实施方案中,核酸靶向核酸是双分子向导rna。在一些实施方案中,核酸靶向核酸是单分子向导rna。双分子向导rna包含两条rna链。第一链在5'至3'方向上包含任选的间隔延伸序列、间隔序列和最小crispr重复序列。第二链包含最小tracrrna序列(与最小crispr重复序列互补)、3’端tracrrna序列和任选的tracrrna延伸序列。单分子向导rna在5'至3'方向上包括任选的间隔延伸序列、间隔序列、最小crispr重复序列、单分子向导接头序列、最小tracrrna序列,3'端tracrrna序列和任选的tracrrna延伸序列。任选的tracrrna延伸可以包括对向导rna贡献附加功能(例如稳定性)的元件。单分子向导接头序列将最小crispr重复序列和最小tracrrna序列连接以形成发夹结构。任选的tracrrna延伸包含一个或更多个发夹。例如,crispr-cas系统中使用的向导rna,或其他较小的rna可以通过化学手段容易地合成,如在下文所说明和在本领域中所描述的。虽然化学合成方法不断扩展,但是通过诸如高效液相色谱(hplc,其避免使用凝胶如page)的方法对这种rna进行纯化倾向于变得更具挑战性,因为多核苷酸长度显著增加到超过约一百个核苷酸。用于产生更大长度的rna的一种方法是产生连接在一起的两个或更多个分子。较长的rna,例如编码cas9核酸内切酶的那些rna,更容易酶促地产生。可以在化学合成和/或酶促生成rna期间或之后引入多种类型的rna修饰,例如增强稳定性、降低先天免疫应答的可能性或程度和/或增强其他属性的修饰,如本领域中所述。间隔延伸序列在核酸靶向核酸的一些实施方案中,间隔延伸序列可以提供稳定性和/或提供用于修饰核酸靶向核酸的位置。在一些实施方案中,提供了间隔延伸序列。间隔物延伸序列可以具有大于1、5、10、15、20、25、30、35、40、45、50、60、70、80、90、100、120、140、160、180、200、220、240、260、280、300、320、340、360、380、400、1000、2000、3000、4000、5000、6000、或7000个或更多个核苷酸的长度。间隔物延伸序列可以具有小于1、5、10、15、20、25、30、35、40、45、50、60、70、80、90、100、120、140、160、180、200、220、240、260、280、300、320、340、360、380、400、1000、2000、3000、4000、5000、6000、或7000个或更多个核苷酸的长度。在一些实施方案中,间隔延伸序列的长度小于10个核苷酸。在一些实施方案中,间隔延伸序列的长度为在10个与30个核苷酸之间。在一些实施方案中,间隔延伸序列的长度在30至70个核苷酸之间。在一些实施方案中,间隔延伸序列包含另一个部分(例如,稳定性控制序列、核糖核酸内切酶结合序列、核糖酶)。在一些实施方案中,所述部分增加了核酸靶向核酸的稳定性。在一些实施方案中,所述部分是转录终止子区段(即,转录终止序列)。在一些实施方案中,所述部分在真核细胞中起作用。在一些实施方案中,所述部分在原核细胞中起作用。在一些实施方案中,所述部分在真核细胞和原核细胞两者中起作用。合适部分的非限制性示例包括:5'帽(例如,7-甲基鸟苷酸帽(m7g));核糖开关序列(例如,以实现经调控的稳定性和/或经调控的蛋白和蛋白复合物可及性);形成dsrna双链体(即,发夹)的序列;使rna靶向亚细胞位置(例如,细胞核、线粒体、叶绿体等)的序列;提供示踪的修饰或序列(例如,与荧光分子的直接缀合、与有利于荧光检测的部分的缀合、允许荧光检测的序列等);和/或为蛋白质(例如,作用于dna的蛋白质,包括转录激活因子、转录阻遏物、dna甲基转移酶、dna去甲基化酶、组蛋白乙酰转移酶、组蛋白去乙酰化酶等)提供结合位点的修饰或序列。间隔序列间隔序列与所关注的靶核酸中的序列杂交。核酸靶向核酸的间隔区通过杂交(即碱基配对)以序列特异性方式与靶核酸相互作用。因此,间隔区的核苷酸序列根据所关注的靶核酸的序列而变化。在本文的crispr/cas系统中,间隔序列被设计为与位于该系统中使用的cas9酶的pam的5'端的靶核酸杂交。每种cas9酶具有其在靶dna中识别的特定pam序列。例如,化脓性链球菌在靶核酸中识别包含序列5'-nrg-3'的pam,其中r包含a或g,其中n是任何核苷酸,n紧接在由间隔序列所靶向的靶核酸序列的3'端。在一些实施方案中,靶核酸序列包含20个核苷酸。在一些实施方案中,靶核酸包含少于20个核苷酸。在一些实施方案中,靶核酸包含至少:5、10、15、16、17、18、19、20、21、22、23、24、25、30或更多个核苷酸。在一些实施方案中,靶核酸包含至多:5、10、15、16、17、18、19、20、21、22、23、24、25、30或更多个核苷酸。在一些实施方案中,靶核酸序列包含紧接在pam的第一核苷酸的5'端的20个碱基。例如,在包含5'-nnnnnnnnnnnnnnnnnnnnnrg-3'(seqidno:143)中,靶核酸包含对应于ns的序列,其中n是任意核苷酸。在一些实施方案中,与靶核酸杂交的间隔序列具有至少约6nt的长度。间隔序列可以为至少约6nt、至少约10nt、至少约15nt、至少约18nt、至少约19nt、至少约20nt、至少约25nt、至少约30nt、至少约35nt或至少约40nt、约6nt至约80nt、约6nt至约50nt、约6nt至约45nt、约6nt至约40nt、约6nt至约35nt、约6nt至约30nt、约6nt至约25nt、约6nt至约20nt、约6nt至约19nt、约10nt至约50nt、约10nt至约45nt、约10nt至约40nt、约10nt至约35nt、约10nt至约30nt、约10nt至约25nt、约10nt至约20nt、约10nt至约19nt、约19nt至约25nt、约19nt至约30nt、约19nt至约35nt、约19nt至约40nt、约19nt至约45nt、约19nt至约50nt、约19nt至约60nt、约20nt至约25nt、约20nt至约30nt、约20nt至约35nt、约20nt至约40nt、约20nt至约45nt、约20nt至约50nt、或约20nt至约60nt。在一些实施方案中,间隔序列包含20个核苷酸。在一些实施方案中,间隔区包含19个核苷酸。在一些实施方案中,间隔序列与靶核酸之间的互补性百分比为至少约30%、至少约40%、至少约50%、至少约60%、至少约65%、至少约70%、至少约75%、至少约80%、至少约85%、至少约90%、至少约95%、至少约97%、至少约98%、至少约99%、或100%。在一些实施方案中,间隔序列与靶核酸之间的互补性百分比为至多约30%、至多约40%、至多约50%、至多约60%、至多约65%、至多约70%、至多约75%、至多约80%、至多约85%、至多约90%、至多约95%、至多约97%、至多约98%、至多约99%、或100%。在一些实施方案中,间隔序列与靶核酸之间的互补性百分比在靶核酸的互补链的靶序列的六个连续的最5'端核苷酸上为100%。在一些情况下,间隔序列与靶核酸之间的互补性百分比在约20个连续的核苷酸上为至少60%。在一些实施方案中,使用计算机程序来设计或选择间隔序列。计算机程序可以使用多个变量,诸如预测的解链温度、二级结构形成和预测的退火温度、序列同一性、基因组结构、染色质可及性、gc%,基因组发生频率(例如,相同或相似但由于错配、插入或缺失而在一个或更多个点中不同的序列的基因组发生频率)、甲基化状态、snp的存在等。最小crispr重复序列在一些实施方案中,最小crispr重复序列是与参考crispr重复序列(例如,来自化脓性链球菌的crrna)具有至少:约30%、约40%、约50%、约60%、约65%、约70%、约75%、约80%、约85%、约90%、约95%或100%的序列同一性的序列。最小crispr重复序列包含可与细胞中最小tracrrna序列杂交的核苷酸。最小crispr重复序列和最小tracrrna序列形成双链体,即碱基配对的双链结构。最小crispr重复序列和最小tracrrna序列一起结合至定点多肽。最小crispr重复序列的至少一部分与最小tracrrna序列杂交。在一些实施方案中,最小crispr重复序列的至少一部分包含与最小tracrrna序列至少:约30%、约40%、约50%、约60%、约65%、约70%、约75%、约80%、约85%、约90%、约95%、或100%的互补性。在一些实施方案中,最小crispr重复序列的至少一部分包含与最小tracrrna序列至多:约30%、约40%、约50%、约60%、约65%、约70%、约75%、约80%、约85%、约90%、约95%、或100%的互补性。最小crispr重复序列的长度可为约7个核苷酸至约100个核苷酸。例如,最小crispr重复序列的长度是约7个核苷酸(nt)至约50nt、约7nt至约40nt、约7nt至约30nt、约7nt至约25nt、约7nt至约20nt、约7nt至约15nt、约8nt至约40nt、约8nt至约30nt、约8nt至约25nt、约8nt至约20nt或约8nt至约15nt、约15nt至约100nt、约15nt至约80nt、约15nt至约50nt、约15nt至约40nt、约15nt至约30nt或约15nt至约25nt。在一些实施方案中,最小crispr重复序列为约9个核苷酸长。在一些实施方案中,最小crispr重复序列为约12个核苷酸长。在一些实施方案中,最小crispr重复序列与参考最小crispr重复序列(例如,来自化脓性链球菌的野生型crrna)在至少6、7或8个连续核苷酸的延伸上至少约60%同一。例如,最小crispr重复序列与参考最小crispr重复序列在至少6、7或8个连续核苷酸的延伸上至少约65%同一、至少约70%同一、至少约75%同一、至少约80%同一、至少约85%同一、至少约90%同一、至少约95%同一、至少约98%同一、至少约99%同一或100%同一。最小tracrrna序列在一些实施方案中,最小tracrrna序列是与参考tracrrna序列(例如,来自化脓性链球菌的野生型tracrrna)具有至少:约30%、约40%、约50%、约60%、约65%、约70%、约75%、约80%、约85%、约90%、约95%或100%的序列同一性的序列。最小tracrrna序列包含与细胞中的最小crispr重复序列杂交的核苷酸。最小tracrrna序列和最小crispr重复序列形成双链体,即碱基配对的双链结构。最小tracrrna序列和最小crispr重复序列一起结合至定点多肽。最小tracrrna序列的至少一部分可以与最小crispr重复序列杂交。在一些实施方案中,最小tracrrna序列与最小crispr重复序列至少:约30%、约40%、约50%、约60%、约65%、约70%、约75%、约80%、约85%、约90%、约95%或100%互补。最小tracrrna序列的长度可为约7个核苷酸至约100个核苷酸。例如,最小tracrrna序列可以是约7个核苷酸(nt)至约50nt、约7nt至约40nt、约7nt至约30nt、约7nt至约25nt、约7nt至约20nt、约7nt至约15nt、约8nt至约40nt、约8nt至约30nt、约8nt至约25nt、约8nt至约20nt或约8nt至约15nt、约15nt至约100nt、约15nt至约80nt、约15nt至约50nt、约15nt至约40nt、约15nt至约30nt或约15nt至约25nt。在一些实施方案中,最小tracrrna序列为约9个核苷酸长。在一些实施方案中,最小tracrrna序列为约12个核苷酸。在一些实施方案中,最小tracrrna由jinek等人,出处同上中描述的tracrrnant23-48组成。在一些实施方案中,最小tracrrna序列与参考最小tracrrna(例如,来自化脓性链球菌的野生型crrna)序列在至少:6、7或8个连续核苷酸的延伸上至少约60%同一。例如,最小tracrrna序列与参考最小tracrrna序列在至少6、7或8个连续核苷酸的延伸上至少:约65%同一、约70%同一、约75%同一、约80%同一、约85%同一、约90%同一、约95%同一、约98%同一、约99%同一或100%同一。在一些实施方案中,最小crisprrna与最小tracrrna之间的双链体包含双螺旋。在一些实施方案中,最小crisprrna与最小tracrrna之间的双链体包含至少约:1、2、3、4、5、6、7、8、9或10个或更多个核苷酸。在一些实施方案中,最小crisprrna与最小tracrrna之间的双链体包含至多约:1、2、3、4、5、6、7、8、9或10个或更多个核苷酸。在一些实施方案中,双链体包含错配(即,双链体的两条链不是100%互补)。在一些实施方案中,双链体包含至少约:1、2、3、4或5或更多个错配。在一些实施方案中,双链体包含至多约:1、2、3、4或5或更多个错配。在一些实施方案中,双链体包含不超过2个错配。凸起(bulges)在一些实施方案中,在最小crisprrna和最小tracrrna之间的双链体中存在“凸起”。该凸起是所述双链体内的核苷酸的未配对区域。在一些实施方案中,该凸起有助于双链体与定点多肽的结合。凸起包括在双链体的一侧上的未配对5'-xxxy-3',其中x是任何嘌呤并且y包含能够与相对链上的核苷酸形成摆动配对的核苷酸;以及在双链体另一侧上的未配对核苷酸区域。双链体两侧上的未配对核苷酸的数目可以是不同的。在一个示例中,凸起包括在凸起的最小crispr重复链上的未配对的嘌呤(例如腺嘌呤)。在一些实施方案中,凸起包含凸起的最小tracrrna序列链的未配对的5'-aagy-3',其中y包含可以与最小crispr重复链上的核苷酸形成摆动配对的核苷酸。在一些实施方案中,双链体的最小crispr重复序列侧上的凸起包含至少:1、2、3、4或5个或更多个未配对的核苷酸。在一些实施方案中,双链体的最小crispr重复序列侧上的凸起包含至多:1、2、3、4或5个或更多个未配对的核苷酸。在一些实施方案中,双链体的最小crispr重复序列侧上的凸起包含1个未配对的核苷酸。在一些实施方案中,双链体的最小tracrrna序列侧上的凸起包含至少:1、2、3、4、5、6、7、8、9或10个或更多个未配对的核苷酸。在一些实施方案中,双链体的最小tracrrna序列侧上的凸起包含至多:1、2、3、4、5、6、7、8、9或10个或更多个未配对的核苷酸。在一些实施方案中,双链体第二侧(例如,双链体的最小tracrrna序列侧)上的凸起包含4个未配对的核苷酸。在一些实施方案中,凸起包括至少一个摆动配对。在一些实施方案中,凸起包括至多一个摆动配对。在一些实施方案中,凸起包含至少一个嘌呤核苷酸。在一些实施方案中,凸起包含至少3个嘌呤核苷酸。在一些实施方案中,凸起序列包含至少5个嘌呤核苷酸。在一些实施方案中,凸起序列包含至少一个鸟嘌呤核苷酸。在一些实施方案中,凸起序列包含至少一个腺嘌呤核苷酸。发夹在多种实施方案中,一个或更多个发夹位于3'端tracrrna序列中的最小tracrrna的3'端。在一些实施方案中,发夹结构从在3'端距最小crispr重复序列和最小tracrrna序列双链体中的最后一个配对核苷酸至少约:1、2、3、4、5、6、7、8、9、10、15或20个或更多个核苷酸处开始。在一些实施方案中,发夹结构从在3'端距最小crispr重复序列和最小tracrrna序列双链体中的最后一个配对核苷酸至多约:1、2、3、4、5、6、7、8、9或10个或更多个核苷酸处开始。在一些实施方案中,发夹结构包含至少约:1、2、3、4、5、6、7、8、9、10、15或20个或更多个连续核苷酸。在一些实施方案中,发夹结构包含至多约:1、2、3、4、5、6、7、8、9、10、15个或更多个连续核苷酸。在一些实施方案中,发夹包含cc二核苷酸(即,两个连续的胞嘧啶核苷酸)。在一些实施方案中,发夹包含双链核苷酸(例如,发夹结构中杂交在一起的核苷酸)。例如,发夹结构包含与3'端tracrrna序列的发夹双链体中的gg二核苷酸杂交的cc二核苷酸。发夹中的一个或更多个可以与定点多肽的向导rna相互作用区域相互作用。在一些实施方案中存在两个或更多个发夹,并且在一些实施方案中存在三个或更多个发夹。3'tracrrna序列在一些实施方案中,3'端tracrrna序列包含与参考tracrrna序列(例如,来自化脓性链球菌的tracrrna)具有至少:约30%、约40%、约50%、约60%、约65%、约70%、约75%、约80%、约85%、约90%、约95%或100%的序列同一性的序列。在一些实施方案中,3'端tracrrna序列的长度为约6个核苷酸至约100个核苷酸。例如,3'端tracrrna序列的长度可以是约6个核苷酸(nt)至约50nt、约6nt至约40nt、约6nt至约30nt、约6nt至约25nt、约6nt至约20nt、约6nt至约15nt、约8nt至约40nt、约8nt至约30nt、约8nt至约25nt、约8nt至约20nt或约8nt至约15nt、约15nt至约100nt、约15nt至约80nt、约15nt至约50nt、约15nt至约40nt、约15nt至约30nt或约15nt至约25nt。在一些实施方案中,3'端tracrrna序列的长度为约14个核苷酸。在一些实施方案中,3'端tracrrna序列与参考3'端tracrrna序列(例如,来自化脓性链球菌的野生型3'端tracrrna序列)在至少:6、7或8个连续核苷酸的延伸上至少约60%同一。例如,3'端tracrrna序列与参考3'端tracrrna序列(例如,来自化脓性链球菌的野生型3'端tracrrna序列)在至少6、7或8个连续核苷酸的延伸上至少:约60%同一、约65%同一、约70%同一、约75%同一、约80%同一、约85%同一、约90%同一、约95%同一、约98%同一、约99%同一、或100%同一。在一些实施方案中,3'tracrrna序列包含多于一个双链化区域(例如,发夹结构、杂交区域)。在一些实施方案中,3'端的tracrrna序列包含两个双链化区域。在一些实施方案中,3'端的tracrrna序列包含茎环结构。在一些实施方案中,3'端tracrrna中的茎环结构包含至少:1、2、3、4、5、6、7、8、9、10、15或20个或更多个核苷酸。在一些实施方案中,3'端tracrrna中的茎环结构包含至多:1、2、3、4、5、6、7、8、9、10个或更多个核苷酸。在一些实施方案中,茎环结构包含功能性部分。例如,茎环结构可以包含适体、核糖酶、蛋白质相互作用发夹结构、crispr阵列、内含子或外显子。在一些实施方案中,茎环结构包含至少约:1、2、3、4或5或更多个功能性部分。在一些实施方案中,茎环结构包含至多约:1、2、3、4或5或更多个功能性部分。在一些实施方案中,3'端的tracrrna序列中的发夹结构包含p结构域。在一些实施方案中,p结构域包含发夹结构中的双链区。tracrrna延伸序列可以提供tracrrna延伸序列,而无论tracrrna是否在单分子向导序列或双分子向导序列的环境中。在一些实施方案中,tracrrna延伸序列具有约1个核苷酸至约400个核苷酸的长度。在一些实施方案中,tracrrna延伸序列的长度大于:1、5、10、15、20、25、30、35、40、45、50、60、70、80、90、100、120、140、160、180、200、220、240、260、280、300、320、340、360、380、400个核苷酸。在一些实施方案中,tracrrna延伸序列的长度为约20至约5000或更多个核苷酸。在一些实施方案中,tracrrna延伸序列的长度大于1000个核苷酸。在一些实施方案中,tracrrna延伸序列的长度小于1、5、10、15、20、25、30、35、40、45、50、60、70、80、90、100、120、140、160、180、200、220、240、260、280、300、320、340、360、380、400个或更多个核苷酸。在一些实施方案中,tracrrna延伸序列的长度可以小于1000个核苷酸。在一些实施方案中,tracrrna延伸序列包含小于10个核苷酸的长度。在一些实施方案中,tracrrna延伸序列为10至30个核苷酸的长度。在一些实施方案中,tracrrna延伸序列为30至70个核苷酸的长度。在一些实施方案中,tracrrna延伸序列包含功能部分(例如,稳定性控制序列、核糖酶、核糖核酸内切酶结合序列)。在一些实施方案中,功能部分包含转录终止子区段(即,转录终止序列)。在一些实施方案中,功能部分的总长度为约10个核苷酸至约100个核苷酸、约10个核苷酸(nt)至约20nt、约20nt至约30nt、约30nt至约40nt、约40nt至约50nt、约50nt至约60nt、约60nt至约70nt、约70nt至约80nt、约80nt至约90nt,或约90nt至约100nt、约15个核苷酸(nt)至约80nt、约15nt至约50nt、约15nt至约40nt、约15nt至约30nt或约15nt至约25nt。在一些实施方案中,所述功能部分在真核细胞中起作用。在一些实施方案中,所述功能部分在原核细胞中起作用。在一些实施方案中,所述功能部分在真核细胞和原核细胞两者中起作用。合适tracrrna延伸功能部分的非限制性示例包括:3'多聚腺苷酸化尾;核糖开关序列(例如,以实现经调控的稳定性和/或经调控的蛋白和蛋白复合物可及性);形成dsrna双链体(即,发夹)的序列;使rna靶向亚细胞位置(例如,细胞核、线粒体、叶绿体等)的序列;提供示踪的修饰或序列(例如,与荧光分子的直接缀合、与有利于荧光检测的部分的缀合、允许荧光检测的序列等);和/或为蛋白质(例如,作用于dna的蛋白质,包括转录激活因子、转录阻遏物、dna甲基转移酶、dna去甲基化酶、组蛋白乙酰转移酶、组蛋白去乙酰化酶等)提供结合位点的修饰或序列。在一些实施方案中,tracrrna延伸序列包含引物结合位点、分子指标(例如,条形码序列)。在一些实施方案中,tracrrna延伸序列包含一个或更多个亲和标签。单分子向导序列的接头序列在一些实施方案中,单分子向导核酸的接头序列的长度为约3个核苷酸至约100个核苷酸。在jinek等人,出处同上中,例如使用简单的4核苷酸“四环”(-gaaa-),science,337(6096):816-821(2012)。说明性接头序列的长度为约3个核苷酸(nt)至约90nt、约3nt至约80nt、约3nt至约70nt、约3nt至约60nt、约3nt至约50nt、约3nt至约40nt、约3nt至约30nt、约3nt至约20nt或约3nt至约10nt。例如,接头序列的长度可为约3nt至约5nt、约5nt至约10nt、约10nt至约15nt、约15nt至约20nt、约20nt至约25nt、约25nt至约30nt、约30nt至约35nt、约35nt至约40nt、约40nt至约50nt、约50nt至约60nt、约60nt至约70nt、约70nt至约80nt、约80nt至约90nt、或约90nt至约100nt。在一些实施方案中,单分子向导核酸的接头序列的长度在4个核苷酸与40个核苷酸之间。在一些实施方案中,接头序列为至少约:100、500、1000、1500、2000、2500、3000、3500、4000、4500、5000、5500、6000、6500或7000个或更多个核苷酸。在一些实施方案中,接头序列为至多约:100、500、1000、1500、2000、2500、3000、3500、4000、4500、5000、5500、6000、6500或7000个或更多个核苷酸。接头序列可以包含多种序列中的任一种,尽管优选接头序列将不包含具有与向导rna的其他部分有同源性的广泛区域的序列,包含具有与向导rna的其他部分有同源性的广泛区域的序列可能会引起分子内结合,这可能会干扰向导序列的其他功能区域。在jinek等人,出处同上中,使用简单的4核苷酸序列-gaaa-,science,337(6096):816-821(2012),但是但是同样可以使用许多其他序列,包括更长的序列。在一些实施方案中,接头序列包含功能部分。例如,接头序列可以包含适体、核糖酶、蛋白质相互作用发夹结构、crispr阵列、内含子以及外显子。在一些实施方案中,接头序列包含至少约:1、2、3、4或5或更多个功能性部分。在一些实施方案中,接头序列包含至多约:1、2、3、4或5或更多个功能性部分。核酸靶向核酸与定点多肽的复合物核酸靶向核酸与定点多肽(例如核酸向导的核酸酶,诸如cas9)相互作用,从而形成复合物。核酸靶向核酸将定点多肽引导至靶核酸。密码子优化在一些实施方案中,根据本领域中用于在含有所关注的靶dna的细胞中表达的方法标准对编码定点多肽的多核苷酸进行密码子优化。例如,如果预期的靶核酸在人细胞中,则考虑将编码cas9的人密码子优化的多核苷酸用于产生cas9多肽。rnp定点多肽和靶向基因组的核酸可以各自单独施用于细胞或患者。另一方面,定点多肽可以与一种或更多种向导rna或一种或更多种crrna以及tracrrna一起预复合。然后可以将预复合的材料施用于细胞或患者。这种预复合的材料被称为核糖核蛋白颗粒(rnp)。编码核酸的系统组分在另一方面,本公开提供了包含编码本公开的核酸靶向核酸的核苷酸序列的核酸、本公开的定点多肽和/或进行本公开的方法的实施方案所必需的任何核酸或蛋白质分子。在一些实施方案中,编码本公开的核酸靶向核酸的核酸、本公开的定点多肽和/或进行本公开的方法的实施方案所必需的任何核酸或蛋白质分子构成了载体(例如,重组表达载体)。如本文所用,术语“载体”是指一种核酸分子。这种核酸分子能够转运其已经连接至的另一种核酸。一种类型的载体是“质粒”,质粒是指环状双链dna环,附加的核酸区段可以连接到所述环状双链dna环中。另一种类型的载体是病毒载体,其中附加的核酸区段可以连接到病毒基因组中。某些载体能够在它们被引入到的宿主细胞中自主复制(例如,具有细菌复制起点和附加型哺乳动物载体的细菌载体)。其他载体(例如非附加型哺乳动物载体)在引入到宿主细胞时整合到宿主细胞的基因组中,从而与宿主基因组一起复制。在一些实施方案中,载体能够向导它们被操作性地连接到基因的表达。此类载体在本文中被称为“重组表达载体”,或更简单地称为“表达载体”,其起到等效功能。术语“操作性地连接”在本文中旨在意味着所关注的核苷酸序列以允许核苷酸序列表达的方式与调控序列连接。术语“调控序列”旨在包括例如启动子、增强子和其他表达控制元件(例如聚腺苷酸化信号)。此类调控序列在本领域中是众所周知的,并且例如描述于goeddel;geneexpressiontechnology:methodsinenzymology185,academicpress,sandiego,ca(1990)中。调控序列包括在许多类型的宿主细胞中向导核苷酸序列的组成型表达的那些序列,以及仅在某些宿主细胞中直接表达核苷酸序列的那些序列(例如组织特异性调控序列)。本领域的技术人员将理解,表达载体的设计可以取决于诸如靶细胞选择、期望的表达水平等因素。所考虑的表达载体包括但不限于基于牛痘病毒、脊髓灰质炎病毒、腺病毒、腺相关病毒、sv40、单纯疱疹病毒、人免疫缺陷病毒、逆转录病毒(例如,鼠白血病病毒,脾坏死病毒,以及衍生自逆转录病毒的载体如劳斯氏肉瘤病毒、哈维肉瘤病毒、禽白血病病毒、慢病毒、人类免疫缺陷病毒,骨髓增殖性肉瘤病毒和乳腺肿瘤病毒)的病毒载体,以及其他重组载体。设想用于真核靶细胞的其他载体包括但不限于载体pxt1、psg5、psvk3、pbpv、pmsg和psvlsv40(pharmacia)。还可以使用其他载体,只要它们与宿主细胞相容即可。在一些实施方案中,载体包含一个或更多个转录和/或翻译控制元件。根据所使用的宿主/载体系统,许多合适的转录和翻译控制元件中的任一者,包括组成型和诱导型启动子、转录增强子元件、转录终止子等,可以用于表达载体中。合适的真核启动子(即在真核细胞中起作用的启动子)的非限制性示例包括来自以下的那些启动子:巨细胞病毒(cmv)立早基因、单纯疱疹病毒(hsv)胸苷激酶、早期和晚期sv40、来自逆转录病毒的长末端重复序列(ltr)、人延伸因子-1启动子(ef1)、包含融合到鸡β-肌动蛋白启动子(cag)的巨细胞病毒(cmv)增强子的混合构建体、鼠干细胞病毒启动子(mscv)、磷酸甘油酸激酶-1位点启动子(pgk)和小鼠金属硫蛋白-i。为了表达小rna(包括与cas核酸内切酶结合使用的向导rna),多种启动子如rna聚合酶iii启动子,包括例如u6和h1,可以是有利的。用于增强这种启动子的使用的描述和参数在本领域中是已知的,并且经常描述附加信息和方法;参见例如ma,h.等人,moleculartherapy-nucleicacids3,e161(2014)doi:10.1038/mtna.2014.12。表达载体还可以含有用于翻译起始的核糖体结合位点和转录终止子。表达载体还可以包括用于扩增表达的适当序列。表达载体还可以包括编码非天然标签(例如,组氨酸标签、血凝素标签、绿色荧光蛋白等)的核苷酸序列,所述非天然标签与定点多肽融合,从而产生融合蛋白。在一些实施方案中,启动子是诱导型启动子(例如热休克启动子、四环素调控的启动子、类固醇调控的启动子、金属调控的启动子、雌激素受体调控的启动子等)。在一些实施方案中,启动子是组成型启动子(例如,cmv启动子、ubc启动子)。在一些实施方案中,启动子是空间上受限和/或时间上受限的启动子(例如,组织特异性启动子、细胞类型特异性启动子等)。在一些实施方案中,编码公开的核酸靶向核酸的核酸和/或定点多肽被包装到递送载体内或其表面上以递送到细胞中。所设想的递送载体包括但不限于纳米球、脂质体、量子点、纳米颗粒、聚乙二醇颗粒、水凝胶和胶束。如本领域所述,可以使用多种靶向部分来增强此类载体与所需细胞类型或位置的优先相互作用。将本公开的复合物、多肽和核酸引入细胞可以通过以下方式进行:病毒或噬菌体感染、转染、缀合、原生质体融合、脂质转染、电穿孔、核转染、磷酸钙沉淀、聚乙烯亚胺(pei)介导的转染、deae-葡聚糖介导的转染、脂质体介导的转染、颗粒枪技术、磷酸钙沉淀、直接微注射、纳米颗粒介导的核酸递送等。递送向导rna多核苷酸(rna或dna)和/或一种或更多种核酸内切酶多核苷酸(rna或dna)可以通过在本领域中已知的病毒或非病毒递送载体递送。或者,一种或更多种核酸内切酶多肽可以通过本领域中已知的病毒或非病毒递送载体(例如电穿孔或脂质纳米颗粒)递送。在另外的替代方面,dna核酸内切酶可以作为一个或更多个多肽而单独地递送或与一个或更多个向导rna或一个或更多个crrna与tracrrna一起预复合地递送。多核苷酸可以通过非病毒递送载体递送,非病毒递送载体包括但不限于纳米颗粒、脂质体、核糖核蛋白、带正电荷的肽、小分子rna缀合物、适体-rna嵌合体以及rna-融合蛋白复合物。一些示例性的非病毒递送载体描述于peer和lieberman,genetherapy,18:1127–1133(2011)(其主要讲述sirna的非病毒递送载体,所述非病毒递送载体还可用于递送其他多核苷酸)。中.多核苷酸,例如向导rna、sgrna和编码核酸内切酶的mrna,可以由脂质纳米颗粒(lnp)递送到细胞或患者中。lnp是指直径小于1000nm、500nm、250nm、200nm、150nm、100nm、75nm、50nm或25nm的任何颗粒。或者,纳米颗粒的大小可以在1-1000nm、1-500nm、1-250nm、25-200nm、25-100nm、35-75nm或25-60nm的范围内。lnp可以由阳离子脂质、阴离子脂质或中性脂质制成。中性脂质,例如促融合磷脂dope或膜组分胆固醇,可以包括在lnp中作为“辅助脂质”,以增强转染活性和纳米颗粒稳定性。阳离子脂质的限制包括低效力,这是由于不良稳定性和快速清除,以及炎症或抗炎反应的产生。lnp还可以由疏水性脂质、亲水性脂质、或疏水性和亲水性脂质两者组成。本领域已知的任何脂质或脂质组合可用来产生lnp。用来产生lnp的脂质的示例是:dotma、dospa、dotap、dmrie、dc-胆固醇、dotap–胆固醇、gap-dmorie–dpype以及gl67a–dope–dmpe–聚乙二醇(peg)。阳离子脂质的示例是:98n12-5、c12-200、dlin-kc2-dma(kc2)、dlin-mc3-dma(mc3)、xtc、md1和7c1。中性脂质的示例是:dpsc、dppc、popc、dope和sm。peg修饰的脂质的示例是:peg-dmg、peg-cerc14和peg-cerc20。脂质可以以任何数量的摩尔比组合以产生lnp。此外,一种或更多种多核苷酸可以以宽范围的摩尔比与一种或更多种脂质组合以产生lnp。如前所述,定点多肽和基因组靶向核酸可以各自单独施用于细胞或患者。另一方面,定点多肽可以与一种或更多种向导rna或一种或更多种crrna以及tracrrna一起预复合。然后可以将预复合的材料施用于细胞或患者。这种预复合的材料被称为核糖核蛋白颗粒(rnp)。rna能够与rna或dna形成特异性相互作用。虽然在许多生物过程中利用这种性质,但这种性质也具有在富含核酸的细胞环境中发生混杂相互作用的风险。这个问题的一个解决方案是形成核糖核蛋白颗粒(rnp),其中rna与核酸内切酶预复合。rnp的另一个益处是保护rna免于降解。rnp中的核酸内切酶可以经修饰或未修饰。同样,grna、crrna、tracrrna或sgrna可以经修饰或未修饰。许多修改是本领域已知的并且可以被使用。核酸内切酶和sgrna一般可以以1:1的摩尔比混合。或者,核酸内切酶、crrna和tracrrna可以通常以1:1:1的摩尔比混合。然而,宽范围的摩尔比可用于产生rnp。重组腺相关病毒(aav)载体可用于递送。产生raav颗粒的技术是在本领域中标准的,其中待包装的aav基因组包含待递送的多核苷酸rep和cap基因并且辅助病毒功能被提供给细胞。raav的产生通常需要在单个细胞内(本文表示为包装细胞)存在以下组分:raav基因组、从raav基因组分离(即不在raav基因组中)的aavrep和cap基因、以及辅助病毒功能。aavrep和cap基因可以来自可以衍生重组病毒的任何aav血清型,并且可以来自与raav基因组itr不同的aav血清型,包括但不限于aav血清型aav-1、aav-2、aav-3、aav-4、aav-5、aav-6、aav-7、aav-8、aav-9、aav-10、aav-11、aav-12、aav-13和aavrh.74。假型raav的产生在例如国际专利申请公开号wo01/83692中公开。参见表2。表2产生包装细胞的方法涉及产生稳定地表达aav颗粒产生的所有必需组分的细胞系。例如,包含没有aavrep和cap基因的raav基因组的一种质粒(或多种质粒)、从raav基因组分离的aavrep和cap基因、以及选择标记如新霉素抗性基因,被整合到细胞的基因组中。已经通过诸如gc加尾(samulski等人,1982,proc.natl.acad.s6.usa,79:2077-2081)、添加含有限制性核酸内切酶切割位点的合成接头(laughlin等人,1983,gene,23:65-73)或通过直接的平末端连接(senapathy和carter,1984,j.biol.chem.,259:4661-4666)的方法将aav基因组引入到细菌质粒中。然后可以用辅助病毒(例如腺病毒)感染包装细胞系。此方法的优点是细胞是可选择的并且适合于raav的大规模生产。合适方法的其他示例使用腺病毒或杆状病毒而不是质粒,来将raav基因组和/或rep和cap基因引入包装细胞中。raav生产的一般原则综述于例如carter,1992,currentopinionsinbiotechnology,1533-539;以及muzyczka,1992,curr.topicsinmicrobial.andimmunol.,158:97-129)中。多种方法描述于ratschin等人,mol.cell.biol.4:2072(1984);hermonat等人,proc.natl.acad.sci.usa,81:6466(1984);tratschin等人,mo1.cell.biol.5:3251(1985);mclaughlin等人,j.virol.,62:1963(1988);以及lebkowski等人,1988mol.cell.biol.,7:349(1988).samulski等人,(1989,j.virol.,63:3822-3828);美国专利no.5,173,414;wo95/13365和相应的美国专利no.5,658.776;wo95/13392;wo96/17947;pct/us98/18600;wo97/09441(pct/us96/14423);wo97/08298(pct/us96/13872);wo97/21825(pct/us96/20777);wo97/06243(pct/fr96/01064);wo99/11764;perrin等人,(1995)vaccine13:1244-1250;paul等人,(1993)humangenetherapy4:609-615;clark等人,(1996)genetherapy3:1124-1132;美国专利no.5,786,211;美国专利no.5,871,982;以及美国专利no.6,258,595中。aav载体血清型可以与靶细胞类型匹配。例如,以下示例性细胞类型可以由所示的aav血清型等转导。参见表3。表3组织/细胞类型血清型肝脏aav8、aav9骨骼肌aav1、aav7、aav6、aav8、aav9中枢神经系统aav5、aav1、aav4rpeaav5、aav4感光细胞aav5肺aav9心脏aav8胰腺aav8肾aav2除了腺相关病毒载体以外,还可以使用其他病毒载体。这种病毒载体包括但不限于慢病毒、甲病毒、肠病毒、瘟病毒、杆状病毒、疱疹病毒、爱泼斯坦巴尔病毒、乳多空病毒(papovavirusr)、痘病毒、牛痘病毒和单纯疱疹病毒。在一些情况下,cas9mrna、靶向il7r基因中的一个或两个位点的sgrna和供体dna可以被各自单独配制到脂质纳米颗粒中,或者全部共配制到一个脂质纳米颗粒中。在一些情况下,cas9mrna可以在脂质纳米颗粒中配制,而sgrna和供体dna可以在aav载体中递送。可用的选项为将cas9核酸酶作为dna质粒、作为mrna或作为蛋白质来递送。向导rna可以从相同的dna表达,或者也可以作为rna递送。rna可以经化学修饰以改变或改善其半衰期,或降低免疫应答的可能性或程度。核酸内切酶蛋白可以在递送之前与grna复合。病毒载体允许有效递送;cas9的分裂形式和cas9的较小直向同源物可以包装在aav中,如hdr的供体可以的一样。还存在一系列可以递送这些组分中的每一种的非病毒递送方法,或者非病毒和病毒方法可以级联使用。例如,纳米颗粒可用于递送蛋白质和向导rna,而aav可用于递送供体dna。基因组编辑的ipsc分化为造血祖细胞或白血细胞本公开的离体方法的另一步骤可以包括将基因组编辑的ipsc分化成造血祖细胞或白血细胞。可以根据在本领域中已知的任何方法进行分化步骤。将基因组编辑的间充质干细胞分化成造血祖细胞或白血细胞本公开的离体方法的另一步骤可以包括将基因组编辑的间充质干细胞分化成造血祖细胞或白血细胞。可以根据在本领域中已知的任何方法进行分化步骤。将细胞植入患者本公开的离体方法的另一步骤可以包括将细胞植入患者体内。这种植入步骤可以使用在本领域中已知的任何植入方法来实现。例如,可以将遗传修饰的细胞直接注射到患者的血液中或以其他方式施用于患者。可以使用选择的标记物离体纯化遗传修饰的细胞。试剂盒本公开提供了用于执行本公开的方法的试剂盒。试剂盒可以包括以下项中的一者或多者:本公开的核酸靶向核酸、编码核酸靶向核酸的多核苷酸、本公开的定点多肽、编码进行本公开的方法的各实施方案所必需的定点多肽和/或任何核酸或蛋白质分子的多核苷酸,或其任何组合。在一些实施方案中,试剂盒包含:(1)包含编码核酸靶向核酸的核苷酸序列的载体,以及(2)包含编码定点多肽的核苷酸序列的载体,以及(3)用于重构和/或稀释载体的试剂。在一些实施方案中,试剂盒包含:(1)包含(i)编码核酸靶向核酸的核苷酸序列和(ii)编码定点多肽的核苷酸序列的载体,以及(2)用于重构和/或稀释载体的试剂。在任何上述试剂盒的一些实施方案中,试剂盒包含单分子向导核酸靶向核酸。在任何上述试剂盒的一些实施方案中,试剂盒包含双分子核酸靶向核酸。在任何上述试剂盒的一些实施方案中,试剂盒包含两个或更多个双分子向导物或单分子向导物。在一些实施方案中,试剂盒包含可以编码核酸靶向核酸的载体。在任何上述试剂盒的一些实施方案中,试剂盒可以进一步包含要插入以实现所需遗传修饰的多核苷酸。试剂盒的各组分可以在单独的容器中;或组合在单个容器中。在一些实施方案中,上文所述的试剂盒还包含一种或更多种附加试剂,其中此类附加试剂选自:缓冲液、用于将试剂盒的多肽或多核苷酸物项引入细胞的缓冲液、洗涤缓冲液、对照试剂、对照载体、对照rna多核苷酸、用于从dna体外产生多肽的试剂、用于测序的接头(adaptor)等。缓冲液可以是稳定缓冲液、重构缓冲液或稀释缓冲液等。除了上述组分之外,试剂盒还可以进一步包括关于使用试剂盒的组分来实践方法的说明书。关于实践方法的说明书通常记录在合适的记录介质上。例如,说明书可以打印在如纸或塑料等基材上。说明书可以作为包装插页存在于试剂盒中,试剂盒的容器或试剂盒的各组分的标签中(即与包装或亚包装相关)。说明书可以作为存在于合适的计算机可读存储介质(例如cd-rom、磁盘、闪存盘等)上的电子存储数据文件来呈现。在一些情况下,实际说明书不存在于试剂盒中,而是可以提供用于(例如,通过互联网)从远程源获得说明书的手段。此实施方案的示例是包括可以查看说明书和/或可以从其下载说明书的网址的试剂盒。与说明书一样,用于获得说明书的此手段可以记录在合适的基材上。向导rna制剂用药物可接受的赋形剂如载体、溶剂、稳定剂、佐剂、稀释剂等配制本发明的向导rna,这取决于特定的施用模式和剂型。通常将向导rna组合物配制成实现生理相容的ph,并且在从约ph3至约ph11、约ph3至约ph7的范围内,具体取决于配方和施用途径。在替代实施方案中,将ph调节至约ph5.0至约ph8的范围。在一些实施方案中,组合物包含治疗有效量的至少一种如本文所述的化合物,以及一种或更多种药学上可接受的赋形剂。任选地,组合物包含本文所述的各化合物的组合,或者可以包括可用于治疗或预防细菌生长的第二活性成分(例如而不限于抗细菌或抗微生物剂),或者可以包括本发明的各试剂的组合。合适的赋形剂包括例如包括大的、缓慢代谢的大分子的载体分子,所述大的、缓慢代谢的大分子为诸如蛋白质、多糖、聚乳酸、聚乙醇酸、聚氨基酸、氨基酸共聚物和无活性病毒颗粒。其他示例性赋形剂包括抗氧化剂(例如而不限于抗坏血酸)、螯合剂(例如而不限于edta)、碳水化合物(例如而不限于糊精、羟基烷基纤维素和羟基烷基甲基纤维素)、硬脂酸、液体(例如而不限于油、水、盐水、甘油和乙醇)、润湿或乳化剂、ph缓冲物质等。遗传修饰的细胞如本文所用,术语“遗传修饰的细胞”是指包含通过基因组编辑(例如,使用crispr/cas系统)引入的至少一种遗传修饰的细胞。在本文的一些实施方案中,遗传修饰的细胞是遗传修饰的祖细胞。本文考虑了包含外源核酸靶向核酸和/或编码核酸靶向核酸的外源核酸的遗传修饰的细胞。关于对γ-球蛋白表达进行去抑制,短语“增加细胞中的γ-球蛋白水平”或“细胞中增加的γ-球蛋白表达”表明细胞或细胞群中的γ-球蛋白在经受基因组编辑的细胞或细胞群中比在没有基因组编辑的可比较的对照群组中高至少2%。在一些实施方案中,相较于可比较的对照处理的群体,γ-球蛋白表达的增加为至少约2%、至少约3%、至少约4%、至少约5%、至少约6%、至少约7%、至少约8%、至少约9%、至少约10%、至少约11%、至少约12%、至少约13%、至少约14%、至少约15%、至少约16%、至少约17%、至少约18%、至少约19%、至少约20%、至少约25%、至少约30%、至少约35%、至少约40%、至少约45%、至少约50%、至少约55%、至少约60%、至少约65%、至少约70%、至少约75%、至少约80%、至少约85%、至少约90%、至少约95%、至少约99%、至少约2倍、至少约3倍、至少约4倍、至少约5倍、至少约6倍、至少约7倍、至少约8倍、至少约9倍、至少约10倍、至少约15倍、至少约20倍、至少约25倍、至少约30倍、至少约35倍、至少约40倍、至少约45倍、至少约50倍、至少约100倍或更多倍。术语“对照处理的群体”在本文中用于描述已经用相同培养基、病毒诱导、核酸序列、温度、汇合度、烧瓶大小、ph等处理的细胞群,不同之处在于添加基因组编辑组分。本领域已知的任何方法可用于测量γ-球蛋白表达的增加,例如对γ-球蛋白进行蛋白质印迹分析或定量γ-球蛋白mrna。如本文所用的术语“分离的细胞”是指已经从其最初存在的生物体取出的细胞,或这种细胞的后代。任选地,细胞已经在体外培养,例如在限定的条件下或在其他细胞存在下培养。任选地,随后将细胞引入第二生物体中或重新引入所述细胞所分离自的生物体(或所述细胞从其传代的生物体)。关于如本文所用的分离的细胞群的术语“分离的群体”是指已经从混合或异质细胞群中取出和分离的细胞群。在一些实施方案中,与细胞所分离或富集自的异质群体相比,分离的群体是基本上纯的细胞群。在一些实施方案中,分离的群体是人类造血祖细胞的分离群体,例如与包含人造血祖细胞和人造血祖细胞所衍生自的细胞的异质细胞群相比,人类造血祖细胞的基本上纯的群体。关于特定细胞群体,术语“显著增强的”是指其中特定类型的细胞的发生率相对于预先存在的或参考水平增加至少2倍、至少3倍、至少4倍、至少5倍、至少6倍、至少7倍、至少8倍、至少9倍、至少10倍、至少20倍、至少50倍、至少100倍、至少400倍、至少1000倍、至少5000倍、至少20000倍、至少100000倍或更多倍的细胞群,具体取决于例如减轻血红蛋白病所需的这种细胞的水平。关于特定细胞群的术语“基本上富集的”是指相对于构成总细胞群的细胞为至少:约10%、约20%、约30%、约40%、约50%、约60%、约70%或更多的细胞群。关于特定细胞群体的术语“基本上富集”或“基本上纯的”是指相对于构成总细胞群的细胞,为至少约75%、至少约85%、至少约90%或至少约95%纯的细胞群。也就是说,关于造血祖细胞群的术语“基本上纯的”或“基本上纯化的”是指含有少于:约20%、约15%、约10%、约9%、约8%、约7%、约6%、约5%、约4%、约3%、约2%、约1%或小于1%的细胞如由本文的术语所定义的造血祖细胞的细胞群体。药学上可接受的载体将祖细胞施用于本文所述的受试者的方法涉及使用包含祖细胞的治疗组合物。治疗组合物含有生理上可耐受的载体以及细胞组合物,以及任选地如本文所述的至少一种附加的生物活性剂,所述生物活性剂溶解或分散在所述治疗组合物中以作为活性成分。在一些实施方案中,除非需要,否则当施用于哺乳动物或人类患者以用于治疗目的时,治疗组合物基本上不具免疫原性。通常,本文所述的祖细胞作为具有药学上可接受的载体的悬浮液施用。本领域的技术人员将认识到,用于细胞组合物中的药学上可接受的载体将不包括用量会实质上干扰要递送至受试者体内的细胞的活力的缓冲剂、化合物、抗冻剂、防腐剂或其他试剂。包含细胞的制剂可以包括例如允许维持细胞膜完整性的渗透缓冲液,以及任选地用以维持细胞活力或增强施用后的植入的营养物。此类制剂和悬浮液是本领域的技术人员已知的和/或可以适用于使用常规实验与如本文所述的祖细胞一起使用。细胞组合物也可以被乳化或呈现为脂质体组合物,条件是乳化程序不会不利地影响细胞活力。细胞和任何其他活性成分可以与赋形剂混合,所述赋形剂是在药学上可接受的并且与活性成分相容,并且其量适用于本文所述的治疗方法。包括在如本文所述的细胞组合物中的附加试剂可以在其中包含多种组分的药学上可接受的盐。药学上可接受的盐包括使用无机酸(如例如盐酸或磷酸)或有机酸(诸如乙酸、酒石酸、扁桃酸等)形成的酸加成盐(与多肽的游离氨基一起形成的)。与游离羧基形成的盐也可以衍生自无机碱(例如氢氧化钠、氢氧化钾、氢氧化铵、氢氧化钙或氢氧化铁)和有机碱(诸如异丙胺、三甲胺、2-乙基氨基乙醇、组氨酸、普鲁卡因等)。生理上可耐受的载体是本领域中熟知的。示例性液体载体是不含除了活性成分和水之外的材料的无菌水溶液,或含有缓冲液如处于生理ph值的磷酸钠、生理盐水或这两者(例如磷酸盐缓冲盐水)。此外,水性载体可以含有多于一种缓冲盐,以及如氯化钠和氯化钾的盐、葡萄糖、聚乙二醇和其他溶质。液体组合物还可以含有作为水的补充或者排除水的液相。这种附加液相的范例是甘油、植物油(例如棉籽油)、以及水油乳液。在本文所述的细胞组合物中使用的活性化合物的有效治疗特定疾病或病症的量将取决于所述疾病或病症的性质,并且可以通过标准临床技术确定。施用和功效如本文所用,术语“施用”、“引入”和“移植”在通过导致所引入的细胞至少部分地定位在所需位点(例如损伤或修复位点)处的方法或途径将细胞(例如如本文所述的祖细胞)放置到受试者体内以产生一种或更多种期望效果的情境中可互换使用。细胞如祖细胞或其分化的子代可以通过导致递送至受试者体内所需位置处的任何适当的途径施用,在所述位置处植入的细胞或细胞组分中的至少一部分保持有活力。在施用于受试者后细胞具活力的时期可以短至几小时(例如二十四小时)至几天,长达几年(即长期植入)。例如,在本文所述方面的一些实施方案中,经由全身施用途径,例如腹膜内或静脉内途径施用有效量的造血祖细胞。术语“个体”、“受试者”、“宿主”和“患者”在本文中可互换使用,并且是指需要诊断、治疗或疗法的任何受试者。在一些实施方案中,受试者是哺乳动物。在一些实施方案中,受试者是人类。当被预防性地提供时,可以将本文所述的祖细胞在血红蛋白病的任何症状之前施用于受试者,例如在从胎γ-球蛋白到主要β-球蛋白的转换开始之前和/或在严重贫血或与血红蛋白病有关的其他症状发生之前。因此,造血祖细胞群的预防性施用用于预防血红蛋白病,如本文所公开的。当被治疗地提供时,在血红蛋白病的症状或指征发作时(或之后)提供造血祖细胞,例如在镰状细胞性贫血或其他scd发作时。在本文所述的方面的一些实施方案中,根据本文所述的方法施用的造血祖细胞群包括从一个或更多个供体获得的同种异体造血祖细胞。如本文所用,“同种异体”是指包含从同一物种的一个或更多个不同供体获得的造血祖细胞的造血祖细胞或生物样品,其中所述一个或更多个不同供体的一个或更多个位点处的基因不同。例如,施用于受试者的造血祖细胞群可以来源于从一个或更多个不相关的供体受试者或从一个或更多个不相同的同胞获得的脐带血。在一些实施方案中,可以使用同基因的造血祖细胞群,例如从遗传上相同的动物或从相同双胞胎获得的那些。在这方面的其他实施方案中,造血祖细胞是自体细胞;即,造血祖细胞是从受试者获得或分离,并且施用于相同的受试者,即供体和受体是相同的。在一个实施方案中,如本文所用的术语“有效量”是指预防或减轻血红蛋白病的至少一种或更多种病征或症状所需的祖细胞群或其子代的量,并且涉及足够量的组合物以提供期望的效果,例如治疗患有血红蛋白病的受试者。因此,术语“治疗有效量”是指当施用于典型受试者(例如患有血红蛋白病或处于血红蛋白病风险下的受试者)时足以促进特定效应的祖细胞或包含祖细胞的组合物的量。如本文所用的有效量还将包括足以预防或延迟疾病症状的发展、改变疾病症状的进程(例如但不限于减缓疾病症状的进展)或逆转疾病症状的量。应当理解,对于任何给定的情况,可以由本领域的普通技术人员使用常规实验来确定适当的“有效量”。为了在本文所述的多个方面中使用,有效量的祖细胞包括至少102个祖细胞、至少5×102个祖细胞、至少103个祖细胞、至少5×103个祖细胞、至少104个祖细胞、至少5×104个祖细胞、至少105个祖细胞、至少2×105个祖细胞、至少3×105个祖细胞、至少4×105个祖细胞、至少5×105个祖细胞、至少6×105个祖细胞、至少7×105个祖细胞、至少8×105个祖细胞、至少9×105个祖细胞、至少1×106个祖细胞、至少2×106个祖细胞、至少3×106个祖细胞、至少4×106个祖细胞、至少5×106个祖细胞、至少6×106个祖细胞、至少7×106个祖细胞、至少8×106个祖细胞、至少9×106个祖细胞、或其倍数。祖细胞来源于一个或更多个供体,或从自体来源获得。在本文所述的各方面的一些实施方案中,将祖细胞在施用于需要其的受试者前在培养物中扩增。如上所述,即使是具有血红蛋白病的患者的细胞中表达的hbf水平的适度和增量增加也可以有益于减轻疾病的一种或更多种症状、增加长期存活和/或减少与其他治疗相关的副作用。在向人类患者施用此类细胞时,产生增加的hbf水平的rbc的存在是有益的。在一些实施方案中,对受试者的有效治疗相对于所治疗受试者体内的总hb产生至少约9%的hbf。在一些实施方案中,hbf将为总hb的至少约14%。在一些实施方案中,hbf将为总hb的至少约20%至30%。类似地,引入即使相对有限的具有显著升高的hbf水平的细胞(称为“f细胞”)的亚群在多种患者中也可是有益的,因为在某些情况下,正常化细胞将相对于患病细胞具有选择性优势。然而,即使是适度水平的具有升高的hbf水平的循环rbc也可有益于减轻患者的血红蛋白病的一个或更多个方面。在一些实施方案中,在此类细胞所施用于的患者体内的约10%、约20%、约30%、约40%、约50%、约60%、约70%、约80%、约90%或更多的rbc不断产生增加水平的hbf,如本文所述。如本文所用,“施用”是指通过导致将细胞组合物至少部分定位在所需位点处的方法或途径将如本文所述的祖细胞组合物递送到受试者中。细胞组合物可以通过任何导致对受试者进行有效治疗的适当途径施用,即施用导致递送至受试者体内的所需位置,其中所述组合物中的至少一部分被递送到所述位置,即至少1×104个细胞在一段时间内被递送到所需的位点。施用模式包括注射、输注、滴注或摄取。“注射”包括但不限于静脉内、肌内、动脉内、鞘内、心室内、囊内、眶内、心内、皮内、腹膜内、经气管、皮下、表皮下、关节内、被膜下、蛛网膜下、脊柱内、脑内脊柱和胸骨内注射和输注。在一些实施方案中,途径是静脉内的。对于细胞的递送,通常优选通过注射或输注施用。在一个实施方案中,本文所述的细胞被全身地施用。如本文所用的短语“全身施用”、“全身地施用”、“外周施用”和“外周地施用”是指施用祖细胞群不是使祖细胞群直接进入靶位点、组织或器官,而是使其进入受试者的循环系统以及因此经受新陈代谢和其他类似过程。包含如本文所述的用于治疗血红蛋白病的组合物的治疗的功效可以由熟练的临床医生确定。然而,如该术语在本文中被使用的,如果任何一种或所有的病症或症状(作为仅一个示例如胎血红蛋白的水平)以有益的方式改变(例如,增加至少10%),另一些临床上认可的疾病症状或标志物被改善或减轻,则治疗被认为是“有效治疗”。还可以通过个体没有恶化来测量功效,如通过没有住院或需要医疗干预所评估的(例如,减少的输血依赖性或疾病的进展停止或至少减缓)。测量这些指标的方法是本领域的技术人员已知和/或在此描述的。治疗包括对个体或动物(一些非限制性示例包括人或哺乳动物)的疾病的任何治疗,并且包括:(1)抑制疾病,例如阻止或减缓症状的进展;或(2)缓解疾病,例如引起症状消退;以及(3)预防或减少症状发生的可能性。根据本发明的治疗通过增加个体中胎血红蛋白的量来减轻与β-血红蛋白病相关的一种或更多种症状。通常与血红蛋白病相关的症状和病征包括例如贫血、组织缺氧、器官功能障碍、异常血细胞比容值、低效红细胞生成、异常网织红细胞(红血球)计数、异常铁负荷、环形铁粒幼红细胞的存在、脾肿大、肝肿大、外周血流受损、呼吸困难、溶血增加、黄疸、贫血痛危象(anemicpaincrise)、急性胸部综合征、脾隔离症、异常勃起、中风、手足综合征和疼痛如心绞痛。定义如本文所用,术语“包括”或“包含”用于提及本发明所必需的组合物、方法及其的一种或更多种相应组分,但是为开放的以包括未指定的要素,无论是否必需。如本文所用,术语“基本上由……组成”是指给定实施方案所需的那些要素。该术语允许存在不会实质上影响本发明的实施方案的一个或更多个基本和新颖或功能特征的附加要素。术语“由……组成”是指如本文所述的组合物、方法及其的一种或更多种相应组分,其排除未在实施方案的描述中叙述的任何要素。如在本说明书和所附权利要求书中所用,不使用数量词修饰时包括多个指代物,除非上下文另外明确指出。本文呈现的某些数值前置有术语“约”。术语“约”在本文中用于为术语“约”之前的数值提供文字支持,以及为近似于该数值的数值提供文字支持,即近似的未记载的数值可以是在呈现其的上下文中基本上等同于明确记载的数值的的数值。当在此给出一系列数值时,考虑该范围的下限和上限之间的每个中间值、为范围的上限和下限的值、以及使用该范围的所有值都被涵盖在本公开中。该范围的下限和上限内的所有可能的子范围也由本公开考虑。实施例通过参考以下提供本发明的说明性非限制性实施方案的实施例将更充分地理解本发明。这些实施例描述了使用crispr/cas体系作为说明性基因组编辑技术,以在β-球蛋白基因簇中产生确定的治疗基因组缺失或单碱基取代,本文统称为“基因组修饰”,其上调hbf的表达。示例性治疗修饰在遗传和/或功能上与在具有血红蛋白病(例如镰状细胞或β-地中海贫血)的个体的造血细胞中观察到的修饰相似或相同,其中修饰导致γ球蛋白以及因此产生的胎血红蛋白的去抑制或引起其重新表达。所限定的治疗性修饰的引入代表了如本文所述和说明的对血红蛋白病潜在改善的新颖治疗策略。实施例1产生靠近于chr11:5224779-5237723的缺失在该实施例中,我们说明了使用本文所述方法以产生靠近于区域chr11:5224779-5237723的一些缺失。在被命名为hpfh-5(或“hpfh西西里”)(在camaschella等人,haematologica,75(suppl5):26-30(1990)中描述)的人类患者中观察到该区域中的缺失。在人患者中观察到的人β-球蛋白位点中的13kb缺失变体与遗传性胎儿血红蛋白持续症(hpfh)的临床表型相关联,其中胎血红蛋白的存在可补充成体血红蛋白合成或功能的缺陷,并改善镰状细胞性贫血或β-地中海贫血中的疾病。在该实施例中,我们说明crispr/cas体系可以用于产生功能上类似于与天然hpfh等位基因(例如hpfh-5)相关联的那些缺失的缺失。向导rna被设计为通过使δ和β球蛋白基因以及γ球蛋白基因3'区域的大部分缺失来消除病原性镰状细胞等位基因。图1a示出了具有突出hpfh-55'和3'靶位点的空心框的人球蛋白位点。13kb缺失起始于ψβ1基因的5'端的3kb处,并结束于β基因末端的3'端的1.7kb处(从β基因聚a信号下游690bp处)。参考图1b。此外,向导rna被设计成靶向贯穿13kb区域内的位点以便确定该位点内较小缺失的治疗潜力。实验方法靶位点的选择针对靶位点扫描β-球蛋白基因簇的区域,其包括与遗传性胎儿血红蛋白持续症-5(hpfh-5)相关的5'和3'区域。扫描每个区域的序列为ngg和/或nrg的前间区序列邻近基序(pam)。鉴定与pam相对应的向导链。对于该说明性示例,在涉及理论结合和实验评估活性的多步骤过程中筛选和选择候选向导。通过说明的方式,具有将特定在靶位点与相邻pam匹配的序列的候选向导可以使用可用于评估脱靶结合的多种生物信息学工具中的一种或更多种来评估其在具有相似序列的脱靶位点处切割的潜力,如下文更详细描述和说明的,以便评估在预期位置以外的染色体位置处的作用的可能性。然后可以通过实验来评估预期具有相对较低的脱靶活性可能性的候选者,以测量其在各个位点处的在靶活性,然后是脱靶活性。优选的向导具有足够高的在靶活性以在所选择的位置达到所期望水平的基因编辑,以及相对较低的脱靶活性,以降低在其它染色体位点处发生改变的可能性。在靶与脱靶活性的比例通常被称为向导的“特异性”。对于预测的脱靶活性的初步筛选,存在许多可用于预测最可能的脱靶位点的已知和可公开获得的生物信息学工具;并且由于与crisprcas9核酸酶系统中的靶位点的结合是由互补序列之间的watson-crick碱基配对驱动的,所以不相似程度(以及因此脱靶结合的可能性降低)基本上与一级序列差异有关:不匹配和凸起,即变为非互补碱基的碱基,以及相对于靶位点在潜在的脱靶位点插入或缺失碱基。称为cosmid(具有不匹配、插入和缺失的crispr脱靶位点)的示例性生物信息学工具(可从网页crispr.bme.gatech.edu获得)汇编了这样的相似之处。获得了选择用于细胞中的特异性向导性rna间隔序列的以下生物信息学输出概述。图1c和图1d中示出了向导rna靶位点相对于用于缺失的5’和3’靶区域的位置。crispr克隆使用表达来自化脓性链球菌的人源化cas9和单分子向导rna的载体组装表达cas9蛋白质和向导链rna的质粒。获得与向导链相对应的互补寡核苷酸(operon或idt),激酶化,退火并克隆到载体中。在细胞中测试包含以下间隔序列的向导rna:hpfh5-4:5’-gctgagttctaaaatcatcg-3’(seqidno:4)hpfh5-5:5’-gctaaaatcatcggggattt-3’(seqidno:5)hpfh5-6:5’-gtaaaatcatcggggatttt-3’(seqidno:6)hpfh5-15:5’-gtgtcttattaccctgtcat-3’(seqidno:15)hpfh5-19:5’-gttggggtgggcctatgaca-3’(seqidno:19)hpfh5-20:5’-gtttggggtgggcctatgac-3’(seqidno:20)前三个间隔序列靶向要被缺失的区域的5'边界,以及最后三个间隔序列靶向要被缺失的区域的3'边界,如图1c和d所示。细胞转染k-562细胞在补充有10%fbs和2mm新鲜l-谷氨酰胺的rpmi培养基中培养,并且在它们接近1×105/ml的汇合时被传代。按照制造商的说明,通过表达hpfh5靶向sgrna的1μg载体以及表达cas9的1000ng质粒,使用amaxanucleofector4d转染20万个k-562细胞。3天后使用quickextractdna提取溶液(epicentre,madison,wi)收集基因组dna,如所述。将hek293t细胞于转染之前24小时在24孔板中接种,密度为每孔80,000个细胞,并在补充有10%fbs和2mm新鲜l-谷氨酰胺的dmem培养基中培养。根据制造商的说明,使用2μl的lipofectamine2000(lifetechnologies公司),用1000ng表达cas9和grna的质粒转染细胞。转染后72小时,使用quickextractdna提取液(epicenter)收获基因组dna。通过测序进行在靶和脱靶突变检测为了对在靶位点和推定的脱靶位点进行测序,鉴定合适的扩增引物,并使用基因组dna用这些引物设置反应,使用quickextractdna提取溶液(epicentre)从转染后三天的被处理细胞收获所述基因组dna。扩增引物包含两侧是衔接子的基因特异性部分。正向引物的5'端包含修饰的正向(读出1)引物结合位点。反向引物的5'端包含相反方向的组合改良反向(读出2)和条形码引物结合位点。通过在琼脂糖凝胶上分离来验证各个pcr反应,然后纯化并重新扩增。第二轮正向引物含有illuminap5序列,其后是经修饰的正向(读出1)引物结合位点的一部分。第二轮反向引物含有illuminap7序列(5'端),其后是6碱基条形码和组合修饰的反向(读出2)条形码引物结合位点。还在琼脂糖凝胶上检查第二轮扩增,然后纯化,并使用nanodrop分光光度计定量。将扩增产物合并以匹配浓度,然后提交给emoryintegratedgenomiccore用于在illuminamiseq机上进行文库准备和测序。测序读数按条形码分类,然后与生物信息学为每种产品提供的参考序列比对。使用前述软件,在推定切割位点的区域中检测比对的测序读出中的插入和缺失率;参见例如lin等人,nucleicacidsres.,42:7473-7485(2014)。然后将在该窗口中检测到的插入和缺失水平与从模拟转染的细胞中分离的基因组dna中的相同位置看到的水平进行比较以最小化测序错误的影响。突变检测测定使用通过nhej的双链断裂的不完美修复产生的突变率来测量cas9和向导rna组合的在靶和脱靶切割活性。按照制造商的说明,使用accuprimetaqdna高保真聚合酶(lifetechnologies,carlsbad,ca)扩增在靶位点,在含有1μl细胞裂解物和1μl每种10μm扩增引物的50μl反应中进行40个循环(94℃,30秒;52-60℃,30秒;68℃,60s)。根据制造商方案进行t7ei突变检测测定[reyon等人,nat.biotechnol.,30:460-465(2012)],将消化物在2%琼脂糖凝胶上分离,并使用imagej[guschin等人,methodsmol.biol.,649:247-256(2010)]进行定量。该测定确定混合细胞群体中插入/缺失(“indel”)的百分比。通过终点pcr检测反转和缺失按照制造商的说明,使用accuprimetaqdna高保真聚合酶(lifetechnologies)进行所有终点pcr反应,在含有1μl细胞裂解物和1μl每种10μm靶区域扩增引物的50μl反应中进行40个循环(94℃,30秒;60℃,30秒;68℃,45秒)。使用液滴数字pcr(ddpcr)进行缺失定量使用biorad(hercules,ca)液滴数字pcr机(ddpcr)qx200定量指示预期染色体缺失的连接的染色体末端的水平。机器允许通过将各个pcr反应断裂为约20,000个液滴进行绝对定量,液滴通过终点pcr使用cybergreen样试剂和可以有效区分pcr阳性和pcr阴性液滴的读出器进行单独测试。使用qiaampdna迷你试剂盒(qiagen,valencia,ca)从k-562细胞中提取用于ddpcr的基因组dna。pcr反应含有2xddpcrevagreensupermix,200ng基因组dna,引物和hindiii(1u/反应)。反应进行40个循环(94℃,30s;55-65℃,30秒;72℃,90秒)。实验数据在图2a和图2b中示出在k562和hek293细胞中5'和3'靶位点上的每个向导rna的在靶切割效率的分析。所有向导rna在两种细胞类型中显示活性。在k562细胞中分别用hpfh5-4(59%)和hpfh5-19(76%)观察到了5'和3'位点的最高活性。在hpfh5-4位点的插入缺失的序列分析证实了多种插入缺失突变与切割和nhej介导的错误修复相符(图2c)。将来自5'和3'靶位点的向导rna对与表达cas9的质粒一起递送至k562和hek293细胞,并且随后通过pcr分析基因组dna中13kb片段的缺失或倒位的存在。图3a和图3b示出了对于所有向导rna组合,检测到缺失和反转事件。对使用hpfh5-4和hpfh5-15向导rna产生的缺失事件的序列分析证实了预期的13kb缺失,并示出了在连接其余染色体末端时产生的普遍接头序列(图4)。使用不同向导rna对产生所期望的13kb缺失等位基因的效率使用ddpcr来定量。图5示出了缺失使用所有向导序列对实现,通过hpfh54-15向导组合在两种细胞类型中实现约12%的最大效率。单独检查hpfh5-4和hpfh5-15向导序列的脱靶切割活性。生物信息学用于预测最可能的脱靶位点(图6a)。使用深度测序询问在这些预测位点处的基因组编辑频率。图6b中的数据示出,对于任一向导rna,尽管有高的在靶活性水平(分别为64%和91%),没有超出背景的脱靶基因组修饰的证据。这表明这两个向导rna的高特异性。在13kb的hpfh-5缺失序列内,可能较小的亚区造成与该基因组变体相关的表型,并且这些较小区域的缺失可代表替代性治疗策略。为了测试这个概念,另外的向导rna被设计用于在位于13kb序列整个长度上靶向位点(图7a),并单独测试其基因编辑效率。图7b示出了多个向导rna使得在整个13kb片段的附加区域能够进行高水平的基因编辑(高达70%)。考虑这些向导序列能够彼此配对以产生具有可行治疗效用的较小缺失。实施例2产生靠近于chr11:5233055-5240389的缺失在该实施例中,我们说明了使用本文所述的方法产生靠近于区域chr11:5233055-5240389的某些缺失。在于染色体11上的人β-球蛋白位点中具有7.2kb缺失(本文中被称作“corfu长”缺失)的人类患者中已经观察到该区域中的缺失。在纯合状态下,这种缺失与完全不存在血红蛋白a和a2以及高水平的胎血红蛋白和hpfh相关[wainscoat等人,ann.nyacadsci445:20(1985)和kulozik等人,blood71:457(1988)]。该缺失描述于图8中。我们进一步确定了γ-球蛋白-bcl11a和gata1的关键调节子的已知结合位点位于7.2kb区域内的3.5kb亚区内(图8)。考虑该较小区域单独缺失(区域chr11:5233055-5240389内的染色体11中的缺失)可足以赋予与较大缺失所观察到的hpfh表型相当的hpfh表型,此外,可以以比较大缺失更高的基因组编辑效率实现。设计了crispr向导rna,以实现在7.2kb和3.5kb区域的各端进行切割,并验证其实现间插片段缺失的能力。测试针对每个corfu缺失的边界的各个向导rna的基因编辑效率。向导rna的间隔序列如图9所示。如实施例1所述产生编码向导rna的载体并将其引入细胞中。图10a-图10c中的数据表明,对每个边界获得的多个功能向导序列在hek293细胞中实现25-50%的基因组编辑。在k562细胞中观察到甚至更高水平的基因组编辑活性(40-80%)(图11a-c)。向导rna对的共同递送导致间插片段的缺失和倒置(图12a和b)。实施例3产生靠近于chr11:5226631-5249422的缺失在该实施例中,我们说明了使用本文所述的方法产生靠近于区域chr11:5226631-5249422的一些缺失。在于染色体11上的人β-球蛋白位点中具有大缺失(本文被称作hpfhkenya样变体)的人类患者中已经观察到该区域中的缺失[huisman等人,arch.biochem.biophys.152:850(1972)和ojwang等人,hemoglobin7:115(1983)]。天然存在的变体表现为是由aγ和β-球蛋白基因的氨基酸80-87之间的非同源交叉和区域chr11:5226631-5249422内的染色体11中间插的约23kb序列的缺失造成的。kenya融合蛋白质含有aγ链的氨基酸残基1-80和β链的氨基酸残基87-146。设计并验证了用于在约23kb区域的每个边界处实现切割的crispr向导rna(图13a和1b)。如实施例1所述产生编码向导rna的载体并将其引入细胞中。向导rna的功能分析表明,在靶标位点的5'和3'边界处都能实现稳健的切割效率(图13c)。预计这些向导序列的组合实现所期望的缺失。实施例4产生靠近于chr11:5249959-5249971的缺失在该实施例中,我们说明了使用本文所述的方法产生靠近于区域chr11:5249959-5249971的一些缺失。在于被鉴定并示出为与hpfh相关的区域5249959-5249971内的染色体11中具有β-球蛋白位点的小缺失变体的人类患者中已经观察到该区域中的缺失[gilman等人,nucleicacidsresearch16(22):10635(1988)]。该缺失跨越γ-球蛋白基因的-102至-114,并且包含据信对于调节γ-基因启动子重要的远端ccaat盒(图14a)。一种方法是在13bp区域内切割该位点,并允许nhej错修复病变,期望在一些情况下可重现精确的13bp缺失。然而,不能保证单独使用nhej的修复结果,并且不太可能以有临床意义的频率发生精确的13bp缺失,而非额外的缺失或插入,这可本身具有所期望的治疗后果。或者,dsb可以由hdr在共同递送的修复模板供体存在的情况下修复,所述供体指定精确的13bp缺失。可以采用创建13bp缺失的第三种方法,其利用了在预期突变位点的微同源性和mmej的修复途径。在本实施例中,对包含并邻近于13bp缺失位点的序列的分析揭示存在两个8bp重复序列,我们预测其可能会在mmej介导的修复期间重组以在存在单个双链断裂的情况下产生13bp缺失(图14b)。我们设计向导rna以非常接近这些重复进行切割(图14b和图14c),并在hek293细胞中测试其驱动细胞中13bp缺失的产生的能力。如实施例1所述产生编码向导rna的载体并将其引入细胞中。所得基因组编辑事件的序列分析揭示两个向导序列,sd1和sd2,介导的dna切割和修复事件中13bp缺失是最常见的结果(图15a和图15b)。对于向导sd2,dna修饰的总等位基因频率为28%,其中这些事件的三分之一(等位基因的9.3%)包含13bp缺失。其余修饰的大部分是1-4个核苷酸的缺失,尽管也检测到其他事件并进行序列确认(图15c)。这些数据教导可以利用微同源性来使单个dna切割事件在内源性人β-球蛋白位点中产生治疗上相关的突变。实施例5产生靠近于chr11:5196709-5239223的缺失在该实施例中,我们说明了使用本文所述的方法产生邻近于区域chr11:5196709-5239223的一些缺失。在于染色体11上的人β-球蛋白位点中具有大缺失(本文被称作hpfh-4(或“hpfh意大利”)等位基因)的人类患者中已经观察到该区域中的缺失[camaschella等人,haematologia75(5):26(1990)],其特征在于完全涵盖较短(13kb)hpfh-5等位基因的区域chr11:5196709-5239223内的染色体11中的40kb缺失(图16a)。考虑可以使用诸如crispr的基因组编辑技术在具有血红蛋白病(例如镰状细胞或β-地中海贫血)的个体的造血细胞中产生相应的或相似的基因组区域或其子集的靶向缺失以使γ球蛋白以及因此产生的胎血红蛋白去抑制或引起其重新表达。实施例6产生靠近于chr11:5225700-5236750的缺失在该实施例中,我们举例说明了本文描述的方法来产生靠近于区域chr11:5225700-5236750的一些缺失。在具有hpfhblack等位基因的人类患者中观察到该区域的缺失[anagnou等人,blood65:1245(1985)],其特征在于在chr11:5225700-5236750区域内的染色体11中的大缺失(图16b),该大缺失与hpfh-4和hpfh-5缺失完全重叠。考虑可以使用诸如crispr的基因组编辑技术在具有血红蛋白病(例如镰状细胞或β-地中海贫血)的个体的造血细胞中产生相应的或相似的基因组区域或其子集的靶向缺失以使γ-球蛋白以及因此产生的胎血红蛋白去抑制或引起其重新表达。实施例7hpfh相关的非缺失突变的产生β-球蛋白位点的gγ或aγ基因中的-175(t至c)点突变与泛细胞型hpfh的表型相关,即在许多细胞中具有相当均匀的分布;参见例如ottolenghi等人,blood71:815(1988)和surrey等人,blood71:807(1988)。hpfh表型被认为是由于破坏调节因子通常所结合的并抑制γ-球蛋白表达的一种或更多种顺式元件,或是由于增强上调γ-球蛋白表达的调节因子的结合。考虑可以使用诸如crispr的基因组编辑技术在具有血红蛋白病(例如镰状细胞或β-地中海贫血)的个体的造血细胞中产生点突变或引起调节因子结合变化的其他修饰以使γ球蛋白以及因此产生的胎血红蛋白去抑制或引起其重新表达。化脓性链球菌cas9的多个推定pam序列的位置临近该靶位点(图16c)。实施例8临床研究与药理学众所周知,hbf水平的增加降低hbs聚合,从而改善sca的表型,减少了临床并发症。在crispr/cas9技术的背景下,或者通过使用本文所述的用于基因编辑的其它核酸内切酶,人类受试者/患者的初步药效学研究的主要目的将是证明γ-球蛋白的成功去抑制以及hbf的伴随增加和有益效果,并确定这种遗传修饰对治疗血红蛋白病的安全性和有效性。基于细胞的研究可以包括野生型细胞,例如正常cd34+hhsc,其通常不表达高水平的hbf,但如本文所述被编辑以增加其hbf水平;以及衍生自具有血红蛋白病(如β-地中海贫血或scd)的患者的细胞,例如cd34+细胞。总红细胞hbf将通过阳离子hplc测量,并且红细胞中hbf的分布将使用facs在f-细胞(具有可检测的hbf水平的细胞)中定量。尽管hbf中甚至小的增量增加已经示出在scd的背景下具有有益效果,如上所述,在一些实施方案中,受试者中总hb的至少约9%将是hbf,其与scd中的死亡率降低相关;参见例如platt等人,nengljmed.330(23):1639-1644(1994)。在一些实施方案中,hbf将为至少约14%,其与额外的临床益处相关,并且在一些实施方案中,hbf将为至少约20%至30%,其与scd背景下表型的极大标准化相关。类似地,引入甚至相对有限的、具有显著升高的hbf水平的细胞的亚群(称为“f-细胞”)在各种患者中可以是有益的,因为在一些情况下,归一化细胞相对于患病细胞将具有选择性优势。即使适度水平的hbf水平升高的循环rbc也可有利于改善患者血红蛋白病的一个或更多个方面。然而,通常考虑,至少十分之一的循环红细胞(rbc)将具有升高的hbf水平,超过四分之一的循环rbc将具有升高的hbf水平,或至少三分之一的循环rbc将具有升高的hbf水平。在一些实施方案中,至少约一半,以及在一些实施方案中至少约四分之三或更多的循环rbc将具有升高的hbf水平。实施例9生物分布将进行初步可行性研究(非glp)以证明nod/scidil2rγ小鼠中cd34+hhsc的移植。将在免疫受损的nod/scidil2rγ小鼠中进行glp生物分布和持续性研究。crispr/cas9修饰的人cd34+hsc将通过静脉内注射(或其他途径,如骨内)施用到nod/scidil2rγ小鼠。未修饰的cd34+hhsc将用作对照。实施例10体内药理学研究在体内药理学的说明性实施例中,将基因编辑的hsc引入免疫缺陷小鼠中,并评估诸如hsc植入的结果。例如,“nsg”或nod严重联合免疫缺陷病γ(nod.cg-prkdcscidil2rgtm1wj1/szj)是迄今为止描述的最为免疫缺陷的近交实验室小鼠品种之一;参见例如shultz等人,nat.rev.immunol.7(2):118-130(2007)。另一种适用于研究造血干细胞移植的免疫受损的小鼠模型是nod/mrkbomtac-prkdcscid小鼠(ww.taconic.com/nodsc)。使用免疫受损的小鼠模型的一个说明性方法是将crispr/cas9修饰的cd34+人hsc注射到免疫受损的nod/scid/il2rγ小鼠中以证明归巢和移植能力。还可以考虑对模型动物的研究,只要这些模型可以合理地预测人类患者一个或更多个方面的病情。发展提供了与多种疾病的某些方面相关的信息的动物模型仍然是本领域常规改进的课题,使用crispr/cas-9基因编辑极大地便于更快创建这种疾病相关的动物模型。实施例11使用编辑的细胞减轻β-地中海贫血使用本文所述和说明的方法,可以产生表达增加水平的hbf的人细胞。此类细胞可以包括例如能够产生红细胞谱系的细胞(例如红血细胞(rbc))的人类造血干细胞(人hsc)。因此,这种hsc可以用于减轻与β-地中海贫血相关的一种或更多种症状。例如,当应用基因组编辑程序以增加患有β-地中海贫血的患者的细胞中的hbf水平时,作为两种有益效果的组合的结果,可以减轻β-地中海贫血的一种或更多种症状或并发症。首先,hbf提供血红蛋白的功能形式,该血红蛋白的功能形式能够在减轻贫血和β-地中海贫血的相关临床病症方面(例如,在重型β-地中海贫血和中间型β-地中海贫血中)(其中正常从hbb基因表达的成体β-球蛋白链不存在或减少)起重要作用。其次,未配对的α-球蛋白链的水平(其是在临床上与β-地中海贫血相关的许多其他问题的起因)降低,这是因为α-球蛋白链可以与由γ-球蛋白基因编码的β-球蛋白链配对,γ-球蛋白基因的表达如本文所述增加。同样如本文所指出的,与正常rbc相比,β-地中海贫血rbc在存活和其它因素方面具有选择性缺点;如本文所述的细胞治疗通过例如增加hbf的水平以及伴随地降低未配对的α-球蛋白链的水平而克服了一些缺点。此外,可以应用其他技术来增强通过如本文所述的基因组编辑修饰的细胞的递送、扩增和/或持久性。这些技术包括消融技术,其中一些驻留细胞在引入细胞之前被消除。例如,在骨髓移植和其中将正常或经校正的细胞引入患者体内的其他程序的情况下,常规地使用此类技术。许多此类程序在本领域中是已知的,并且结合人类患者的治疗常规地实践。使用这种技术来减轻β-地中海贫血的一个说明性和非限制性实施例如下。在自体程序中,对来自患有β-地中海贫血的患者的细胞进行基因组编辑。由于患者自身的细胞已经匹配,因此它们不会引起与使用同种异体细胞相关的潜在问题。对这种细胞进行离体校正之后将它们重新引入患者体内提供了一种减轻疾病的手段。作为可使用的细胞的一个说明性实施例,来自患有β-地中海贫血的患者的外周血干细胞(pbsc)可以来源于血流。可使用称为清血法(apheresis)或白细胞去除术(leukapheresis)的过程来获得pbsc。在清血法前的4或5天,可以向患者施用药物以增加释放到血流中的干细胞的数量。在清血法中,通过臂中的大静脉或中心静脉导管(放置于颈部、胸部或腹股沟区域中的大静脉中的柔性管)来去除血液。使血液穿过去除干细胞的机器。作为可以使用的细胞的另一个说明性实施例,可以使用熟知的技术来从患者的骨髓收获造血干细胞(hsc)。cd34是与造血干细胞相关的抗原,并且cd34+hsc的分离同样可以通过熟知的和临床上验证的方法完成。例如,可以使用已经fda批准用于多种移植环境中并且可从miltenyibiotec商购获得的磁珠分离方法,以及用于处理和维持这种细胞的制备物。为了如本文所述治疗患有β-地中海贫血的人类患者,可以使用被调整用以反映患者体重的cd34+hsc群体,例如,包括约一千万个cd34+hsc/千克体重的群体。然后使用本文所述的基因组编辑方法来修饰此细胞群。例如,如果cas9是基因组编辑核酸内切酶,则可以通过使用多种已知技术转染mrna来将蛋白质引入cd34+hsc;以及引入(在转染中潜在地同时)靶向如本文所述的位点的向导rna(其可以是单分子向导物或双分子向导物)。根据所使用的程序,随后可以使用一部分细胞(例如,原始细胞中的一半)来重新引入到患者体内。如果要使用消融来增强新引入的细胞的植入,则在引入基因组编辑的hsc之前,可以使患者经历例如轻度骨髓调节。在任何调节之后,可以将基因组编辑的hsc的群体重新引入患者体内,例如通过输血。随着时间的推移,hsc产生红细胞谱系的细胞,包括红血细胞(rbc)。在所得rbc中,在β-地中海贫血的情况下的基因组编辑导致hbf水平增加,以及未配对的α-球蛋白链的伴随减少;由此减轻了与β-地中海贫血相关的一种或更多种症状或并发症。实施例12使用编辑的细胞减轻镰状细胞性贫血使用本文所述和说明的方法,可以产生表达增加水平的hbf的人细胞。此类细胞可以包括例如能够产生红细胞谱系的细胞(例如红血细胞(rbc))的人类造血干细胞(人hsc)。因此,这种hsc可以用于减轻与镰状细胞病相关的一种或更多种症状,诸如镰状细胞性贫血。例如,当应用基因组编辑程序以增加患有镰状细胞性贫血(sca)的患者的细胞中的hbf水平时,可以减轻sca的一种或更多种症状或并发症。在一些实施方案中,突变β-球蛋白基因的至少一个拷贝被敲低或消除,从而导致两个有益效果的组合。首先,hbf提供血红蛋白的功能形式,该血红蛋白的功能形式可以在减轻sca的贫血和相关临床病症方面起重要作用。其次,从突变体β-球蛋白表达的镰状细胞血红蛋白(hbs)的水平降低或消除。hbs的存在导致在临床上与sca相关的许多问题,并且即使hbs存在的适度减少也可以用于减少或实质上防止镰状化,如在本文和在领域中所述。同样如本文所指出的,与正常rbc相比,镰状细胞rbc在存活和其它因素方面具有选择性缺点;如本文所述的细胞治疗通过例如增加hbf的水平,以及在突变β-球蛋白基因被敲低或消除的实施方案中伴随地降低hbs的水平而克服了一些缺点。此外,可以应用其他技术来增强通过如本文所述的基因组编辑修饰的细胞的递送、扩增和/或持久性。这些技术包括消融技术,其中一些驻留细胞在引入细胞之前被消除。例如,在骨髓移植和其中将正常或经校正的细胞引入患者体内的其他程序的情况下,常规地使用此类技术。许多此类程序在本领域中是已知的,并且结合人类患者的治疗常规地实践。使用这种技术来减轻sca的一个说明性和非限制性实施例如下。在自体程序中,对来自患有sca的患者的细胞进行基因组编辑。由于患者自身的细胞已经匹配,因此它们不会引起与使用同种异体细胞相关的潜在问题。对这种细胞进行离体校正之后将它们重新引入患者体内提供了一种减轻疾病的手段。作为可以使用的细胞的一个说明性实施例,来自患有sca的患者的pbsc可以来源于血流,或者可以从患者的骨髓收获hsc,各自如在先前实施例中使用熟知技术所描述的。然后可以使用如前述实施例所述的程序和熟知的技术中衍生出cd34+细胞。为了如本文所述治疗患有sca的人类患者,可以使用被调整用以反映患者体重的cd34+hsc群体,例如,包括约一千万个cd34+hsc/千克体重的群体。然后使用本文所述的基因组编辑方法来修饰此细胞群。例如,如果cas9是基因组编辑核酸内切酶,则可以通过使用多种已知技术转染mrna来将蛋白质引入cd34+hsc;以及引入(在转染中潜在地同时)靶向如本文所述的位点的向导rna(其可以是单分子向导物或双分子向导物)。根据所使用的程序,随后可以使用一部分细胞(例如,原始细胞中的一半)来重新引入到患者体内。如果要使用消融来增强新引入的细胞的植入,则在引入基因组编辑的hsc之前,可以使患者经历例如轻度骨髓调节。在任何调节之后,可以将基因组编辑的hsc的群体重新引入患者体内,例如通过输血。随着时间的推移,hsc产生红细胞谱系的细胞,包括红血细胞(rbc)。在所得rbc中,在sca的情况下的基因组编辑导致hbf水平增加,以及在其中突变β-球蛋白基因被敲低或消除的实施方案中伴随地降低hbs的水平;由此减轻了与β-地中海贫血相关的一种或更多种症状或并发症。实施例13γ-球蛋白基因的去抑制从5名健康供体获得了cd34+细胞,然后筛选基础水平的γ球蛋白转录物,并与cas9mrna和与hbb位点相对应的以下向导物一起电穿孔以重新唤醒胎血红蛋白表达:hpfhcs-02和hpfhcs-06以产生corfu小3.5kb缺失;hpfhcl-01和hpfhcl-08以产生corfu大7.2kb缺失;hpfhk-02和hfphk-17以产生kenya约20kb的缺失;hpfhsd_02,其在两个位置处切割以产生4.9kb的小缺失;hpfhsd_01,其在两个位置处切割,以产生4.9kb的小缺失;hpfh5-04和hpfh5-15以产生hpfh5约12.9kb缺失。编辑后,这些cd34+细胞分化成红细胞系祖细胞,以分析与α-球蛋白和β-球蛋白转录物相关的γ-球蛋白转录物的表达。在不同实验中进行hpfh5缺失。这些实验的结果示于图17至图19和图21中。图17a至图17c分别示出了多个供体的γ/α-球蛋白、α-球蛋白和γ-球蛋白转录物的基础水平。图18a至图18c分别示出与未编辑的对照相比,在各个供体的每一个中使用上述corfu短、corfu长、kenya和小缺失向导物时的γ-球蛋白、α-球蛋白和β-球蛋白转录物的水平。图19示出了与未编辑的对照相比,在各个供体的每一个中使用上述corfu短、corfu长、kenya和小缺失向导物时的γ-球蛋白与α-球蛋白转录物水平的比率。图21a至图21c分别示出了与未编辑的外周血对照相比,针对两个供体中的每一个使用上述corfu长和hpfh5向导物时的α-球蛋白、γ-球蛋白和γ/α-球蛋白转录物水平。综上所述,corfu短、corfu长、kenya、小缺失和hpfh5向导物在这个实施例中去抑制了γ-球蛋白基因。实施例14通过crispr/cas9介导的基因组编辑去抑制g-球蛋白表达:向导鉴定和验证crispr/cas9向导rna被设计用于产生遗传性胎儿血红蛋白持续(hpfh)缺失。然后将这些向导rna进行生物信息学筛选,以获得潜在的脱靶位点。再筛选向导rna的活性(切割活性)和缺失频率。然后筛选向导rna的脱靶活性。最后,筛选向导rna的常见单核苷酸多态性(snp)。图22-34中示出了这些试验的结果。图22b和图23a至图23d示出了hpfh缺失的最佳grna。图25a示出缺失大小对缺失频率没有影响。图25b示出了grna对的平均nhej活性和缺失频率的弱相关性。图25c至图25d示出pam取向对缺失频率的影响。图26是示出向导rna对缺失频率的影响的图。图27a至图27d示出了向导rna对缺失频率的剂量曲线。图28是示出如通过深度测序确定的在靶(hpfh5-4on;hpfh5-15on)和预测的脱靶位点处的基因组编辑频率的图。多种在靶和脱靶序列与图6a中相同。图29是示出在k562细胞中进行的对小缺失向导rnahpfhsd_02的测试的图。具体地,示出了向导rnahpfhsc_02的插入缺失频率。全部缺失的约50%是13bp。质粒dna对应更高的量(约30%)。图30a是示出corfu大缺失向导rnacl01和cl08的单独经修饰等位基因频率的图。图30b是示出corfu大缺失向导rnacl01和cl08组合的缺失频率的图。图31a至图31c示出了k562细胞中dna、rna和蛋白质之间在插入频率和缺失频率方面的差异。图32是示出如通过tide分析测量的向导rnacl01、cl08、k2和sd2的插入缺失频率的图。图33是示出corfu缺失和kenya缺失在24小时的hpfh缺失频率的图。图34b至图34c示出了hbf表达细胞。总之,本实施例中的corfu短、corfu长、kenya、小缺失和hpfh5向导去抑制γ-球蛋白基因并上调hbf。关于说明性实施例的说明虽然本公开提供了对用于说明本发明的各个方面和/或其可能应用的多种具体实施方案的描述,但是应当理解,本领域技术人员将想到许多变化和修改。因此,本文描述的本发明应被理解为至少与所要求保护的范围一样宽,而不是由本文提供的特定说明性实施方案更狭义地限定。本申请中引用的所有文献的全部内容通过引用并入本文,特别注意其引用的公开内容。序列表<110>克里斯珀医疗股份公司<120>治疗血红蛋白病的材料和方法<130>32265/49064pct2<150>62/119,754<151>2015-02-23<160>192<170>patentinversion3.5<210>1<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>1gcttccattctaacccacat20<210>2<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>2gtactgagttctaaaatcat20<210>3<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>3gactgagttctaaaatcatc20<210>4<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>4gctgagttctaaaatcatcg20<210>5<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>5gctaaaatcatcggggattt20<210>6<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>6gtaaaatcatcggggatttt20<210>7<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>7gaaaatcatcggggattttg20<210>8<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>8ggagatttcacattaaatgt20<210>9<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>9gatgccaatgtgggttagaa20<210>10<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>10gattagtgtaatgccaatgt20<210>11<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>11gcatttaatgtgaaatctca20<210>12<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>12gaattagtgtaatgccaatg20<210>13<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>13gggactgagaagaatttgaa20<210>14<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>14gctgagaagaatttgaaagg20<210>15<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>15gtgtcttattaccctgtcat20<210>16<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>16ggtcataggcccaccccaaa20<210>17<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>17gggaagtcccattcttcctc20<210>18<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>18gatgtttaagattagcattc20<210>19<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>19gttggggtgggcctatgaca20<210>20<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>20gtttggggtgggcctatgac20<210>21<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>21gtgggacttccatttggggt20<210>22<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>22gatgggacttccatttgggg20<210>23<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>23gagaatgggacttccatttg20<210>24<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>24gaagaatgggacttccattt20<210>25<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>25ggaagaatgggacttccatt20<210>26<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>26gaaacatcctgaggaagaat20<210>27<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>27gtaaacatcctgaggaagaa20<210>28<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>28ggctaatcttaaacatcctg20<210>29<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>29gtggtatgggaggtatacta20<210>30<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>30gatctcgaactcctaacatc20<210>31<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>31ggtatacctcccataccatg20<210>32<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>32ggagtgcaatggcatgatcc20<210>33<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>33gagcattgctatggttgccc20<210>34<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>34ggaattcaccccaccagtgc20<210>35<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>35gacagaccagcacgttgccc20<210>36<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>36gcagctcctgggcaacgtgc20<210>37<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>37gttagcaaaagggcctagct20<210>38<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>38gattattctgagtccaagct20<210>39<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>39ggctgctggtggtctaccct20<210>40<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>40ggtagaccaccagcagccta20<210>41<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>41gtagaccaccagcagcctaa20<210>42<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>42gccaccagcagcctaagggt20<210>43<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>43gggtgggaaaatagaccaat20<210>44<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>44gcccaaagtgtgactatcaa20<210>45<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>45gcccattgatagtcacactt20<210>46<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>46gccaaagtgtgactatcaat20<210>47<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>47gctatcaatggggtaatcag20<210>48<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>48ggtaatcagtggtgtcaaat20<210>49<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>49gacctgtctcaaccctcatc20<210>50<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>50gacctgatgagggttgagac20<210>51<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>51gcacacacgcagaaagtgtt20<210>52<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>52gtggttcttctatggctatc20<210>53<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>53gtgcctatgtatgattatag20<210>54<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>54gtatcagaatggccctagtc20<210>55<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>55gatcagaatggccctagtct20<210>56<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>56gtctaagtatacccagacta20<210>57<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>57gctctaagtatacccagact20<210>58<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>58gctagtctgggtatacttag20<210>59<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>59gttcagtatgtctgaatgaa20<210>60<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>60gaaattaaagccaaatcttg20<210>61<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>61ggaattaattcctcaagatt20<210>62<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>62gttaaaacaaagtataggaa20<210>63<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>63ggtacatgtacaagttatat20<210>64<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>64gacacattgtcagtatattc20<210>65<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>65gatccttctaattttaccta20<210>66<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>66gtgccataggtaaaattaga20<210>67<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>67gtgagcaccatttttgccat20<210>68<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>68gatggcaaaaatggtgctca20<210>69<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>69gcacccattaatgccttgta20<210>70<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>70gaaccgtacaaggcattaat20<210>71<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>71ggaaccgtacaaggcattaa20<210>72<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>72gaaagcaagggaaccgtaca20<210>73<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>73gtccctatctgtagagcctc20<210>74<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>74gagcctctcccatacccatg20<210>75<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>75gctccacatgggtatgggag20<210>76<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>76gtgtctctccacatgggtat20<210>77<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>77gttgtctctccacatgggta20<210>78<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>78gttctaagtgcagaattagc20<210>79<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>79ggcggtggggagatatgtag20<210>80<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>80gtgctgaaagagatgcggtg20<210>81<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>81gctgctgaaagagatgcggt20<210>82<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>82gactgctgaaagagatgcgg20<210>83<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>83ggtgttttaggctaatatag20<210>84<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>84gtcaaattttggtggtgata20<210>85<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>85gtacaatagtataacccctt20<210>86<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>86gcatttgtggatactattaa20<210>87<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>87gtaatagtatccacaaatgc20<210>88<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>88gatcaagcatccagcatttg20<210>89<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>89gtgtcatttttaacaggtag20<210>90<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>90ggtaaattcttaaggccatg20<210>91<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>91ggatcaaataacagtcctca20<210>92<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>92gtctgttaattccaaagact20<210>93<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>93gctgaaatgattttacacat20<210>94<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>94gaggatgagccacatggtat20<210>95<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>95gatgagccacatggtatggg20<210>96<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>96ggaggtatactaaggactct20<210>97<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>97gtttggggtgggcctatgac20<210>98<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>98ggtaggtagatgctagattc20<210>99<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>99gtcttattcaatacctaggt20<210>100<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>100gcaccataagggacatgata20<210>101<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>101gatgtcccttatggtgcttc20<210>102<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>102gcagtagaggtatggtttcc20<210>103<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>103gatctagcatctacctacct20<210>104<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>104gattactggtggtctaccct20<210>105<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>105gtagaccaccagtaatctga20<210>106<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>106gcctaccctcagattactgg20<210>107<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>107gtggtatgggaggtatacta20<210>108<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>108gatctcgaactcctaacatc20<210>109<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>109ggtatacctcccataccatg20<210>110<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>110gctaaaatcatcggggattt20<210>111<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>111ggtgtgctggcccgcaactt20<210>112<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>112gtggggcagaagtcgttgct20<210>113<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>113gctggcccgcaactttggca20<210>114<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>114gaaccgtacaaggcattaat20<210>115<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>115gaaagcaagggaaccgtaca20<210>116<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>116ggaaccgtacaaggcattaa20<210>117<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>117gtcaatggtacttgtgagcc20<210>118<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>118gccactcaagagatatggtg20<210>119<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>119gcaagccccctgtttggatc20<210>120<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>120gtgcctacaagccccctgtt20<210>121<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>121gcctcgagactaaaggcaac20<210>122<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>122gtcttcagcctacaacatac20<210>123<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>123gcccttcaagcactagtcac20<210>124<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>124ggccagtgactagtgcttga20<210>125<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>125gctcgaggcaacttagacaa20<210>126<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>126gccagtgactagtgcttgaa20<210>127<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>127gctcgagactaaaggcaaca20<210>128<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>128gcagtgactagtgcttgaag20<210>129<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>129gttagcaaaagggcctagct20<210>130<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>130gtgcctagtacattactatt20<210>131<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>131gacagaccagcacgttgccc20<210>132<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>132gtacacatattgaccaaatc20<210>133<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>133gcagctcctgggcaacgtgc20<210>134<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>134gacgaatgattgcatcagtg20<210>135<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>135gattattctgagtccaagct20<210>136<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>136ggtgtgctggcccatcactt20<210>137<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>137gttaagttcatgtcatagga20<210>138<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>138gtttgccttgtcaaggctat20<210>139<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>139gttgtcaaggctattggtca20<210>140<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>140gttgaccaatagccttgaca20<210>141<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>141gaaggctattggtcaaggca20<210>142<211>20<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>142gctattggtcaaggcaaggc20<210>143<211>23<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<220><221>modified_base<222>(1)..(21)<223>a,c,t,g,unknownorother<220><221>misc_feature<222>(1)..(21)<223>nisa,c,g,ort<400>143nnnnnnnnnnnnnnnnnnnnnrg23<210>144<211>57<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>144aaaatcgatactgagttctaaaatcatcggggattttggggactatgtcttacttca57<210>145<211>48<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>145gaaggtaaaccccacccggatactgtcccattattctgtcatcactta48<210>146<211>53<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>146aagacatagtccccaaaatccccgatgattttagaactcagtatcgattttaa53<210>147<211>50<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>147aagacatagtccccaaaatccctgattttagaactcagtatcgattttaa50<210>148<211>50<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>148aagacatagtccccaaaatccccgattttagaactcagtatcgattttaa50<210>149<211>50<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>149aagacatagtccccaaaatccccgattttagaactcagtatcgattttaa50<210>150<211>48<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>150aagacatagtccccaaaatccccgattagaactcagtatcgattttaa48<210>151<211>52<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>151aagacatagtccccaaaatccccgtgattttagaactcagtatcgattttaa52<210>152<211>94<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>152tgtgaaatctcaaggaagtatgaagtaagacatagtccccaaaatccccgatcataggcc60caccccaaatggaagtcccattcttcctcaggat94<210>153<211>94<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>153tgtgaaatctcaaggaagtatgaagtaagacatagtccccaaaatccccgatcataggcc60caccccaaatggaagtcccattcttcctcaggat94<210>154<211>94<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>154tgtgaaatctcaaggaagtatgaagtaagacatagtccccaaaatccccgatcataggcc60caccccaaatggaagtcccattcttcctcaggat94<210>155<211>94<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>155tgtgaaatctcaaggaagtatgaagtaagacatagtccccaaaatccccgatcataggcc60caccccaaatggaagtcccattcttcctcaggat94<210>156<211>94<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>156tgtgaaatctcaaggaagtatgaagtaagacatagtccccaaaatccccgatcataggcc60caccccaaatggaagtcccattcttcctcaggat94<210>157<211>94<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>157tgtgaaatctcaaggaagtatgaagtaagacatagtccccaaaatccccgatcataggcc60caccccaaatggaagtcccattcttcctcaggat94<210>158<211>93<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>158tgtgaaatctcaaggaagtatgaagtaagacatagtccccaaaatcccagacataggccc60accccaaatggaagtcccattcttcctcaggat93<210>159<211>82<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>159tgtgaaatctcaaggaagtatgaagtaagacatagtccccaataggcccaccccaaatgg60aagtcccattcttcctcaggat82<210>160<211>94<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>160tgtgaaatctcaaggaagtatgaagtaagacatagtccccaaaatccccgatgcataggc60ccaccccaaatggaagtcccattcttcctcagga94<210>161<211>94<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>161tgtgaaatctcaaggaagtatgaagtaagacatagtccccaaaatccccgattttcatag60gcccaccccaaatggaagtcccattcttcctcag94<210>162<211>23<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>162actgagttctaaaatcatcgggg23<210>163<211>23<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>163cctgatctcttaaatcatcgtgg23<210>164<211>23<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>164ctgtcttattaccctgtcatagg23<210>165<211>23<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>165tttgcttattaccctgtcataag23<210>166<211>23<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>166ctgatttataaccctgtcatcgg23<210>167<211>23<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>167atgtgtcatcaccctgtcatcag23<210>168<211>23<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>168gtgtcttctttccctgtcattgg23<210>169<211>23<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>169gtgtcttcttcccctgtcatgag23<210>170<211>23<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>170tactcttattcccctgtcatcag23<210>171<211>23<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>171cttttttattaacctgtcattgg23<210>172<211>23<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>172atgagttattagcctgtcattgg23<210>173<211>23<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>173cattcttattacactgtcatcag23<210>174<211>23<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>174tagttttattacactgtcatcag23<210>175<211>23<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>175atgtcttgatagcctgtcataag23<210>176<211>12<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>176ccttgccttgac12<210>177<211>21<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>177ccttgccttgaccaatagcct21<210>178<211>18<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>178aaggcaaacttgaccaat18<210>179<211>22<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>179tgacaaggcaaacttgaccaat22<210>180<211>110<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticpolynucleotide<400>180gccggcccctggcctcactggatactctaagactattggtcaagtttgccttgtcaaggc60tattggtcaaggcaaggctggccaacccatgggtggagtttagccaggga110<210>181<211>110<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticpolynucleotide<400>181tccctggctaaactccacccatgggttggccagccttgccttgaccaatagccttgacaa60ggcaaacttgaccaatagtcttagagtatccagtgaggccaggggccggc110<210>182<211>75<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>182aagactattggtcaagtttgccttgtcaaggctattggtcaaggcaaggctggccaaccc60atgggtggagtttag75<210>183<211>36<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>183aagactattggtcaagtttgcctgggtggagtttag36<210>184<211>63<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>184aagactattggtcaagtttgccttgatcaaggcaaggctggccaacccatgggtggagtt60tag63<210>185<211>62<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>185aagactattggtcaagtttgccttgtcaaggcaaggctggccaacccatgggtggagttt60ag62<210>186<211>61<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>186aagactattggtcaagtttgccttgtcaaggctagctggccaacccatgggtggagttta60g61<210>187<211>67<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>187aagactattggtcaagtttgccttgtcaaggcaaggcaaggctggccaacccatgggtgg60agtttag67<210>188<211>70<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>188aagactattggtcaagtttgccttgtcaaggcttcaaggcaaggctggccaacccatggg60tggagtttag70<210>189<211>72<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>189aagactattggtcaagtttgccttgtcaaggctattcaaggcaaggctggccaacccatg60ggtggagtttag72<210>190<211>74<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>190aagactattggtcaagtttgccttgtcaaggctattggcaaggcaaggctggccaaccca60tgggtggagtttag74<210>191<211>78<212>dna<213>artificialsequence<220><223>descriptionofartificialsequence:syntheticoligonucleotide<400>191gggggccccttccccacactatctcaatgcaaatatctgtctgaaagggtccctggctaa60actccacccatgggttgg78<210>192<211>9<212>prt<213>unknown<220><223>descriptionofunknown:homingendonucleasesequence<400>192leualaglyleuileaspalaaspgly15当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1