用于同时检测样品中多个核酸序列的方法与流程
发明领域本发明涉及检测样品中的核酸序列例如检测临床样品中的病原生物的
技术领域:
。更具体地,本发明涉及通过扩增和检测来自所述病原生物的特异性核酸序列来检测由临床标本中病原生物如病毒或细菌引起的感染的领域。发明背景通常通过在有利于这样的生物生长的条件下培养临床样品并通过包括显微镜的许多不同技术监测生长和检测生物的或多或少的特异性代谢物,来检测传染性病原体。在过去十年中,已经在微生物实验室中实施了核酸扩增测试,以迅速可靠地鉴定病原体。核酸扩增测试可用于直接在临床标本中检测微生物的存在而无需培养。最初,通过扩增靶核酸序列并通过使用凝胶电泳和dna结合荧光染料的可视化检测所得到的dna来实现鉴定。核酸扩增测试彻底改变了临床诊断世界,因为与传统培养测定相比,它们提高了几个数量级的灵敏度和速度。通常,核酸扩增测试由靶特异性核酸扩增步骤和或多或少的通用检测步骤组成。本文下面是可用的扩增技术和检测平台的简要概述。pcr目前仍然是扩增靶序列的第一选择,扩增各种病原体的能力取决于通用、随机或多重扩增技术。在通用pcr测试中,从一系列相关病原体扩增靶序列只需要一个或两个引物对即可。需要保守核苷酸序列的区域,并且通常使用简并引物对。存在几种随机扩增技术,其使用随机八聚体或在其3'端含有随机的5-8个核苷酸延伸并且在其5'端具有规定的序列的引物。随机扩增与taq聚合酶或基于等温聚合酶的扩增酶如klenowdna聚合酶或φ29dna聚合酶组合进行。多重pcr涉及在一个扩增反应中靶向不同序列的若干引物对的组合。多重pcr需要仔细优化,以使其作为单对扩增反应具有可比较的灵敏度和特异性。多重连接依赖性探针扩增(mlpa)技术(schoutenetal;wo01/61033,schoutenetal.,nucl.acidsres.2002,vol30no12;e57)是能够同时扩增不同靶的多重pcr方法。在mlpa中,在相同的反应中加入两个在靶dna上彼此紧邻相互杂交的寡核苷酸。寡核苷酸中的一个是合成的,并且具有40-60个核苷酸(nt)的大小,而另一个寡核苷酸具有100至400nt的大小,并且需要在m13载体中的克隆步骤以最终产生单链探针dna。mlpa由三个步骤组成:第一是将探针杂交到其靶区域的退火步骤,第二是将两个探针共价连接在一起的连接步骤,第三是最终pcr,以仅使用两个通用引物获得靶区域的指数扩增。目前,靶特异性多重pcr扩增是病原体检测测定的标准方法。由于核酸扩增测试的极端灵敏性,必须注意避免这些试验中的污染。所扩增核酸的检测最初通过使用凝胶电泳和嵌入dna染料的大小测定进行。当扩增和检测步骤组合成一个步骤时,采取了最小化污染的第一步。结果,消除了扩增后处理步骤,从而增加了测定的可靠性。此类测定通常被称为封闭系统。已经开发了封闭系统扩增技术如实时pcr(ratcliffetal.currissuesmolbiol.2007,9(2):87-102)和nasba(loensetal.,jclinmicrobiol.2006,44(4);1241–1244)和lamp(saitoretal.,jmedmicrobiol.2005,54;1037-41),并使用嵌入荧光染料或荧光标记的探针。等温lamp技术允许通过使用实时浊度计的分光光度分析实时检测。目前,这些测定中的大多数是生物特异性的,仅在怀疑特定病原体时才有用。这大大限制了这些测定的范围。然而,临床症状很少归因于单一病原体。因此,本领域需要允许同时鉴定和区分多个病原体的测定。这种多参数测定使临床医生能够进行更快更好的治疗,并有助于临床管理和公共卫生的改善。为此,已经开发了用于同时测试多于一个生物的技术。这些技术之一是多重实时pcr。然而,目前只有五种彩色寡探针多重检测是可能的,其中一种颜色原则上预留用于内部对照以监测抑制,甚至可以充当共同扩增的竞争者(molenkampetal.j.virolmethods,2007,141:205-211)。这大大限制了同时测试的病原体的数量。呼吸道感染的区域是特别希望对于几种病原体立即进行快速、可靠和特异性多重测定的区域的例子。急性呼吸道感染是成人和儿童中最普遍的急性感染类型。涉及的病原体数量众多。呼吸道感染(rti)通常分为上呼吸道感染(urti)和下呼吸道感染(lrti)。urti包括鼻漏,结膜炎,咽炎,中耳炎和鼻窦炎,而lrti包括肺炎,毛细支气管炎和支气管炎。病毒和细菌二者都会引起急性rti,而病原体的数量大并且是多样的。涉及rti的非典型病毒和细菌包括甲型流感病毒和乙型流感病毒(infa和b),副流感病毒1、2、3和4(piv-1、-2、-3和4),呼吸道合胞病毒a和b(rsva和b),鼻病毒,冠状病毒229e,oc43和nl63(cor-229e、-oc43和nl63),严重急性呼吸综合征冠状病毒(sars-cov),人偏肺病毒(hmpv),腺病毒,肺炎支原体(mycoplasmapneumoniae),肺炎衣原体(chlamydiapneumoniae),嗜肺军团菌(legionellapneumophila)和百日咳鲍特菌(bordetellapertussis)。这些感染中的许多仅在临床特征上是无法区分的,并且需要快速的实验室测试来优化患者管理和感染控制。病毒培养仍然是呼吸道病毒实验室诊断的金标准。然而,病毒培养相对较慢,因此常规诊断是次优的。尽管对这些病毒中的一些病毒有可用的快速抗原检测测试,但是这些测试已经显示出比病毒培养更不灵敏和更不具有特异性。目前,迫切需要以多重形式同时检测呼吸道病毒的灵敏和特异性的方法。在单个反应中能够检测两个或更多个靶是非常有利的,因为这将在临床诊断中提供明显的优点。它将简化测定,增加通量,最大限度地减少临床标本的消耗,并且特别是多重测定比单一(monoplex)测定更具成本效益。为了区别检测临床标本中的呼吸道病毒,以下检测平台已被使用:(i)凝胶电泳。使用琼脂糖凝胶电泳作为检测装置,已经开发了使用三或四个引物对的几种嵌套多重逆转录酶(rt)-pcr测定。osiowy等人(j.clin.microbiol.1998,36;3149-3154)使用5个引物对,其从呼吸道合胞病毒a和b,副流感病毒1、2和3以及1型至7型的腺病毒扩增rna。pcr产物的大小从84个至348个碱基对变化。与直接免疫荧光(dif)测定或间接免疫荧光(iif)测定相比,该多重rt-pcr方法获得了91%的灵敏度值和87%的特异性值。coiras等人(j.med.virology,2004,72;484-495)使用相同的方法,能够在两个多重rt嵌套pcr测定中同时检测14个呼吸道病毒。它们包括冠状病毒229e和oc43,鼻病毒,肠病毒,副流感病毒4和内部对照,但忽略了腺病毒1至7。该测定评估了两岁以下婴儿的鼻和喉咙拭子(swap)和鼻咽抽吸物。似乎该多重测定比常规病毒培养和免疫荧光测定更灵敏,其优点是可以在同一时间和用单个技术测试所有病毒。另外,在9.5%的样品中发现双重感染。erdman等人(j.clin.microbiol.2003,41;4298-4303)最近开发了用于检测呼吸道合胞病毒a和b,副流感病毒1、2和3以及甲型流感病毒和乙型流感病毒的基于自动荧光毛细管电泳的针对6个常见呼吸道病毒的rt-pcr测定和genescan软件。使用其中每个引物组的正链引物用荧光染料6-羧基荧光素(6-fam)进行5'端标记的引物,进行一步rt-pcr反应。总的来说,这种rt-pcr测定在92%的通过培养或dif染色也是阳性的样品中是阳性的。上述参考文献是其中使用凝胶电泳分析大量样品的技术的实例。凝胶电泳作为检测技术的缺点在于它是费力和耗时的,因此相当昂贵。此外,因为在pcr分析后必须打开每个样品,交叉污染的风险增加。(ii)二次酶杂交。在这种方法中,多重pcr测定与酶联免疫吸附测定(elisa)组合。通过在微量滴定板的包被链霉抗生物素蛋白的孔中的微孔杂交分析来进行多重pcr产物的检测。加入特异性针对扩增的靶序列的生物素化的捕获探针,并进行过氧化物酶标记的杂交反应。随后,通过酶标仪/分光光度计测量光密度。根据光密度截止值,将样品分类为pcr阳性或阴性。已经开发并相对金标准验证了用于快速鉴定引起呼吸道感染的七个或九个微生物的多重rt-pcr酶杂交测定。市售的六重(hexaplex)测定(prodesse,inc.,milwaukee,wisconsin)(fanetal,clin.infect.dis.1998,26;1397-1402,kehletal.,j.clin.microbiol.2001,39;1696-1701和lioliosetal.,j.clin.microbiol.2001,39;2779-2783)针对副流感病毒1、2和3,呼吸道合胞病毒a和b以及甲型流感病毒和乙型流感病毒,而九重(nineplex)测定包含相同的rna病毒减去副流感2并且呼吸道合胞病毒a和b合并在一个引物对中,但包括肠病毒,腺病毒和两个细菌肺炎支原体和肺炎衣原体(grondahletal.,j.clin.microbiol.1997,37;1-7和puppeetal.,j.clin.virol.2003,30;165-174)。根据病毒,六重测定的分析灵敏度已经显示为100-140个拷贝/ml,而与呼吸道合胞病毒和副流感病毒1的培养相比,九重测定不太灵敏,而对副流感病毒3,甲型流感病毒和乙型流感病毒,腺病毒和肠病毒则更灵敏。对连续稀释的病毒培养上清液测量分析灵敏度。九重测定和六重测定对临床标本的敏感性和特异性在rna病毒之间变化,发现灵敏度在86%-100%之间,特异性为80%-100%。将两种测定与单一rt-pcrelisa和其他单一rt-pcr测试进行比较,并且大致具有相同的质量。尽管基于elisa的测定允许高度多重分析,但它们具有与基于凝胶电泳的测定相同的缺点。测定费力又费时,反应容器必须在pcr后打开,从而增加交叉污染的风险。(iii)使用不同的荧光染料测量发射。荧光报告系统如实时荧光定量pcr最近已被引入诊断实验室中。实时pcr在单个管中将dna扩增与产物的检测相结合。检测是基于与产品增加成正比的荧光变化。实时pcr同时检测多个靶的能力受限于可明确解析的荧光发射峰的数量。目前,只有四种颜色的寡探针多重检测是可能的,其中一种颜色原则上预留用于内部对照以监测抑制,甚至可以充当共同扩增的竞争者。已经开发了针对呼吸道病原体的许多单一或双重(duplex)实时pcr测定,其或基于自制(vaneldenetal.,j.clin.microbiol.2001,39;196-200,huetal,j.clin.microbiol.2003,41;149-154)或市售测定(prodesse,inc.,milwaukee,wisconsin)。其中针对呼吸道病毒的第一多重实时pcr测定之一是由templeton等人(j.clin.microbiol.2004,42;1564-1569)开发的。开发了用于在双管多重反应中检测7个呼吸系统rna病毒(甲型流感病毒和乙型流感病毒,呼吸道合胞病毒,副流感病毒1、2、3和4)的实时多重pcr测定。每个测定最初设置为单一测定,然后合并为两个多重测定:一个包括甲型流感病毒和乙型流感病毒和呼吸道合胞病毒,而另一个包含副流感病毒1、2、3和4,两个测定具有相同的pcr操作方案,因此它们可以平行运行。没有观察到非特异性反应或任何测定间交叉扩增,通过两个多重反应只扩增正确的病毒。通过病毒培养进行临床评价,并对相同的样品通过if和多重pcr进行确认。病毒培养得到19%的阳性样品,而多重测定得到24%的阳性。多重pcr阳性标本包括病毒培养阳性的所有样品和其他样品。通过第二次pcr测定来测试其他样品,并且可以显示这些样品为真阳性。为了通过实时pcr同时检测12个呼吸系统rna病毒,gunson等人(j.clin.virol.2005,33;341-344)开发了四个三重反应:(i)甲型流感病毒和乙型流感病毒和人偏肺病毒,(ii)呼吸道合胞病毒a和b和鼻病毒,(iii)副流感病毒1、2和3和(iv)冠状病毒229e、oc43和nl63。这4个测定几乎涵盖了一套完整的呼吸系统rna病毒,据说这些测定的实施可以改善患者管理、感染控制程序和监控系统的有效性。实时pcr测定允许分析而无需对样品进行任何后pcr处理。这减少了交叉污染的风险,并且不需要额外的处理时间。然而,当前测定的复杂性限于每个反应最多四个探针。此外,复杂的分析需要更多的反应,从而增加成本。(iv)由固定在固体表面上的寡核苷酸或pcr扩增子组成的微阵列。用于诊断目的的微阵列需要(a)基因组特异性探针以捕获未知靶序列,或(b)扩增的靶序列中存在的通用zipcode,从而揭示该病原体在临床标本中的存在。通过扫描阵列表面或对阵列表面进行成像来显示结合的探针和样品中靶序列之间的杂交。dna微阵列提供高度平行病毒筛选以同时检测数百个病毒的可能性。相关病毒血清型可以通过每个病毒产生的独特的杂交模式进行区分。高密度阵列能够区分数万个不同的靶,而至多数百个靶的低密度阵列在临床诊断中更为合适。第一个用于诊断病毒学的阵列是由wang等人(proc.natl.acad.sci.usa99;15678-15692)构建的。他们最初构建了从约140个病毒基因组序列设计的1600个独特的70聚体寡核苷酸探针的微阵列,其中主要关注呼吸道病原体。使用随机标记的pcr程序扩增病毒rna,并且用常见感冒患者的鼻灌洗标本验证该阵列。该阵列检测到含有少至100个感染性颗粒的呼吸系统病原体。使用特异性pcr引物用rt-pcr确认数据。仅观察到其近亲病毒的交叉杂交。已经构建了低密度阵列用于流感(kessleretal.,j.clin.microbiol.2004,42;2173-2185)和急性呼吸道疾病相关腺病毒(linetal.,j.clinmicrobiol.2005,42;3232-3239)的检测、分型和亚型分型。当直接分析临床样品时,流感病毒芯片显示检测到少至1×102至5×102个流感病毒颗粒,而腺病毒微阵列的灵敏度为103个基因组拷贝。使用多重和随机扩增程序。呼吸道病原体识别中的一个非常新的发展是使用重新测序微阵列(linetal.,genomeres.,2006,16:527-535和wangetal,bioinformatics200622(19):2413-2420;doi:10.1093/bioinformatics/btl396)。微生物序列的指数增加的可用性使得可以使用直接测序来进行常规病原体诊断。然而,这需要快速获得病原体序列信息。重新测序微阵列使用平铺的25聚体或29聚体的105至106个探针,靶基因的两条链的每个碱基含有一个完全匹配的探针和三个错配的探针。已经公开了定制设计的affymetrix重新测序呼吸系统病原微阵列(rpmv.1)。该rpmv.1阵列包含14个病毒和细菌物种。使用随机扩增操作方案,并且在两个研究中,不仅在种的水平上而且在株的水平获得鉴定。这对于监测疫情爆发特别有意义。第二个rpm芯片(v.2)的开发已经开始,包括54个细菌和病毒物种。然而,必须改进灵敏度和测定速度,以提供病原体检测的诊断平台。适合临床诊断的dna阵列的最接近的前体是反向杂交线探针或印迹。已经描述了用于突变检测和基因分型的线性探针/印迹测定,并且也可商购获得(来自innogenetics,belgium的线探针测定(lipa))。通常,使用通用扩增技术,但是迄今尚未公开针对呼吸系统病毒病原体的线探针印迹的研究。基于微阵列的测定允许高度复杂的分析。然而,它们需要专门的设备和充分的后pcr处理。(v)珠或微球系统在这些产品中,通过流式细胞仪进行检测。在这样的系统中,用两个光谱不同的荧光染料对微球进行内部染色。使用精确量的这些荧光染料中的每一种,产生由具有特异性光谱性质的100个不同的微球组组成的阵列。由于这种不同的光谱性质,可以组合微球,允许在单个反应中同时测量至多100个不同的靶。对于使用微球的核酸检测,使用标记的pcr扩增的靶dna与携带特异性针对每个靶序列的寡核苷酸捕获探针产物的微球的直接杂交。检测由两个激光器进行,一个用于识别不同的珠组,另一个用于测定特异性靶序列。基于微球的悬浮阵列技术(如xmaptm系统)提供了高通量多重核酸测试的新平台。与平面微阵列相比,它们具有更快的杂交动力学和阵列制备中更大灵活性的优点。最近,已经开发了一种称为tag-ittm平台的新型基于微球的通用阵列平台,并用于检测和区分19个呼吸系统病毒。tag-ittm阵列平台特征在于通用的、最小的交叉杂交标签,用于通过杂交到互补的抗标签偶联的微球上捕获反应产物。luminex平台上的呼吸系统病毒组由tmbiosciences(toronto,canada)开发,并在鼻咽拭子和抽吸物上得到验证。获得96.1%的总体灵敏度,并通过单一pcr和dfa确认数据。另外检测到(294个标本样品中的)12个双重感染。与基于微阵列的测定一样,这些测定允许高度复杂的分析,但需要专门的设备和充分的后pcr处理。(vi)质谱系统这些是其中标签通过uv照射释放并随后通过质谱仪分析的测定。针对病原体的保守区域设计的寡核苷酸引物用5'c6间隔区和氨基己基修饰合成,并通过光可切割链接(masscode)标签共价缀合。已经建立了64个不同标签的文库。个体引物组中的正向和反向引物用不同的分子标签进行标记。特定病原体靶的扩增导致质谱仪中的双重信号,其允许评估特异性。质谱是均匀的溶液测定形式,其允许在单个反应中同时检测多个核酸序列,从而与基于单个反应的检测平台相比,减少了时间、人工和成本。已经开发了一类新的分子标记,称为可切割质谱标签(cmst),用于同时进行数据采集。cmst技术的一个应用称为masscode(qiagen,hilden,germany),并且用于呼吸系统病原体的差异检测。cmst的一般结构是高度模块化的,并且包括对光不稳的接头、质谱灵敏度增强剂和可变质量单位,所有这些都通过围绕中心赖氨酸残基构建的支架连接。cmst通过光可切割接头连接到pcr引物的寡核苷酸的5'-端。增强剂和可变质量单位的组合指定每个个体cmst的最终质量。目前,已经开发了64个不同的masscode标签的文库,并且可以应用各种质谱法电离方法。质谱检测的一大优点是速度。分析只需几秒钟。briese等人(emerg.infect.dis.,2005,11;310-313)开发了一种诊断测定,其包含代表22个呼吸系统病原体的30个基因靶。测试来自库痰、鼻拭子和肺洗液的核酸,并将其与病毒分离和常规嵌套rt-pcr进行比较。获得了一致的结果。每个样品的检测阈值在100到500个拷贝之间。质谱是一种非常快速的技术,使得能够进行高度复杂的分析。然而,这个应用需要专门和昂贵的硬件,目前在标准微生物实验室中并不常见。鉴定和区分rti的病原体的上述多参数方法是向前迈进的一大步,但仍然具有局限性。它们需要太多时间(>10小时),需要大量亲自动手的时间,具有有限的多参数特性和/或需要昂贵的设备或大的设置成本来进行测试。发明概述在本文中,我们描述了一种新的多参数方法来检测和区分临床样本中的各种病原体。本发明属于检测样品中的核酸序列例如检测临床样品中的病原生物的
技术领域:
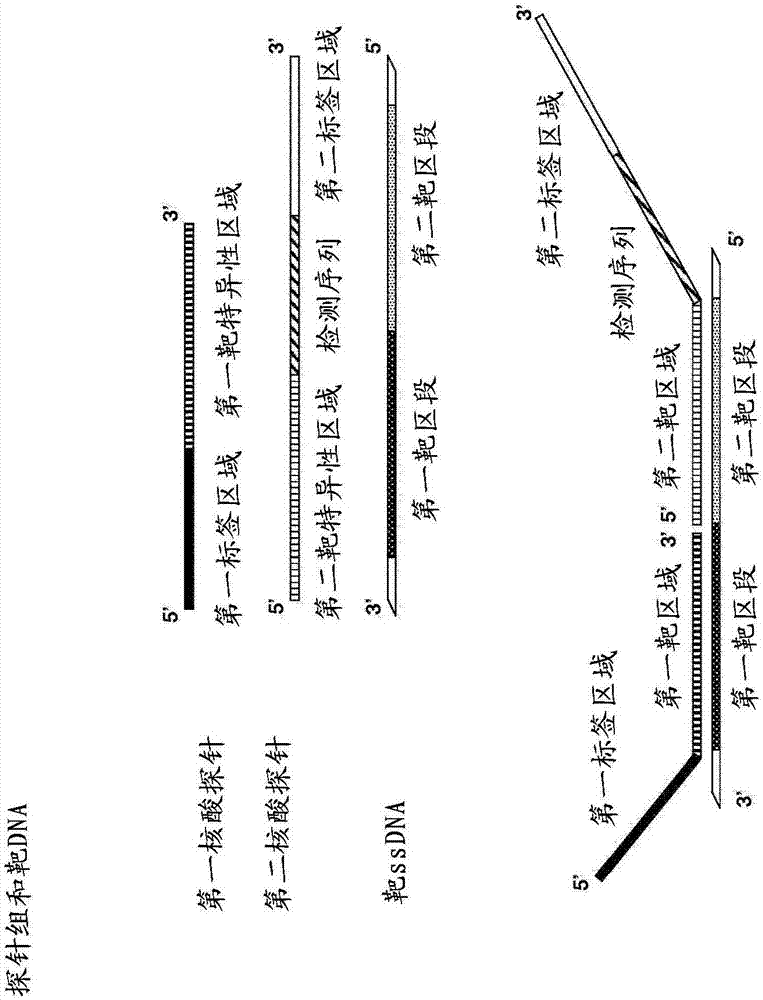
。更具体地,本发明涉及通过扩增和检测来自所述病原生物的特异性核酸序列来检测由临床标本中病原生物如病毒或细菌引起的感染的领域。它提供了可以在单个单管测定中结合实时探针检测来测定约30个不同靶核酸序列的多重测定。本发明涉及能够同时检测样品中的多个不同靶dna模板的方法,每个dna模板包含第一靶区段和第二靶区段,两个靶区段的组合对于特定靶dna模板是特异性的,其中第一和第二靶特异性区段基本上彼此相邻并且其中第一靶区段位于所述第二靶区段的3',所述方法包括以下步骤:a)任选的逆转录和/或预扩增步骤,b)使至少一个dna模板与多个不同探针组接触,每个探针组对于一个靶dna模板是特异性的,并且允许至少一个dna模板与特异性针对该至少一个dna模板的探针组杂交,c)形成包含该特异性探针组的连接的探针组件,d)扩增该连接的探针组件以获得至少一个扩增子,e)通过进行实时解链曲线分析来检测该至少一个扩增子的存在,其中供体或受体标记被并入所述第一或第二标签区域中,基本上与所述检测区域相邻,并且其中通过以下步骤进行步骤e):-提供多个检测探针,其包含:-与并入第一或第二标签区域中的标记互补的至少一个荧光供体标记或至少一个受体标记,-可与所述检测序列特异性杂交的核酸区域-允许该至少一个扩增子与多个检测探针杂交-通过测量受体标记的荧光来监测在至少一个预先选择的温度标记的检测探针的杂交,其中所述标记的检测探针的杂交指示样品中靶dna模板的存在。而且,本发明涉及用于进行这样的方法的试剂盒,其包含:a)多个不同探针组b)dna连接酶活性的源c)dna聚合酶活性的源d)包含至少一个供体标记的至少一个引物e)包含至少一个荧光标记的多个检测探针,f)用于进行该方法的说明。在优选实施方案中,本发明涉及于进行这样的方法的试剂盒,其包含:a)多个不同探针组b)dna聚合酶活性的源c)包含至少一个供体标记的至少一个引物d)包含至少一个荧光标记的多个检测探针,e)用于进行该方法的说明。附图说明图1显示了根据本发明的方法的任选的逆转录和预扩增步骤的示意图。图2显示了在根据本发明的方法中使用的探针组及其与至少一个dna模板的第一和第二靶区段杂交的示意图。图3显示了其中探针组并入连接的探针组件中的步骤的示意图。作为这样一个步骤的优选实例,其显示了杂交探针如何通过连接酶的作用连接。图4提供了从2个不同的dna模板a和b开始形成连接的探针组件的扩增子的示意图。图5显示了对连接的探针组件实时pcr检测的示意图。这里举例说明使用杂交探针的检测。图6显示了使用具有不同解链温度的两种不同探针x和y的解链曲线分析的示意图。这在解链曲线分析中产生了可区分的信号。图7显示了含有人偏肺病毒的临床样品的三个通道的解链曲线分析。人偏肺病毒检测探针用cy5标记并具有70℃的理论解链温度。图8显示了两个样品的解链曲线分析。样品1含有肺炎支原体,样品2含有呼吸道合胞病毒a。两个样品均掺有内部扩增对照。图9显示了含肺炎支原体样品的定量数据。反应1含有未稀释的样品,反应2含有10倍稀释的样品。图10显示了含有甲型流感病毒和肺炎衣原体共感染的样品的解链曲线分析的效果。图11显示了来自含肺炎支原体样品的单管反应的解链曲线分析。图12提供了根据本发明的方法的示意图,其中通过第一和第二核酸探针与其相应的靶dna模板链的同时杂交形成连接的探针组件。图13显示了含有双重感染甲型流感和牛/肠病毒的样品的解链曲线分析的效果。在该实例中连接的探针组件通过第一和第二核酸探针与其相应的靶dna模板链的同时杂交而形成。图14显示了含有腺病毒和甲型流感病毒共感染的样品的解链曲线分析的效果。在该实例中连接的探针组件通过第一和第二核酸探针与其相应的靶dna模板链的同时杂交而形成。图15显示了病原体阴性样品的两个反应的解链曲线分析的效果;阴性结果由内部对照和区分两种反应混合物的不同扩增对照的存在来验证。发明详述在一个方面,本发明涉及能够同时检测样品中的多个不同靶dna模板的方法,每个dna模板包含第一靶区段和第二靶区段,两个靶区段的组合对于特定靶dna模板是特异性的,其中第一和第二靶特异性区段基本上彼此相邻并且其中第一靶区段位于所述第二靶区段的3',所述方法包括以下步骤:a)任选的逆转录和/或预扩增步骤,b)使至少一个dna模板与多个不同探针组接触,每个探针组对于一个靶dna模板是特异性的,并且允许至少一个dna模板与特异性针对该至少一个dna模板的探针组杂交,c)形成包含该特异性探针组的连接的探针组件,d)扩增该连接的探针组件以获得至少一个扩增子,e)通过进行实时解链曲线分析来检测该至少一个扩增子的存在。这样的方法已经被公开于ep1130113a1。其中描述了通过进行实时解链曲线分析检测多重连接依赖性扩增(mlda)获得的扩增子。在这种分析中,提供了许多不同的填充片段,其导致扩增子本身的不同解链行为。然后,这样的扩增子可能例如长度不同,这使得它们易于使用简单的凝胶电泳进行检测。或者,也可以使用一些聚合酶的5'核酸酶活性检测扩增子。公开了其他实时检测方法,其不依赖于寡核苷酸的破坏,而是依赖于使用分子信标。这样的检测需要含有荧光团和淬灭剂的探针。ep1130113中公开的第三种选择是使用由两个实体组成的检测探针,每个实体与扩增子上存在的序列互补,每个含有荧光部分,其中当两个探针实体与扩增子结合后发生荧光共振能量转移。现在已经发现,检测扩增子的另一种方法提供了更可靠和稳健的测定,其允许甚至在单管或双管系统中至多30个不同靶序列的真正的多重检测。这在本文中也被称为与固有地是开放系统的上述现有技术相反的封闭系统。根据本发明的方法提供了更大的自由度来工程改造探针组件,因此导致更可靠的测定,其能够在大量不同扩增子之间更好地区分。在wo2009007438中也已经公开了这样的检测方法,其中本发明的方法特别不同在于用于形成连接的探针组件的方法。在wo2009007438中,通过所使用的探针在与其靶序列杂交时直接连接而形成连接的探针组件。在本发明的方法中,通过将一个探针与其dna对应物和另一个探针与其互补链的杂交而形成组件。事实上,探针组件通过其在反向链上互补序列连接到第二探针。这在本申请的图12中示出。随后,通过反应中使用的聚合酶扩增杂交的探针。除了在本发明的方法中不需要另外的酶如连接酶的优点之外,还有降低的污染风险,因为不应再次打开管以添加这样的另外的连接酶。此外,与wo2009007438的方法相比,发现本发明的方法发生得更快(即约1.5小时),这是分子诊断中的巨大优点,以便对患者进行快速诊断。最后,出乎意料地发现,与使用连接制备探针组件的wo2009007438的方法相比,使用扩增用于制备探针组件的测试的特异性没有显著降低。本发明涉及能够同时检测样品中的多个不同靶dna模板的方法,每个dna模板包含第一靶区段和第二靶区段,两个靶区段的组合对于特定靶dna模板是特异性的,其中第一和第二靶特异性区段基本上彼此相邻并且其中第一靶区段位于所述第二靶区段的3',所述方法包括以下步骤:a)任选的逆转录和/或预扩增步骤,b)使至少一个dna模板与多个不同探针组接触,每个探针组对于一个靶dna模板是特异性的,并且允许至少一个dna模板与特异性针对该至少一个dna模板的探针组杂交,c)形成包含该特异性探针组的连接的探针组件,d)扩增该连接的探针组件以获得至少一个扩增子,e)通过进行实时解链曲线分析来检测该至少一个扩增子的存在,其中供体或受体标记被并入所述第一或第二标签区域中,基本上与所述检测区域相邻,并且其中通过以下步骤进行步骤e):-提供多个检测探针,其包含:-与并入第一或第二标签区域中的标记互补的至少一个荧光供体或至少一个受体标记,-可与所述检测序列特异性杂交的核酸区域-允许该至少一个扩增子与多个检测探针杂交-通过测量受体标记的荧光来监测在至少一个预先选择的温度标记的检测探针的杂交,其中所述标记的检测探针的杂交指示样品中靶dna模板的存在。具体地,本发明提供用于检测和区分临床样品中的多个病源生物的方法,临床样品包含衍生自多个病源生物的多个不同靶rna模板和/或多个不同靶dna模板,每个靶dna模板包含第一靶区段和第二靶区段,第一靶区段和第二靶区段二者的组合对于特定靶dna模板是特异性的,其中第一靶区段和第二靶区段基本上彼此相邻并且其中第一靶区段位于所述第二靶区段的3',所述方法包括以下步骤:(a)多个不同靶rna模板逆转录为多个不同靶dna模板的逆转录步骤和多个不同靶dna模板的预扩增步骤;(b)使所述多个不同靶dna模板与多个不同探针组接触,每个探针组对于多个不同靶dna模板中的特定靶dna模板或其互补物是特异性的,并允许特定靶dna模板与其杂交,每个探针组包括:具有可与所述特定靶dna模板的第一靶区段杂交的第一靶区域和第一标签区域的第一核酸探针,其中所述第一标签区域包含第一标签序列,以及具有可与所述特定靶dna模板的第二靶区段的互补物杂交的第二靶区域和第二标签区域的第二核酸探针,其中所述第二标签区域包含第二标签序列,并且其中所述第一核酸探针或所述第二核酸探针中的至少一个含有位于靶与第一或第二标签序列之间的检测序列;和其中每个标签区域位于相应靶区域的5'(c)形成多个连接的探针组件,其中所述多个连接的探针组件包含多个不同探针组,并且通过将每个探针组的所述第一核酸探针与其相应的靶dna模板链杂交和将每个探针组的所述第二核酸探针与其相应的反向靶dna模板链杂交而形成;(d)扩增多个连接的探针组件以获得多个扩增子,其中通过允许多个连接的探针组件与多个核酸引物对接触来扩增多个连接的探针组件,每个核酸引物对包含:引物1和引物2,其中引物1或引物2中的至少一个在其3'端或其3'端附近包含至少一个内部供体或受体荧光标记,从而在所述多个连接的探针组件扩增后提供多个内部标记的扩增子,其中所述内部供体或受体荧光标记被并入所述第一或第二标签区域中,并且基本上与所述检测序列相邻;和(e)在单个反应容器中检测和区分所述多个内部标记的扩增子,其中通过进行实时解链曲线分析来检测和区分所述多个内部标记的扩增子,所述实时解链曲线分析包括:(1)提供多个标记的检测探针,每个标记的检测探针对于特定内部标记的扩增子的特定检测序列是特异性的,并且包含:与通过所述多个核酸引物对并入第一或第二标签区域中的内部供体或受体荧光标记互补的至少一个荧光供体或受体标记,其中使用单对供体和受体标记来检测多个内部标记的扩增子,其中每个类似标记的检测探针在与特定内部标记的扩增子的特定检测序列杂交后显示不同解链温度,使得所述多个内部标记的扩增子是可区分的;和可与特定内部标记的扩增子的所述特定检测序列特异性杂交的核酸区域,(2)允许所述多个内部标记的扩增子与所述多个标记的检测探针杂交,和(3)通过测量在不同荧光检测通道内预先选择的增加的温度范围内受体标记的荧光来监测所述多个标记的检测探针的杂交和不同解链温度,其中:所述多个标记的检测探针的杂交指示临床样品中多个不同病源生物的存在,不同解链温度与多个标记的检测探针的不同荧光标记的组合允许检测和能够区分临床样品中的多个不同病源生物,从而可以检测和区分临床样品中的至多约30个不同病源生物。在另一具体实施方案中,本发明提供了用于在单个反应容器中在临床样品中检测和区分多个拷贝数小于6000的病源生物的方法,所述临床样品包含多个不同的靶rna模板和/或多个衍生自多种病源生物的不同靶dna模板,每个靶dna模板包含第一靶区段和第二靶区段,第一靶区段和第二靶区段二者的组合对于特定靶dna模板是特异性的,其中所述第一靶区段和所述第二靶区段基本上彼此相邻,并且其中所述第一靶区段位于所述第二靶区段的3',所述方法包括以下步骤:(a)在单个反应容器中的多个不同靶rna模板逆转录为多个不同靶dna模板的逆转录步骤和/或任选的在单个反应容器中的多个不同靶dna模板的预扩增步骤;(b)使所述多个不同靶dna模板在单个反应容器中与多个不同探针组接触,每个探针组对于多个不同靶dna模板中的特定靶dna模板或其互补物是特异性的,并允许特定靶dna模板与其杂交,每个探针组包括:具有可与所述特定靶dna模板的第一靶区段杂交的第一靶区域和第一标签区域的第一核酸探针,其中所述第一标签区域位于所述第一靶区域的5'并且包含第一标签序列,以及具有可与所述特定靶dna模板的第二靶区段杂交的第二靶区域和第二标签区域的第二核酸探针,其中所述第二标签区域位于所述第二靶区域的3'并且包含第二标签序列,并且其中所述第一核酸探针或第二核酸探针中的至少一个含有位于第二标签序列的5'或者位于第一标签序列的3'的检测序列;(c)在单个反应容器中形成多个连接的探针组件,其中所述多个连接的探针组件包含多个不同探针组,并且通过将每个探针组的所述第一核酸探针与其相应的靶dna模板链杂交和将每个探针组的所述第二核酸探针与其相应的反向靶dna模板链杂交而形成;(d)在单个反应容器中扩增多个连接的探针组件以获得多个扩增子,其中通过允许多个连接的探针组件与多个核酸引物对接触来扩增多个连接的探针组件,每个核酸引物对包含:引物1和引物2,其中引物1或引物2中的至少一个在其3'端或其3'端附近包含至少一个内部供体或受体荧光标记,从而在所述多个连接的探针组件扩增后提供多个内部标记的扩增子,其中所述内部供体或受体荧光标记被并入所述第一或第二标签区域中,并且基本上与所述检测序列相邻;和(e)在单个反应容器中检测和区分所述多个内部标记的扩增子,其中通过进行实时解链曲线分析来检测和区分所述多个内部标记的扩增子,所述实时解链曲线分析包括:(1)提供多个标记的检测探针,每个标记的检测探针对于特定内部标记的扩增子的特定检测序列是特异性的,并且包含:与通过所述多个核酸引物对并入第一或第二标签区域中的内部供体或受体荧光标记互补的至少一个荧光供体或受体标记,其中使用单对供体和受体标记来检测多个内部标记的扩增子,其中每个类似标记的检测探针在与特定内部标记的扩增子的特定检测序列杂交后显示不同解链温度,使得所述多个内部标记的扩增子是可区分的;和可与特定内部标记的扩增子的所述特定检测序列特异性杂交的核酸区域,(2)允许所述多个内部标记的扩增子与所述多个标记的检测探针杂交,和(3)通过测量在不同荧光检测通道内预先选择的增加的温度范围内受体标记的荧光来监测所述多个标记的检测探针的杂交和不同解链温度,其中:所述多个标记的检测探针的杂交指示临床样品中多个不同病源生物的存在,不同解链温度与多个标记的检测探针的不同荧光标记的组合允许检测和能够区分临床样品中的多个不同病源生物,从而可以检测和区分临床样品中的至多约30个不同病源生物;并且步骤(a)-(e)在封闭系统中进行。根据本发明的方法能够同时检测样品中的多个不同靶dna模板。这意味着该方法具有同时检测样品中多个靶dna模板的潜力。如果样品只含有一个靶dna模板,则该方法当然检测该一个模板,然后不使用特异性针对其他模板的探针。在许多临床样品中,足够拷贝的dna模板可用于进行根据本发明的方法而无需任选的预扩增步骤。然而,在一些临床样品中,没有足够拷贝的dna模板可供使用。在这种情况下,必须进行任选的预扩增步骤。此外,当模板是rna模板时,必须通过逆转录步骤将其转化为dna模板。这两个步骤是本领域已知的,本领域技术人员会知晓进行它们的方式。该步骤的示意图在图1中提供。在sambrooketal.,2000.molecularcloning:alaboratorymanual(thirdedition)coldspringharborlaboratorypress中可以找到另外的指导。然后通过使至少一个dna模板与多个不同的探针组接触来进行根据本发明的方法(图2)。该多个探针组是特异性针对一组特定dna模板的预定探针组。可以基于经常在临床疾病中一起发现的病原体的多样性有利地选择探针组。例如,对于呼吸道感染的筛查和分型,可以有利地将特异性针对甲型流感病毒和乙型流感病毒,副流感病毒1、2、3和4,呼吸道合胞病毒a和b,鼻病毒,冠状病毒229e,oc43和nl63,人偏肺病毒,腺病毒,肺炎支原体,肺炎衣原体,嗜肺军团菌和百日咳鲍特菌的探针组进行组合。此外,该方法可有利地用于检测感染,如血液中的感染。例如,为了对血液相关病毒进行筛查和分型,组合探针组以同时检测人免疫缺陷病毒,乙型肝炎病毒和丙型肝炎病毒可能是有利的。如果样品中存在针对其设计的探针组的一个或多个dna模板,那么特异性针对靶dna模板的探针组将与dna模板的第一和第二靶区段进行特异性杂交(图2)。本领域技术人员将知晓在dna模板上选择可用作第一和第二靶区段的合适区域时适用的限制。最重要的是,这应该是保守区域,使得模板的自然变异性不会造成假阴性结果。在schoutenetal.,wo01/61033,schoutenetal.,nucl.acidsres.2002,vol30no12;e57和sambrooketal.,2000.molecularcloning:alaboratorymanual(thirdedition)coldspringharborlaboratorypress中可以找到关于靶区域选择的另外的指导。然后允许特异性探针组形成连接的探针组件(图3、12)。这可以通过连接酶链反应实现,其导致连接的探针组件的多个拷贝,或通过单个连接酶步骤实现(如图3所示),然后扩增连接的探针组件(图4)。或者,连接的探针组件可以通过第一和第二核酸探针与其相应的靶dna模板链的同时杂交形成(图12)。在本文中,使每个探针组的第一核酸探针与第一靶dna模板链杂交,并使每个探针组的第二核酸探针与其反向靶dna模板链杂交。然后,在随后将扩增引物杂交到所述探针的标签区域之后,连接的探针组件被扩增。当进行单个连接酶步骤时,选择不太低的温度,即50至65℃可能是有利的。t4连接酶在42至47℃的温度下进行,因此在需要高测定特异性时不太适合。使用最小的可能体积也是有利的。温度稳定和对温度不稳定的连接酶同等地适用。在schoutenetal.,wo01/61033,schoutenetal.,nucl.acidsres.2002,vol30no12;e57和sambrooketal.,2000.molecularcloning:alaboratorymanual(thirdedition)coldspringharborlaboratorypress中可找到选择合适的连接酶及其使用条件的另外的指导。扩增的连接探针组件的检测有利地实时进行(图5),优选在封闭系统中进行,例如在实时pcr装置中使用荧光共振能量转移(fret)探针。为此,可以进行解链曲线分析(图6)。在这种情况下,随着温度的升高监测荧光,当探针解链离开时,获得荧光的降低(调查参见mackayetal2002,nucleicacidsres.30;1292-1305)。根据本发明的方法,即使与mlpa相比也具有优异的灵敏度。标准mlpa需要大约6000个单拷贝靶来获得可重现的结果。源自病毒和细菌感染患者的样品平均含有少得多的拷贝。更详细地并且如图2所示,本发明涉及如上所述的方法,其中步骤b)是通过使至少一个dna模板与多个不同的探针组接触而进行的,每个探针组特异性针对一个靶dna模板并允许所述至少一个dna模板与特异性针对所述至少一个dna模板的探针组杂交,并且每个探针组包含:-第一核酸探针,其具有-可与第一靶区段杂交的第一靶区域-包含第一标签序列的第一标签区域,其在第一靶区域的5'-第二核酸探针,其具有-可与第二靶区段杂交的第二靶区域-包含第二标签序列的第二标签区域,其在第二靶区域的3'其中所述第一和第二核酸探针中的至少一个含有位于所述第二标签序列的5'或位于所述第一标签序列的3'的检测序列。在另一具体实施方案中并且如图12所示,本发明涉及如上所述的方法,其中步骤b)是通过使至少一个dna模板与多个不同的探针组接触而进行的,每个探针组特异性针对一个靶dna模板并允许所述至少一个dna模板与特异性针对所述至少一个dna模板的探针组杂交,并且每个探针组包含:-第一核酸探针,其具有-可与第一靶区段杂交的第一靶区域-包含第一标签序列的第一标签区域,其在第一靶区域的5'-第二核酸探针,其具有-可与第二靶区段杂交的第二靶区域-包含第二标签序列的第二标签区域,其在第二靶区域的5'其中所述第一和第二核酸探针中的至少一个含有位于标签和靶序列之间的检测序列。本发明还涉及如上所述的方法,其中步骤c)是通过使第一和第二核酸探针如与所述靶dna模板杂交则彼此共价连接从而形成侧接第一和第二标签区域的至少一个连接的探针组件而进行的。本发明还涉及如上所述的方法,其中步骤c)是通过使第一和第二核酸探针与所述靶dna模板杂交从而形成侧接各自第一和第二标签区域的包含靶dna模板和第一和第二核酸探针的至少一个连接的探针组件而进行的。在本文中,允许每个探针组的第一核酸探针与第一靶dna模板链杂交,允许每个探针组的第二核酸探针与其反向靶dna模板链杂交。本发明还涉及如上所述的方法,其中通过以下步骤进行步骤d):-允许所述至少一个连接的探针组件与包含引物1和引物2的核酸引物对接触,其中-引物1包含可与第一标签序列的互补序列杂交的第一核酸序列,和-引物2包含可与第二标签序列杂交的第二核酸序列-扩增所述至少一个连接的探针组件以获得至少一个扩增子,所述扩增子包含-第一标签区域或其至少一部分-第一靶区域-第二靶区域-检测区域-第二标签区域或其至少一部分或其互补物。本发明还涉及如上所述的方法,其中通过以下步骤进行步骤e):-通过以下步骤检测所述至少一个扩增子的存在-提供多个检测探针,所述检测探针包含-至少一个荧光标记-可与所述检测序列特异性杂交的核酸区域-使所述至少一个扩增子与所述多个检测探针杂交-通过测量标记的荧光来监测在至少一个预先选择的温度检测探针的杂交,其中所述检测探针的杂交指示样品中靶dna模板的存在。如本文所使用的短语“同时检测”表示可以在一个并且同一分析中检测多个、即大于1个不同的靶。为此目的,根据本发明的方法为每个待扩增的核酸模板提供不同的靶区域对。这预先假定靶dna模板的核酸序列的至少一部分,例如dna或rna病毒或细菌的基因组是已知的。从该已知的核酸序列,本领域技术人员可以选择合适的第一和第二靶区段用于特异性探针的杂交。第一和第二靶区段优选足够长(典型地约20至40个核酸)以允许探针在升高的温度下进行杂交和退火。本领域技术人员会注意第一和第二靶区段彼此充分不同以允许与探针组的特异性杂交。出于相同的原因,本领域技术人员将从待检测的各种核酸模板中选择足够不同的第一和第二靶区段。用于这样做的手段和方法在本领域中是容易获得的并且是本领域技术人员已知的。在schoutenetal.,wo01/61033,schoutenetal.,nucl.acidsres.2002,vol30no12;e57和sambrooketal.,2000.molecularcloning:alaboratorymanual(thirdedition)coldspringharborlaboratorypress中可以找到选择靶序列的另外的指导。基本上相邻地选择第一和第二靶区段。这意味着与这些区域杂交的第一和第二核酸探针定位成使得当存在合适的连接酶时它们可以容易地共价偶联。为了允许通过连接基本相邻的探针的连接,一种可能性是产生在杂交后不留下缺口的探针。然而,还可以提供与所述靶核酸的至少一个相邻部分互补的至少一个另外的单链核酸,由此所述附加核酸与所述相邻部分的杂交允许两个相邻探针的连接。在本发明的该实施方案中,通过所述另外的单链核酸的杂交来填充探针与靶核酸杂交后的缺口。连接和扩增后,得到的扩增子将包含所述另外的单链核酸的序列。可以选择使所述相邻部分相对较小,从而导致所述靶核酸的所述一个相邻部分与包含与所述靶核酸的所述相邻部分同源但包含差异在一个或多个核苷酸的序列的核酸之间的杂交效率的差异增加。在本发明的另一个实施方案中,在所述连接之前,通过延伸扩增杂交探针或相邻部分的另外的核酸填充部分的3'端来填充所述靶核酸上探针之间的缺口。如本文所用,在核酸杂交的上下文下,术语“互补”或“互补核酸”是指在正常杂交条件下能够与另一核酸杂交的核酸。它可能会在少数几个位点出现错配。在荧光标签的上下文中,术语“互补”是指供体或受体标记。供体标记与受体标记互补,反之亦然。如本文所用,“寡核苷酸”表示长度在10至至少800个核苷酸之间的任何短区段核酸。可以在包括化学合成、质粒或噬菌体dna的限制性内切核酸酶消化、dna复制、逆转录或其组合的任何事件中产生寡核苷酸。可以例如通过添加甲基、生物素或地高辛部分、荧光标签或通过使用放射性核苷酸修饰一个或多个核苷酸。如本文所用,术语“引物”是指无论是如在纯化限制性消化物中天然存在还是以合成方式产生的寡核苷酸,当其置于合成诱导与核酸链互补的引物延伸产物的条件下,即在适当的缓冲液(“缓冲液”包括ph,离子强度,辅因子等)的不同核苷酸三磷酸和聚合酶存在下,并在合适的温度下,其能够作为核酸序列合成起始点。可以例如通过添加甲基、生物素或地高辛部分、荧光标签或通过使用放射性核苷酸修饰引物的一个或多个核苷酸。引物序列不需要反映模板的确切序列。例如,非互补核苷酸片段可以连接到引物的5'端,引物序列的其余部分与链基本上互补。在上述步骤d)中获得的扩增子有利地包含第一和第二标签区域。或者,取决于所使用的引物对长度,扩增子也可以仅含有第一和第二标签区域的一部分。包含在扩增子中的所述第一和第二标签区的一部分的最小长度对应于所使用的引物长度。如本文所用,术语“靶序列”是指待分析样品中待检测和/或定量的特异性核酸序列。如本文所用,术语“热启动”是指用于在扩增反应中防止聚合酶活性直到达到一定温度的方法。如本文所用,术语“限制性内切核酸酶”和“限制酶”是指各自在特异性核苷酸序列处或其附近切割双链dna的细菌酶。如本文所用,术语“pcr”是指聚合酶链反应(mulis等人的美国专利号4,683,195、4,683,202和4,800,159)。pcr扩增过程导致长度由寡核苷酸引物的5'端限定的离散dna片段的指数增加。如本文所用,术语“杂交”和“退火”用于指互补核酸的配对。在本领域技术中的分子生物学和重组dna技术的常规技术在文献中得到充分解释。参见例如sambrook,fritschandmaniatis,molecularcloning;alaboratorymanual,secondedition(1989)及系列methodsinenzymology(academicpress,inc.)和sambrooketal.,2000.molecularcloning:alaboratorymanual(thirdedition)coldspringharborlaboratorypress。根据本发明的方法的描述不应该如此狭窄地解释为不能检测其它核酸模板,例如rna模板。本领域技术人员将理解本发明的公开内容为包括rna序列例如rna模板的扩增,例如通过引入预扩增步骤,以产生含有对应于样品例如临床样品中的rna序列的dna模板的样品。由此获得的dna样品可以用于如上所述的方法。因此,本文公开的根据本发明的方法允许检测可以通过扩增样品例如临床样品中多个不同核酸如dna或rna而获得的多个不同dna模板。当多个不同核酸衍生自诸如细菌、病毒、藻类、寄生虫、酵母和真菌的微生物时,这样的方法可能是特别有利的。如果这样的微生物是病原性的,可以检测各种不同的核酸模板的快速可靠的方法可能是特别有利的。在许多情况下,样品可能含有干扰随后的扩增和/或检测步骤的物质。在根据本发明的方法中,可以通过在杂交前从样品中提取多个dna模板来避免这种干扰。因此,本发明涉及如上所述的方法,其中在允许dna模板与多个不同探针杂交之前,从样品中提取多个dna模板。在根据本发明的方法中,使用包含在第一和第二核酸探针中的标签序列来扩增第一和第二靶特异性序列。当以使得它们不与待检测的任何靶dna模板杂交的方式选择这些标签序列是有利的。应当注意,这不是强制性的,并且该方法也可以在没有这种修改的情况下进行。然而,在如上上述的方法中改进了该方法的灵敏度和特异性,其中第一和第二标签序列中的至少一个具有以使得第一和第二标签序列不与多个不同靶序列杂交的方式选择的核酸序列。通常,合适的标签序列具有约50的g/c含量,约20至30个核苷酸的长度和60至80℃的tm。为了提高该方法的特异性和灵敏度,还可以特别有利的是,不仅第一和/或第二标签序列不与待检测的任何靶dna模板杂交,而且整个标签区域不杂交。因此,根据本发明的方法涉及如上所述的方法,其中第一标签区域和第二标签区域中的至少一个具有以使得第一和第二标签区域不与多个不同靶序列杂交的方式选择的核酸序列。根据本发明方法的该实施方案中的标签序列用于扩增连接的探针组件的目的。多个不同探针组的标签序列可以全部不同;然而,为了方便起见,标签序列可以有利地是通用的,使得每个不同的连接的探针组件可以用一个并且同一个核酸引物对扩增。因此,在一个实施方案中,本发明涉及如上所述的方法,其中第一和第二标签序列中的至少一个是通用序列。为了避免疑义,上述描述意味着所有第一标签序列是相同的,并且所有第二标签序列是相同的,并不必然第一和第二序列是相同的,尽管在不影响该方法有用性的情况下也可以是这种情况。当待连接的两个寡核苷酸与靶核酸杂交时,两个片段之间的共价磷酸酯连接可以通过连接酶酶促形成。尽管共价偶联两种核酸的其它方法是可用的(例如连接酶链反应),但是当第一和第二探针通过连接酶彼此连接时,本发明的方法是最有利地进行的。因此,在具体实施方案中,本发明涉及如上所述的方法,其中所述第一和第二核酸探针彼此共价连接,以便通过具有连接酶活性的酶形成所述至少一个连接的探针组件。在具体实施方案中,本发明还涉及如上所述的方法,其中所述第一和第二核酸探针与其相应的dna靶模板杂交以形成所述至少一个连接的探针组件。在这里,使每个探针组的第一核酸探针与第一靶dna模板链杂交,并使每个探针组的第二核酸探针与其反向靶dna模板链杂交。在一个实施方案中,可以使用与空间上彼此接近但不相邻的模板杂交的探针,以允许探针的最接近连接。在这种情况下,在3'端具有靶特异性序列的探针可以通过聚合酶在合适的缓冲液和四种dntp的存在下伸长,以使两个探针连接成为可能。作为替代,探针之间的缺口可以由可以连接到探针的互补寡核苷酸填充。dna连接酶是能够在互补链上的相邻位点处结合的两个寡核苷酸之间形成共价磷酸酯连接的酶。这些酶使用nad或atp作为辅因子来密合双链dna中的切口。或者,修饰的dna端的化学自动连接可用于连接在互补链上的相邻位点处结合的两个寡核苷酸(xu,y.&kool,e.t.(1999),nucleicacidres.27,875-881)。还可以设想,根据本发明的方法可以通过rna或dna引物进行。为了方便起见,dna引物更经常使用,因为它们比rna引物更稳定。因此,本发明涉及如上所述的方法,其中核酸引物1和引物2中的至少一个是dna引物。检测序列可以位于两个标签序列之间的任何位置;然而,如果检测序列被设计为与第一或第二标签序列基本上相邻,则其具有干扰杂交和/或连接反应的机会最小。检测序列的这种定位具有额外的优点,即检测序列可用于涉及fret探针的检测反应中。因此,根据本发明的方法可以被表征为如上所述的方法,其中检测序列与第一或第二标签序列基本上相邻。在这方面,术语“基本上相邻”是指当与检测序列杂交的端标记探针能够与标签序列端的标签能量转移时,可能发生能量转移。更优选的是,检测序列紧邻第二标签序列。在后一种情况下,从内部标记的探针组件到荧光标记的检测探针的能量转移是最有效的。通常,当标记之间的距离小于5至10个、优选小于5例如4、3、2、1或0个核苷酸时,能量转移是最佳的。检测序列可以是以除了检测探针之外其不干扰该方法中使用的任何其它试剂的方式选择的随机序列。有利地,检测序列以检测探针的tm在50至75℃的方式来选择。这样的检测方法需要使用内部标记的扩增区域或扩增子。这样的扩增子可以通过在根据本发明的方法中提供标记的引物获得。然后,可以用供体或受体荧光标记对这些引物进行标记;相反,荧光标记的检测探针当然应该包含互补供体或受体荧光标记。因此,本发明还涉及如上所述的方法,其中引物1或引物2中的至少一个在其3'端或其3'端附近包含至少一个内部供体或受体荧光标记,从而在扩增所述至少一个连接的探针组件后提供至少一个内部标记的扩增子。最好使标记位于引物的3'端,因为然后它将与检测探针的互补标记最接近接触。实例提供了一种方法,其中引物2在其3'端包含内部标记。此外,还例举了一种方法,其中所述至少一个内部标记的扩增子由所述多个检测探针检测,所述检测探针在其3'端或其3'端附近提供有至少一个互补供体或受体荧光标记。为了检测相邻的荧光分子,可以激发供体荧光标记,并且可以测量受体的荧光。仍然在反应容器中的非相邻供体和受体标记不会对信号有贡献,因为供体和受体之间的距离太大。以这种方式,仅检测到允许形成内部标记的探针组件的那些dna模板(图5)。因此,根据本发明的方法允许同时实时检测多个不同的dna模板。因此,根据本发明的方法可以表征为如上所述的方法,其中至少一个内部标记的扩增子的检测包括激发供体荧光标记并测量受体荧光标记的荧光的步骤。在具体实施方案中,根据本发明的方法还提供了通过以使得它们可以通过与检测序列杂交时其解链温度的差异来区分的方式选择检测探针来区分多个连接的探针组件的机会。这可以通过设计具有不同长度或核苷酸组成的检测探针来实现。因此,本发明涉及如上所述的方法,其中以使得当与检测序列杂交时各个检测探针可以通过它们在解链温度上的差异来彼此区分的方式选择所述多个检测探针。该实施方案使得可以检测到的靶dna模板的数目成倍。在实时测定中;相差只有几摄氏度的探针的解链曲线可以很容易地区分开来。在这里,我们举例说明了使用解链温度相差3至5摄氏度的探针。因此,在有利的实施方案中,本发明涉及如上所述的方法,其中所述至少一个预先选择的温度由至少3摄氏度的温度范围组成,并且其中进行解链曲线分析以检测检测探针去杂交时荧光的降低。为了进一步增加可能被检测到的靶dna模板的数量,可以使用不同的荧光标记。因此,本发明涉及如上所述的方法,其中不同的荧光标记用于增加待检测的不同靶dna模板的数量。为了监测从初级样品提取核酸的效率,并监测可能的预扩增反应的效率,可以在提取前将内部对照(ic)添加到初级样品中。ic应包含已知序列的核酸,其不存在于待检测的任何病原体中,也不存在于可能存在于初级样品中的任何其它序列中。以与可能存在于样品中病原体相同的方式检测ic序列。如果样品对于测定中可能检测到的病原体是阴性的,则ic应仍然是阳性的,以确保(i)来自样品的核酸提取的充分性,和(ii)当进行时反应的适当进展。因此,阴性结果得到验证。调整添加到样品中ic的量,使得排除被ic竞争掉量小的病原体核酸的假阴性结果。因此,如果样品中存在病原体,则可能出现ic的异常竞争并被允许,并且当检测到病原体但是ic没有检测到,则认为该反应是有效的。如果待检测的靶序列的数量超过可通过最大数量的不同解链温度和/或可用的荧光标记的最大数目获得的多重检测能力,则用特定样品进行的反应数量可以翻倍、三倍、四倍等,这导致双倍、三倍、四倍等多重检测能力。多重反应的数量可以遵循预扩增步骤,其含有在所有反应中检测到的所有病原体的预扩增引物。或者,如果期望的多重检测超过预扩增步骤的多重检测能力,则该预扩增步骤也可以分成多于一个分开的反应,每个反应含有一个或多个病原体特异性引物对。为了简化解链峰的制造和解释,检测探针的荧光标记和解链温度在每个最终反应中可以是相同的,而给定的检测序列可以与不同反应的不同靶特异性序列连接。另外,直接扩增所述初级反应产物的引物1和2可以在不同反应之间相同。为了防止导致误诊的用户混合不同反应,每个反应可以含有包含第一和第二标签区域以及在每个反应之间不同的检测区的合成核酸(从这里开始称为扩增对照(ac)),导致通过其可以区分不同反应的这些扩增对照的不同解链温度。此外,当必要时,ac可以包含部分随机或非病原性序列。当在单个预扩增步骤之后进行多个最终反应时,病原体存在于样品中,并且如上所述在预扩增步骤期间发生ic的完全异常竞争,ic信号将不存在于每个最终反应中。因此,仅在含有特异性针对存在的病原体的内引物的特定反应混合物中才能观察到阳性信号,其他反应混合物根本不显示任何信号。由于没有来自其他反应混合物的信号,ic的竞争异常和由于其他原因导致的不适当的反应进展无法区分。然而,反应混合物中存在的ac信号确保了该反应的适当进展。ac仅在反应期间存在,并且不在预扩增步骤期间存在;因此它们对于预扩增步骤期间的可能出现的异常竞争并不是灵敏的。在某个实施方案中,软件工具可以具有试剂盒,以允许对解链峰的明确和用户独立的解释以及与所检测的解链峰相关联的病原体的自动分配。在上述多重反应混合物的情况下使用软件工具时,软件工具应能够检测特定样品位置中包含的反应混合物。这通过上述ac在每个反应中显示不同的解链峰来实现。为了避免污染,限制在最终检测发生之前必须打开反应容器的次数可能是有利的。由于本发明中采用的步骤的具体数量和顺序以及反应条件,我们能够将连续步骤的数目限制在至多两个(预扩增步骤和实际反应)。在优选实施方案中,根据本发明的方法甚至可以在单个封闭的反应容器中进行,从而消除任何污染的风险。因此,本发明涉及如上所述的方法,其中杂交和连接步骤在一个反应容器中进行。或者,本发明涉及如上所述的方法,其中预扩增和实际反应在一个反应容器中进行。本发明人已经发现,本方法的第一杂交步骤可以在低盐浓度下和mg离子优选mgcl2存在下进行,从而允许连接反应在与杂交反应相同的反应容器中进行。更具体地,本发明涉及如上所述的方法,其中杂交步骤在mg离子的存在下并且在小于200mmkcl的存在下进行。此外,扩增和连接步骤可以在一个反应容器中进行。如果甚至条件进一步得到优化,甚至在一个反应容器中也可以进行整个反应。对于这种单管反应,以下条件是有利的。对于扩增步骤,优选使用热启动酶。这具有提供更长的激活期的优点,使得扩增过程允许足够的时间进行杂交和连接步骤而不干扰扩增步骤。这进一步增加了该方法的可靠性并且允许更加增加的灵敏度。因此,本发明涉及如上所述的方法,其中热启动酶用于扩增步骤。单管或一步反应通常也被称为封闭系统。该术语用于表示系统产生扩增子,而不需要重新打开容器用于添加试剂,也可以指在同一容器中检测扩增子而不需要在扩增步骤后重新打开容器。优选地,它是指在加入起始试剂后不需要打开容器一次即可产生和检测扩增子的系统。在本发明的上下文中,一步反应是指在一个反应容器中至少进行步骤b、c、d和e的反应,而不需要在这些步骤之间打开容器,例如,用于添加或除去反应化合物。双管或两步反应可以被认为是半封闭系统。该术语表示如上所述的反应可以在其中反应容器在步骤b至e之间仅打开一次的单个反应容器中进行。优选在步骤b和c之间完成。一步测定的mg浓度优选选择在0.5和5mm之间,并且nacl浓度在0和250mm之间是最佳的,优选在25和150mm之间。当体积保持尽可能小,例如5至25微升时也是有利的。因此,本发明涉及如上所述的方法,其中杂交、连接和扩增步骤在一个反应容器中进行。甚至被证明可以在单个反应容器中进行整个方法,即可以组合杂交、连接、扩增和检测步骤,而不需要打开反应容器甚至仅一次。因此,本发明涉及如上所述的方法,其中检测步骤与扩增步骤在同一容器中进行。根据本发明的方法可有利地用于任何应用,其中必须检测和/或鉴定病原体,例如病毒,细菌,真菌,酵母,藻类,原生动物和多细胞寄生虫,以及正常和突变的宿主序列及其任何组合。它也可以用于任何应用,其中必须对病原体例如病毒,细菌,真菌,寄生虫进行分型。它也可以用于任何应用,其中必须对病原体例如病毒,细菌,真菌,寄生虫或宿主筛选特异性遗传特征。具体的应用可见于疟疾患者血液中疟原虫物种的筛查和分型,疟蚊物种的检测和它们宿主倾向性的确定以及疟原虫物种子孢子的筛查,蜱叮咬相关物种如ehrlichia和anaplasma物种的筛查和分型,怀孕妇女中与胎儿异常相关的病原体如巨细胞病毒(cmv)和其他人疱疹病毒(hhv),细小病毒b19,弓形虫(toxoplasmagondii),梅毒螺旋体(treponemapallidum)和风疹病毒的筛查和分型,艰难梭菌(clostridiumdifficile)的筛查和分型,脑脊液中的脑膜炎相关病原体如hhv1至8,肠病毒,parechoviruslisteriamonocytogenes,葡萄球菌属(staphylococcus)物种,流感嗜血杆菌(haemophilusinfluenzae),肺炎链球菌(streptococcuspneumoniae),无乳链球菌(streptococcusagalactiae),脑膜炎奈瑟氏球菌(neisseriameningitidis),伯氏疏螺旋体(borreliaburgdorferisensulato),肺炎克雷伯菌(klebsiellapneumonia)k1,新型隐球菌(cryptococcusneoformans),cryptococcusgattii和结核分枝杆菌(mycobacteriumtuberculosis)的筛查和分型,脐带血中病毒感染如人疱疹病毒(hhv)和eb病毒(ebv)的筛查和分型,地下水中人肠病毒的筛查和分型,血液传播病毒像人免疫缺陷病毒,乙型肝炎病毒和丙型肝炎病毒的筛查和分型,流感病毒的筛查和(亚)分型,人免疫缺陷病毒和肝炎病毒中的抗性相关多态性的筛查,分枝杆菌物种的筛查和分型和结核分枝杆菌复合物成员的鉴定,人乳头状瘤病毒的筛查和分型,对受水稻tungro疾病影响的水稻植物上的病毒如ricetungrobaccilliformvirus,ricetungrosphericalvirus的筛查和分型,百日咳鲍特菌(bordetellapertussis)菌株的筛查和分型和对于特定的遗传性质的筛查,通常与性传播疾病相关的病毒、细菌和寄生虫如人乳头瘤病毒,沙眼衣原体(chlamydiatrachomatis),淋病奈瑟氏球菌(neisseriagonorrhoeae),生殖支原体(mycoplasmagenitalium),阴道毛滴虫(trichomonasvaginalis),梅毒螺旋体的筛查和分型,通常与皮肤癣菌病相关的皮肤癣菌物种的筛查和分型,耐甲氧西林金黄色葡萄球菌(staphylococcusaureus)的筛查和分型,通常与食源性感染和毒素相关的沙门氏菌属(salmonellaspp.)、弯曲菌属(campylobacterspp.)、诺如病毒等病原体的筛查和分型,通常与牛和猪呼吸系统感染相关的病原体的筛查和分型,牡蛎样品中的病毒如诺如病毒,轮状病毒,星状病毒,甲型肝炎病毒和肠病毒的筛查和分型,通常与龋齿相关的细菌如突变型链球菌(streptococci)和乳杆菌(lactobacilli)的筛查和分型,热带病(例如非洲锥虫病,登革热,利什曼病,血吸虫病,查加斯病,麻风病,淋巴丝虫病,霍乱,黄热病等)的筛查和分型,人畜共患病(例如沙门氏菌病和弯曲菌病,布鲁氏菌病,狂犬病,钩端螺旋体病,志贺氏菌病,棘球蚴病,弓形虫病等)的筛查和分型,通常与胃肠炎相关的病原体(例如轮状病毒,诺如病毒,腺病毒,札幌病毒,星状病毒等)的筛查和分型,以及多药耐药细菌菌株的筛查和分型。用于检测衍生自病原体的dna模板的诊断方法通常以试剂盒的形式提供。因此,本发明还涉及一种用于进行如上所述的方法的试剂盒,其包含:a)多个不同的探针组b)dna连接酶活性的源c)dna聚合酶活性的源d)包含至少一个供体标记的至少一个引物e)包含至少一个荧光标记的多个检测探针,f)用于进行该方法的说明。取决于所选择的具体实施方案,试剂盒可以包含额外的探针,引物,标记和未标记,如具体实施方案所要求的。特别有利的试剂盒包含具有不同解链温度的检测探针,优选地,这些检测探针具有彼此至少3摄氏度不同的解链温度。实施例提供以下实施例以帮助理解本发明而不意图限制本发明。在不脱离本发明的精神的情况下,对所阐述的方法进行修改目前在本领域技术人员的可及范围内。在下面的实施例中,描述了能够同时检测临床样品中的多个不同靶的方法。当至少部分组合时,这些具体实施例形成根据本发明的检测临床样品、在这种情况下是鼻咽灌洗液中多个不同病原体(pathogen或diseaseagent)的测定。该方法能够同时检测选自甲型和乙型流感病毒,呼吸道合胞病毒a和b,人偏肺病毒,肺炎衣原体,肺炎支原体和嗜肺军团菌中的至少两种病原体。实施例1-9表示根据本发明第一实施方案的方法的实验设置和结果,其中通过连接第一和第二核酸探针形成连接的探针组件。实施例10-17表示根据本发明第二实施方案的方法的实验设置和结果,其中通过将第一和第二核酸探针与其相应的靶dna模板链杂交形成连接的探针组件。在这里,使每个探针组的第一核酸探针与第一靶dna模板链杂交,并使每个探针组的第二核酸探针与其反向靶dna模板链杂交。实施例1探针和引物的设计病毒和细菌序列获自genbank和losalamos数据库。对于每个病毒的一组序列进行比对(clustalxv.1.8.1)以鉴定高度保守的区域。由于一些靶病原体是rna病毒,因此进行逆转录酶步骤之后进行预扩增步骤,以获得多个不同的靶dna模板。基于这些高度保守的区域,设计了逆转录酶(rt)-pcr引物和连接探针。表1列出了作为第一和第二核酸探针杂交的模板的序列的靶区域、genbank登录号和位置。选择基质蛋白基因(m1)中的保守区域作为扩增和检测甲型流感病毒的靶。这些引物适用于各种菌株的扩增,包括但不限于,h3n2,h2n2,h4n2,h3n8,h4n6,h6n3,h5n2,h3n6,h6n8,h5n8,h1n1,h7n1,h6n2,h9n2,h6n1,h7n3,h11n1,h5n3,h8n4,h5n1,h4n9,h4n8,h4n1,h10n8,h10n7,h11n6,h12n4,h11n9,h6n6,h2n9,h7n7,h10n5,h12n5,h11n2。还选择了基质蛋白基因(m1)中的保守区域作为扩增和检测乙型流感病毒的靶。选择主要核衣壳蛋白基因(n)中的保守区域作为扩增和检测呼吸道合胞病毒a和b以及人偏肺病毒的靶。用于扩增人偏肺病毒的引物和用于其检测的探针适用于扩增和检测所有四个遗传谱系(a1,a2,b1,b2)。选择主要外膜基因(ompa)的保守区域作为扩增和检测肺炎衣原体的靶。选择细胞粘附素(cytadhesin)p1基因(p1)中的保守区域作为扩增和检测肺炎支原体的靶。选择巨噬细胞感染增强子基因(mip)中的保守区域作为扩增和检测嗜肺军团菌的靶。选择多聚蛋白基因(pp)中的保守区域作为用作内部对照的脑心肌炎病毒的扩增和检测的靶。表1.呼吸系统病毒和细菌的靶基因表2列出了用于逆转录和后续预扩增的rt-pcr引物(正向和反向)的序列。表2.呼吸系统病毒和细菌的rt-pcr引物所使用的第一和第二核酸探针的序列列于表3中。如上表1对于引物所示,这些探针组适用于检测各种菌株。例如,流感探针适用于扩增各种菌株,包括但不限于,h3n2,h2n2,h4n2,h3n8,h4n6,h6n3,h5n2,h3n6,h6n8,h5n8,h1n1,h7n1,h6n2,h9n2,h6n1,h7n3,h11n1,h5n3,h8n4,h5n1,h4n9,h4n8,h4n1,h10n8,h10n7,h11n6,h12n4,h11n9,h6n6,h2n9,h7n7,h10n5,h12n5,h11n2。人偏肺病毒的探针适用于检测所有四个遗传谱系。第一和第二标签序列在表3中加下划线,而靶特异性区域没有。检测序列为斜体。用于扩增连接的探头组件的引物如表4所示。反向引物携带内部标记。表4引物1和引物2表3.用于检测呼吸系统病毒和细菌的第一和第二核酸探针所使用的检测探针的序列和标记列于表5中。每个标记位于3'端表5.标记的检测探针病原体标记解链温度(理论)序列(5’→3’)seqidn°甲/乙型流感病毒rox55℃tgaccactctcctagt39呼吸道合胞病毒arox60℃actggaccattaggcatg40呼吸道合胞病毒bcy555℃agatttacctgtggaga41人偏肺病毒cy570℃gcgcgaagagttctattgcatccgt42肺炎衣原体rox70℃gaccgctaatccacgtaacgacctg43肺炎支原体rox65℃agcggactctaaggacgga44嗜肺军团菌cy560℃cattaagaccactctggct45内部扩增对照(iac)ir70065℃ggtcttcgcccagaagctgct46除了用于检测甲型流感病毒和乙型流感病毒的流感病毒的检测探针,每个检测探针对于所示的单个病原体是特异性的。实施例2.样品制备。使用鼻咽灌洗液作为临床标本。使用magnapure核酸系统(rochediagnostics,almere,thenetherlands)作为提取方法。应用总核酸分离试剂盒和总核酸裂解提取magnapure操作方案。根据制造商的说明进行提取。简言之,使用200μl起始物质,纯化的核酸以100μl的终体积洗脱。在开始提取之前,向裂解的样品中加入5μl(约150个拷贝)的内部扩增对照(iac)。实施例3.预扩增将提取的具有iac的核酸置于分开的反应管中,并将如表2所示的引物混合物与用于逆转录的试剂一起加入,然后进行预扩增(rt-pcr)。虽然可以使用本领域已知的用于rt-pcr的任何程序,但是在该实施例中使用以下程序。在含有5μlonesteprt-pcr缓冲液(12.5mmmgcl2;ph8.7(20℃))、1μl脱氧核苷三磷酸(dntp)混合物(含有1.6μm的每种dntp)、2.5μl引物混合物(含有2μm的每种引物)、1μl的onesteprt-pcr酶混合物、5.5μl无rnase的水和10μl提取的具有iac的核酸模板的25μl中进行onesteprt-pcr(qiagen,hilden,germany)。通过向一个反应管中加入无rnase的水代替核酸模板来制备空白反应对照。将反应管置于biometrat1thermocycler(biometra,germany)中,程序如下:在50℃逆转录30分钟,95℃初始pcr活化15分钟,然后30个循环:在94℃30秒、在55℃30秒以及72℃60秒。实施例4.多个不同靶dna模板的连接与检测扩增后通过向各个反应管中加入100μlte(10mmtris-hcl,1mmedtaph8.0)将rt-pcr反应稀释5倍。在8μl的最终体积中进行杂交,所述8μl的最终体积由2μl5倍稀释的rt-pcr反应、最终浓度为0.28mkcl、56mmtris-hclph8.5、0.19mmedta的缓冲液组分和探针的完全混合物组成,每个探针的终浓度为1-4fmol。将反应管置于rotor-gene6000实时系统(corbett,sydney,australia)中,程序如下:98℃初始5分钟变性步骤,然后在60℃杂交1小时。在40μl的最终体积中进行组合连接和pcr,所述40μl的最终体积由8μl杂交反应、终浓度为2mmmgcl2、3.8mmtris-hclph8.2、0.16mmnad的缓冲液组分、400μm的每种dntp、1u连接酶-65、2utaq聚合酶、0.1μm正向引物、0.2μm内部fam标记的反向引物、8个0.1μm3'端标记的检测探针(表5)和0.1xgreeni(invitrogen,breda,thenetherlands)组成。使用以下pcr条件:在95℃初始变性2分钟,然后40个循环:在94℃变性30秒、60℃退火30秒以及在72℃延伸1分钟。在每个退火步骤结束时测量荧光。在每个通道中的激发在470nm,在510nm、610nm、660nm和710nm检测发射。添加0.1xsybrgreen可以检测510nm通道中的扩增曲线,与检测探针的标记无关。扩增程序之后是解链程序。在95℃变性2分钟并在45℃再退火90秒后记录解链曲线。在0.2℃/秒加热至80℃过程中检测到荧光,当探针解链离开时,测量到荧光的降低。在四个通道中测量荧光。在每个通道中的激发在470nm,在510nm、610nm、660nm和710nm检测发射。结果示于图7中。图7显示了反应的解链曲线分析。fam/rox通道(470/610nm)和fam/cy5通道(470/710nm)仅显示背景读数,并且没有检测到解链峰。在fam/cy5通道(470/660nm)中,在70℃下检测到解链峰,对应于人偏肺病毒检测探针的解链温度。实施例5:在一个反应中分开的通道中检测两个不同靶dna模板在该实施例中,使用含有包含病毒靶序列的dna或体外合成rna的样品。反应1含有肺炎支原体的dna,反应2含有具有呼吸道合胞病毒a靶序列的rna。两个样品还含有内部扩增对照。以与样品dna或rna的浓度相当的浓度加入内部扩增对照。如实施例3和4所述进行预扩增和连接和检测。fam/rox通道(470/610nm)和fam/ir700通道(470/710nm)的解链数据的结果示于图8中。fam/rox通道显示在65℃反应1中的解链峰和在59℃反应2中的解链峰。这对应于肺炎支原体检测探针和呼吸道合胞病毒a检测探针的理论解析温度。fam/ir700通道在两个样品中都显示出67℃的解链峰。这对应于内部扩增对照的检测探针的理论解链温度。实施例6.靶dna的定量。在该实施例中,证明了基于扩增曲线的输入dna浓度的定量。含有肺炎支原体dna的样品10倍稀释。分析未稀释和稀释的样品。如实施例3和4所述进行预扩增和连接和检测。图9显示两个样品的fam/rox通道(470/610nm)中的扩增曲线和解链数据。解链数据的分析表明在反应中单个产物是否被扩增。对于两个样品,在fam/rox通道中仅检测到一个解链峰。其他通道仅显示背景读数。由于fam/rox通道中检测到的信号仅来自一个产品,因此解释该通道中的扩增曲线是有效的。指示每个样品的阈值循环(ct)。两个样品之间的ct值差异为2.2,对应于输入dna的倍数差异为22.2=4.6。实施例7.在一个通道中检测两个不同靶dna模板。此实施例给出了在同一通道中检测到两个dna靶时数据的效果。样品制备、预扩增、连接和检测必须按照实施例2、3和4进行。典型的结果看起来如图10所示。在55℃和70℃可以鉴定两个解链峰。该通道和在55℃的峰对应于甲型流感病毒和乙型流感病毒检测探针的rox标记和解链温度。在70℃的第二个峰对应于rox标记的肺炎支原体检测探针的解链温度。实施例8.在单个封闭反应容器中多个不同靶dna模板的杂交、连接和检测。在该实施例中,描述了能够在单个反应容器中同时检测临床样品中的多个不同靶的应用。在这种情况下,使用含有肺炎支原体dna的样品。引物和探针与实施例1中所述的相同,样品制备和预扩增条件如实施例2和3所述。由提取的具有iac的核酸进行rt-pcr反应,并在扩增后向各个反应管中加入100μl10mmtris-hcl、1mmedtaph8.0进行5倍稀释。整个方法在单个反应容器中进行并杂交、连接、扩增和检测在一个程序步骤中组合而不打开反应容器。制备20μl反应混合物,其由2μl5倍稀释的rt-pcr反应、最终浓度为3mmmgcl2、10mmtris-hclph8.2、0.2mmnad、50mmkcl、200μm的每种dntp的缓冲液组分、完全的探针混合物,每个探针的终浓度为1-4fmol、0.1μm正向引物、0.2μm内部fam标记的反向引物、1utaq连接酶、0.5uhotstartaqdna聚合酶(qiagen,hildengermany)、8个3'端标记的检测探针(每个探针0.1μm)(表5)和0.1xgreeni(invitrogen,breda,thenetherlands)组成。将反应管置于rotor-gene6000实时系统(corbett,sydney,australia)中,程序如下:98℃初始5分钟变性步骤,然后在60℃杂交1小时,在95℃下变性和初始激活热启动taq聚合酶15分钟,然后40个循环:在94℃30秒,60℃退火30秒,72℃延伸1分钟。在每个退火步骤结束时测量荧光。在每个通道中的激发在470nm,在510nm、610nm、660nm和710nm检测发射。添加0.1xsybrgreen可以检测510nm通道中的扩增曲线,与检测探针的标记无关。扩增程序之后是解链程序。在95℃变性2分钟并在45℃再退火90秒后记录解链曲线。在0.2℃/s加热至80℃过程中检测到荧光,当探针解链离开时,测量到荧光的降低。在四个通道中测量荧光。在每个通道中的激发为在470nm,在510nm、610nm、660nm和710nm检测发射。fam/rox通道(470/610nm)中的解链曲线分析结果如图11所示。数据显示在64.1℃的解链峰,其对应于肺炎支原体检测探针的解链温度。其他通道显示背景读数。实施例9临床验证用根据本发明的方法总共分析了128个临床标本。使用针对甲型、乙型流感病毒和a亚型h5n1,副流感病毒1、2、3和4,呼吸道合胞病毒a和b,鼻病毒,冠状病毒229e,oc43和nl63,腺病毒和人偏肺病毒的探针。这种多参数呼吸系统测试的灵敏度与每个个体病毒的单一实时pcr一样好。扩增产物的鉴定是通过在封闭系统中使用检测探针进行解链曲线分析。实施例10:探针和引物的设计病毒和细菌序列获自genbank和losalamos数据库。对于每个微生物的一组序列进行比对(clustalwmultiplealignment)以鉴定高度保守的区域。由于一些靶病原体是rna病毒,因此进行逆转录酶步骤之后进行预扩增步骤,以获得多个不同的靶dna模板。基于这些高度保守的区域,设计了逆转录酶(rt)-pcr引物和连接探针。表6列出了作为第一和第二引物杂交模板的序列的靶区域、genbank登录号和位置。选择基质蛋白基因(m1)中的保守区域作为扩增和检测甲型流感病毒的靶。这些引物适用于各种菌株的扩增,包括但不限于,h3n2,h2n2,h4n2,h3n8,h4n6,h6n3,h5n2,h3n6,h6n8,h5n8,h1n1,h7n1,h7n9,h6n2,h9n2,h6n1,h7n3,h11n1,h5n3,h8n4,h5n1,h4n9,h4n8,h4n1,h10n8,h10n7,h11n6,h12n4,h11n9,h6n6,h2n9,h7n7,h10n5,h12n5,h11n2,h10n8。还选择了基质蛋白基因(m1)中的保守区域作为扩增和检测乙型流感病毒的靶。选择主要核衣壳蛋白基因(n)中的保守区作为扩增和检测rsva和b和hmpv的靶。用于扩增hmpv的引物适用于扩增和检测所有四个遗传谱系(a1,a2,b1,b2)。选择编码多聚蛋白基因的保守区域作为扩增和检测肠病毒的靶。选择编码多聚蛋白基因的保守区域作为扩增和检测鼻病毒的靶。选择编码主要外膜蛋白(ompa)的基因中的保守区域作为扩增和检测肺炎衣原体的靶。选择细胞粘附素蛋白1基因(p1)中的保守区域作为扩增和检测肺炎支原体的靶。选择巨噬细胞感染增强子基因(mip)中的保守区域作为扩增和检测嗜肺军团菌的靶。选择is481区域的保守区域作为扩增和检测百日咳鲍特菌的靶。选择编码噬菌体外壳蛋白和裂解蛋白的相邻基因中的保守区域作为扩增和检测用作内部对照的噬菌体ms2的靶。表7列出了用于逆转录和随后预扩增的rt-pcr引物(正向和反向)的序列。表6.呼吸系统病毒和细菌的靶基因表7.用于病毒和细菌核酸的预扩增的(逆转录酶)pcr引物。第一和第二核酸探针的序列列于表8中。如上表7对于引物所示,这些探针组适用于检测各种菌株。例如,流感探针适用于扩增各种菌株,包括但不限于,h3n2,h2n2,h4n2,h3n8,h4n6,h6n3,h5n2,h3n6,h6n8,h5n8,h1n1,h7n1,h6n2,h9n2,h6n1,h7n3,h11n1,h5n3,h8n4,h5n1,h4n9,h4n8,h4n1,h10n8,h10n7,h11n6,h12n4,h11n9,h6n6,h2n9,h7n7,h10n5,h12n5,h11n2。hmpv的探针适用于检测所有四个遗传谱系。用于扩增初级反应产物(连接的探针组件)的引物示于表9中。反向引物携带内部标记。表8.用于检测呼吸系统病毒和细菌的第一和第二核酸探针。第一和第二标签序列加下划线,而靶特异性区域没有。为了明确区分,检测序列为斜体,侧接有连字符。表9.引物1和引物2所使用的检测探针的序列和标记列于表10。每个标记位于3'端。除了检测鼻病毒和肠病毒的探针之外,每个检测探针对于单个病原体是特异性的。表10.*表示硫代磷酸酯键,其是第一个和第二个核苷酸之间的特殊键,并保护探针免受taq聚合酶的5'-3'核酸外切酶活性。实施例11.样品制备。手动样品制备将重复悬浮于1ml通用运输介质(utm,copan)中的鼻咽拭子作为临床标本。提取的样品输入体积为200μl。所有样品均掺入5μl的ms2内部对照,并根据制造商的说明书使用qiaampminelutevirusspinkit提取,最终洗脱体积为60μl。通过仅提取utm并掺入5μl的ms2内部对照制备阴性对照样品。自动样品制备将重复悬浮于1ml通用运输介质(utm,copan)中的鼻咽拭子作为临床标本。提取的样品输入体积为200μl。根据boom(boom1990)的操作方案,使用easymag(biomérieux),根据制造商的说明书中的“generic”操作方案,使用磁性二氧化硅进行自动核酸提取。裂解在机上进行,洗脱体积为60μl。将内部对照(每个样品5.5μl)与二氧化硅一起加入。实施例12.预扩增虽然可以使用本领域已知的用于rt-pcr的任何程序,但是在该实施例中使用以下程序。使用lifetechnologies的1xfastvirus1-stepmastermix,200nm的如表7所示的每个预扩增引物和10μl所提取的核酸进行one-steprt-pcr。将反应管置于biometrat1thermocycler(biometra,germany)中,程序如下:在50℃逆转录10分钟,在95℃初始pcr活化2分钟,然后40个循环:在94℃20秒、在55℃20秒,72℃35秒。实施例13.连接的探针组件的杂交、形成和在单个封闭反应容器中检测多个不同的靶dna模板连接的探针组件的组合形成和扩增以及扩增的连接的探针组件的检测在25μl的最终体积中进行,所述25μl的最终体积由5μl预扩增反应产物、最终浓度为1.5mmmgcl2、10mmtris-hclph8.5、50mmkcl的缓冲液组分、200μm的每种dntp、1.25utaq聚合酶、80nm的每个第一核酸探针(表8)、20nm的每个第二核酸探针(表8)、50nm的引物1(表9)、400nm的内部fam标记的引物2(表9)和13个200nm3'端标记的检测探针(表10)(integrateddnatechnologies)组成。使用以下pcr条件:95℃初始变性2min,接着(i)10个循环:94℃变性15秒,55℃退火15秒,72℃延伸15秒,(ii)23个循环:94℃变性15秒,50℃退火15秒,72℃延伸15秒。在每个退火步骤结束时测量荧光。在每个通道中的激发在470nm,在510nm、610nm和660nm检测发射。扩增程序之后是解链程序。在95℃变性2分钟并在40℃再退火90秒后记录解链曲线。在1℃/秒加热至90℃过程中检测荧光,当探针解链离开时,测量荧光的降低。在三个通道中测量荧光。在每个通道中的激发在470nm,在510nm、610nm和660nm处检测到发射。实施例14.在一个反应中分开的通道中检测两个不同呼吸道病毒的rna。在该实施例中,显示含有甲型流感病毒和肠病毒/鼻病毒的临床标本。按照实施例2、4和5进行样品制备、预扩增、连接和检测。在三个通道中测量荧光。在每个通道中的激发在470nm,在510nm(fam)、610nm(rox)和660nm(cy5)检测发射。解链数据的结果如图13所示。rox通道显示出与甲型流感病毒的检测探针的理论解链温度相对应的75℃的解链峰。cy5通道显示对应于肠病毒/鼻病毒检测探针的理论解析温度65℃的解链峰。fam通道显示对应于内部对照的检测探针的理论解链温度的73℃的负解链峰,和对应于混合物1的扩增对照的63℃的负解链峰。实施例15.在一个通道中检测两个不同靶dna模板。该实施例给出了在同一通道中检测到两个dna靶时数据的效果。样品制备、预扩增、连接和检测必须按照实施例2、3和4进行。典型的结果看起来如图14所示。可以在rox通道中鉴定61℃和75℃的两个解链峰。61℃的峰对应于腺病毒检测探针的解链温度。75℃的第二个峰对应于甲型流感病毒检测探针的解链温度。实施例16.两个不同反应混合物中扩增对照的外观当在两种混合物中测试样品(在这种情况下为在测定中检测到的任何病原体为阴性的样品)时,该实施例(图15)给出了数据的效果,其中每个混合物包含可识别的扩增对照,其允许由解链温度的差异区分混合物1和2。样本的阴性结果通过两种混合物中内部对照的存在以及相应的扩增对照的存在来验证。实施例17:临床验证根据实施例2、4和5分析了来自“genomicstocombatresistanceagainstantibioticsforcommunity-acquiredlowerrespiratorytractinfection(lrti)ineurope(grace)”(finch2012)的联合体(consortium)的总共195个鼻咽拭子。在本发明的允许检测22个呼吸系统病原体的实施方案中测试这些样品。在多重预扩增步骤之后,以两个混合物的方式进行反应。混合物1含有腺病毒,肺炎病毒,甲型流感病毒和乙型流感病毒,呼吸道合胞病毒a和b,鼻病毒/肠病毒(不区分),百日咳博德特氏菌,嗜肺军团菌,肺炎支原体和嗜酸性肺炎链球菌的组件探针和检测探针。混合物2含有a(h1n1)pdm09,副流感病毒1-2-3-4,博卡病毒,冠状病毒nl63/hku1(不区分),冠状病毒oc43和冠状病毒229e的组件探针和检测探针。本发明该实施方案的总体(平均)灵敏度为91.1%,特异性为99.6%。序列表<110>pathofinderb.v.<120>用于同时检测样品中多个核酸序列的测定<130>path-012<140>us14/694,008<141>2015-04-23<160>118<170>bissap1.2<210>1<211>22<212>dna<213>人工<220><221>来源<222>1..22<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>1caagaccaatcctgtcacctct22<210>2<211>25<212>dna<213>人工<220><221>来源<222>1..25<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>2atcgatggcgcatgcaactggcaag25<210>3<211>25<212>dna<213>人工<220><221>来源<222>1..25<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>3atgtcgctgtttggagacacaattg25<210>4<211>24<212>dna<213>人工<220><221>来源<222>1..24<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>4gcatcttttgttttttatccattc24<210>5<211>27<212>dna<213>人工<220><221>来源<222>1..27<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>5tcccataatatacaagtatgatctcaa27<210>6<211>24<212>dna<213>人工<220><221>来源<222>1..24<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>6aacccagtgaatttatgattagca24<210>7<211>26<212>dna<213>人工<220><221>来源<222>1..26<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>7tgtggtatgctattaatcactgaaga26<210>8<211>20<212>dna<213>人工<220><221>来源<222>1..20<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>8ggagccacttctcccatctc20<210>9<211>23<212>dna<213>人工<220><221>来源<222>1..23<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>9caaagaggcaagaaaaacaatgg23<210>10<211>25<212>dna<213>人工<220><221>来源<222>1..25<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>10gcctggctcttctgactgtggtctc25<210>11<211>20<212>dna<213>人工<220><221>来源<222>1..20<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>11ggaacaaagtctgcgaccat20<210>12<211>25<212>dna<213>人工<220><221>来源<222>1..25<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>12aaacaatttgcatgaagtctgagaa25<210>13<211>20<212>dna<213>人工<220><221>来源<222>1..20<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>13ggttcttcaggctcaggtca20<210>14<211>20<212>dna<213>人工<220><221>来源<222>1..20<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>14ggggtgcgtacaataccatc20<210>15<211>21<212>dna<213>人工<220><221>来源<222>1..21<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>15ttagtgggcgatttgtttttg21<210>16<211>20<212>dna<213>人工<220><221>来源<222>1..20<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>16atagcgtcttgcatgccttt20<210>17<211>19<212>dna<213>人工<220><221>来源<222>1..19<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>17acatgtaaccgcccccatt19<210>18<211>20<212>dna<213>人工<220><221>来源<222>1..20<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>18tccacgcacgcactactatg20<210>19<211>51<212>dna<213>人工<220><221>来源<222>1..51<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>19gggttccctaagggttggaccatgcacgctcaccgtgcccagtgagcgagg51<210>20<211>83<212>dna<213>人工<220><221>来源<222>1..83<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>20actgcagcgtagacgctttgtccaaaatgccctcaatgggaatgactaggagagtggtca60tctagattggatcttgctggcac83<210>21<211>46<212>dna<213>人工<220><221>来源<222>1..46<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>21gggttccctaagggttggagacagaagatggagaaggcaaagcaga46<210>22<211>79<212>dna<213>人工<220><221>来源<222>1..79<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>22actagcagaaaaattacactgttggttcggtgggaaagaaactaggagagtggtcatcta60gattggatcttgctggcac79<210>23<211>53<212>dna<213>人工<220><221>来源<222>1..53<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>23gggttccctaagggttggaggctcttagcaaagtcaagttgaatgatacactc53<210>24<211>87<212>dna<213>人工<220><221>来源<222>1..87<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>24aacaaagatcaacttctgtcatccagcaaatacaccatccaacggacatgcctaatggtc60cagttctagattggatcttgctggcac87<210>25<211>56<212>dna<213>人工<220><221>来源<222>1..56<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>25gggttccctaagggttggagtccaggttaggaagggaagacactataaagatactt56<210>26<211>86<212>dna<213>人工<220><221>来源<222>1..86<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>26aaagatgctggatatcatgttaaagctaatggagtagatataacaatctccacaggtaaa60tcttctagattggatcttgctggcac86<210>27<211>58<212>dna<213>人工<220><221>来源<222>1..58<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>27gggttccctaagggttggagctcatgcatcccacaaaatcagaggccttcagcaccag58<210>28<211>105<212>dna<213>人工<220><221>来源<222>1..105<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>28acacaccaataattttattatgtgtaggtgccttaatattcactaaactagcatcaaacg60gatgcaatagaactcttcgcgctctagattggatcttgctggcac105<210>29<211>52<212>dna<213>人工<220><221>来源<222>1..52<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>29gggttccctaagggttggaccatacattggagtacaatggtctcgagcaact52<210>30<211>94<212>dna<213>人工<220><221>来源<222>1..94<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>30tttgatgctgataacatccgcattgctcagccaaaactacctacagcaggtcgttacgtg60gattagcggtctctagattggatcttgctggcac94<210>31<211>46<212>dna<213>人工<220><221>来源<222>1..46<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>31gggttccctaagggttggagtggcttgtggggcagttaccaagcac46<210>32<211>88<212>dna<213>人工<220><221>来源<222>1..88<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>32gagtgacggaaacacctcctccaccaacaacctcgcgcctaatacttccgtccttagagt60ccgcttctagattggatcttgctggcac88<210>33<211>51<212>dna<213>人工<220><221>来源<222>1..51<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>33gggttccctaagggttggagctgttatggggcttgcaatgtcaacagcaat51<210>34<211>89<212>dna<213>人工<220><221>来源<222>1..89<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>34ggctgcaaccgatgccacatcattagctacagacaaggataagttgtagccagagtggtc60ttaatgtctagattggatcttgctggcac89<210>35<211>53<212>dna<213>人工<220><221>来源<222>1..53<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>35gggttccctaagggttggagcagtcaggtgagcacccagacttgcctccttgt53<210>36<211>85<212>dna<213>人工<220><221>来源<222>1..85<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>36gagaggccgcaccttggtagtaaatagacacatggccgagtagcagcttctgggcgaaga60cctctagattggatcttgctggcac85<210>37<211>19<212>dna<213>人工<220><221>来源<222>1..19<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>37gggttccctaagggttgga19<210>38<211>23<212>dna<213>人工<220><221>来源<222>1..23<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>38gtgccagcaagatccaatctaga23<210>39<211>16<212>dna<213>人工<220><221>来源<222>1..16<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>39tgaccactctcctagt16<210>40<211>18<212>dna<213>人工<220><221>来源<222>1..18<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>40actggaccattaggcatg18<210>41<211>17<212>dna<213>人工<220><221>来源<222>1..17<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>41agatttacctgtggaga17<210>42<211>25<212>dna<213>人工<220><221>来源<222>1..25<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>42gcgcgaagagttctattgcatccgt25<210>43<211>25<212>dna<213>人工<220><221>来源<222>1..25<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>43gaccgctaatccacgtaacgacctg25<210>44<211>19<212>dna<213>人工<220><221>来源<222>1..19<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>44agcggactctaaggacgga19<210>45<211>19<212>dna<213>人工<220><221>来源<222>1..19<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>45cattaagaccactctggct19<210>46<211>21<212>dna<213>人工<220><221>来源<222>1..21<223>/organism="人工"/note="人工引物或探针"/mol_type="未分配的dna"<400>46ggtcttcgcccagaagctgct21<210>47<211>27<212>dna<213>人工序列<220><221>来源<222>1..27<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>47tcccataatatacaagtatgatctcaa27<210>48<211>24<212>dna<213>人工序列<220><221>来源<222>1..24<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>48aacccagtgaatttatgattagca24<210>49<211>29<212>dna<213>人工序列<220><221>来源<222>1..29<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>49gacatgaccttcgaggtggaccccatgga29<210>50<211>29<212>dna<213>人工序列<220><221>来源<222>1..29<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>50gacatgacttttgaggtggatcccatgga29<210>51<211>21<212>dna<213>人工序列<220><221>来源<222>1..21<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>51ttatgtggtggcgttgccggc21<210>52<211>21<212>dna<213>人工序列<220><221>来源<222>1..21<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>52ttacgtggtagcgttaccggc21<210>53<211>23<212>dna<213>人工序列<220><221>来源<222>1..23<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>53caaagaggcaagaaaaacaatgg23<210>54<211>25<212>dna<213>人工序列<220><221>来源<222>1..25<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>54gcctggctcttctgactgtggtctc25<210>55<211>26<212>dna<213>人工序列<220><221>来源<222>1..26<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>55tgtggtatgctattaatcactgaaga26<210>56<211>20<212>dna<213>人工序列<220><221>来源<222>1..20<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>56ggagccacttctcccatctc20<210>57<211>18<212>dna<213>人工序列<220><221>来源<222>1..18<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>57tcaggccccctcaaagcc18<210>58<211>17<212>dna<213>人工序列<220><221>来源<222>1..17<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>58ggcacggtgagcgtgaa17<210>59<211>25<212>dna<213>人工序列<220><221>来源<222>1..25<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>59atgtcgctgtttggagacacaattg25<210>60<211>24<212>dna<213>人工序列<220><221>来源<222>1..24<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>60gcatcttttgttttttatccattc24<210>61<211>17<212>dna<213>人工序列<220><221>来源<222>1..17<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>61ggggcttgcaatgtcaa17<210>62<211>18<212>dna<213>人工序列<220><221>来源<222>1..18<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>62cactcatagcgtcttgca18<210>63<211>23<212>dna<213>人工序列<220><221>来源<222>1..23<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>63ggcatcaagcaccgctttacccg23<210>64<211>26<212>dna<213>人工序列<220><221>来源<222>1..26<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>64cggtgttgggagttctggtaggtgtg26<210>65<211>15<212>dna<213>人工序列<220><221>来源<222>1..15<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>65gcctgcgtggctgcc15<210>66<211>14<212>dna<213>人工序列<220><221>来源<222>1..14<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>66cctgcgtggcggcc14<210>67<211>14<212>dna<213>人工序列<220><221>来源<222>1..14<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>67gctgcgtggcggcc14<210>68<211>23<212>dna<213>人工序列<220><221>来源<222>1..23<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>68gaaacacggacacccaaagtagt23<210>69<211>24<212>dna<213>人工序列<220><221>来源<222>1..24<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>69agaacttaatgtgatctgtaacgt24<210>70<211>21<212>dna<213>人工序列<220><221>来源<222>1..21<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>70cgagaccattgtactccaatg21<210>71<211>20<212>dna<213>人工序列<220><221>来源<222>1..20<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>71gcagacggtcgcggataacg20<210>72<211>23<212>dna<213>人工序列<220><221>来源<222>1..23<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>72cgaaccaggtgaggttgccaatg23<210>73<211>25<212>dna<213>人工序列<220><221>来源<222>1..25<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>73gcaatgcaaggtctcctaaaagatg25<210>74<211>30<212>dna<213>人工序列<220><221>来源<222>1..30<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>74ggaagatcaatacataaagagttgaacttc30<210>75<211>54<212>dna<213>人工序列<220><221>来源<222>1..54<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>75gtggcagggcgctacgtacaagctcttagcaaagtcaagttgaatgatacactc54<210>76<211>69<212>dna<213>人工序列<220><221>来源<222>1..69<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>76ggacgcgccagcaagatccaatctagacatgcctaatggtccgctggatgacagaagttg60atctttgtt69<210>77<211>38<212>dna<213>人工序列<220><221>来源<222>1..38<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<220><221>misc_feature<222>32<223>/note="肌苷"<400>77gtggcagggcgctacgtacaagtgcacagccncaccgc38<210>78<211>42<212>dna<213>人工序列<220><221>来源<222>1..42<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<220><221>misc_feature<222>30<223>/note="肌苷"<220><221>misc_feature<222>36<223>/note="肌苷"<220><221>misc_feature<222>39<223>/note="肌苷"<400>78gtggcagggcgctacgtacaaggagtgcancagccncancgc42<210>79<211>69<212>dna<213>人工序列<220><221>来源<222>1..69<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<220><221>misc_feature<222>52<223>/note="肌苷"<220><221>misc_feature<222>57..58<223>/note="肌苷"<400>79ggacgcgccagcaagatccaatctagaccaatcgcaatcgtggcgcaggtanacggnntc60gatgacgcc69<210>80<211>73<212>dna<213>人工序列<220><221>来源<222>1..73<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<220><221>misc_feature<222>56<223>/note="肌苷"<220><221>misc_feature<222>59<223>/note="肌苷"<220><221>misc_feature<222>61..62<223>/note="肌苷"<220><221>misc_feature<222>70<223>/note="肌苷"<400>80ggacgcgccagcaagatccaatctagaccaatcgcaatcgtggcgtgcgcaggtanacng60nntcgatgangcc73<210>81<211>42<212>dna<213>人工序列<220><221>来源<222>1..42<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>81gtggcagggcgctacgtacaagtcagaggccttcagcaccag42<210>82<211>80<212>dna<213>人工序列<220><221>来源<222>1..80<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<220><221>misc_feature<222>73<223>/note="肌苷"<400>82ggacgcgccagcaagatccaatctagatagaccagaagtgaacgcatctgcacctacaca60taataaaattatnggtgtgt80<210>83<211>54<212>dna<213>人工序列<220><221>来源<222>1..54<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>83gtggcagggcgctacgtacaagggttaggaagggaagacactataaagatactt54<210>84<211>83<212>dna<213>人工序列<220><221>来源<222>1..83<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>84ggacgcgccagcaagatccaatctagaactaggagagtggtcaggattggcccattagct60ttaacatgatatccagcatcttt83<210>85<211>45<212>dna<213>人工序列<220><221>来源<222>1..45<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<220><221>misc_feature<222>40<223>/note="肌苷"<400>85gtggcagggcgctacgtacaagcttgaggctctcatggantggct45<210>86<211>80<212>dna<213>人工序列<220><221>来源<222>1..80<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<220><221>misc_feature<222>64<223>/note="肌苷"<400>86ggacgcgccagcaagatccaatctagacacggcaggtccggtatcagttgcttcgaggtg60acangattggtcttgtcttt80<210>87<211>47<212>dna<213>人工序列<220><221>来源<222>1..47<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<220><221>misc_feature<222>28<223>/note="肌苷"<400>87gtggcagggcgctacgtacaagtcattnacagaagatggagaaggca47<210>88<211>93<212>dna<213>人工序列<220><221>来源<222>1..93<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>88ggacgcgccagcaagatccaatctagatagctcgccatcacacgtctcgctgttgaccat60ctcgacagtgtaatttttctgctagttctgctt93<210>89<211>93<212>dna<213>人工序列<220><221>来源<222>1..93<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>89ggacgcgccagcaagatccaatctagatagctcgccatcacacgtctcgctgttgaccat60ctcgacagtgtaatttttctgctagttctgctt93<210>90<211>73<212>dna<213>人工序列<220><221>来源<222>1..73<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>90ggacgcgccagcaagatccaatctagagaagtcagtgtcgtgctataagacaacttatcc60ttgtctgtagcta73<210>91<211>43<212>dna<213>人工序列<220><221>来源<222>1..43<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>91gtggcagggcgctacgtacaagccgaacgcttcatccagtcgg43<210>92<211>65<212>dna<213>人工序列<220><221>来源<222>1..65<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>92ggacgcgccagcaagatccaatctagaactagtagtcaggcacgcgtaagcccactcacg60caagg65<210>93<211>50<212>dna<213>人工序列<220><221>来源<222>1..50<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<220><221>misc_feature<222>25<223>/note="肌苷"<220><221>misc_feature<222>35..36<223>/note="肌苷"<220><221>misc_feature<222>38<223>/note="肌苷"<220><221>misc_feature<222>42<223>/note="肌苷"<400>93gtggcagggcgctacgtacaagccncgtgtgctcnntntgantcctccgg50<210>94<211>50<212>dna<213>人工序列<220><221>来源<222>1..50<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<220><221>misc_feature<222>34..35<223>/note="肌苷"<220><221>misc_feature<222>42<223>/note="肌苷"<400>94gtggcagggcgctacgtacaagcgcatgtgcttnnttgtgantcctccgg50<210>95<211>66<212>dna<213>人工序列<220><221>来源<222>1..66<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<220><221>misc_feature<222>56<223>/note="肌苷"<400>95ggacgcgccagcaagatccaatctagatgtccttgggtcctcttggaggttagccncatt60cagggg66<210>96<211>61<212>dna<213>人工序列<220><221>来源<222>1..61<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<220><221>misc_feature<222>27<223>/note="肌苷"<220><221>misc_feature<222>34<223>/note="肌苷"<220><221>misc_feature<222>49..51<223>/note="肌苷"<220><221>misc_feature<222>53<223>/note="肌苷"<400>96gtggcagggcgctacgaacaagggtgngaagagnctattgagctacttnnnantcctccg60g61<210>97<211>78<212>dna<213>人工序列<220><221>来源<222>1..78<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<220><221>misc_feature<222>52..54<223>/note="肌苷"<400>97ggacgcgccagcaagatccaatctagatgtccttgggtcctcttgggctccnnngttagg60attagccgcattcagggg78<210>98<211>45<212>dna<213>人工序列<220><221>来源<222>1..45<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>98gtggcagggcgctacgtacaaggcgtagcaacagctactggaaca45<210>99<211>79<212>dna<213>人工序列<220><221>来源<222>1..79<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>99ggacgcgccagcaagatccaatctagacaggtcgttacgtggattagcggcattcatgat60aattgatggtcgcagactt79<210>100<211>38<212>dna<213>人工序列<220><221>来源<222>1..38<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<220><221>misc_feature<222>29<223>/note="肌苷"<400>100gtggcagggcgctacgtacaagggccccnatcgccctc38<210>101<211>93<212>dna<213>人工序列<220><221>来源<222>1..93<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<220><221>misc_feature<222>84<223>/note="肌苷"<400>101ggacgcgccagcaagatccaatctagacattgcgtcaagttgcttatcaatgttgagcgt60aagatggaccagggcgaagtacgnttcaaacgg93<210>102<211>45<212>dna<213>人工序列<220><221>来源<222>1..45<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>102gtggcagggcgctacgtacaagtttgaatggccggcgtctattag45<210>103<211>70<212>dna<213>人工序列<220><221>来源<222>1..70<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>103ggacgcgccagcaagatccaatctagaagcagcttctgggcgaagaccacgcagcaaact60ccggcatcta70<210>104<211>21<212>dna<213>人工序列<220><221>来源<222>1..21<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>104gtggcagggcgctacgaacaa21<210>105<211>27<212>dna<213>人工序列<220><221>来源<222>1..27<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>105ggacgcgccagcaagatccaatctaga27<210>106<211>18<212>dna<213>人工序列<220><221>来源<222>1..18<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>106tcacgagctacagcaggt18<210>107<211>23<212>dna<213>人工序列<220><221>来源<222>1..23<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>107gtggtcttcgcccagaagctgct23<210>108<211>15<212>dna<213>人工序列<220><221>来源<222>1..15<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>108ggaccattaggcatg15<210>109<211>16<212>dna<213>人工序列<220><221>来源<222>1..16<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>109ccacgattgcgattgg16<210>110<211>16<212>dna<213>人工序列<220><221>来源<222>1..16<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>110ccacgattgcgattgg16<210>111<211>24<212>dna<213>人工序列<220><221>来源<222>1..24<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>111gccaatcctgaccactctcctagt24<210>112<211>27<212>dna<213>人工序列<220><221>来源<222>1..27<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>112gaagcaactgataccggacctgccgtg27<210>113<211>37<212>dna<213>人工序列<220><221>来源<222>1..37<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>113cgagatggtcaacagcgagacgtgtgatggcgagcta37<210>114<211>14<212>dna<213>人工序列<220><221>来源<222>1..14<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>114acgacactgacttc14<210>115<211>17<212>dna<213>人工序列<220><221>来源<222>1..17<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>115cgtgcctgactactagt17<210>116<211>19<212>dna<213>人工序列<220><221>来源<222>1..19<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>116ccaagaggacccaaggaca19<210>117<211>23<212>dna<213>人工序列<220><221>来源<222>1..23<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>117ccgctaatccacgtaacgacctg23<210>118<211>38<212>dna<213>人工序列<220><221>来源<222>1..38<223>/organism="人工序列"/note="引物"/mol_type="未分配的dna"<400>118gtcttacgctcaacattgataagcaacttgacgcaatg38当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1