TCR文库的制作方法
本发明涉及一种展示多种不同T细胞受体(TCR)的颗粒文库,其中多种TCR基本上由包含α链和β链的TCR组成,所述α链包含为TRAV12-2基因产物的α链可变结构域,所述β链包含为单个TRBV基因的基因产物的β链可变结构域,其中当TRAV12-2来自天然库时,β链可变结构域不包含来自天然库的TRBV6基因产物。
背景技术:
T细胞受体(TCR)介导T细胞对特异性主要组织相容性复合物(MHC)-限制性肽抗原进行识别,并且对于免疫系统的细胞武器的功能而言是必要的。在人体中,MHC分子也被称为人类白细胞抗原(HLA),这两个术语在本说明书中作同义使用。术语“肽抗原”、“肽-MHC”和“肽-HLA”是指由TCR识别的抗原。TCR仅以膜结合形式存在,因此TCR在历史上难以分离。大多数TCR由两条二硫键连接的多肽链,即α链和β链组成。
本说明书使用国际免疫遗传学(IMGT)的TCR命名法和TCR序列的IMGT公共数据库链接来描述TCR。天然α-β异源二聚体TCR具有α链和β链。广泛地,每条链均包含可变区、连接区和恒定区,并且β链通常还包含可变区和连接区之间的短多变区,但是该多变区通常被认为是连接区的一部分。每个可变区包含嵌入框架序列中的三个高变CDR(互补决定区);CDR3被认为是抗原识别的主要介导物。存在几种类型的α链可变区(Vα)和几种类型的β链可变区(Vβ),它们通过框架、CDR1和CDR2序列以及部分限定的CDR3序列来区分。Vα类型在IMGT命名法中由独特的TRAV编号来指代。因此,“TRAV12-2”定义了一个具有独特框架和CDR1、CDR2序列以及CDR3序列的TCR Vα区,所述CDR3序列部分限定为一个在TCR之间保守的氨基酸序列,但也包含一个在TCR之间变化的氨基酸序列。以相同的方式,“TRBV9”定义了一个具有独特框架和CDR1、CDR2序列,但仅具有一个部分限定的CDR3序列的TCR Vβ区。已知在α和β基因座内分别有54个α可变基因(其中44个是功能性的)和67个β可变基因(其中42个是功能性的)(Scaviner D.和Lefranc M.P.(2000)Exp Clin Immunogenet,17(2),83-96;Folch G.和Lefranc M.P.(2000)Exp Clin Immunogenet,(2000)17(1),42-54;“T cell Receptor Factsbook”,(2001)LeFranc and LeFranc,Academic Press,ISBN 0-12-441352-8)。正如本领域技术人员已知的那样,功能性的定义可以改变。因此,为了清楚起见,本说明书始终参考在国际免疫遗传学(IMGT)网站www.imgt.org(访问日期:2015年8月17日)上所载的TCR命名法。
所述TCR的连接区由独特的IMGT TRAJ和TRBJ命名法进行类似的定义,并且其恒定区由IMGT TRAC和TRBC命名法进行定义(Scaviner D.和Lefranc M.P.(2000)Exp Clin Immunogenet,17(2),97-106;Folch G.和Lefranc M.P.(2000)Exp Clin Immunogenet,17(2),107-14;“T cell Receptor Factsbook”,(2001)LeFranc and LeFranc,Academic Press,ISBN 0-12-441352-8)。
β链多变区在IMGT命名法中由缩写TRBD表示,并且如上所述,串联的TRBD/TRBJ区通常一起被认为是连接区。
编码TCRα和β链的基因库位于不同的染色体上,并且含有分离的V、(D)、J和C基因片段,所述基因片段在T细胞发育期间通过重排而汇聚在一起。由于54个TCRα可变基因和61个αJ基因之间,或者67个β可变基因、2个βD基因和13个βJ基因之间可以产生大量的潜在重组事件,这导致了T细胞α和β链具有非常高的多样性。所述重组过程并不精确,使得在CDR3区内引入更多的多样性。每个α和β可变基因还可以包含等位基因变体,在IMGT命名法中分别被命名为TRAVxx*01和*02,或TRBVx-x*01和*02,从而进一步增加了变异量。同样,一些TRBJ序列有两种已知的变体(请注意,没有限定符“*”则表示相关序列中只有一种等位基因是已知的)。已经估计由重组和胸腺选择而产生的人类TCR天然库包含有由CDR3多样性而确定的约106个独特β链序列(Arstila,T.P.,et al.(1999)Science,286(5441),958-61),可能甚至更高(Robins,H.S.et al..(2009)Blood,114(9),4099-4107)。估计每条β链与至少25种不同的α链配对,从而产生更多的多样性(Arstila,T.P.,et al.(1999)Science,286(5441),958-61)。
在本说明书和权利要求书中,术语“TCRα(或阿尔法)可变域”因此指代TRAV和TRAJ区的串联;仅指TRAV区;或指TRAV和部分TRAJ区,并且术语“TCRα(或阿尔法)恒定结构域”指胞外TRAC区,或指C端截短的或全长的TRAC序列。类似地,术语“TCRβ(或贝塔)可变域”可以指代TRBV和TRBD/TRBJ区的串联;仅指TRBV和TRBD区;仅指TRBV和TRBJ区;或指TRBV和部分TRBD和/或TRBJ区,术语“TCRβ(或贝塔)恒定结构域”是指胞外TRBC区,或指C端截短的或全长的TRBC序列。
由IMGT命名法定义的独特序列是TCR领域技术人员广泛熟知并且可得到的。例如,可以在IMGT公共数据库中检索到它们。《T细胞受体资料手册》(“T cell Receptor Factsbook”,(2001)LeFranc and LeFranc,Academic Press,ISBN 0-12-441352-8)也公开了由IMGT命名法定义的序列,但由于其出版日期和随后的时间延迟,其中的信息有时需要参考IMGT数据库进行确认。
长期以来,需要鉴定基本上由天然α和β链序列组成的可特异性结合至特定抗原的TCR,从而可以研发例如TCR或其可溶性类似物来为潜在治疗提供基础。由已鉴定的TCR识别的抗原,可能与某种疾病,如癌症、病毒感染、炎性疾病、自身免疫疾病、寄生虫感染和细菌感染相关。因此,所述疗法可以用于治疗上述疾病。
此外,一旦鉴定了TCR并且确定其序列,则可根据需要引入能导致亲和性或半衰期增加的突变(如WO2012/013913中所述)。
传统上,鉴定特异性结合至疾病相关抗原(如癌症、病毒、自身免疫、炎症、寄生虫或细菌抗原)的TCR的尝试一直受限于使用从志愿者供体采集的血液样品。这些样品用于分离T细胞及其结合疾病相关抗原的相应的TCR。这种方法通常需要至少20个供体才可合理期望获得成功。该过程耗时长且劳动强度大,并且无法保证能够鉴定到抗原结合TCR。在鉴定到功能性TCR的情况中,所述TCR通常在体外对抗原具有弱亲和性、低特异性和/或不能恰当折叠。能够筛选的T细胞的多样性受限于供体内的T细胞多样性。一些疾病相关抗原,包括大多数癌症抗原都是自身抗原;由于胸腺选择去除了识别自身抗原的TCR,针对疾病相关抗原有特异性的TCR可能不存在于供体的天然库中,或者可能对抗原具有弱亲和性。
设计一个用于分离具有抗原结合特异性的新型TCR的文库的尝试已经持续多年。由于TCR链比较不稳定并且通常不能正确展示,因此TCR文库比类似的抗体文库更加难以创建。TCR文库的构建涉及非常多的复杂因素。优选保留CDR3长度变化(如其在天然库中的存在形态)。任何文库的实质部分通常都会因终止密码子、移码、折叠问题和可能根本不能结合HLA复合物的TCR链组合而失败。考虑到可变α基因和可变β基因以及J和D基因的巨大数量,想要产生并鉴定功能性折叠α链和功能性折叠β链(二者共同形成以所需特异性结合至抗原肽的TCR)的几率是极低的。
在构建文库上已经做出了许多尝试。下文首先描述的是基于合成的TCR文库;也就是说,文库中的TCR含有突变,且突变一般包含在使用随机突变从体外引入的CDR中。因此,上述文库中包含的任何单条TCR链的序列可能与在天然库中存在的任何TCR链均不对应。由于在合成文库中仅存在某些突变,因此整个文库与天然库并不对应。在之前公开的合成文库中,将随机突变引入到单个已知TCR的α和β链的CDR区中,使得所述文库中的所有TCR含有相同的α和β框架序列,但是具有随机生成的CDR序列。针对文库的进一步分析证明,鉴定抗原特异性TCR是不成功的。具体而言,据发现,较大部分TCR链是非功能性的,而原因各不相同:在许多情况中,序列是截短的或含有移码;而在其他情况中,尽管鉴定出全长TCR链,但它们无法正确折叠;最后,从文库中分离的TCR在进一步测试时不能特异性结合至抗原。这些合成文库中的非天然多样性被认为是文库不成功的原因之一。引入非天然突变可能干扰了TCR本身功能。此外,与天然TCR谱系相比,CDR3中引入的多样性可能有限。如天然库中的CDR3序列长度所例证的那样,在T细胞中的TCR组装期间,在CDR3序列中生成了巨大的多样性。通过使文库基于特定位置突变,CDR3序列的多样性可能受到非常多的限制,尤其在CDR3序列长度方面。最后,非天然TCR序列将不会经过在体内发生的胸腺选择过程。
上述理由在一定程度上解释了(不受理论限制)为何构建下述的希望从中鉴定出特异性结合TCR的文库的尝试是不成功的。
WO2005/116646描述了一种基于已知(天然)TCR的文库,其中6个CDR经单独或组合突变,即文库中所有TCR都是非天然的,但以天然鉴定的TCR框架区为基础。WO2005/114215还涉及从这种文库中获得的产物。用多种其他抗原(除了原始TCR结合的抗原以外)筛选该文库。然而,这导致仅分离出一条生产性全长TCR序列。在其他实验中,发现这种TCR具有交叉反应性。
因此,已经构建了基于体外突变TCR的文库,但是其还不能允许分离具有足够抗原结合特异性的新TCR以供使用。
已经构建了基于完整天然库的文库,其中天然衍生的α和β链随机混合(如下所述),但是未能成功鉴定任何的特异性结合抗原的TCR。
具体而言,WO2005/116074描述了核蛋白文库,其各自在表面上展示包含天然TCRα可变域序列或天然β可变域序列的多肽。该公开中所描述的文库由多条α和β链构建;从用于生成所述文库的mRNA池扩增43个Vα类基因和37个Vβ类基因。该文献显示,三轮噬菌体展示导致与测试肽结合的克隆的分离。所述克隆被描述为在ELISA筛选期间鉴定,由强ELISA信号所确定。然而,当测试所述克隆结合替代的肽-HLA时,也观察到强的ELISA信号;因此,所述TCR克隆对肽无特异性。对该文库的进一步分析显示了与上述合成文库类似的问题,即它们含有大比例的非生产性TCR链以及不能正确折叠的TCR。因此所述文库不能用于鉴定新的抗原结合TCR。
因此,需要能够更可靠地鉴定包含α链可变域和β链可变域的、功能性TCR的TCR文库,所述文库可以使用多种肽抗原进行筛选以鉴定此类有用的TCR。然后鉴定的TCR可以以其天然亲和性使用,或者可用于,例如噬菌体展示成熟,以增强亲和性。
技术实现要素:
本发明在第一方面提供了一种展示多种不同T细胞受体(TCR)的颗粒文库,其中多种TCR基本上由包含α链和β链的TCR组成,所述α链包含为TRAV12‐2基因产物的α链可变结构域,所述β链包含为单个TRBV基因的基因产物的β链可变结构域;其中当TRAV12‐2来自天然库时,β链可变结构域不包含来自天然库的TRBV6基因产物。可变结构域如上所述,即它们也可分别包含完整或部分TRAJ或TRBD和/或TRBJ区。
单个TRBV基因可以是以下之一:
α和/或β可变域的CDR3序列可以从天然库获得。可选地,α和/或β可变域的CDR3序列可以人工设计并且可以包含非天然突变。所述框架、CDR1和/或CDR2可以包含非天然的突变。
α链可变域和β链可变域可展示为单一多肽链。
TCR在颗粒上展示并且可以包含在α链的恒定区和β链的恒定区之间的非天然二硫键。例如,WO 03/020763中描述了此种非天然二硫键。可选地,在颗粒上展示的TCR可以包含在α链的恒定区和β链的恒定区之间的天然二硫键。
每条α链和每条β链可包含二聚化结构域,其优选是异源的。此种异源结构域可以是亮氨酸拉链、5H3结构域或疏水性富脯氨酸反结构域,或本领域已知的其他类似方案。
形成文库的颗粒可以是噬菌体颗粒。
可选地,该文库可以是核糖体文库。可选地,该文库可以是酵母展示文库,因此所述颗粒可以是酵母细胞。所述颗粒可以是哺乳动物细胞。
该文库可能适合用肽抗原进行筛选。此种肽抗原可以包含HLA,如HLA-A,B或C,如HLA-A2。
本发明的另一方面提供了从本发明第一方面的文库获得的包含TCRα链可变域和TCRβ链可变域的分离的T细胞受体(TCR),所述TCRα链可变域包括TRAV12-2基因产物,且所述TCRβ链可变域包括为单个TRBV基因的基因产物的β链可变域。所述TCR可以是可溶的,或者可以适合在细胞上表达。本发明还包括编码所述TCR的TCRα链可变域和/或β链可变域的核酸。
在另一方面,本发明提供第一方面的文库的用途,其用于鉴定特异性结合至肽抗原的TCR。肽抗原可以用于筛选本发明的文库以获得与其结合的TCR。肽抗原可以包含HLA,如HLA-A、B、C、G或E,或非经典HLA如CD1。肽抗原可以包含HLA-A2。
另一方面提供了一种获得特异性结合肽抗原的TCR的方法,包括用肽抗原筛选上述第一方面的文库,所述方法包括:a)使用肽抗原作为标靶淘选文库;b)重复步骤a)一次或多次;c)筛选步骤a)或b)中鉴定的噬菌体克隆;及d)鉴定特异性结合肽抗原的TCR。该肽抗原可以包含HLA,如HLA-A、B、C、G或E,或非经典HLA如CD1。肽抗原可以包含HLA-A2。
在另一方面,本发明涉及一种制备颗粒文库的方法,所述文库展示多种不同的TCR,所述方法包括:i)获得编码不同TRAV12-2α链可变域的多种核酸;ii)获得编码单个TRBV基因的不同TRBVβ链可变域的多种核酸;iii)将编码TRAV12-2α链可变域的核酸克隆到表达载体中;iv)将编码TRBVβ链可变域的核酸克隆到相同或不同的载体中;及v)在颗粒中表达载体,由此产生基本上由包含由所述核酸编码的α链可变域和β链可变域的TCR组成的文库。
还提供了本发明的另一种制备颗粒文库的方法,所述文库展示多种不同的TCR,所述方法包括:i)使用与编码TRAV12-2α链可变域的核酸杂交的引物,来获得编码不同TRAV12-2α链可变域的多种核酸;ii)使用与编码TRBVβ链可变域的核酸杂交的引物,来获得编码单个TRBV基因的不同TRBVβ链可变域的多种核酸;iii)将编码TRAV12-2α链可变域的核酸克隆到表达载体中;iv)将编码TRBVβ链可变域的核酸克隆到相同或不同的载体中;及v)在颗粒中表达载体,由此产生基本上由TCR组成的文库,所述TCR包含由与所述引物杂交的所述核酸编码的α链可变域和β链可变域。
单个TRBV基因可以是以下之一:
正向引物可以设计为与TRAV12-2基因座或选定的TRBV基因座杂交。反向引物可以设计为分别至少部分与α或β恒定区杂交,使得所得PCR产物含有可变区,至连接区和至少部分恒定区。转录、翻译或翻译后事件可能导致连接和/或恒定区(包括在β链序列情况下的多变区)的部分或全部的截短或删除。
在步骤(i)和/或步骤(ii)中编码TRAV12-2或TRBV的多种核酸中的每个核酸的全部或部分可以通过合成和/或人工设计来获得。
框架区、CDR1、CDR2和/或CDR3的全部或部分可以通过合成和/或人工设计来获得。至少步骤(i)和步骤(ii)的核酸的CDR3序列可以人工设计,或者可以来自天然库。
步骤(i)和步骤(ii)的核酸序列可以从天然库获得,或者可以部分或完全地人工设计。
在一些情况下,可以在步骤iii)之前将非天然突变引入核酸序列。可以在步骤i)和/或步骤ii)之后,或在步骤iii)和/或步骤iv)之后引入突变。
在制备本发明的文库的任一方法中,TCRα链可变域和TCRβ链可变域优选从相同的载体表达,即编码每个α链和β链可变域的核酸被克隆到相同的载体中。α链可变域和β链可变域可以表达成单一多肽或者分别的多肽。
在另一方面本发明提供了一种获得特异性结合肽抗原的T细胞受体的方法,包括用肽抗原筛选本发明的第一或第二方面的文库。肽抗原可以包括HLA-A、B、C、G或E,或非经典HLA如CD1。肽抗原可以包括HLA-A2。
根据本发明在其表面上显示TCR的颗粒也包括在本发明的范围内。
本发明的文库是非天然存在的,因为其包括非天然存在的TCR或者那些在本说明书中使用时被认为是“分离的”TCR;并且相应地,由于这些TCR是非天然存在的,或者如在本说明书中使用时被认为是“分离的”,因此本发明的TCR同样是可专利的主题。类似地,本发明的细胞和颗粒是可专利的主题,因为通过在其表面上展示或表达本发明的TCR,所述细胞或颗粒是非天然存在的,或者如在本说明书中使用的那样被认为是“分离的”。
还应注意的是,在本公开中,尤其是在权利要求书和/或说明书段落中,诸如“包含”等术语具有对其赋予的通常含义,例如,它们可以意指“包括”等;且诸如“基本上由...组成”等术语具有对其赋予的通常含义,例如,它们允许未明确列举的要素,但排除出现在现有技术中或影响本发明基本特征或新颖性特征的要素。
这些及其他实施方式在下述说明中公开、或者从下述说明中明白可见并且包含在其中。
附图说明
下文的详细描述以示例的形式给出,但并非旨在将本发明仅限制为所述特定实施方式,最好可以结合附图对其进行理解,其中:
图1概述了用于创建文库的克隆策略;
图2详述了在文库构建中使用的通用引物序列;
图3显示了通过ELISA筛选对来自含有TRAV12-2和TRBV5文库的带有抗原特异性TCR的噬菌体颗粒进行检测;
图4显示了通过ELISA筛选对来自含有TRAV12-2和TRBV7的文库的带有抗原特异性TCR的噬菌体颗粒进行检测;
图5显示了通过ELISA筛选对来自含有TRAV12-2和TRBV11-1的文库的带有抗原特异性TCR的噬菌体颗粒进行检测;
图6显示了通过ELISA筛选对来自含有TRAV12-2和TRBV11-3的文库的带有抗原特异性TCR的噬菌体颗粒进行检测;
图7显示了通过ELISA筛选对来自含有TRAV12-2和TRBV14文库的带有抗原特异性TCR的噬菌体颗粒进行检测;
图8显示了通过ELISA筛选对来自含有TRAV12-2和TRBV20-1的文库的带有抗原特异性TCR的噬菌体颗粒进行检测;
图9显示了通过ELISA筛选对来自含有TRAV12-2和TRBV27的文库的带有抗原特异性TCR的噬菌体颗粒进行检测;
图10显示了通过ELISA筛选对来自含有TRAV12-2和TRBV30的文库的带有抗原特异性TCR的噬菌体颗粒进行检测;
图11显示了从含有TRAV12-2和TRBV5的文库和含有TRAV12-2和TRBV30的文库中获得的TCR的Biacore数据。
具体实施方式
根据本发明,提供了一种展示多种不同T细胞受体(TCR)的颗粒文库,其中多种TCR基本上由包含α链和β链的TCR组成,所述α链包含为TRAV12-2基因产物的α链可变结构域,所述β链包含为单个TRBV基因的基因产物的β链可变结构域,其中当TRAV12-2来自天然库时,β链可变结构域不包含来自天然库的TRBV6基因产物。
单个TRBV基因可以是下表列出的已知的TRBV基因中的一种。
具体而言,TRBV基因可以是TRBV5、TRBV7、TRBV11‐1、TRBV11‐3、TRBV14、TRBV20‐1、TRBV27或TRBV30。
优选地,TRBV基因可以是功能性的。
“基本上由......组成”表示文库中的大部分TCR包含TRAV12‐2和特定的TRBV,但由于在制备文库时引物的非特异性杂交,或者在α或β可变基因座基因中的基因之间存在高度同源性的区域,小部分TCR可包含不同的α或β链可变结构域。针对大部分的数量可以如下定义。
所述多种TCR可由80%的含有α链可变结构域和β链可变结构域的TCR组成,所述α链可变结构域包含TRAV12‐2基因产物,所述β链可变结构域包含TRBV基因产物。所述多种TCR可由85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的、含有α链可变结构域(包含TRAV12‐2基因产物)和β链可变结构域(包含特定的TRBV基因产物)的TCR组成。
所述多种TCR中剩余的20%或以下可以包含与TRBVβ链可变结构域基因产物配对的不同的α链可变结构域基因产物,以及与TRAV12‐2可变结构域基因产物配对的不同的β链可变结构域基因产物。或者不同的TRAV基因产物与不同的TRBV基因产物配对。
因此,本发明的文库可以含有多种TCR,每种TCR具有以下α链和β链V、J、(D)和C基因用法:
α链---TRAV12-2/TRAJxx/TRAC;和
β链---TRBVyy/TRBDx/TRBJxx/TRBC1、TRBC2或C1和C2的嵌合体,
其中xx分别是61个αJ基因或13个βJ基因中的任何基因,Dx代表2个βD基因中的任一个,并且yy是TRBV基因中的任一个。如上所述,J、D或C区可以各自是全部的或部分存在或缺失。
优选地,V、D、J和C基因是人类的。
“基因产物”也表示可包括翻译后修饰的多肽,其被所示基因的核酸序列编码。如本领域技术人员已知的那样,每个TCRα或β链可变域基因在CDR3区含有变异,如上所述,这意味着TRAV12-2或特定TRBV的基因产物也将大幅变化。
α和/或β链序列可以从天然库获得。“来自天然库”是指:对于例如序列长度和氨基酸组成而言,至少在多种TCR内的CDR3序列直接对应于天然库的那些序列。在这种情况下,α和β链可变域可以从已从人类供体扩增的DNA序列表达。换而言之,文库的TCR的α和/或βCDR3结构域的多样性已经在体内T细胞发育期间自然产生。此外,这意味着文库中所有α和β链的序列将在胸腺选择期间被选择。与最初存在体内(即在供体内)中相比,在文库创建期间发生的这些α和β链的随机组合可能产生αβ链组合的替代库。可以通过例如从供体mRNA产生cDNA来间接获得DNA序列。然后cDNA序列可以用作模板来产生DNA序列,从中产生多种不同的TCR。
可选地,可以人工设计α和/或β链序列。“人工设计”是指多种TCR内的CDR3序列的多样性可能与天然库并不对应。在这种情况下,可以通过例如DNA合成,使用简并寡核苷酸,如NNK、NNN或NNS,掺入CDR3序列内限定的位置,或通过引入如下定义的非天然突变来产生序列。优选地,该文库中人工设计的CDR3序列的多样性,在例如序列长度和氨基酸组成的变化方面,设计为与天然库的序列类似。优选地,文库中人工设计的CDR3序列的总多样性大于从天然库获得的总多样性。
因此,“人工设计”是指该序列与来自天然库的TRAV12-2或特定TRBV的基因产物的氨基酸序列相同或相似(即具有90%的序列同一性)。该序列可能与天然库中存在的任何TRAV12-2或TRBV基因产物并非100%相同。例如且如同本领域技术人员已知的那样,该序列可能已经针对密码子使用、折叠能力、稳定性、去除切割位点、去除/添加糖基化或酰胺化或其他翻译后修饰位点进行优化。这种修饰可以是氨基酸取代、添加或删除,即通过引入一个或多个非天然突变,其包含在“人工设计”的定义中。如本领域技术人员所理解的那样,取代可以是保守氨基酸取代或非保守氨基酸取代。通常,这种修饰发生在TRAV12-2和/或TRBV基因产物的框架区内。
可以通过本领域已知的任何方式引入非天然突变。非天然突变可以随机生成,或者特异性限定,或者同时通过两者生成。例如,可以使用位点饱和诱变在限定位置处纳入随机生成的突变,其中天然氨基酸编码序列被所有其他天然存在氨基酸的编码序列替换;从而在限定位置处产生另外的文库多样性。该方法可能涉及使用简并合成寡核苷酸作为引物的PCR扩增来复制目标DNA。优选地,这种突变在α和/或β链可变区的CDR区内进行。可选地,或附加地,可使用例如市售试剂盒(例如Stratagene公司的Quik Change Site Directed Mutagensis试剂盒)在特定位置处引入限定的突变(包括插入和删除)。
该文库可展示TCR,其中10%、15%、20%、25%、30%、40%、50%、60%、70%、75%、80%、85%、90%、95%或100%的α链可变域或β链可变域包含非天然突变。
本发明的文库优选包含至少1×10 8个展示αβTCR链组合的颗粒。
该文库可以是噬菌体颗粒的文库。在WO2004/044004中描述了噬菌体展示。
可选地,该文库是核糖体文库。核糖体展示是本领域已知的。颗粒可以是完整的核糖体复合物或其部分。
可以使用酵母展示系统,这意味着文库可以是酵母细胞文库。
适用于创建TCR文库的另一种展示方法是哺乳动物细胞展示。该系统使用逆转录病毒载体来将TCRα和β链引入TCR阴性T细胞杂交瘤。该方法还在Chervin et al.(2008)J Immunol Methods,339,175-84;和Kessels et al.(2000)Proc Natl Acad Sci U S A,97,14578-83)中有进一步描述。
如前所述,能够展示异二聚体或单链TCR的任何颗粒文库都包含在本发明中。
单链TCR包括Vα-L-Vβ、Vβ-L-Vα、Vα-Cα-L-Vβ、Vα-L-Vβ-Cβ或Vα-Cα-L-Vβ-Cβ类型的αβTCR多肽,可选地,其中Vα和Vβ分别为TCR的α和β可变区,Cα和Cβ分别为TCR的α和β恒定区,L为接头序列。单链TCR在WO2004/033685;WO98/39482;WO01/62908;Weidanz et al.(1998)J Immunol Methods 221(1-2):59-76;Hoo et al.(1992)Proc Natl Acad Sci U S A,89(10):4759-4763;Schodin(1996)Mol Immunol 33(9):819-829)中有进一步描述。
α和/或β链恒定结构域可相对于原始/天然存在的TRAC/TRBC序列截短。另外,当TRAC/TRBC存在时可含有修饰。α链胞外序列可以包含与原始/天然存在的TRAC相关的修饰,从而参考IMGT编号的TRAC的氨基酸T48被C48取代。类似的,β链胞外序列可包含与原始/天然存在的TRBC1或TRBC2相关的修饰TRBC1或TRBC2的S57(参考IMGT编号)被C57取代,并且C75被A75取代,N89被D89取代。这些与天然α和β链细胞外序列相关的半胱氨酸取代能够形成非天然链间二硫键,其使得重折叠的可溶性TCR(即通过重折叠胞外α和β链形成的TCR)稳定化。这种非天然二硫键有助于在噬菌体上展示正确折叠的TCR(Li,Y.et al.Nat Biotechnol 2005:23(3),349-54)。另外,使用稳定的二硫键连接的可溶性TCR能够更方便地评估结合亲和性和结合半衰期。在WO03/020763中描述了可选的替代物。可选地,α和β恒定结构域可以通过与自然界中发现的二硫键相对应的二硫键连接。
为了进一步,或可选地,使异二聚体TCR稳定化,每条α链和每条β链可以包含二聚化结构域,其可相对于天然TCR链序列异源。
特别地,二聚化结构域可以是亮氨酸拉链。该术语描述了以特定方式相互作用以形成异二聚体的螺旋肽对。发生相互作用是因为沿各拉链肽的一侧上有互补的疏水残基。肽的性质更倾向于形成异二聚体,远胜于形成螺旋的同二聚体。亮氨酸拉链可以是合成的或天然产生的,如在WO99/60120中所描述的那样。替代性的二聚化结构域包括二硫键桥形成元件。可选地,它可以由负责参与信号转导的蛋白质中观察到的蛋白质-蛋白质相互作用的SH3结构域和疏水性/富脯氨酸的反结构域提供(由Schlessinger综述,(Schlessinger,J.,Curr Opin Genet Dev.1994Feb;4(1):25-30)。参与信号转导级联的蛋白质之间发现的其他天然蛋白质-蛋白质相互作用,依赖于翻译后修饰的氨基酸和特异性识别此种经修饰的残基的蛋白质模块之间的结合。此类翻译后修饰的氨基酸和蛋白质模块可以形成本发明的文库的TCR链的二聚化结构域。
抛开理论限制,本发明的文库的大小,即在其中表现的与完整的(或接近完整的)库相关的α和β链可变结构域基因的数量的减少,被认为是从本发明的文库中能够鉴定出特异性功能性TCR的可能的原因之一。在前述较大的“天然”文库中,可能某些α链与某些β链不配对,从而文库的大部分都是非功能性的。可能是某些链型在噬菌体表面无法正确折叠。可能每条α链的表达或展示不够频繁而无法与“理想”β链配对,反之亦然,因此减少了鉴定出包含α和β链的特异性功能性TCR的几率。发明人已经发现TRAV12‐2链在与任何单个β链配对时特别适合于产生文库,而通过该文库可以常规鉴定出有用的TCR。
在另一方面,本发明提供了一种分离的T细胞受体(TCR),其包含TCRα链可变结构域和TCRβ链可变结构域,所述TCRα链可变结构域包含TRAV12-2基因产物,所述TCRβ链可变结构域包含从根据本发明第一方面的文库中分离的TRBV基因产物。
“分离的”是指TCR从其天然环境中取出,即不是在体内T细胞上天然展示的TCR。
TCR可以特异性结合至肽抗原。如通过,例如但不限于,ELISA或BiaCore所测定的那样,从本发明的文库中获得的这种TCR可以强亲和性和高特异性结合至肽抗原。可通过进一步的亲和性成熟来获取TCR,使得结合亲和性和/或半衰期增加。TCR可以是可溶的,即其可以从跨膜结构域上切割(如WO 03/020763中所述)。如上所述,TCR可含有非天然二硫键。TCR可以与可检测的标记物或治疗剂融合,所述标记物包括但不限于荧光标记物、放射性标记物、酶、核酸探针和造影剂,,所述治疗剂包括但不限于免疫调节剂、放射性化合物、酶(例如穿孔素)或化疗剂(例如顺铂)(WO2010/133828)。TCR可以在细胞表面非天然表达,优选哺乳动物细胞,更优选免疫细胞,甚至更优选T细胞。
可以通过任何合适的方法来确定结合亲和性(与平衡常数KD成反比)和结合半衰期(表示为T1/2)。应该理解的是,TCR的亲和性翻倍导致KD减半。T1/2计算为ln2除以解离速率(koff)。所以T1/2的翻倍导致koff减半。通常通过TCR的可溶形式,即截短以去除疏水性跨膜结构域残基的那些形式,来测量TCR的KD和koff值。因此,应该理解的是,如果给定的TCR的可溶形式满足其对肽抗原具有结合亲和性和/或结合半衰期的要求,则该TCR满足该要求。优选地,使用相同的试验方案在限定的温度下多次测量给定的TCR的结合亲和性或结合半衰期,并取结果的平均值。更优选地,通过在25℃的温度下的表面等离子体共振来测量结合亲和性或结合半衰期。在实施例12中给出了一种优选的方法。
为了本发明的目的,如上所述,TCR是具有至少一个TCRα和至少一个TCRβ可变域的部分。通常它将同时包含TCRα可变域和TCRβ可变域。它们可以是αβ异二聚体,或者可以是单链形式,其表示单个多肽同时含有α链和β链(如WO 2004/033685中所述)。单链TCR包括Vα-L-Vβ、Vβ-L-Vα、Vα-Cα-L-Vβ、Vα-L-Vβ-Cβ或Vα-Cα-L-Vβ-Cβ类型的αβTCR多肽(可选地在相反方向),其中Vα和Vβ分别为TCRα和β可变区,Cα和Cβ分别为TCRα和β恒定区,且L为接头序列。可选地,TCR可以包含通过一对C端二聚化肽(例如亮氨酸拉链)二聚化为TCRβ链胞外结构域的TCRα链胞外结构域,此种TCR在WO 99/60120中有描述。为了在过继性治疗中使用,αβ异二聚体TCR可以例如以具有胞质和跨膜结构域的全长链转染到细胞(如T细胞)中。如果需要,各个恒定结构域的残基之间可以存在引入的二硫键(参见例如WO 2006/000830)。可选地,α和β恒定结构域可以通过与自然界中发现的二硫键相对应的二硫键连接。
本发明包括编码本发明TCR的TCRα链可变域和/或TCRβ链可变域的核酸。α和β链可以从分开的各核酸或从单个核酸分子表达。如果从相同的核酸分子表达,则α和β链可以表达为独立的多肽或单链。
该核酸包含TRAV12-2序列和/或TRBV核酸序列。该核酸也可以包含TRAJ序列和/或TRBD/TRBJ序列。该核酸还可以包含TRAC和/或TRBC1或TRBC2核酸序列,或其部分序列。
在本发明的另一方面,提供了第一方面的文库用于鉴定特异性结合至肽抗原的TCR的用途。如上所述,由于各种原因,需要特异性结合至肽抗原的TCR。
在另一方面,本发明提供了一种制备根据本发明第一方面所述文库的方法,所述方法包括:i)获得编码不同TRAV12-2α链可变域的多种核酸;ii)获得编码特定TRBV基因的不同TRBVβ链可变域的多种核酸;iii)将编码的TRAV12-2α链可变域的核酸克隆到表达载体中;iv)将编码TRBVβ链可变域的核酸克隆到相同或不同的载体中;及v)在颗粒中表达载体,由此产生基本上由TCR组成的文库,所述TCR包含由所述核酸编码的α链可变域和β链可变域。
可以使用从供体血液获得的mRNA地通过PCR完全或部分获得核酸。可选地,可以完全或部分地通过合成手段获得核酸,例如使用固相DNA合成,如由生命技术公司(Life Technologies)有偿进行的。可以通过复制/扩增核苷酸序列反式cDNA(从来自供体的T细胞库的mRNA制备)来获得步骤i)和ii)的核酸。获得的核酸可编码不同的TRAV12-2α或TRBVβ链可变域,所获的编码不同的TRAV12-2α或TRBVβ链可变结构域的核酸可能是唯一获得的核酸,即步骤i)可能涉及获得编码不同TRAV12-2α链可变结构域的唯一核酸,且步骤ii)可能涉及获得编码特定TRBV基因的不同TRBVβ链可变结构域的唯一核酸。所产生的文库可以是基本上由包含α链可变结构域和β链可变结构域的TCR组成的文库,其中α链可变结构域包含TRAV12-2基因产物,且β链可变结构域包含特定的TRBV基因产物。
本发明还提供了一种制备颗粒文库的方法,所述文库展示多种不同的TCR,所述方法包括:i)使用与编码TRAV12-2α链可变域的核酸杂交的引物,获得编码不同TRAV12-2α链可变域的多种核酸;ii)使用与编码单个TRBV基因的TRBVβ链可变域的核酸杂交的引物,获得编码不同TRBVβ链可变域的多种核酸;iii)将编码TRAV12-2α链可变域的核酸克隆到表达载体中;iv)将编码TRBVβ链可变域的核酸克隆到相同或不同的载体中;及v)在颗粒中表达载体,由此产生基本上由TCR组成的文库,所述TCR包含由与所述引物杂交的核酸编码的α链可变域和β链可变域。
单个TRBV基因可以是下表列出的已知的TRBV基因中的一种:
具体而言,TRBV基因可以是TRBV5、TRBV7、TRBV11-1、TRBV11-3、TRBV14、TRBV20-1、TRBV27或TRBV30。
根据杂交反应发生的条件以及杂交核酸序列的组成和长度,两条单链序列将互相杂交,即使两条序列之间不存在100%序列同一性。
一般而言,杂交温度和杂交缓冲液的离子强度(如Mg2+浓度)将决定杂交的严格度。高严格度(例如高杂交温度和低盐杂交缓冲液)使得杂交仅发生在高度相似的核酸序列之间,而低严格度(例如较低温度和高盐)允许在序列相似性较低时杂交。本领域技术人员可以很容易进行关于达到一定程度严格度的杂交条件的计算,并在Sambrook et al.,(1989)“Molecular Cloning”,第二版,Cold Spring Harbor Laboratory,Plainview,NY(第9和11章)中也有描述。本领域技术人员将能够根据灵敏度和特异性测试的结果优化杂交条件。
下面是用于本发明的一组示例性杂交条件:
极高严格度(检测有至少90%同一性的序列)
杂交:5x SSC,65℃下16小时
洗涤两次:2x SSC,室温(RT)下各15分钟
洗涤两次:0.5x SSC,65℃下各20分钟
高严格度(检测有至少80%同一性的序列)
杂交:5x-6x SSC,65℃-70℃下16-20小时
洗涤两次:2x SSC,室温下各5-20分钟
洗涤两次:1x SSC,55℃-70℃下各30分钟
低严格度(检测有至少50%同一性的序列)
杂交:6x SSC,室温至55℃下16-20小时
至少洗涤两次:2x-3x SSC,室温至55℃下各20-30分钟,。
本文公开的引物可以在低严格度、高严格度和极高严格度条件下与编码TRAV12-2α链可变结构域或TRBVβ链可变结构域的核酸杂交。
该引物可以高严格度结合至编码α和β链可变域的序列。然而,该引物可以结合至与TRAV12-2和/或TRBV具有高度同源性的一些其他基因座。
步骤i)和ii)的核酸可以来自天然库。可选地,其可以人工设计。可以在步骤iii)之前或在步骤iii)之后将非天然突变引入α或β可变域,即可在克隆入载体之前向核酸序列引入非天然突变。可选地,可以在步骤iii)和/或iv)的克隆步骤之后引入非天然突变。
编码TRAV12-2或TRBV的每种核酸的全部或部分可以通过合成获得和/或人工设计。具体地,可以部分或完全合成地和/或人工设计来获得可变域、框架区、CDR1、CDR2和/或CDR3序列。
“合成”是指已经化学合成(即不是通过PCR或其他生物技术)的序列。全部或部分合成的α或β链序列可以是化学合成的。
TRAV12-2可变域的扩增可以来自预先制备的、本身来源于供体mRNA的cDNA文库,其中正向引物被设计为特异性结合至目标基因座。可以将反向引物设计为特异性结合至(至少部分)TCRα恒定区,使得所得的PCR产物含有TRAV12-2核酸序列,部分的或全部的连接区或不含有连接区,以及至少部分恒定区。这类引物设计确保了所捕获α链可变域CDR3区的变化性和多样性,从而在本发明的文库中表现出大量独特的TCRα链序列。可选地,CDR3区可以独立扩增,例如使用特异性结合至CDR3任一侧框架序列的引物。得到的PCR产物可以连接至不具有CDR3的TRAV12-2序列。
类似地,TRBV可变域的扩增可来自可用的cDNA文库,其中正向引物被设计为特异性结合至目标基因座。可以将反向引物设计为特异性结合至TCRβ恒定区,使得所得的PCR产物含有特定TRBV核酸序列,部分的或全部的连接区或不含有连接区(含有D和J基因座),以及至少部分恒定区。这类引物设计确保捕获β链可变域CDR3区的变化和多样性,从而在本发明的文库中表现出大量独特的TCRβ链序列。可选地,CDR3区可以独立扩增,例如使用特异性结合至CDR3任一侧框架序列的引物。得到的PCR产物可以连接至不具有CDR3的TRBV序列。
从至少一个供体获得mRNA。“来自至少一个供体”是指全部或部分α或β链可变域的多肽序列基本与其在作为mRNA来源的供体的T细胞中天然产生的多肽序列相同。优选地,供体是人类。单个或多个供体的组织类型可能是已知的。单个或多个供体可以是HLA-A2阳性的。
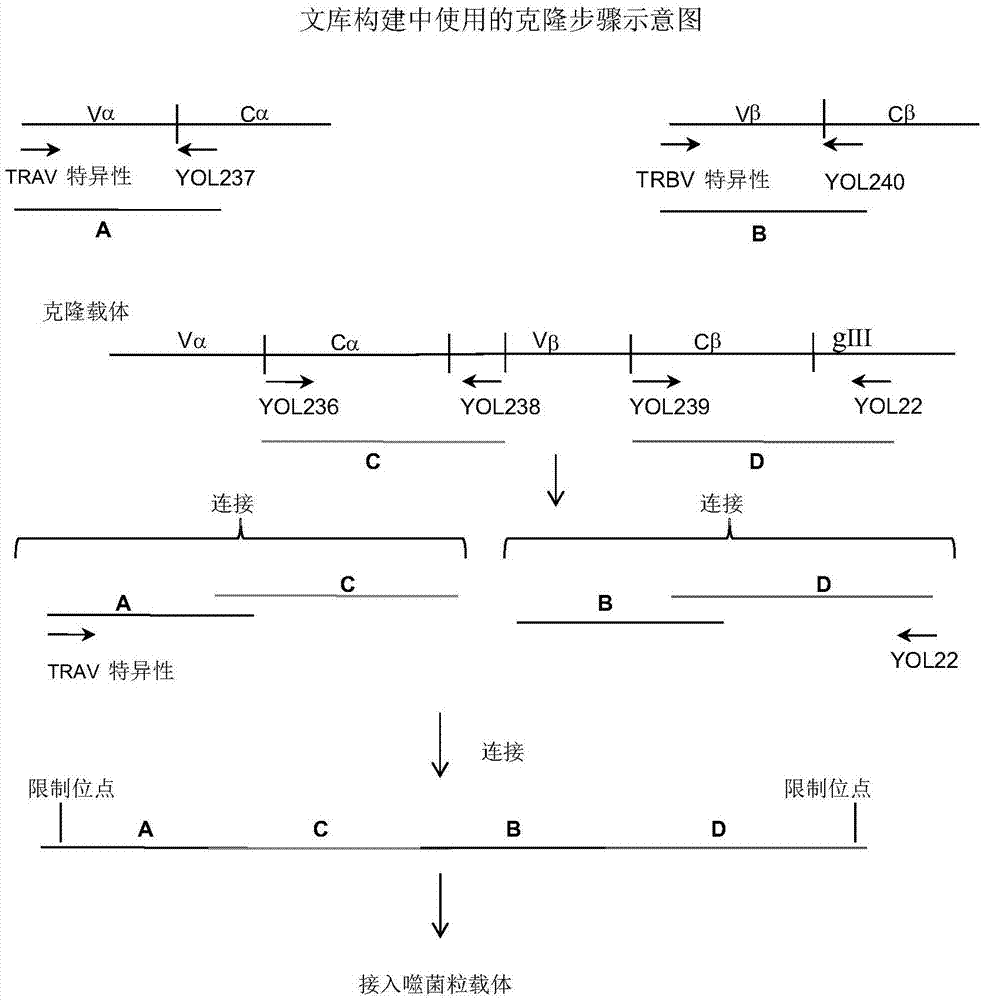
所得的PCR产物若含有完整恒定基因序列,则可以直接连接至噬菌体载体,前提是载体和引物序列中存在所需的连接或重组序列。可选地,可以分别用含有α恒定结构域基因序列和β恒定结构域基因序列的序列将α和βPCR产物连接在一起,以获得完全TCR链序列。α链和β链可以随机连接在一起,以增加噬菌体文库的多样性。然后可以将完整序列克隆到噬菌体载体中,以表达为开放读码框。图1显示了产生本发明文库的合适的克隆策略的例子。
可选地,也可以使用其他颗粒展示形式来产生本发明的文库。这类方法是本领域技术人员已知的,并且可以包括但不限于在核糖体颗粒或酵母细胞上展示。
这些展示方法分为两大类,即体外展示和体内展示。
所有体内展示方法都依赖于这样一个步骤,其中将通常在可复制颗粒(如质粒或噬菌体复制子)的基因组核酸中编码的文库或与所述核酸共同编码的文库导入细胞以表达蛋白质或多肽(Plückthun(2001)Adv Protein Chem 55 367-403)。已证明有多种复制子/宿主系统适用于蛋白质或多肽的体内展示。这些包括:
噬菌体/细菌细胞
质粒/CHO细胞
基于酵母2μm质粒/酵母细胞的载体
杆状病毒/昆虫细胞
质粒/细菌细胞
逆转录病毒载体/哺乳动物细胞
体内展示方法包括细胞表面展示方法,其中将质粒引入编码融合蛋白的宿主细胞中,所述融合蛋白由与细胞表面蛋白或多肽融合的目标蛋白质或多肽组成。该融合蛋白的表达使得在细胞表面上展示目标蛋白质或多肽。展示这些目标蛋白质或多肽的细胞随后可经过选择过程,如FACS,并且可以对所选一个或多个细胞中获得的质粒进行分离和测序。已经针对哺乳动物细胞(Higuschi(1997)J Immunol.Methods 202 193-204)、酵母细胞(Shusta(1999)J Mol Biol292 949-956)和细菌细胞(Sameulson(2002)J.Biotechnol 96(2)129-154)设计了细胞表面展示系统。在酵母细胞表面展示单链TCR是本领域已知的(WO01/48145)
已经发表了针对各种体内展示技术的多篇综述。例如(Hudson(2002)Expert Opin Biol Ther(2001)1(5)845-55)和(Schmitz(2000)21(Supp A)S106-S112)。
体外展示方法基于使用核糖体来将mRNA文库翻译成蛋白质或多肽变体的多样化阵列。通过两种方法中的一种来维持所形成的蛋白质或多肽与编码这些分子的mRNA之间的连接。常规的核糖体展示采用编码短(通常40-100个氨基酸)接头序列和待展示的蛋白质或多肽的mRNA序列。接头序列给予所展示的蛋白质或多肽足够的空间来重新折叠而不受核糖体空间位阻。mRNA序列缺少‘终止’密码子,这确保表达的蛋白质或多肽和RNA保持接合在核糖体颗粒上。相关的mRNA展示方法要基于对编码目标蛋白质或多肽和携带嘌呤霉素部分的DNA接头的mRNA序列的制备。一旦核糖体到达mRNA/DNA连接处,翻译停止,并且嘌呤霉素与核糖体形成共价连接。关于这两种相关的体外展示方法的综述参见(Amstutz(2001)Curr Opin Biotechnol 12 400-405)。
特别优选的是噬菌体展示技术,其基于噬菌体颗粒表达与其表面蛋白融合的异源肽或多肽的能力(Smith(1985)Science 217 1315-1317)。该过程非常普遍,并且在多肽单体展示领域为人熟知。二聚体蛋白(例如异二聚体TCR)的展示在本领域也十分成熟(WO04/044004)
有两种同时适用于单体和二聚体展示的主要过程:首先(方法A),通过将与编码噬菌体外壳蛋白(例如编码蛋白质P3或P8的DNA)的DNA融合的编码异源肽或多肽的DNA插入载体(噬菌粒)中。然后通过用噬菌粒转染细菌细胞来进行展示异源肽或多肽的噬菌体颗粒的表达,然后用“辅助噬菌体”来感染转化的细胞。辅助噬菌体用作未由产生功能性噬菌体颗粒所需的噬菌粒所编码的噬菌体蛋白的来源。
其次(方法B),通过将编码异源肽或多肽的DNA插入与编码噬菌体外壳蛋白的DNA融合的完整噬菌体基因组中。然后通过用噬菌体基因组感染细菌细胞来进行展示异源肽或多肽的噬菌体颗粒的表达。该方法作为“单步骤”过程而比第一种方法更具优势。然而,可被成功包装到所得噬菌体颗粒中的异源DNA序列的尺寸减小。M13、T7和Lambda是适用于该方法的噬菌体的示例。
方法B的变化形式涉及将编码核苷酸结合结构域的DNA序列添加至编码待展示异源肽的噬菌体基因组中的DNA,并进一步将相应的核苷酸结合位点添加至噬菌体基因组。这使得异源肽直接结合至噬菌体基因组。然后将该肽/基因组组合物包装成展示异源肽的噬菌体颗粒。该方法在WO 99/11785中有充分的描述。
然后可以回收噬菌体颗粒并用于研究异源肽或多肽的结合特征。一旦分离,就可以从肽或多肽展示噬菌体颗粒中回收噬菌粒或噬菌体DNA,并且该DNA可以通过PCR复制。PCR产物可用于对由给定噬菌体颗粒展示的异源肽或多肽进行测序。
单链抗体及其片段的噬菌体展示已成为研究这些多肽的结合特征的常规手段。有众多书籍对噬菌体展示技术和噬菌体生物学进行综述(参见例如,“Phage Display-A Laboratory Manual”,Barbas et al.,(2001)Cold Spring Harbor Laboratory Press)。
第三种噬菌体展示方法(方法C)依赖于这样一个事实,即在所需的位置处有半胱氨酸残基的异源多肽可通过噬菌粒或噬菌体基因组以可溶形式表达,并且由此与在表面暴露位置处也有半胱氨酸残基的修饰的噬菌体表面蛋白通过在两个半胱氨酸之间形成二硫键而结合。WO 01/05950详细描述了该替代性连接方法用于表达单链抗体衍生肽的用途。
如上所述,本发明的αβ异二聚体TCR可以在其恒定结构域之间引入(非天然)二硫键。这可以通过将扩增的核酸序列连接到修饰的恒定基因序列来制备本发明的文库的方法期间实现。此类序列可以包括具有TRAC恒定结构域序列和TRBC1或TRBC2恒定结构域序列的那些序列(除参照IMGT编号的TRAC的Thr48和TRBC1或TRBC2的Ser57被半胱氨酸残基置换以外),所述半胱氨酸在TRAC恒定结构域序列与文库的TCR的TRBC1或TRBC2恒定结构域序列之间形成二硫键。
在具有或者不具有前述段落中提及的引入的链间键的情况下,本发明的αβ异二聚体TCR可以具有TRAC恒定结构域序列和TRBC1或TRBC2恒定结构域序列,并且TCR的TRAC恒定结构域序列和TRBC1或TRBC2恒定结构域序列可以通过TRAC的外显子2的Cys4与TRBC1或TRBC2的外显子2的Cys2之间的天然二硫键连接。
可选地,TCRα链可变域和TCRβ链可变域可以表达为单链多肽。这种构型可以包括突变的氨基酸残基之间的非天然二硫键。
本发明还提供了获得特异性结合肽抗原的T细胞受体的方法,包括用肽抗原筛选根据本发明第一方面的文库。
筛选可包括以下一个或多个步骤
a)使用肽抗原作为靶点来淘选文库
b)重复步骤a)一次或多次
c)筛选步骤a)或b)中鉴定的噬菌体克隆,
d)鉴定特异性结合肽抗原的TCR。
根据步骤(b),步骤(a)可以重复一次、两次、三次、四次、五次或六次。其可以重复多达10次。步骤(a)可以重复多达20次。
“淘选”表示使噬菌体克隆接触抗原并将结合的噬菌体克隆与未结合的噬菌体克隆分离开来。这可以包括将抗原固定在固体支持物如管道、磁珠、柱基质或BiaCore传感器芯片上。可以通过非特异性吸附或通过使用特异性接合标签如生物素化抗原和链霉亲和素包被的表面来介导抗原接合。替代性方法可以包括在完整细胞上进行淘选(Hoogenboom,H.R.et al.(1998)Immunotechnology,4(1),1-20)。洗掉未结合的噬菌体克隆(即不显示结合至抗原的TCR的噬菌体)。然后结合的噬菌体克隆的洗脱可以通过:TCRβ链和基因III之间的蛋白酶位点(例如胰蛋白酶)的酶切割;极端的pH值;或过量抗原竞争来进行。这些噬菌体克隆可经过另外几轮淘选,或者进行筛选实验以鉴定具有最佳结合特性的克隆。
可以通过,例如基于ELISA的方法,用包被的抗原或完整细胞,并且可以以96孔形式来进行筛选;在使用全细胞的情况下,可以使用流式细胞术进行筛选。可以使用表面等离子体共振(例如在BiaCore仪器上)或使用石英晶体微天平来进行对结合亲和性和动力学的筛选。在(Pande,J.et al.(2010)Biotechnol Adv 28(6):849-58)中描述了筛选方法。如本领域技术人员已知的那样,用于筛选这种类型的生物分子相互作用的其他合适的方法是可用的,包括:来自ForteBIO的Octet系统,其利用生物膜层干涉量度法(BLI)来实时测量生物分子相互作用,并提供关于亲和性和动力学的信息;扩增发光邻近均相测定法(例如AlphaScreen TM),其中潜在相互作用的分子接合至“供体”和“受体”珠,所述“供体”和“受体”珠靠近时具有特定荧光性质;闪烁迫近测定法,其中通过紧密接近的分子之间的β粒子的转移来评估相互作用;其他的光学界面测定法在例如在WO 2004/044004中有描述。
可以通过测试鉴定出的TCR对至其他肽(除了用于筛选文库的肽抗原之外)的结合来确定特异性。如果与其他肽发生结合,则可认为TCR是非特异性的。可以使用上述方法评估特异性。
肽抗原可以是已知的抗原,如Bridgeman,J.S.et al.(2012)Immunology,135(1),9-18所述的那样。筛选本发明的文库的方法也可与新型肽抗原一起使用,以鉴定可用于治疗领域的特异性结合的TCR。
本发明的最后一个方面提供了在其表面上展示根据本发明的TCR的分离细胞,即包含获自本发明的第一方面的文库的、含有TRAV基因产物的TCRα链可变域和含有TRBV基因产物的TCRβ链可变域的分离T细胞受体(TCR),其中TCR特异性结合肽抗原。该细胞可以是T细胞。细胞可以是人、鼠或其他动物细胞。
有许多方法适用于用编码本发明TCR的DNA或RNA转染T细胞(参见例如Robbins等,(2008)J.Immunol.180:6116-6131)。表达本发明的TCR的T细胞适用于疾病(例如癌症、病毒感染、自身免疫性疾病、炎性疾病、寄生虫感染和细菌感染)基于过继性治疗的疗法。如本领域技术人员所知,有许多合适的方法可以进行过继性治疗(参见例如Rosenberg等,(2008)Nat Rev Cancer 8(4):299-308)。
为了用于过继性治疗,本发明还包括含有TCR表达载体的细胞,所述TCR表达载体在单个开放读码框或两个不同的开放读码框中包含编码本发明的TCR的核酸。在本发明的范围内还包括含有第一表达载体和第二表达载体的细胞,所述第一表达载体包含编码本发明的TCR的α链的核酸,而所述第二表达载体包含编码本发明的TCR的β链的核酸。可选地,一个载体可以同时表达本发明的TCR的α链和β链。
用于在过继性治疗中使用的本发明的TCR,可在由转染的T细胞表达时经糖基化。众所周知,可以通过转染的基因的突变来改变转染的TCR的糖基化模式(Kuball J et al.(2009),J Exp Med206(2):463-475)。
为了对患者给药,可以在药物组合物中提供用本发明的TCR转染的T细胞与药学上可接受的载体。根据本发明所述的细胞通常作为无菌的药物组合物(通常包含药学上可接受的载体)的一部分来提供。该药物组合物可以是任何合适的形式(取决于将其施用于患者所需的方法)。它可以以单位剂型提供,通常以密封的容器提供中并且可以作为试剂盒的一部分提供。这种试剂盒通常(但不一定)包括使用说明。其可以包括多种所述单位剂型。给药的合适组合物和方法为本领域技术人员所知,例如参见Johnson et al.,Blood(114):535-46(2009),参考临床试验编号NCI-07-C-0175和NCI-07-C-0174。
该药物组合物可以适于通过任何合适的途径给药,例如通过肠胃外(包括皮下、肌内、静脉内或腹膜内)、吸入或口服途径。这类组合物可以通过药学领域已知的任何方法制备,例如通过将活性成分与一种或多种载体或一种或多种赋形剂在无菌条件下混合。
本发明物质的剂量可在广泛的范围之间变化,取决于待治疗的疾病或病症(例如癌症、病毒感染、自身免疫性疾病、炎性疾病、细菌感染或寄生虫感染)与待治疗的个体的年龄和状况等。例如,ImmTAC试剂(与抗CD3结构域融合的可溶性TCR)的合适剂量范围可以是25ng/kg至50μg/kg之间。由药剂师最终确定使用的合适剂量。
本发明的TCR也可以用成像化合物来标记,例如适用于诊断目的的标记物。此类标记的高亲和性TCR可用于检测选自以下的TCR配体的方法:CD1-抗原复合物、细菌超抗原和MHC-肽/超抗原复合物,该方法包括将TCR配体与对TCR配体有特异性的高亲和性TCR(或多聚体高亲和性TCR复合物)接触;并检测与TCR配体的结合。在例如采用生物素化的异二聚体形成的四聚体高亲和性TCR复合物中,可以采用荧光链霉亲和素(可商购)来提供可检测标记物。荧光标记的四聚体适用于FACS分析,例如用于检测携带肽抗原(对于高亲和性TCR有特异性)的抗原呈递细胞。
本发明的高亲和性TCR(或其多价复合物)可以,可选地或另外地,与治疗剂结合(例如通过共价或以其它方式连接),所述治疗剂可以是例如用于细胞杀伤的毒性部分或免疫刺激剂(如白细胞介素或细胞因子)。与非多聚野生型或高亲和性T细胞受体异二聚体相比,本发明的多价高亲和性TCR复合物可以具有强化的TCR配体结合能力。因此,根据本发明所述的多价高亲和性TCR复合物,特别适用于在体外或体内追踪或靶向那些呈递特定抗原的细胞,并且还可用作生产具有此类用途的其他多价高亲和性TCR复合物的中间体。因此,可以在药学上可接受的制剂里提供所述高亲和性TCR或多价高亲和性TCR复合物,用于在体内使用。
本发明的高亲和性TCR可用于生产可溶性双特异性试剂。在一个优选的实施方式中,所述可溶性双特异性试剂是ImmTAC试剂。ImmTAC试剂包含可溶性TCR,其通过接头与抗CD3特异性抗体片段融合。WO10/133828中描述了包括如何生产这些试剂的其他细节。
本发明各方面的优选或可选特征可经必要的修改用于各其他方面。因此,虽然已经详细描述了本发明及其优点,但应当理解,可以对本文进行各种改变、替换和更改而不背离所附权利要求所限定的本发明的原理和范围。
本发明将以实施例进行进一步说明,所述实施例仅用于说明目的,并非旨在以任何方式限制本发明。
实施例
实施例1
-------------------------------------------
制备用于构建TCR噬菌体展示文库的cDNA
从外周血淋巴细胞(PBL)中分离mRNA
从已知HLA类型的志愿者供体获得的PBL中提取RNA。根据制造商推荐的方案使用TRI试剂(Sigma,目录号T9424)来进行RNA提取。随后按照制造商的指示,使用μMACSTM mRNA分离试剂盒(Miltenyi,目录号130-075-101)来分离mRNA。
从mRNA制备cDNA
根据制造商推荐的方案,使用SMARTScribe TM逆转录酶(Clontech,639536)从mRNA合成cDNA。使用S.N.A.P.凝胶纯化试剂盒(Invitrogen,45-0078)来进一步纯化cDNA。
实施例2
噬菌体文库的构建
图1显示了文库构建的概况,并且在图2中详细描述了相应的引物序列。使用TRAV或TRBV特异性正向引物和反向引物,通过PCR从纯化的cDNA扩增TCR链,所述反向引物在TRAC区(引物YOL237)或TRBC区(引物YOL 240)内退火。参考人TCR链的已知序列来设计引物组(“T Cell Receptor Facts Book”,Lefranc and Lefranc,Publ.Academic Press 2001)。所得的PCR产物由完全可变域序列和截短的恒定结构域组成(图1中标记为A和B)。使用TRAC的引物YOL236和YOL238,及TRBC2的引物YOL239和YOL22(在图1中标记为C和D),通过PCR从分离的克隆载体中扩增含有非天然半胱氨酸残基的TRAC和TRBC2结构域的剩余C端部分。然后将纯化的A/C和B/D片段在单独的反应中通过它们的重叠引物区(分别为YOL237/YOL236和YOL240/YOL239)连接在一起。将所得的A-C和B-D片段凝胶纯化,并使用TRAV特异性正向引物和YOL22反向引物通过重叠PCR连接在一起,其中TRBV和YOL238引物区提供重叠序列。该最终连接反应导致在α链和β链之间产生随机重组。将片段接入被称为pIM672(pIM672基于之前所述的pEX922载体,参见WO2005116074)的合适的噬菌粒载体中,然后将其用于转化高转化效率的电感受态TG1大肠杆菌细胞。在30℃下将培养物接种到2xTYEag(EzMix,Sigma,目录号Y2627,加100μg/ml氨苄青霉素和2%葡萄糖)琼脂平板上过夜,并将所得细胞菌苔刮入小体积的含有100μg/ml氨苄青霉素、20%甘油和2%葡萄糖的2xTYag培养基中。将文库的甘油储液在-80℃下保存。
实施例3
文库繁殖和淘选
噬菌体颗粒的繁殖
使用足够覆盖文库多样性的噬菌体文库甘油储液来接种2xYTag培养基,至0.05的初始OD600。然后将培养物培养至约为0.5的OD600。然后以约20:1噬菌体:大肠杆菌的感染率加入辅助噬菌体。然后反转混合培养物并在37℃下培养30分钟。将培养物离心并将沉淀于2xYTak(2xYTag但不含葡萄糖并加入50μg/ml卡那霉素)中重悬,随后在26℃下摇晃培养16小时。
噬菌体颗粒的分离
培养物经合并、离心、收集上清液并经0.45μm过滤。将洗脱物与7ml PEG/NaCl(20%PEG-8000(Sigma目录号5413),2.5M NaCl)混合并在冰上培养30分钟。然后将样品离心得到沉淀,并丢弃上清液。将沉淀在10ml PBS(Dulbeccos Sigma目录号D8537-无Mg,无Ca)中重悬并再次离心。收集所得的上清液,与5ml PEG/NaCl混合并在冰上储存30分钟。离心后,沉淀在3ml PBS中重悬,重离心并收集上清液。使用Nanodrop分光光度计来测定噬菌体浓度的估值,其中每ml的噬菌体数量=OD260×(22.14×1010)。
淘选
将纯化的噬菌体颗粒与3%MPBS缓冲液(PBS(Dulbeccos Sigma目录号D8537-无Mg,无Ca)加3%奶粉,先与链霉亲和素包被的顺磁珠一起培养,然后用15mM EDTA处理,之后广泛透析,最后在0.22μm处过滤)混合并在室温下培养1小时。然后加入10%(v/v)吐温-20,及100nM或1μM生物素化的肽-HLA。样品在室温下混合60分钟。通过加入在3%MPBS缓冲液中预封闭的链霉亲和素包被的顺磁珠来获取噬菌体-生物素化-HLA复合物,并在室温下培养7分钟。捕获后,使用磁力浓缩器(Dynal公司)分离珠子并用3%MPBS(未经EDTA处理)洗涤三次,并用PBS-0.1%吐温洗涤两次。噬菌体颗粒在0.5ml TBSC(10mM Tris,pH7.4,137mM NaCl,1mM CaCl2和0.1mg/ml胰蛋白酶)中在室温下洗脱25分钟,并在37℃下轻缓旋转5分钟。
使用洗脱的噬菌体颗粒来感染早对数期的TG1大肠杆菌细胞。将培养物在37℃下培育30分钟,随后以1μl、0.1μl和0.01μl连续稀释系接种于YTEag(1L MQ-水中10g胰蛋白胨、5g酵母提取物、8g NaCl、15g细菌琼脂,加100μg/ml氨苄青霉素和2%葡萄糖)。将剩余的培养物浓缩并且也接种到YTEag上。平板在30℃下培养16小时。次日,将平板上的菌落加入到2xTYag中,在干冰上冷冻并在-80℃下储存用于下一轮淘选。通过PCR分析来自每个选择的菌落以检查全长插入物。
在第三轮选择后,从琼脂平板上刮下菌落,并用于以每孔一个克隆来接种在96孔Cellstar细胞培养平板中的无菌2xTYag。平板在26℃下摇晃培养16小时。然后使用这些培养物来接种在96孔板中的新鲜2xTYag培养基,并在37℃下摇晃培养30分钟直至OD600=0.5。然后将辅助噬菌体以20:1的噬菌体-大肠杆菌感染率加入到每个孔中,并在37℃下不摇晃地培养平板30分钟。通过离心收集沉淀并在2xYTak中重悬。平板在26℃下摇晃培养16小时。然后将细胞离心为沉淀并收集上清液用于ELISA筛选。
实施例4
通过ELISA筛选对带有抗原特异性TCR的噬菌体颗粒进行检测
通过ELISA筛选来鉴定结合至给定肽-HLA复合物的噬菌体克隆,并随后测试其针对一组替代性肽-HLA复合物的特异性。使用生物素化肽-HLA来制备ELISA平板。使用抗Fd抗体(Sigma,目录号B7786),然后使用单克隆抗兔IgG过氧化物酶缀合物(γ链特异性克隆RG96)(Sigma,目录号A1949)来进行检测。使用KPL实验室TMB微孔过氧化物酶底物系统(目录号50-76-00)检测结合的抗体。在该孔中出现蓝色则表明噬菌体克隆已与同源肽-HLA结合,而含有替代性肽-HLA复合物的孔中没有颜色则表明没有结合,因此与同源抗原的结合是特异性的。
实施例5
包含TRAV12-2基因产物和单一TRBV基因产物的文库的构建和淘选
构建包含TRAV12-2和以下TRBV基因产物的文库:
·TRBV5
·TRBV7
·TRBV11-1
·TRBV11-3
·TRBV14
·TRBV20-1
·TRBV27
·TRBV30
根据实施例1和2中所述的方法并参考图2中的引物序列来制备文库,除了以下情况:
对于TRBV5文库,用下列正向引物:gcccagccggccatggcccagaaggaggtggagcagaattc(SEQ ID No:16)来扩增TRAV12-2片段。
对于TRBV7文库,使用YOL236(图2)和下列反向引物:gggcagccctgatttgtcttgttgggcttcataattgaa(SEQ ID No:17)从克隆载体扩增α恒定结构域和TRBV区的第一部分(图1,分别为片段C以及片段B的第一部分)作为单个片段。所得的片段编码整个α恒定结构域及TRBV至CDR2的序列。使用下列正向引物:cagggcccagagtttctgacttacttcaattat(SEQ ID No:18)和YOL240反向引物(图2)从cDNA扩增TRBV的剩余部分(片段B的第二部分),包括CDR3区域。所得的片段B包含TRBV7-6和TRBV7-7。如实施例1中所述,然后将这两个片段连同片段A和D一起经由重叠引物区进行连接。
对于TRBV11-1文库,分两个步骤来制备TRBV片段(在图1中表示为B)。在步骤1中,使用以下引物从cDNA扩增TRBV11-1的框架、CDR1和CDR2序列:TRBV特异性正向引物gaagctgaagttgcccagtcc(SEQ ID No:19)和TRBV反向引物gtctactcctttgagcctctctgc(SEQ ID No:20)。两种引物都在3'端经PTO修饰以降低酶促降解(PTO修饰指示在寡核苷酸的磷酸基中包含硫代磷酸酯键,其中硫原子替代非桥接氧)。在步骤2中,使用在3'端进行了PTO修饰的下述正向引物gcagagaggctcaaaggagtagac(SEQ ID No:21)和YOL240反向引物(如图2所示)从cDNA扩增CDR3区。这两个部分通过其重叠引物区而连接在一起,以产生单个TRBV片段(片段B),然后如实施例1中所述将其缝合至其他片段。
对于TRBV20-1文库,使用YOL236(图2)和下列反向引物:agttgtggcctgaaagtccagggaacggcactcgat(SEQ ID No:22)从克隆载体扩增α恒定结构域和TRBV区的第一部分(图1,分别为片段C以及片段B的第一部分)作为单个片段。所得的片段因此编码整个α恒定结构域及TRBV20-1至CDR1的序列。使用下列正向引物:atcgagtgccgttccctggactttcaggccacaact(SEQ ID No:23)和YOL240反向引物(图2)从cDNA扩增TRBV的剩余部分(片段B的第二部分),包括CDR3区。如实施例1中所述,然后将这两个片段连同片段A和D一起经由重叠引物区进行连接。
根据实施例3用肽-HLA复合物淘选每个文库,并且根据实施例4所述进行ELISA筛选。图3至10显示了从本发明的文库获得的TCR的典型的特异性ELISA。这些数据证实本发明的文库可用于分离抗原特异性TCR。
来自ELISA阳性噬菌体克隆的TCR的DNA序列可以通过使用本领域技术人员已知的方法进行测序来获得。
实施例6
对获自文库的TCR的Biacore分析
方法
通过使用BIAcore 3000设备的表面等离子体共振,来测定从文库中分离的TCR的抗原亲和性,并报告为平衡解离常数(KD)。将从噬菌体克隆获得的TCR序列用于采用Boulter et al.,Protein Eng,2003.16:707-711中描述的方法来产生可溶形式的TCR。如Garboczi et al.,Proc Natl Acad Sci USA 1992.89:3429-3433和O'Callaghan et al.,Anal Biochem 1999.266:9-15所述制备生物素化特异性和对照pMHC单体,并固定在链霉亲和素偶联的CM-5传感器芯片上。所有测量均在25℃下的补充有0.005%吐温(Sigma)的PBS缓冲液(Sigma)中以恒定流速进行。为了测量亲和性,令可溶TCR的连续稀释系流过固定的pMHC,并测定每种浓度下的平衡响应值。通过绘制随蛋白质浓度变化的特异性平衡结合,然后以最小二乘法拟合至朗格缪尔(Langmuir)结合等式(假设1:1相互作用),来确定平衡解离常数(KD)。图11提供了从本发明的文库获得的TCR的代表性Biacore数据。
序列表
<110> 艾达普特免疫有限公司
英美偌科有限公司
<120> TCR文库
<130> P100066GB/AC
<140> GB1516272.0
<141> 2015-09-15
<160> 23
<170> SIPOSequenceListing 1.0
<210> 1
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> YOL22 Reverse Primer sequence
<400> 1
cattttcagg gatagcaagc 20
<210> 2
<211> 47
<212> DNA
<213> Artificial Sequence
<220>
<223> TRAV12-2 Primer sequence
<400> 2
ctcgcggccc agccggccat ggcccagaag gaggtggagc agaattc 47
<210> 3
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> TRBV5 Primer Sequence
<400> 3
ctattctcac agcgcgcagg acgctggagt cacccaaag 39
<210> 4
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> TRBV7 Primer Sequence
<400> 4
ctattctcac agcgcgcagg gtgctggagt ctcccag 37
<210> 5
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> TRBV11-1 Primer Sequence
<400> 5
ctattctcac agcgcgcagg aagctgaagt tgcccagtcc 40
<210> 6
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> TRBV11-3 Primer Sequence
<400> 6
ctattctcac agcgcgcagg aagctggagt ggttc 35
<210> 7
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> TRBV14 Primer Sequence
<400> 7
ctattctcac agcgcgcagg aagctggagt tactcagttc 40
<210> 8
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> TRBV20-1 Primer Sequence
<400> 8
ctattctcac agcgcgcagg gtgctgtcgt ctctcaacat 40
<210> 9
<211> 38
<212> DNA
<213> Artificial Sequence
<220>
<223> TRBV27 Primer Sequence
<400> 9
ctattctcac agcgcgcagg aagcccaagt gacccaga 38
<210> 10
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> TRBV30 Primer Sequence
<400> 10
ctattctcac agcgcgcagt ctcagactat tcatcaatgg 40
<210> 11
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> YOL237 Reverse Primer Sequence
<400> 11
gagtctctca gctggtacac gg 22
<210> 12
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> YOL240 Reverse Primer Sequence
<400> 12
agtgtggcct tttgggtgtg 20
<210> 13
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> YOL236 Forward Primer Sequence
<400> 13
ccgtgtacca gctgagagac tc 22
<210> 14
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> YOL238 Reverse Primer Sequence
<400> 14
gcgcgctgtg agaatagaaa g 21
<210> 15
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> YOL239 Forward Primer Sequence
<400> 15
cacacccaaa aggccacact 20
<210> 16
<211> 41
<212> DNA
<213> Artificial Sequence
<220>
<223> TRAV12-2 Fragment Forward Primer sequence
<400> 16
gcccagccgg ccatggccca gaaggaggtg gagcagaatt c 41
<210> 17
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> TRBV7 Fragment Including CDR2 Region Reverse Primer sequence
<400> 17
gggcagccct gatttgtctt gttgggcttc ataattgaa 39
<210> 18
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> TRBV7 Fragment Including CDR3 Region Forward Primer sequence
<400> 18
cagggcccag agtttctgac ttacttcaat tat 33
<210> 19
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> TRBV Specific Forward Primer sequence
<400> 19
gaagctgaag ttgcccagtc c 21
<210> 20
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> TRBV Reverse Primer sequence
<400> 20
gtctactcct ttgagcctct ctgc 24
<210> 21
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> CDR3 Region Forward Primer sequence
<400> 21
gcagagaggc tcaaaggagt agac 24
<210> 22
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> TRBV20-1 Fragment Including CDR1 Region Reverse Primer sequence
<400> 22
agttgtggcc tgaaagtcca gggaacggca ctcgat 36
<210> 23
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> TRBV20-1 Fragment Including CDR3 Region Forward Primer sequence
<400> 23
atcgagtgcc gttccctgga ctttcaggcc acaact 36
- 还没有人留言评论。精彩留言会获得点赞!