鞘内施用腺伴随病毒载体用于基因治疗的制作方法
申请人在此通过引用并入随同提交的序列表材料。
发明背景
生物医学和药物研究人员已致力于设计新的且更有效的治疗剂来治疗影响中枢神经系统的疾病。然而,中枢神经系统本身的生物学特性,包括血脑屏障在保护大脑方面的有效性,对药物递送提出了严峻的挑战。这导致缺乏许多中枢神经系统疾病诸如中风、神经变性疾病和脑肿瘤的可用治疗。
神经变性是神经元的结构或功能的进行性丧失(包括神经元的死亡)的涵盖性术语。许多神经变性疾病,包括肌萎缩性侧索硬化症(als)、帕金森氏病、阿尔茨海默氏病和亨廷顿氏病是神经变性过程的结果。已经描述了用于治疗此类神经变性疾病的各种疗法,包括单克隆抗体疗法。
腺伴随病毒(aav)是一种复制缺陷型细小病毒,其单链dna基因组长度为约4.7kb,包括145个核苷酸反向末端重复(itrs)。aav血清型2(aav2)基因组的核苷酸序列呈现于srivastava等人,jvirol,45:555-564(1983)中,其通过ruffing等人,jgenvirol,75:3385-3392(1994)校正。itrs内含有指导病毒dna复制(rep)、封装/包装和宿主细胞染色体整合的顺式作用序列。三个aav启动子(针对它们的相对图谱位置命名为p5、p19和p40)驱动编码rep和cap基因的两个aav内部开放阅读框的表达。两个rep启动子(p5和pi9),连同单一aav内含子的差异剪接(在核苷酸2107和2227处),导致从rep基因产生四种rep蛋白(rep78、rep68、rep52和rep40)。rep蛋白具有最终负责复制病毒基因组的多种酶促特性。cap基因由p40启动子表达并编码三种衣壳蛋白vp1、vp2和vp3。选择性剪接和非共有翻译起始位点负责产生三种相关的衣壳蛋白。单一共有聚腺苷酸化位点位于aav基因组的图谱位置95处。aav的生命周期和遗传学综述于muzyczka,currenttopicsinmicrobiologyandimmunology,158:97-129(1992)中。aav基因组的编码序列可以反式提供,使得可能生成携带目标基因、而不是内源病毒基因的aav载体。这些aav载体能够在体内进行基因转移。存在多种aav的血清型,并且提供不同的组织嗜性。已知血清型包括例如aavl、aav2、aav3、aav4、aav5、aav6、aav7、aav8、aav9、aav10和aav1。aav9描述于美国专利号7,198,951和gao等人,j.virol,78:6381-6388(2004)。
已经描述了携带靶向sod1的单链可分泌scfv抗体的aav载体的鞘内施用作为als的潜在治疗方法。参见,例如,ppatel等人,moleculartherapy(2014):22,3,498-510。
需要用于将免疫球蛋白构建体(包括但不限于全长抗体)递送至中枢神经系统的其他方法。
发明概述
提供了经配制用于鞘内递送至中枢神经系统的包含aav载体的组合物,其中所述组合物包含至少一个表达盒和药学上可接受的载体和/或赋形剂,所述表达盒含有与其表达控制序列可操作连接的编码用于递送至cns的免疫球蛋白产物的序列。在一个方面,所述组合物在不存在血脑屏障的化学或物理破坏的情况下施用。在一个实例中,免疫球蛋白构建体包含经修饰以对新生fc受体(fcrn)具有降低的或不可测量的亲和力的免疫球蛋白。
在一个方面,描述了经配制用于鞘内递送用于治疗中枢神经系统的病况的包含aav载体的组合物,其中所述组合物包含aav载体,所述aav载体包含靶向中枢神经系统的细胞的aav衣壳,所述衣壳其中已经包装至少一个aav反向末端重复序列和与其表达控制序列可操作连接的编码免疫球蛋白构建体的序列,所述组合物进一步包含药学上可接受的载体和/或赋形剂。合适地,编码的免疫球蛋白被突变或工程改造以对新生fc受体(fcrn)具有降低的或不可测量的亲和力。
本文提供了包含编码免疫球蛋白构建体的至少一种aav载体储备物的组合物可用于治疗神经病症和/或中枢神经系统的感染性疾病的用途。
在另一个方面,提供了治疗阿尔茨海默氏病的方法,其涉及向有需要的主体鞘内递送如本文所述的aav载体组合物,其中至少一种aav载体储备物表达对aβ、β分泌酶和/或tau蛋白特异性的免疫球蛋白。
在又另一个方面,提供了治疗als的方法,其涉及鞘内递送如本文所述的aav载体组合物,其中至少一种aav载体储备物表达对als酶超氧化物歧化酶1(sod1)及其变体(条件是抗sod1抗体不是scfv片段)、derlin-1-结合区域特异性的免疫球蛋白和/或针对神经突增生抑制剂的抗体构建体。
在又另一个实施方案中,提供了用于治疗帕金森氏病或相关突触核蛋白病的方法,其涉及鞘内递送如本文提供的aav载体组合物,其中至少一种aav载体储备物编码一种或多种富含亮氨酸的重复激酶2抗体、dardarin(lrrk2)抗体、α-突触核蛋白抗体和/或dj-1(park7)抗体。
在又一个进一步方面,本文提供了治疗多发性硬化症的方法,其涉及鞘内递送如本文提供的aav载体组合物,在所述组合物中,至少一种载体储备物编码针对a4-整联蛋白、cd20、cd25、il12、p40+il23p40、lingo-1、cd40和rhigm22、cd52、il17、cd19和/或sema4d中的一种或多种的免疫球蛋白。
在另一个方面,提供了用于治疗中枢神经系统的感染性疾病的方法,其涉及鞘内递送如本文提供的aav载体组合物,在所述组合物中,至少一种载体储备物编码针对引起所述感染性疾病的病原体的免疫球蛋白。实例非限制性地包括一种或多种针对结核分枝杆菌(结核病)、脑膜炎奈瑟氏菌(脑膜炎)、肺炎链球菌、单核细胞增生李斯特菌(李斯特菌病)、伯氏疏螺旋体(莱姆病)、人缺陷病毒(获得性免疫缺陷综合征)、疱疹家族病毒、水痘带状疱疹病毒、eb病毒(ebv)、巨细胞病毒和/或jc病毒中的一种或多种的免疫球蛋白。免疫球蛋白靶标的其他实例在本申请中别处提供,并且通过引用并入本文。
在一个方面,提供了用于治疗朊病毒相关疾病的方法,其包括鞘内递送aav载体组合物,其中至少一种载体储备物编码针对一种或多种主要朊病毒蛋白或prpsc的免疫球蛋白。
从以下发明详述,本发明的还有其他方面和优点将是显而易见的。
附图简述
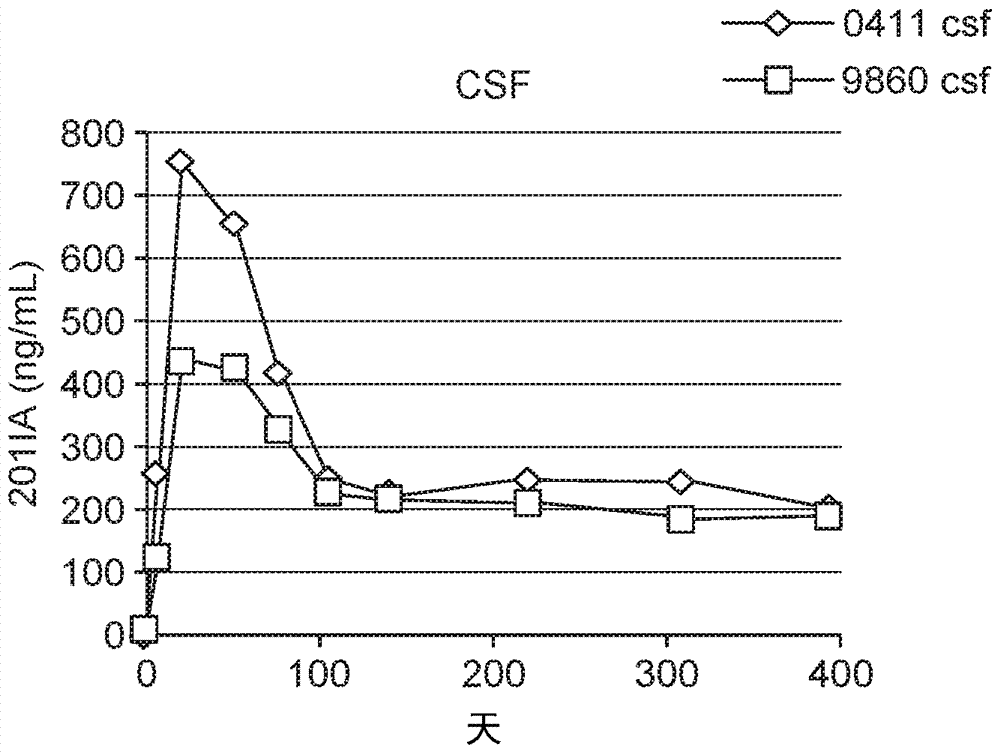
图1a是显示接受通过枕骨下穿刺鞘内递送的表达鸡β-肌动蛋白(cb)启动子下的免疫粘附素的aav9载体的两只动物的脑脊液(csf)中的恒河猴来源的免疫粘附素(201ia)的浓度的图。在载体施用后定期收集csf,其中400天是所示的最后收集时间点。csf的规模最高达800ng/ml。
图1b是显示接受通过枕骨下穿刺鞘内递送的表达鸡β-肌动蛋白(cb)启动子下的免疫粘附素的aav9载体的两只动物的血清中的恒河猴来源的免疫粘附素(201ia)的浓度的图。在载体施用后定期收集血清,其中400天是所示的最后收集时间点。血清的浓度规模最高达120µg/ml。
发明详述
本文所述的组合物和方案可用于将免疫球蛋白构建体递送至中枢神经系统。本文所述的组合物包含aav,所述aav具有用于鞘内递送至中枢神经系统(cns)的免疫球蛋白构建体。
如本文所用,免疫球蛋白构建体(包括如本文定义的抗体或抗体片段)编码结合位于中枢神经系统内的细胞表面抗原或受体的基于多肽的部分。这种受体可以位于已经感染中枢神经系统的细菌、病毒、真菌或其他病原体上,和/或为与中枢神经系统的病症和/或这种病原体相关的蛋白,例如分泌的蛋白和/或蛋白聚集体。
术语“免疫球蛋白”在本文用于包括抗体、其功能片段和免疫粘附素。抗体可以各种形式存在,包括例如多克隆抗体、单克隆抗体、骆驼单结构域抗体、胞内抗体(“胞内体”)、重组抗体、多特异性抗体、抗体片段,诸如fv、fab、f(ab)2、f(ab)3、fab'、fab'-sh、f(ab')2、单链可变片段抗体(scfv)、串联/双-scfv、fc、pfc'、scfvfc(或scfv-fc)、二硫化物fv(dsfv)、双特异性抗体(bc-scfv),诸如bite抗体;骆驼抗体、表面重构抗体(resurfacedantibodies)、人源化抗体、完全人抗体、单结构域抗体(sdab,也称为nanobody®)、嵌合抗体、包含至少一个人恒定区的嵌合抗体等。“抗体片段”是指结合其靶标(例如细胞表面抗原或受体)的免疫球蛋白可变区的至少一部分。
在一个实施方案中,如本文所述的组合物提供aav介导的免疫球蛋白的递送,所述免疫球蛋白包括片段可结晶区域(fc部分),包括诸如存在于全长抗体、双特异性抗体、免疫粘附素[含有免疫球蛋白恒定结构域和通常还有铰链和fc区]、单克隆抗体、重链骆驼或鲨免疫球蛋白中。aav递送全长抗体和两种抗体组合的用途已经描述于,例如,2015年5月13日提交的"compositionscomprisingaavexpressingdualantibodyconstructsandusesthereof",国际申请号pct/us15/30533、2015年4月24日提交的"methodsandcompositionsfortreatingmetastaticbreastcancerandothercancersinthebrain",国际申请号pct/us15/27491(其通过引用并入本文)中。任选地,组合物可以含有两种或更多种不同的如本文所述的aav-免疫球蛋白构建体。
在另一个实施方案中,本文所述的组合物提供了aav介导的免疫球蛋白的递送,所述免疫球蛋白排除fc部分,例如为fab(片段抗原-结合片段,通常通过ab的木瓜蛋白酶消化而形成),f(ab')2片段(含有两个抗原结合片段,通常通过ab的胃蛋白酶消化而形成),fab'片段(通常通过还原f(ab')2而形成),fab’-sh,f(ab)3(三特异性抗体片段),fv(仅含有两个可变结构域的免疫球蛋白),单一结构域抗体(sdab或vhh纳米抗体),例如骆驼或鲨鱼抗体,或scfv构建体。这种组合物可以包括两种或更多种不同的aavscfv构建体。
当关于蛋白或核酸使用时,术语“异源”表示蛋白或核酸包含两种或更多种在自然界中未以彼此相同的关系发现的序列或子序列。例如,通常重组产生核酸,其具有来自不相关基因的两个或更多个序列,其排列产生新的功能性核酸。例如,在一个实施方案中,核酸具有来自一个基因的启动子,其被排列以指导从不同基因表达编码序列。因此,关于编码序列,启动子是异源的。
如本文所用,“表达盒”是指包含免疫球蛋白基因(例如,免疫球蛋白可变区、免疫球蛋白恒定区、全长轻链、全长重链或免疫球蛋白构建体的另一个片段)、启动子且可包括其其他调节序列的核酸分子,所述盒可以经由遗传元件(例如质粒)递送至包装宿主细胞并包装至病毒载体的衣壳(例如,病毒颗粒)中。通常,用于生成病毒载体的这种表达盒含有本文所述的免疫球蛋白序列,其侧接病毒基因组的包装信号和其他表达控制序列、诸如本文所述的那些。
如本文所用,“载体储备物”或“aav载体储备物”是指其中已经包装编码如本文所定义的免疫球蛋白的非aav序列的aav病毒颗粒的基因组拷贝的群体。合适地,载体储备物包括足够数量的重组aav载体的基因组拷贝(gc)以实现期望的生理效应。在获得期望的生理效应的情况下,组合物、剂量或方案中的载体储备物的量可以被称为“有效量”的raav载体或载体储备物。
除非另有指明,“中枢神经系统”是指脊髓和大脑,并且与排除脊髓和大脑的“外周神经系统”形成对照。在中枢神经系统内存在不同的细胞类型,包括神经元细胞和神经胶质细胞。成熟系统中的神经胶质包括星形胶质细胞、少突胶质细胞和小神经胶质细胞。用于本发明中的载体的aav衣壳优选选自将在中枢神经系统的这些细胞类型中的至少一种中转导和/或表达的那些。
如本文所用,术语“鞘内递送”或“鞘内施用”是指经由注射入椎管、更具体地注射入蛛网膜下腔、使得其到达脑脊液(csf)的药物的施用途径。鞘内递送可以包括腰椎穿刺、心室内(包括脑室内(icv))、枕骨下/脑池内和/或c1-2穿刺。例如,可以通过腰椎穿刺的方式引入材料用于扩散整个蛛网膜下腔。在另一个实例中,注射可以进入小脑延髓池。
如本文所用,术语“脑池内递送”或“脑池内施用”是指将药物直接施用入小脑延髓池(cisternamagnacerebellomedularis)的脑脊液中的途径,更具体地经由枕骨下穿刺或通过直接注射入小脑延髓池或经由永久定位管。
如上所述,术语“约”当用于修饰数值时意指±10%的变化,除非另外指明。
如整个本说明书和权利要求中所用,术语“包含(comprise)”和“含有(contain)”以及其变体(包括“包含(comprises)”、“包含(comprising)”、“含有(contains)”和“含有(containing)”)以及其他变体包括其他组分、要素、整数、步骤等。术语“由...组成(consistsof)”或“由...组成(consistingof)”排除其他组分、要素、整数、步骤等。
为了从aav载体表达,可以工程改造或选择编码可用于治疗一种或多种神经变性病症的免疫球蛋白的核酸构建体,用于经由本发明的aav组合物递送。此类病症可以包括但不限于传染性海绵状脑病(例如creutzfeld-jacob疾病),帕金森氏病,肌萎缩性侧索硬化症(als),多发性硬化症,阿尔茨海默氏病,亨廷顿病,卡纳万氏病,创伤性脑损伤,脊髓损伤(ati335,novartis的抗nogo1),偏头痛(alderbiopharmaceuticals的ald403;eli的ly2951742;labrysbiologics的rn307),溶酶体贮积病,中风以及影响中枢神经系统的感染性疾病。
还有其他核酸可以编码针对含有富含亮氨酸的重复和免疫球蛋白样结构域的蛋白1(lingo-1)的免疫球蛋白,所述lingo-1是nogo受体的功能性组分并且与具有多发性硬化症、帕金森氏病或特发性震颤的患者中的特发性震颤相关。一种这种市售抗体是ocrelizumab(biogen,biib033)。参见例如美国专利8,425,910。
在一个实施方案中,核酸构建体编码可用于具有als的患者的免疫球蛋白构建体。合适抗体的实例包括针对als酶超氧化物歧化酶1(sod1)及其变体(例如als变体g93a,c4f6sod1抗体)的抗体;ms785,其针对derlin-1结合区域);针对神经突增生抑制剂(nogo-a或网状内皮素4)的抗体,例如gsk1223249,ozanezumab(人源化的,gsk,也被描述为可用于多发性硬化症)。
可以设计或选择编码可用于具有阿尔茨海默氏病的患者的免疫球蛋白的核酸序列。此类抗体构建体包括例如adumanucab(biogen),bapineuzumab(elan;针对aβ的氨基末端的人源化mab);solanezumab(elililly,针对可溶性aβ的中心部分的人源化mab);gantenerumab(chugai和hoffmann-laroche,是针对aβ的氨基末端和中心部分的完整人mab);crenezumab(genentech,人源化mab,其作用于单体和构象表位,包括aβ的寡聚和原纤维形式;ban2401(esaico.,ltd,人源化免疫球蛋白g1(igg1)mab,其选择性结合aβ原纤维并被认为增强aβ原纤维的清除和/或中和其对脑中的神经元的毒性作用);gsk933776(针对aβ的氨基末端的人源化igg1单克隆抗体);aab-001,aab-002,aab-003(fc-工程改造的bapineuzumab);sar228810(针对原纤维和低分子量aβ的人源化mab);biib037/bart(针对不溶性原纤维人aβ的完整人igg1,biogenidec),抗aβ抗体诸如m266,tg2576(对于aβ寡聚体的相对特异性)[brody和holtzman,annurevneurosci,2008;31:175-193]。其他抗体可以靶向β-淀粉样蛋白、aβ、β分泌酶和/或tau蛋白。
在本文实施例中举例说明了三种抗aβscfv构建体,其被设计为消除全长抗淀粉样蛋白β(aβ)的fc区。设计这些构建体以降低淀粉样蛋白相关成像异常的风险和/或限制血管暴露于高单克隆抗体浓度。在一个实施方案中,说明性scfv结合纤维状β-淀粉样蛋白。参见例如,具有seqidno:8的氨基酸序列的scfv,其中重链可变区具有氨基酸21至143的序列,且轻链可变区具有氨基酸159至265的序列。还涵盖该scfv的变体。例如,关于seqidno:8,不同的信号序列(氨基酸1-20),可以用不同的接头取代gly-ser接头(氨基酸144-158),或者可以移除his标签(氨基酸266至271)。本文提供了编码本文所述的scfv氨基酸序列的核酸序列。参见例如,seqidno:7。
在另一个实施方案中,说明性scfv针对寡聚的、可溶性和纤维状β-淀粉样蛋白。参见,例如,seqidno:10的氨基酸序列,其具有氨基酸21至131的重链可变区和氨基酸147至258的轻链可变区。还涵盖该scfv的变体。例如,关于seqidno:10,不同的信号序列(氨基酸1-20),可以用不同的接头取代gly-ser接头(氨基酸132至146),或者可以移除his标签(氨基酸259-264)。本文提供了编码本文所述的scfv氨基酸序列的核酸序列。参见,例如,seqidno:9。
在又另一个实施方案中,说明性scfv针对可溶性β-淀粉样蛋白。参见例如,seqidno:12的氨基酸序列,其具有氨基酸21至131的重链可变区和氨基酸147至258的轻链可变区。还涵盖该scfv的变体。例如,关于seqidno:12,不同的信号序列(氨基酸1-20),可以用不同的接头取代gly-ser接头(氨基酸132至146),或者可以移除his标签(氨基酸259-264)。本文提供了编码本文所述的scfv氨基酸序列的核酸序列。参见,例如,seqidno:11。
在还有其他实施方案中,抗β-淀粉样蛋白抗体衍生自靶向β-淀粉样蛋白的igg4单克隆抗体以使效应子功能最小化,或者选择除了缺乏fc区的的scfv以外的构建体以避免淀粉样蛋白相关的成像异常(aria)和炎性反应。在这些实施方案中的某些中,scfv构建体中的一种或多种的重链可变区和/或轻链可变区用于如本文提供的另一种合适的免疫球蛋白构建体中。与含有fc区的免疫球蛋白相比,这些scfv和其他工程改造的免疫球蛋白可以降低血清中免疫球蛋白的半衰期。降低抗淀粉样蛋白分子的血清浓度可以进一步降低aria的风险,因为血清中极高水平的抗淀粉样蛋白抗体可以使具有高淀粉样斑块负荷的大脑血管不稳定,引起血管渗透性。
编码用于治疗具有帕金森氏病的患者的其他免疫球蛋白构建体的核酸可以被工程改造或设计为表达构建体,包括例如富含亮氨酸的重复激酶2、dardarin(lrrk2)抗体;抗突触核蛋白和α-突触核蛋白抗体和dj-1(park7)抗体。其他抗体可以包括prx002(prothena和roche)帕金森氏病和相关突触核蛋白病。这些抗体,特别是抗突触核蛋白抗体也可用于治疗一种或多种溶酶体贮积病。
可以设计或选择编码用于治疗多发性硬化症的免疫球蛋白构建体的核酸构建体。此类免疫球蛋白可以包括或衍生自抗体,诸如2006年批准的那他珠单抗(人源化抗a4-整联蛋白,inata,tysabri,biogenidecandelanpharmaceuticals),阿仑单抗(campath-1h,人源化抗cd52),利妥昔单抗(rituzin,嵌合抗cd20),达利珠单抗(zenepax,人源化抗cd25),ocrelizumab(人源化,抗cd20,roche),优特克单抗(cnto-1275,人抗il12p40+il23p40);抗lingo-1和ch5d12(嵌合抗cd40)和rhigm22(髓鞘再生单克隆抗体;acordaandthemayofoundationformedicaleducationandresearch)。如本文所述,还可以经由aav载体递送其他抗a4-整联蛋白抗体、抗cd20抗体、抗cd52抗体、抗il17、抗cd19、抗sema4d和抗cd40抗体。
本发明还考虑aav介导的针对中枢神经系统的各种感染的抗体的递送。此类感染性疾病可以包括真菌性疾病,诸如隐球菌性脑膜炎,脑脓肿,脊髓硬膜外感染,其由例如新型隐球菌(cryptococcusneoformans)、粗球孢子菌(coccidioidesimmitis)、毛霉目(mucorales)、曲霉菌属种(aspergillusspp)和念珠菌属种(candidaspp)引起;原生动物的,诸如弓形体病,疟疾和原发性阿米巴脑膜脑炎,其由病原体诸如刚地弓形虫(toxoplasmagondii)、有钩绦虫(taeniasolium)、恶性疟原虫(plasmodiumfalciparus)、曼森裂头绦虫(spirometramansonoides)(sparaganoisis)、棘球绦虫属种(引起神经性包囊病)和脑阿米巴病引起;细菌的,诸如例如结核病、麻风病、神经梅毒、细菌性脑膜炎、莱姆病(伯氏疏螺旋体)、落基山斑疹热(立克次体)、cns诺卡氏菌病(诺卡氏菌属种)、cns结核病(结核分枝杆菌),cns李斯特菌病(单核细胞增生性李斯特菌)、脑脓肿和神经疏螺旋体病;病毒性感染,诸如例如病毒性脑膜炎、东方马脑炎(eee)、圣路易斯脑炎、西尼罗河病毒和/或脑炎、狂犬病、加利福尼亚脑炎病毒、lacrosse脑炎、麻疹脑炎、脊髓灰质炎,其可以由疱疹家族病毒(hsv)、hsv-1、hsv-2(新生儿单纯疱疹性脑炎)、水痘带状疱疹病毒(vzv)、bickerstaff脑炎、eb病毒(ebv)、巨细胞病毒(cmv,诸如tcn-202,由theraclonesciences开发中)、人疱疹病毒6(hhv-6)、b病毒(猴疱疹病毒)、黄病毒脑炎、日本脑炎、穆雷谷热、jc病毒(进行性多灶性脑白质病)、nipah病毒(niv)、麻疹(亚急性硬化性全脑炎)引起;和其他感染,诸如例如,亚急性硬化性全脑炎、进行性多灶性脑白质病;人免疫缺陷病毒(获得性免疫缺陷综合症(aids));酿脓链球菌(streptococcuspyogenes)和其他β-溶血性链球菌(例如,与链球菌感染相关的儿科自身免疫性神经精神病学疾病,pandas)和/或syndenham氏舞蹈病和guillain-barre综合征以及朊病毒。
合适的抗体构建体的实例可以包括例如在wo2007/012924a2,2015年1月29日(其通过引用并入本文)中所述的那些。
例如,其他核酸序列可以编码抗朊病毒免疫球蛋白构建体。此类免疫球蛋白可以针对主要朊病毒蛋白(prp,朊病毒蛋白或蛋白酶抗性蛋白,也称为cd230(分化簇230))。提供了prp的氨基酸序列,例如http://www.ncbi.nlm.nih.gov/protein/np_000302,其通过引用并入本文。该蛋白可以以多种同种型(正常prpc、致病prpsc和位于线粒体中的同种型)存在。错误折叠版本prpsc与各种认知病症和神经变性疾病、诸如creutzfeldt–jakob疾病、牛海绵状脑病、gerstmann–sträussler–scheinker综合征、致命性家族性失眠症和库鲁病相关。
一旦选择靶标和免疫球蛋白,可以获得和/或合成所选免疫球蛋白(例如重链和/或轻链)的编码序列。对蛋白、肽或多肽(例如,作为免疫球蛋白)进行测序的方法是本领域技术人员众所周知的。一旦蛋白序列已知,就存在基于网络和市售的计算机程序以及基于服务的公司(将氨基酸序列反向翻译成核酸编码序列)。参见例如,emboss的backtranseq,http://www.ebi.ac.uk/tools/st/;geneinfinity(http://www.geneinfinity.org/sms/sms_backtranslation.html);expasy(http://www.expasy.org/tools/)。在一个实施方案中,设计rna和/或cdna编码序列用于在人细胞中进行最佳表达。
密码子优化的编码区可以通过各种不同的方法设计。这种优化可以使用在线可得的方法(例如geneart)、公开的方法或提供密码子优化服务的公司(例如dna2.0(menlopark,ca))进行。一种密码子优化算法描述于例如在美国国际专利公开号wo2015/012924(其通过引用并入本文)中。还参见例如美国专利公开号2014/0032186和美国专利公开号2006/0136184。合适地,修饰产物的开放阅读框(orf)的整个长度。然而,在一些实施方案中,仅可以改变orf的片段。通过使用这些方法之一,可以将频率应用于任何给定的多肽序列,并产生编码多肽的密码子优化的编码区的核酸片段。
许多选项可用于对密码子进行实际改变或合成如本文所述设计的密码子优化的编码区。此类修饰或合成可以使用本领域普通技术人员众所周知的标准和常规分子生物学操作来进行。在一种方法中,通过标准方法合成长度各自为80-90个核苷酸且跨越期望序列的长度的一系列互补寡核苷酸对。合成这些寡核苷酸对,使得它们在退火后形成含有粘性末端的80-90个碱基对的双链片段,例如,合成该对中的每个寡核苷酸以延伸超出与该对中的另一个寡核苷酸互补的区域的3、4、5、6、7、8、9、10个或更多个碱基。设计每对寡核苷酸的单链末端以与另一对寡核苷酸的单链末端退火。使寡核苷酸对退火,然后使这些双链片段中的约5-6个经由粘性单链末端退火在一起,然后将它们连接在一起并克隆至标准细菌克隆载体中,例如可得自invitrogencorporation,carlsbad,calif的topo®载体。然后通过标准方法对构建体进行测序。制备由80至90个碱基对片段连接在一起的5至6个片段组成(即约500个碱基对的片段)的这些构建体中几个,使得整个期望的序列以一系列质粒构建体表示。然后用适当的限制酶切割这些质粒的插入物并连接在一起以形成最终构建体。然后将最终构建体克隆至标准的细菌克隆载体中,并测序。额外方法对于技术人员将立即显而易见。此外,基因合成容易商业获得。
本文所述的免疫球蛋白基因可用于表达“野生型”、公开或市售或其他已知的恒定免疫球蛋白结构域,或可以被工程改造以降低对存在于免疫球蛋白上的fc结合位点的亲和力或消除与存在于免疫球蛋白上的fc结合位点的结合。存在几种不同类型的fc受体,其根据它们识别的抗体的类型分类。如本文所用,“fcrn”是指结合igg的新生fc受体。其在结构上与mhci类蛋白类似。在人中,其由fcgrt基因编码。fc受体位于各种细胞类型(包括例如血脑屏障的上皮细胞)上。如本文所用的术语“fcrn结合结构域”是指直接或间接结合fcrn的蛋白结构域。fcrn可以是哺乳动物fcrn。在进一步实施方案中,fcrn是人fcrn。直接结合fcrn的fcrn结合结构域是抗体fc区。同时,能够结合具有人fcrn结合活性的多肽诸如白蛋白或igg的区域可以经由白蛋白、igg等间接结合人fcrn。因此,这种人fcrn结合区域可以是结合具有人fcrn结合活性的多肽的区域。如本文所用的术语“fc区”是指直接结合fcrn、哺乳动物fcrn或人fcrn的fcrn结合结构域。具体而言,fc区是抗体的fc区。fc区可以是哺乳动物fc区或更特别是人fc区。具体而言,fc区可以位于人免疫球蛋白(ch2和ch3)的第二和第三恒定结构域内。此外,fc区可以是ch2和ch3的铰链。在一个实施方案中,免疫球蛋白构建体是igg。在一个进一步实施方案中,fc区是人igg1的fc区。同样可以使用其他ig同种型。
用于制备这种比对的方法和计算机程序是可用的,并且是本领域技术人员众所周知的。取代还可以写成(由单字母代码标识的氨基酸)-位置#-(由单字母代码标识的氨基酸),其中第一氨基酸是被取代的氨基酸,且第二氨基酸是在指定位置的取代氨基酸。如本文所用的术语“取代”和“氨基酸的取代”和“氨基酸取代”是指氨基酸序列中的氨基酸被另一氨基酸替代,其中后者不同于被替代的氨基酸。用于替代氨基酸的方法是本领域技术人员众所周知的,并且包括但不限于编码氨基酸序列的核苷酸序列的突变。在igg中进行氨基酸取代的方法描述于例如wo2013/046704中,其通过引入并入,用于讨论氨基酸修饰技术,尽管该文献描述了增加fcrn亲和力,而不是减少或去除如本文所述的结合亲和力。
上述术语“氨基酸取代”及其同义词意欲涵盖通过用另一取代氨基酸替代氨基酸来修饰氨基酸序列。取代可以是保守取代。关于两个氨基酸,术语保守意指氨基酸具有本领域技术人员公认的共同特性。关于两个氨基酸,术语非保守是指在本领域技术人员所认识的至少一种特性上具有差异的氨基酸。例如,此类特性可以包括具有疏水性非酸性侧链的氨基酸、具有疏水性侧链的氨基酸(其可以进一步区分为酸性或非酸性)、具有脂肪族疏水性侧链的氨基酸、具有芳香族疏水性侧链的氨基酸、具有极性中性侧链的氨基酸、具有带电侧链的氨基酸、具有带电酸性侧链的氨基酸和具有带电碱性侧链的氨基酸。天然存在的和非天然存在的氨基酸都是本领域已知的,并且可以在实施方案中用作取代氨基酸。因此,保守氨基酸取代可以涉及用具有疏水性侧链的不同氨基酸改变具有疏水性侧链的第一氨基酸;而非保守氨基酸取代可以涉及用具有不同侧链(例如碱性疏水性侧链或亲水性侧链)的不同氨基酸改变具有酸性疏水性侧链的第一氨基酸。本领域技术人员可以确定还其他的保守或非保守的改变。
在还有其他实施方案中,给定位置处的取代将是氨基酸或氨基酸组中的一个,其对于本领域技术人员实现本文所鉴定的目标是显而易见的。
在一个实施方案中,将如本文所定义的免疫球蛋白构建体工程改造,使得切除位于免疫球蛋白fc区的保守区上的天然序列以消除与fcrn的结合并最小化或消除蛋白性免疫球蛋白构建体穿过血脑屏障(离开cns区域)且进入全身循环的转运。在一个实例中,这可以通过改变fcrn结合结构域的一个或多个氨基酸,例如通过修饰所选氨基酸的密码子来实现。参见,例如,us8618252b2。
另外或可替代地,选择经工程改造以具有增强的效应子功能的免疫球蛋白构建体(例如fc变体)。参见,例如,t.matsushita,koreanjhematol,2011sep;46(3):148-150;美国专利6,946,292。
本文用于鉴定突变体的位置的重链氨基酸编号基于eu编号系统[imgt独特编号,edelman,g.m.等人,proc.natl.acad.usa,63,78-85(1969);http://www.imgt.org/-imgtscientificchart/-numbering/hu_ighgnber.html]]并且是指在fcrn结合结构域中(特别是在fc区中)的位置。以类似的方式,取代被表示为例如“eu387r”或“eu440e”,其中在“eu”之后给出的数字指示根据eu编号的取代的位置,并且在数字之后的字母是以单字母代码给出的取代的氨基酸。其他编号系统包括,例如,kabat,e.a.,t.t.wu,h.m.perry,k.s.gottesman,c.foeler.(1991)sequencesofproteinsofimmunologicalinterest.no.91-3242u.s.publichealthservices,nationalinstitutesofhealth,bethesda)。
所述启动子可以选自不同来源,例如人巨细胞病毒(cmv)立即早期增强子/启动子、sv40早期增强子/启动子、jc多瘤病毒启动子、髓鞘碱性蛋白(mbp)或胶质原纤维酸性蛋白(gfap)启动子、单纯疱疹病毒(hsv-1)潜伏相关启动子(lap)、劳氏肉瘤病毒(rsv)长末端重复序列(ltr)启动子、神经元特异性启动子(nse)、血小板衍生生长因子(pdgf)启动子、hsyn、黑色素浓集激素(mch)启动子、cba、基质金属蛋白启动子(mpp)和鸡β-肌动蛋白启动子。
在某些实施方案中,本文所述的表达盒含有位于重链和轻链的编码区之间的至少一个内部核糖体结合位点,即ires。或者,重链和轻链可以通过弗林蛋白酶-2a自我切割肽接头分离[参见,例如,radcliffeandmitrophanous,genetherapy(2004),11,1673-1674。表达盒可以含有至少一种增强子,即cmv增强子。还有其他增强子元件可以包括例如载脂蛋白增强子、斑马鱼增强子、gfap增强子元件和脑特异性增强子(诸如wo2013/1555222中所述)、土拨鼠肝炎后转录后调控元件。另外或可替代地,可以选择其他,例如,杂合人巨细胞病毒(hcmv)-立即早期(ie)-pdgr启动子或其他启动子-增强子元件。为了增强表达,其他元件可以是内含子(如promega™内含子或嵌合的鸡珠蛋白样人免疫球蛋白内含子)。
在两个或更多个核酸或多肽序列的上下文中,术语“同一的”或百分比“同一性”是指两个或更多个序列或子序列在指定区域(例如,当在比较窗口或指定区域上比较和比对以获得最大对应性时,本文提供的任何一个修饰的orf)是相同的或具有相同的氨基酸残基或核苷酸的特定百分比(即,约70%同一性,优选75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更高同一性,如使用具有下述默认参数的blast或blast2.0序列比较算法,或通过手动比对和视觉检查(参见例如ncbi网站等)所测量。作为另一个实例,可以使用gcg版本6.1中的程序fasta来比较聚核苷酸序列。fasta提供查询和搜索序列之间最佳重叠区域的比对和百分比序列同一性。例如,核酸序列之间的百分比序列同一性可以使用如在gcg版本6.1(其通过引用并入本文)中提供的具有其默认参数(字长6和评分矩阵的nopam因子)的fasta来确定。通常,这些程序以默认设置使用,但本领域技术人员可以根据需要改变这些设置。或者,本领域技术人员可以利用另一种算法或计算机程序,其提供至少与参考算法和程序提供一样的同一性或比对水平。该定义也指或可以应用于序列的互补序列。该定义还包括具有缺失和/或添加的序列,以及具有取代的序列。如下所述,优选的算法可以负责缺口(gap)等问题。优选地,同一性存在于至少约25、50、75、100、150、200个氨基酸或核苷酸长度的区域上,并且通常在225、250、300、350、400、450、500个氨基酸或核苷酸长度的区域上或在氨基酸或核酸序列的全长上。
通常,当基于氨基酸序列准备比对时,比对含有相对于参考aav序列如此鉴定的插入和缺失,并且氨基酸残基的编号基于为比对提供的参考标度。然而,任何给定的aav序列可以具有比参考标度更少的氨基酸残基。在本发明中,当讨论亲本序列时,术语“相同位置”或“对应位置”是指相对于比对序列的参考标度,在每个序列中位于相同残基编号的氨基酸。然而,当从比对中取出时,每种蛋白可能具有位于不同残基编号的这些氨基酸。使用各种公共或市售的多序列比对程序中的任何一种来进行比对。序列比对程序可用于氨基酸序列,例如“clustalx”、“map”、“pima”、“msa”、“blockmaker”、“meme”和“match-box”程序。通常,这些程序中的任何一个都以默认设置使用,但本领域技术人员可以根据需要改变这些设置。或者,本领域技术人员可以利用另外的算法或计算机程序,其提供至少与参考算法和程序提供的一样的同一性或比对水平。参见例如j.d.thomson等人,nucl.acids.res.,“acomprehensivecomparisonofmultiplesequencealignments”,27(13):2682-2690(1999)。
在另一个实施方案中,提供了对fcrn的亲和力消除并保留生理有效活性的修饰的免疫球蛋白。可以选择一个或多个氨基酸修饰以消除与fcrn的功能性结合。在一个实施方案中,突变将免疫球蛋白对fcrn的结合亲和力降低至天然蛋白的小于10%。合适地,具有这些突变的免疫球蛋白基本上正常结合所有其他fc受体。一旦选择氨基酸序列,就可以设计核酸序列和/或可以如上所述工程改造前述序列。通过使用本领域技术人员已知的定点诱变或其他基因工程技术工程改造核酸编码区来进行这些修饰。
在一个实施方案中,本文所述的免疫球蛋白基因被工程改造至可用于生成aav载体的遗传元件(例如,质粒)中,所述遗传元件转移其上携带的免疫球蛋白构建体序列。本文的实施例和seqidno:13、14和15分别说明具有三种不同的针对aβ靶标的scfv构建体的aav载体基因组。可以通过任何合适的方法,包括转染、电穿孔、脂质体递送、膜融合技术、高速dna包被的小丸、病毒感染和原生质体融合将所选载体递送至aav包装细胞。也可以制备稳定的包装细胞。用于制备此类构建体的方法对于具有核酸操作技能的人员是已知的,并且包括基因工程改造、重组工程改造和合成技术。参见例如molecularcloning:alaboratorymanual,green和sambrook编,coldspringharborpress,coldspringharbor,ny(2012)。
aav载体
如本文所述的aav载体可以包含一个或多个核酸序列,其各自编码免疫球蛋白构建体的重链和/或轻链多肽或其他多肽中的一种或多种。合适地,组合物含有一种或多种aav载体,其含有编码在体内形成免疫球蛋白构建体的所有多肽的核酸序列。例如,全长抗体由四种多肽组成:两个拷贝的重(h)链多肽和两个拷贝的轻(l)链多肽。每条重链含有一个n-端可变(vh)区和三个c-端恒定(ch1、ch2和ch3)区,并且每条轻链含有一个n-端可变(vl)区和一个c-端恒定(cl)区。每对轻链和重链的可变区形成抗体的抗原结合位点。在这方面,如本文所述的aav载体可以包含编码免疫球蛋白构建体的重链多肽(例如恒定可变区)和轻链多肽的单一核酸序列。或者,aav载体可以包含编码至少一种重链恒定多肽和至少一种重链可变多肽的第一表达盒和编码免疫球蛋白构建体的恒定和可变轻链多肽的第二表达盒。在又另一个实施方案中,aav载体可以包含编码第一重链多肽的第一表达盒、编码第二重链多肽的第二表达盒,并且编码轻链多肽的第三表达盒由两条重链共有。在另一个实施方案中,aav载体可以表达1、2、3或4个scfv开放阅读框(orf),其各自可以是相同的或不同的。
通常,用于aav载体的表达盒包含aav5’反向末端重复序列(itr)、编码免疫球蛋白构建体的序列和任何调节序列以及aav3’itr。然而,这些元件的其他配置可以是合适的。已经描述了称为δitr的5'itr的缩短版本,其中缺失d序列和末端决定位点(terminalresolutionsite,trs)。在其他实施方案中,使用全长aav5’和3’itrs。
在待产生假型aav的情况下,表达中的itr选自不同于衣壳的aav来源的来源。例如,可以选择aav2itr用于与具有靶向cns或cns内的组织或细胞的特定效力的aav衣壳一起使用。在一个实施方案中,为了方便起见使用来自aav2的itr序列或其缺失形式(∆itr),以加速法规审批。然而,可以选择来自其他aav来源的itr。在itr的来源来自aav2且aav衣壳来自另一种aav来源的情况下,所得载体可以被称为假型的。然而,可以使用aavitr的其他来源。
缩写“scaav”是指自我互补的。“自我互补的aav”是指其中由重组aav核酸序列携带的编码区已被设计为形成分子内双链dna模板的构建体。感染后,不是等待细胞介导的第二条链的合成,而是scaav的两个互补的一半将缔合以形成一个准备立即复制和转录的双链dna(dsdna)单位。参见例如dmmccarty等人,“self-complementaryrecombinantadeno-associatedvirus(scaav)vectorspromoteefficienttransductionindependentlyofdnasynthesis”,genetherapy,(august2001),vol8,number16,第1248-1254页。自我互补的aav描述于例如美国专利号6,596,535;7,125,717;和7,456,683,其各自以其整体通过引用并入本文。
表达盒通常含有作为表达控制序列的一部分,例如位于所选5’itr序列和编码免疫球蛋白构建体的序列之间的启动子序列。可以在本文所述的载体中使用组织型特异性启动子、组成型启动子、可调节启动子[参见,例如,wo2011/126808和wo2013/04943]或对生理学线索响应的启动子。除了启动子以外,表达盒和/或载体可以含有其他适当的转录起始、终止、增强子序列,有效的rna加工信号诸如剪接和聚腺苷酸化(polya)信号;稳定胞质mrna的序列;增强翻译效力的序列(即,kozak共有序列);增强蛋白稳定性的序列;以及当期望时,增强编码的产物的分泌的序列。
这些控制序列“可操作连接”至免疫球蛋白构建体基因序列。如本文所用,术语“可操作连接”是指与目标基因邻接的表达控制序列,以及反式或在远处起作用以控制目标基因的表达控制序列两者。
在一个实施方案中,提供了自我互补的aav。该病毒载体可以含有∆5’itr和aav3’itr。在另一个实施方案中,提供了单链aav病毒载体。用于生成和分离适合递送至主体的aav病毒载体的方法是本领域已知的。参见,例如,美国专利7790449;美国专利7282199;wo2003/042397;wo2005/033321,wo2006/110689;和us7588772b2]。在一种系统中,生产者细胞系用编码侧接itr的转基因的构建体和编码rep和cap的构建体瞬时转染。在第二种系统中,稳定供应rep和cap的包装细胞系用编码侧接itr的转基因的构建体瞬时转染。在这些系统中的每一种中,aav病毒粒子响应于辅助性腺病毒或疱疹病毒感染而产生,需要将raav从污染性病毒中分离出来。更近来,已经开发了不需要用辅助病毒感染来恢复aav的系统-所需的辅助功能(即,腺病毒e1、e2a、va和e4或疱疹病毒ul5、ul8、ul52和ul29以及疱疹病毒聚合酶)也由系统反式提供。在这些较新的系统中,辅助功能可以通过用编码所需辅助功能的构建体瞬时转染细胞来提供,或者细胞可以被工程改造以稳定地含有编码辅助功能的基因,其表达可以在转录水平或转录后水平进行控制。在又另一种系统中,通过用基于杆状病毒的载体感染将侧接itr和rep/cap基因的转基因引入昆虫细胞中。关于这些生产系统的综述,通常参见例如zhang等人,2009,“adenovirus-adeno-associatedvirushybridforlarge-scalerecombinantadeno-associatedvirusproduction,”humangenetherapy20:922-929,其各自的内容以其整体通过引用并入本文。制备和使用这些和其他aav生产系统的方法也描述于以下美国专利中,其各自的内容以其整体通过引用并入本文:5,139,941;5,741,683;6,057,152;6,204,059;6,268,213;6,491,907;6,660,514;6,951,753;7,094,604;7,172,893;7,201,898;7,229,823;和7,439,065。
可以通过将多于一个转录单位组合至单个表达盒中、因此减少需要的调节序列的量来保留可用的包装空间。例如,单一启动子可以指导编码两个或三个或更多个基因的单一rna的表达,并且下游基因的翻译由ires序列驱动。在另一个实例中,单一启动子可以指导rna的表达,所述rna在单个开放阅读框(orf)中含有通过编码自我切割肽(例如,2a)和/或蛋白酶识别位点(例如,弗林蛋白酶)的序列与彼此分开的两个或三个或更多个基因。因此orf编码单一多蛋白,其在翻译期间或翻译之后被切割成单个蛋白(诸如例如,重链和轻链)。然而,应该注意的是,尽管这些ires和多蛋白系统可以用于节约aav包装空间,但它们仅可用于表达可由相同启动子驱动的组分。在另一个替代方案中,可以通过提供可以退火以形成头至尾多联体的两个基因组的aavitrs来增加aav的转基因能力。在一个进一步替代方案中,可以选择双向启动子。
在以下实例中,描述了具有aav9衣壳的aav载体。如本文所用,“aav9衣壳”是指具有genbank登录号:aas99264(其通过引用并入本文)的氨基酸序列的aav9。本发明涵盖从该编码序列的一些变化,其可以包括与genbank登录号:aas99264和us7906111(也是wo2005/033321)中提及的氨基酸序列具有至少约95%、至少约97%或至少约99%同一性的序列。已经描述了生成衣壳的方法,因此编码的序列,和用于产生raav病毒载体的方法。参见,例如,gao,等人,proc.natl.acad.sci.u.s.a.100(10),6081-6086(2003)和us2013/0045186a1。aav9载体的生成描述于例如美国专利号7,906,111(其通过引用并入本文)中。然而,可以选择aav衣壳和其他病毒元件的其他来源,其他免疫球蛋白构建体和其他载体元件也可以如此。生成aav载体的方法已广泛描述于文献和专利文件中,包括例如wo2003/042397;wo2005/033321、wo2006/110689;us7588772b2。aav衣壳的来源可以选自靶向cns、cns内的特定细胞和/或特定抗原或受体的aav。合适的aav可以包括例如aav9[us7,906,111;us2011-0236353-a1]、rh10[wo2003/042397]和/或hu37[参见,例如us7,906,111;us2011-0236353-a1]。然而,其他aav,包括例如aav1、aav2、aav3、aav4、aav5、aav6、aav7、aav8、[美国专利7790449;美国专利7282199]以及其他aav可被选择用于制备本文所述的aav载体。
用途和方案
合适地,设计本发明的组合物,使得aav载体携带编码免疫球蛋白构建体的核酸表达盒和指导其免疫球蛋白在所选细胞中表达的调节序列。在将载体施用至cns中后,载体将表达盒递送至cns并在体内表达蛋白性免疫球蛋白构建体。描述了本文描述的组合物用于治疗目的的用途,以及这些组合物在各种方案中的用途,其可以任选地涉及递送一种或多种其他活性剂。
如上所述,组合物可以含有如本文所述的单一类型的aav载体,其含有用于体内递送免疫球蛋白构建体的表达盒。或者,组合物可以含有两种或更多种不同的aav载体,其各自其中已经包装不同的表达盒。例如,两种或更多种不同的aav可以具有不同的表达盒,其表达免疫球蛋白多肽,其在体内组装以形成单一功能性免疫球蛋白构建体。在另一个实例中,两种或更多种aav可以具有不同的表达盒,其表达针对不同靶标的免疫球蛋白多肽,例如两种提供两种功能性免疫球蛋白构建体(例如第一免疫球蛋白构建体和第二免疫球蛋白构建体)。在又另一个替代方案中,两种或更多种不同的aav可以表达针对相同靶标的免疫球蛋白构建体(其中免疫球蛋白构建体之一已被修饰以消除fcrn结合)和保留其能力或具有增强的结合fcrn的能力的第二免疫球蛋白构建体。这种组合物可用于同时提供在cns中保留增加的抗体和用于全身递送免疫球蛋白构建体的抗体。
除了本文所述的一种或多种组合之外,本文所述的方案还包括与生物药物、小分子药物或其他疗法中的一种或多种的其他组合。如本文所述的生物药物基于肽、多肽、蛋白、酶、核酸分子、载体(包括病毒载体)等。
合适地,本文所述的组合物包含有效量的一种或多种悬浮于药学上合适的载体中和/或与合适的赋形剂混合的aav,其经设计用于经由注射、渗透泵、鞘内导管递送至主体或用于通过另一种装置或途径递送。在一个实例中,组合物被配制用于鞘内递送。在一个实施方案中,鞘内递送涵盖注射入椎管,例如蛛网膜下腔。然而,可以选择其他递送途径和用于aav组合物的药学上可接受的载体,包括例如颅内、鼻内、脑池内、脑脊液内递送以及在其他合适的直接或全身途径中,即ommaya储库。
可以配制组合物以含有约1x109个基因组拷贝(gc)至约5x1014个gc的范围内的量的aav。在一个实例中,载体为约3x1013个gc,但其他量诸如约1x109个gc、约5x109个gc、约1x1010个gc、约5x1010个gc、约1x1011个gc、约5x1011个gc、约1x1012个gc、约5x1012个gc或约5x1013个gc。此类组合物可以含有表达针对所选靶标的免疫球蛋白的单一aav储备物。在另一个实施方案中,此类组合物可以含有两种共表达免疫球蛋白的aav储备物,其在靶向的宿主细胞中组装以形成针对所选靶标的期望的免疫球蛋白(例如,全长抗体)。在另一个实施方案中,组合物可以含有两种或更多种aav储备物,其各自都表达不同的免疫球蛋白构建体。在此类组合物中,表达的蛋白可以组合以形成单一免疫球蛋白或可以表达两种或更多种具有不同靶标的免疫球蛋白。这些不同的靶标可以针对相同细胞类型或相同病毒(或其他靶标)上的不同配体,或针对两种完全不同的病原体。组合物被设计用于鞘内递送。在一个实施方案中,进行脊髓抽液,其中移取约15ml(或更少)至约25mlcsf,并且其中将载体悬浮于相容的载体中并递送至主体。
可以将raav(优选地悬浮于生理相容的载体中且任选地与一种或多种赋形剂混合)施用至人或非人哺乳动物患者。考虑到转移病毒针对的适应症,本领域技术人员可以容易地选择合适的载体。例如,一种合适的载体包括盐水,其可以用各种缓冲溶液(例如磷酸盐缓冲盐水)配制。其他示例性载体包括elliot’sb、无菌盐水、乳糖、蔗糖、麦芽糖和水。对载体的选择不是对本发明的限制。任选地,除了raav和载体之外,本发明的组合物可以包含其他常规药物成分,诸如防腐剂或化学稳定剂。
在一个实施方案中,本文所述的组合物用于制备用于治疗中枢神经系统病症和疾病的药物。
在另一个方面,提供了治疗阿尔茨海默氏病的方法,其涉及向有需要的主体鞘内递送如本文所述的aav载体组合物,其中至少一种aav载体储备物表达对aβ、β分泌酶和/或tau蛋白特异性的免疫球蛋白。
在又另一个实施方案中,提供了用于治疗帕金森氏病或相关突触核蛋白病的方法,其涉及鞘内递送如本文提供的aav载体组合物,其中至少一种aav载体储备物编码一种或多种富含亮氨酸的重复激酶2抗体、dardarin(lrrk2)抗体、α-突触核蛋白抗体和/或dj-1(park7)抗体。
在又一个进一步方面,本文提供了治疗多发性硬化的方法,其涉及鞘内递送如本文提供的aav载体组合物,在所述组合物中,至少一种载体储备物编码针对a4-整联蛋白、cd20、cd25、il12、p40+il23p40、lingo-1、cd40和rhigm22、cd52、il17、cd19和/或sema4d中的一种或多种的免疫球蛋白。
在又另一个方面,提供了治疗als的方法,其涉及鞘内递送如本文所述的aav载体组合物,其中至少一种aav载体储备物表达对als酶超氧化物歧化酶1(sod1)及其变体、derlin-1-结合区特异性的免疫球蛋白和/或针对神经突增生抑制剂的抗体构建体。在一个实施方案中,所述载体和/或组合物包含抗sod1免疫球蛋白fc片段。
在一个方面,提供了用于治疗朊病毒相关疾病的方法,其包括鞘内递送aav载体组合物,其中至少一种载体储备物编码针对一种或多种主要朊病毒蛋白或prpsc的免疫球蛋白。
用于评价针对本文所述的各种病症和疾病的治疗的有效性的动物模型是可用的。参见,例如,用于评价帕金森氏病的动物模型,描述于例如dvandam和p.p.dedeyn,brjpharmacol,2011oct;164(4):1285-1300。用于评价帕金森氏病药物的模型也已描述于例如httran等人,cellreports,vol.7,issue6,p2054-2065,june26,2014.,rmransajoff,natureneuroscience,aug2012,vol15,1074-1077(多发性硬化症);m.apouladi等人,natuerreviewsneuroscience,14,708-721(2013)(亨廷顿氏病);nfernandez-borges,等人.,curtopmedchem2013;13(19):2504-21(抗朊病毒药物);pmcgoldrick,等人,biochimicaetbiophysicaacta(bba)-molecularbasisofdisease,vol.1832,issue9,september2013,pp.1421-1436和jmmoser等人,molgenetgenomics,2013jun;288(5-6):207-29(als)。还有其他模型是本领域技术人员已知的。
在另一个方面,提供了用于治疗中枢神经系统的感染性疾病的方法,其涉及鞘内递送如本文提供的aav载体组合物,在所述组合物中,至少一种载体储备物编码针对引起所述感染性疾病的病原体的免疫球蛋白。实例非限制性地包括一种或多种针对结核分枝杆菌(结核病)、脑膜炎奈瑟氏菌(脑膜炎)、肺炎链球菌、单核细胞增生李斯特菌(李斯特菌病)、伯氏疏螺旋体(莱姆病)、人缺陷病毒(获得性免疫缺陷综合征)、疱疹家族病毒、水痘带状疱疹病毒、eb病毒(ebv)、巨细胞病毒和/或jc病毒中的一种或多种的免疫球蛋白。
任选地,如本文所述的aav组合物在不存在额外的外在药理学或化学药剂或血脑屏障的其他物理破坏的情况下施用。
在组合疗法中,在用另一种药剂开始疗法之前、期间或之后、以及其任何组合,即在开始疗法之前和期间、之前和之后、期间和之后、或者之前、期间和之后施用本文所述的aav递送的免疫球蛋白构建体。
本文所述的组合物可以用于涉及其他活性剂的共同施用的方案中。可以使用任何合适的方法或途径来施用此类其他药剂。施用路径包括例如全身性施用、口服施用、静脉内施用、腹膜内施用、皮下施用、或肌肉内施用。任选地,本文所述的aav组合物还可以通过这些途径之一来施用。
以下实施例仅是说明性的,并且不是对本文所述的发明的限制。
实施例
实施例1:抗siv免疫粘附素的aav9介导的递送的cns表达
为了评估在itaav递送之后cns中长期抗体产生的潜能,将编码免疫粘附素的载体施用于两种非人灵长类动物,并且通过elisa评估csf中转基因产物的浓度。如先前针对aav8所述构建表达恒河猴来源的免疫粘附素(201ia)的aav9载体,除了使用cb启动子(具有巨细胞病毒增强子的鸡β肌动蛋白)代替巨细胞病毒启动子[greigja,等人(2014)intramuscularinjectionofaav8inmiceandmacaquesisassociatedwithsubstantialhepatictargetingandtransgeneexpression.plosone9(11):e112268]和用aav9衣壳取代aav8。
简而言之,将用于恒河猴抗sivmac251gp120igg-201(glamann等人jvirol.1998;74(15):7158–7163.doi:10.1128/jvi.74.15.7158-7163.2000.更新)免疫粘附素(201ia)的密码子优化的核苷酸序列克隆至aav表达构建体中。所述构建体侧接aav2反向末端重复,且含有cb7启动子、嵌合内含子和兔球蛋白聚腺苷酸化序列(paav.cb7.ci.201ia.rbg)。证实该质粒的序列[seqidno:1(seqidno:2对应于编码的201ia序列)]。
通过枕骨下穿刺将raav9.cb7.ci.201ia.rbg载体(3x1012gc/kg)递送至两只食蟹猴(id0411和9860)的脑脊液(csf)中。在载体施用后定期收集血清和csf。如下通过elisa测定csf和血清中201ia的浓度。
除非另有说明,否则所有程序均在室温下进行。用biotek405ts微孔板洗涤器使用pbs+0.05%tween-20洗涤板。将在pbs中稀释至2μg/ml的mac251gp120(immunetechnologycorp.)在costar®96孔easywash™elisa测定板(corning)上在4℃下孵育过夜。然后将板用201iaelisa封闭缓冲液(pbs+5%热灭活的胎牛血清+1mmedta+0.07%tween-20)封闭。将稀释的样品添加至板并在板上稀释2倍至少四次。将板在37℃下孵育1小时,并再次在201iaelisa封闭缓冲液中封闭。然后将板与pbs中稀释的affinipure多克隆山羊抗人igg-生物素(jacksonimmunoresearchlabs)孵育,然后与pbs中稀释的链霉抗生物素蛋白-辣根过氧化物酶(abcam)孵育。使用3,3′,5,5′-四甲基联苯胺(tmb)底物来显现板。在用h2so4停止比色反应之后,使用spectramaxm3(moleculardevices)读板器在450nm处读板。
图1a和1b中举例说明两种动物各自的结果,其分别显示从施用至给药后第400天脑脊液和血清中的201ia的浓度。csf的浓度标度以ng/ml呈现,而血清中的表达水平以微克(μg)/ml测量。
实施例2-201ia的i253a突变以废除fcrn结合
从geneart(lifetechnologies)获得与201ia基因互补的长度为768bp、但含有对应于重链氨基酸序列的i253a或h453a(kabat编号)的突变的核苷酸序列。该序列侧接与paav.cb7.ci.201ia.rbg中的那些匹配的pst1和bstz17i限制性位点。如制造商所述,通过使用所示的酶(neb)限制性消化和连接(takarainc.)分别将突变的序列克隆至paav.cb7.ci.201ia.rbg中。sanger测序(genewiz)用于证实paav.cb7.ci.201ia.rbg[seqidno:1(seqidno:2对应于编码的201ia序列)]、paav.cb7.ci.201ia(i253a).rbg[seqidno:3(编码seqidno:4)]和paav.cb7.ci.201ia(h435a).rbg[seqidno:5(编码seqidno:6]在期望的突变的任一侧的互补性。
使用这些质粒使用实施例1中描述的方法来生成aav9载体。
实施例3-生成示例性aav.scfv构建体
制备三种说明性scfv构建体。设计这些构建体以消除fc区,同时仍结合全长抗淀粉样蛋白β(aβ)。设计这些构建体以降低淀粉样蛋白相关成像异常的风险和/或限制血管暴露于高单克隆抗体浓度。
a.aducanumabscfv
全长抗体已被描述为靶向纤维状aβ[weiner等人,naturereviews,immunology,6:404-416(may2006)]。为了生成scfv,基于who公开的序列[internationalnonproprietarynamesforpharmaceuticalsubstances(inn),whodruginformation,vol27,no.4,2013,p401-402;还参见wo2014/089500a1]鉴定重组、完全人抗aβigg1mab(aducanumab,biogen)的可变重结构域(seqidno:8的aa21-143)和可变轻结构域(seqidno:8的aa159-265)的氨基酸序列,将其密码子优化同时反向翻译成核酸序列。重和轻结构域与编码gly-ser接头(ggggsggggsggggs,seqidno:8的aa144-158)的核酸序列连接。将il-2分泌信号肽(seqidno:8的aa1-20)的编码序列与核酸序列的5'末端融合,并将6xhis标签的编码序列置于核酸的3'末端以促进纯化。我们设计的这些序列由商业合同设施订购并合成。除了其他质粒元件外,质粒还含有由以下组成的aav载体基因组:seqidno:13:5'aav2-itr、cmvie启动子、cb启动子、鸡β-肌动蛋白内含子、scfv序列、兔β-珠蛋白polya和aav2-3'itr。该质粒在包装293细胞系中与表达aav9衣壳的质粒共表达。在包装293细胞系中还共表达将含有scfv编码序列和侧接aavitrs的其他调节元件的aav基因组包装至aav9衣壳中所需的aavrep功能和腺病毒辅助功能。所得重组载体具有aav9衣壳和包装的aav载体基因组[seqidno:13],被称为aav9.cb7.ci.aducanumabscfv.rgb。
在本文描述的研究中,如通过数字微滴pcr(ddpcr)测定的载体产量是5.89x1013基因组拷贝(gc/ml)。使用用于测定aav载体基因组滴度的ddpcr技术通过m.lock等人,hugenetherapymethods,humgenethermethods.2014apr;25(2):115-25.doi:10.1089/hgtb.2013.131.epub2014feb14中的ddpcr来评价载体批次。
b.crenezumabscfv
crenezumab是由acimmune开发并授权给genentech的针对人1-40和1-42aβ(也称为β-淀粉样蛋白)的重组人源化单克隆抗体。crenezumab也被称为mabt5102a。其特征在于具有wo2015/120233a1的图2的hvr区域序列和其中的seqidno:2-9的序列。该抗体已被描述为能够靶向寡聚的、可溶性和纤维状aβ。
crenezumabscfv如上对于aducanumab所述制备,其取代crenezumab的可变重链[seqidno:10的aa21-131]和可变轻链[seqidno:10的aa147-258]的氨基酸序列,其基于who公开的序列[internationalnonproprietarynamesforpharmaceuticalsubstances(inn),whodruginformation,vol25,no.2,pp.163-164(2011);还参见wo2015/120233a]合成。所得重组载体具有aav9衣壳和包装的aav载体基因组[seqidno:14],被称为aav9.cb7.ci.crenezumabscfv.rgb。
在本文描述的研究中,如通过ddpcr测定的载体产量是3.79x1013gc/ml。将载体批次纯化并如m.lock等人,hugenetherapymethods,humgenethermethods.2014apr;25(2):115-25.doi:10.1089/hgtb.2013.131.epub2014feb14所述使用ddpcr进行评价。
c.solanezumabscfv
solanezumab是可得自elililly的重组人源化单克隆抗体。尽管其已被描述为与crenezumab高度同源,但其仅靶向可溶性aβ,而crenezumab靶向寡聚的、可溶性和纤维状aβ。solanezumab的特征在于具有us7,195,761的hvr区域序列。用于构建scfv的序列获得自internationalnonproprietarynamesforpharmaceuticalsubstances(inn),whodruginformation,vol.23,no.3,pp.263-264(2009)。
solanezumabscfv如上对于aducanumab所述制备,其取代solanezumab的可变重链[seqidno:12的aa21-131]和可变轻链[seqidno:12的aa147-258]的氨基酸序列,其基于who公开的序列[internationalnonproprietarynamesforpharmaceuticalsubstances(inn),whodruginformation,vol25,no.2,pp.163-164(2011);还参见wo2015/120233a]合成。所得重组载体具有aav9衣壳和包装的aav载体基因组[seqidno:15],被称为aav9.cb7.ci.solanezumabscfv.rgb。
在本文描述的研究中,ddpcr产量为5.25x1013gc/ml。将载体批次纯化并如所述通过ddpcr进行滴定。
d.检测抗aβscfv的elisa
上述部分a-c的scfv表达质粒用于产生elisa标准品,然后将质粒包装在aav9衣壳中。为了纯化scfvelisa标准品,将paav质粒转染至293细胞中。scfv分泌至细胞培养上清液中。利用在scfv末端的his-标签,通过使上清液在his-trap柱[gehealthcare]上运行来纯化scfv。剩余的上清液流过柱。然后使用高盐缓冲液洗脱结合的his-标签化的物质(scfv)。
使用以下酶联免疫吸附测定法(elisa)来检测抗aβscfv。将板在4℃下使用“混合种类”淀粉样蛋白(肽的聚集形式和单体形式)用1μg/ml重组人淀粉样蛋白β包被过夜。该测定如下进行。用pbs+0.05%tween-20洗涤5次。在室温下用pbs+1%牛血清白蛋白封闭1小时。添加血清/脑裂解物/纯化的抗aβscfv。用his标签构建本实施例的部分a、b和c中描述的三种scfv以促进elisa分析,并且使用his-trap柱[his-trapff1ml柱,gehealthcare]根据制造商的说明书[http://www.gelifesciences.com/-webapp/wcs/stores/servlet/catalog/en/gelifesciencesus/products/alternativeproductstructure_17555/17524701]从293上清液中纯化它们。在37℃下孵育1小时。用pbs+0.05%tween-20洗涤5次。添加在pbs中1:10,000稀释的抗his标签抗体(abcamab1187)。在室温下孵育1小时。用pbs+0.05%tween-20洗涤5次。在室温下用tmb底物(3,3’,5,5’-四甲基联苯胺)显色30分钟。添加2nh2so4以终止反应。在读板器上在450nm和540nm处读取。
e.3xtg小鼠模型中抗aβscfv的初步研究
将aav9.cb7.aducanumabscfv.rbg、aav9.cb7.crenezumabscfv.rbg、aav9.cb7.solanezumabscfv.rbg或pbs以1x1011gc/小鼠脑室内(icv)施用于6周龄3xtg小鼠(mmrrcrepository/jackson)[参见例如r.sternicdzuk,等人,“characterizationofthe3xtg-admousemodelofalzheimer'sdisease:part2.behavioralandcognitivechanges”.brainres.2010aug12;1348:149-55.doi:10.1016/j.brainres.2010.06.011.epub2010jun15和r.sterniczuk,等人,“characterizationofthe3xtg-admousemodelofalzheimer'sdisease:part1.circadianchanges.”,brainres.2010aug12;1348:139-48.doi:10.1016/j.brainres.2010.05.013.epub2010may31.],每个给药组6只小鼠。对照小鼠(不发展阿尔茨海默氏病):b6129sf2/j(jackson)。每月取血清以监测scfv表达。小鼠在载体施用后6个月时经历morris水迷宫和/或y-迷宫交替行为测试[参见例如webster等人usingmicetomodelalzheimer'sdementia:anoverviewoftheclinicaldiseaseandthepreclinicalbehavioralchangesin10mousemodels.frontgenet.2014;5:88.]。设计morris水迷宫以测量空间记忆、运动控制和认知绘图[whishaw,i.q.(1995)."acomparisonofratsandmiceinaswimmingpoolplacetaskandmatchingtoplacetask:somesurprisingdifferences".physiology&behavior.58(4):687–693.doi:10.1016/0031-9384(95)00110-5;crusio,wim(1999)."methodologicalconsiderationsfortestinglearninginmice".incrusio,w.e.;gerlai,r.t.handbookofmolecular-genetictechniquesforbrainandbehaviorresearch(第1版).amsterdam:elsevier.pp.638–651.]。y迷宫用于评价大鼠和小鼠中的空间工作记忆,特别是自发交替任务。该迷宫可以是商业购买的,例如来自panlab。在12个月龄时使小鼠安乐死用于aβ病理学的组织学评估,并且通过elisa从脑裂解物定量scfv/淀粉样蛋白。其他初步研究检查scfv在年龄较大的小鼠中的效力(在~4-6个月(其为该模型中的可见aβ病理发病的平均年龄)时给药)。将小鼠组在6周龄和4-6个月龄时给药,用于tau蛋白病理学的组织学评估,随后使小鼠在15个月龄时安乐死。
本申请含有序列和序列表,其在此通过引用并入。本申请中引用的所有出版物、专利和专利申请(包括2015年10月28日提交的优先权美国专利申请62/247498)都在此以其整体通过引用并入,如同每个单独的公开或专利申请被明确且单独地提到通过引用并入。尽管已经出于清楚理解的目的通过实例说明和实例的方式详细描述了前述发明,但鉴于本发明的教导,对于本领域普通技术人员显而易见的是,在不脱离所附权利要求的精神或范围的情况下,可以对其进行某些变化和改变。
表格(序列表自由文本)
对于含有数字标识符<223>下的自由文本的序列提供以下信息。
序列表
<110>thetrusteesoftheuniversityofpennsylvania
<120>鞘内施用腺伴随病毒载体用于基因治疗
<130>16-7656pct
<150>62/247498
<151>2015-10-28
<160>15
<170>patentinversion3.5
<210>1
<211>6694
<212>dna
<213>人工序列
<220>
<223>paav.cb7.ci.201ia.rbg
<220>
<221>polya_信号
<222>(60)..(186)
<220>
<221>misc_feature
<222>(275)..(404)
<223>3'itr(互补序列)
<220>
<221>复制起点
<222>(581)..(1036)
<220>
<221>misc_feature
<222>(3226)..(3355)
<223>5'itr
<220>
<221>misc
<222>(3423)..(3804)
<223>cmvie启动子
<220>
<221>启动子
<222>(3807)..(4088)
<220>
<221>tata_信号
<222>(4061)..(4064)
<220>
<221>内含子
<222>(4183)..(5155)
<220>
<221>cds
<222>(5161)..(6690)
<223>201ia
<400>1
gtacctctagagtcgacccgggcggcctcgaggacggggtgaactacgcctgaggatccg
60
atctttttccctctgccaaaaattatggggacatcatgaagccccttgagcatctgactt
120
ctggctaataaaggaaatttattttcattgcaatagtgtgttggaattttttgtgtctct
180
cactcggaagcaattcgttgatctgaatttcgaccacccataatacccattaccctggta
240
gataagtagcatggcgggttaatcattaactacaaggaacccctagtgatggagttggcc
300
actccctctctgcgcgctcgctcgctcactgaggccgggcgaccaaaggtcgcccgacgc
360
ccgggctttgcccgggcggcctcagtgagcgagcgagcgcgcagccttaattaacctaat
420
tcactggccgtcgttttacaacgtcgtgactgggaaaaccctggcgttacccaacttaat
480
cgccttgcagcacatccccctttcgccagctggcgtaatagcgaagaggcccgcaccgat
540
cgcccttcccaacagttgcgcagcctgaatggcgaatgggacgcgccctgtagcggcgca
600
ttaagcgcggcgggtgtggtggttacgcgcagcgtgaccgctacacttgccagcgcccta
660
gcgcccgctcctttcgctttcttcccttcctttctcgccacgttcgccggctttccccgt
720
caagctctaaatcgggggctccctttagggttccgatttagtgctttacggcacctcgac
780
cccaaaaaacttgattagggtgatggttcacgtagtgggccatcgccctgatagacggtt
840
tttcgccctttgacgttggagtccacgttctttaatagtggactcttgttccaaactgga
900
acaacactcaaccctatctcggtctattcttttgatttataagggattttgccgatttcg
960
gcctattggttaaaaaatgagctgatttaacaaaaatttaacgcgaattttaacaaaata
1020
ttaacgcttacaatttaggtggcacttttcggggaaatgtgcgcggaacccctatttgtt
1080
tatttttctaaatacattcaaatatgtatccgctcatgagacaataaccctgataaatgc
1140
ttcaataatattgaaaaaggaagagtatgagtattcaacatttccgtgtcgcccttattc
1200
ccttttttgcggcattttgccttcctgtttttgctcacccagaaacgctggtgaaagtaa
1260
aagatgctgaagatcagttgggtgcacgagtgggttacatcgaactggatctcaacagcg
1320
gtaagatccttgagagttttcgccccgaagaacgttttccaatgatgagcacttttaaag
1380
ttctgctatgtggcgcggtattatcccgtattgacgccgggcaagagcaactcggtcgcc
1440
gcatacactattctcagaatgacttggttgagtactcaccagtcacagaaaagcatctta
1500
cggatggcatgacagtaagagaattatgcagtgctgccataaccatgagtgataacactg
1560
cggccaacttacttctgacaacgatcggaggaccgaaggagctaaccgcttttttgcaca
1620
acatgggggatcatgtaactcgccttgatcgttgggaaccggagctgaatgaagccatac
1680
caaacgacgagcgtgacaccacgatgcctgtagcaatggcaacaacgttgcgcaaactat
1740
taactggcgaactacttactctagcttcccggcaacaattaatagactggatggaggcgg
1800
ataaagttgcaggaccacttctgcgctcggcccttccggctggctggtttattgctgata
1860
aatctggagccggtgagcgtgggtctcgcggtatcattgcagcactggggccagatggta
1920
agccctcccgtatcgtagttatctacacgacggggagtcaggcaactatggatgaacgaa
1980
atagacagatcgctgagataggtgcctcactgattaagcattggtaactgtcagaccaag
2040
tttactcatatatactttagattgatttaaaacttcatttttaatttaaaaggatctagg
2100
tgaagatcctttttgataatctcatgaccaaaatcccttaacgtgagttttcgttccact
2160
gagcgtcagaccccgtagaaaagatcaaaggatcttcttgagatcctttttttctgcgcg
2220
taatctgctgcttgcaaacaaaaaaaccaccgctaccagcggtggtttgtttgccggatc
2280
aagagctaccaactctttttccgaaggtaactggcttcagcagagcgcagataccaaata
2340
ctgttcttctagtgtagccgtagttaggccaccacttcaagaactctgtagcaccgccta
2400
catacctcgctctgctaatcctgttaccagtggctgctgccagtggcgataagtcgtgtc
2460
ttaccgggttggactcaagacgatagttaccggataaggcgcagcggtcgggctgaacgg
2520
ggggttcgtgcacacagcccagcttggagcgaacgacctacaccgaactgagatacctac
2580
agcgtgagctatgagaaagcgccacgcttcccgaagggagaaaggcggacaggtatccgg
2640
taagcggcagggtcggaacaggagagcgcacgagggagcttccagggggaaacgcctggt
2700
atctttatagtcctgtcgggtttcgccacctctgacttgagcgtcgatttttgtgatgct
2760
cgtcaggggggcggagcctatggaaaaacgccagcaacgcggcctttttacggttcctgg
2820
ccttttgctggccttttgctcacatgttctttcctgcgttatcccctgattctgtggata
2880
accgtattaccgcctttgagtgagctgataccgctcgccgcagccgaacgaccgagcgca
2940
gcgagtcagtgagcgaggaagcggaagagcgcccaatacgcaaaccgcctctccccgcgc
3000
gttggccgattcattaatgcagctggcacgacaggtttcccgactggaaagcgggcagtg
3060
agcgcaacgcaattaatgtgagttagctcactcattaggcaccccaggctttacacttta
3120
tgcttccggctcgtatgttgtgtggaattgtgagcggataacaatttcacacaggaaaca
3180
gctatgaccatgattacgccagatttaattaaggccttaattaggctgcgcgctcgctcg
3240
ctcactgaggccgcccgggcaaagcccgggcgtcgggcgacctttggtcgcccggcctca
3300
gtgagcgagcgagcgcgcagagagggagtggccaactccatcactaggggttccttgtag
3360
ttaatgattaacccgccatgctacttatctaccagggtaatggggatcctctagaactat
3420
agctagtcgacattgattattgactagttattaatagtaatcaattacggggtcattagt
3480
tcatagcccatatatggagttccgcgttacataacttacggtaaatggcccgcctggctg
3540
accgcccaacgacccccgcccattgacgtcaataatgacgtatgttcccatagtaacgcc
3600
aatagggactttccattgacgtcaatgggtggactatttacggtaaactgcccacttggc
3660
agtacatcaagtgtatcatatgccaagtacgccccctattgacgtcaatgacggtaaatg
3720
gcccgcctggcattatgcccagtacatgaccttatgggactttcctacttggcagtacat
3780
ctacgtattagtcatcgctattaccatggtcgaggtgagccccacgttctgcttcactct
3840
ccccatctcccccccctccccacccccaattttgtatttatttattttttaattattttg
3900
tgcagcgatgggggcggggggggggggggggcgcgcgccaggcggggcggggcggggcga
3960
ggggcggggcggggcgaggcggagaggtgcggcggcagccaatcagagcggcgcgctccg
4020
aaagtttccttttatggcgaggcggcggcggcggcggccctataaaaagcgaagcgcgcg
4080
gcgggcggggagtcgctgcgacgctgccttcgccccgtgccccgctccgccgccgcctcg
4140
cgccgcccgccccggctctgactgaccgcgttactcccacaggtgagcgggcgggacggc
4200
ccttctcctccgggctgtaattagcgcttggtttaatgacggcttgtttcttttctgtgg
4260
ctgcgtgaaagccttgaggggctccgggagggccctttgtgcggggggagcggctcgggg
4320
ggtgcgtgcgtgtgtgtgtgcgtggggagcgccgcgtgcggctccgcgctgcccggcggc
4380
tgtgagcgctgcgggcgcggcgcggggctttgtgcgctccgcagtgtgcgcgaggggagc
4440
gcggccgggggcggtgccccgcggtgcggggggggctgcgaggggaacaaaggctgcgtg
4500
cggggtgtgtgcgtgggggggtgagcagggggtgtgggcgcgtcggtcgggctgcaaccc
4560
cccctgcacccccctccccgagttgctgagcacggcccggcttcgggtgcggggctccgt
4620
acggggcgtggcgcggggctcgccgtgccgggcggggggtggcggcaggtgggggtgccg
4680
ggcggggcggggccgcctcgggccggggagggctcgggggaggggcgcggcggcccccgg
4740
agcgccggcggctgtcgaggcgcggcgagccgcagccattgccttttatggtaatcgtgc
4800
gagagggcgcagggacttcctttgtcccaaatctgtgcggagccgaaatctgggaggcgc
4860
cgccgcaccccctctagcgggcgcggggcgaagcggtgcggcgccggcaggaaggaaatg
4920
ggcggggagggccttcgtgcgtcgccgcgccgccgtccccttctccctctccagcctcgg
4980
ggctgtccgcggggggacggctgccttcgggggggacggggcagggcggggttcggcttc
5040
tggcgtgtgaccggcggctctagagcctctgctaaccatgttcatgccttcttctttttc
5100
ctacagctcctgggcaacgtgctggttattgtgctgtctcatcattttggcaaagaattc
5160
atggagttcgggctgagctgggtctttctggtggccctgctgaaggga
5208
metglupheglyleusertrpvalpheleuvalalaleuleulysgly
151015
gtccagtgcgaggtgcagctgctggaatccggacctggcctggtgaaa
5256
valglncysgluvalglnleuleugluserglyproglyleuvallys
202530
ccatctgagacactgagtctgacttgtgctgtctccggcctgtctatc
5304
prosergluthrleuserleuthrcysalavalserglyleuserile
354045
agctccgatttctcctgggcatggattaggcagacccccggcaaggcc
5352
serseraspphesertrpalatrpileargglnthrproglylysala
505560
ctggaatatgtggggtacatccgcgggaacaccggagatacatactat
5400
leuglutyrvalglytyrileargglyasnthrglyaspthrtyrtyr
65707580
aatcctagtctgaagtcaaggctgactatctcaaaggacaccagcaaa
5448
asnproserleulysserargleuthrileserlysaspthrserlys
859095
aaccaaatctacctgaatctgtctagtgtcaccgctggcgatgccgcc
5496
asnglniletyrleuasnleuserservalthralaglyaspalaala
100105110
gtgtactattgcgcaagggaccgggtgtgcgacgatgactacggatac
5544
valtyrtyrcysalaargaspargvalcysaspaspasptyrglytyr
115120125
tattacaccgaggtgtgcttcggcctggattcttgggggcagggaatc
5592
tyrtyrthrgluvalcyspheglyleuaspsertrpglyglnglyile
130135140
gtggtcacagtgtcaagcggcggaggaggcagcggaggaggagggtcc
5640
valvalthrvalserserglyglyglyglyserglyglyglyglyser
145150155160
ggaggcgggggatctgcagaactggtcatgacacagtccccactgagc
5688
glyglyglyglyseralagluleuvalmetthrglnserproleuser
165170175
ctgtccgtcgctccaggacagactgcatctattagttgtcgatcctct
5736
leuservalalaproglyglnthralaserilesercysargserser
180185190
cagtccctggactatgctaacggcaatacctacctgtcttggtttcac
5784
glnserleuasptyralaasnglyasnthrtyrleusertrpphehis
195200205
cagcgaccaggacagccacctcggagactgatctatcagatttccaac
5832
glnargproglyglnproproargargleuiletyrglnileserasn
210215220
agagattctggagtgcccgacaggttctcaggcagcggagcaggaact
5880
argaspserglyvalproaspargpheserglyserglyalaglythr
225230235240
gagtttaccctgcgaatcagtcggatggaatcagatgacgtggggatc
5928
gluphethrleuargileserargmetgluseraspaspvalglyile
245250255
tactactgcggacaggggaccacattcccacggacatttggacagggc
5976
tyrtyrcysglyglnglythrthrpheproargthrpheglyglngly
260265270
actaaggtggagatcaaaacctgtggaggaggaagcaagccaccaacc
6024
thrlysvalgluilelysthrcysglyglyglyserlysproprothr
275280285
tgccctccatgtacatctcccgaactgctgggcgggcctagcgtgttc
6072
cysproprocysthrserprogluleuleuglyglyproservalphe
290295300
ctgtttccccctaagcctaaagatacactgatgattagtagaacccca
6120
leupheproprolysprolysaspthrleumetileserargthrpro
305310315320
gaggtcacatgcgtggtcgtggacgtgtcccaggaagatcctgacgtg
6168
gluvalthrcysvalvalvalaspvalserglngluaspproaspval
325330335
aagttcaactggtacgtgaatggcgccgaggtgcaccatgctcagact
6216
lyspheasntrptyrvalasnglyalagluvalhishisalaglnthr
340345350
aaaccacgcgaaacccagtataatagtacataccgagtcgtgtcagtc
6264
lysproarggluthrglntyrasnserthrtyrargvalvalserval
355360365
ctgacagtgactcaccaggattggctgaacggcaaggagtatacctgc
6312
leuthrvalthrhisglnasptrpleuasnglylysglutyrthrcys
370375380
aaggtgtctaacaaggccctgcccgcccctatccagaaaacaattagc
6360
lysvalserasnlysalaleuproalaproileglnlysthrileser
385390395400
aaggacaaagggcagccacgggaaccccaggtgtacactctgccaccc
6408
lysasplysglyglnproarggluproglnvaltyrthrleupropro
405410415
tcaagagaggaactgactaagaaccaggtcagcctgacctgtctggtg
6456
serargglugluleuthrlysasnglnvalserleuthrcysleuval
420425430
aaaggcttctaccccagcgatatcgtcgtggagtgggaaagttcaggc
6504
lysglyphetyrproseraspilevalvalglutrpglusersergly
435440445
cagcctgagaatacttacaagactacccctccagtgctggatagcgac
6552
glnprogluasnthrtyrlysthrthrproprovalleuaspserasp
450455460
gggtcctatttcctgtacagcaagctgacagtggacaaatcccgctgg
6600
glysertyrpheleutyrserlysleuthrvalasplysserargtrp
465470475480
cagcagggaaacgtcttttcctgttctgtgatgcatgaggccctgcac
6648
glnglnglyasnvalphesercysservalmethisglualaleuhis
485490495
aatcattacacccagaagagtctgtcactgagccccggcaaatgag
6694
asnhistyrthrglnlysserleuserleuserproglylys
500505510
<210>2
<211>510
<212>prt
<213>人工序列
<220>
<223>合成构建体
<400>2
metglupheglyleusertrpvalpheleuvalalaleuleulysgly
151015
valglncysgluvalglnleuleugluserglyproglyleuvallys
202530
prosergluthrleuserleuthrcysalavalserglyleuserile
354045
serseraspphesertrpalatrpileargglnthrproglylysala
505560
leuglutyrvalglytyrileargglyasnthrglyaspthrtyrtyr
65707580
asnproserleulysserargleuthrileserlysaspthrserlys
859095
asnglniletyrleuasnleuserservalthralaglyaspalaala
100105110
valtyrtyrcysalaargaspargvalcysaspaspasptyrglytyr
115120125
tyrtyrthrgluvalcyspheglyleuaspsertrpglyglnglyile
130135140
valvalthrvalserserglyglyglyglyserglyglyglyglyser
145150155160
glyglyglyglyseralagluleuvalmetthrglnserproleuser
165170175
leuservalalaproglyglnthralaserilesercysargserser
180185190
glnserleuasptyralaasnglyasnthrtyrleusertrpphehis
195200205
glnargproglyglnproproargargleuiletyrglnileserasn
210215220
argaspserglyvalproaspargpheserglyserglyalaglythr
225230235240
gluphethrleuargileserargmetgluseraspaspvalglyile
245250255
tyrtyrcysglyglnglythrthrpheproargthrpheglyglngly
260265270
thrlysvalgluilelysthrcysglyglyglyserlysproprothr
275280285
cysproprocysthrserprogluleuleuglyglyproservalphe
290295300
leupheproprolysprolysaspthrleumetileserargthrpro
305310315320
gluvalthrcysvalvalvalaspvalserglngluaspproaspval
325330335
lyspheasntrptyrvalasnglyalagluvalhishisalaglnthr
340345350
lysproarggluthrglntyrasnserthrtyrargvalvalserval
355360365
leuthrvalthrhisglnasptrpleuasnglylysglutyrthrcys
370375380
lysvalserasnlysalaleuproalaproileglnlysthrileser
385390395400
lysasplysglyglnproarggluproglnvaltyrthrleupropro
405410415
serargglugluleuthrlysasnglnvalserleuthrcysleuval
420425430
lysglyphetyrproseraspilevalvalglutrpglusersergly
435440445
glnprogluasnthrtyrlysthrthrproprovalleuaspserasp
450455460
glysertyrpheleutyrserlysleuthrvalasplysserargtrp
465470475480
glnglnglyasnvalphesercysservalmethisglualaleuhis
485490495
asnhistyrthrglnlysserleuserleuserproglylys
500505510
<210>3
<211>6694
<212>dna
<213>人工序列
<220>
<223>编码201ia(i253a)突变体的质粒
<220>
<221>misc
<222>(1)..(130)
<223>5'itr
<220>
<221>misc_feature
<222>(198)..(579)
<223>cmvie启动子
<220>
<221>启动子
<222>(582)..(863)
<223>cb启动子
<220>
<221>tata_信号
<222>(836)..(839)
<220>
<221>内含子
<222>(958)..(1930)
<223>鸡β-肌动蛋白内含子
<220>
<221>cds
<222>(1936)..(3465)
<223>cmvie启动子
<220>
<221>polya_信号
<222>(3529)..(3655)
<223>兔球蛋白polya
<220>
<221>misc_feature
<222>(3744)..(3873)
<223>3'itr(互补序列)
<220>
<221>复制起点
<222>(4050)..(4505)
<220>
<221>misc_feature
<222>(4636)..(5493)
<223>ap(r)marker
<220>
<221>复制起点
<222>(5667)..(6255)
<400>3
ctgcgcgctcgctcgctcactgaggccgcccgggcaaagcccgggcgtcgggcgaccttt
60
ggtcgcccggcctcagtgagcgagcgagcgcgcagagagggagtggccaactccatcact
120
aggggttccttgtagttaatgattaacccgccatgctacttatctaccagggtaatgggg
180
atcctctagaactatagctagtcgacattgattattgactagttattaatagtaatcaat
240
tacggggtcattagttcatagcccatatatggagttccgcgttacataacttacggtaaa
300
tggcccgcctggctgaccgcccaacgacccccgcccattgacgtcaataatgacgtatgt
360
tcccatagtaacgccaatagggactttccattgacgtcaatgggtggactatttacggta
420
aactgcccacttggcagtacatcaagtgtatcatatgccaagtacgccccctattgacgt
480
caatgacggtaaatggcccgcctggcattatgcccagtacatgaccttatgggactttcc
540
tacttggcagtacatctacgtattagtcatcgctattaccatggtcgaggtgagccccac
600
gttctgcttcactctccccatctcccccccctccccacccccaattttgtatttatttat
660
tttttaattattttgtgcagcgatgggggcggggggggggggggggcgcgcgccaggcgg
720
ggcggggcggggcgaggggcggggcggggcgaggcggagaggtgcggcggcagccaatca
780
gagcggcgcgctccgaaagtttccttttatggcgaggcggcggcggcggcggccctataa
840
aaagcgaagcgcgcggcgggcggggagtcgctgcgacgctgccttcgccccgtgccccgc
900
tccgccgccgcctcgcgccgcccgccccggctctgactgaccgcgttactcccacaggtg
960
agcgggcgggacggcccttctcctccgggctgtaattagcgcttggtttaatgacggctt
1020
gtttcttttctgtggctgcgtgaaagccttgaggggctccgggagggccctttgtgcggg
1080
gggagcggctcggggggtgcgtgcgtgtgtgtgtgcgtggggagcgccgcgtgcggctcc
1140
gcgctgcccggcggctgtgagcgctgcgggcgcggcgcggggctttgtgcgctccgcagt
1200
gtgcgcgaggggagcgcggccgggggcggtgccccgcggtgcggggggggctgcgagggg
1260
aacaaaggctgcgtgcggggtgtgtgcgtgggggggtgagcagggggtgtgggcgcgtcg
1320
gtcgggctgcaaccccccctgcacccccctccccgagttgctgagcacggcccggcttcg
1380
ggtgcggggctccgtacggggcgtggcgcggggctcgccgtgccgggcggggggtggcgg
1440
caggtgggggtgccgggcggggcggggccgcctcgggccggggagggctcgggggagggg
1500
cgcggcggcccccggagcgccggcggctgtcgaggcgcggcgagccgcagccattgcctt
1560
ttatggtaatcgtgcgagagggcgcagggacttcctttgtcccaaatctgtgcggagccg
1620
aaatctgggaggcgccgccgcaccccctctagcgggcgcggggcgaagcggtgcggcgcc
1680
ggcaggaaggaaatgggcggggagggccttcgtgcgtcgccgcgccgccgtccccttctc
1740
cctctccagcctcggggctgtccgcggggggacggctgccttcgggggggacggggcagg
1800
gcggggttcggcttctggcgtgtgaccggcggctctagagcctctgctaaccatgttcat
1860
gccttcttctttttcctacagctcctgggcaacgtgctggttattgtgctgtctcatcat
1920
tttggcaaagaattcatggagttcgggctgagctgggtctttctggtggcc
1971
metglupheglyleusertrpvalpheleuvalala
1510
ctgctgaagggagtccagtgcgaggtgcagctgctggaatccggacct
2019
leuleulysglyvalglncysgluvalglnleuleugluserglypro
152025
ggcctggtgaaaccatctgagacactgagtctgacttgtgctgtctcc
2067
glyleuvallysprosergluthrleuserleuthrcysalavalser
303540
ggcctgtctatcagctccgatttctcctgggcatggattaggcagacc
2115
glyleuserileserseraspphesertrpalatrpileargglnthr
45505560
cccggcaaggccctggaatatgtggggtacatccgcgggaacaccgga
2163
proglylysalaleuglutyrvalglytyrileargglyasnthrgly
657075
gatacatactataatcctagtctgaagtcaaggctgactatctcaaag
2211
aspthrtyrtyrasnproserleulysserargleuthrileserlys
808590
gacaccagcaaaaaccaaatctacctgaatctgtctagtgtcaccgct
2259
aspthrserlysasnglniletyrleuasnleuserservalthrala
95100105
ggcgatgccgccgtgtactattgcgcaagggaccgggtgtgcgacgat
2307
glyaspalaalavaltyrtyrcysalaargaspargvalcysaspasp
110115120
gactacggatactattacaccgaggtgtgcttcggcctggattcttgg
2355
asptyrglytyrtyrtyrthrgluvalcyspheglyleuaspsertrp
125130135140
gggcagggaatcgtggtcacagtgtcaagcggcggaggaggcagcgga
2403
glyglnglyilevalvalthrvalserserglyglyglyglysergly
145150155
ggaggagggtccggaggcgggggatctgcagaactggtcatgacacag
2451
glyglyglyserglyglyglyglyseralagluleuvalmetthrgln
160165170
tccccactgagcctgtccgtcgctccaggacagactgcatctattagt
2499
serproleuserleuservalalaproglyglnthralaserileser
175180185
tgtcgatcctctcagtccctggactatgctaacggcaatacctacctg
2547
cysargserserglnserleuasptyralaasnglyasnthrtyrleu
190195200
tcttggtttcaccagcgaccaggacagccacctcggagactgatctat
2595
sertrpphehisglnargproglyglnproproargargleuiletyr
205210215220
cagatttccaacagagattctggagtgcccgacaggttctcaggcagc
2643
glnileserasnargaspserglyvalproaspargpheserglyser
225230235
ggagcaggaactgagtttaccctgcgaatcagtcggatggaatcagat
2691
glyalaglythrgluphethrleuargileserargmetgluserasp
240245250
gacgtggggatctactactgcggacaggggaccacattcccacggaca
2739
aspvalglyiletyrtyrcysglyglnglythrthrpheproargthr
255260265
tttggacagggcactaaggtggagatcaaaacctgtggaggaggaagc
2787
pheglyglnglythrlysvalgluilelysthrcysglyglyglyser
270275280
aagccaccaacctgccctccatgtacatctcccgaactgctgggcggg
2835
lysproprothrcysproprocysthrserprogluleuleuglygly
285290295300
cctagcgtgttcctgtttccccctaagcctaaagatacactgatggcc
2883
proservalpheleupheproprolysprolysaspthrleumetala
305310315
agtagaaccccagaggtcacatgcgtggtcgtggacgtgtcccaggaa
2931
serargthrprogluvalthrcysvalvalvalaspvalserglnglu
320325330
gatcctgacgtgaagttcaactggtacgtgaatggcgccgaggtgcac
2979
aspproaspvallyspheasntrptyrvalasnglyalagluvalhis
335340345
catgctcagactaaaccacgcgaaacccagtataatagtacataccga
3027
hisalaglnthrlysproarggluthrglntyrasnserthrtyrarg
350355360
gtcgtgtcagtcctgacagtgactcaccaggattggctgaacggcaag
3075
valvalservalleuthrvalthrhisglnasptrpleuasnglylys
365370375380
gagtatacctgcaaggtgtctaacaaggccctgcccgcccctatccag
3123
glutyrthrcyslysvalserasnlysalaleuproalaproilegln
385390395
aaaacaattagcaaggacaaagggcagccacgggaaccccaggtgtac
3171
lysthrileserlysasplysglyglnproarggluproglnvaltyr
400405410
actctgccaccctcaagagaggaactgactaagaaccaggtcagcctg
3219
thrleuproproserargglugluleuthrlysasnglnvalserleu
415420425
acctgtctggtgaaaggcttctaccccagcgatatcgtcgtggagtgg
3267
thrcysleuvallysglyphetyrproseraspilevalvalglutrp
430435440
gaaagttcaggccagcctgagaatacttacaagactacccctccagtg
3315
gluserserglyglnprogluasnthrtyrlysthrthrproproval
445450455460
ctggatagcgacgggtcctatttcctgtacagcaagctgacagtggac
3363
leuaspseraspglysertyrpheleutyrserlysleuthrvalasp
465470475
aaatcccgctggcagcagggaaacgtcttttcctgttctgtgatgcat
3411
lysserargtrpglnglnglyasnvalphesercysservalmethis
480485490
gaggccctgcacaatcattacacccagaagagtctgtcactgagcccc
3459
glualaleuhisasnhistyrthrglnlysserleuserleuserpro
495500505
ggcaaatgaggtacctctagagtcgacccgggcggcctcgaggacggggtgaacta
3515
glylys
510
cgcctgaggatccgatctttttccctctgccaaaaattatggggacatcatgaagcccct
3575
tgagcatctgacttctggctaataaaggaaatttattttcattgcaatagtgtgttggaa
3635
ttttttgtgtctctcactcggaagcaattcgttgatctgaatttcgaccacccataatac
3695
ccattaccctggtagataagtagcatggcgggttaatcattaactacaaggaacccctag
3755
tgatggagttggccactccctctctgcgcgctcgctcgctcactgaggccgggcgaccaa
3815
aggtcgcccgacgcccgggctttgcccgggcggcctcagtgagcgagcgagcgcgcagcc
3875
ttaattaacctaattcactggccgtcgttttacaacgtcgtgactgggaaaaccctggcg
3935
ttacccaacttaatcgccttgcagcacatccccctttcgccagctggcgtaatagcgaag
3995
aggcccgcaccgatcgcccttcccaacagttgcgcagcctgaatggcgaatgggacgcgc
4055
cctgtagcggcgcattaagcgcggcgggtgtggtggttacgcgcagcgtgaccgctacac
4115
ttgccagcgccctagcgcccgctcctttcgctttcttcccttcctttctcgccacgttcg
4175
ccggctttccccgtcaagctctaaatcgggggctccctttagggttccgatttagtgctt
4235
tacggcacctcgaccccaaaaaacttgattagggtgatggttcacgtagtgggccatcgc
4295
cctgatagacggtttttcgccctttgacgttggagtccacgttctttaatagtggactct
4355
tgttccaaactggaacaacactcaaccctatctcggtctattcttttgatttataaggga
4415
ttttgccgatttcggcctattggttaaaaaatgagctgatttaacaaaaatttaacgcga
4475
attttaacaaaatattaacgcttacaatttaggtggcacttttcggggaaatgtgcgcgg
4535
aacccctatttgtttatttttctaaatacattcaaatatgtatccgctcatgagacaata
4595
accctgataaatgcttcaataatattgaaaaaggaagagtatgagtattcaacatttccg
4655
tgtcgcccttattcccttttttgcggcattttgccttcctgtttttgctcacccagaaac
4715
gctggtgaaagtaaaagatgctgaagatcagttgggtgcacgagtgggttacatcgaact
4775
ggatctcaacagcggtaagatccttgagagttttcgccccgaagaacgttttccaatgat
4835
gagcacttttaaagttctgctatgtggcgcggtattatcccgtattgacgccgggcaaga
4895
gcaactcggtcgccgcatacactattctcagaatgacttggttgagtactcaccagtcac
4955
agaaaagcatcttacggatggcatgacagtaagagaattatgcagtgctgccataaccat
5015
gagtgataacactgcggccaacttacttctgacaacgatcggaggaccgaaggagctaac
5075
cgcttttttgcacaacatgggggatcatgtaactcgccttgatcgttgggaaccggagct
5135
gaatgaagccataccaaacgacgagcgtgacaccacgatgcctgtagcaatggcaacaac
5195
gttgcgcaaactattaactggcgaactacttactctagcttcccggcaacaattaataga
5255
ctggatggaggcggataaagttgcaggaccacttctgcgctcggcccttccggctggctg
5315
gtttattgctgataaatctggagccggtgagcgtgggtctcgcggtatcattgcagcact
5375
ggggccagatggtaagccctcccgtatcgtagttatctacacgacggggagtcaggcaac
5435
tatggatgaacgaaatagacagatcgctgagataggtgcctcactgattaagcattggta
5495
actgtcagaccaagtttactcatatatactttagattgatttaaaacttcatttttaatt
5555
taaaaggatctaggtgaagatcctttttgataatctcatgaccaaaatcccttaacgtga
5615
gttttcgttccactgagcgtcagaccccgtagaaaagatcaaaggatcttcttgagatcc
5675
tttttttctgcgcgtaatctgctgcttgcaaacaaaaaaaccaccgctaccagcggtggt
5735
ttgtttgccggatcaagagctaccaactctttttccgaaggtaactggcttcagcagagc
5795
gcagataccaaatactgttcttctagtgtagccgtagttaggccaccacttcaagaactc
5855
tgtagcaccgcctacatacctcgctctgctaatcctgttaccagtggctgctgccagtgg
5915
cgataagtcgtgtcttaccgggttggactcaagacgatagttaccggataaggcgcagcg
5975
gtcgggctgaacggggggttcgtgcacacagcccagcttggagcgaacgacctacaccga
6035
actgagatacctacagcgtgagctatgagaaagcgccacgcttcccgaagggagaaaggc
6095
ggacaggtatccggtaagcggcagggtcggaacaggagagcgcacgagggagcttccagg
6155
gggaaacgcctggtatctttatagtcctgtcgggtttcgccacctctgacttgagcgtcg
6215
atttttgtgatgctcgtcaggggggcggagcctatggaaaaacgccagcaacgcggcctt
6275
tttacggttcctggccttttgctggccttttgctcacatgttctttcctgcgttatcccc
6335
tgattctgtggataaccgtattaccgcctttgagtgagctgataccgctcgccgcagccg
6395
aacgaccgagcgcagcgagtcagtgagcgaggaagcggaagagcgcccaatacgcaaacc
6455
gcctctccccgcgcgttggccgattcattaatgcagctggcacgacaggtttcccgactg
6515
gaaagcgggcagtgagcgcaacgcaattaatgtgagttagctcactcattaggcacccca
6575
ggctttacactttatgcttccggctcgtatgttgtgtggaattgtgagcggataacaatt
6635
tcacacaggaaacagctatgaccatgattacgccagatttaattaaggccttaattagg
6694
<210>4
<211>510
<212>prt
<213>人工序列
<220>
<223>合成构建体
<400>4
metglupheglyleusertrpvalpheleuvalalaleuleulysgly
151015
valglncysgluvalglnleuleugluserglyproglyleuvallys
202530
prosergluthrleuserleuthrcysalavalserglyleuserile
354045
serseraspphesertrpalatrpileargglnthrproglylysala
505560
leuglutyrvalglytyrileargglyasnthrglyaspthrtyrtyr
65707580
asnproserleulysserargleuthrileserlysaspthrserlys
859095
asnglniletyrleuasnleuserservalthralaglyaspalaala
100105110
valtyrtyrcysalaargaspargvalcysaspaspasptyrglytyr
115120125
tyrtyrthrgluvalcyspheglyleuaspsertrpglyglnglyile
130135140
valvalthrvalserserglyglyglyglyserglyglyglyglyser
145150155160
glyglyglyglyseralagluleuvalmetthrglnserproleuser
165170175
leuservalalaproglyglnthralaserilesercysargserser
180185190
glnserleuasptyralaasnglyasnthrtyrleusertrpphehis
195200205
glnargproglyglnproproargargleuiletyrglnileserasn
210215220
argaspserglyvalproaspargpheserglyserglyalaglythr
225230235240
gluphethrleuargileserargmetgluseraspaspvalglyile
245250255
tyrtyrcysglyglnglythrthrpheproargthrpheglyglngly
260265270
thrlysvalgluilelysthrcysglyglyglyserlysproprothr
275280285
cysproprocysthrserprogluleuleuglyglyproservalphe
290295300
leupheproprolysprolysaspthrleumetalaserargthrpro
305310315320
gluvalthrcysvalvalvalaspvalserglngluaspproaspval
325330335
lyspheasntrptyrvalasnglyalagluvalhishisalaglnthr
340345350
lysproarggluthrglntyrasnserthrtyrargvalvalserval
355360365
leuthrvalthrhisglnasptrpleuasnglylysglutyrthrcys
370375380
lysvalserasnlysalaleuproalaproileglnlysthrileser
385390395400
lysasplysglyglnproarggluproglnvaltyrthrleupropro
405410415
serargglugluleuthrlysasnglnvalserleuthrcysleuval
420425430
lysglyphetyrproseraspilevalvalglutrpglusersergly
435440445
glnprogluasnthrtyrlysthrthrproprovalleuaspserasp
450455460
glysertyrpheleutyrserlysleuthrvalasplysserargtrp
465470475480
glnglnglyasnvalphesercysservalmethisglualaleuhis
485490495
asnhistyrthrglnlysserleuserleuserproglylys
500505510
<210>5
<211>6694
<212>dna
<213>人工序列
<220>
<223>含有201ia(h435)突变体的工程改造的质粒
<220>
<221>misc_feature
<222>(275)..(404)
<223>3'itr(互补序列)
<220>
<221>复制起点
<222>(2198)..(2786)
<220>
<221>misc_feature
<222>(3226)..(3355)
<220>
<221>misc_feature
<222>(3423)..(3804)
<223>cmvie启动子
<220>
<221>启动子
<222>(3807)..(4088)
<223>cb启动子
<220>
<221>tata_信号
<222>(4061)..(4064)
<223>兔球蛋白polya
<220>
<221>内含子
<222>(4183)..(5155)
<223>鸡β-肌动蛋白内含子
<220>
<221>cds
<222>(5161)..(6690)
<223>cmvie启动子
<400>5
gtacctctagagtcgacccgggcggcctcgaggacggggtgaactacgcctgaggatccg
60
atctttttccctctgccaaaaattatggggacatcatgaagccccttgagcatctgactt
120
ctggctaataaaggaaatttattttcattgcaatagtgtgttggaattttttgtgtctct
180
cactcggaagcaattcgttgatctgaatttcgaccacccataatacccattaccctggta
240
gataagtagcatggcgggttaatcattaactacaaggaacccctagtgatggagttggcc
300
actccctctctgcgcgctcgctcgctcactgaggccgggcgaccaaaggtcgcccgacgc
360
ccgggctttgcccgggcggcctcagtgagcgagcgagcgcgcagccttaattaacctaat
420
tcactggccgtcgttttacaacgtcgtgactgggaaaaccctggcgttacccaacttaat
480
cgccttgcagcacatccccctttcgccagctggcgtaatagcgaagaggcccgcaccgat
540
cgcccttcccaacagttgcgcagcctgaatggcgaatgggacgcgccctgtagcggcgca
600
ttaagcgcggcgggtgtggtggttacgcgcagcgtgaccgctacacttgccagcgcccta
660
gcgcccgctcctttcgctttcttcccttcctttctcgccacgttcgccggctttccccgt
720
caagctctaaatcgggggctccctttagggttccgatttagtgctttacggcacctcgac
780
cccaaaaaacttgattagggtgatggttcacgtagtgggccatcgccctgatagacggtt
840
tttcgccctttgacgttggagtccacgttctttaatagtggactcttgttccaaactgga
900
acaacactcaaccctatctcggtctattcttttgatttataagggattttgccgatttcg
960
gcctattggttaaaaaatgagctgatttaacaaaaatttaacgcgaattttaacaaaata
1020
ttaacgcttacaatttaggtggcacttttcggggaaatgtgcgcggaacccctatttgtt
1080
tatttttctaaatacattcaaatatgtatccgctcatgagacaataaccctgataaatgc
1140
ttcaataatattgaaaaaggaagagtatgagtattcaacatttccgtgtcgcccttattc
1200
ccttttttgcggcattttgccttcctgtttttgctcacccagaaacgctggtgaaagtaa
1260
aagatgctgaagatcagttgggtgcacgagtgggttacatcgaactggatctcaacagcg
1320
gtaagatccttgagagttttcgccccgaagaacgttttccaatgatgagcacttttaaag
1380
ttctgctatgtggcgcggtattatcccgtattgacgccgggcaagagcaactcggtcgcc
1440
gcatacactattctcagaatgacttggttgagtactcaccagtcacagaaaagcatctta
1500
cggatggcatgacagtaagagaattatgcagtgctgccataaccatgagtgataacactg
1560
cggccaacttacttctgacaacgatcggaggaccgaaggagctaaccgcttttttgcaca
1620
acatgggggatcatgtaactcgccttgatcgttgggaaccggagctgaatgaagccatac
1680
caaacgacgagcgtgacaccacgatgcctgtagcaatggcaacaacgttgcgcaaactat
1740
taactggcgaactacttactctagcttcccggcaacaattaatagactggatggaggcgg
1800
ataaagttgcaggaccacttctgcgctcggcccttccggctggctggtttattgctgata
1860
aatctggagccggtgagcgtgggtctcgcggtatcattgcagcactggggccagatggta
1920
agccctcccgtatcgtagttatctacacgacggggagtcaggcaactatggatgaacgaa
1980
atagacagatcgctgagataggtgcctcactgattaagcattggtaactgtcagaccaag
2040
tttactcatatatactttagattgatttaaaacttcatttttaatttaaaaggatctagg
2100
tgaagatcctttttgataatctcatgaccaaaatcccttaacgtgagttttcgttccact
2160
gagcgtcagaccccgtagaaaagatcaaaggatcttcttgagatcctttttttctgcgcg
2220
taatctgctgcttgcaaacaaaaaaaccaccgctaccagcggtggtttgtttgccggatc
2280
aagagctaccaactctttttccgaaggtaactggcttcagcagagcgcagataccaaata
2340
ctgttcttctagtgtagccgtagttaggccaccacttcaagaactctgtagcaccgccta
2400
catacctcgctctgctaatcctgttaccagtggctgctgccagtggcgataagtcgtgtc
2460
ttaccgggttggactcaagacgatagttaccggataaggcgcagcggtcgggctgaacgg
2520
ggggttcgtgcacacagcccagcttggagcgaacgacctacaccgaactgagatacctac
2580
agcgtgagctatgagaaagcgccacgcttcccgaagggagaaaggcggacaggtatccgg
2640
taagcggcagggtcggaacaggagagcgcacgagggagcttccagggggaaacgcctggt
2700
atctttatagtcctgtcgggtttcgccacctctgacttgagcgtcgatttttgtgatgct
2760
cgtcaggggggcggagcctatggaaaaacgccagcaacgcggcctttttacggttcctgg
2820
ccttttgctggccttttgctcacatgttctttcctgcgttatcccctgattctgtggata
2880
accgtattaccgcctttgagtgagctgataccgctcgccgcagccgaacgaccgagcgca
2940
gcgagtcagtgagcgaggaagcggaagagcgcccaatacgcaaaccgcctctccccgcgc
3000
gttggccgattcattaatgcagctggcacgacaggtttcccgactggaaagcgggcagtg
3060
agcgcaacgcaattaatgtgagttagctcactcattaggcaccccaggctttacacttta
3120
tgcttccggctcgtatgttgtgtggaattgtgagcggataacaatttcacacaggaaaca
3180
gctatgaccatgattacgccagatttaattaaggccttaattaggctgcgcgctcgctcg
3240
ctcactgaggccgcccgggcaaagcccgggcgtcgggcgacctttggtcgcccggcctca
3300
gtgagcgagcgagcgcgcagagagggagtggccaactccatcactaggggttccttgtag
3360
ttaatgattaacccgccatgctacttatctaccagggtaatggggatcctctagaactat
3420
agctagtcgacattgattattgactagttattaatagtaatcaattacggggtcattagt
3480
tcatagcccatatatggagttccgcgttacataacttacggtaaatggcccgcctggctg
3540
accgcccaacgacccccgcccattgacgtcaataatgacgtatgttcccatagtaacgcc
3600
aatagggactttccattgacgtcaatgggtggactatttacggtaaactgcccacttggc
3660
agtacatcaagtgtatcatatgccaagtacgccccctattgacgtcaatgacggtaaatg
3720
gcccgcctggcattatgcccagtacatgaccttatgggactttcctacttggcagtacat
3780
ctacgtattagtcatcgctattaccatggtcgaggtgagccccacgttctgcttcactct
3840
ccccatctcccccccctccccacccccaattttgtatttatttattttttaattattttg
3900
tgcagcgatgggggcggggggggggggggggcgcgcgccaggcggggcggggcggggcga
3960
ggggcggggcggggcgaggcggagaggtgcggcggcagccaatcagagcggcgcgctccg
4020
aaagtttccttttatggcgaggcggcggcggcggcggccctataaaaagcgaagcgcgcg
4080
gcgggcggggagtcgctgcgacgctgccttcgccccgtgccccgctccgccgccgcctcg
4140
cgccgcccgccccggctctgactgaccgcgttactcccacaggtgagcgggcgggacggc
4200
ccttctcctccgggctgtaattagcgcttggtttaatgacggcttgtttcttttctgtgg
4260
ctgcgtgaaagccttgaggggctccgggagggccctttgtgcggggggagcggctcgggg
4320
ggtgcgtgcgtgtgtgtgtgcgtggggagcgccgcgtgcggctccgcgctgcccggcggc
4380
tgtgagcgctgcgggcgcggcgcggggctttgtgcgctccgcagtgtgcgcgaggggagc
4440
gcggccgggggcggtgccccgcggtgcggggggggctgcgaggggaacaaaggctgcgtg
4500
cggggtgtgtgcgtgggggggtgagcagggggtgtgggcgcgtcggtcgggctgcaaccc
4560
cccctgcacccccctccccgagttgctgagcacggcccggcttcgggtgcggggctccgt
4620
acggggcgtggcgcggggctcgccgtgccgggcggggggtggcggcaggtgggggtgccg
4680
ggcggggcggggccgcctcgggccggggagggctcgggggaggggcgcggcggcccccgg
4740
agcgccggcggctgtcgaggcgcggcgagccgcagccattgccttttatggtaatcgtgc
4800
gagagggcgcagggacttcctttgtcccaaatctgtgcggagccgaaatctgggaggcgc
4860
cgccgcaccccctctagcgggcgcggggcgaagcggtgcggcgccggcaggaaggaaatg
4920
ggcggggagggccttcgtgcgtcgccgcgccgccgtccccttctccctctccagcctcgg
4980
ggctgtccgcggggggacggctgccttcgggggggacggggcagggcggggttcggcttc
5040
tggcgtgtgaccggcggctctagagcctctgctaaccatgttcatgccttcttctttttc
5100
ctacagctcctgggcaacgtgctggttattgtgctgtctcatcattttggcaaagaattc
5160
atggagttcgggctgagctgggtctttctggtggccctgctgaaggga
5208
metglupheglyleusertrpvalpheleuvalalaleuleulysgly
151015
gtccagtgcgaggtgcagctgctggaatccggacctggcctggtgaaa
5256
valglncysgluvalglnleuleugluserglyproglyleuvallys
202530
ccatctgagacactgagtctgacttgtgctgtctccggcctgtctatc
5304
prosergluthrleuserleuthrcysalavalserglyleuserile
354045
agctccgatttctcctgggcatggattaggcagacccccggcaaggcc
5352
serseraspphesertrpalatrpileargglnthrproglylysala
505560
ctggaatatgtggggtacatccgcgggaacaccggagatacatactat
5400
leuglutyrvalglytyrileargglyasnthrglyaspthrtyrtyr
65707580
aatcctagtctgaagtcaaggctgactatctcaaaggacaccagcaaa
5448
asnproserleulysserargleuthrileserlysaspthrserlys
859095
aaccaaatctacctgaatctgtctagtgtcaccgctggcgatgccgcc
5496
asnglniletyrleuasnleuserservalthralaglyaspalaala
100105110
gtgtactattgcgcaagggaccgggtgtgcgacgatgactacggatac
5544
valtyrtyrcysalaargaspargvalcysaspaspasptyrglytyr
115120125
tattacaccgaggtgtgcttcggcctggattcttgggggcagggaatc
5592
tyrtyrthrgluvalcyspheglyleuaspsertrpglyglnglyile
130135140
gtggtcacagtgtcaagcggcggaggaggcagcggaggaggagggtcc
5640
valvalthrvalserserglyglyglyglyserglyglyglyglyser
145150155160
ggaggcgggggatctgcagaactggtcatgacacagtccccactgagc
5688
glyglyglyglyseralagluleuvalmetthrglnserproleuser
165170175
ctgtccgtcgctccaggacagactgcatctattagttgtcgatcctct
5736
leuservalalaproglyglnthralaserilesercysargserser
180185190
cagtccctggactatgctaacggcaatacctacctgtcttggtttcac
5784
glnserleuasptyralaasnglyasnthrtyrleusertrpphehis
195200205
cagcgaccaggacagccacctcggagactgatctatcagatttccaac
5832
glnargproglyglnproproargargleuiletyrglnileserasn
210215220
agagattctggagtgcccgacaggttctcaggcagcggagcaggaact
5880
argaspserglyvalproaspargpheserglyserglyalaglythr
225230235240
gagtttaccctgcgaatcagtcggatggaatcagatgacgtggggatc
5928
gluphethrleuargileserargmetgluseraspaspvalglyile
245250255
tactactgcggacaggggaccacattcccacggacatttggacagggc
5976
tyrtyrcysglyglnglythrthrpheproargthrpheglyglngly
260265270
actaaggtggagatcaaaacctgtggaggaggaagcaagccaccaacc
6024
thrlysvalgluilelysthrcysglyglyglyserlysproprothr
275280285
tgccctccatgtacatctcccgaactgctgggcgggcctagcgtgttc
6072
cysproprocysthrserprogluleuleuglyglyproservalphe
290295300
ctgtttccccctaagcctaaagatacactgatgattagtagaacccca
6120
leupheproprolysprolysaspthrleumetileserargthrpro
305310315320
gaggtcacatgcgtggtcgtggacgtgtcccaggaagatcctgacgtg
6168
gluvalthrcysvalvalvalaspvalserglngluaspproaspval
325330335
aagttcaactggtacgtgaatggcgccgaggtgcaccatgctcagact
6216
lyspheasntrptyrvalasnglyalagluvalhishisalaglnthr
340345350
aaaccacgcgaaacccagtataatagtacataccgagtcgtgtcagtc
6264
lysproarggluthrglntyrasnserthrtyrargvalvalserval
355360365
ctgacagtgactcaccaggattggctgaacggcaaggagtatacctgc
6312
leuthrvalthrhisglnasptrpleuasnglylysglutyrthrcys
370375380
aaggtgtctaacaaggccctgcccgcccctatccagaaaacaattagc
6360
lysvalserasnlysalaleuproalaproileglnlysthrileser
385390395400
aaggacaaagggcagccacgggaaccccaggtgtacactctgccaccc
6408
lysasplysglyglnproarggluproglnvaltyrthrleupropro
405410415
tcaagagaggaactgactaagaaccaggtcagcctgacctgtctggtg
6456
serargglugluleuthrlysasnglnvalserleuthrcysleuval
420425430
aaaggcttctaccccagcgatatcgtcgtggagtgggaaagttcaggc
6504
lysglyphetyrproseraspilevalvalglutrpglusersergly
435440445
cagcctgagaatacttacaagactacccctccagtgctggatagcgac
6552
glnprogluasnthrtyrlysthrthrproprovalleuaspserasp
450455460
gggtcctatttcctgtacagcaagctgacagtggacaaatcccgctgg
6600
glysertyrpheleutyrserlysleuthrvalasplysserargtrp
465470475480
cagcagggaaacgtcttttcctgttctgtgatgcatgaggccctgcac
6648
glnglnglyasnvalphesercysservalmethisglualaleuhis
485490495
aatgcttacacccagaagagtctgtcactgagccccggcaaatgag
6694
asnalatyrthrglnlysserleuserleuserproglylys
500505510
<210>6
<211>510
<212>prt
<213>人工序列
<220>
<223>合成构建体
<400>6
metglupheglyleusertrpvalpheleuvalalaleuleulysgly
151015
valglncysgluvalglnleuleugluserglyproglyleuvallys
202530
prosergluthrleuserleuthrcysalavalserglyleuserile
354045
serseraspphesertrpalatrpileargglnthrproglylysala
505560
leuglutyrvalglytyrileargglyasnthrglyaspthrtyrtyr
65707580
asnproserleulysserargleuthrileserlysaspthrserlys
859095
asnglniletyrleuasnleuserservalthralaglyaspalaala
100105110
valtyrtyrcysalaargaspargvalcysaspaspasptyrglytyr
115120125
tyrtyrthrgluvalcyspheglyleuaspsertrpglyglnglyile
130135140
valvalthrvalserserglyglyglyglyserglyglyglyglyser
145150155160
glyglyglyglyseralagluleuvalmetthrglnserproleuser
165170175
leuservalalaproglyglnthralaserilesercysargserser
180185190
glnserleuasptyralaasnglyasnthrtyrleusertrpphehis
195200205
glnargproglyglnproproargargleuiletyrglnileserasn
210215220
argaspserglyvalproaspargpheserglyserglyalaglythr
225230235240
gluphethrleuargileserargmetgluseraspaspvalglyile
245250255
tyrtyrcysglyglnglythrthrpheproargthrpheglyglngly
260265270
thrlysvalgluilelysthrcysglyglyglyserlysproprothr
275280285
cysproprocysthrserprogluleuleuglyglyproservalphe
290295300
leupheproprolysprolysaspthrleumetileserargthrpro
305310315320
gluvalthrcysvalvalvalaspvalserglngluaspproaspval
325330335
lyspheasntrptyrvalasnglyalagluvalhishisalaglnthr
340345350
lysproarggluthrglntyrasnserthrtyrargvalvalserval
355360365
leuthrvalthrhisglnasptrpleuasnglylysglutyrthrcys
370375380
lysvalserasnlysalaleuproalaproileglnlysthrileser
385390395400
lysasplysglyglnproarggluproglnvaltyrthrleupropro
405410415
serargglugluleuthrlysasnglnvalserleuthrcysleuval
420425430
lysglyphetyrproseraspilevalvalglutrpglusersergly
435440445
glnprogluasnthrtyrlysthrthrproprovalleuaspserasp
450455460
glysertyrpheleutyrserlysleuthrvalasplysserargtrp
465470475480
glnglnglyasnvalphesercysservalmethisglualaleuhis
485490495
asnalatyrthrglnlysserleuserleuserproglylys
500505510
<210>7
<211>819
<212>dna
<213>人工序列
<220>
<223>工程改造的编码aducanumabscfv的序列
<400>7
atgtacaagatgcagctgctgagctgtatcgccctgaccctggtgctggtggccaactct
60
caggtgcagctggtggaatctggcggcggagtggtgcagcctggcagaagcctgagactg
120
agctgtgccgccagcggcttcgccttcagcagctacggaatgcactgggtgcgccaggcc
180
cctggcaaaggactggaatgggtggccgtgatttggttcgacggcaccaagaagtactac
240
accgacagcgtgaagggccggttcaccatcagccgggacaacagcaagaacaccctgtac
300
ctgcagatgaataccctgcgggccgaggacaccgccgtgtactactgcgccagagacaga
360
ggcatcggcgccagacggggcccttactacatggacgtgtggggcaagggcaccaccgtg
420
acagtgtctggcggaggcggaagtggcggagggggatcaggcgggggaggcagcgatatt
480
cagatgacccagagccccagcagcctgagcgcctctgtgggcgacagagtgaccatcacc
540
tgtcgggccagccagagcatcagctcctacctgaactggtatcagcagaagcccggcaag
600
gcccccaagctgctgatctatgccgcctccagtctgcagagcggcgtgcccagcagattt
660
tctggcagcggctccggcaccgacttcaccctgacaatcagctccctgcagcccgaggac
720
ttcgccacctactactgccagcagagctacagcacccccctgacattcggcggaggcacc
780
aaggtggaaatcaagcaccaccaccatcaccactgatga
819
<210>8
<211>271
<212>prt
<213>人工序列
<220>
<223>aducanumabscfv构建体氨基酸序列
<220>
<221>信号
<222>(1)..(20)
<223>il2分泌信号
<220>
<221>misc_feature
<222>(21)..(143)
<220>
<221>misc_feature
<222>(144)..(158)
<223>gly-ser接头
<220>
<221>misc_feature
<222>(159)..(265)
<223>轻链可变区
<220>
<221>misc_feature
<222>(266)..(271)
<223>6xhis标签(explasy)
<400>8
mettyrlysmetglnleuleusercysilealaleuthrleuvalleu
151015
valalaasnserglnvalglnleuvalgluserglyglyglyvalval
202530
glnproglyargserleuargleusercysalaalaserglypheala
354045
phesersertyrglymethistrpvalargglnalaproglylysgly
505560
leuglutrpvalalavaliletrppheaspglythrlyslystyrtyr
65707580
thraspservallysglyargphethrileserargaspasnserlys
859095
asnthrleutyrleuglnmetasnthrleuargalagluaspthrala
100105110
valtyrtyrcysalaargaspargglyileglyalaargargglypro
115120125
tyrtyrmetaspvaltrpglylysglythrthrvalthrvalsergly
130135140
glyglyglyserglyglyglyglyserglyglyglyglyseraspile
145150155160
glnmetthrglnserproserserleuseralaservalglyasparg
165170175
valthrilethrcysargalaserglnserilesersertyrleuasn
180185190
trptyrglnglnlysproglylysalaprolysleuleuiletyrala
195200205
alaserserleuglnserglyvalproserargpheserglysergly
210215220
serglythraspphethrleuthrileserserleuglnprogluasp
225230235240
phealathrtyrtyrcysglnglnsertyrserthrproleuthrphe
245250255
glyglyglythrlysvalgluilelyshishishishishishis
260265270
<210>9
<211>798
<212>dna
<213>人工序列
<220>
<223>工程改造的crenezumabscfv构建体
<400>9
atgtacaagatgcagctgctgagctgtatcgccctgaccctggtgctggtggccaactct
60
gaagtgcagctggtggaaagcggcggaggcctggtgcagcctggcggatctctgagactg
120
agctgtgccgccagcggcttcaccttcagcagctacggcatgagctgggtgcgccaggcc
180
cctggaaaaggcctggaactggtggcctccatcaacagcaatggcggcagcacctactac
240
cccgacagcgtgaagggccggttcaccatctcccgggacaacgccaagaacagcctgtac
300
ctgcagatgaactccctgcgggccgaggacaccgccgtgtactattgtgccagcggcgac
360
tattggggccagggcacaaccgtgacagtgtctggcggaggcggatctgggggcggagga
420
tcaggcgggggaggatctgatatcgtgatgacccagagccccctgagcctgcctgtgaca
480
cctggcgaacctgccagcatcagctgcagatccagccagagcctggtgtacagcaacggc
540
gacacctacctgcactggtatctgcagaagcccggccagagccctcagctgctgatctac
600
aaggtgtccaaccggttcagcggcgtgcccgacagattttctggcagcggctccggcacc
660
gacttcaccctgaagatcagccgggtggaagccgaggacgtgggcgtgtactactgcagc
720
cagtccacccacgtgccctggacatttggacagggcaccaaggtggaaatcaagcaccac
780
caccatcaccactgatga
798
<210>10
<211>264
<212>prt
<213>人工序列
<220>
<223>工程改造的crenezumabscfv氨基酸序列
<220>
<221>信号
<222>(1)..(20)
<223>il-2分泌信号
<220>
<221>misc_feature
<222>(21)..(131)
<223>重链可变区
<220>
<221>misc_feature
<222>(131)..(146)
<223>gly-ser接头
<220>
<221>misc_feature
<222>(147)..(258)
<223>轻链可变区
<220>
<221>misc_feature
<222>(259)..(264)
<223>6xhis标签
<400>10
mettyrlysmetglnleuleusercysilealaleuthrleuvalleu
151015
valalaasnsergluvalglnleuvalgluserglyglyglyleuval
202530
glnproglyglyserleuargleusercysalaalaserglyphethr
354045
phesersertyrglymetsertrpvalargglnalaproglylysgly
505560
leugluleuvalalaserileasnserasnglyglyserthrtyrtyr
65707580
proaspservallysglyargphethrileserargaspasnalalys
859095
asnserleutyrleuglnmetasnserleuargalagluaspthrala
100105110
valtyrtyrcysalaserglyasptyrtrpglyglnglythrthrval
115120125
thrvalserglyglyglyglyserglyglyglyglyserglyglygly
130135140
glyseraspilevalmetthrglnserproleuserleuprovalthr
145150155160
proglygluproalaserilesercysargserserglnserleuval
165170175
tyrserasnglyaspthrtyrleuhistrptyrleuglnlysprogly
180185190
glnserproglnleuleuiletyrlysvalserasnargphesergly
195200205
valproaspargpheserglyserglyserglythraspphethrleu
210215220
lysileserargvalglualagluaspvalglyvaltyrtyrcysser
225230235240
glnserthrhisvalprotrpthrpheglyglnglythrlysvalglu
245250255
ilelyshishishishishishis
260
<210>11
<211>798
<212>dna
<213>人工序列
<220>
<223>工程改造的solanezumabscfv构建体核酸序列:
<400>11
atgtacaagatgcagctgctgagctgtatcgccctgaccctggtgctggtggccaactct
60
gaagtgcagctggtggaaagcggcggaggcctggtgcagcctggcggatctctgagactg
120
agctgtgccgccagcggcttcaccttcagccggtacagcatgagctgggtgcgccaggcc
180
cctggcaaaggactggaactggtggcccagatcaacagcgtgggcaacagcacctactac
240
cccgacaccgtgaagggccggttcaccatcagcagagacaacgccaagaacaccctgtac
300
ctgcagatgaacagcctgcgggccgaggacaccgccgtgtactattgtgccagcggcgac
360
tattggggccagggcacactcgtgacagtgtctggcggaggcggatctgggggcggagga
420
tcaggcgggggaggatctgatgtcgtgatgacccagagccccctgagcctgcctgtgaca
480
ctgggacagcctgccagcatcagctgcagaagcagccagagcctgatctacagcgacggc
540
aacgcctacctgcactggtttctgcagaaacccggccagtcccccagactgctgatctat
600
aaggtgtccaaccggttcagcggcgtgcccgacagattttctggcagcggctccggcacc
660
gacttcaccctgaagatcagccgggtggaagccgaggacgtgggcgtgtactactgcagc
720
cagtccacccacgtgccctggacatttggacagggcaccaaggtggaaatcaagcaccac
780
caccatcaccactgatga
798
<210>12
<211>264
<212>prt
<213>人工序列
<220>
<223>工程改造的solanezumabscfv构建体氨基酸序列
<220>
<221>信号
<222>(1)..(20)
<223>il2分泌信号
<220>
<221>misc_feature
<222>(21)..(131)
<223>重链可变区
<220>
<221>misc_feature
<222>(132)..(146)
<220>
<221>misc_feature
<222>(147)..(258)
<223>轻链可变区
<220>
<221>misc_feature
<222>(259)..(264)
<223>6xhis标签
<400>12
mettyrlysmetglnleuleusercysilealaleuthrleuvalleu
151015
valalaasnsergluvalglnleuvalgluserglyglyglyleuval
202530
glnproglyglyserleuargleusercysalaalaserglyphethr
354045
pheserargtyrsermetsertrpvalargglnalaproglylysgly
505560
leugluleuvalalaglnileasnservalglyasnserthrtyrtyr
65707580
proaspthrvallysglyargphethrileserargaspasnalalys
859095
asnthrleutyrleuglnmetasnserleuargalagluaspthrala
100105110
valtyrtyrcysalaserglyasptyrtrpglyglnglythrleuval
115120125
thrvalserglyglyglyglyserglyglyglyglyserglyglygly
130135140
glyseraspvalvalmetthrglnserproleuserleuprovalthr
145150155160
leuglyglnproalaserilesercysargserserglnserleuile
165170175
tyrseraspglyasnalatyrleuhistrppheleuglnlysprogly
180185190
glnserproargleuleuiletyrlysvalserasnargphesergly
195200205
valproaspargpheserglyserglyserglythraspphethrleu
210215220
lysileserargvalglualagluaspvalglyvaltyrtyrcysser
225230235240
glnserthrhisvalprotrpthrpheglyglnglythrlysvalglu
245250255
ilelyshishishishishishis
260
<210>13
<211>3165
<212>dna
<213>人工序列
<220>
<223>aav载体基因组cb7.ci.aducanumabscfvrbg
<220>
<221>重复区
<222>(1)..(130)
<223>5"aav2-itr
<220>
<221>重复区
<222>(198)..(579)
<223>cmvie启动子
<220>
<221>启动子
<222>(582)..(863)
<223>cb启动子
<220>
<221>tata_信号
<222>(836)..(838)
<220>
<221>内含子
<222>(958)..(1930)
<223>鸡β-肌动蛋白启动子
<220>
<221>misc_feature
<222>(1958)..(2001)
<220>
<221>misc_feature
<222>(2002)..(2370)
<223>aducanumab重链可变区
<220>
<221>misc_feature
<222>(2371)..(2415)
<223>gly-ser接头
<220>
<221>misc_feature
<222>(2416)..(2736)
<223>aducanumab轻链可变区
<220>
<221>misc_feature
<222>(2737)..(2754)
<223>his标签
<220>
<221>polya_信号
<222>(2821)..(2947)
<223>兔球蛋白polya
<220>
<221>重复区
<222>(3036)..(3165)
<223>3'itr
<400>13
ctgcgcgctcgctcgctcactgaggccgcccgggcaaagcccgggcgtcgggcgaccttt
60
ggtcgcccggcctcagtgagcgagcgagcgcgcagagagggagtggccaactccatcact
120
aggggttccttgtagttaatgattaacccgccatgctacttatctaccagggtaatgggg
180
atcctctagaactatagctagtcgacattgattattgactagttattaatagtaatcaat
240
tacggggtcattagttcatagcccatatatggagttccgcgttacataacttacggtaaa
300
tggcccgcctggctgaccgcccaacgacccccgcccattgacgtcaataatgacgtatgt
360
tcccatagtaacgccaatagggactttccattgacgtcaatgggtggactatttacggta
420
aactgcccacttggcagtacatcaagtgtatcatatgccaagtacgccccctattgacgt
480
caatgacggtaaatggcccgcctggcattatgcccagtacatgaccttatgggactttcc
540
tacttggcagtacatctacgtattagtcatcgctattaccatggtcgaggtgagccccac
600
gttctgcttcactctccccatctcccccccctccccacccccaattttgtatttatttat
660
tttttaattattttgtgcagcgatgggggcggggggggggggggggcgcgcgccaggcgg
720
ggcggggcggggcgaggggcggggcggggcgaggcggagaggtgcggcggcagccaatca
780
gagcggcgcgctccgaaagtttccttttatggcgaggcggcggcggcggcggccctataa
840
aaagcgaagcgcgcggcgggcggggagtcgctgcgacgctgccttcgccccgtgccccgc
900
tccgccgccgcctcgcgccgcccgccccggctctgactgaccgcgttactcccacaggtg
960
agcgggcgggacggcccttctcctccgggctgtaattagcgcttggtttaatgacggctt
1020
gtttcttttctgtggctgcgtgaaagccttgaggggctccgggagggccctttgtgcggg
1080
gggagcggctcggggggtgcgtgcgtgtgtgtgtgcgtggggagcgccgcgtgcggctcc
1140
gcgctgcccggcggctgtgagcgctgcgggcgcggcgcggggctttgtgcgctccgcagt
1200
gtgcgcgaggggagcgcggccgggggcggtgccccgcggtgcggggggggctgcgagggg
1260
aacaaaggctgcgtgcggggtgtgtgcgtgggggggtgagcagggggtgtgggcgcgtcg
1320
gtcgggctgcaaccccccctgcacccccctccccgagttgctgagcacggcccggcttcg
1380
ggtgcggggctccgtacggggcgtggcgcggggctcgccgtgccgggcggggggtggcgg
1440
caggtgggggtgccgggcggggcggggccgcctcgggccggggagggctcgggggagggg
1500
cgcggcggcccccggagcgccggcggctgtcgaggcgcggcgagccgcagccattgcctt
1560
ttatggtaatcgtgcgagagggcgcagggacttcctttgtcccaaatctgtgcggagccg
1620
aaatctgggaggcgccgccgcaccccctctagcgggcgcggggcgaagcggtgcggcgcc
1680
ggcaggaaggaaatgggcggggagggccttcgtgcgtcgccgcgccgccgtccccttctc
1740
cctctccagcctcggggctgtccgcggggggacggctgccttcgggggggacggggcagg
1800
gcggggttcggcttctggcgtgtgaccggcggctctagagcctctgctaaccatgttcat
1860
gccttcttctttttcctacagctcctgggcaacgtgctggttattgtgctgtctcatcat
1920
tttggcaaagaattcgccaccatgtacaagatgcagctgctgagctgtatcgccctgacc
1980
ctggtgctggtggccaactctcaggtgcagctggtggaatctggcggcggagtggtgcag
2040
cctggcagaagcctgagactgagctgtgccgccagcggcttcgccttcagcagctacgga
2100
atgcactgggtgcgccaggcccctggcaaaggactggaatgggtggccgtgatttggttc
2160
gacggcaccaagaagtactacaccgacagcgtgaagggccggttcaccatcagccgggac
2220
aacagcaagaacaccctgtacctgcagatgaataccctgcgggccgaggacaccgccgtg
2280
tactactgcgccagagacagaggcatcggcgccagacggggcccttactacatggacgtg
2340
tggggcaagggcaccaccgtgacagtgtctggcggaggcggaagtggcggagggggatca
2400
ggcgggggaggcagcgatattcagatgacccagagccccagcagcctgagcgcctctgtg
2460
ggcgacagagtgaccatcacctgtcgggccagccagagcatcagctcctacctgaactgg
2520
tatcagcagaagcccggcaaggcccccaagctgctgatctatgccgcctccagtctgcag
2580
agcggcgtgcccagcagattttctggcagcggctccggcaccgacttcaccctgacaatc
2640
agctccctgcagcccgaggacttcgccacctactactgccagcagagctacagcaccccc
2700
ctgacattcggcggaggcaccaaggtggaaatcaagcaccaccaccatcaccactgatga
2760
ggtacctctagagtcgacccgggcggcctcgaggacggggtgaactacgcctgaggatcc
2820
gatctttttccctctgccaaaaattatggggacatcatgaagccccttgagcatctgact
2880
tctggctaataaaggaaatttattttcattgcaatagtgtgttggaattttttgtgtctc
2940
tcactcggaagcaattcgttgatctgaatttcgaccacccataatacccattaccctggt
3000
agataagtagcatggcgggttaatcattaactacaaggaacccctagtgatggagttggc
3060
cactccctctctgcgcgctcgctcgctcactgaggccgggcgaccaaaggtcgcccgacg
3120
cccgggctttgcccgggcggcctcagtgagcgagcgagcgcgcag
3165
<210>14
<211>3144
<212>dna
<213>人工序列
<220>
<223>paav.cb7.ci.crenezumabscfv.rbg
<220>
<221>重复区
<222>(1)..(130)
<223>aav2-5'itr
<220>
<221>重复区
<222>(198)..(579)
<223>cmvie启动子
<220>
<221>启动子
<222>(582)..(863)
<223>cb启动子
<220>
<221>tata_信号
<222>(836)..(839)
<220>
<221>内含子
<222>(958)..(1930)
<220>
<221>misc_feature
<222>(1942)..(2001)
<223>il-2分泌信号
<220>
<221>misc_feature
<222>(2001)..(2334)
<223>crenezumab重链可变区
<220>
<221>misc_feature
<222>(2335)..(2379)
<223>gly-ser接头
<220>
<221>misc_feature
<222>(2380)..(2715)
<223>crenezumab轻链可变区
<220>
<221>misc_feature
<222>(2716)..(2733)
<223>his标签
<220>
<221>polya_信号
<222>(2800)..(2926)
<223>兔球蛋白polya
<220>
<221>重复区
<222>(3015)..(3144)
<223>aav2-3'itr
<400>14
ctgcgcgctcgctcgctcactgaggccgcccgggcaaagcccgggcgtcgggcgaccttt
60
ggtcgcccggcctcagtgagcgagcgagcgcgcagagagggagtggccaactccatcact
120
aggggttccttgtagttaatgattaacccgccatgctacttatctaccagggtaatgggg
180
atcctctagaactatagctagtcgacattgattattgactagttattaatagtaatcaat
240
tacggggtcattagttcatagcccatatatggagttccgcgttacataacttacggtaaa
300
tggcccgcctggctgaccgcccaacgacccccgcccattgacgtcaataatgacgtatgt
360
tcccatagtaacgccaatagggactttccattgacgtcaatgggtggactatttacggta
420
aactgcccacttggcagtacatcaagtgtatcatatgccaagtacgccccctattgacgt
480
caatgacggtaaatggcccgcctggcattatgcccagtacatgaccttatgggactttcc
540
tacttggcagtacatctacgtattagtcatcgctattaccatggtcgaggtgagccccac
600
gttctgcttcactctccccatctcccccccctccccacccccaattttgtatttatttat
660
tttttaattattttgtgcagcgatgggggcggggggggggggggggcgcgcgccaggcgg
720
ggcggggcggggcgaggggcggggcggggcgaggcggagaggtgcggcggcagccaatca
780
gagcggcgcgctccgaaagtttccttttatggcgaggcggcggcggcggcggccctataa
840
aaagcgaagcgcgcggcgggcggggagtcgctgcgacgctgccttcgccccgtgccccgc
900
tccgccgccgcctcgcgccgcccgccccggctctgactgaccgcgttactcccacaggtg
960
agcgggcgggacggcccttctcctccgggctgtaattagcgcttggtttaatgacggctt
1020
gtttcttttctgtggctgcgtgaaagccttgaggggctccgggagggccctttgtgcggg
1080
gggagcggctcggggggtgcgtgcgtgtgtgtgtgcgtggggagcgccgcgtgcggctcc
1140
gcgctgcccggcggctgtgagcgctgcgggcgcggcgcggggctttgtgcgctccgcagt
1200
gtgcgcgaggggagcgcggccgggggcggtgccccgcggtgcggggggggctgcgagggg
1260
aacaaaggctgcgtgcggggtgtgtgcgtgggggggtgagcagggggtgtgggcgcgtcg
1320
gtcgggctgcaaccccccctgcacccccctccccgagttgctgagcacggcccggcttcg
1380
ggtgcggggctccgtacggggcgtggcgcggggctcgccgtgccgggcggggggtggcgg
1440
caggtgggggtgccgggcggggcggggccgcctcgggccggggagggctcgggggagggg
1500
cgcggcggcccccggagcgccggcggctgtcgaggcgcggcgagccgcagccattgcctt
1560
ttatggtaatcgtgcgagagggcgcagggacttcctttgtcccaaatctgtgcggagccg
1620
aaatctgggaggcgccgccgcaccccctctagcgggcgcggggcgaagcggtgcggcgcc
1680
ggcaggaaggaaatgggcggggagggccttcgtgcgtcgccgcgccgccgtccccttctc
1740
cctctccagcctcggggctgtccgcggggggacggctgccttcgggggggacggggcagg
1800
gcggggttcggcttctggcgtgtgaccggcggctctagagcctctgctaaccatgttcat
1860
gccttcttctttttcctacagctcctgggcaacgtgctggttattgtgctgtctcatcat
1920
tttggcaaagaattcgccaccatgtacaagatgcagctgctgagctgtatcgccctgacc
1980
ctggtgctggtggccaactctgaagtgcagctggtggaaagcggcggaggcctggtgcag
2040
cctggcggatctctgagactgagctgtgccgccagcggcttcaccttcagcagctacggc
2100
atgagctgggtgcgccaggcccctggaaaaggcctggaactggtggcctccatcaacagc
2160
aatggcggcagcacctactaccccgacagcgtgaagggccggttcaccatctcccgggac
2220
aacgccaagaacagcctgtacctgcagatgaactccctgcgggccgaggacaccgccgtg
2280
tactattgtgccagcggcgactattggggccagggcacaaccgtgacagtgtctggcgga
2340
ggcggatctgggggcggaggatcaggcgggggaggatctgatatcgtgatgacccagagc
2400
cccctgagcctgcctgtgacacctggcgaacctgccagcatcagctgcagatccagccag
2460
agcctggtgtacagcaacggcgacacctacctgcactggtatctgcagaagcccggccag
2520
agccctcagctgctgatctacaaggtgtccaaccggttcagcggcgtgcccgacagattt
2580
tctggcagcggctccggcaccgacttcaccctgaagatcagccgggtggaagccgaggac
2640
gtgggcgtgtactactgcagccagtccacccacgtgccctggacatttggacagggcacc
2700
aaggtggaaatcaagcaccaccaccatcaccactgatgaggtacctctagagtcgacccg
2760
ggcggcctcgaggacggggtgaactacgcctgaggatccgatctttttccctctgccaaa
2820
aattatggggacatcatgaagccccttgagcatctgacttctggctaataaaggaaattt
2880
attttcattgcaatagtgtgttggaattttttgtgtctctcactcggaagcaattcgttg
2940
atctgaatttcgaccacccataatacccattaccctggtagataagtagcatggcgggtt
3000
aatcattaactacaaggaacccctagtgatggagttggccactccctctctgcgcgctcg
3060
ctcgctcactgaggccgggcgaccaaaggtcgcccgacgcccgggctttgcccgggcggc
3120
ctcagtgagcgagcgagcgcgcag
3144
<210>15
<211>3144
<212>dna
<213>人工序列
<220>
<223>paav.cb7.ci.solanezumabscfv.rbg
<220>
<221>重复区
<222>(1)..(130)
<223>aav2-5'itr
<220>
<221>重复区
<222>(198)..(579)
<223>cmvie启动子
<220>
<221>启动子
<222>(582)..(863)
<223>cb启动子
<220>
<221>tata_信号
<222>(836)..(839)
<220>
<221>内含子
<222>(958)..(1930)
<223>鸡β-肌动蛋白内含子
<220>
<221>misc_feature
<222>(1942)..(2001)
<223>il-2分泌信号
<220>
<221>misc_feature
<222>(2002)..(2334)
<223>solanezumab重链可变区
<220>
<221>misc_feature
<222>(2335)..(2379)
<223>gly-ser接头
<220>
<221>misc_feature
<222>(2380)..(2715)
<223>solanezumab轻链可变区
<220>
<221>polya_信号
<222>(2800)..(2926)
<223>兔球蛋白polya
<220>
<221>misc_feature
<222>(3015)..(3144)
<223>aav2-3'itr
<400>15
ctgcgcgctcgctcgctcactgaggccgcccgggcaaagcccgggcgtcgggcgaccttt
60
ggtcgcccggcctcagtgagcgagcgagcgcgcagagagggagtggccaactccatcact
120
aggggttccttgtagttaatgattaacccgccatgctacttatctaccagggtaatgggg
180
atcctctagaactatagctagtcgacattgattattgactagttattaatagtaatcaat
240
tacggggtcattagttcatagcccatatatggagttccgcgttacataacttacggtaaa
300
tggcccgcctggctgaccgcccaacgacccccgcccattgacgtcaataatgacgtatgt
360
tcccatagtaacgccaatagggactttccattgacgtcaatgggtggactatttacggta
420
aactgcccacttggcagtacatcaagtgtatcatatgccaagtacgccccctattgacgt
480
caatgacggtaaatggcccgcctggcattatgcccagtacatgaccttatgggactttcc
540
tacttggcagtacatctacgtattagtcatcgctattaccatggtcgaggtgagccccac
600
gttctgcttcactctccccatctcccccccctccccacccccaattttgtatttatttat
660
tttttaattattttgtgcagcgatgggggcggggggggggggggggcgcgcgccaggcgg
720
ggcggggcggggcgaggggcggggcggggcgaggcggagaggtgcggcggcagccaatca
780
gagcggcgcgctccgaaagtttccttttatggcgaggcggcggcggcggcggccctataa
840
aaagcgaagcgcgcggcgggcggggagtcgctgcgacgctgccttcgccccgtgccccgc
900
tccgccgccgcctcgcgccgcccgccccggctctgactgaccgcgttactcccacaggtg
960
agcgggcgggacggcccttctcctccgggctgtaattagcgcttggtttaatgacggctt
1020
gtttcttttctgtggctgcgtgaaagccttgaggggctccgggagggccctttgtgcggg
1080
gggagcggctcggggggtgcgtgcgtgtgtgtgtgcgtggggagcgccgcgtgcggctcc
1140
gcgctgcccggcggctgtgagcgctgcgggcgcggcgcggggctttgtgcgctccgcagt
1200
gtgcgcgaggggagcgcggccgggggcggtgccccgcggtgcggggggggctgcgagggg
1260
aacaaaggctgcgtgcggggtgtgtgcgtgggggggtgagcagggggtgtgggcgcgtcg
1320
gtcgggctgcaaccccccctgcacccccctccccgagttgctgagcacggcccggcttcg
1380
ggtgcggggctccgtacggggcgtggcgcggggctcgccgtgccgggcggggggtggcgg
1440
caggtgggggtgccgggcggggcggggccgcctcgggccggggagggctcgggggagggg
1500
cgcggcggcccccggagcgccggcggctgtcgaggcgcggcgagccgcagccattgcctt
1560
ttatggtaatcgtgcgagagggcgcagggacttcctttgtcccaaatctgtgcggagccg
1620
aaatctgggaggcgccgccgcaccccctctagcgggcgcggggcgaagcggtgcggcgcc
1680
ggcaggaaggaaatgggcggggagggccttcgtgcgtcgccgcgccgccgtccccttctc
1740
cctctccagcctcggggctgtccgcggggggacggctgccttcgggggggacggggcagg
1800
gcggggttcggcttctggcgtgtgaccggcggctctagagcctctgctaaccatgttcat
1860
gccttcttctttttcctacagctcctgggcaacgtgctggttattgtgctgtctcatcat
1920
tttggcaaagaattcgccaccatgtacaagatgcagctgctgagctgtatcgccctgacc
1980
ctggtgctggtggccaactctgaagtgcagctggtggaaagcggcggaggcctggtgcag
2040
cctggcggatctctgagactgagctgtgccgccagcggcttcaccttcagccggtacagc
2100
atgagctgggtgcgccaggcccctggcaaaggactggaactggtggcccagatcaacagc
2160
gtgggcaacagcacctactaccccgacaccgtgaagggccggttcaccatcagcagagac
2220
aacgccaagaacaccctgtacctgcagatgaacagcctgcgggccgaggacaccgccgtg
2280
tactattgtgccagcggcgactattggggccagggcacactcgtgacagtgtctggcgga
2340
ggcggatctgggggcggaggatcaggcgggggaggatctgatgtcgtgatgacccagagc
2400
cccctgagcctgcctgtgacactgggacagcctgccagcatcagctgcagaagcagccag
2460
agcctgatctacagcgacggcaacgcctacctgcactggtttctgcagaaacccggccag
2520
tcccccagactgctgatctataaggtgtccaaccggttcagcggcgtgcccgacagattt
2580
tctggcagcggctccggcaccgacttcaccctgaagatcagccgggtggaagccgaggac
2640
gtgggcgtgtactactgcagccagtccacccacgtgccctggacatttggacagggcacc
2700
aaggtggaaatcaagcaccaccaccatcaccactgatgaggtacctctagagtcgacccg
2760
ggcggcctcgaggacggggtgaactacgcctgaggatccgatctttttccctctgccaaa
2820
aattatggggacatcatgaagccccttgagcatctgacttctggctaataaaggaaattt
2880
attttcattgcaatagtgtgttggaattttttgtgtctctcactcggaagcaattcgttg
2940
atctgaatttcgaccacccataatacccattaccctggtagataagtagcatggcgggtt
3000
aatcattaactacaaggaacccctagtgatggagttggccactccctctctgcgcgctcg
3060
ctcgctcactgaggccgggcgaccaaaggtcgcccgacgcccgggctttgcccgggcggc
3120
ctcagtgagcgagcgagcgcgcag
3144
- 还没有人留言评论。精彩留言会获得点赞!