一组胃癌基因的应用的制作方法
本发明属于肿瘤基因检测技术领域,具体涉及一组胃癌相关基因的应用。
背景技术:
胃癌是一种起始于胃黏膜组织上皮细胞的恶性肿瘤。胃癌一直是世界上最常见的恶性肿瘤之一,发病率仅次于肺癌、乳腺癌、结肠直肠癌和前列腺癌,在世界癌症发病率中排名第五位,胃癌的发病率和死亡率一直居高不下;虽然近十年来世界胃癌总发病率和死亡率有所下降,但患胃癌的绝对人数依旧呈上升趋势,每年有近百万的新增病例。我国每年新发病例约40万例,占世界总发病例数的42%。从国家卫生和计划生育委员会官方网站公布数据显示来看:2005年城市和农村居民胃癌死亡率分别为18.12/10万和19.05/10万,2006年为19.66/10万和22.09/10万,2007年为22.87/10万和率23.35/10万,2008年为18.60/10万和26.33/10万,2009年为18.17/10万和23.10/10万,2010年为18.63/10万和22.57/10万,2011年为19.66/10万和22.09/10万,均居恶性肿瘤死因第三位。对我国胃癌进行调查表明胃癌基本均居恶性肿瘤发病率及死亡率前三位,胃癌仍然是当今我国肿瘤防治的重点。
随着科技水平的不断发展,胃癌早期诊断水平有了一定提高,使得胃癌五年生存率有了大幅的提高。但对于进展期的胃癌,其五年生存率仅为29.3%,主要原因是胃癌早期不易诊断,发现较晚,从而错过了最佳的治疗时间,并且胃癌容易复发和转移。胃癌的治疗主要分为手术治疗、放射治疗、化学治疗、靶向治疗等。化疗是晚期/转移胃癌病人的主要治疗方案,但往往伴严重副作用。近年来以曲妥珠单抗为代表的靶向药物开启了胃癌靶向治疗的新时代。目前曲妥珠单抗联合化疗已成为人表皮生长因子受体2(HER2/ERBB2)基因扩增或过度表达阳性患者的首选治疗。
胃癌是一种多基因控制的疾病,是多种促癌和抗癌基因与体内微环境的共同作用,使胃黏膜发生早期病变到异常增生,最终发展到胃癌。在这整个过程中都存在相关基因特征性差异表达。临床胃癌分期和分化程度一直缺少相应的分子标记物对其进行区分。近年来越来越多的证据显示胃癌组织的分子生物学特征也对预后起着重要作用。比如,大约10–30%的胃癌患者具有HER2/ERBB2基因的扩增或过度表达,后者和胃癌的预后和淋巴结转移密切相关。也有证据提示p53蛋白的积累与胃癌的预后呈负相关。另外,转录因子缺氧诱导因子1α(HIF-1α)在胃癌细胞中高表达,并且在TNM分类早期胃癌患者中表达更高,可能与胃癌的早期发展相关。
在目前的癌症研究中,芯片技术和二代测序技术已经成为研究胃癌体细胞遗传异质性和复杂性的重要工具,为开发诊断、治疗和预后相关生物标志物提供了巨大的信息。基因表达分析可将同一肿瘤分成不同亚型并对其预后进行研究。在基因表达分析技术的帮助下,可构建基因的相关网络,后者被证明对研讨癌症的发生发展具有重要的意义。比如以CDKNIA为节点构建了一胃癌调控网络,并筛选出7个和胃癌发生相关的基因:MMP7,SPARC,SOD2,INHBA,IGFBP7,NEK6和LUM。结果显示这7个基因随着病情进展被激活,表明它们可能与癌症的发展有关。
在其它肿瘤方面,美国Genomic Health公司研发的Oncotype DX和挪威Agendia公司研发的MammaPrint基因检测技术可对乳腺癌的复发与转移的预后进行评估,并为病人是否需要化疗提供指导信息。Oncotype DX是对ER阳性、淋巴结阴性乳腺癌石蜡包埋组织标本RNA使用实时定量PCR(RT-PCR)进行21个基因表达的定量检测,包括16个与复发相关的目标基因(与增殖、侵袭、HER2、激素等)和5个参考基因。将乳腺癌患者10年的复发风险分为低(RS<18)、中(RS18~31)和高(RS≥31)三组,判断患者是否需要化疗。通常对低RS的患者不推荐化疗,而推荐其用于高RS的病人。中RS主要基于患者的年龄和健康状况推荐是否进行化疗。MammaPrint是基于70个基因的表达来预测ER阳性和ER阴性并淋巴结阴性乳腺癌患者的复发,在预测转移和生存期方面优于临床病理学指标。两项检测均获美国FDA批准上市。另外,Oncotype DX被列为NCCN指南推荐和美国医疗保险的乳腺癌检测项目。虽然遗传学和基因组学是相关的,但提供不同的信息。基因测试通常筛查可能发展一种疾病或癌症的遗传危险因素,而基因组测试,比如Oncotype DX,评估一组重要癌症相关基因的活性以揭示一特定个体肿瘤的生物学特性,可更准确的预测该肿瘤的行为。
Genomic Health公司也开发了针对前列腺癌和结肠癌的Oncotype DX基因检测项目,但迄今为至世界上尚未有针对胃癌预后的类似检测。因此,很有必要在现有知识和技术的基础上,设计研发一项胃癌的多基因表达谱和预后评分系统。
技术实现要素:
解决的技术问题:本发明应用国际通用肿瘤数据库,通过建立多步分析法来全面确定相关的249个胃癌生物标志物;然后用逐步的多变量聚类技术(multivariate clustering technique)来确定和胃癌预后相关的关键基因。基于这些分析,我们创建了53个基因表达谱和预后评分系统,并成功的将其应用于胃癌临床数据的生存期预测。此方法可用于协助胃癌病人的治疗选择以及预测对治疗干预的反应,从而判断患者从化疗/靶向治疗获益程度、达到避免过度用药、降低医疗成本的目的。
技术方案:为了实现以上目的,本发明采用的技术方案为:
一项用于评价胃癌预后的多基因表达谱和评分系统。本发明包括了53个胃癌预后相关基因和对其在临床样本中表达水平的检测,然后通过计算预后评分来预测临床预后。
作为优选方案,我们首先通过正常和胃癌组织之间的比较确定了在胃癌中有显著差异表达的基因。我们开发了一个多步骤的策略,以寻找能分辨胃癌患者预后好坏的关键基因标签(gene signature)。我们使用了两个公开可用的国际肿瘤数据库:(1)由RNA测序建立的癌症基因数据库(The Cancer Genome Atlas,TCGA);(2)由Affymetrix芯片(Affymetrix Genechip arrays,HG-U133 Plus 2.0)建立的人胃肿瘤和正常组织库GSE30727。我们发现TCGA里的688基因和GSE30727的3239基因均达到我们的选择标准,即2倍的表达变化和调整p值<0.05。其中有276个基因在TCGA和GSE30727数据库之间相重叠,包括57个胃癌表达下调基因和219个胃癌表达上调基因。
作为优选方案,我们进一步评估了上述276个基因的差异表达在胃癌临床进展中的重要性。我们利用生存预后在线工具Kaplan-Meier曲线(http://kmplot.com/analysis/index.php?p=service&cancer=gastric)分析了它们在大型公共临床芯片胃癌数据库中对胃癌患者预后的应用价值。基于其表达的水平,这些基因被分成两组(高和低表达)。随后,使用Kaplan-Meier曲线(图1)显示这些基因的高或低表达水平对胃癌病人五年存活率的影响,现其中249个基因与总体存活率显著相关联。这一结果表明,这些分子标志物可以有效的预测胃癌患者的治疗预后。最后,我们根据单因素分析得出的p值来将基因对临床预后的重要性进行排名(表一),作为后续选择基因的依据。
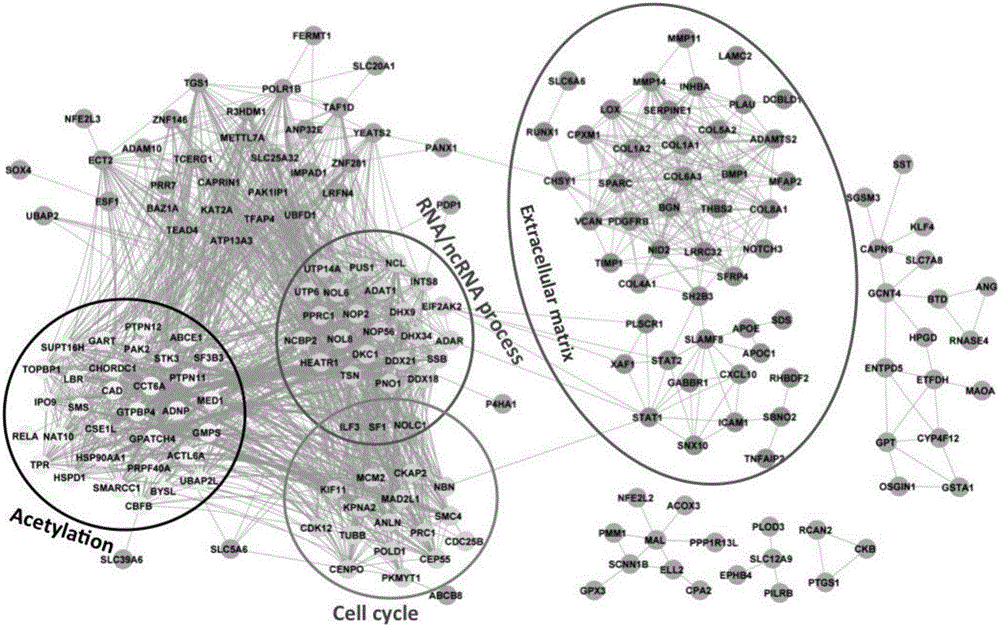
作为优选方案,我们建立了249个基因在胃癌中的共表达网络(geneco-expression network),以更好地揭示这些基因的生物学功能和胃癌发展的分子机制。通过应用生物信息数据库The Database for Annotation,Visualization and Integrated Discovery(DAVID),观察到这些基因显著集中在调节细胞增殖,粘附和迁移,RNA/ncRNA过程,乙酰化,细胞外基质等生物学功能(图2),所有这些都是癌症的特征。接下来我们用相关网络分析软件(http://baderlab.org/Software/ExpressionCorrelation)和TCGA数据,来识别和构建胃癌患者的功能相关的基因共表达网络(图2)。
作为优选方案,基于上述结果,我们开发了一项胃癌预后评分系统。我们应用逐步的典型判别分析(canonical discriminant analysis)来标识能够以100%的准确性鉴别病人预后好或差的基因标签,最后确定了53个特定的胃癌预后生物标志物基因,评分系统获得了100%的预后预测准确率,具体包括:(1)细胞周期相关基因:CEP55,MCM2,PRC1,SCNN1B,TUBB;(2)乙酰化相关基因:ADNP,ABCE1,CBFB,CHORDC1,CCT6A,GART,SMS;(3)RNA/ncRNA通路相关基因:NOL8,NCL,PNO1;(4)细胞外基质相关基因:APOE,APOC1,CXCL10,COL6A3,CPXM1,GABBR1,INHBA,LAMC2,MMP14,TNFAIP2;(5)其它基因:ADH1C,ALDH6A1,ATP13A3,BAZ1A,BCAR3,CAPRIN1,CXCL1,CCT2,ECHD2,ETFDH,ENC1,EPHB4,FHOD1,FGFR4,KAT2A,KLF4,LRRC41,LIMK1,OSMA,PTGS1,PGRMC2,P4HA1,PDP1,PRR7,SCC12A9,SLC20A1,TGS1和TCERG1(图3)。
所述胃癌预后评分系统用预测分值来计算患者的生存概率。预测分值被定义为通过典型判别函数为基础的基因表达水平的线性组合。计算公式见下:
注:见表二。
如果预后评分≤-2,我们定义患者具有好预后的基因标签(goodsignature);反之预后评分>-2,我们定义患者具有差预后的基因标签(bad signature)(见图4)。我们用TCGA数据库的数据评估了此预后评分系统的精准性。图5显示前者比后者生存时间显著延长。超过50%的前者在100个月后仍然存活,而所有携带有差预后基因标签的患者在80个月内均死亡。总之,我们的试验结果显示在好和差预后中,预后评分的分布是差别明显的(图5),这表明所述预后评分系统具有区分好和差预后的良好能力。我们运用GSE15459数据库的数据同样得到了类似的精准性结果(见实施例2,图6)。
作为优选方案,我们根据不同的检测技术平台,包括但不限于实时荧光定量PCR、基因芯片、二代高通量测序、Panomics、Nanostring技术,通过采集胃癌病人肿瘤组织的RNA,包括但不限于新鲜活检组织,术后组织,固定后的组织和石蜡包埋的组织,设计开发了相应的测量试剂盒以及相应的评分系统。本发明开发的试剂盒针对不同的技术平台,设计了相应的基因引物(实时荧光定量PCR)和靶针(基因芯片、二代测序、Panomics和Nanostring技术)。
我们此发明中定义的预测分值(≤-2和>-2)是根据以二代测序为基础的TCGA数据库的数据而制定的。预测分值的绝对值和分数划界可依不同的检测技术平台而不同,需要分别确定。
有益效果:
虽然一些分子特征研究已经在胃癌中进行,但很少有研究试图找出和胃癌预后相关联的基因标签,更尚未见预后评分系统在临床运用的报道。本发明通过使用多组学数据成功寻找到预测胃癌患者总生存期的一组53个重要生物标志物基因,并首次建立了基于53个基因标签的预后评分系统。我们也证实了所述系统的预测分值能够明确的区分预后好差。此发明可用于帮助对胃癌病人的治疗选择以及预测对治疗干预的反应,从而判断患者从化疗和靶向治疗中获益、避免过度用药、降低医疗成本,最后达到个体化医疗的目的。
附图说明
图1:胃癌相关基因的Kaplan-Meier生存曲线举例。p值通过对比两组之间差别检定(log-rank test)而得到。
图2:用于本发明的胃癌基因的共表达网络图。
图3:本发明胃癌预后评分系统中的53基因及相关功能/通路。
图4:本发明预后评分在好和差胃癌预后间的分布图。
图5:Kaplan-Meier生存曲线显示预后分数与TCGA库中胃癌总体生存显著相关。
图6:Kaplan-Meier生存曲线显示预后分数与GSE15459库中胃癌总体生存显著相关。
图7:基于已报道的19基因和7个基因标签的分析不能预测病人的整体生存(TCGA数据)。
具体实施方式:
下面结合附图和具体实施例,进一步阐明本发明,应理解这些实施例仅用于说明本发明而不用于限制本发明的范围,在阅读了本发明之后,本领域技术人员对本发明的各种等价形式的修改均落于本申请所附权利要求所限定的范围。
实施例1
运用TCGA公共数据库胃癌患者进行系统验证:
将所述预后评分系统应用于253例拥有生存数据的TCGA胃癌患者。预后评分被用来预测每个个体患者的生存概率。我们根据预后评分将患者分成两组。如果预测分值≤-2,我们定义患者具有好预后的基因标签;如果预测分值>-2,我们定义病人具有差预后的基因标签。如图5所示,前者比后者的生存时间显著延长。前者超过50%的患者在100个月后仍然存活,而后者所有患者在80个月之内均死亡。
文献中已有运用表达差异的方式显示基因或多基因组与胃癌预后的相关性。一个问题是我们的53个基因评分系统是否优于上述单基因或基因组的系统。我们首先进行了单变量Cox回归分析,显示上面所述276个来自TCGA的单基因与胃癌总体存活仅呈弱关联。然后我们使用先前报道过的胃癌基因标签来计算预测分值,包括一19个基因组(Cui J等,Gene-Expression Signatures Can Distinguish Gastric Cancer Grades and Stages.PLoS ONE.2011;6:e17819)和7-基因标签(Takeno A等,Integrative approach for differentially overexpressed genes in gastric cancer by combining large-scale gene expression profiling and network analysis.British J.Cancer.2008;99:1307-15)。如图7所示,这两个多基因组标签的评分分析不能清楚的预测TCGA数据里病人的整体生存。
实施例2
运用GSE15459公共数据库胃癌患者进行生存期验证:
应用同样的方法,我们验证了所述预后评分系统在GSE15459公共数据库的应用价值。虽然此数据库的胃癌组织基因表达值是由Affymetrix芯片技术测定的,造成了表达水平基线和刻度上的差异,由此导致预测分值绝对值的不同,但本发明评分系统仍然可以成功的预测胃癌的预后(图6)。
实施例3
预测临床胃癌病人的预后效果:
采集临床接收的胃癌病人的肿瘤组织并提取RNA,肿瘤组织可包括新鲜活检组织,术后组织,固定后的组织和石蜡包埋的组织。然后用本发明开发的试剂盒与相应的仪器定量检测所述预后评分系统53基因的表达水平。将53基因的表达水平输入本发明建立的预后评分公式:
在计算出病人的预测分值后,医生根据分数值来预测病人的预后情况(见实施例1),比如5年生存率。目前我们通过回顾性研究建立了模型,成功的在不同数据库进行了验证。并开始了前瞻性研究来进一步加以完善评分系统。
实施例4
预测临床胃癌病人对HER2/ERBB2靶向治疗(比如但不限于拉帕替尼和曲妥珠单抗)的反应:
作为预后和预测生物标志物,大约10–30%的胃癌具有HER2/ERBB2扩增或过度表达。目前仅部分胃癌HER2/ERBB2阳性病人对HER2靶向治疗有效,为减少无效或过度应用靶向药、降低医疗成本,本发明通过下如下实施来预测临床胃癌病人对HER2/ERBB2靶向药(比如但不限于拉帕替尼和曲妥珠单抗)的反应:
对临床接收的HER2/ERBB2阳性胃癌病人采集肿瘤组织并提取RNA,肿瘤组织可包括新鲜活检组织,术后组织,固定后的组织和石蜡包埋的组织。然后用本发明开发的试剂盒与相应的仪器定量检测预后评分系统53个基因的表达水平。然后将53基因的表达水平输入本发明建立的预后评分公式:
在计算出病人的预测分值后,医生根据分数值来考虑病人是否应接受HER2/ERBB2靶向治疗。对预测分值标示好预后的病人,可以建议医生酌情考虑HER2/ERBB2靶向治疗的必要性,达到避免过度用药、降低医疗成本,最后达到精准或个体化医疗的目的。
实施例5
预测临床胃癌病人对化疗药物5-FU的反应:
目前胃癌化疗总有效率大约在30%左右。为减少无效或过度用药、降低医疗成本,本发明通过以下方案实施来预测临床胃癌病人对化疗药物5-FU的反应:
对临床接收的胃癌病人采集肿瘤组织并提取RNA,肿瘤组织可包括新鲜活检组织,术后组织,固定后的组织和石蜡包埋的组织。然后用本发明开发的试剂盒与相应的仪器定量检测53基因的表达水平。将53基因的表达水平输入本发明建立的预后评分公式:
在计算出病人的预测分值后,医生根据分数值来考虑病人是否应接受5-FU化疗。对预测分值标示好预后的病人,可以建议医生酌情考虑5-FU治疗的必要性。对预测分值标示差预后的病人,可以建议医生酌情考虑增大5-FU或其它化疗药物的治疗强度。
表1.K-M绘图分析结果总结
(如果基因具有多个Affymetrix探针,结果最显著的被列在此表中)
表2.典型判别函数系数
- 还没有人留言评论。精彩留言会获得点赞!