牡丹PsDREB1基因的抗干旱和高盐启动子及其表达和应用的制作方法
本发明公开了一种牡丹psdreb1启动子derbp的克隆、载体构建及其功能分析的方法,属于植物基因工程研究领域。
背景技术:
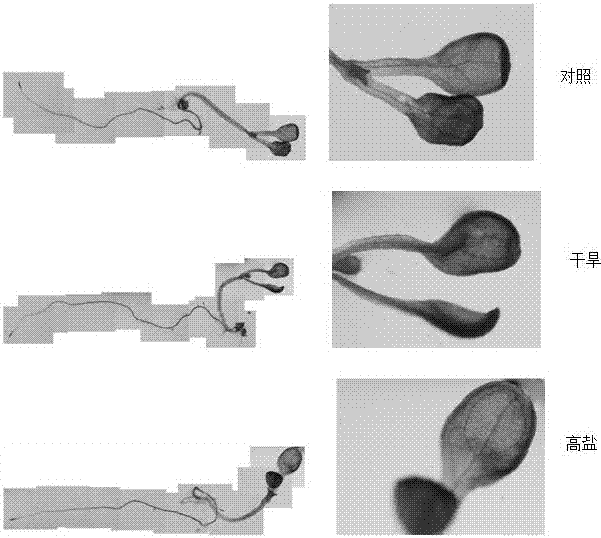
:牡丹(paeoniasuffruticosa)是我国的传统名花。随着我国园林花卉产业的发展,牡丹的美化价值和药用价值等也日益受到重视。然而,环境因素使其种植和应用具有一定的局限性。为了能够在江南和华南地区种植出雍容华贵的牡丹花,定向培育具有较强抗逆性的牡丹花品种已成为一项非常重要和紧迫的工作。前期,我们已经获得了牡丹抗逆转录因子psdreb1基因,并明确了该基因可以提高植物抗干旱和高盐的能力。但其具体的作用机制尚未明确。启动子是存在于基因上游、rna聚合酶能够识别并与之结合、从而起始基因转录的一段核苷酸序列,该序列包含了大量的调控下游基因表达的顺式作用元件。技术实现要素:针对目前牡丹psdreb1启动子方面的研究在国内仍为空白这一现状,本发明的目的在于提供一种牡丹psdreb1基因的抗干旱和高盐启动子及其表达和应用。为达到上述目的,本发明采用的实施方案是:一种牡丹psdreb1基因的抗干旱和高盐启动子,所述启动子drebp,核苷酸序列如seqidno:1所示,启动子区域含有三个abre顺式作用元件,能特异性地对aba进行应答反应,含有一个与干旱诱导相关的myb结合位点。一种采用所述的启动子构建的表达载体,启动子序列drebp构建到植物表达载体pbi121上,从而获得重组载体pbi121-drebp-gus。一种根据所述的启动子的应用,通过调控启动子drebp的表达,从而调控牡丹抗干旱和高盐的能力。本发明的有益效果是:本发明首次从牡丹中克隆dreb1转录因子基因的启动子drebp;本发明分析了drebp启动子的顺式作用元件,发现其含有典型的tatabox、caatbox以及与逆境胁迫响应相关联的结构域;通过转化拟南芥进行功能验证,发现该启动子属于组成型启动子,并受逆境如干旱、高盐等胁迫的诱导。附图说明图1为重组载体pbi121-drebp-gus构建流程图;图2为转drebp拟南芥阳性植株不同组织器官和不同发育时期的gus染色定性分析结果;其中,a部分组织器官:分别为a根、b茎、c叶、d花、e果荚、f种子;b部分5天幼苗的状态;c部分10天幼苗的状态;d部分25天幼苗的状态;图3为转drebp拟南芥阳性植株在干旱、高盐胁迫下的gus染色定性分析结果;图4a为转drebp拟南芥阳性植株根、茎、叶gus定量分析结果;图4b为转drebp拟南芥阳性植株干旱、高盐gus定量分析结果。具体实施方式启动子的克隆,对于研究植物基因表达调控和构建表达载体以及后续转化模式植物进行功能验证至关重要。对启动子进行功能分析,能够揭示植物响应逆境胁迫的作用机制。为了进一步揭示psdreb1基因提高植物抗干旱和高盐能力的分子机制,我们试图找到牡丹psdreb1相关的启动子,并进行克隆、载体构建及功能分析。启动子克隆的成功与否,与引物设计、克隆的方法等都有很大关系,如果引物设计不合理将不能成功地获得启动子序列;如果克隆的方法不当,可能获得的启动子序列不完全或者拿不到想要的启动子序列。我们主要采用取以下的步骤:(1)启动子克隆:根据牡丹psdreb1基因序列设计上游引物,采用染色体步移法克隆获得其上游启动子序列drebp,如seqidno:1所示;(2)载体构建:将上述获得的启动子序列drebp构建到植物表达载体pbi121上,从而获得重组载体pbi121-drebp-gus;(3)遗传转化:将2构建的重组载体通过蘸花法转化拟南芥,获得转基因后代;(4)功能验证:将已转入drebp-pbi121的拟南芥阳性植株进行gus染色的定性及定量分析,明确其表达模式。实施例1牡丹psdreb1启动子序列drebp的克隆于2016年1月取3年生牡丹品种‘洛阳红’的幼嫩叶片为材料,采用omega公司生产的trizol试剂盒提取总dna,以此为模板。根据psdreb1基因全长序列,设计反向引物sp1-caacagaaggggatcagcgaag(seqidno:2),sp2-ctttggttttacctcttgctcgt(seqidno:3),sp3-cggatttctcattttcccatttc(seqidno:4),使用takara公司的genomewalkingkit(codeno.6108),用上述模板进行反应。经过pcr扩增获得大小约2kb的产物。使用takara公司的minibestaragrosegeldnaextractionkitver4.0(codeno.9762)切胶回收上述pcr产物,命名为p1。使用takara公司的dnaligationkit(codeno.6022)中的solutioni,将p1产物与pmd19-tvector(codeno.6013)连接,热转化至e.colicompetentcellsjm109(codeno.9052)中,涂布平板,37℃过夜培养。挑选阳性菌落植菌,提取阳性克隆质粒。使用引物f1-ctgctgatttggtatttgga(seqidno:5),r1-ttcctggatctctattctga(seqidno:6)对获得的质粒进行测序。测序结果如seqidno:1所示,将测序获得的序列与已知的psdreb1进行blast比对,结果表明获得的序列是已知psdreb1基因的上游启动子序列,该序列全长为2245bp,命名为drebp。启动子序列分析:使用在线分析软件plantcare和place程序分析所获得的启动子drebp序列的顺式作用元件和调控元件,在线分析网址分别为:www.plantcare.com/encyclopedia和www.dna.affrc.go.jp/place。分析结果显示:drebp启动子序列含有65个与转录起始相关的tatabox结构,22个与组织特异性相关的caatbox结构,15个与环境胁迫相关的元件如光胁迫响应元件atct-motif、box4、boxi、g-box、gag-motif、gt1-motif、i-box、sp1、tct-motif和chs-cma,热胁迫响应元件hse、干旱胁迫响应元件myb、真菌诱导响应元件box-w1等,另外还有一些植物激素感应元件如感应脱落酸aba的abre、感应茉莉酸甲酯meja的cgtca和tgacg结构、感应赤霉素ga的garemotif以及感应水杨酸sa的tcamotif等。具体如表1所示。表1牡丹psderb1启动子drebp序列结构分析结果元件名称数量序列功能abre3gacacgtacgt,tacgtg,gccacgtaca受干旱、aba、盐胁迫的诱导are2tggttt厌氧菌诱导的调控元件atct-motif1aatctaatcc受光的诱导box41attaat受光的诱导boxi1tttcaaa受光的诱导box-w11ttgacc受真菌的诱导caat-box22caat,caaat启动子增强区域cgtca-motif1cgtca茉莉酸甲酯响应的调控元件g-box3cacgta,gtacgtg,tacgtg光感应的调控元件gag-motif1agagagt受光的诱导gare-motif1tctgttg赤霉素响应的调控元件gt1-motif3ggttaa光感应的调控元件hse2aaaaaatttc受高温胁迫诱导i-box1aagataaggct受光的诱导mbs1caactgmyb结合位点,干旱诱导相关mbsii1aaaagttagttamyb结合位点,类黄酮合成相关skn-1motif1gtcat胚乳特异性表达元件sp13cc(g/a)ccc光感应的调控元件tata-box65taata,taaaaataa,tacaaaa…转录起始tca-element1gagaagaata水杨酸响应元件tct-motif1tcttac受光的诱导tgacg-motiv1tgacg茉莉酸甲酯响应的调控元件chs-cma2ttacttaa,gcaattcc受光的诱导实施例2重组载体pbi121-drebp-gus的构建insertdna(目的dna)的制备:以上述获得的启动子drebp质粒为模板,采用引物对f2(seqidno:7)/r2(seqidno:8)使用takara公司的primestar®®hs(premix)(codeno.r040a)进行pcr扩增,扩增体系为:drebp质粒(50倍稀释液)1μl,f2(20pmol/μl)0.5μl,r2(20pmol/μl)0.5μl,primestarhs(premix)25μl,加dh2o至50μl;反应条件:98℃10s,55℃15s,72℃90s,共30个循环。取5μlpcr产物进行琼脂糖凝胶电泳,并使用takaraminibestagarosegeldnaextractionkitver.4.0(codeno.9762)切胶回收目的片段,命名为insertdna。vectordna(载体dna)的制备:以质粒pbi121为模板,采用限制性内切酶hindiii和xbai进行双酶切,酶切反应体系为:pbi121质粒dna30μl,10×buffer5μl,hindiii(10u/μl)1.5μl,xbair2(10u/μl)1.5μl,加dh2o至50μl;反应条件:37℃16h。取5μlpcr产物进行琼脂糖凝胶电泳,并使用takaraminibestagarosegeldnaextractionkitver.4.0(codeno.9762)切胶回收载体片段,命名为vectordna。重组质粒的构建:使用in-fusion®®hdcloningkit(clontechcodeno.639633),将insertdna和vectordna连接,连接反应体系如下:insertdna(约50ng/μl)1μl,vectordna(约80ng/μl)1μl,5×in-fusionhdenzymepremix2μl,加dh2o至10μl。反应条件:50℃,15min。将连接产物取1μl热转化至e.colicompetentcelljm109(codeno.9052)中,涂布平板,37℃过夜培养。挑取阳性克隆,提取质粒并使用引物vp1(seqidno:9)/vp2(seqidno:10)对质粒进行测序,测序获得的结果经blast比对,确认为成功构建的重组质粒,并命名为pbi121-drebp-gus。构建流程如图1所示。实施例3遗传转化将上述构建好的重组载体质粒采用电转法转化农杆菌gv3101,然后采用clough和bent(1998)报道的蘸花法转入拟南芥。采集拟南芥转化苗的f1代种子,在含有40mg/l卡那霉素(kan)的1/2ms培养基上进行初步筛选培养,筛选出的阳性植株再使用引物对vp1/vp2进行pcr鉴定,鉴定为阳性的植株继续种植从而收集f2代种子。延续上述方法,继续获得纯合的f3代种子。实施例4功能分析无菌播种:将获得的拟南芥转化苗的f3代种子经1%的naclo表面消毒10min,无菌水清洗6遍后,播种在1/2ms+蔗糖20g/l+琼脂粉7.5g/l(ph5.8)的培养基上,先22℃暗培养48h后再进行光培养。不同组织器官的表达模式分析:将光培养40天的拟南芥转化苗,使用中科瑞泰(北京)生物科技有限公司的gus染色试剂盒(货号:rtu4032)进行染色。具体染色方法如下:gus染色液配制:将1mlx-gluc染色液加入到50mlgus染色缓冲液中,即配成gus染色溶液。染色步骤:1)将准备好的拟南芥转化苗浸泡在gus染色液中,于25℃染色过夜;2)染过色的植株依次转入25%、50%、75%、95%的乙醇中,分别在25℃摇床上120rpm振荡脱色20min;3)显微镜下观察拟南芥转化苗根、茎、叶、花、果荚、种子等不同组织器官的gus染色结果。染色结果显示:转入drebp的拟南芥植株,在根、茎、叶、花、果荚中都有gus基因的表达,而在种子里无表达,其中根部表达量最高,其次是叶片和茎干,如图2a所示。定量分析以25d转化苗作为材料,使用上海江莱生物科技有限公司的植物guselisa试剂盒(cat.no.jl13690)进行测定,具体方法参照说明书,gus定量结果与染色结果吻合,即根部表达最强,其次是叶片和茎干,如图4a所示。不同发育时期的表达模式分析:将无菌培养5d、10d和25d的拟南芥转化苗,使用上述方法分别进行gus染色并在显微镜下观察染色结果,结果显示:拟南芥转化苗在不同发育时期均有不同的表达模式,5d发芽期,只在主根的成熟区进行表达,其他部位不表达,如图2b所示;10d幼苗期,在主根的成熟区和伸长区均有表达,叶片也有表达,茎干部分有轻微的染色,说明在茎部位表达很少,如图2c所示;25d花苞期,gus染色布满整个植株,根、茎、叶、花苞都有gus表达,其中又以根部表达最强、其次是叶片和花苞、茎干表达较其他部位稍弱,如图2d所示。不同逆境胁迫下的表达模式分析:采集无菌播种25d的拟南芥转化苗,分别将其根系浸泡在含有10%的聚乙二醇(peg6000)、300μm的nacl溶液中,以模拟干旱和高盐胁迫,以蒸馏水作为对照,分别处理2h。取处理后的植株分别进行gus染色定性分析和gus定量分析。gus染色结果显示:干旱和高盐胁迫处理后,gus染色程度明显高于对照,如图3所示;gus定量分析结果显示:与gus染色结果相吻合,即经干旱和高盐处理后gus表达量高于对照,且高盐胁迫下的表达量最高,如图4b所示。sequencelisting<110>浙江省萧山棉麻研究所<120>牡丹psdreb1基因的抗干旱和高盐启动子及其表达和应用<160>10<170>patentinversion3.5<210>1<211>2245<212>dna<213>paeoniasuffruticosa<400>1cgcgacgcatgcgcatccttttactcatttaccccaaacaaactaaccctagttaactga60actccgtttcactttcaccgacgttaaccatggttagttaaacccagcccttgatttgcg120taatgaaactctgttccccactcgagaatcgagggaatccaaacgtctctatctctcgct180acaattttttttctttctgggactggcgccgcaattgaagaagagaagcttttgtcctca240gtggccacttgattatggctctcaaccatggtttcgttaaaaactaaccatagttatcag300ttgatcattcagcaaactttcaaccaacgcactttcccacttaggcttattttaatcggg360ttttggtattggttttaataaataaaaaaggtctaatttagatgagattgtgtggataag420aatgatgccgttaaagattagtattatccattgtccaaccactactataaccatttcact480gtgatgatgtaacgtgcttatgtacggcgcacccatgtcaatcttaatcagtaaattaaa540aactatttttttttataaaatttaatctatttggttttttacccggtagttgttatttac600attatttcgctttcatccaattattcatttaaggtcctgtttgggatagcttctgcttat660gcttataaacagtaaaagtagagaagcagaagccagtgcagcttaaaactgctgatttgg720tatttggaaactgtttttggaagtgctttttagtaatatgttaaccagccttaaaaataa780attttgtataaaaactgcttatttttctgctttttgagaaaaagctatcccttaggtgct840tctgccaaaaaacactttttggcttttaagcactttttagtcaagtatgccaaacaccgt900aaaagcacttatatatgaaaataagtgcttataagcagcttataagcttagccaaacagg960gcctaagtctgactaacttttatattttcacacctacattctatggttatttattattat1020cgttcgagtttttaattcattgaattaaatttgaatttactagaatcttaaatcgagatg1080atgagtgcaccttacatctacacacctataatccatggctcttaactttctatgccagac1140tcttcaccactatagttcgagtctcgatctcatcgaactaagtttcaattcactcaaatc1200ctaaatcgagatgttgagtgcaccttaactttctaagtcagacttttcaccactagactt1260atcttagtagtttaatttaatatatttgtttacagtcatatttgtcgatacgatttttca1320accatctaatttattttttttataaaatcattgtccaaccctaattcataatactcaacc1380ttaattcaaaatactaaggtaagagaaaggacaaaaatgtattaaattctaaaggtaata1440tactcaatcagtcaatcacttacaaacaagtatgggtagtgtgaaggtaatatactcaat1500ccgtcaatcacttacaaacaagtatgggtagtgtgttatctccacccaaatgttggtggt1560caataaatatgaagttcatgtacgtgtcgctatagtattggatttgatttatgagtttag1620tttttaaaaaaaataaagaattaggtatctttcaagttgacaaaggttaaaaaaaaagaa1680gtgataactaagtaaaaagaaatatgaaaatagtgagaagaaagagagtaataattaagt1740aaagaaatgggaaaatgagaaatccgtgcgcctggaagtttatataagaggatgggtggg1800ggagggtacgagcaagaggtaaaaccaaagtgaggccacgaaaaaagagtgatacagttt1860ctctcactttctgcaactgcttttgatcatcatcttcttcttctcccccatccccacttc1920gctgatccccttctgttgtctcctgtctttcaattaattgttttgggtattgtttttttg1980tgatttttgtttgatatttgtgggttaaaaaaagcaattccagaaaggtgtttttgaaag2040tcaggtgattcagaatagagatccaggaaatatttttctttaatttgtagttttatcttt2100aattcaactacgagggcttctttttgtgtcctcaggttgtatttttgttttgtgcagata2160aagaaagtttttttcttttcctttttagcttgtatttccgtagatctttctctggttttt2220catatatatatttagtcgaggaagc2245<210>2<211>21<212>dna<213>artificialsequence<220><223>sp1<400>2caacagaaggggatcagcgaa21<210>3<211>23<212>dna<213>artificialsequence<220><223>sp2<400>3ctttggttttacctcttgctcgt23<210>4<211>23<212>dna<213>artificialsequence<220><223>sp3<400>4cggatttctcattttcccatttc23<210>5<211>20<212>dna<213>artificialsequence<220><223>f1<400>5ctgctgatttggtatttgga20<210>6<211>20<212>dna<213>artificialsequence<220><223>r1<400>6ttcctggatctctattctga20<210>7<211>54<212>dna<213>artificialsequence<220><223>f2<400>7tgattacgccaagcttgcatgcctgcaggtcccccgcgacgcatgcgcatcctt54<210>8<211>48<212>dna<213>artificialsequence<220><223>r2<400>8ccggggatcctctagagtccccgcttcctcgactaaatatatatatga48<210>9<211>20<212>dna<213>artificialsequence<220><223>vp1<400>9cgcaattaatgtgagttagc20<210>10<211>20<212>dna<213>artificialsequence<220><223>vp2<400>10ccaacgctgatcaattccac20当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1