一种基于猪ERV转座子插入多态性研发新型分子标记的方法与流程
本发明涉及分子生物学和生物信息学领域,具体涉及一种基于猪erv转座子插入多态性研发新型分子标记的方法。
背景技术:
dna分子标记技术是一种以基因组dna多态性为基础的新型遗传标记技术,是遗传变异在dna水平上的直接反映。与其他几种遗传标记——形态学标记、生物化学标记、细胞学标记相比,dna分子标记具有的优越性有:大多数分子标记为共显性,对隐性的性状的选择十分便利;基因组变异极其丰富,分子标记的数量几乎是无限的;在生物发育的不同阶段,不同组织的dna都可用于标记分析;分子标记揭示来自dna的变异;表现为中性,不影响目标性状的表达,与不良性状无连锁;检测手段简单、迅速。
随着分子生物学技术的发展,现在dna分子标记技术已有数十种,广泛应用于遗传育种、基因组作图、基因定位、物种亲缘关系鉴别、基因库构建、基因克隆等方面。第一类:是以分子杂交为核心的分子标记,包括rflp、dna指纹技术等,这类分子标记被称为第一代分子标记;第二类:是以pcr为核心的分子标记,包括随机扩增多态性rapd、简单序列重复ssr、扩增片段长度多态性aflp、序列标签位点sts等,为第二代分子标记;第三类:是一些新型的分子标记,如:snp标记、表达序列标签est标记等,也以pcr技术为基础,为第三代分子标记。还存在其他类型的dna标记,但他们是上述dna标记类型的组合。例如,cap为酶切扩增多态性dna,其中pcr产物在扩增后要进行酶切消化。其中的引物对可以根据重复元件和aflp引物或不同重复元件的序列来设计。
转座子在生物基因组中广泛存在,是一类在基因组上可以自由跳跃(复制或移位)的移动dna序列,首先由mc.clintock于20世纪40年代在玉米染色体中发现,以后又陆续在细菌、真菌及昆虫等各种生物中发现。由于转座子能在其寄主的基因组中复制、移动,因此,像其它的突变源一样,转座子不仅促进了物种间基因组的分化,而且还产生了物种内丰富的遗传多样性,对物种、品种、亚种、品系形成发挥了重要作用。利用转座子插入多态性(tip)建立新型分子标记技术在遗传进化研究中为我们理解高等生物基因组的进化提供了新视角,同时,tip技术也成为生物多样性、遗传进化研究和分子育种的重要工具。人类转座子解析研究最为深入和广泛。研究表明:人类基因组中的活性转座子主要为l1、alu和sva,这些活性转座子在不同人群中产生了大量的多态,witherspoon等学者(witherspoon,d.j.etal.genomeresearch23,1170-81(2013))仅对196个来源于不同地区和群体的人基因组分析时,一次就获得了5799个alu插入位点,其中多态位点高达2524个。目前发现在人类基因组中至少有8350个tip座位(hancks,d.c.etal.currentopinioningenetics&development22,191-203(2012))。鼠的tip比人类更丰富,因为鼠的世代间隔短,且基因组中活性转座子更多,主要包括erv(鼠内源性病毒)、l1、b1/sine和b2/sine。
猪基因组学研究目前面临的主要挑战是虽然获得了大量数量性状变异的qtl,但缺乏足够密度的分子标记,难以进行基因精细定位,无法有效开展分子标记辅助选择。而tip具有理想分子标记的诸多优点:如多态性高、共显性、基因组分布广泛、检测手段简单快速、重复性好和开发成本低等。并且相较其他标记的来源,erv的插入对猪基因组结构、基因表达的影响大于snp,微卫星等,因此基于猪erv转座子插入多态研发的分子标记必将大大推动分子育种的进程和猪功能基因学的研究;同时,tip分子标记应用于猪群体遗传学研究也将加深人类对猪生物多样性、起源进化和品种种质特性形成的理解和认识。
技术实现要素:
本发明是针对现有分子标记的不足,基于猪erv转座子在猪基因组上具有含量丰富,分布广泛,并且在不同的个体间存在插入多态性的特点提供一种研发分子标记的方法。
本发明通过以下技术方案实现:
一种基于猪erv转座子的新型分子标记的获取方式,包括以下步骤:
(1)利用erv的ltr序列作为查询序列,在生物学公用数据库中对猪公开基因组进行检索,寻找插入位点;
(2)针对寻找到的erv的每个插入位点,分别向上游和下游延伸300-500个核苷酸序列,然后分别下载每条序列;
(3)将步骤(2)获得的序列去除冗余并进行多序列比对,每条插入位点序列保留300-500个erv插入位点侧翼区核苷酸序列和ltr序列;
(4)根据步骤(3)获得的序列信息设计检测引物,作为待验证分子标记引物,其中使一侧引物位于erv插入位点基因组侧翼序列中,另一侧引物位于erv的ltr序列中;
(5)利用步骤(4)所得的待验证分子标记引物pcr扩增不同品种,或同一品种不同个体基因组样品,选取能清晰扩出条带且有多态性的引物组合,获得分子标记。
其中,erv家族的成员序列有差异,可以用任何一个作为查询序列,获得的位点都属于erv转座子的插入位点。所述erv的ltr核苷酸序列包括但不限于seqidno.1、seqidno.2、seqidno.3、seqidno.4、seqidno.5、seqidno.6、seqidno.7、seqidno.8、seqidno.9、seqidno.10、seqidno.11、seqidno.12、seqidno.13、seqidno.14、seqidno.15、seqidno.16、seqidno.17、seqidno.18、seqidno.19或seqidno.20。
上述方法的一个实例是:
(1)利用erv的ltr核苷酸序列seqidno.1作为查询序列,在ensembl数据和ucsc数据库对猪公开的考基因组进行比对(blat)搜索,寻找erv在基因组上的插入位点,选取比对上的长度大于95%且相似度大于98%的比对位点,获得53个插入位点;
(2)根据53个插入位点分别向上游和下游延伸300核苷酸,然后分别下载每条序列;共53条序列;
(3)将获得的53条序列利用cd-hit程序按照相似性95%进行合并,去除冗余,并进行多序列比对,每条插入位点序列保留300-500个erv插入位点侧翼区核苷酸序列和ltr序列,最终获得37条序列。
(4)截取erv插入位点序列作为设计引物的模板,根据序列设计检测引物,其中使一侧引物位于erv插入位点的基因组侧翼序列中,另一侧引物位于erv的ltr序列中。
(5)利用待验证分子标记引物pcr扩增不同品种,或同一品种不同个体基因组样品,选取能清晰扩出条带且有多态性的引物组合,获得分子标记,共获得23个标记,对应的引物名称,引物组合,引物序列如表1所示。
其中,步骤(5)中pcr扩增的操作可以是包括以下步骤:
1)人工合成上述37对待验证分子标记引物;
2)提取不同品种的池dna以及不同品种的不同个体基因组;
3)以不同品种的池dna以及不同品种的不同个体基因组为模板进行pcr扩增;
4)对pcr扩增结果进行琼脂糖凝胶电泳检测。
本发明主要通过获取erv插入位点序列,设计各位点检测引物,pcr扩增,琼脂糖凝胶电泳等步骤获得转座子插入情况,据此建立基于转座子插入多态性检测方法。为猪的品系鉴定,育种中的标记辅助选择提供有用的分子标记。
附图说明
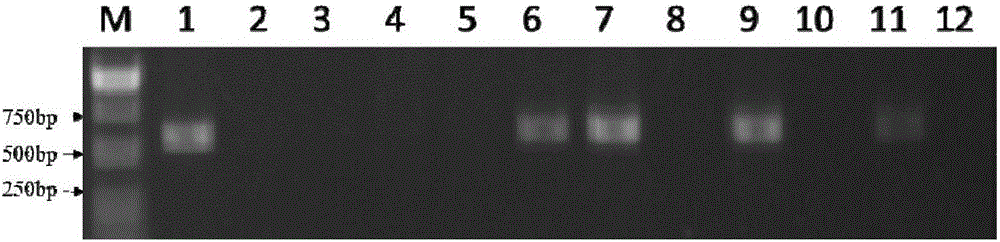
图1标记erv-tip-5在11个不同品种中的扩增结果。marker为宝生物的dl5000,从左到右1-12分别是洛克猪、长白猪、大白猪、梅山猪、姜曲海猪、苏姜猪、藏猪、野猪、巴马香猪、皖南花猪、烟台黑猪混合基因组及空白对照,混合基因组均为5个个体的等质量混合基因组。
图2标记erv-tip-7个不同品种中的扩增结果。marker为宝生物的dl5000,从左到右1-12分别是洛克猪、长白猪、大白猪、梅山猪、姜曲海猪、苏姜猪、藏猪、野猪、巴马香猪、皖南花猪、烟台黑猪混合基因组及空白对照,混合基因组均为5个个体的等质量混合基因组。
图3标记erv-tip-6在9个不同品种的不同个体中的检测结果。图3中marker为诺唯赞的dl2000plus,从左到右分别是野猪、大白、杜洛克、梅山、二花脸、藏猪、荣昌、巴马香猪、苏姜猪,其中藏猪3头,其余品种均为5头。
图4标记erv-tip-14在9个不同品种的不同个体中的检测结果。图4中marker为宝生物的dl5000,从左到右分别是野猪、大白、杜洛克、梅山、二花脸、藏猪、荣昌、巴马香猪、苏姜猪,其中藏猪3头,其余品种均为5头。
具体实施方式
在进一步描述本发明具体实施方式之前,应理解,本发明的保护范围不局限于下述特定的具体实施方案;还应当理解,本发明实施例中使用的术语是为了描述特定的具体实施方案,而不是为了限制本发明的保护范围。
一、参考基因组中erv插入位点序列获取,具体过程如下:
1、erv转座子在基因组中插入位点检索:利用利用erv的ltr序列作为查询序列,作为查询序列,在ensembl数据库(http://www.ensembl.org/)和ucsc数据库(http://genome.ucsc.edu/)中利用blat程序对猪的参考基因组(sgcgsscrofa10.2/susscr3)进行比对搜索,运行参数使用数据库中各自的默认设置,寻找line1在基因组上的插入位点。
2、在两个数据库中每个erv插入位点序列获取:在ensembl数据库中检索插入位点后,选取比对上的长度大于95%且相似度大于98%的比对位点,分别向上游和下游延伸300-500个核苷酸序列,然后分别下载每条序列,同理在ucsc数据库中进行再次检索,剔除与ensembl数据库中一致的位点,然后向每个插入位点的5’端上游延伸300核苷酸,下载每条序列,最后合并两个数据库下载的序列。
3、将获得的序列利用cd-hit-est程序(http://weizhongli-lab.org/cdhit_suite/cgi-bin/index.cgi?cmd=cd-hit-est)将sequenceidentitycut-off参数设置为0.95,其他参数使用默认值进行去冗余,并进行多序列比对,每条插入位点序列保留300-500个erv插入位点侧翼区核苷酸序列和ltr序列。
二、引物的设计
利用获得的序列,使用batchprimer3(http://batchprimer3.bioinformatics.ucdavis.edu/cgi-bin/batchprimer3/batchprimer3.cgi)在线软件进行引物设计,使其中使一侧引物位于erv插入位点基因组侧翼序列中,另一侧引物位于erv的ltr序列中。产物长度设置为400-600bp,其他参数使用软件默认设置。设计好的各引物用olige7进行分析评价,然后由北京六合华大基因科技股份有限公司合成。
三、分子标记在11个不同品种中检测
1、基因组的准备:
(1)用takaraminibestuniversalgenomicdnaextractionkitver.5.0提取杜洛克猪、长白猪、大白猪、梅山猪、姜曲海猪、苏姜猪、藏猪、野猪、巴马香猪、皖南花猪、烟台黑猪各5个个体基因组,主要步骤如下:
a)取2-25mg的耳组织,剪碎,加入180微升的buffergl、20微升的proteinasek和10微升rnasea(10mg/ml),于56℃水浴温浴至组织完全裂解。
b)向裂解液中加入200微升buffergb和200微升100%乙醇,充分吸打混匀。
c)将spincolumn安置于collectiontube上,溶液移至spincolumn中,12000rpm离心2分钟,弃滤液。
d)将500微升的bufferwa加入至spincolumn中,12000rpm离心1分钟,弃滤液。
e)将700微升的bufferwb加入至spincolumn中,12000rpm离心1分钟,弃滤液。
f)重复操作步骤6.
g)将spincolumn安置于collectiontube上,12000rpm离心2分钟。
h)将spincolumn安置于新的1.5ml的离心管上,在spincolumn膜的中央处加入50-200微升的灭菌水或elutionbuffer,室温静置5分钟。
i)12000rpm离心2分钟洗脱dna.
(2)调整各个基因组浓度成为40ng/μl,取每个个体基因组50μl混合为品种基因组,共11种混合基因组,以此作为模板进行pcr扩增。
2、pcr扩增
设计以下pcr反应体系:
(1)在灭菌pcr管中配制20μl反应体系
(2)将pcr管置于pcr仪中,进行如下反应程序
注:1:南京诺唯赞生物科技有限公司
3、琼脂糖凝胶检测
(1)称取1.5g琼脂糖放入三角烧瓶中,加入100ml1*tae电泳缓冲液,置于微波炉中使之完全溶解.
(2)待凝胶冷却至50℃-60℃时,将琼脂糖胶倒入插有胶梳的模具中,厚度一般在4-5mm,并检查有无气泡存在。室温放置30min待凝胶完全凝固后,小心拔出胶梳,浸没于加有1*tae电泳缓冲液的电泳槽中。
(3)用微量上样枪吸取6μlpcr扩增产物加入凝胶孔,同时点4μldna标准分子量maker作为参照。
(4)连接电泳槽与电泳仪,用120v的恒压电泳,根据指示剂的迁移位置,判断电泳情况。
(5)电泳结束后将凝胶置于盛有溴化乙锭溶液的塑料盒中,染色10分钟,将凝胶拿出放到紫外灯下观察并拍照记录。
四、分子标记在9个不同品种中检测
1、基因组的准备:
(1)选取9个品种(野猪、大白、杜洛克、梅山、二花脸、荣昌、巴马香猪、苏姜猪、藏猪),藏猪三头个体,其余每种5头个体,采集耳组织,用takaraminibestuniversalgenomicdnaextractionkitver.5.0提取各自基因组,主要步骤如下:
a)取2-25mg的耳组织,剪碎,加入180微升的buffergl、20微升的proteinasek和10微升rnasea(10mg/ml),56℃水浴至组织完全裂解。b)向裂解液中加入200微升buffergb和200微升100%乙醇,充分混匀。c)将spincolumn安置于collectiontube上,溶液移至spincolumn中,12000rpm离心2分钟,弃滤液。d)向spincolumn中加入500微升bufferwa,12000rpm离心1分钟,弃滤液。e)将700微升的bufferwb加入至spincolumn中,12000rpm离心1分钟,弃滤液。f)重复操作步骤6。g)将spincolumn安置于collectiontube上,12000rpm离心2分钟。h)将spincolumn安置于新的1.5ml的离心管上,在spincolumn膜的中央处加入50-200微升的灭菌水或elutionbuffer,室温静置5分钟。i)12000rpm离心2分钟洗脱dna.
(2)调整各个基因组浓度成为40ng/l,以此作为模板进行pcr扩增。
2、pcr扩增
设计以下pcr反应体系:
(1)在灭菌pcr管中配制20μl反应体系
(2)将pcr管置于pcr仪中,进行如下反应程序
注:1:南京诺唯赞生物科技有限公司
3、琼脂糖凝胶检测
(1)制作1.5%的琼脂糖凝胶:称取1.5g琼脂糖放入三角烧瓶中,加入100ml1×tae溶液,放于微波炉中加热使之完全溶解,取出冷却至约60℃时,将琼脂糖胶倒入插有胶梳的模具中,并检查有无气泡存在。室温放置30min待凝胶完全凝固后,小心拔出胶梳,浸没于加有1×tae电泳缓冲液的电泳槽中。
(2)pcr产物电泳:用移液器吸取6μlpcr扩增产物依次加入到凝胶孔中,同时点4μldnamaker作为参照,120v恒压电泳,待指示剂到达胶块的2/3处停止电泳,然后将凝胶放到溴化乙锭溶液中,染色10分钟,将凝胶拿出放到紫外灯下观察并拍照记录。
五、结果分析
部分检测结果如图1,图2,图3,图4所示。图1,图2所示基于erv插入多态性的分子标记在不同品种间存在良好的多态性,图3,图4所示基于erv插入多态性的分子标记在不同个体间存在良好的多态。
六、结论
经过两轮实验验证,获得表1所示的23个基于erv插入多态性的分子标记,能够在品种内或品种间产生清晰且具有多态的检测结果,可以为品种鉴定提供鉴定方法,可以作为个体鉴定,分子辅助育种的良好标记,具有检测成本低,重复性好,结果清晰等优点;且为共显性,对隐性的性状的选择十分便利;在生物发育的不同阶段,不同组织的dna都可用于标记分析;不影响目标性状的表达,与不良性状无连锁;检测手段简单、迅速。
表1基于erv转座子插入多态性标记引物
sequencelisting
<110>扬州大学
<120>一种基于猪erv转座子插入多态性研发新型分子标记的方法
<130>
<160>49(序列总个数)
<170>patentinversion3.3
<210>1
<211>629
<212>dna
<213>猪(susscrofa)
<400>1
tgaaaggatgaaaatgcaacctaaccctcccagaacccaggaagttaataaaaagctcta60
aatgcccccgaattccagaccctgctggctgccagtaaataggtagaaggtcacacttcc120
tattgttccagggcctgctatcctggcctaagtaagataacaggaaatgagttgactaat180
cgcttatctggattctgtaaaactgactggcaccatagaagaattgattacacattgaca240
gccctagtgacctatctcaactgcaatctgtcactctgcccaggagcccacgcagatgcg300
gacctccggagctattttaaaatgattggtccacggagcgcgggctctcgatattttaaa360
atgattggtccacggagcgcgggctctcgatattttaaaatgattggtttgtgacgcaca420
ggctttgttgtgaaccccataaaagctgtcccgattccgcactcggggccgcagtcctct480
acccctgcgtggtgtacgactgtgggccccagcgcgcttggaataaaaatcctcttgctg540
tttgcatcaagaccgcttctcgtgagtgatttggggtgtcgcctcttccgagcccggacg720
agggggattgttcttttactggcctttca629
<210>2
<211>429
<212>dna
<213>猪(susscrofa)
<400>2
tgaaatggagttttaactaaagaacaacccccccttggctaagaaggcagctgcaagcct60
ttccagctgtgctcatcactatgccacccttcttgtaataatgtaacaactgtgtgctct120
ctgtccaagccagcagccctgctaatcaggtacccagcattgcttatcagttccgcttct180
gtaaccttgcttgctcagtttcttagggaggaacactgtctgcttggggatgggggaggt240
ccgggatgcttacatgggaaaatcaagaccttgtagtgtataaaagttactgagaacaaa300
gacctggtgctcagactctggaggtgactcctctgagcccgcaccggtgctgaataaacc360
ttgcttttccatatctccgagtgctgtttgtctccttccgttgccacggtttttgcggtt420
tccgcaaca429
<210>3
<211>347
<212>dna
<213>猪(susscrofa)
<400>3
tgtggtaggggccctcccagaacaaagggaaggcaacgtggcacaaccagcatctgtgcg60
cgcatgcgcaactctgtggcagcatgacataatcagtatccatgtgcaactttgtagcat120
agaccatccaaccagaatttacagaatgtatgttaccttataagggggacaaagggacca180
aagtgtaaaagaaaaaaaataacggagggtatataagacagcgccccgctgcgttcgggg240
ctcagcctttggatatgaatccgctgagccagtgccggcacgaataaacgctgcttcctg300
gcaaaaagcctcggtgtcccgtctctctgtatgagaatcctgcaaca347
<210>4
<211>448
<212>dna
<213>猪(susscrofa)
<400>4
tgtaagggaaaatggcccgaccaaaaggtcaaaaggagacgccccagccatgatgactaa60
gcaaaatccagccaaactgcccctaaccaatcaactacctctaaccacccctcccctaca120
catttgcttctgtaaaactcactcctacgtctattactttgccagttctcactccagtcc180
accaagcatggacccggaagaactaacacataaaagccttgtgaaacccctctttggcgc240
tcagacttcagagagctatctcctctgagcccgccggcgtaataaacctgagttctccaa300
ctctccgagtgctcgcttggtttctcgccaggtgaaagagctgctggagctggtacactg360
cggctacagagctgctggagctggtacactgtggccacagggccgctggagctgatacac420
tccggccttgggctgctggcagtcgcca448
<210>5
<211>429
<212>dna
<213>猪(susscrofa)
<400>5
tgtaggggtgtggaaaagggaatagggagcttgacaagagcgaagccatcttgctgccac60
cgtccatcttggtgaacttaaggtgactcatacgagggctctgagaaaaacaaagtcatt120
aggacttgagaaactccgggccccgattgttctaaagaaattcaaaaatagctagattgt180
acttgaggttaatcattaaagtaacaatataaggcattctgtacagctatacatctttct240
gtatcctgcatgtaactaaaggttaatgtagcatgttagacacgtaagcaagcactataa300
atgtatctccccctttctgttcggggtccatgattgggaagcgattccccgtggacccgc360
cggcgtaataaagctgttctccatcttcctcgagtgtcctctgaggtgatttctgggaat420
tctacaaca429
<210>6
<211>720
<212>dna
<213>猪(susscrofa)
<400>6
tgcctgggccttgttaacatttcttagctcaggacataggcctgggcctcgttaactttc60
cacagggccggtgtaatgtaatcagcctggcacctaagtctctggcctcagcccccttcc120
ttagaagaacaaagttagctgataagggggactgcagatgctacaaggttacccatctaa180
ttacctctttggcgctggcagaagatgccggcttgaaagtcggaaaatatggcccttact240
ataaatcattattgttaacctaaactctattcttagttaaggtttgaagcaatttgactg300
ctgctttgcttttgcgtccctgcttatgcatgtgcacccctccccacctggatgtaagtt360
tgcctttgtaaatgcggtcggtttcttagttgggagcttcacactcaagccgcttttagg420
cagtgtgtggccctggccagtaataaagactcctaatttgttttttcctggggtggagtg480
gtctctgttcttttaccgcacatttggagaccccagtgagatagagaccctccagcttct540
ggaactcgaacacctccagatcggcgaggggaaaggggtcccttccttaaggggcgtgcg600
ccctgatgatgttccagggcccgaggaaaagggcacccgaactcctctgtgctaatccca660
ggagaagagcgacttcaccccgaaattagaaaacggagtctcccgggtctggaaatcctc720
<210>7
<211>702
<212>dna
<213>猪(susscrofa)
<400>7
tgaaaggatgaaaatgcaacctgactctcccagaacccaggaagttaataagaagctcta60
aatgccctcgaattccagaccctgttccctataggtaaaagatcatactttttgctgttt120
tagggcttgctttctgctctgtacaaaactttgtggaaggggaaaaacaggcccctgagt180
atgtgcctctatgcttgaaacttcttgaaactgctcctaactgcttgtttggcttctgta240
aacctgcttgcataagataaaaagaggagaagtcaatcgcctaaccgaccccagtaagat300
cgggcgtgccacaaaatgttgaaaatcctgataaatatatcttggtgacaatatgtctcc360
cccacccagagacaggcacaaacatgtaactccagaacaacttaaaattaattggtccac420
aaagcgcgggctctcgaagttttgaattgactggtttgcgatattttaaaaatgattagt480
ttgtaaaagcgcgggctttgttgtgaaccccataaaagctgtcccgactccacactcggg540
gccgcagtcctctacccctgcgtggcgtacgactgtgggccccagcgcgctcggaataaa600
aatcctcttgctgtttgcatcaagaccgcttctcgtgagtgatttggggtgtcgcctctt660
ccgagtccggacgagagggattttcactcgactggcctttca702
<210>8
<211>408
<212>dna
<213>猪(susscrofa)
<400>8
tgtaggagaaactgtcctgataaaaatgtagaaaggagagaccctgccatgatgactaag60
caaaacccggctaactacccctaaccacccctcccccacgcatctgcttctgtaaacttg120
ccagttctcactctggtctgccaagcatggacccggaagaactagcccataaaagccttg180
tgaaacccctctttggggctcagactctggagagtgatctcctctgagcccgccgtcata240
ataaaacctgagttctcctcctctctgagtgctcgcttggtttctcgctgggtgaaagag300
ctgatctgctgtagccacagagctgctggagctggtacactatagccatagggctgtcgc360
cagagctgatatgctgtggcgcagagctgctgggcagctgccgtaaca408
<210>9
<211>430
<212>dna
<213>猪(susscrofa)
<400>9
tgtaggagaaaatgtcccaataaaatgtagaaaggagagaccccagccatgatgactaag60
caaagtctagccaactgccccaaccagtcctcccccatgcatctgcttctgtaaatttgt120
ttccgcatctactaccttgcctgacgtcactccagtccaactagccaagcttggacccgg180
aagacgtagcccataaaagccttgtgaaacccttcttccgggctcagactctggagagcg240
atctcctctgagcccgccggcgtaataaacctgagttctccaactctccgagtgctcgct300
tggtttctcgccgggtaaaagagctgatccactacggccacagagctactggagctggta360
cgctacagccacggggctgtcgccagagctgatacgctgcagcgcagggctgctgggtat420
cgccgtaaca430
<210>10
<211>268
<212>dna
<213>猪(susscrofa)
<400>10
tggagcaaaccctaactctaagcccctattggacaaggtccgatgtaggtgctgggcaag60
gccaatagggagaaggactgatatccagaccccatgctggtacccctccctgtctttaag120
agataacacctccttagaaaattagaaggcgggaactcttcctctccctctcagtgcctg180
tgctgggagctgtctgccttcctgtaatacagacttgttcagttgccgcctgggtgttcg240
caaagttcattcttcagtttcatgaaca300
<210>11
<211>308
<212>dna
<213>猪(susscrofa)
<400>11
tgtaagcgagcaaccgttagtgagcgacctttagtggtcctgctcagccactggtcagca60
ttgcagtgggcccttcccccaagcaagtgacagttagtgagtgaccgttggcccctcccc120
ctccccttcctctgtataaaaggagcctgaattcggactgaggcaagatggttttttgag180
acactagtctgccatcttctcggtctgctagctttccgattaaagtcgttattccttgcc240
ccaacaactcgtctccgaatttattggcctgtcgtgcggcgagcagagcgaacttgggtt300
cggtatca308
<210>12
<211>359
<212>dna
<213>猪(susscrofa)
<400>12
tgtggaaagtaacataatgtcttttctatcaaaggaaatcttggttactccattttgctc60
cggtttcggctgaaacgcccctgcgacccccttcctctttacctcttttcccagcaacaa120
tttgtttcaaattagccaaccgggaagaatgtgcgccctgacccgaccaatgggaagggg180
acaggtanatcacctgcgttagggataaataggggagggtcctttgttcggcgcgcgcgc240
tttttggagtacccgcgcccttctgcagaagtaaagagccttgtcgagatttctccttgt300
ccacgtgtctcactttccgacactgacgacccgagccagagttatcttaatttccaaca359
<210>13
<211>318
<212>dna
<213>猪(susscrofa)
<400>13
tgaagggaagcccaggtcacccaggaggagatattgagaataacttctgccaactccagg60
caggaacagttcctgacggcgccggacacacaatcatgcccctgtcaactgaccacactc120
gctaaactgtaactgatagaaataacgttgacaaaaaataggctttctaaactcccctat180
atactctagaagctccaagaccctggagcaacagtctggcctcaaagttgactgatgcct240
ctccttgctgattaaaggcaatctggttctgttgagaccgtctcatttctttctgtaacc300
acctctcttttcctgaca318
<210>14
<211>252
<212>dna
<213>猪(susscrofa)
<400>14
tgtatgatagggagttcccgtcgtggcgcagtggttaacaaatccgactaggaaccatga60
ggttgcgggtttggtccctgcccttgctcagtgggttaacgatccggcgttgccgtgagc120
tgtggtgtaggttgcagacgcggctcggatcccgcgttgctgtggctctggcgtaggccg180
gtggctacagctccgattcgacccctagcctgggaacctccatatgccgcgggagcggcc240
caagaaatagca152
<210>15
<211>281
<212>dna
<213>猪(susscrofa)
<400>15
tggaaagatattccatgttcatggattggaaaaatcaatattgtgaaaatggacatacta60
cccaaagcaatctacagattcaatgcaatccctatcaagttacccatgacatttttcaca120
gaactagaacaaacaatccaaatatttatatggaacgaccaaagacccacaattgccaaa180
gcaatcctgagaaacaaaaaccaagcaggaggcataactctcccagacttcaggcaatat240
tacaaagccacagccatcaaaactgtgtggtactggtacaa281
<210>16
<211>352
<212>dna
<213>猪(susscrofa)
<400>16
tgttggagaccactggactgctgaactctgtagaaagcaaacttagtgtgtggcaaagcc60
agaaaagcatgtatgccaaaagagttctcaagtacagggtctccttcaaagacaagataa120
aggacttatgcattaaccacaatattctataaatagctgcactgtgtcctaagggtatgc180
accagtaacttccttgctaatagtttcctctgatatcattaagagatagctttgtcatag240
cctatgcctgttcttgcccactaacctacatacatcaccttccttaacaaaagtcaggtg300
gtctaaagactagggcctttgttctgctgaatggagtgcttcctggtgcgca352
<210>17
<211>184
<212>dna
<213>猪(susscrofa)
<400>17
tgtaccacatagcgaactagggtgatacctgtgtgattgaaatctcacaaatgacacaaa60
ataagaggcttctggcttcctacttgctaaatgtatgccactcagtaaaagaacactagc120
cccaatgatgttttgcacaggaaggcaacctttgcctttttccacgtaggcctcttgtat180
aaca240
<210>18
<211>498
<212>dna
<213>猪(susscrofa)
<400>18
tgttgcaagacatgtttccttgaaaactccatcttgtccttccttactttatcccatctt60
ccttcttggaaaaacagtcacagtgctggtaaatgacttaagcttaagagcaaagaccta120
aaggcataaatgacttaagcttaagtactgttatgtgcctagaggtcagagtaagagctt180
aaccctttaccacctaagtggctttctaaatagtaaaagaacttatatcatccacccata240
gccactcctcacttaatctcacaataaaagcagtgtggtttcttgaggcagtgctcttgg300
tcccggagatctggagttccccggcgctcttggtccctgagaccttgagtcccgcactct360
tggtccctgagaccttgagtcccccggctcccacctttacttaagataaatgtctctgtg420
tcttgttttatgttaactatttttccttaagttccacagcacccgttcttccccatcctg480
ctgagctggccccggcaa498
<210>19
<211>104
<212>dna
<213>猪(susscrofa)
<400>19
tcaccagcaaagcccattaacaattccccagaaataaagatagaatctgggtaataaaat60
taagtctgacctttttcttttcctttgtgcttcttctagtgaca104
<210>20
<211>119
<212>dna
<213>猪(susscrofa)
<400>20
tggactctaggatagacatttacaaccagccacctatctatactacagcaggatagatag60
aaataacagcaggcacaaggcaaataactggtacctgtcttctgtgaacttgttaaaca119
<210>21
<211>22
<212>dna
<213>猪(susscrofa)
<400>21
ctggttcacatgtgcaagattt22
<210>22
<211>20
<212>dna
<213>猪(susscrofa)
<400>22
gcttcctcggacatttctca20
<210>23
<211>20
<212>dna
<213>猪(susscrofa)
<400>23
tggggttctttcaattttcc20
<210>24
<211>20
<212>dna
<213>猪(susscrofa)
<400>24
tgcaagcactgtctcgtcat20
<210>25
<211>20
<212>dna
<213>猪(susscrofa)
<400>25
ggagtgatgtcaggcaaggt20
<210>26
<211>20
<212>dna
<213>猪(susscrofa)
<400>26
gggttacctggaggatcagc20
<210>27
<211>20
<212>dna
<213>人工序列
<400>27
caaaaggggaaagaaagcaa20
<210>28
<211>20
<212>dna
<213>人工序列
<400>28
tatagcctaggccggcatct20
<210>29
<211>20
<212>dna
<213>人工序列
<400>29
gttcccattgtggctcagtt20
<210>30
<211>20
<212>dna
<213>人工序列
<400>30
ttcttcagtcccatcctgct20
<210>31
<211>20
<212>dna
<213>人工序列
<400>31
tggaggtgtccttgatctcc20
<210>32
<211>20
<212>dna
<213>人工序列
<400>32
aggtgcagcgctagaaagac20
<210>33
<211>20
<212>dna
<213>人工序列
<400>33
gttttcctggctgcattttt20
<210>34
<211>20
<212>dna
<213>人工序列
<400>34
ggctcttcatctgcctgttc20
<210>35
<211>20
<212>dna
<213>人工序列
<400>35
ggtatgaggcccacttctca20
<210>36
<211>21
<212>dna
<213>人工序列
<400>36
ttgcacagagattgtggactt21
<210>37
<211>21
<212>dna
<213>人工序列
<400>37
gacgcattaaggggattatca21
<210>38
<211>20
<212>dna
<213>人工序列
<400>38
gaggatatggcttcctccaa20
<210>39
<211>20
<212>dna
<213>人工序列
<400>39
caccatagttcgtggcaatg20
<210>40
<211>20
<212>dna
<213>人工序列
<400>40
cattgaaacagcccttctga20
<210>41
<211>20
<212>dna
<213>人工序列
<400>41
aggaaacctacagccacctg20
<210>42
<211>20
<212>dna
<213>人工序列
<400>42
ccaggctgaatcatttgacc20
<210>43
<211>20
<212>dna
<213>人工序列
<400>43
ctgctgcacctgtctctctg20
<210>44
<211>20
<212>dna
<213>人工序列
<400>44
cctgggcagagtgacagatt20
<210>45
<211>20
<212>dna
<213>人工序列
<400>45
ggcagagtgacagattgcag20
<210>46
<211>21
<212>dna
<213>人工序列
<400>46
tggtgccagtcagttttacag21
<210>47
<211>20
<212>dna
<213>人工序列
<400>47
ttcctctatggtgccagtca20
<210>48
<211>20
<212>dna
<213>人工序列
<400>48
ctcggcagagtgacagattg20
<210>49
<211>20
<212>dna
<213>人工序列
<400>49
aatccggataagcgattggt20
- 还没有人留言评论。精彩留言会获得点赞!