利用酵素进行恒温核酸杂交的方法与流程
本发明涉及一种核酸杂交方法,特别涉及一种恒温核酸杂交方法,该方法利用酵素,即重组酶,以取代现有的以温度使核酸双链分离与接合的操作步骤。
背景技术:
核酸杂交技术(nucleicacidhybridization)是利用具有互补核苷酸序列的两条单链核苷酸分子片段,在适当条件下,通过氢键结合,形成dna-dna,dna-rna或rna-rna杂交的双链(doublestrand)分子。分子杂交可在dna-dna,dna-rna或rna-rna的二条单链(singlestrand)之间进行。由于dna一般都以双链形式存在,因此在进行核酸分子杂交时,需先将双链dna(doublestranddna)分子解离成为单链(singlestrand),这一过程称为变性(denature),现有技术是通过加热或提高ph值来实现。
核酸分子杂交可按作用环境大致分为固相杂交和液相杂交两种类型。固相杂交是将参与反应的一条核酸链先固定在固体支持物上,另一条待测反应核酸链游离在溶液中。固体支持物可以是硝酸纤维素滤膜、尼龙膜、乳胶颗粒、磁珠和微孔板等。液相杂交所参与反应的两条核酸链都游离在溶液中。常用的固相杂交类型有:菌落原位杂交、斑点杂交、southern印迹杂交、northern印迹杂交、组织原位杂交(insituhybridization)等。现有的固相膜的核酸分子杂交方法基本步骤如下:1.待测核酸(dna)变性反应:在低盐浓度(0.1molssc溶液含15mmolnacl、1.5mmol柠檬酸三钠,ph7.0)溶液中下加热100℃10分钟使双股dna解离成单股dna;2.变性后之单股dna迅速冰浴保存,避免单股dna黏回成双股dna;3.将单股dna固定于硝酸纤维素膜上:硝酸纤维素滤膜(孔径0.45μm)先在蒸馏水中充分浸泡,再用6xssc溶液浸泡30分钟至2小时,之后将dna样品转移或加至硝酸纤维素膜上,先在室温干燥4小时,然后再以80℃干燥2小时;4.预杂交反应:将已预热至60℃的预杂交溶液(6xssc溶液、0.5%sds、100μg/ml鲑鱼精子dna)加至硝酸纤维素滤膜上以68℃反应3至12小时;5.核酸杂交反应:杂交反应在60~70℃中进行,所需时间视探针和检测靶dna的性质而定,一般约需4至20小时。杂交反应溶液的组成是6xssc、0.01mol核酸探针,0.5%sds、100μg/ml变性的鲑鱼精子dna;6.膜清洗步骤:以2xssc和0.5%sds溶液,室温下漂洗5分钟,再置换成2xssc和0.1%sds溶液,室温下漂洗15分钟(轻轻摇动),再以0.1xssc和0.5%sds溶液于低于核酸探针tm值12℃以上的温度下漂洗2小时;7.呈色反应:依探针标记物进行呈色反应。
综上所述,现有的核酸杂交反应,需要通过控制温度来使核酸双股分离(变性)与接合(复性),反应需较多的操作步骤,并需耗费长达数小时以上的反应时间,对于许多急于知晓检验结果的试验,无法于短时间内完成。因此,有必要开发一种可简化核酸杂交反应操作步骤与时间的方法,其具有大幅减低所需时间与仪器成本的特色与效果。
技术实现要素:
本发明提供了一种利用酵素来进行核酸杂交反应的方法,以改善现有进行核酸杂交反应时,需先以物理法加热至高温(约95℃)使待测核酸变性(denature)成单股线性结构,再加入核酸探针后,再降低温度(约65℃)以进行碱基配对(basepairing)黏合作用(annealing)等多重步骤且需依赖仪器以维持特定温度及耗时的缺失。
本发明的主要反应组成份包含重组酶(recombinase)以及杂交反应溶液。杂交反应方法主要包括下列步骤:混合待测核酸、核酸探针(核酸探针与待测核酸的某一段核苷酸序列互补,长度为15~100核苷酸,最佳为20~60核苷酸)、重组酶及杂交反应溶液;于低恒温环境下,利用重组酶将核苷酸探针黏合至待测核酸的互补序列上,形成核酸探针-待测核酸复合体,其中所利用的重组酶具有使同源性核酸碱基配对(homologouspairing)与核酸链置换(strandexchange)的功能,可进行dna-dna或rna-dna杂交反应。
重组酶可由原核生物(prokaryotes)或真核生物(eukaryotes)中分离或纯化而来,可为野生型(wildtype)(shibatat.etal.,methodinenzymology,100:197(1983))或变异型(mutanttypes)(如reca803:madirajum.etal.,proc.natl.acad.sci.usa,85:6592(1988))的reca441(kawashimah.etal.,mol.gen.genet.,193:288(1984),etc.)、uvsx蛋白(yoncsakit.ctal.,eur.j.biochem.,148:127(1985))、bacillussuhilisreca蛋白(lovettc.m.etal.,j.biol.chem.,260:3305(1985))、ustilagoreel蛋白(kmiece.b.etal.,cell,29:367(1982))、热稳定性细菌(如thermusaquaticus或thermusthermophilus)reca-like蛋白(angove.etal.,j.bacteriol.,176:1405(1994);kator.etal.,j.biochem.,114:926(1993))、或酵母菌、鼠、人来源的reca-like蛋白(shinoharaa.etal.,naturegenetics,4:239(1993)),以及其他如rad51、rad51b、rad51c、rad51d、rad51e、xrcc2或dmc1等。
本发明的重组酶可应用于液相溶液或固相基质(如塑料、纸、玻璃、磁珠、尼龙模(nylon)或硝化纤维膜(nitrocellulose)等),将核酸探针或待测核酸固定在固相基质表面上进行核酸杂交反应。进行核酸杂交反应的方法包括原位杂交技术(insituhybridization)、southern杂交(southernblotting)、northern杂交(northernblotting)及微列阵杂交分析(microarrayhybridization)等。进行核酸杂交所需反应时间为5~60分钟,最佳反应时间为10~60分钟;所需的反应温度范围为30~50℃,最佳反应温度范围为30~45℃。
本发明的特色与效果包括无须改变温度,可于低恒温环境下进行核酸杂交反应,降低仪器需求与适用材质范围,亦可将待测核酸与探针同时混合并整合成一步骤反应,且实施本发明的时间仅需30分钟至1小时,大幅降低时间需求,更能同时进行多探针杂合反应。
【附图说明】
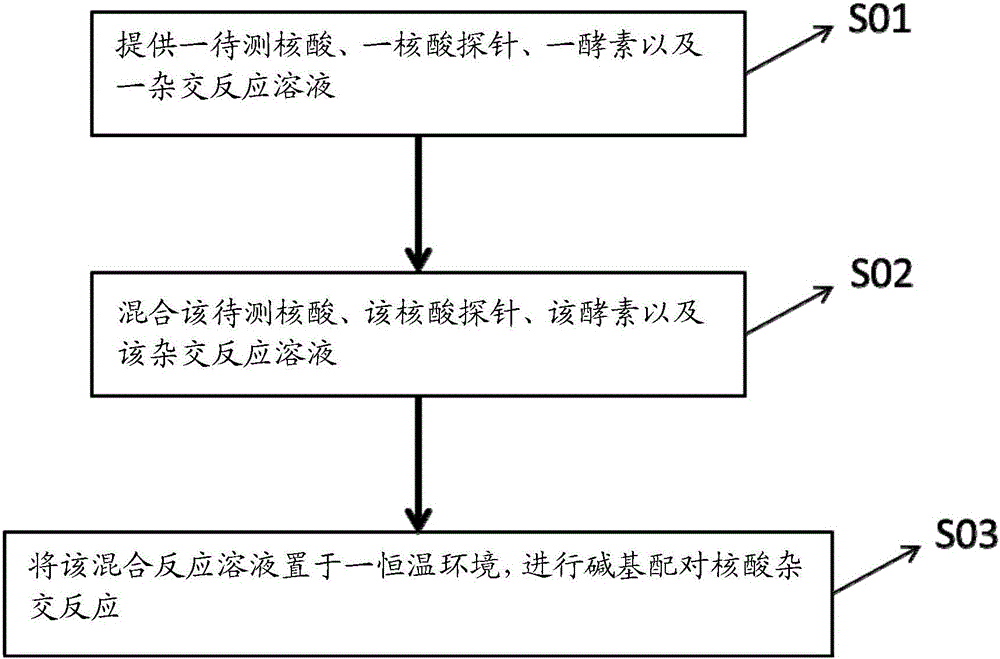
图1为本发明利用酵素进行恒温核酸杂交方法的流程示意图;
图2为本发明实施例1的待测核酸分布示意图;
图3a~图3d为本发明实施例1利用酵素进行dna-dna杂交反应的结果图;
图4为本发明实施例2利用酵素进行核酸杂交反应用以鉴别hpv-52病毒型别的结果图;
图5为本发明实施例2利用酵素进行核酸杂交反应用以鉴别hpv-33与58病毒型别的结果图;
图6为本发明实施例2利用酵素进行核酸杂交反应用以鉴别hpv-56病毒型别的结果图。
【符号说明】
s01~s03步骤
0196微孔盘
02点样的结核杆菌核酸
03点样的大肠杆菌核酸
04点样的真核a549细胞株核酸
【具体实施方式】
为充分了解本发明的目的、特征及功效,现通过下述具体实施例,并配合所附的附图,对本发明做一详细说明,说明如后:
请参阅图1。本发明是利用一酵素,在低恒温环境下,待测核酸与核酸探针的碱基配对杂交作用可一步骤完成,减少现有杂交方法所需的步骤与时间。本发明的核酸杂交方法,包括步骤s01,提供一待测核酸、一核酸探针、一酵素(即重组酶)以及一杂交反应溶液;步骤s02,再将前述提供的待测核酸、核酸探针、重组酶以及杂交反应溶液充分混合;以及步骤s03,将混合均匀的反应溶液置于低恒温环境下,30~45℃,进行碱基配对核酸杂交反应。进行碱基配对核酸杂交反应的方法包括原位杂交技术(insituhybridization)、southern印迹杂交、northern印迹杂交及微列阵杂交分析(microarrayhybridization)等。前述的待测核酸为单股(singlestrand)或双股(doublestrand)的dna或rna。前述的核酸探针可以是dna或寡核苷酸探针,但不限于这两者。探针标记方法包括放射性同位素(如:32p和35s)以及非放射性探针标记(金属如hg、荧光物质如fitc、半抗原如毛地黄素(dig)、生物素(biotin)、酶类如辣根过氧化物酶(hrp)、半乳糖苷酶或碱性磷酸酶(akp))。
酵素(重组酶)的制备
从国际数据库(如ncbi)搜寻酵素数据,以及与酵素相对应的dna序列;设计该dna序列专用的引物以pcr的方式对该段序列进行扩增;使用电泳分析确认pcr产物的长度,将正确位置的pcr产物从电泳胶上切下,之后以胶提取的方式将目标pcr产物进行纯化,并将纯化后的pcr产物进行dna测序以确定pcr产物的正确性,测序结果如核苷酸及氨基酸序列表的seqidno:44所记载。
将纯化后的pcr产物以ta-克隆的方式与一个合适的dna载体制备成一第一质粒,第一质粒具有抵抗氨苄青霉素及大量复制的特性,再将该质粒转染e.colidh5α感受态细胞;转染后,e.coli具有独立大量复该质粒的能力,因此可获得大量的目标dna序列;再以质粒提取的方式将包含目标dna序列的第一质粒提取出来;接着以5’ndei限制性内切酶和3’bamhi限制性内切酶将目标dna从第一质粒上切下来。同时将第二个载体(pet14b)以相同的限制性内切酶处理。由于目标dna序列与第二个载体(pet14b)具有相同的酶切位点,因此在连接酶的帮助下组合成第二质粒;重组后的第二质粒再次进行第二次转染作用,此次选取的细胞系为bl21-de3细胞系。含有第二质粒的细胞系,可通过iptg诱导t7聚合酶表达,而t7聚合酶可大量转录目标基因,也将会促使目标基因的蛋白大量产生。
将转染作用后的bl21-de3细胞进行大量培养,之后进行iptg诱导,可大量表达目标蛋白,之后可以保留蛋白活性的裂解液进行细胞裂解处理,以保留蛋白活性;提取蛋白后,进行纯化。在本发明所使用的第二质粒上同时标记六个组氨酸(6xhistag)及sumo蛋白(smallubiquitin-likemodifierproteins),通过辨识6xhistag的特异性抗体接合磁珠(antibody-coatedbeads)进行纯化;再使用特定的洗脱缓冲液,将抗原抗体的专一性键结中断,接着以离心或磁力的方式去除磁珠,仅保留目标蛋白。此目标蛋白的氨基酸序列如核苷酸及氨基酸序列表的seqidno:45所记载。
以下提供两则利用前述目标蛋白亦即酵素(重组酶)进行恒温dna-dna杂交反应的实施例。
实施例1
实施例1的待测dna核酸为结核杆菌(t.b.)dna(100ng)、大肠杆菌dna(100ng)与真核(人类)a549细胞株(a549cellline)dna(100ng)。核酸探针分别为结核杆菌专一性探针(100nmole)、大肠杆菌专一性探针(100nmole)、甘油醛3-磷酸脱氢酶基因(glyceraldehyde3-phosphatedehydrogenase;gapdh)专一性探针(100nmole),各核酸探针皆标记有生物素。
请参阅图2,图2是待测核酸分布示意图。将各待测dna固定于pc材质的96微孔盘01,第一列为点样的结核杆菌核酸02,第二列为点样的大肠杆菌核酸03,第三列为点样的真核a549细胞株核酸04。在96微孔盘01的不同微孔中,分别加入核酸探针(100nmole)、重组酶(120ng/ul)及杂交反应溶液50ul(tris(ph7.9)50mm、potassiumacetate100mm、dtt2mm、peg20k5%、atp3mm、phosphocreatine50mm、creatinekinase100ng/ul、mgac140mm)混合均匀。将含有混合溶液的96微孔盘01置于40℃下,进行杂交反应30分钟。
去除未反应的上清液,再以500μl的清洗溶液(w)(0.1倍ssc+0.1%sds)清洗3次,将未配对的核酸探针与反应混合液洗去。加入呈色溶液200μl(含5%已偶联抗生物素蛋白(avidin)的乳胶(latex))。去除未反应的上清液,再以500μl的清洗溶液(w)(0.1倍ssc+0.1%sds)清洗3次,将未结合的呈色溶液洗去。反应结果呈现于图3。
请参阅图3,图3a的微孔盘为阴性对照组,即不加入任何核酸探针,图3b的微孔盘为加入结核杆菌专一性探针(100nmole),图3c的微孔盘为加入大肠杆菌专一性探针(100nmole),图3d的微孔盘为加入类gapdh基因专一性探针(100nmole)。参阅图3b至图3d可知,由于重组酶能够结合核酸探针并解开双股dna,使核酸探针能于待测dna上滑行,当遇到相对应的核苷酸序列时即可产生碱基配对作用而附着于待测dna上,再经过核酸探针上的标志物进行呈色反应,即可进行结果判读。因此,通过本发明方法的确能够鉴别配对有核酸探针的待测dna,进而确认待测dna是否具有所欲检测的标的序列。另一方面,于图3a中,由于未含有核酸探针,故重组酶不会产生非专一性碱基配对作用而有任何核酸探针附着于待测dna上。因此,通过本发明方法的确具备可行性,不会有假阳性的情况发生。
实施例2
实施例2是针对36型基因型人类乳突瘤病毒(hpvtype6,11,16,18,26,31,32,33,35,39,42,43,44,45,51,52,53,54,55,56,58,59,61,62,66,67,68,69,70,72,74,mm4,mm7,mm8,cp8061,cp8304)的专一性基因片段进行检测,子宫颈抹片检体中的hpvdna以生物素标记的my11/gp6+混合引物进行核酸扩增反应,再与测试片上hpv基因型特定寡核醣核酸探针进行杂交反应,最后经由与streptavidin接合乳胶作用产生呈色反应来判别人类乳突瘤病毒基因型。实施例2所采用的引物与核酸探针的序列如核苷酸及氨基酸序列表所记载。序列表中,seqidno:1是hpv-6核酸探针;seqidno:2是hpv-11核酸探针;seqidno:3是hpv-16(a)核酸探针;seqidno:4是hpv-18核酸探针;seqidno:5是hpv-26核酸探针;seqidno:6是hpv-31核酸探针;seqidno:7是hpv-32核酸探针;seqidno:8是hpv-33核酸探针;seqidno:9是hpv-35核酸探针;seqidno:10是hpv-39核酸探针;seqidno:11是hpv-42核酸探针;seqidno:12是hpv-43核酸探针;seqidno:13是hpv-44核酸探针;seqidno:14是hpv-45核酸探针;seqidno:15是hpv-51核酸探针;seqidno:16是hpv-52核酸探针;seqidno:17是hpv-53核酸探针;seqidno:18是hpv-54核酸探针;seqidno:19是hpv-55核酸探针;seqidno:20是hpv-56核酸探针;seqidno:21是hpv-58核酸探针;seqidno:22是hpv-59核酸探针;seqidno:23是hpv-61核酸探针;seqidno:24是hpv-62核酸探针;seqidno:25是hpv-66核酸探针;seqidno:26是hpv-67核酸探针;seqidno:27是hpv-68核酸探针;seqidno:28是hpv-69核酸探针;seqidno:29是hpv-70核酸探针;seqidno:30是hpv-72核酸探针;seqidno:31是hpv-74核酸探针;seqidno:32是hpv-mm4(82)核酸探针;seqidno:33是hpv-mm7(83)核酸探针;seqidno:34是hpv-mm8(84)核酸探针;seqidno:35是hpv-cp8061(71)核酸探针;seqidno:36是hpv-cp8304(81)核酸探针;seqidno:37是hpv-pani核酸探针;seqidno:38是hpv-panii核酸探针;seqidno:39是ic:gapdh核酸探针2;seqidno:40是my11f-引物;seqidno:41是生物素-hpv-r1引物;seqidno:42是gapdh-f2引物;seqidno:43是生物素-gapdh-r2引物。
将36型基因型人类乳突瘤病毒的专一性核酸探针与dnaloadingdye以10:1混匀后以点片机点样于nc膜(sartoriusstedimbiotech,unisartcn140unbacked,catno:1un14ar100270nt)上,各个探针分布矩阵如下表1所示:
表1
每一测试片加入250ul杂交反应溶液(含重组酶20iu、杂交反应试剂a(2mguscninpbs)与杂交反应试剂b(5%bsa/0.05%nan3/pbs,ph7.4-7.6,以1:1比例混合,须于使用前新鲜配置)及25ulgapdh-核酸扩增产物与25ulhpv-核酸扩增产物。本实施例中,待测hpv病毒核酸分别为hpv-33,52,56,58型病毒核酸。
杂交反应溶液于40℃、300rpm震荡下进行dna杂交反应30分钟。吸除杂交反应溶液,以400ul洗涤液(1%tritonx-100inpbs)于37℃、300rpm震荡下清洗芯片5分钟两次。加入300ul红色乳胶呈色液,于37℃、300rpm震荡下进行反应5分钟。吸除红色乳胶呈色液,以400ul清洗液,于37℃、300rpm震荡下清洗芯片5分钟两次。将测试片于37℃烘干10分钟后,以判读仪(bpchr-210reader)将检体类型定出。反应结果呈现于图4、图5及图6。
请参阅图4至图6。在图中4,箭头所指的点为hpv-52型的病毒核酸。在图5中,箭头所指的点分别为hpv-33与58型的病毒核酸。在图6中,箭头所指的点为hpv-56型的病毒核酸。
参阅图4至第图6可知,由于重组酶能够结合核酸探针并解开双股dna,使核酸探针能于待测dna上滑行,当遇到相对应的核苷酸序列时即可产生碱基配对作用而附着于待测dna上,再经过核酸探针上的标志物进行呈色反应,即可进行结果判读。因此,通过本发明方法的确能够鉴别配对有核酸探针的待测dna,进而确认待测dna是否具有所欲检测的标的序列。此外,相对于其他病毒型别位点中,由于未含有该型别病毒核酸,故重组酶不会产生非专一性碱基配对作用而有任何核酸探针附着于待测病毒核酸上。因此,通过本发明方法的确具备可行性,不会有假阳性的情况发生。
本发明在上文中以较佳实施例揭露,然本领域技术人员应理解的是,该实施例仅用于描述本发明,而不应解读为限制本发明的范围。应注意的是,举凡与该实施例等效的变化与置换,均应设为涵盖于本发明的范畴内。因此,本发明的保护范围当以权利要求书所界定的范围为准。
序列表
<110>芯世代生技医药股份有限公司
<120>利用酵素进行恒温核酸杂交的方法
<160>45
<170>patent-in3.5
<210>1
<211>30
<212>dna
<213>artificialsequence
<400>1
aactacatcttccacatacaccaactgcac30
<210>2
<211>30
<212>dna
<213>artificialsequence
<400>2
gtctaaatctgctacatacactaaatccgt30
<210>3
<211>30
<212>dna
<213>artificialsequence
<400>3
atgtgctgccatatctacttcagaatctgt30
<210>4
<211>30
<212>dna
<213>artificialsequence
<400>4
tacacagtctcctgtacctgggcagtcatt30
<210>5
<211>30
<212>dna
<213>artificialsequence
<400>5
ttatctgcagcatctgcatccacttgcttc30
<210>6
<211>30
<212>dna
<213>artificialsequence
<400>6
tgctgcaattgcaaacagtgatacagtaca30
<210>7
<211>30
<212>dna
<213>artificialsequence
<400>7
caactgaagacacatacaagtctatgtttg30
<210>8
<211>30
<212>dna
<213>artificialsequence
<400>8
cacacaagtaactagtgacagtacctgtaa30
<210>9
<211>30
<212>dna
<213>artificialsequence
<400>9
gtgttctgctgtgtcttctagtgatttatg30
<210>10
<211>30
<212>dna
<213>artificialsequence
<400>10
tctatagagtcttccataccttctgtctgt30
<210>11
<211>30
<212>dna
<213>artificialsequence
<400>11
catctggtgatacatatacagctgtctacc30
<210>12
<211>30
<212>dna
<213>artificialsequence
<400>12
gaccctactgtgcccagtacatatctgcaa30
<210>13
<211>30
<212>dna
<213>artificialsequence
<400>13
acacagtcccctccgtctacatattctact30
<210>14
<211>30
<212>dna
<213>artificialsequence
<400>14
aatcctgtgccaagtacatatgacgccact30
<210>15
<211>30
<212>dna
<213>artificialsequence
<400>15
gccactgctgcggtttccccaacaacacaa30
<210>16
<211>30
<212>dna
<213>artificialsequence
<400>16
ggttaaaaaggaaagcacatataaagcact30
<210>17
<211>30
<212>dna
<213>artificialsequence
<400>17
gtctacatataattcaaagcaaattgctga30
<210>18
<211>30
<212>dna
<213>artificialsequence
<400>18
atccacgcaggatagctttaataagtctat30
<210>19
<211>30
<212>dna
<213>artificialsequence
<400>19
caactcagtctccatctacaacattacagc30
<210>20
<211>30
<212>dna
<213>artificialsequence
<400>20
ctacagaacagttaagtaaatatgctgcta30
<210>21
<211>30
<212>dna
<213>artificialsequence
<400>21
cactgaagtaactaaggaaggtacgtactg30
<210>22
<211>30
<212>dna
<213>artificialsequence
<400>22
acttcttctattcctaatgtatacattatg30
<210>23
<211>30
<212>dna
<213>artificialsequence
<400>23
tacatccccccctgtatctgaatatctact30
<210>24
<211>22
<212>dna
<213>artificialsequence
<400>24
cgcctccactgctgcatactgc22
<210>25
<211>30
<212>dna
<213>artificialsequence
<400>25
tgcagctaaaagcacattaactaattgtac30
<210>26
<211>30
<212>dna
<213>artificialsequence
<400>26
tgaggaaaaatcagaggctacatatattaa30
<210>27
<211>30
<212>dna
<213>artificialsequence
<400>27
actactgaatcagctgtaccaaatatgttc30
<210>28
<211>30
<212>dna
<213>artificialsequence
<400>28
ctgcatctgccacttttaaaccattctact30
<210>29
<211>30
<212>dna
<213>artificialsequence
<400>29
gcaccgaaacggccatacctgctgcacaat30
<210>30
<211>30
<212>dna
<213>artificialsequence
<400>30
agcgtcctctgtatcagaatatacctgcct30
<210>31
<211>30
<212>dna
<213>artificialsequence
<400>31
tactacacaatcccctcctgctgctgccac30
<210>32
<211>30
<212>dna
<213>artificialsequence
<400>32
ctgttactcaatctgttgcacaaatgcgcc30
<210>33
<211>30
<212>dna
<213>artificialsequence
<400>33
ctgctacacaggctaatgaatacagcactg30
<210>34
<211>30
<212>dna
<213>artificialsequence
<400>34
ctaccaacaccgaatcagaatatacagctg30
<210>35
<211>30
<212>dna
<213>artificialsequence
<400>35
ccaaaactgttgagtctacatatagtgctg30
<210>36
<211>30
<212>dna
<213>artificialsequence
<400>36
ctacatctgctgctgcagaatacagtgcta30
<210>37
<211>32
<212>dna
<213>artificialsequence
<400>37
actgtkgtrgatacyacycgyagtacgcacag32
<210>38
<211>18
<212>dna
<213>artificialsequence
<400>38
ggcatttgytggtttgtt18
<210>39
<211>25
<212>dna
<213>artificialsequence
<400>39
caccaactgcttagcacccaayaat25
<210>40
<211>20
<212>dna
<213>artificialsequence
<400>40
cmcagggwcataayaatggg20
<210>41
<211>29
<212>dna
<213>artificialsequence
<400>41
ataaaytgyaaatcatattcytctgaaaa29
<210>42
<211>20
<212>dna
<213>artificialsequence
<400>42
cccatgttcgtcatgggtgt20
<210>43
<211>19
<212>dna
<213>artificialsequence
<400>43
catgagtccttccacgata19
<210>44
<211>1185
<212>dna
<213>artificialsequence
<400>44
catatgagcgacctgaaaagccgtctgattaaggcgagcaccagcaaactgaccgcggaa60
ctgaccgcgagcaaattcttcaacgaaaaagatgtggttcgtaccaaaatcccgatgatg120
aacattgagctgagcggcgagatcaccggtggcatgcagagcggcctgctgattctggcg180
ggtccgagcaagagcttcaagagcaactttggtctgacgatggtgagcagctacatgcgt240
caatatccggatgcggtttgcctgttctacgacagcgaatttggcatcaccccggcgtat300
ctgcgtagcatgggtgttgatccggagcgtgtgattcacaccccggttcagagcctggaa360
caactgcgtatcgatatggtgaaccagctggacgcgattgagcgtggcgaaaaagtggtt420
gtgtttatcgacagcctgggtaacctggcgagcaagaaggaaaccgaagatgcgctgaac480
gagaaggttgtgagcgacatgacccgtgcgaaaaccatgaagagcctgtttcgtatcgtt540
accccgtacttcagcaccaaaaacattccgtgcatcgcgattaaccacacctacgaaacc600
caggaaatgtttagcaagaccgtgatgggtggtggcaccggtccgatgtacagcgcggat660
accgtttttatcattggtaaacgtcaaatcaaggacggtagcgacctgcaaggttatcaa720
ttcgttctgaacgtggagaaaagccgtaccgtgaaggagaagagcaagttctttatcgac780
gttaagttcgatggcggtattgacccgtacagcggcctgctggacatggcgctggaactg840
ggctttgttgtgaaaccgaagaacggttggtatgcgcgtgagttcctggatgaggaaacc900
ggtgaaatgattcgtgaggagaagagctggcgtgcgaaggacaccaactgcaccaccttc960
tggggcccgctgtttaaacaccagccgttccgtgacgcgatcaagcgtgcgtaccaactg1020
ggtgcgatcgatagcaacgagattgttgaggcggaagtggacgaactgattaacagcaag1080
gtggagaaattcaagagcccggaaagcaaaagcaagagcgcggcggacctggaaaccgac1140
ctggaacaactgagcgacatggaggaatttaatgaataaggatcc1185
<210>45
<211>392
<212>prt
<213>artificialsequence
<400>45
hismetseraspleulysserargleuilelysalaserthrser15
lysleuthralagluleuthralaserlysphepheasnglulys30
aspvalvalargthrlysileprometmetasnilegluleuser45
glygluilethrglyglymetglnserglyleuleuileleuala60
glyproserlysserphelysserasnpheglyleuthrmetval75
sersertyrmetargglntyrproaspalavalcysleuphetyr90
aspserglupheglyilethrproalatyrleuargsermetgly105
valaspprogluargvalilehisthrprovalglnserleuglu120
glnleuargileaspmetvalasnglnleuaspalailegluarg135
glyglulysvalvalvalpheileaspserleuglyasnleuala150
serlyslysgluthrgluaspalaleuasnglulysvalvalser165
aspmetthrargalalysthrmetlysserleupheargileval180
thrprotyrpheserthrlysasnileprocysilealaileasn195
histhrtyrgluthrglnglumetpheserlysthrvalmetgly210
glyglythrglypromettyrseralaaspthrvalpheileile225
glylysargglnilelysaspglyseraspleuglnglytyrgln240
phevalleuasnvalglulysserargthrvallysglulysser255
lysphepheileaspvallyspheaspglyglyileaspprotyr270
serglyleuleuaspmetalaleugluleuglyphevalvallys285
prolysasnglytrptyralaargglupheleuaspglugluthr300
glyglumetilearggluglulyssertrpargalalysaspthr315
asncysthrthrphetrpglyproleuphelyshisglnprophe330
argaspalailelysargalatyrglnleuglyalaileaspser345
asngluilevalglualagluvalaspgluleuileasnserlys360
valglulysphelysserprogluserlysserlysseralaala375
aspleugluthraspleugluglnleuseraspmetglugluphe390
asnglu392
- 还没有人留言评论。精彩留言会获得点赞!