水稻基因组重组核酸片段RecCR012612及其检测方法与流程
本申请涉及全基因组选择育种技术。具体而言,本申请涉及利用全基因组选择育种技术选育具有稻瘟病抗性功能的重组核酸片段的水稻植株,以及由此而获得的重组核酸片段及其检测方法。
背景技术:
长期以来,传统育种的选择方法主要依赖于田间表现型的评价,根据育种家个人经验进行取舍,其最大的缺点在于耗时长,效率较低。要提高选择的效率,最理想的方法应是能够直接对基因型进行选择。随着分子生物技术的发展,分子标记为实现对基因型的直接选择提供了可能。近年来,已开始应用分子标记辅助选择方法来改良个别目标性状,能够显著的缩短育种年限。
稻瘟病是水稻最严重的病害之一,全球每年由稻瘟病引起的水稻产量损失占11%~30%,因此稻瘟病及其抗性的研究显得尤为重要。随着对稻瘟病研究的逐步深入,许多水稻抗稻瘟病基因dna片段相继被定位和克隆。其中,水稻第6染色体的pi2区间被定位和克隆了很多稻瘟病抗性基因,如pi2、piz-t、pi9、pigm、pi50,该区间包含一个稻瘟病抗性基因的基因簇(qu等,genetics.2006,172:1901-1914;wang等,phytopathology.2012,102:779-786;xiao等,molbreeding.2012,30:1715-1726;liu等,molgenetgenomics.2002,267:472-480;jiang等,rice.2012,5:29-35;zhu等,theorapplgenet.2012,124:1295-1304;deng等,theorapplgenet.2006,113:705-713)。
技术实现要素:
一方面,本申请提供了水稻基因组重组核酸片段,其包含以下重组核酸片段,或者由以下重组核酸片段组成:
-第一重组核酸片段,其选自:i)包含seqidno:1所示序列第51至75位和第422至445位核苷酸的序列或其片段或其变体或其互补序列;ii)包含seqidno:1所示序列第63至433位核苷酸的序列或其片段或其变体或其互补序列;iii)包含seqidno:1所示序列第51至75位和第422至445位核苷酸的序列或其片段或其变体或其互补序列,以及seqidno:1所示序列第8至28位、第519至541位和第630至659位核苷酸中的至少一种序列或其片段或其变体或其互补序列;或iv)包含seqidno:1所示的序列或其片段或其变体或其互补序列;和/或
-第二重组核酸片段,其选自:v)包含seqidno:2所示序列第16至42位和第259至289位核苷酸的序列或其片段或其变体或其互补序列;vi)包含seqidno:2所示序列第39至261位核苷酸的序列或其片段或其变体或其互补序列;vii)包含seqidno:2所示序列第16至42位和第259至289位核苷酸的序列或其片段或其变体或其互补序列,以及seqidno:2所示序列第4至24位和第295至324位核苷酸中的至少一种序列或其片段或其变体或其互补序列;或viii)包含seqidno:2所示的序列或其片段或其变体或其互补序列。
在一实施方案中,本申请提供的水稻基因组重组核酸片段包含选自i)、ii)、iii)、iv)中任一序列的第一重组核酸片段,或者由选自i)、ii)、iii)、iv)中任一序列的第一重组核酸片段组成。
在另一实施方案中,本申请提供的水稻基因组重组核酸片段包含选自v)、vi)、vii)、viii)中任一序列的第二重组核酸片段,或者由选自v)、vi)、vii)、viii)中任一序列的第二重组核酸片段组成。
在又一实施方案中,本申请提供的水稻基因组重组核酸片段包含选自i)、ii)、iii)、iv)中任一序列的第一重组核酸片段与选自v)、vi)、vii)、viii)中任一序列的第二重组核酸片段的组合,或者由选自i)、ii)、iii)、iv)中任一序列的第一重组核酸片段与选自v)、vi)、vii)、viii)中任一序列的第二重组核酸片段的组合组成。
此外,本申请提供了检测所述重组核酸片段的引物,其中所述引物包含:
-检测第一重组核酸片段的引物,其选自:(i)特异性识别seqidno:1所示序列第51至75位核苷酸的序列的引物,和特异性识别seqidno:1所示序列第422至445位核苷酸的序列的引物;(ii)特异性识别seqidno:1所示序列第63至433位核苷酸的序列的引物;(iii)特异性识别seqidno:1所示序列第51至75位核苷酸的序列的引物,和特异性识别seqidno:1所示序列第422至445位核苷酸的序列的引物,以及特异性识别seqidno:1所示序列第8至28位、第519至541位和第630至659位核苷酸中至少一种序列的引物;或(iv)特异性识别seqidno:1所示序列的引物;和/或,
-检测第二重组核酸片段的引物,其选自:(v)特异性识别seqidno:2所示序列第16至42位核苷酸的序列的引物,和特异性识别seqidno:2所示序列第259至289位核苷酸的序列的引物;(vi)特异性识别seqidno:2所示序列第39至261位核苷酸的序列的引物;(vii)特异性识别seqidno:2所示序列第16至42位核苷酸的序列的引物,和特异性识别seqidno:2所示序列第259至289位核苷酸的序列的引物,以及特异性识别seqidno:2所示序列第4至24位和第295至324位核苷酸中至少一种序列的引物;或(viii)特异性识别seqidno:2所示序列的引物。
在一实施方案中,用于扩增第一重组核酸片段的引物对,例如用于扩增seqidno:1所示序列的引物对为,例如,5’-gccctaaaagtatccaagttgtg-3’,和5’-atggggatagtctgcgatttctt-3’;以及用于测序第一重组核酸片段的引物,例如用于测序seqidno:1所示序列第8至28位和第51至75位核苷酸的序列的引物为,例如,5’-aaacggaaaccagtgaccc-3’;用于测序seqidno:1所示序列第422至445位、第519至541位和第630至659位核苷酸的序列的引物为,例如,5’-cacaggcagcaacaaggt-3’。
在另一实施方案中,用于扩增第二重组核酸片段的引物对,用于扩增seqidno:2所示序列的引物对为,例如5’-cacttcgtttactcggactggtt-3’,和5’-gtatgcccactgtagactttgta-3’。用于测序第二重组核酸片段的引物,例如,用于测序seqidno:2所示序列第4至24位、第16至42位、第259至289位和第295至324位核苷酸的序列的引物为,例如,5’-cttcgctctatgtcaggtc-3’。
另一方面,本申请提供了选育含有所述重组核酸片段的水稻植株的方法,其中所述重组核酸片段具有稻瘟病抗性功能,并且所述方法包括以下步骤:1)将轮回亲本水稻‘y58s’与供体水稻‘谷梅4号’进行杂交,将所得到的杂交种与轮回亲本进行回交,获得回交一代,利用正向选择标记pi31和负向选择标记pi2s67、pi2s122对其进行抗稻瘟病基因组片段的单侧同源重组片段筛选,并利用水稻全基因组育种芯片对其进行背景选择;2)选择背景回复较好的重组单株与轮回亲本再次进行回交,获得回交二代,利用正向选择标记pi31对其进行检测,选择含有抗稻瘟病基因组片段的重组单株,然后利用水稻全基因组育种芯片对其进行背景选择;3)选择背景回复好的重组单株与轮回亲本又一次进行回交,获得回交三代,利用正向选择标记pi31和负向选择标记pi2s67、pi2s122对其进行抗稻瘟病基因组片段的另一侧同源重组片段筛选,并利用水稻全基因组育种芯片对其进行背景选择;以及4)选择导入片段小,且背景回复好的重组单株,将选中的重组单株自交一次,获得自交种,利用正向选择标记pi31对其进行检测,并利用水稻全基因组育种芯片对其进行背景选择,最终获得含纯合基因组重组核酸片段且背景回复的水稻植株。
在一实施方案中,利用分子标记对重组植株进行前景选择时采用的扩增引物,包括扩增分子标记pi31的引物对,其包括正向引物:5’-atccaaacccgttgttgcac-3’,反向引物:5’-cggcaattgccacgatgata-3’;扩增分子标记pi2s67的引物对,其包括:正向引物:5’-ccgatgcaagaacaagctaa-3’,反向引物:5’-ccaccacatcaccagtgttt-3’;以及扩增分子标记pi2s122的引物对,其包括:正向引物:5’-gacttgaaaaccagtgcgtg-3’,反向引物:5’-cctacctaatggaaaggattgc-3’。
又一方面,本申请提供了检测所述重组核酸片段的方法,其包括采用本申请所提供的引物,以待测基因组为模板进行pcr反应,并分析pcr产物的步骤。具体地,所述方法以待测样品基因组dna为模板,利用前述扩增引物进行pcr扩增,然后利用前述测序引物对获得的扩增产物进行测序,若测序结果与seqidno:1和/或2所示序列或其区段一致或互补,则待测样品中含有seqidno:1/或2所示序列的同源重组核酸片段。
通过检测确定了待测样品中含有seqidno:1和/或seqidno:2所示序列或其区段的重组核酸片段,即可确定待测样品中包含具有稻瘟病抗性功能的重组核酸片段。
此外,本申请还提供了检测重组核酸片段的试剂盒,其包括如前述的引物。
进一步地,本申请还提供了筛选含有重组核酸片段的水稻植株或种子的方法,其包括检测待测水稻植株的基因组中是否含有如前所述的重组核酸片段的步骤。
在一实施方案中,采用如前所述的引物来检测待测水稻植株的基因组中是否含有如前所述的重组核酸片段。在另一实施方案中,采用如前所述的检测重组核酸片段的方法来检测待测水稻植株的基因组中是否含有如前所述的重组核酸片段。在又一实施方案中,采用如前所述的试剂盒来检测待测水稻植株的基因组中是否含有如前所述的重组核酸片段。
在又一方面,本申请提供了通过所述方法筛选得到的含有本申请公开的重组核酸片段的水稻植株或其种子。
本申请提供的基于全基因组选择育种技术的选育含有抗稻瘟病基因组重组核酸片段的水稻植株的方法,具有快速、准确、稳定的优势。仅通过五世代转育,即可仅将目标基因组片段导入受体材料,并同时实现背景的回复。本申请改良的受体材料为‘y58s’为具有广适性的光温敏不育系,其稻瘟病抗性表现欠缺。利用上述方法,可以在保留‘y58s’原有优点的情况下大幅度提高其稻瘟病抗性。同时,本申请提供的重组核酸片段与稻瘟病抗性紧密相关,可作为抗性资源应用于其他品种的培育。
附图说明
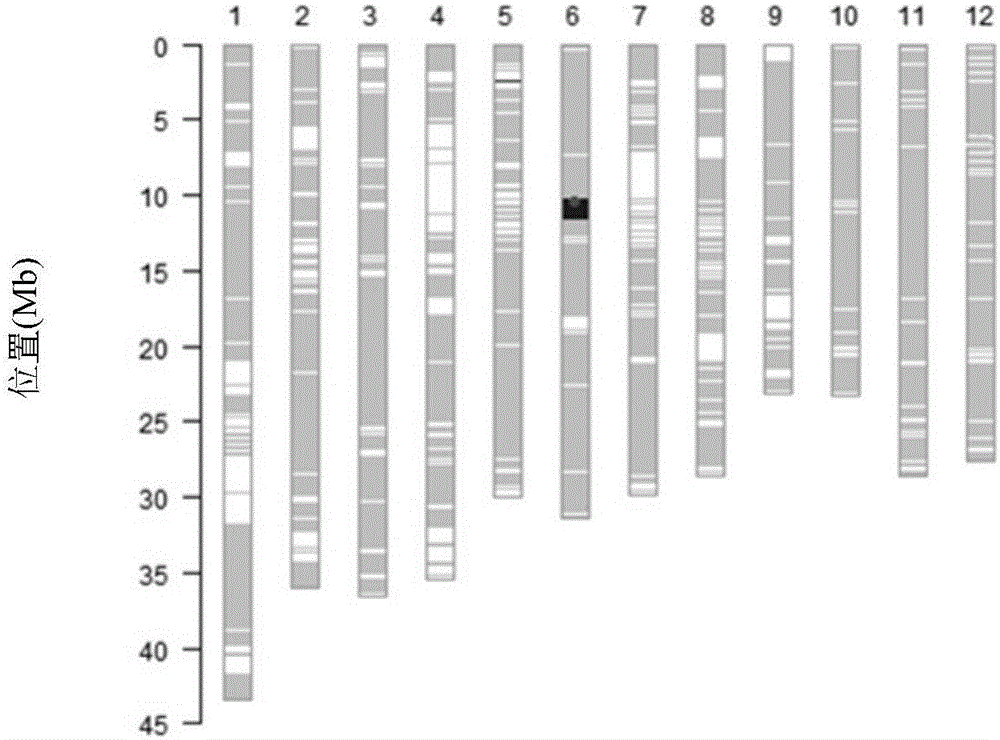
图1为本申请实施例1中cr012612水稻rice60k全基因组育种芯片检测结果;其中,横坐标数字所指示方框依次表示水稻12条染色体,纵坐标数字为水稻基因组上的物理位置[以兆碱基(mb)为单位],灰色线条代表受体亲本‘y58s’基因型,黑色线条代表供体亲本‘谷梅4号’基因型,白色线条代表两亲本基因型一致即无多态性区段。图中第6号染色体黑色圆点所示线条显示区段即为导入的抗稻瘟病基因组重组核酸片段reccr012612。
图2为本申请实施例3中cr012612稻瘟病抗性室内鉴定结果;图中所示叶片依次为:(a)稻瘟病感病品种丽江新团黑谷;(b)原品种‘y58s’;(c)改良新品系cr012612;(d)稻瘟病抗病品种谷梅4号。
具体实施方式
提供以下定义和方法用以更好地界定本申请以及在本申请实践中指导本领域普通技术人员。除非另作说明,术语按照相关领域普通技术人员的常规用法理解。
如本文所用,“核苷酸序列”包括涉及单链或双链形式的脱氧核糖核苷酸或核糖核苷酸多聚物,并且除非另有限制,核苷酸序列以5’至3’方向从左向右书写,包括具有天然核苷酸基本性质的已知类似物(例如,肽核酸),所述类似物以与天然存在的核苷酸类似的方式与单链核酸杂交。
在一些实施方案中,可以对本申请的核苷酸序列进行改变,以进行保守氨基酸替换。在某些实施方案中,可以依照单子叶密码子偏好性对本申请的核苷酸序列进行不改变氨基酸序列的替换,例如可以用单子叶植物偏好的密码子替换编码同一氨基酸序列的密码子,而不改变该核苷酸序列所编码的氨基酸序列。
具体地,本申请涉及对seqidno:1或2进一步优化所得的核苷酸序列。该方法的更多细节描述于murray等(1989)nucleicacidsres.17:477-498。优化核苷酸序列可用于提高抗稻瘟病基因组重组核酸片段在水稻中的表达。
在一些实施方案中,本申请涉及seqidno:1或2所示序列或其区段的变体。一般来讲,特定核苷酸序列的变体将与该特定核苷酸序列具有至少约70%、75%、80%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%、99.5%或99.9%或更高的序列同一性,或以上的互补序列。这样的变体序列包括一个或多个核酸残基的添加、缺失或替换,从而可以导致相应的氨基酸残基的添加、移除或替换。通过本领域内已知的序列比对程序包括杂交技术确定序列同一性。实施方案的核苷酸序列变体与本申请的序列的差异可以少至1-15个核苷酸、少至1-10个(例如6-10个),少至5个,少至4、3、2或甚至1个核苷酸。
本申请还涉及包含seqidno:1或seqidno:2所示序列中特定位点的序列或其片段或其变体或其互补序列,例如,包含seqidno:1所示序列第51至75位和第422至445位核苷酸的序列或其片段或其变体或其互补序列(包含上游同源重组区段的边界snp/indel位点);包含seqidno:1所示序列第63至433位核苷酸的序列或其片段或其变体或其互补序列(包含上游同源重组区段及其边界snp/indel位点);包含seqidno:1所示序列第51至75位和第422至445位核苷酸的序列或其片段或其变体或其互补序列,以及以下中的任一种或多种:seqidno:1所示序列第8至28位、第519至541位和第630至659位核苷酸的序列或其片段或其变体或其互补序列(包含上游同源重组区段的边界snp/indel位点,以及分别来源于受体片段和/或供体片段的snp/indel位点)。或者,包含seqidno:2所示序列第16至42位和第259至289位核苷酸的序列或其片段或其变体或其互补序列(包含下游同源重组区段的边界snp/indel位点);包含seqidno:2所示序列第39至261位核苷酸的序列或其片段或其变体或其互补序列(包含下游同源重组区段及其边界snp/indel位点);包含seqidno:2所示序列第16至42位和第259至289位核苷酸的序列或其片段或其变体或其互补序列,以及以下中的任一种或多种:seqidno:2所示序列第4至24位和第295至324位核苷酸的序列或其片段或其变体或其互补序列(包含下游同源重组区段的边界snp/indel位点,以及分别来源于供体片段和/或受体片段的snp/indel位点)。
根据包含上述特定位点的片段或区段,能够特异性地鉴定出相应的seqidno:1和/或seqidno:2所示序列。进一步地,通过鉴定出含有seqidno:1和/或seqidno:2所示序列的重组核酸片段,即可确定待测样品中包含具有稻瘟病抗性功能的重组核酸片段。
如本文所用,“水稻”是任何水稻植株并包括可以与水稻育种的所有植物品种。如本文所用,“植株”或“植物”,包括整株植物、植物细胞、植物器官、植物原生质体、植物可以从中再生的植物细胞组织培养物、植物愈伤组织、植物丛和植物或植物部分中完整的植物细胞,所述植物部分例如胚、花粉、胚珠、种子、叶、花、枝、果实、茎杆、根、根尖、花药等。
在本申请的方法可以适用于任何需要选育的水稻品种。也就是说,可以将任何缺少某种有利性状的优良品种(即综合性状较好,预计有发展前途的品种)用作轮回亲本。用另一具有该受体所缺少的有利性状的品种作为供体亲本,并且所提供的有利性状最好是显性单基因控制的。在本申请的实施方案中,采用水稻‘y58s’作为轮回亲本,采用已被证实具有良好的稻瘟病抗性的水稻‘谷梅4号’作为供体。
在本申请所提供的重组植株的选育方法中,利用分子标记对重组植株进行前景选择。前景选择的可靠性主要取决于标记与目标基因间连锁的紧密程度,为提高选择的准确率,一般同时用两侧相邻的两个标记对目标基因进行跟踪选择。
在本申请的实施方案中,采用的前景选择标记包括正向选择标记和负向选择标记。在具体实施方案中,经优化筛选使用的正向前景选择标记是与目标基因组片段紧密连锁的标记pi31,负向选择标记是位于目标片段上游的标记pi2s67,以及位于目标片段下游的标记pi2s122。
在本申请的实施方案中,利用上述前景选择标记进行同源重组的检测时,一侧或单侧同源重组的判断标准是pi31检出与‘谷梅4号’相同带型,且pi2s67或pi2s122检出与‘y58s’相同带型;两侧或双侧同源重组的判断标准是pi31检出与‘谷梅4号’相同带型,且pi2s67和pi2s122检出与‘y58s’相同带型。
在本申请中,可以使用任何一种可利用的芯片进行本申请所提供的育种方法中的背景选择。在优选的实施方案中,可以采用本申请人在中国专利申请cn102747138a中公开的水稻全基因组育种芯片rice6k,或者在pct国际申请wo/2014/121419中公开的水稻全基因组育种芯片rice60k。这两份申请文件中的全部内容整体并入本文作为参考。
以下实施例仅用于说明而非限制本申请范围的目的。若未特别指明,实施例均按照常规实验条件,如sambrook等分子克隆实验手册(sambrookj&russelldw,molecularcloning:alaboratorymanual,2001),或按照制造厂商说明书建议的条件。
本申请中所使用的水稻植株材料信息均可参见中国水稻品种及其系谱数据库(http://www.ricedata.cn/variety/index.htm)。
本申请中所提到的水稻基因组物理位置均参照水稻日本晴基因组msu/tigr注释第6.1版(http://rice.plantbiology.msu.edu/)。
实施例1选育导入抗稻瘟病基因组片段的重组植株
本实施例中使用的材料为水稻‘y58s’及水稻‘谷梅4号’。
水稻‘谷梅4号’具有良好的稻瘟病抗性,并推测可能是第6号染色体的pi2区间对该材料的稻瘟病抗性起到了关键作用。
在重组植株的选育过程中,利用分子标记对重组植株进行前景选择,对所采用的前景选择分子标记进行了筛选。参照水稻日本晴基因组msu/tigr注释第6.1版,下载第6染色体9,559,000至10,990,000dna序列。使用ssrlocator软件对上述序列中的ssr位点进行扫描。利用primerpremier3.0软件对寻找出的ssr位点设计引物,共设计引物162对。通过pcr的方法,筛选上述引物对在‘谷梅4号’及‘y58s’中的多态性,最终挑选出在两份材料中具有多态性、扩增效率高的前景选择分子标记,分别是正向选择标记pi31和负向选择标记pi2s67、pi2s122。用于pcr扩增上述分子标记的具体引物信息见表1。
表1前景选择分子标记引物信息
将水稻‘谷梅4号’中前述基因所在的基因组片段导入到水稻‘y58s’中,具体过程如下:
以‘y58s’为轮回亲本,‘谷梅4号’为供体亲本进行杂交,将所得到的杂交种与轮回亲本‘y58s’进行回交,获得bc1f1种子,育苗后利用正向选择标记pi31和负向选择标记pi2s67、pi2s122进行重组单株选择,筛选出4个在目标基因组dna片段一侧同源重组的单株,即pi31检出与‘谷梅4号’相同带型,且pi2s67或pi2s122检出与‘y58s’相同带型,并利用水稻全基因组育种芯片rice6k(cn102747138a)对其进行背景选择(yu等,plantbiotechnologyjournal.2014,12:28-37)。
在筛选出的4个单侧同源重组单株中比较芯片结果,选择背景回复最好的重组单株(此世代背景回复值超过75%),使其与轮回亲本‘y58s’再次进行回交,获得bc2f1种子,育苗后利用正向选择标记pi31对其进行检测,选择含有目标基因组片段的重组单株,即pi31检出与‘谷梅4号’相同带型,利用水稻全基因组育种芯片rice6k对其进行背景选择。
选择背景回复较好的单株(此世代背景回复值超过87.5%),使其与轮回亲本‘y58s’又一次进行回交,获得bc3f1种子,育苗后利用正向选择标记pi31和负向标记pi2s67、pi2s122对收获的种子进行目标基因组片段另一侧同源重组片段的筛选,获得3个在目标片段两侧重组的单株,即pi31检出与‘谷梅4号’相同带型,且pi2s67和pi2s122检出与‘y58s’相同带型。
利用水稻全基因组育种芯片rice60k(wo/2014/121419)对上述3个双侧交换单株进行背景和目标片段选择(chen等,molecularplant.2014,7:541-553),筛选到导入目标片段较小,且背景回复好的目标单株一个(此世代背景回复值超过93.75%)。
将选中的单株自交一次,获得bc3f2,育苗后利用正向选择标记pi31对其进行检测,选择含有目标基因组片段的单株,即pi31检出与‘谷梅4号’相同带型,利用水稻全基因组育种芯片rice60k对其进行背景选择。
最终获得目标片段纯合,且背景回复(背景回复值超过99%)的株系一个,命名为cr012612。芯片检测结果见图1。
实施例2导入稻瘟病抗性基因组片段后同源重组片段的确定
为了确定导入的稻瘟病抗性基因组片段大小,对‘y58s’导入片段的纯合单株进行了目标基因组片段两侧同源重组片段的测序。将cr012612所含的抗稻瘟病基因组重组核酸片段命名为reccr012612。
通过水稻全基因组育种芯片rice60k检测结果初步确定,reccr012612上游同源重组片段位于标记r0610187834ct和f0610202034ac之间,下游同源重组片段位于标记f0610692034ag和r0610699627ag之间。
同时,使用miseq测序技术对‘y58s’、‘谷梅4号’和cr012612三个样本进行全基因组测序。使用truseqnanodnaltkit(illumina)试剂盒进行文库建立,使用libraryquantificationkit–universal(kapabiosystems)试剂盒进行定量,使用miseqv2reagentkit(illumina)试剂盒进行测序反应。使用miseq台式测序仪(illumina)进行检测。具体步骤及方法参见各试剂盒及测序仪使用说明书。
根据前述snp芯片及miseq测序结果,将reccr012612上游同源重组片段进一步确定在第6染色体的10193487bp到10202034bp区间,下游同源重组片段定位在第6染色体的10692034bp到10698736区间。
在此基础上,参照水稻日本晴基因组msu/tigr注释第6.1版,下载对应区段dna序列。使用primerpremier5.0软件设计扩增及测序引物,设计要求为引物长22nt左右、gc含量40-60%且没有错配。
以受体亲本‘y58s’和供体亲本‘谷梅4号’为对照,对reccr012612上下游同源重组片段设计扩增引物,使用高保真酶kodfxneo(toyobo)进行扩增,使用两步法或三步法寻找最佳扩增条件,确保扩增产物在琼脂糖凝胶电泳检测中显示为单一明亮条带。筛选出1对引物用于上游同源重组片段的扩增,其反应条件为:94℃2min;98℃10sec,61℃30sec,68℃330sec,37个循环;20℃1min。筛选出1对引物用于下游同源重组片段的扩增,其反应条件为:94℃2min;98℃10sec,61℃30sec,68℃330sec,37个循环;20℃1min。
另外,以扩增产物为模板,利用sanger测序法进行测序,对上游同源重组片段共设计17条测序引物,根据实际测序效果,挑选出2条用于snp或indel位点的测序。对下游同源重组片段共设计18条测序引物,根据实际测序效果,挑选出1条用于snp或indel位点的测序。具体的扩增引物及测序引物序列见表2。
表2抗稻瘟病基因组重组核酸片段扩增及测序引物信息
reccr012612上游同源重组片段测序长度为663bp(seqidno:1)。1-63bp为受体‘y58s’的基因组区段,与供体‘谷梅4号’比较,存在1个snp,1个indel。64-432bp这一369bp区段为同源重组区段。433-663bp为供体‘谷梅4号’基因组片段,与受体‘y58s’比较,存在3个snp。
reccr012612下游同源重组片段测序长度为328bp(seqidno:2)。1-39bp为供体‘谷梅4号’的基因组区段,与受体‘y58s’比较,存在4个snp,1个indel。40-260bp这一221bp区段为同源重组区段。261-328bp为受体‘y58s’基因组片段,与供体‘谷梅4号’比较,存在9个snp,3个indel。
cr012612、‘y58s’和‘谷梅4号’snp或indel位点差异见表3。表中“位置”为:上游同源重组片段snp或indel位点差异的位置相对于seqidno:1而言,下游同源重组片段snp或indel位点差异的位置相对于seqidno:2而言。
表3cr012612、‘y58s’和‘谷梅4号’snp或indel位点差异
实施例3‘y58s’导入抗稻瘟病基因组片段后的抗性鉴定
为了鉴定抗性效果,对本申请选育的新品系cr012612、轮回亲本‘y58s’、稻瘟病抗病品种谷梅4号(作为阳性对照),以及稻瘟病感病品种丽江新团黑谷(作为阴性对照)进行室内种植,将其培养至3-4叶期后采用如下方法进行鉴定:
选取2015年来自宜昌分离的m15bb-1-1、m15bb-1-2、m15bb-2-1、m15bb-3-1、m15bb-4-1、m15bb-5-1、m15bb-6-1共7个菌株作为接种菌株。菌株采用高粱粒法-20℃保存,使用前将保存的高粱粒取出至马铃薯葡萄糖培养基(pda)平板活化(pda:去皮马铃薯200g,葡萄糖20g,琼脂粉15g,蒸馏水定容至1l),28℃光照培养5天后取直径5mm的新鲜菌丝块转接至高粱粒培养基中(高粱粒500g加入1.5l蒸馏水,煮至沸腾后滤去液体,将高粱粒捞出装入250ml三角瓶,100ml/瓶,湿热灭菌20分钟),10块/瓶,接菌2天后每天将高粱粒摇散,28℃黑暗培养至菌丝长满高粱粒。然后将高粱粒摊在无菌纱布上,盖上无菌的潮湿纱布,在25℃,rh≥95%,12h光照条件下培养4-5天至大量孢子产生,用无菌水(含0.02%吐温20)洗下孢子,调整浓度至5×105个/ml。
用上述分生孢子悬浮液喷雾接种cr012612、‘y58s’、谷梅4号及丽江新团黑谷,接种三个重复。接种后罩上透明罩子,28℃黑暗培养24h,然后16h光照培养5天后调查。
调查标准为0级(高抗,hr):没有症状;1级(抗,r):很小的褐色病斑;2级(中抗,mr):直径约为1mm的褐色病斑;3级(ms,中感):直接约为2-3mm的带圆形病斑,中央灰白色,边缘褐色;4级(感,s):长约1-3cm的椭圆形病斑,中央灰白色,边缘褐色;5级(高感,hs):长且宽的大椭圆形病斑,病斑融合成片,至叶片枯死。其中0-2级为抗病,3-5级为感病。接种结果见表4和图2。
表4接种稻瘟菌后的抗性表现
虽然,上文中已经用一般性说明及具体实施方案对本申请作了详尽的描述,但在本申请基础上,可以对之作一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本申请精神的基础上所做的这些修改或改进,均属于本申请要求保护的范围。
序列表
<110>中国种子集团有限公司
<120>水稻基因组重组核酸片段reccr012612及其检测方法
<160>15
<170>siposequencelisting1.0
<210>1
<211>663
<212>dna
<213>水稻(oryzasativa)
<220>
<221>gene
<222>(1)..(63)
<223>来源于‘y58s'基因组区段
<220>
<221>misc_recomb
<222>(64)..(432)
<223>同源重组区段
<220>
<221>gene
<222>(433)..(663)
<223>来源于‘谷梅4号'基因组区段
<400>1
ggctgctgctgctgctgccgcctcccctccctctcgatcgatcagacatcggtgggaggc60
ggtccggcgcaacacaggcagcaacaaggtcgccgctgcttgctgctactactgctcccc120
ggcgccgccgctttcgctttgttgttgacgcggtccatcatgtgcaggtcgtccttggcc180
tgccgcgccgccctgtccaccacgctcctcggcaccgggcaccagtgccgctgcgccagg240
ctcagcatgcacggcgacgtcctcagcacgaacccgcagcattgctccatcaccctgcat300
gcggagcagaggtgaataaacaatatatattcatcatccatccattcaaaatttcgaacc360
aaaataaggaaaaacactggtgatgtggtggcatggagtcgtgggtgcgtgaaagtgaat420
attacgtgtttgagggagggctttcaacgggatttcagttagattaattagggatgggtc480
actggtttccgtttcttagaacgtgtgttaggggggtgaatgtggtgttacgtgcgacca540
tgcatatagcagagtatgcgtgtgcgcgtttgccatgtaggaaaaagaaaaagaaccaac600
atttcaatccctggtatcactactagcatgaaggttgtgtttagttcacgccaaaattga660
aag663
<210>2
<211>328
<212>dna
<213>水稻(oryzasativa)
<220>
<221>gene
<222>(1)..(39)
<223>来源于‘谷梅4号'基因组区段
<220>
<221>misc_recomb
<222>(40)..(260)
<223>同源重组区段
<220>
<221>gene
<222>(261)..(328)
<223>来源于‘y58s'基因组区段
<400>2
aaattccacacgaaagaaggtgggagagagagatgaatcagaaagaggatgtggttgctc60
accaggatcccctcccccttctctgctcgccagatggagatcggcgcccctcccctgttc120
atgcgcgagatggcgcaatgagcggagacgagacgggagggaatggggagacactcggct180
actctctacctttatctcaaatttactatgttaccctttcttttatgatagactggtggc240
ccacgtgtcagcagaactcagagaaaaaaaatacaataaactcaagttgggattgtgttg300
acagtaaatttctcggagcaccgacaac328
<210>3
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>3
gccctaaaagtatccaagttgtg23
<210>4
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>4
atggggatagtctgcgatttctt23
<210>5
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>5
aaacggaaaccagtgaccc19
<210>6
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>6
cacaggcagcaacaaggt18
<210>7
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>7
cacttcgtttactcggactggtt23
<210>8
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>8
gtatgcccactgtagactttgta23
<210>9
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>9
cttcgctctatgtcaggtc19
<210>10
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>10
atccaaacccgttgttgcac20
<210>11
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>11
cggcaattgccacgatgata20
<210>12
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>12
ccgatgcaagaacaagctaa20
<210>13
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>13
ccaccacatcaccagtgttt20
<210>14
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>14
gacttgaaaaccagtgcgtg20
<210>15
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>15
cctacctaatggaaaggattgc22
- 还没有人留言评论。精彩留言会获得点赞!