用于从福尔马林固定的石蜡包埋的生物样品中表征抗药性细菌的方法和组合物与流程
所描述的方法和组合物一般涉及使用聚合酶链反应(pcr)扩增来自福尔马林固定的石蜡包埋(ffpe)活组织检查样品的痕量dna序列。特别地,该方法和材料促进pcr扩增人活组织检查ffpe保存样品内的痕量细菌dna序列。更具体地,该方法和组合物适于表征人胃活组织检查ffpe样品中存在的多药抗性幽门螺杆菌(helicobacterpylori)(幽门螺杆菌(h.pylori))。
背景技术:
幽门螺杆菌是主要在胃中发现的重要人病原体。幽门螺杆菌在胃炎、消化性溃疡和胃癌中起重要作用。幽门螺杆菌的抗药性和多药抗性菌株越来越常见,并且用于治疗幽门螺杆菌的一线治疗通常涉及三联抗生素治疗,包括同时施用质子泵抑制剂(ppi)和另外两种抗生素,通常是克拉霉素和甲硝唑或阿莫西林。然而,这种治疗仅在它靶向针对其的临床分离物不是克拉霉素抗性或甲硝唑或青霉素类药物如阿莫西林抗性时才有效。可以使用其它抗生素,但是在每种情况下,了解折磨患者的幽门螺杆菌菌株是否倾向于对任何特定抗生素抗性以提供有效的治疗处理是至关重要的。已开发了用于检测幽门螺杆菌存在的许多诊断方法,包括尿素呼吸测试、快速尿素酶测试、以及用于血清和粪便样品的抗幽门螺杆菌抗体测试、以及广泛范围的免疫组织学染色方法。在大多数情况下,此类方法需要使用培养的分离物或最近获得的临床样品。由于幽门螺杆菌在生长和运输方面的特殊要求,幽门螺杆菌的培养和抗生素敏感性测试通常不以常规实践执行。这很困难且并非总是成功的。此外,不存在允许确定检测到的任何幽门螺杆菌的抗药性谱的良好方法。能够检测幽门螺旋杆菌同时确定检测到的细菌的抗药性谱的稳健且廉价的方法代表了超过现有技术的显著改进。
历史上,活组织检查组织,特别是胃活组织检查组织保存在福尔马林固定的石蜡包埋样品(ffpe)“块”中。此类块不需要特殊处理或贮存,并且代表了在临床组织学实验室中保存人体活组织检查材料的常见方法。ffpe经过长时间段保持包埋组织的超微结构关系,样品不经受外源污染,并且ffpe促进组织学染色的处理,并且顺应许多基于抗体的分析。不幸的是,允许固定细胞物质(主要是通过交联大分子中的含氮基团)的福尔马林固定降低了许多核酸方法的功效,所述核酸方法例如ffpe活组织检查样品中特定基因序列的聚合酶链反应(pcr)扩增。用于pcr扩增的dna依赖性dna聚合酶既不能穿过也不能精确复制化学交联的或含有许多经修饰的含氮碱基的dna序列。
幸运的是,大多数ffpe样品中的交联水平并不完全排除核酸分析。本文描述的方法和组合物改善了超过现有方法来自活组织检查ffpe样品的此类交联的和在其他方面损坏的痕量dna的pcr扩增功效。通常,来自ffpe保存的样品的dna的pcr扩增靶向分析活组织检查中存在的人dna序列,并且存在相对大量的可以扩增的靶dna。这很重要,因为通过福尔马林的化学交联是随机发生的,并且在pcr扩增反应内具有任何给定靶dna序列的足够高的拷贝数,所以很可能所需靶dna序列的多个拷贝中的至少一个或一些组合可用于扩增,并且因此顺应基于pcr的分析。商业试剂盒可用于直接分析从ffpe活组织检查样品中回收的人dna,例如illumina的truseqcustomampliconlowinputkit(illumina)。然而,当应用于较不丰富的dna序列,例如在ffpe保存的组织内以低拷贝数存在的细菌病原体的那些时,情况是非常不同的。在其中靶向用于pcr扩增的dna序列处于极低拷贝数的情况下,包含靶dna的每个和每一个拷贝的交联或加合物的可能性是主要问题。为了最大化成功pcr扩增的机会,有必要将通过pcr扩增产生的dna片段(扩增子)的大小限制在分析所需的最小值,仅使用产生此类最小扩增子的最有利的引物序列,并且产生尽可能多的来自靶dna的单个等分试样的诊断性扩增子。
对此类小扩增子大小的要求限制了沿着靶序列分布引物的选择的数目,并且需要使用多个密切相关的引发位点来确保没有单个位点被交联或引物结合序列内的破坏性加合物的存在妥协。因为dna序列不是完全异源的,并且因为最佳引物结合位点趋于支持更高的gc含量,尤其是在引物序列的3'末端处,用于生成一组扩增子的最佳引物趋于聚集到给定靶序列的窄区域内。为了确保每个扩增子由所需的引物对特异性地且单独地产生,通常每个扩增子必须个别地产生,以避免在pcr扩增过程期间通过扩增子链的交叉杂交形成引物二聚体或重组扩增产物。实际上说,这要求在多个pcr反应中稀释靶dna,这是靶dna的无效使用,并且对于可以从ffpe活组织检查样品中回收的微量细菌dna是不实际的。此处公开的更有效的方法涉及将pcr引物对分离成相互相容的库,使得存在在pcr扩增期间在新生扩增子之间的引物二聚体形成或交叉杂交的最小风险。这种引物对的分离合并允许在临床实验室背景中,通过基于pcr扩增的方法分析含有稀有dna序列的ffpe样品的有效和自动化处理。
技术实现要素:
在一个方面,本发明提供了一组pcr扩增引物,用于表征ffpe保存组织中的抗药性幽门螺杆菌的存在。图2中列出了由两对引物(每对中一个正向和一个反向)组成的引物,用于分离已知抗药性幽门螺杆菌菌株特有的基因。此处呈现的示例性基因包括16srrna(与四环素抗性相关)、23srrna(与克拉霉素抗性相关)、pbp1(与对青霉素抗生素的抗性相关)、gyra(与氟代醌(fluoroquinone)抗生素的抗性相关)、rpob(与利福布汀抗性相关)和rdxa(涉及对甲硝唑的抗性)。本领域技术人员应理解,涉及对其它抗生素的抗性的其它遗传基因座是已知的,并且可以包括在此处所述的那些中或取代此处所述的那些。实际上,对于每个靶基因使用两个引物对最小化在靶dna序列中发现的任何特定损伤(例如交联或加合物)将抑制来自两个扩增子的pcr扩增的机会,因为此类损伤不太可能在两个不同的引物结合位点处发生,并且涉及靶(模板)dna内的靶基因的所有拷贝。然而,使用靶向细菌染色体的相同区域然而产生不同但重叠的片段的两对引物,需要pcr扩增反应分开进行,以便避免产生与每个引物对设计为产生的全长扩增子不匹配的杂合扩增子。因此,必须对于每组引物对执行至少两次pcr扩增反应。相比之下,可以合并靶向不同基因并且产生没有同源序列的扩增子的pcr引物对,并且因此尽可能有效地利用可以从ffpe保存的组织中扩增的有限量的靶细菌dna。本发明教导了将靶向重叠扩增子组的引物对分离成分开的库,并且对分离的库执行单一pcr扩增反应以产生所需的扩增子。在此处提供的一个实施例中,使用从ffpe胃活组织检查样品中提取的dna,可以仅从两个pcr反应产生对于上文列出的五种不同类型的抗药性幽门螺杆菌基因诊断性的多达10个扩增子。
本发明的一个实施例是用于在样品内检测多个基因中的突变的方法,该方法包括a)鉴定适合于产生扩增子的pcr引物对,所述扩增子包含含有一个或多个突变的每个基因的区域,b)将包含一个或多个引物的pcr引物对分离成分开的pcr引物对库,所述一个或多个引物干扰通过另一个pcr引物对的扩增子生成,其中分开的pcr引物对库各自包含多个pcr引物对;c)从每个分开的pcr引物对库和由样品中分离的靶dna生成扩增子;d)将从每个分开的pcr引物对库和靶dna产生的所有扩增子组合成样品扩增子库,将独特的索引序列加入样品扩增子库内的扩增子,以生成加索引的样品扩增子库,任选地还将加索引的样品扩增子库与来自不同样品的一个或多个差异索引的样品扩增子库组合,并且同时测序所有加索引的样品扩增子;并且e)通过参考相应的野生型基因序列,鉴定来自样品的加索引的测序的扩增子内的突变。
在本发明的一个实施例中,样品是活组织检查样品,并且在另一个实施例中,活组织检查是胃活组织检查。在一个进一步的实施例中,活组织检查样品包含福尔马林固定的石蜡包埋的活组织检查样品。在另一个实施例中,样品含有幽门螺杆菌(helicobacterpylori)(幽门螺杆菌(h.pylori))。
在另一个实施例中,多个基因包含选自以下的基因:幽门螺杆菌16srrna、23srrna、gyra、rpob、pbp1和rdxa。在进一步的实施例中,鉴定的突变是幽门螺杆菌23srrna基因的a2142g、a2143g和/或a2142c突变;幽门螺杆菌16srrna基因的a928c、ag926-927gt、a926g/a928c和/或aga926-928ttc突变;和/或编码dna促旋酶亚基a的幽门螺杆菌gyra基因的c261a、c261g、g271a和/或g271t突变;在编码dna指导的rna聚合酶的β/β'亚基的幽门螺杆菌rpob基因的密码子525和545之间;编码青霉素结合蛋白1的幽门螺杆菌pbp1基因中的c1242a或c1242g突变;或幽门螺杆菌rdxa基因内。在另一个实施例中,鉴定的突变产生由rdxa编码的幽门螺杆菌氧不敏感性(i型)napd(p)h硝基还原酶的功能丧失。
在本发明的一个实施例中,扩增子长度不超过230个碱基对。在另一个实施例中,扩增子长度大于130个碱基对。在一个进一步的实施例中,包含干扰通过另一个pcr引物对的扩增子生成的一个或多个引物的pcr引物对,通过形成交叉对引物二聚体或通过形成交叉对截短的扩增子来干扰。
本发明的另一个实施例涉及用于在患者衍生的样品内检测抗药性幽门螺杆菌的存在的方法,该方法包括:a)由从患者衍生的样品分离的dna生成扩增子;以及i)pcr引物对库1,其包含引物seqidno.1-10;ii)pcr引物对库2,其包含引物seqidno.11-22;iii)pcr引物对3,包含引物seqidno.23-28;iv)pcr引物对库4,其包含引物seqidno.29-32;v)pcr引物对库5,其包含引物seqidno.33-38;vi)pcr引物对库6,其包含引物seqidno.39-44;b)将步骤a)中从pcr引物对库1-6产生的所有扩增子组合成样品扩增子库,将独特的索引序列加入样品扩增子库内的扩增子,以生成加索引的样品扩增子库,任选地还将加索引的样品扩增子库与来自不同患者衍生的样品的一个或多个差异索引的样品扩增子库组合,并且同时测序所有加索引的样品扩增子;c)通过参考seqidno.47-51鉴定测序的加索引的样品扩增子内的突变,并且d)通过步骤c)中鉴定的突变的存在或不存在,来确定患者衍生的谱中存在的幽门螺杆菌的抗药性谱。
本发明的另外一个实施例涉及用于在患者衍生的样品内检测抗药性幽门螺杆菌的存在的方法,该方法包括:a)由从患者衍生的样品分离的dna生成扩增子;以及i)pcr引物对库1,其包含引物seqidno.23-28;ii)pcr引物对库2,其包含引物seqidno.29-32;iii)pcr引物对库3,其包含引物seqidno.33-38;iv)pcr引物对库4,其包含引物seqidno.39-44;b)将步骤a)中从pcr引物对库1-4产生的所有扩增子组合成样品扩增子库,将独特的索引序列加入样品扩增子库内的扩增子,以生成加索引的样品扩增子库,任选地还将加索引的样品扩增子库与来自不同患者衍生的样品的一个或多个差异索引的样品扩增子库组合,并且同时测序所有加索引的样品扩增子;c)通过参考seqidno.47-51鉴定测序的加索引的样品扩增子内的突变,并且d)通过步骤c)中鉴定的突变的存在或不存在,来确定患者衍生的谱中存在的幽门螺杆菌的抗药性谱。
在一个进一步的实施例中,使用针对库内的单个扩增子的一个末端引物作为正向测序引物,以及针对该扩增子的另一个末端引物作为反向测序引物,可以通过经典的桑格测序方法来测序此处所述的任何扩增子库。可替代地,对于扩增子库内的每个个别扩增子的每个所需反应特异性的独特测序引物可以用于相同目的。以这种方式,每个扩增子可以从扩增子库直接测序。扩增子库可以与来自相同ffpe提取的活组织检查样品的其它扩增子库组合或不组合,并且组合的扩增子在下一代测序(ngs)制备中通过添加衔接子和索引标签制备用于测序。源自单个ffpe活组织检查样品的加上标签的扩增子库还可以与来自不同ffpe活组织检查样品的差异标记的扩增子库组合,并且组合的样品扩增子库通过高通量多重测序方法直接测序。
附图说明
图1描绘了幽门螺杆菌rdxa基因并且示出了所述扩增子的相对排列。
图2是说明书和实例中公开的各个引物的表。
图3是电泳凝胶,证实合并所有rdxa特异性引物生成不正确大小的扩增子,而此处所述的分离的引物库生成正确大小的扩增子。
图4鉴定了与23srrna194-f和170r-f的比对测序的23srrna(在ffpe样品ffpe_k和l中)23srrna克拉霉素序列中的代表性突变。红框显示了23srrna上的突变基因座。
图5是使用分离合并和合并的扩增子策略,通过来自16个ffpe样品的下一代测序(ngs)数据分析的16个ffpe样品的汇总数据表,其中六个不同基因各自中的突变是鉴定的抗药性幽门螺杆菌特有的。通过桑格测序证实了通过ngs鉴定的所有突变。
图6是使用分离合并和合并的扩增子策略,通过来自24个ffpe样品的ngs数据分析的24个ffpe样品的汇总数据表,其中六个不同基因各自中的突变是鉴定的抗药性幽门螺杆菌特有的。数据表列出了通过ngs鉴定的基因突变以及其突变频率。
具体实施方式
本发明提供了用于扩增ffpe活组织检查样品中存在的痕量dna序列的方法和组合物。这些方法和组合物可用于确定来自ffpe胃活组织检查样品的幽门螺杆菌的药物和多药抗性谱的存在并将其表征。
本文所述的方法和组合物涉及从ffpe活组织检查样品中存在的痕量细菌dna生成扩增子。此类dna尤其经受损伤,并且受损dna的pcr扩增需要特殊的步骤,以确保从靶dna扩增所需的序列。这些步骤包括最小化从扩增反应中回收的每个扩增子的长度,确定靶向dna序列内的最佳引物序列,并且策略性地合并重叠的扩增子和引物以避免彼此干扰。称为分离合并的过程。在典型的pcr扩增方案中,其中dna完整性可以被假定为相对较高的,引物选择不像已知模板dna被交联或碱性加合物的存在损坏时那样受限制,如同ffpe衍生的dna一样。此外,在典型的活组织检查样品中发现的细菌dna的绝对水平比宿主生物的dna低多个数量级。这意味着靶向用于扩增的序列是样品中总dna的少数,并且从此类稀有dna序列中成功扩增一个或多个扩增子需要最佳的引物以及可能的靶dna的最有效使用。此外,为了在商业临床实验室中普遍有用,此类方法应该尽最大可能与已建立的技术和试剂相容并利用其。
使用qiagenqiaampdspdnaffpetissuekit(qiagen,目录号60404),遵循制造商的说明从ffpe胃活组织检查样品中提取总dna。简言之,将五个5μm厚的单个ffpe活组织检查样品切片置于单个管中,并且用二甲苯脱石蜡,用蛋白酶k在65℃下处理一小时,以裂解脱石蜡的样品内的细胞,然后保持在90℃下另外一小时,以使细胞结构去交联且使蛋白酶k变性。将脱石蜡且裂解的样品冷却至室温,遵循制造商推荐的程序,应用于minielutepcrpurification柱(qiagen,目录号28004)。将minielute柱洗涤两次,并且用洗脱缓冲液从柱中回收从ffpe活组织检查样品中回收的dna。用qubit2.0荧光计(thermofisherscientific)定量回收的dna,并且用nanodrop仪器(thermofisherscientific)测定dna纯度。
为了验证来自每个活组织检查样品的总dna样品内幽门螺杆菌序列的存在,使用pcr引物seqidno:45和46执行幽门螺杆菌23srrna基因的特异性125碱基对片段独特高度保守区域的pcr扩增。通过blast分析[altschul,s.f.,gish,w.,miller,w.,myers,e.w.&lipman,d.j.(1990)"basiclocalalignmentsearchtool."j.mol.biol.215:403-410],将pcr产物纯化且测序,并且证实为对于幽门螺杆菌是特异性的。正确的扩增产物(扩增子)的不存在指示样品中不存在可用的幽门螺杆菌dna。
进一步调查的确产生125碱基对幽门螺杆菌特异性pcr扩增子的样品,以确定回收的dna的质量。为了确定从含有幽门螺杆菌的ffpe胃活组织检查样品中提取的dna用于pcr扩增和测序的适合性,开发了多重pcr定量测定。该定量测定涉及用一组pcr引物pcr扩增编码甘油醛-3-磷酸脱氢酶的人gapdh基因,所述pcr引物组能够产生100、200、300、400和500个碱基对片段的扩增子梯。没有显著损伤、具有大片段大小和以相对高浓度的ffpedna产生扩增子梯的所有五个“梯级”,而高度受损和显著片段化的dna将不产生任何预期的扩增子。根据我们用这种方法积累的经验,如果该测试产生扩增子梯的2到5个条带,则我们将从ffpe胃活组织检查样品中回收的dna评定为良好,如果它只产生单个条带,则评定为中等,并且如果完全没有观察到条带,则评定为差。跨越众多ffpe提取的活组织检查样品观察到的扩增子条带数目的总体频率指示,将分析扩增子大小限制在约200个碱基对或更少,提供了在产生尽可能多的连续序列和避免模板dna中的pcr扩增终止损伤之间的最佳平衡。
使用在100mmtris-hcl、500mmkcl和25mmmgcl2缓冲液中的1utaqdna聚合酶、10mmdntp混合物,使用新近制备的或冷冻的幽门螺杆菌染色体dna(作为阳性对照)以及从ffpe胃活组织检查样品(实验样品)提取的样品执行pcr扩增反应。
对于通过桑格测序方法分析的样品,如指示的使用图1中的pcr引物(seqidno1-44),其中热循环参数是:在95℃下初始变性10分钟,随后为在95℃下30秒、在55℃下30秒和在72℃下30秒的35个循环,然后在72℃下最后延伸10分钟。遵循制造商的说明,用minielutepcrpurification柱纯化来自每个扩增反应的所得到的扩增子。
遵循制造商的方案,用bigdyeterminatorv3.1cyclesequencingkit(appliedbiosystems,目录号4337455)处理纯化的扩增子用于桑格测序。pcr测序反应用下述热参数执行:95℃1分钟,然后是95℃30秒、56℃30秒和60℃1分钟的25个循环。遵循制造商的说明,用agencourtcleanseq试剂盒(beckmancoulterlifesciences目录号a29151)处理引物延伸反应。在abi3500geneticanalyzer(appliedbiosystems)上装载样品且分析。用3500seriesdatacollectionsoftware(appliedbiosystems)收集原始序列数据,并且使用sequencherv5.4软件(genecodescorp.)组装且针对参考序列比对。
在设计用于ngs测序的样品的情况下,将illumina突出端衔接子序列加入图2中列出的基因座特异性引物序列(seqidno1-44)中。正向突出端序列(加入基因座特异性正向引物序列的5'侧)是tcgtcggcagcgtcagatgtgtat-aagagacag(seqidno.47),并且反向突出端序列(加入基因座特异性反向引物序列的5'侧)是gtctcgtgggctcggag-atgtgtataagagacag(seqidno.48)。第一轮pcr扩增热循环参数为:在95℃下初始变性11分钟,随后为在95℃下30秒、在59℃下1分钟和在72℃下1分钟的35个循环,然后在72℃下最后延伸10分钟。第二阶段pcr反应(涉及多重指数衔接子的添加)涉及热循环参数:98℃30秒,98℃20秒、60℃30秒和72℃45秒的17个循环,以及在72℃下5分钟的最后延伸。然后遵循制造商的说明,用agencourtampurexp试剂盒(beckmancoulterlifesciences目录号a63880)处理文库,在必要时稀释,在2100bioanalyzer(agilenttechnologies)上定量,并且装载到illuminamiseq测序仪器(illumina,inc.)上。使用nextgenev2.4.1.1软件(softgenetics)执行数据分析。
在下述实例中示出了本发明,所述实例阐述以帮助理解本发明,但不应解释为以任何方式限制如在下述权利要求中限定的本发明的范围。
实例1
pcr引物和扩增子的分离合并,以表征来自多个遗传基因座的抗药性
在本发明的一个方面,可以通过分离用于生成覆盖每个靶向基因座的诊断性扩增子的pcr引物来同时表征编码不同形式的抗药性的多个基因座。包含seqidno:1和2的pcr引物对用于产生跨越幽门螺杆菌16srrna基因的位置926-928的168碱基对扩增子(16srrna168)。事实上,这些位置中的任何突变都产生低水平的四环素抗性,而三重突变aga926-928ttc与非常高水平的四环素抗性相关。包含seqidno:11和12的第二引物对产生162碱基对扩增子(16srrna162),其也涵盖幽门螺杆菌16srrna的位置926-928。包含seqidno:3和4的另一个pcr引物对产生跨越幽门螺杆菌23srrna基因的位置2142和2143的194碱基对扩增子(23srrna194)。这些位置的突变,特别是a2142g、a2142c和a2143g突变与克拉霉素抗性相关。包含seqidno:13和14的第二引物对产生170碱基对扩增子(23srrna170),其也跨越幽门螺杆菌23srrna的位置2142和2143。包含seqidno:5和6的另一个pcr引物对产生193碱基对扩增子(gyra193),其跨越编码由gyra基因编码的幽门螺杆菌促旋酶的a亚基的氨基酸位置87至91的区域。已知asn87至lys或tyr的突变以及asp91至gly、asn或tyr的突变个别地或一起产生氟代醌抗生素的抗性。跨越幽门螺杆菌gyra基因的该区域的第二pcr引物对,包含seqidno:15和16,产生139碱基对扩增子(gyra139)。包含seqidno:17和18跨越相同区域的第三pcr引物对产生137碱基对扩增子(gyra137)。包含seqidno:7和8的另一个pcr引物对产生159碱基对扩增子(pbpa159),其涵盖编码幽门螺杆菌pbp1基因的氨基酸位置414的序列。通常在青霉素结合蛋白1的位置414处发现的丝氨酸至精氨酸的突变产生对阿莫西林和其它青霉素类抗生素的抗性。另一个pcr引物对,seqidno:19和20产生140碱基对扩增子(pbpa140),其还涵盖编码幽门螺杆菌青霉素结合蛋白1的位置414的序列。包含seqidno:9和10的另一个pcr引物对产生228碱基对扩增子,其涵盖编码rna聚合酶的β/β'亚基的幽门螺杆菌rpob基因的密码子525至545。该区域内任何密码子的突变可以赋予针对利福布汀及其它利福霉素样抗生素的抗性。包含seqidno:21和22的pcr引物对还产生扩增子(rpob-r-167),其为167个碱基对并且涵盖rpob内的关键密码子。
将靶向潜在编码抗药性突变的特定基因区域的每个pcr引物对分离成分开的pcr引物对库,其含有靶向不同基因的一个或多个独特引物对。因此,每个库的pcr扩增产生对于每个库内的多个基因特异性的扩增子,并且最小化pcr扩增假象的机会,所述假象例如由重叠扩增子序列内的同源配对引起的引物二聚体或交叉对扩增子截短。如图2所示,库5gf包含pcr引物对16srrna168、23srrna194、gyra193、pbpa159和rpob228。当用这些引物对扩增ffpe衍生的幽门螺杆菌靶dna时,产生159、168、193、194和228个碱基对的五个独特的扩增子。
扩增子库5gr(图2)包含pcr引物对16srrna162r、23srrna170r、gyra139r、gyra137r、pbpa140和rpob-r-167。当用这些引物对扩增ffpe衍生的幽门螺旋杆菌靶dna时,产生多达8个扩增子。其中四个是140、162、167和170个碱基对的独特扩增子(分别对应于pbpa特异性扩增子、16srrna特异性扩增子、rpob特异性扩增子和23srrna特异性扩增子)。剩余的四个扩增子代表库5gr中存在的两对gyra特异性pcr引物对的排列。这些引物对在相同方向上彼此部分重叠,使得可以从两个引物对产生四种可能的扩增子。因为引物的部分重叠限制于具有相同链取向(方向)的引物,所以不存在引物二聚体形成的风险,并且因为引物之间的扩增序列是相同的(除了扩增序列的绝对长度之外),所以扩增子之间的交叉杂交将不产生新的序列。因此,两个半独特性引物对可以容纳在扩增子库内。在这种情况下,这些引物可以产生131、137、139和145个碱基对的多达四个同源物扩增子。这些扩增子的序列从末端到末端与每个其它扩增子中的序列相同-除了少数碱基对从较短扩增子的末端缺失之外,所述少数碱基对存在于较长的扩增子中。
每个测序的扩增子的比对允许鉴定赋予抗药性的突变。图4是在两个独立的ffpe衍生样品(“e”和“k”)中鉴定的23srrna中的a2142g突变的例子,其指示折磨这些样品由其衍生的患者的幽门螺杆菌菌株的治疗不太可能响应克拉霉素。顶部序列是已知的克拉霉素抗性幽门螺杆菌菌株,而紧接在对照下方的两个序列是来自样品“e”的独立pcr扩增子,并且最后两个序列是来自样品“f”的独立扩增子。
实例2
pcr引物和扩增子的分离合并,以表征源自调查基因的整个编码序列的抗药性
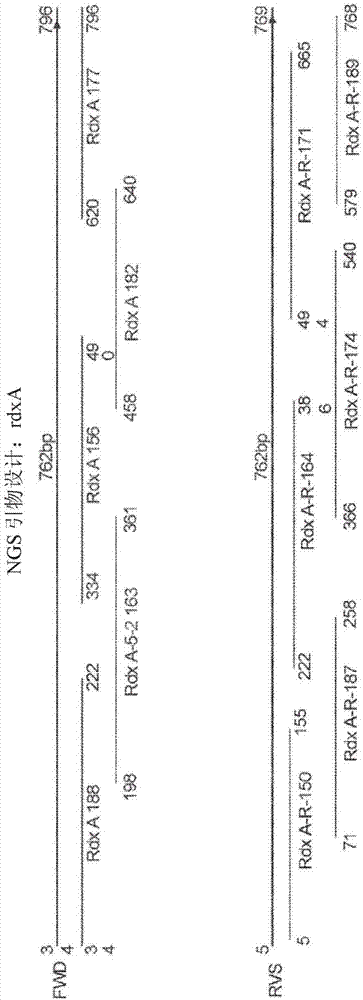
在本发明的另一个方面,分离的pcr引物库允许有效覆盖整个基因。由于编码细菌氧不敏感性(i型)nad(p)h硝基还原酶的幽门螺杆菌rdxa基因中的任何功能缺失丧失,可以发生对甲硝唑的抗性。甲硝唑通过该酶的作用而被激活,并且因此,编码该酶的rdxa基因内的任何移码或点突变都有可能赋予对甲硝唑的抗性。与先前描述的实施例不同,没有单个短扩增子可以涵盖幽门螺杆菌中遇到的已知的甲硝唑抗性突变谱。为了解决这个问题,设计了产生重叠扩增子的两个系列的pcr引物对,使得每个系列内的引物产生短扩增子,其一起覆盖rdxa基因的整个编码区。两个系列之间的引物位于独特的位置中,但一些可以在rdxa基因的编码序列内相对于其它系列中的类似引物在一个方向上仅偏移几个碱基(以类似于先前段落中描述的扩增子库5gr内的部分重叠gyra引物的方式)。该策略降低了靶dna中存在的单一交联或加合物完全阻断扩增子产生的机会。源自由一个pcr引物系列产生的扩增子的序列可以与从另一个pcr引物系列产生的扩增子组装,以确保从总体引物库组实现rdxa基因的完全覆盖。将每个系列的引物置于每个系列的两个扩增子库之一内,使得系列内可以重叠且由易于形成引物二聚体的引物产生的扩增子被分离到分开的库内。在幽门螺杆菌rdxa基因的情况下,一系列五个短扩增子共同跨越整个rdxa编码序列。这五个扩增子,从5'到3'包含rdxa188扩增子(由pcr引物对seqidno:23和24产生)、rdxa-5-2-163扩增子(由pcr引物对产生seqidno:29)和30)、rdxa156扩增子(由pcr引物对seqidno:25和26产生)、rdxa182扩增子(由pcr引物对seqidno:31和32产生)和rdxa177扩增子(由pcr引物对seqidno:27和28产生)。在该系列内,产生扩增子rdxa188、rdxa156和rdxa177的pcr引物对被置于命名为rdxa-f1的单个库内,而产生扩增子rdxa-5-2-163和rdxa182的pcr引物对被组合成命名为rdxa-f2的不同库(图2)。在另一个系列中,六个短扩增子用于跨越整个幽门螺杆菌rdxa基因。5'至3'排列的这些扩增子包含rdxa-r-150扩增子(由pcr引物对seqidno:33和34产生)、rdxa-r-187扩增子(由pcr引物对seqidno:39和40产生)、rdxa-r-164扩增子(由pcr引物对seqidno:35和36产生)、rdxa-r0174扩增子(由pcr引物对seqidno:41和42产生、rdxa-r-171个扩增子(由pcr引物对seqidno:37和38产生)和rdxa-r-189扩增子(由pcr引物对seqidno:43和44产生)。在该系列内,产生扩增子rdxa-r-150、rdxa-r-164和rdxa-r-171的pcr引物对组合成命名为rdxa-r1的一个库,而产生扩增子rdxa-r-187、rdxa-r-174和rdxa-r-189的pcr引物对组合成命名为rdxa-r2的一个不同的库(图2)。这些pcr引物库用于从由ffpe活组织检查样品中提取的幽门螺杆菌dna生成其同源扩增子。取决于扩增子如何测序,来自每个pcr反应的扩增子可以进一步组合,使得每个系列由单个扩增子库表示。
如图3所示,适当的分离合并产生正确的(预测的)扩增子,而当存在于单个库中的相同引物产生由不正确的扩增子和引物二聚体组成的一系列pcr产物。两个ffpe样品,“ffpe_c”和“ffpe_e”如所述的进行处理,并且充当用于分析rdxa基因的靶dna。泳道6(从最左边的泳道计数为泳道1)是双链dna尺寸标记物(ready-to-use100bpdnaladder,biotium,inc.)。尺寸标记物在每一侧上侧面为样品特异性合并的扩增产物。标记为“rdx-all”的泳道1和7是pcr扩增反应的结果,其含有跨越上述整个rdxa基因的扩增子的所有pcr引物。标记为“f1”的泳道(泳道2和8)对应于包含rdxa188、rdxa156和rdxa177扩增子引物的rdxa-f1库,并且两个样品泳道均含有如预测的150至200个碱基对的扩增子。标记为“f2”的泳道(泳道3和9)对应于包含rdxa-5-2-163和rdxa182扩增子引物的rdxa-f2库,并且两个样品泳道均含有正确大小的扩增子。注意到也存在约60个碱基对的次要产物,然而该扩增子不干扰测序正确扩增子,并且可能代表所需扩增子之一之内或之间的内部重组产物。标记为“r1”的泳道(泳道4和10)对应于包含rdxa-r-150、rdxa-r-164和rdxa-r-171扩增子引物的rdxa-r1库,并且所需的扩增子在凝胶上在150至约170对碱基对之间成簇。标记为“r2”的泳道(泳道5和11)对应于包含rdxa-r-187、rdxa-r-174和rdxa-r-189扩增子引物的rdxa-r2库,产生适当大小的扩增子。
图5示出了该方法基于单一ngs分析确定源自ffpe样品的多种分离物中的抗药性和多药抗性的存在和模式的能力。在这种情况下,将如实例1中所述分析的5个分开基因以及如实例2中所述分析的rdxa基因整理成单个报告,概述每个患者衍生的ffpe活组织检查样品中幽门螺杆菌对六种药物各自的潜在抗性谱。
图6还示出了该方法基于如实例1和2中所述的ngs分析确定源自ffpe样品的多种分离物中的抗药性和多药抗性的存在和模式的能力。该表列出了在每个样品中发现的基因突变以及每个突变的频率。结果显示ngs分析可以区分没有任何突变的ffpe样品相对于具有突变的样品。例如,在24个样品中,10个(42%)没有突变(编号5、6、8、9、13、17和21-24)。然而,结果还显示ngs分析可以检测一个ffpe样品内的单个突变和多个突变两者。例如,14个样品(58%)具有至少一个基因突变(编号1-4、7、10-12、14-16和18-20)。在这14个样品中,11个样品在单个基因中具有突变(编号2、4、7、11-12、14-16、18、19和20),并且3个样品在多个基因中具有突变(编号1、3和10)。
在具有单基因突变的样品中,5个样品仅具有gyra基因突变(编号2、4、15、18和19),2个样品仅具有rdxa基因突变(编号7和20),并且4个样品仅具有23srrna基因突变(编号11、12、14和16)。在仅具有gyra突变的样品中,样品之一具有关于gyra基因的两个突变(编号19)。如上文讨论的,gyra基因突变的存在指示氟喹诺酮类抗生素抗性。在仅具有rdxa基因突变的样品中,样品之一具有在rdxa基因内的两个突变(编号7)。如上文讨论的,rdxa基因突变的存在指示对甲硝唑的抗性。
具有多个基因突变的3个样品在23srrna和gyra(编号1和3)以及gyra和rdxa(编号10)两者中具有突变。同时发生的23srrna和gyra突变指示克拉霉素和氟喹诺酮类抗生素抗药性两者;然而,同时发生的gyra和rdxa突变指示氟喹诺酮类抗生素抗性和甲硝唑抗性两者。
序列表
<110>美国分子实验室公司(americanmolecularlaboratories,inc.)
<120>用于从福尔马林固定的石蜡包埋的生物样品中表征抗药性细菌的方法和组合物
<130>116110-5001p
<150>62/334,185
<151>2016-05-10
<160>52
<170>patentinversion3.5
<210>1
<211>17
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>1
taacgcattaagcatcc17
<210>2
<211>18
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>2
ccagacactccactattt18
<210>3
<211>15
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>3
ccgacctgcatgaat15
<210>4
<211>15
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>4
agccaaagcccttac15
<210>5
<211>19
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>5
tatgcgatgcatgaattag19
<210>6
<211>19
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>6
catcaatagagccaaagtt19
<210>7
<211>17
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>7
ttgataatggctattcc17
<210>8
<211>15
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>8
ggctcaaggcttctt15
<210>9
<211>20
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>9
gacaagctcactaccatgag20
<210>10
<211>18
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>10
cacatccctggcttcaaa18
<210>11
<211>17
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>11
ctagcggattctctcaa17
<210>12
<211>17
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>12
cagtaatgcagctaacg17
<210>13
<211>17
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>13
catcaagggtggtatct17
<210>14
<211>17
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>14
ttgtagtggaggtgaaa17
<210>15
<211>18
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>15
cgttatcgccatcaatag18
<210>16
<211>19
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>16
ggtgatgtgattggtaaat19
<210>17
<211>18
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>17
ccatcaatagagccaaag18
<210>18
<211>15
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>18
atcgtgggtgatgtg15
<210>19
<211>17
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>19
ttgataatggctattcc17
<210>20
<211>16
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>20
ggttacaagccctaaa16
<210>21
<211>20
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>21
tgggacaaattcggccataa20
<210>22
<211>17
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>22
tttcatgggcggtcagc17
<210>23
<211>22
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>23
tggtaattgtttcgttagggat22
<210>24
<211>18
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>24
tggcgatttcagcgattt18
<210>25
<211>18
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>25
aagcgcttcagcgttaat18
<210>26
<211>18
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>26
tgcatgctgtggttgaat18
<210>27
<211>24
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>27
gaagagcgtatcaataagcctaaa24
<210>28
<211>22
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>28
atgccactccttgaactttaat22
<210>29
<211>23
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>29
agcctccaataatgcaactatcc23
<210>30
<211>22
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>30
cataccaccattaacgctgaag22
<210>31
<211>18
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>31
atgcttggcgtgagattc18
<210>32
<211>21
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>32
ggcttattgatacgctcttct21
<210>33
<211>20
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>33
atgccactccttgaacttta20
<210>34
<211>18
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>34
gcatgcttgatcgctttg18
<210>35
<211>22
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>35
atgcaactatccaatcccatta22
<210>36
<211>24
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>36
ccggagtcttataaagttagagtg24
<210>37
<211>18
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>37
gtgcgctgcaatttgttt18
<210>38
<211>19
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>38
ttaaacgagcgccattctt19
<210>39
<211>21
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>39
caaccaagtaatcgcatcaac21
<210>40
<211>18
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>40
catgggcgtgagcttaat18
<210>41
<211>25
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>41
ctaactttataagactccggataga25
<210>42
<211>22
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>42
tgtgatggttactgataaggat22
<210>43
<211>19
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>43
ctggcgatttcagcgattt19
<210>44
<211>22
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>44
tggtaattgtttcgttagggat22
<210>45
<211>22
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>45
acaacccagactaccaaataag22
<210>46
<211>19
<212>dna
<213>人工序列
<220>
<223>pcr引物
<400>46
gtgagctgttacgctttct19
<210>47
<211>500
<212>dna
<213>幽门螺杆菌(helicobacterpylori)
<400>47
gtaatccgtagagatcaagaggaatactcattgcgaggcgacctgctggaacattactga60
cgctgattgcgcgaaagcgtggggagcaaacaggattagataccctggtagtccacgccc120
taaacgatggatgctagttgttggagggcttagttttccagtaatgcagctaacgcatta180
agcatcccgcctggggagtacggtcgcaagattaaaatcaaaggaatagacggggacccg240
cacaagcggtggagcatgtggtttaattcgaagatacacgaagaaccttacctaggcttg300
acattgagagaatccgctagaaatagtggagtgtctggcttgccagaccttgaaaacagg360
tgctgcacggctgtcgtcagctcgtgtcgtgagatgttgggttaagtcccgcaacgagcg420
caaccccttttcttagttgctaacaggttatgctgagaactctaaggatactgcctccgt480
aaggaggaggaaggtgggga500
<210>48
<211>1500
<212>dna
<213>幽门螺杆菌(helicobacterpylori)
<400>48
caagtgataataaaagggggtagagccctgattgggctagggctgctcgccgcggtacca60
aaccctatcaaacttcgaataccttttatcgtatcttgggagtcaggcggtgggtgataa120
aatcaatcgtcaaaaggggaacaacccagactaccaaataaggtccctaagttctattct180
gagtggaaaaagatgtgtggctactcaaacaaccaggaggttggcttagaagcagccatc240
ctttaaagaaagcgtaacagctcactggtctagtggtcatgcgctgaaaatataacgggg300
ctaagatagacaccgaatttgtagattgtgttaaacacagtggtagaagagcgttcatac360
cagcgttgaaggtataccggtaaggagtgctggagcggtatgaagtgagcatgcaggaat420
gagtaacgataagatatatgagaattgtatccgccgtaaatctaaggtttcctacgcgat480
ggtcgtcatcgtagggttagtcgggtcctaagccgagtccgaaaggggtaggtgatggca540
aattggttaatattccaataccgactgtggagcgtgatggggggacgcatagggttaagc600
gagctagctgatggaagcgctagtctaagggcgtagattggagggaaggcaaatccacct660
ctgtatttgaaacccaaacaggctctttgagtccttttaggacaaagggagaatcgctga720
taccgtcgtgccaagaaaagtctctaagcatatccatagtcgtccgtaccgcaaaccgac780
acaggtagatgagatgagtattctaaggcgcgtgaaagaactctggttaaggaactctgc840
aaactagcaccgtaagttcgcgataaggtgtgccacagcgatgtggtctcagcaaagagt900
ccctcccgactgtttaccaaaaacacagcactttgccaactcgtaagaggaagtataagg960
tgtgacgcctgcccggtgctcgaaggttaagaggatgcgtcagtcgcaagatgaagcgtt1020
gaattgaagcccgagtaaacggcggccgtaactataacggtcctaaggtagcgaaattcc1080
ttgtcggttaaataccgacctgcatgaatggcgtaacgagatgggagctgtctcaaccag1140
agattcagtgaaattgtagtggaggtgaaaattcctcctacccgcggcaagacggaaaga1200
ccccgtggacctttactacaacttagcactgctaatgggaatatcatgcgcaggataggt1260
gggaggctttgaagtaagggctttggctcttatggagccatccttgagataccacccttg1320
atgtttctgttagctaactggcctgtgttatccacaggcaggacaatgcttggtgggtag1380
tttgactggggcggtcgcctcctaaaaagtaacggaggcttgcaaaggttggctcattgc1440
ggttggaaatcgcaagttgagtgtaatggcacaagccagcctgactgtaagacatacaag1500
<210>49
<211>550
<212>dna
<213>幽门螺杆菌(helicobacterpylori)
<400>49
atgcaagataattcagtcaatgaaacaaaaaatattgtagaagtggggattgattcttct60
attgaagagagctatttagcttattccatgagcgtgatcatagggcgcgctttaccggac120
gctagagatggcttaaagcccgtgcataggcgtattttgtatgcgatgcatgaattaggc180
cttacttcaaaagtcgcttacaaaaaaagcgctaggatcgtgggtgatgtgattggtaaa240
taccacccccatggcgataatgcggtttatgatgcgctagtgagaatggcgcaagatttt300
tccatgcgtttggaattagtggatgggcagggcaactttggctctattgatggcgataac360
gccgcagcgatgcgttacactgaagccagaatgactaaggcgagtgaagaaattttaagg420
gatattgataaagacaccattgattttgtgcctaattatgacgataccttaaaagagcca480
gatattttaccaagccgtctgcctaaccttttagtcaatggggctaatgggatcgctgtg540
gggatggcga550
<210>50
<211>500
<212>dna
<213>幽门螺杆菌(helicobacterpylori)
<400>50
aactaacgcgtctaatgaagatgaagacaacttaaacgctagcatgatcgttacagacac60
gagcaccggtaagattttagctttagtgggggggattgattataaaaaaagcgctttcaa120
tcgcgccacgcaagccaaacggcagtttgggagcgcgataaagccttttgtgtatcagat180
cgcttttgataatggctattccacgacttctaaaatccctgataccgcgcgaaactttga240
aaatggcaattatagtaaaaacagtgaacaaaaccacgcatggcaccccagcaattattc300
tcgcaagtttttagggcttgtaaccttgcaagaagccttgagccattcgttaaatctagc360
cacgatcaatttaagcgatcagcttggctttgaaaaaatttatcaatctttaagcgatat420
ggggtttaaaaacctccctaaggacttgtctattgtgttagggagctttgctatctcacc480
cattgatgcagctgaaaagt500
<210>51
<211>600
<212>dna
<213>幽门螺杆菌(helicobacterpylori)
<400>51
atgaagatattatcaccaccgttaaatacctcatgaagatcaaaaacaatcaaggcaaga60
ttgatgacagggaccacttgggcaatcgtaggattagggcggtaggggaattgttggcca120
atgaattgcattcaggtttagtgaaaatgcaaaagaccattaaagacaagctcactacca180
tgagcggggcttttgattcgctcatgccccatgacttggtcaattctaaaatgatcacaa240
gcaccatcatggaatttttcatgggcggtcagctctcgcaatttatggatcaaacgaatc300
ccttgagtgaggttacgcacaagcgccgcctttcagcgctcggcgaaggggggttggtga360
aagacagagtggggtttgaagccagggatgtgcaccccacgcattatggccgaatttgtc420
ccattgagaccccagaaggtcaaaatatcggtctgatcaacaccctttccactttcacaa480
gagtgaatgatttaggctttattgaagccccttataaaaaggttgtggatggcaaggtcg540
tgggtgagacgatttatttgaccgctattcaagaagacagccacatcatcgctcccgcaa600
<210>52
<211>1032
<212>dna
<213>幽门螺杆菌(helicobacterpylori)
<400>52
atttgagcatggggcagattttaagcttatttatggtaattgtttcgttagggattttat60
tgtatgctacaaaaaattctaaaaaaataaaggaaaatcaatgaaatttttggatcaaga120
aaaaagaagacaattattaaacgagcgccattcttgcaagatgtttgatagccattatga180
gttttctagcacagaattagaagaaatcgctgaaatcgccaggctatcgccaagctctta240
caacacgcagccatggcattttgtgatggttactgataaggatttaaaaaaacaaattgc300
agcgcacagctatttcaatgaagagatgattaaaagcgcttcagcgttaatggtggtatg360
ctctttaagacccagcgagttgttaccacacggccactacatgcaaaatctctatccgga420
gtcttataaagttagagtgatcccctcttttgctcaaatgcttggcgtgagattcaacca480
cagcatgcaaagattagaaagctatattttagagcaatgctatatcgctgtggggcaaat540
ttgcatgggcgtgagcttaatgggattggatagttgcattattggaggctttgatccttt600
aaaggtgggcgaagttttagaagagcgtatcaataagcctaaaatcgcatgcttgatcgc660
tttgggcaaagggtggcagaagcgagtcaaaaatcaagaaaatcaaaagttgatgcgatt720
acttggttgtgattaaacaaaatcaaaaactttttaactataatcaaacctaaattaaag780
ttcaaggagtggcattttgtttaaaagaatggttttaatcgctcttttaggggtgttttc840
aagcgtttcattaagcgctaagagtcttttaagagatgatgggattttagtctctgattt900
aaagggcatgaaatcagaactatctgatgctcctgcttgggtttttgaagacgctaaagc960
cccctacgaagaaatgggcgtggcgtatatccctgttaataataaatatttagggattga1020
gcaagcgacctt1032
- 还没有人留言评论。精彩留言会获得点赞!