超嗜热脂肪酶LipK及其相关生物材料与应用的制作方法
本发明涉及生物技术领域中超嗜热脂肪酶lipk及其相关生物材料与应用。
背景技术
脂肪酶(ec3.1.1.3)被认为是最重要的商业酶之一,在快速增长的生物技术领域引起了极大的关注。脂肪酶可以在油和水的界面催化三酰基甘油水解,释放二酰基甘油酯,长链脂肪酸(>10个碳)和甘油。在水解过程中,脂肪酶与底物酰基结合形成脂肪酶-酰基复合物,然后将酰基转移到水分子的羟基基团上从而实现水解。在非水溶条件下,脂肪酶可以将羧酸的酰基转移到亲核试剂。
微生物来源的脂肪酶广泛用于各领域,包括工业、食品、饲料和医疗领域。与传统化学催化剂相比,脂肪酶使用领域更为广泛且可重复使用,对环境友好等优点而被重视,但脂肪酶大都对极端温度以及苛刻反应ph比较敏感,必须根据特定的工业环境选择具有特定酶学性质的脂肪酶。根据最适反应温度可以将脂肪酶分为低温、中温、嗜热和超嗜热酶,目前大多数工业生产都是在中高温碱性条件下进行的(45℃以上,ph7以上)。通常在实际应用中使用的化学类催化剂不具备生物学活性,这是酶制剂在工业反应中的一大优势,而在苛刻的工业条件下稳定存在并且持续发挥着催化活性的脂肪酶就显得凤毛麟角了,尤其是超过80℃的最适温度的超嗜热脂肪酶几乎难以找到。许多脂肪酶参与的工业催化反应过程都要求较高的反应温度和较好的热稳定性,如纸架脱脂脱墨、皮毛脱脂以及饲料生产,因此超嗜热脂肪酶在工业领域具有着重要的意义。
技术实现要素:
本发明所要解决的一个技术问题是提供超嗜热脂肪酶。
本发明所提供的超嗜热脂肪酶是a1)-a5)中的任一蛋白质:
a1)氨基酸序列是seqidno.2的蛋白质;
a2)氨基酸序列是seqidno.2的第53-587位的蛋白质;
a3)氨基酸序列是seqidno.2的第54-587位的蛋白质;
a4)在a2)或a3)所示的蛋白质的羧基端或/和氨基端融合蛋白标签得到的融合蛋白;
a5)将a2)或a3)所示的蛋白质经过一个或几个氨基酸残基的取代和/或缺失和/或添加得到的具有超嗜热脂肪酶活性的蛋白质。
上述超嗜热脂肪酶中,a1)的蛋白质名称为rtlipk-his,a2)的蛋白质名称为rtlipk,a3)的蛋白质名称为tlipk。tlipk是来源于trichodermalentiformeaccc30425的成熟的蛋白质。rtlipk是在tlipk的氨基末端添加甲硫氨酸残基保持tlipk的其它氨基酸残基不变得到的重组蛋白质。rtlipk-his是在tlipk的氨基末端添加seqidno.2的第1-53位氨基酸残基,在tlipk的羧基末端添加seqidno.2的第588-595位氨基酸残基,保持tlipk的其它氨基酸残基不变得到的重组蛋白质。
上述超嗜热脂肪酶中,seqidno.2由595个氨基酸残基组成。
上述超嗜热脂肪酶中,蛋白标签是指利用dna体外重组技术,与目的蛋白一起融合表达的一种多肽或者蛋白,以便于目的蛋白的表达、检测、示踪和/或纯化等。
实验证明所述蛋白质具有脂肪酶活性。所述蛋白质在80℃具有脂肪酶的最高酶活力,即所述蛋白质的脂肪酶酶反应的最适温度为80℃。所述蛋白质在ph值为9.5具有脂肪酶的最高酶活力,即所述蛋白质的脂肪酶酶反应的最适ph为9.5。rtlipk-his在37℃、ph值为9-10条件下的脂肪酶酶活是在37℃、ph值为9.5条件下的脂肪酶酶活的92%以上。rtlipk-his在80-90℃、ph值为9.5条件下的脂肪酶酶活是在80℃、ph值为9.5条件下的脂肪酶酶活的90%以上。
与所述超嗜热脂肪酶相关的生物材料也属于本发明的保护范围。
与所述超嗜热脂肪酶相关的生物材料可为下述b1)-b7)中至少一种:
b1)编码所述超嗜热脂肪酶的核酸分子;
b2)含有b1)所述核酸分子的表达盒;
b3)含有b1)所述核酸分子的重组载体、或含有b2)所述表达盒的重组载体;
b4)含有b1)所述核酸分子的重组微生物、或含有b2)所述表达盒的重组微生物、或含有b3)所述重组载体的重组微生物;
b5)含有b1)所述核酸分子的转基因植物细胞系、或含有b2)所述表达盒的转基因植物细胞系、或含有b3)所述重组载体的转基因植物细胞系;
b6)含有b1)所述核酸分子的转基因植物组织、或含有b2)所述表达盒的转基因植物组织、或含有b3)所述重组载体的转基因植物组织;
b7)含有b1)所述核酸分子的转基因植物器官、或含有b2)所述表达盒的转基因植物器官、或含有b3)所述重组载体的转基因植物器官。
上述生物材料中,b1)所述核酸分子可为如下b11)-b13)中任一所示的基因:
b11)编码序列是seqidno.1的dna分子;
b12)核苷酸序列是seqidno.1的第157-1761位的dna分子;
b13)核苷酸序列是seqidno.1的第160-1761位的dna分子。
上述生物材料中,b11)是rtlipk-his的优化基因,其名称为rtlipk-his-y基因;b12)是rtlipk的优化基因,其名称为rtlipk-y基因;b13)是tlipk的优化基因,其名称为tlipk-y基因。其中,序列表中的序列1(seqidno.1)由1788个核苷酸组成。
上述生物材料中,b3)所述的重组载体具体可为pet30a(+)-rtlipk-his-y,pet30a(+)-rtlipk-his-y是用核苷酸序列是seqidno.1的第151-1767位的dna分子替换pet30a(+)的ecori和xhoi识别位点间的片段(包括ecori识别位点和xhoi识别位点在内的小片段),保持pet30a(+)的其它序列不变,得到的重组表达载体。
上述生物材料中,b4)的重组微生物可为将所述的蛋白质的编码基因导入受体微生物,得到表达所述蛋白质的重组微生物,所述受体微生物可为c1)-c4)中的任一种:
c1)原核微生物;
c2)肠杆菌科细菌;
c3)埃希氏菌属细菌;
c4)大肠杆菌,如大肠杆菌e.colibl21(de3)。
上述生物材料中,b4)的重组微生物具体可为将所述pet30a(+)-rtlipk-his导入大肠杆菌e.colibl21(de3)得到的表达氨基酸序列是seqidno.2的重组蛋白质(名称为rtlipk-his)的重组大肠杆菌。
上述生物材料中,b5)至b7)可包括繁殖材料也可不包括繁殖材料。
所述蛋白质在作为脂肪酶(如超嗜热脂肪酶)中的应用和所述生物材料在制备脂肪酶(如超嗜热脂肪酶)中的应用也属于本发明的保护范围。
本发明还提供了制备脂肪酶的方法。
本发明所提供的制备脂肪酶的方法,包括:使所述蛋白质的编码基因在生物中进行表达,得到脂肪酶;所述生物为微生物、植物或非人动物。
上述方法中,所述微生物可为c1)-c4)中的任一种:
c1)原核微生物;
c2)肠杆菌科细菌;
c3)埃希氏菌属细菌;
c4)大肠杆菌,如大肠杆菌e.colibl21(de3)。
上述方法中,使所述蛋白质的编码基因在生物中进行表达包括将所述的蛋白质的编码基因导入受体微生物,得到表达所述蛋白质的重组微生物,培养所述重组微生物,表达得到所述蛋白质。
上述方法中,使所述受体微生物可为上述c1)-c4)中的任一种。
上述方法中,所述重组微生物具体可为将所述pet30a(+)-rtlipk-his导入大肠杆菌e.colibl21(de3)得到的表达氨基酸序列是seqidno.2的重组蛋白质(名称为rtlipk-his)的重组大肠杆菌。
上文中,所有应用的直接目的可以是非疾病治疗目的、非疾病预后目的和/或非疾病诊断目的。
本文中,所述超嗜热脂肪酶是指酶反应的最适温度为80℃以上的脂肪酶,该最适温度是酶反应时间为15分钟的最适温度。
实验证明,本发明的超嗜热脂肪酶的脂肪酶酶反应的最适温度为80℃,脂肪酶酶反应的最适ph为9.5。rtlipk-his在37℃、ph值为9-10条件下的脂肪酶酶活是在37℃、ph值为9.5条件下的脂肪酶酶活的92%以上。rtlipk-his在80-90℃、ph值为9.5条件下的脂肪酶酶活是在80℃、ph值为9.5条件下的脂肪酶酶活的90%以上。本发明的超嗜热脂肪酶是碱性超嗜热脂肪酶。与密码子优化前的基因rtlipk-his-w基因相比,密码子优化后的基因rtlipk-his-y基因使脂肪酶rtlipk-his产量提高了5.5倍;与密码子优化前的基因rtlipk-w基因相比,密码子优化后的基因rtlipk-y基因使脂肪酶rtlipk-his产量提高了5.5倍;与密码子优化前的基因tlipk-w基因相比,密码子优化后的基因tlipk-y基因使脂肪酶rtlipk-his产量提高了5.5倍。本发明可用于纸架脱脂脱墨、皮毛脱脂以及饲料生产中。
附图说明
图1为ph对rtlipk-his活性的影响。
图2为温度对rtlipk-his活性的影响。
具体实施方式
下面结合具体实施方式对本发明进行进一步的详细描述,给出的实施例仅为了阐明本发明,而不是为了限制本发明的范围。以下提供的实施例可作为本技术领域普通技术人员进行进一步改进的指南,并不以任何方式构成对本发明的限制。
下述实施例中的实验方法,如无特殊说明,均为常规方法。下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
下述实施例中的木霉(trichodermalentiforme)accc30425于2008年6月8日收藏于中国微生物菌种保藏管理委员会农业微生物中心(又称中国农业微生物菌种保藏管理中心,简称accc,地址:北京市海淀区中关村南大街12号,中国农业科学院农业资源与农业区划研究所,邮编100081),自该收藏日起公众可从中国微生物菌种保藏管理委员会农业微生物中心获得该菌株。
下述实施例中,脂肪酶表达载体pet30a(+)和感受态细胞e.colibl21(de3)为transgenbiotechco.(中国北京)产品。
实施例1、制备脂肪酶
1制备重组菌
1.1制备含有密码子优化基因的重组菌e.colibl21(de3)/pet30a(+)-rtlipk-his-y
根据木霉(trichodermalentiforme)accc30425全基因组测序技术注释获得的脂肪酶基因cdna,手动删除信号肽,得到tlipk基因,将该tlipk基因命名为tlipk-w基因。tlipk-w基因是将seqidno.3的第1-159位核苷酸缺失,并将seqidno.3的第1762-1785位核苷酸缺失,保持seqidno.3的其它核苷酸不变得到的dna分子。tlipk-w基因是木霉(trichodermalentiforme)accc30425的原始序列,没有进行密码子优化。tlipk-w基因编码氨基酸序列是seqidno.2的第54-587位的蛋白质(其名称为tlipk)。tlipk是野生型蛋白质。
选择合适酶切位点(ecori和xhoi),删除tlipk-w基因原有终止密码子,将所需合成的成熟肽链的dna序列信息,送往上海捷瑞生物公司,合成密码子优化的适合原核表达的核苷酸序列(合成产物)。将该合成产物用ecori和xhoi进行酶切,回收目的片段,用ecori和xhoi进行酶切pet30a(+),回收载体大片段;将回收的目的片段与回收的载体大片段连接,得到连接产物,将连接产物转化到感受态细胞e.colibl21(de3)中,得到重组大肠杆菌。分离该重组大肠杆菌中的重组质粒并测序。将测序结果表明是用核苷酸序列是seqidno.1的第151-1767位的dna分子替换pet30a(+)的ecori和xhoi识别位点间的片段(包括ecori识别位点和xhoi识别位点在内的小片段),保持pet30a(+)的其它序列不变,得到的重组表达载体命名为pet30a(+)-rtlipk-his-y。将含有pet30a(+)-rtlipk-his-y的重组大肠杆菌命名为e.colibl21(de3)/pet30a(+)-rtlipk-his-y。
pet30a(+)-rtlipk-his-y含有rtlipk-his基因,将该rtlipk-his基因命名为rtlipk-his-y基因,rtlipk-his-y基因的核苷酸序列是seqidno.1,是密码子优化基因,rtlipk-his-y基因编码氨基酸序列是seqidno.2的蛋白质(其名称为rtlipk-his);pet30a(+)-rtlipk-his-y含有rtlipk基因,将该rtlipk基因命名为rtlipk-y基因,rtlipk-y基因的核苷酸序列是seqidno.1的第157-1761位,是密码子优化基因,rtlipk-y基因编码氨基酸序列是seqidno.2的第53-587位的蛋白质(其名称为rtlipk);pet30a(+)-rtlipk-his-y含有tlipk基因,将该tlipk基因命名为tlipk-y基因,tlipk-y基因的核苷酸序列是seqidno.1的第160-1761位,是密码子优化基因,tlipk-y基因编码氨基酸序列是seqidno.2的第54-587位的蛋白质(其名称为tlipk)。
pet30a(+)-rtlipk-his-y在e.colibl21(de3)中可表达氨基酸序列是seqidno.2的蛋白质(其名称为rtlipk-his)。bl21(de3)/pet30a(+)-rtlipk-his-y可产生脂肪酶rtlipk-his。
1.2制备含有未进行密码子优化基因的重组菌e.colibl21(de3)/pet30a(+)-rtlipk-his-w
按照步骤1.1的方法构建含有pet30a(+)-rtlipk-his-w的重组大肠杆菌,命名为e.colibl21(de3)/pet30a(+)-rtlipk-his-w。
pet30a(+)-rtlipk-his-w是用核苷酸序列是seqidno.3的第151-1767位的dna分子替换pet30a(+)的ecori和xhoi识别位点间的片段(包括ecori识别位点和xhoi识别位点在内的小片段),保持pet30a(+)的其它序列不变,得到的重组表达载体命名为pet30a(+)-rtlipk-his-w。
pet30a(+)-rtlipk-his-w含有rtlipk-his基因,将该rtlipk-his基因命名为rtlipk-his-w基因,rtlipk-his-w基因的核苷酸序列是seqidno.3,是未进行密码子优化基因,rtlipk-his-w基因编码氨基酸序列是seqidno.2的蛋白质(其名称为rtlipk-his);pet30a(+)-rtlipk-his-w含有rtlipk基因,将该rtlipk基因命名为rtlipk-w基因,rtlipk-w基因的核苷酸序列是seqidno.3的第157-1761位,是未进行密码子优化基因,rtlipk-w基因编码氨基酸序列是seqidno.2的第53-587位的蛋白质(其名称为rtlipk);pet30a(+)-rtlipk-his-w含有tlipk基因,将该tlipk基因命名为tlipk-w基因,tlipk-w基因的核苷酸序列是seqidno.3的第160-1761位,是密码子优化基因,tlipk-w基因编码氨基酸序列是seqidno.2的第54-587位的蛋白质(其名称为tlipk)。
pet30a(+)-rtlipk-his-w在e.colibl21(de3)中可表达氨基酸序列是seqidno.2的蛋白质(其名称为rtlipk-his)。bl21(de3)/pet30a(+)-rtlipk-his-w可产生脂肪酶rtlipk-his。
1.3制备含有空载体的重组菌e.colibl21(de3)/pet30a(+)
将pet30a(+)转化到感受态细胞e.colibl21(de3)中,得到重组大肠杆菌。分离该重组大肠杆菌中的重组质粒并测序。将含有pet30a(+)的重组大肠杆菌命名为e.colibl21(de3)/pet30a(+),作为空载体对照重组菌。
2表达碱性超嗜热脂肪酶
将e.colibl21(de3)/pet30a(+)-rtlipk-his-y、e.colibl21(de3)/pet30a(+)-rtlipk-his-w和e.colibl21(de3)/pet30a(+)这三种菌株分别按0.5%的接种量单独接种于30ml的lb小瓶液体培养基(含有50μg/ml硫酸卡那霉素)之中,在37℃、220rpm的振荡摇床中培养活化12-16小时。然后取适量活化好的菌液按1%的接种量,接种到300ml的大瓶lb培养液(含50μg/ml硫酸卡那霉素)之中,在37℃、220rpm的振荡摇床中,连续培养2.5-3小时(利用紫外分光光度计确定培养菌液od600值为0.8,以含50μg/ml硫酸卡那霉素的lb液体培养基为空白对照),加入iptg(经过0.22μm滤膜过滤灭菌)至iptg的含量为0.6mm,在30℃、220rpm的振荡摇床中诱导培养6小时。将上述诱导培养菌液转移至离心杯,于4000rpm转速,离心10分钟,弃上清液,用5ml缓冲液重悬菌体,并回收菌体,将重悬菌体收集至10ml离心管内。在冰水浴条件下,用超声波细胞破碎仪对重悬混浊液进行超声波细胞破碎。设置破碎仪器功率为200w,超声波工作时间4秒,间隔时间3秒,破碎时间为30分钟。破碎完成后,立即于转速12,000rpm、4℃离心菌液10分钟,保留上清液,去除细胞碎片等杂质,该上清液即为诱导表达所获得的粗酶液。
将该粗酶液溶于ph为8.0的20mmtris-hcl缓冲溶液中,得到待测酶液。
采用对硝基苯酚(p-np)法测定待测酶液的脂肪酶活性。具体方法如下:配制p-np标准溶液(p-np的浓度为8mm,用ph为8.0的20mmtris-hcl缓冲溶液配制);将p-np标准溶液用ph为8.0的20mmtris-hcl缓冲溶液稀释成适当的梯度,分别测定吸光度,绘制吸光度-浓度关系曲线。取2.4ml底物溶液(称取适量对硝基苯酚棕榈酸酯p-npp,溶解于ph为8.0的20mmtris-hcl缓冲溶液,p-npp的浓度为0.8mm),37℃下预热5min后加入0.1ml待测酶液,37℃下反应15min后立即加入2.5ml无水乙醇混合均匀,放置5min终止反应;用0.1mlph为8.0的20mmtris-hcl缓冲溶液替换0.1ml待测酶液,其他条件不变,即得空白;于410nm测定吸光度。对照标准曲线算出生成的对硝基苯酚浓度,进而计算出酶活力。酶活定义以及计算公式:脂肪酶酶活力单位定义为:在37℃下,ph8.0的条件下,每分钟释放出1μmol对硝基苯酚的酶量定义为1个脂肪酶活力单位(u)。计算公式:x=cv/tvˊ(式中:x为脂肪酶活力,u/ml;c为对硝基苯酚浓度,μmol/l;v反应液终体积,ml;vˊ为待测酶液的用量,ml;t为作用时间,min。实验重复三次。根据待测酶液来自的菌体数量,计算e.colibl21(de3)/pet30a(+)-rtlipk-his-y、e.colibl21(de3)/pet30a(+)-rtlipk-his-w和e.colibl21(de3)/pet30a(+)的脂肪酶产量,结果表明bl21(de3)/pet30a(+)-rtlipk-his-y产生的脂肪酶rtlipk-his产量为0.13u/108cfubl21(de3)/pet30a(+)-rtlipk-his-y,e.colibl21(de3)/pet30a(+)-rtlipk-his-w产生的脂肪酶rtlipk-his产量为0.02u/108cfue.colibl21(de3)/pet30a(+)-rtlipk-his-w,e.colibl21(de3)/pet30a(+)不产脂肪酶。
bl21(de3)/pet30a(+)-rtlipk-his-y的脂肪酶rtlipk-his产量是e.coli
bl21(de3)/pet30a(+)-rtlipk-his-w的脂肪酶rtlipk-his产量的6.5倍。
说明与密码子优化前的基因rtlipk-his-w基因相比,密码子优化后的基因rtlipk-his-y基因使脂肪酶rtlipk-his产量提高了5.5倍;与密码子优化前的基因rtlipk-w基因相比,密码子优化后的基因rtlipk-y基因使脂肪酶rtlipk-his产量提高了5.5倍;与密码子优化前的基因tlipk-w基因相比,密码子优化后的基因tlipk-y基因使脂肪酶rtlipk-his产量提高了5.5倍。
实施例2、rtlipk-his是碱性超嗜热脂肪酶
将e.colibl21(de3)/pet30a(+)-rtlipk-his-y按0.5%的接种量接种于30ml的lb小瓶液体培养基(含有50μg/ml硫酸卡那霉素)之中,在37℃、220rpm的振荡摇床中培养活化12-16小时。然后取适量活化好的菌液按1%的接种量,接种到300ml的大瓶lb培养液(含50μg/ml硫酸卡那霉素)之中,在37℃、220rpm的振荡摇床中,连续培养2.5-3小时(利用紫外分光光度计确定培养菌液od600值为0.8,以含50μg/ml硫酸卡那霉素的lb液体培养基为空白对照),加入iptg(经过0.22μm滤膜过滤灭菌)至iptg的含量为0.6mm,在30℃、220rpm的振荡摇床中诱导培养6小时。将上述诱导培养菌液转移至离心杯,于4000rpm转速,离心10分钟,弃上清液,用5ml缓冲液重悬菌体,并回收菌体,将重悬菌体收集至10ml离心管内。在冰水浴条件下,用超声波细胞破碎仪对重悬混浊液进行超声波细胞破碎。设置破碎仪器功率为200w,超声波工作时间4秒,间隔时间3秒,破碎时间为30分钟。破碎完成后,立即于转速12,000rpm、4℃离心菌液10分钟,保留上清液,去除细胞碎片等杂质,该上清液即为诱导表达所获得的含有脂肪酶rtlipk-his的粗酶液。
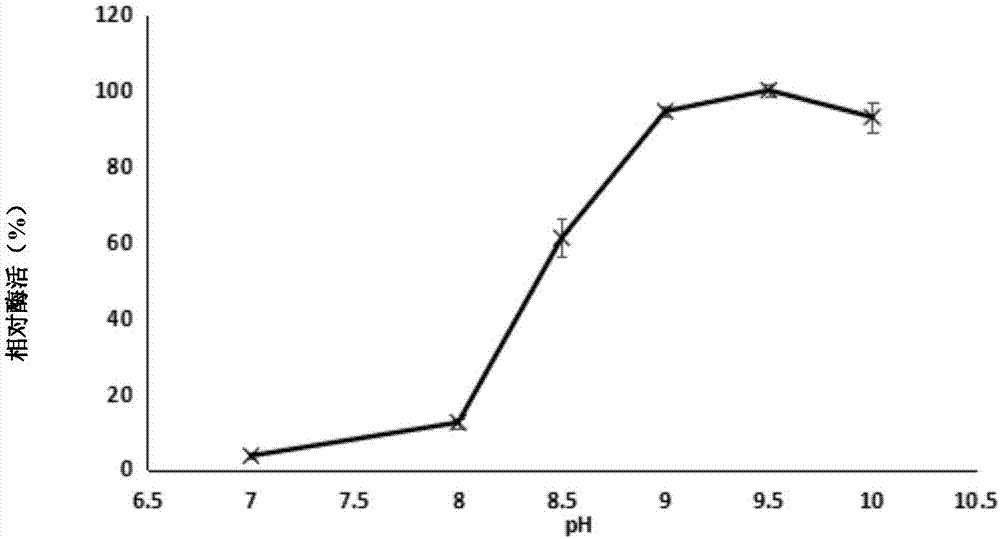
1、ph对脂肪酶rtlipk-his活性的影响
将含有脂肪酶rtlipk-his的粗酶液溶于ph值分别为7,8,8.5,9,9.5和10的20mmtris-hcl缓冲溶液中,得到相应ph值的待测酶液。
在37℃(待测温度)下,按照实施1的对硝基苯酚法在以下缓冲液中测定脂肪酶rtlipk-his活性的最适ph值:ph值为7、8、8.5、9、9.5或10的20mmtris-hcl。即用该缓冲溶液作为溶剂配制底物溶液和p-np标准溶液。其中,底物为对硝基苯酚棕榈酸酯(p-npp)。实验重复三次。脂肪酶酶活力单位定义为:在37℃下,ph9.5的条件下每分钟释放出1μmol对硝基苯酚的酶量定义为1个脂肪酶活力单位(u)。其它操作均与实施例1的对硝基苯酚法相同。
结果表明108cfubl21(de3)/pet30a(+)-rtlipk-his-y所产生的脂肪酶rtlipk-his在37℃,ph9.5的脂肪酶活力为1.07±0.02u。将脂肪酶rtlipk-his在37℃,ph值为9.5的脂肪酶活力定义为100%,来比较ph值对酶活的影响。脂肪酶rtlipk-his在中性与酸性条件下处于较差状态,同时从ph7开始稳定增加,直到ph达到最佳ph值为9.5。脂肪酶rtlipk-his在37℃、ph值为9时的相对酶活是94.43%,在37℃、ph值为10时的相对酶活是92.91%(图1)。说明脂肪酶rtlipk-his在37℃、ph值为9-10具有92%以上的酶活,是碱性脂肪酶。
2、温度对脂肪酶rtlipk-his活性的影响
将含有脂肪酶rtlipk-his的粗酶液溶于ph值为9.5(ph9.5)的20mmtris-hcl缓冲溶液中,得到待测酶液。
在ph值为9.5下,采用对硝基苯酚法测定待测酶液的脂肪酶酶活。
用ph9.5的20mmtris-hcl缓冲溶液作为溶剂配制底物溶液和p-np标准溶液。其中,底物为对硝基苯酚棕榈酸酯(p-npp)。
取2.4ml底物溶液,待测温度(分别为40℃,50℃,60℃,65℃,70℃,80℃和90℃)预热5min后加入0.1ml待测酶液,相应待测温度下反应15min后立即加入2.5ml无水乙醇混合均匀,放置5min终止反应;用0.1mlph9.5的20mmtris-hcl缓冲溶液替换0.1ml待测酶液,其他条件不变,即得空白;于410nm测定吸光度。对照标准曲线算出生成的对硝基苯酚浓度,进而计算出酶活力。实验重复三次。脂肪酶酶活力单位定义为:在80℃下,ph9.5的条件下每分钟释放出1μmol对硝基苯酚的酶量定义为1个脂肪酶活力单位(u)。
结果表明108cfubl21(de3)/pet30a(+)-rtlipk-his-y所产生的脂肪酶rtlipk-his在80℃,ph9.5的脂肪酶活力为1.27±0.02u。将脂肪酶rtlipk-his在80℃,ph值为9.5的脂肪酶活力定义为100%,来比较温度对酶活的影响。脂肪酶rtlipk-his的最适温度为80℃,其脂肪酶活性在低温条件下处于较差状态,随着温度的升高,从40℃开始,酶活随着温度提高逐渐增大,当温度达到80℃,酶活达到最高活性在最适温度的超高温区保持稳定,但由于超热失活,超过80℃后酶活开始降低,脂肪酶rtlipk-his在90℃、ph值为9.5时的相对酶活是90.62%(图2)。说明脂肪酶rtlipk-his在80-90℃、ph值为9.5具有90%以上的酶活,是碱性超嗜热脂肪酶。
以上对本发明进行了详述。对于本领域技术人员来说,在不脱离本发明的宗旨和范围,以及无需进行不必要的实验情况下,可在等同参数、浓度和条件下,在较宽范围内实施本发明。虽然本发明给出了特殊的实施例,应该理解为,可以对本发明作进一步的改进。总之,按本发明的原理,本申请欲包括任何变更、用途或对本发明的改进,包括脱离了本申请中已公开范围,而用本领域已知的常规技术进行的改变。按以下附带的权利要求的范围,可以进行一些基本特征的应用。
<110>中国农业科学院农业资源与农业区划研究所
<120>超嗜热脂肪酶lipk及其相关生物材料与应用
<130>gncfh181285
<160>3
<170>patentinversion3.5
<210>1
<211>1788
<212>dna
<213>人工序列(artificialsequence)
<400>1
atgcaccatcatcatcatcattcttctggtctggtgccacgcggttctggtatgaaagaa60
accgctgctgctaaattcgaacgccagcacatggacagcccagatctgggtaccgacgac120
gacgacaaggccatggctgatatcggatccgaattcatggaatgtctgccgcaggttgat180
ctgggctatgaaattcatgaagccctgaattataatgaaaccaccgatctgtataccttt240
agtaatattcgctatgcacagccgccggtgggtgatctgcgctttagcgccccgaaaccg300
ccgaccggtcgtaatcatgttgttcagaaaggtaatggctcaattattatgtgtccgcag360
ggtagtccgggttggggtagcgcaaatgcagattttggccatgcctttattaatggcaat420
ctgagtgattataattataccgcagaacatgcagcccaggaactgaccaccaaaaaaaat480
gcacagaccgtgctggaactgccgcagaccgaagattgtctgtttctggatgtggttgtt540
ccgcgtgcagtgtttgaacgtcgtaattctcgcgccaaaaaagccccggtgttagtgtgg600
atgcatggcggcggctatatttttggctataaaaatgatgttgtggatgccaccgatccg660
gcaggtctgattaatgcaagtcgtagcgatggctcagatggctttatttatgtgagctta720
aattatcgcttaggtgcctttggttggttagcaggcccgagcttagaagcagcaaatggt780
accgcaaatgcaggcttacatgatcagcatctggccttacagtgggttcagaaaaatatt840
catctgtttggcggcgatccgaaaaaagtgaccctgagcggcctgagcgcaggcggcggt900
agcaccgttcatcatattaccgcctatggtggcaaaaaaggtccgccgccgtttgcacgc960
gcctttattaatagtccgggctattttccgattggctctaattttcaggccgaaaatatt1020
acccagtattttctgaaatttctgaatgcaagctcaattgaacaggcacgtaaactgccg1080
gcacgcgcactgatggaagcaaatgttgcccatacctctagcgtggaaaccaattttgtg1140
tatggtccggcggtggatggcgattttgttccggccctgccgggcttaagtctgttgtct1200
ggtaattttcataaaaatattgatctgctgctgtcacataccctgtatgaaggcggcgaa1260
tatagtcagccgtttgttcagaatgataatgattttaaatcactgttacatcagctgtat1320
ccgggcattgtggatgaagttgccaattatattattaaagacctgtatccgggtaaaggt1380
gccgtgcgtacaatcgatttcatgggcgatagcatttttgcatgtaataccaattggtta1440
gcacgcgcctttgccaatcagacctataatatgcagtttaatatttctccgccgtttcat1500
gcctatgatgaatcatatatttggaaaggctatggtcgtaataattttggtgccccggtg1560
attgaatcactggccacctcttggcagggctatattaccaattttattaaaaccggcaat1620
ccgaatggtcgcggtttaccgccgtttccgcagcagggccagaatgcaagtgccaatatt1680
ctgaccccggatggcattattaccggtcaggatagtaccgcaggtcatcgctgtgaatgg1740
ttacagaaaggcctgtttattctcgagcaccaccaccaccaccactga1788
<210>2
<211>595
<212>prt
<213>人工序列(artificialsequence)
<400>2
methishishishishishisserserglyleuvalproargglyser
151015
glymetlysgluthralaalaalalysphegluargglnhismetasp
202530
serproaspleuglythraspaspaspasplysalametalaaspile
354045
glysergluphemetglucysleuproglnvalaspleuglytyrglu
505560
ilehisglualaleuasntyrasngluthrthraspleutyrthrphe
65707580
serasnileargtyralaglnproprovalglyaspleuargpheser
859095
alaprolysproprothrglyargasnhisvalvalglnlysglyasn
100105110
glyserileilemetcysproglnglyserproglytrpglyserala
115120125
asnalaasppheglyhisalapheileasnglyasnleuserasptyr
130135140
asntyrthralagluhisalaalaglngluleuthrthrlyslysasn
145150155160
alaglnthrvalleugluleuproglnthrgluaspcysleupheleu
165170175
aspvalvalvalproargalavalphegluargargasnserargala
180185190
lyslysalaprovalleuvaltrpmethisglyglyglytyrilephe
195200205
glytyrlysasnaspvalvalaspalathraspproalaglyleuile
210215220
asnalaserargseraspglyseraspglypheiletyrvalserleu
225230235240
asntyrargleuglyalapheglytrpleualaglyproserleuglu
245250255
alaalaasnglythralaasnalaglyleuhisaspglnhisleuala
260265270
leuglntrpvalglnlysasnilehisleupheglyglyaspprolys
275280285
lysvalthrleuserglyleuseralaglyglyglyserthrvalhis
290295300
hisilethralatyrglyglylyslysglyproproprophealaarg
305310315320
alapheileasnserproglytyrpheproileglyserasnphegln
325330335
alagluasnilethrglntyrpheleulyspheleuasnalaserser
340345350
ilegluglnalaarglysleuproalaargalaleumetglualaasn
355360365
valalahisthrserservalgluthrasnphevaltyrglyproala
370375380
valaspglyaspphevalproalaleuproglyleuserleuleuser
385390395400
glyasnphehislysasnileaspleuleuleuserhisthrleutyr
405410415
gluglyglyglutyrserglnprophevalglnasnaspasnaspphe
420425430
lysserleuleuhisglnleutyrproglyilevalaspgluvalala
435440445
asntyrileilelysaspleutyrproglylysglyalavalargthr
450455460
ileaspphemetglyaspserilephealacysasnthrasntrpleu
465470475480
alaargalaphealaasnglnthrtyrasnmetglnpheasnileser
485490495
proprophehisalatyraspglusertyriletrplysglytyrgly
500505510
argasnasnpheglyalaprovalilegluserleualathrsertrp
515520525
glnglytyrilethrasnpheilelysthrglyasnproasnglyarg
530535540
glyleupropropheproglnglnglyglnasnalaseralaasnile
545550555560
leuthrproaspglyileilethrglyglnaspserthralaglyhis
565570575
argcysglutrpleuglnlysglyleupheileleugluhishishis
580585590
hishishis
595
<210>3
<211>1788
<212>dna
<213>人工序列(artificialsequence)
<400>3
atgcaccatcatcatcatcattcttctggtctggtgccacgcggttctggtatgaaagaa60
accgctgctgctaaattcgaacgccagcacatggacagcccagatctgggtaccgacgac120
gacgacaaggccatggctgatatcggatccgaattcatggaatgccttcctcaagtagac180
ttgggatacgagatccatgaggcactcaactataacgagacgaccgacctctacaccttt240
agcaacattcgatatgcccagcctcctgtcggtgacctgcgcttctccgctccaaagcca300
ccaacaggccgaaatcatgtggtgcaaaagggcaatgggtcaataatcatgtgccctcag360
gggtcgccaggctggggcagcgcgaatgcggatttcggtcatgcgtttataaatggaaat420
ttgtctgattacaattacacagcagagcacgcagctcaggaattgaccacaaagaagaat480
gcacagactgtcctggagctcccccagacagaagattgcctgtttctcgacgtcgttgtt540
cctcgtgctgtgtttgagcgccgaaactcgagagctaagaaagctcctgtattggtctgg600
atgcatgggggcggctacatcttcggatacaaaaatgatgtcgttgatgctactgacccg660
gctggtcttatcaatgcaagccgaagcgatggctcagacggcttcatctatgtatcttta720
aactaccggcttggagcttttggctggcttgctgggccgtctcttgaagccgcaaatggc780
actgcaaatgccggcctccacgaccaacatcttgctcttcagtgggtgcagaagaacatt840
catctctttggaggagatccaaagaaggtgacgcttagcggcctttcggccggcggtggc900
agtacagttcatcatatcacagcatacggtggcaagaagggaccacctccctttgccaga960
gccttcatcaactctcctggatactttcccatcggctccaacttccaagccgaaaatatc1020
acacaatatttcctcaagtttcttaatgcgagttcaatcgagcaagctcgtaagctccca1080
gccagagctttgatggaggcaaacgttgcgcatacgtccagcgttgagaccaactttgtc1140
tacggtcccgcggttgatggagactttgtgcctgctttgccagggttatcgctcctttct1200
ggaaactttcacaaaaacatcgatttactgctcagtcatacgctgtatgaaggcggcgag1260
tattcgcagccttttgtgcagaacgacaatgacttcaaatccctccttcatcagctctac1320
ccaggcatcgtggatgaagtggccaattacatcatcaaagatctctatcctgggaaaggc1380
gcagtgcggaccattgattttatgggcgatagtatatttgcatgcaacacgaactggctt1440
gccagggcgtttgccaatcagacatacaacatgcagttcaacatcagcccccctttccat1500
gcatatgacgaatcgtacatctggaagggatacgggagaaataatttcggagctcccgta1560
atagagtcactggcaacatcttggcaaggatatatcaccaattttatcaagactggcaat1620
ccgaatggtcgtggcctgccgccattccctcagcaaggacagaacgcgagcgccaatatc1680
ttgactccagatggtatcatcactggacaagattccactgccgggcatagatgtgagtgg1740
ctacagaagggtctttttatcctcgagcaccaccaccaccaccactga1788
- 还没有人留言评论。精彩留言会获得点赞!