桃转录因子PpHBG7基因、蛋白、其重组表达载体及应用的制作方法
本发明涉及桃转录因子pphb.g7基因、蛋白、其重组表达载体及应用,属于植物基因工程领域。
背景技术
果实成熟是指在果实生长发育停止后发生的复杂有序的一系列生理生化反应,涉及色泽、风味、香气、质地等多方面,并且果实成熟直接影响到果品的商品价值、采后储藏和市场竞争力。(giovannonietal,2004;lietal,2010;田世平,2013)。果实成熟在番茄、苹果、香蕉、桃等中有广泛的研究。根据果实在成熟过程中是否出现出现呼吸峰,被分为呼吸跃变型和非呼吸跃变型(lelievrej,1997)。对于跃变型果实,果实成熟过程中会伴随着大量的乙烯释放,并且外源乙烯处理可促进果实成熟。
转录因子(transcriptionfactor,tf)是生物体内重要的调控因子,在植物生长发育和形态建成方面有着至关重要的作用(杨致荣等,2004),其能够激活或抑制基因的转录效应,调节基因的表达,或者响应环境胁迫和激素诱导等(liuetal.,2010;孙禹,2013)。转录因子一般由dna结合区、转录调控区(包括激活区或抑制区)、核定位信号和寡聚化位点4个功能区间组成。转录因子通过这些功能区域与启动子顺式元件作用或与其他转录因子的功能区域相互作用来调控基因的表达(刘强等,2000)。根据dna结合功能域的不同,可以将转录因子分为以下几类:mads-box结构域、亮氨酸拉链结构域(leucinezipper,lz)、碱性螺旋-环-螺旋结构域(bhlh)、zinc-finger结构域、myb结构域、ap2/erf结构域、同源异型域(hd)等(liujetal,1993;liuletal,2010)。
同源异型盒(homeobox,hb)蛋白含有典型的同源异型结构域(homedomain,hd),其主要是含有一段保守的dna序列,长度为183bp,编码61个氨基酸残基(mukherjeeketal,2006)。根据同源异型域的位置、与其他结构域的关联和基因结构等特征,将hb家族划分为hd-zip、phd、bell、wox、knox、ddt和sawadee家族(f.d.arieletal,2007;a.bhattacharjee,etal,2015;k.mukherjeeetal,2009)。vollbrecht等在玉米中克隆得到了一个与玉米叶节突变有关的基因knotted-1,knotted-1中存在一段与已报道的同源异型盒高度同源的dna基序(e.vollbrechtetal,1991)。knox家族中的基因参与调节叶发育(e.shanietal,2009)、分生组织活性(e.belles-boix,2006;a.t.groover,2006)、参与花序结构的形成(s.p.venglatetal,2002)、细胞分化(j.du,s.d.mansfield,etal,2009;g.mele,etal,2003)、以及促进细胞分裂素和赤霉素积累(t.sakamotoetal,2006;f.m.rosinetal,2003)。belll家族中的基因与茎生长(m.e.byrneetal,2003;c.gomez-menaetal,2008)、花发育(s.kanraretal,2008;m.proveniersetal,2007)、胚胎发生和无性繁殖(n.a.horst,2016)有关。此外,wox基因家族中的基因也参与了叶发育(m.dai,2007;m.nakata,2012)、调节分生组织活性(x.wuetal,2005)、促进细胞增殖(y.hirakawa,2010),影响果实的胚胎形成(m.romera-branchatetal,2003)、芽生冠根发育(y.zhaoetal,2009)和胁迫防御能力(s.chengetal,2016)。
hd-zip家族包含四个亚家族i、ii、iii和iv,所有基因都含有亮氨酸拉链结构域,并且与其他家族有着相似的功能作用(f.d.arieletal,2007;a.bhattacharjee,etal,2015;k.mukherjeeetal,2009)。据报道,在hd-zipi亚家族中,拟南芥athb1、athb3、athb13、athb20和athb23具有调节叶的发育作用(t.aoyamaetal,1995),athb4、athb6、athb7和athb12可以响应非生物胁迫(c.a.dezaretal,2005;a.s.olssonetal,2004;e.sodermanetal)。在hd-zipii亚家族中,向日葵hb10(hahb10)促进拟南芥早期开花(e.c.ruedaetal,2005)。在hd-zipiii中,athb15参与原形成层和木质部细胞的分化(k.ohashi-itoetal,2003),atphb和atphv直接诱导茎尖分生组织的发育和侧向器官极性(s.p.griggetal,2009)。在hd-zipiv中,atglabra2影响籽油的含量(b.shenetal,2006)。显然,除hd-zip-iv家族外,其他三个hd-zip亚家族中的成员在植物生长发育中的作用与其他家族具有重叠的功能。
目前,hb基因在果实发育和成熟过程中的研究甚少,仅报道了番茄lehb-1对果实成熟的调节作用。本研究首先对桃果实中的hb基因进行了系统发育树的构建和基因表达分析,以筛选出与果实成熟相关的候选基因。经qrt-pcr鉴定后,将候选基因瞬时转化到成熟前期的桃果实中,通过测定果实成熟度相关基因的表达水平和乙烯产量,对其基因功能进行评价。最后,用双荧光素酶和酵母单杂交法检测了hb基因与乙烯生物合成基因aco和acs的启动子的相互作用。本研究首次报道了桃果实中的hb基因,为进一步阐明果实成熟的分子调控网络提供了依据。
技术实现要素:
本发明目的是提供一种促进乙烯生物合成的转录因子pphb.g7基因,属于hb家族成员,其核苷酸序列如序列表seqidno.1所示,编码区序列cds长度为936bp,编码311个氨基酸,其编码的氨基酸序列为序列表seqidno.2,等电点为6.09,分子量为35.04kd。
本发明还提供了含有本发明所述pphb.g7基因的重组表达载体。
所述的重组表达载体,优选以pcambia1301为出发载体,所述pphb.g7基因的插入点为xbal和hindiii之间。
本发明还提供了含有本发明所述pphb.g7基因的宿主菌以及克隆本发明所述pphb.g7基因的cdna序列的引物对,上游引物pphb.g7-f1序列如seqidno.3所示,下游引物pphb.g7-r1序列如seqidno.4所示。
本发明的另一目的是提供该基因的应用。
包括pphb.g7基因在促进桃乙烯合成中的应用。
以及重组表达载体在促进桃乙烯合成中的应用。
通过软件分析并结合转录组数据构建系统发育树并对相关基因进行qrt-pcr分析。
利用qrt-pcr技术分析,pphb.g7与乙烯生物合成有关。
构建pphb.g7超表达载体和沉默载体,通过农杆菌介导瞬时转化桃果实中表明,pphb.g7的超表达和沉默分别增加和减少ppacs1,ppaco1基因的表达水平和乙烯产量。
利用双荧光素酶报告基因系统分析了pphb.g7和ppacs1,ppaco1基因的互作关系,结果表明pphb.g7和ppacs1,ppaco1基因的启动子相互作用。
利用酵母单杂交分析揭示了pphb.g7通过结合ppacs1和ppaco1的启动子介导其表达。
与现有技术比,本发明具有以下优点和效果:
pphb.g7基因的发现,为促进乙烯合成的分子育种提供新的基因资源,为实施绿色农业提供新的遗传资源,该资源的开发利用有利提供桃果实的商品价值,延长果实货架期,有利于降低农业成本和实现环境友好。
附图说明
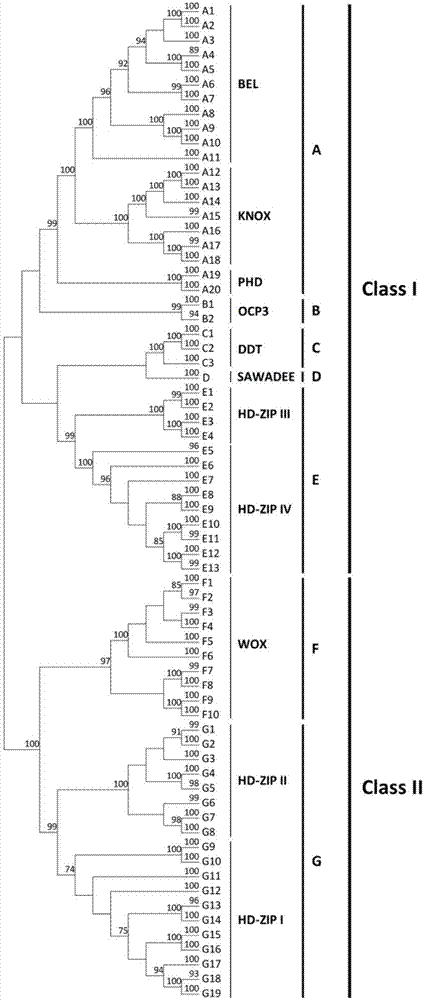
图1为hb基因家族图谱,398个hb基因分为两个类i和ii,它们分别为5个组(a~e)和2个组(f和g)。除d组外又进一步被分为67个簇,分别是a1→a20、b1、b2、c1→c3、e1→e13、f1→f10和g1→g19。
图2a为南山甜桃、朝晖两个桃品种的热图分析,两个基因pphb.g2和pphb.g7在果实成熟期间中的表达高于果实发育期间,而五个基因pphb.a11、pphb.e1、pphb.e12、pphb.e13和pphb.f4在果实成熟期间表达水平最低。
图2b为pphb.a11、pphb.e12、pphb.e13在石质硬化(s2)、预熟(s3)、成熟(s4)阶段表达量差异并不显著,而其他四个基因pphb.g2、pphb.g7、pphb.e1、pphb.f4与转录组测序数据有相似的表达模式。
图3为hd-zipi(g9→g19)亚家族的11个hb基因。
图4为hd-zipii(g1→g8)亚家族的8个hb基因。
图5为10个桃品种中pphb.g7的表达量在果实成熟期中均有较高的表达水平。在多数品种中pphb.g2的表达量在果实成熟前期与成熟果实期之间略有差异。
图6a为本发明克隆基因pphb.g7超表达和沉默载体分别通过农杆菌瞬时转化侵染桃果实后乙烯释放量分析示意图。
图6b为本发明克隆基因pphb.g7超表达和沉默载体分别通过农杆菌瞬时转化侵染桃果实后pphb.g7和ppacs1、ppaco1的qrt-pcr表达量分析图。小写字母a、b、c代表p值<0.05的显著差异。
图7a为双荧光素酶报告基因系统分析克隆基因pphb.g7对ppacs1、ppaco1启动子的调节作用,ppacs1和ppaco1启动子驱动的luc基因在拟南芥原生质体中表达量高于对照组,小写字母a和b代表p值<0.05的显著差异。
图7b为酵母单杂实验结果,表明克隆基因pphb.g7和ppacs1启动子的片段1和片段2相互作用。
图7c为酵母单杂实验结果,表明克隆基因pphb.g7和ppaco1启动子的片段3和片段4相互作用。
具体实施方式
以下结合具体实施例对本发明做出详细的描述。根据以下描述和实施例,本领域技术人员可以确定本发明的基本特征,并且在不偏离本发明精神和范围的情况下,可以对本发明做出各种改变和修改,以使其适用各种用途和条件。
实施例1桃和其他果树hb家族的鉴定
根据桃基因组(第1版)分离出大量的hb基因,并从蔷薇科基因组数据库(gdr;http://www.rosaceae.org/)中下载其核苷酸和氨基酸序列,将所获得的氨基酸序列进行筛选,获得桃基因组中hb家族的成员。同时对梨(pyrusbretschneideri;http://peargenome.njau.edu.cn/)、树莓((rubusoccidentalis;gdr)、草莓(fragaria×ananassa)、柑橘(citrussinensis)、葡萄(vitisvinifera)、番木瓜(caricapapaya)等基因库中搜索并下载hb家族所有成员。最后所有搜到的基因在美国国家生物技术信息中心(ncbi;https://www.ncbi.nlm.nih.gov))中进一步比对其同源性。
实施例2序列和系统发育树分析
将上述筛选后基因的氨基酸序列下载,并通过culstalx方式对齐,使用软件mega6构建基于兆加权邻接(nj)的系统发生树。从桃基因组中分离到70个hb基因,并将其与梨、草莓、柑橘、番木瓜和葡萄中的hb基因一起构建系统进化树。结果表明:398个hb基因可分为两个类i和ii,它们分别有5个组(a~e)和2个组(f和g)(图1)。这些基因除d组外又进一步被分为67个簇,分别是a1→a20、b1、b2、c1→c3、e1→e13、f1→f10和g1→g19。ncbi中序列比对结果表明a组包含以前报道的三个家族bel(a1→a11)、knox(a12→a18)和phd(a19和a20)。c、d和f组分别与ddt、sawdee和wox家族相似。e组包含两个亚家族hd-zipiii和iv,g组包含hd-zipi和ii。另外b组中的hb基因与aba和ja诱导的防御功能相关(j.garcía-andradeetal,2011;v.ramírez,aetal,2009;v.ramírez,setal,2009)。为了便于对桃中hb基因的描述,将所有70个基因基于聚类编码命名(如表1所示),例如g7簇中的hb基因被命名为pphb.g7。
表1桃中70个hb基因聚类编码命名表
实施例3hb基因在桃果实中的表达谱
根据转录组数据,采用白香露、早白凤、迟园蜜、春美、霞晖2号、雨花露、春雪、白凤、湖景蜜露、霞晖6号10个品种,在果实发育期(s1-s3)和成熟(s4)期转录水平上分析70个基因的表达水平。结果表明:两个基因pphb.g2和pphb.g7在果实成熟期间中的表达高于果实发育期间,而五个基因pphb.a11、pphb.e1、pphb.e12、pphb.e13和pphb.f4在果实成熟期间表达水平最低(图2a)。为了验证这7个基因的表达量,采用qrt-pcr技术检测了这7个基因在中油4号桃果实发育期和果实成熟期的表达水平。结果表明,pphb.a11、pphb.e12、pphb.e13在石质硬化(s2)、预熟(s3)、成熟(s4)阶段表达量差异并不显著。而其他四个基因pphb.g2、pphb.g7、pphb.e1、pphb.f4与转录组测序数据有相似的表达模式(图2b)。因此,在桃果实中pphb.g2、pphb.g7、pphb.e1和pphb.f4四个基因可能与果实成熟有关。
实施例4验证pphb.g7参与果实成熟
lehb-1已经被报道参与了番茄果实成熟的调控(z.f.linetal,2008),但hd-zipi亚家族中的桃hb基因(g9→g19)(图3)明显与果实成熟不相关(图2)。hd-zipii亚家族的pphb.g2(ppa007421m)和pphb.g7(ppa008984m(图4)可能参与桃果实的成熟。为了进一步验证pphb.g2、pphb.g7与桃果实成熟的关系,对另外10个桃品种的成熟前期和成熟时期的果实进行了qrt-pcr检测。因此,所有10个桃品种中pphb.g7的表达量均在果实成熟期中有较高的表达水平。而pphb.g2在多数品种表达量在果实成熟前期与成熟果实之间略有差异(图5)。结果表明,pphb.g7与桃果实成熟有关。
实施例5pphb.g7基因分离克隆与载体构建
1、采用多酚多糖类物质的植物总rna提取试剂盒(购自tiangen公司,按照试剂盒说明书操作)从桃的果肉中抽提rna,经反转录得到的第一链cdna用于扩增pphb.g7基因全长。50ul的反应体系中包括2ul上述引物,17ulddh2o,25ul2×buffer,1uldntp,2ulcdna模板,1ul高保真酶(phantasuper-fidelitydnapolymerase)(购自vazyme公司)。pcr反应在eppendorpcr扩增仪上按以下程序完成:95℃,预变性3分钟,95℃变性30秒,60℃退火30秒,72℃延伸1分钟,30个热循环,循环完成后72℃延伸10分钟,20℃5分钟。
2、pcr产物经1%琼脂糖凝胶电泳后,将产生的目的条带按照axyprepdna凝胶回收试剂盒说明书操作回收。回收纯化后的pcr产物和已用xbai和hindiii双酶切完成的pcambia1301通过重组酶进行重组(购自vazyme公司),重组体系按照一步克隆重组试剂盒说明书操作。将重组产物采用热击法(参照《分子克隆实验手册》第三版,科学出版社,2002)转化大肠杆菌dh5ɑ,均匀涂在含有50mg/l卡娜霉素的lb固体平板上,筛选阳性克隆,挑取6个阳性克隆测序。测序结果表明,本发明扩增的目的片段长度为936bp,其核苷酸序列为序列表seqidno.1所示。通过序列比对分析,确定该序列是本发明需要的目的基因。
实施例6pphb.g7基因超表达和沉默载体的构建
1、根据psak277载体的多克隆位点和pphb.g7基因的编码区序列上的酶切位点分析,选择xbai和hindiii作为内切酶。按照一般设计引物原则用primer5.0软件设计出带有酶切位点的引物,其引物对序列如下所示:
pphb.g7-f1:tttaagcttagagagagatgagttttgatgaag(seqidno.3)
pphb.g7-r1:ttttctagatttagcaagctgtagatggatgggt(seqidno.4)
以测序正确的保存菌液提取质粒为模板,进行含有限制性内切酶位点基因的克隆。pcr扩增的退火温度为60℃,pcr反应体系及扩增程序同实施例1。psak277载体用和两个酶(购自neb公司)进行双酶切,酶切总体系为50ul:载体质粒5ul,cutsmartbuffer缓冲液5ul,xbai和hindiii各1ul,ddh2o38ul。放于37℃酶切3-4小时,酶切产物用axyprep清洁试剂盒进行纯化。(操作过程按照说明书进行)。将pcr产物和纯化后双酶切产物重组连接,并转化大肠杆菌dh5ɑ,均匀涂在含有50mg/l卡那霉素的lb固体平板上,筛选阳性克隆,抽提质粒进行酶切及pcr鉴定,测序结果确定没有读码框突变,获得含有插入片段的重组载体,将其命名为超表达载体pcambia1301-pphb.g7。应用冻融法将重组质粒导入农杆菌eha105中。
2、根据trv载体的多克隆位点和pphb.g7基因的编码区序列上的酶切位点分析,选择kpni和ecori作为内切酶。设计引物方法和构建trv-pphb.g7沉默载体方法均同上。并将沉默载体质粒利用电激法转入农杆菌gv3101中。
设计出带有酶切位点的引物,其引物对序列如下所示:
trv-f2:accgaattcacaccaccctcaatcccaagc(seqidno.5)
trv-r2:ctcggtacctttagcaagctgtagatggatgggt(seqidno.6)
实施例7桃果实的瞬时转化
1、分别挑取含超表达载体psak277-pphb.g7的农杆菌单菌落于含有卡那霉素和利福平抗性的液体培养基中、沉默载体trv-pphb.g7的农杆菌单菌落于含有壮观霉素和利福平抗性的液体培养基中,同时以含空载体psak277和trv1+trv2的农杆菌为对照。28℃,220rpm培养24-48h。然后,将各农杆菌在含40ml液体培养基的三角瓶中扩大培养至od600为0.8-1.0。离心机4℃条件下,5000g,离心10分钟,弃上清收集菌液,然后重新悬浮于侵染液中(10mmmgcl2,10mmmes,ph=5.7,200μm乙酰丁香酮),5000g,离心10分钟,弃上清收集菌液,并重复一次。最后用侵染液悬浮放置ty-200s摇床(南京普阳)每分钟60rpm摇3h,静置1h。
2、取上述各菌液在商品采收前7天用1ml注射器注射桃果实霞晖8号。所有的分析都是通过三个生物学重复获得的,每个生物学重复至少包含四个桃果实。
实施例8乙烯测量
使用乙烯探测器测量了每个被侵染的果实乙烯的释放量(购自深圳普利通电子科技有限公司)。在室温下至少六个已被侵染的桃果实被独立地放入一个3升的密闭罐中3小时,然后用软木塞把罐子密封起来,用软管输送气体。在对乙烯检测器进行零校准后,探头与软管连接以接收释放的气体。测定释放的乙烯产量,并将浓度显示在监视器上。结果显示注射含pphb.g7超表达载体的桃果实乙烯的释放量高于注射含pphb.g7沉默载体的桃果实乙烯的释放量(图6a),结果表明pphb.g7具有促进乙烯合成的作用。
实施例9qrt-pcr分析瞬时转化桃果实中pphb.g7、ppacs1和ppaco1的表达水平
取细菌覆盖的果肉进行qrt-pcr检测。采用多酚多糖类物质的植物总rna提取试剂盒(购自tiangen公司,按照试剂盒说明书操作)从案例3中被侵染的桃果肉中抽提rna,经反转录得到cdna。qrt-pcr是在ligthtcycler480ii/96热循环中进行的。试剂盒是lightcycler480sybrgreenimaster及qpcr96孔板均有roche公司提供。qpcr反应体系:sybrgreenimaster10ul,ddh204ul,正向引物2.5ul,反向引物2.5ul,cdna1ul。反应程序为:95℃变性15s,60℃退火30s,45个循环。所有分析均由三个独立的生物学重复完成。桃ppa008668m基因作为内参基因。所有引物序列都在表2中。qrt-pcr结果显示在注射含pphb.g7超表达载体的果肉中ppacs1和ppaco1的表达量上升,在注射含pphb.g7沉默载体的果肉中ppacs1和ppaco1的表达量下降(图6b)。这些结果表明,pphb.g7可以通过刺激ppacs1和ppaco1的活性来介导乙烯生物合成。
实施例10原生质体分离和双荧光素酶测定
1、从桃果肉中扩增acs和aco启动子密码子上游2000bp序列,扩增产物插入到pgrilii0800-luc载体中,并构建pgrilii0800-luc-acs和pgrilii0800-luc-aco两个载体。构建载体的方法同实施案例2。
扩增引物序列为
ppacs1-f:tgggtacctgcagtatgtccgttccttggc(seqidno.7)
ppacs1-r:tcaagcttggttccaaagaatactcacacacaag(seqidno.8)
ppaco1-f:tgggtaccaagagacatatcaggtgatgaaagaacg(seqidno.9)
ppaco1-r:tcaagcttgtgaagtggagtttggtgtgg(seqidno.10)
2、从3至5周龄的每天光照8小时的植物中提取的原生质体。将重组质粒转入原生质体暗培养18小时。使用双荧光素酶
实施例11酵母单杂交
从桃果肉中克隆出acs和aco基因启动子中克隆了2条约2000bp的序列。并将启动子分成小片段分别连接pabai载体中。同时,将分离的pphb.g7基因的全长序列插入到pgadt7载体中。酵母单杂交(y1h)检测使用酵母单杂交文库筛选系统进行。结果表明pphb.g7通过结合ppacs1和ppaco.a1的启动子介导其表达(图7b、c)。所有引物均在表2中列出。
表2引物序列列表
参考文献
[1]giovannoni,j.j.geneticregulationoffruitdevelopmentandripening.plantcell16suppl.suppl_1(2003):s170.
[2]xianli,changjiexu,schuylers.korban.regulatorymechanismsoftexturalchangesinripeningfruits.criticalreviewsinplantsciences29.4(2010):222-243.
[3]田世平.果实成熟和衰老的分子调控机制.植物学报48.5(2013):481-488.
[4]jean‐marclelièvre,alainlatchè,brianjones,mondherbouzayen,jean‐claudepech.ethyleneandfruitripening.physiologiaplantarum101.4(2006):727-739.
[5]杨致荣,王兴春,李西明.高等植物转录因子的研究进展.遗传26.3(2004):403-408..
[6]liul,whitemj,macraeth.transcriptionfactorsandtheirgenesinhigherplantsfunctionaldomains,evolutionandregulation.[j].europeanjournalofbiochemistry,2010,262(2):247-257,febsjournal262.2(2010):247-257.
[7]孙禹.水稻hb转录因子家族基因克隆与oshb40、oshb102功能初步研究.diss.吉林大学,2013
[8]刘强,张贵友."植物转录因子的结构与调控作用."科学通报45.14(2000):1465-1474.
[9]jingdongliu,thomase.wilson,jeffreymilbrandt."identifyingdna-bindingsitesandanalyzingdna-bindingdomainsusingayeastselectionsystem."methods5.2(1993):125–137.
[10]mukherjee,k,andt.r.bürglin."mekhla,anoveldomainwithsimilaritytopasdomains,isfusedtoplanthomeodomain-leucinezipperiiiproteins."plantphysiology140.4(2006):1142-1150.
[11]f.d.ariel,p.a.manavella,c.a.dezar,r.l.chan,thetruestoryofthehd-zipfamily,trendsplantsci.12(2007)419-426.
[12]a.bhattacharjee,r.ghangal,r.garg,m.jain,genome-wideanalysisofhomeoboxgenefamilyinlegumes:identification,geneduplicationandexpressionprofiling,plosone10(2015)e0119198.
[13]k.mukherjee,l.brocchieri,t.r.burglin,acomprehensiveclassificationandevolutionaryanalysisofplanthomeoboxgenes,mol.biol.evol.26(2009)2775-2794.
[14]e.vollbrecht,b.veit,n.sinha,s.hake,thedevelopmentalgeneknotted-1isamemberofamaizehomeoboxgenefamily,nature350(1991)241-243
[15]e.shani,y.burko,l.ben-yaakov,y.berger,z.amsellem,a.goldshmidt,e.sharon,n.ori,stage-specificregulationofsolanumlycopersicumleafmaturationbyclass1knotted1-likehomeoboxproteins,plantcell21(2009)3078-3092.
[16]e.belles-boix,o.hamant,s.m.witiak,h.morin,j.traas,v.pautot,knat6:anarabidopsishomeoboxgeneinvolvedinmeristemactivityandorganseparation,plantcell18(2006)1900-1907.
[17]a.t.groover,s.d.mansfield,s.p.difazio,g.dupper,j.r.fontana,r.millar,y.wang,thepopulushomeoboxgenearborknox1revealsoverlappingmechanismsregulatingtheshootapicalmeristemandthevascularcambium,plantmol.biol.61(2006)917-932.
[18]s.p.venglat,t.dumonceaux,k.rozwadowski,l.parnell,v.babic,w.keller,r.martienssen,g.selvaraj,r.datla,thehomeoboxgenebrevipedicellusisakeyregulatorofinflorescencearchitectureinarabidopsis,procnatlacadsciusa99(2002)4730-4735.
[19]j.du,s.d.mansfield,a.t.groover,thepopulushomeoboxgenearborknox2regulatescelldifferentiationduringsecondarygrowth,plantj.60(2009)1000-1014
[20]g.mele,n.ori,y.sato,s.hake,theknotted1-likehomeoboxgenebrevipedicellusregulatescelldifferentiationbymodulatingmetabolicpathways,genesdev,17(2003)2088-2093.
[21]t.sakamoto,h.sakakibara,m.kojima,y.yamamoto,h.nagasaki,y.inukai,y.sato,m.matsuoka,ectopicexpressionofknotted1-likehomeoboxproteininducesexpressionofcytokininbiosynthesisgenesinrice,plantphysiol.142(2006)54-62.
[22]f.m.rosin,j.k.hart,h.t.horner,p.j.davies,d.j.hannapel,overexpressionofaknotted-likehomeoboxgeneofpotatoaltersvegetativedevelopmentbydecreasinggibberellinaccumulation,plantphysiol.132(2003)106-117.
[23]m.e.byrne,a.t.groover,j.r.fontana,r.a.martienssen,phyllotacticpatternandstemcellfatearedeterminedbythearabidopsishomeoboxgenebellringer,dev.130(2003)3941-3950.
[24]c.gomez-mena,r.sablowski,arabidopsisthalianahomeoboxgene1establishesthebasalboundariesofshootorgansandcontrolsstemgrowth,plantcell20(2008)2059-2072
[25]s.kanrar,m.bhattacharya,b.arthur,j.courtier,h.m.smith,regulatorynetworksthatfunctiontospecifyflowermeristemsrequirethefunctionofhomeoboxgenespennywiseandpound-foolishinarabidopsis,plantj.54(2008)924-937.
[26]m.proveniers,b.rutjens,m.brand,s.smeekens,thearabidopsistalehomeoboxgeneath1controlsfloralcompetencythroughpositiveregulationofflc,plantj.52(2007)899-913.
[27]n.a.horst,a.katz,i.pereman,e.l.decker,n.ohad,r.reski,asinglehomeoboxgenetriggersphasetransition,embryogenesisandasexualreproduction,nat.plants2(2016)15209.
[28]m.dai,y.hu,y.zhao,h.liu,d.x.zhou,awuschel-likehomeoboxgenerepressesayabbygeneexpressionrequiredforriceleafdevelopment,plantphysiol.144(2007)380-390.
[29]m.nakata,n.matsumoto,r.tsugeki,e.rikirsch,t.laux,k.okada,rolesofthemiddledomain-specificwuschel-relatedhomeoboxgenesinearlydevelopmentofleavesinarabidopsis,plantcell24(2012)519-535.
[30]x.wu,t.dabi,d.weigel,requirementofhomeoboxgenestimpy/wox9forarabidopsismeristemgrowthandmaintenance,curr.biol.15(2005)436-440.
[31]y.hirakawa,y.kondo,h.fukuda,tdifpeptidesignalingregulatesvascularstemcellproliferationviathewox4homeoboxgeneinarabidopsis,plantcell22(2010)2618-2629.
[32]m.romera-branchat,j.j.ripoll,m.f.yanofsky,s.pelaz,thewox13homeoboxgenepromotesreplumformationinthearabidopsisthalianafruit,plantj.73(2013)37-49.
[33]y.zhao,y.hu,m.dai,l.huang,d.x.zhou,thewuschel-relatedhomeoboxgenewox11isrequiredtoactivateshoot-bornecrownrootdevelopmentinrice,plantcell21(2009)736-748.
[34]s.cheng,d.x.zhou,y.zhao,wuschel-relatedhomeoboxgenewox11increasesricedroughtresistancebycontrollingroothairformationandrootsystemdevelopment,plantsignalbehav.11(2016)e1130198.
[35]t.aoyama,c.h.dong,y.wu,m.carabelli,g.sessa,i.ruberti,g.morelli,n.h.chua,ectopicexpressionofthearabidopsistranscriptionalactivatorathb-1altersleafcellfateintobacco,plantcell7(1995)1773-1785.
[36]c.a.dezar,g.m.gago,d.h.gonzalez,r.l.chan,hahb-4,asunflowerhomeobox-leucinezippergene,isadevelopmentalregulatorandconfersdroughttolerancetoarabidopsisthalianaplants,transgenicres.14(2005)429-440
[37]a.s.olsson,p.engstrom,e.soderman,thehomeoboxgenesathb12andathb7encodepotentialregulatorsofgrowthinresponsetowaterdeficitinarabidopsis,plantmol.biol.55(2004)663-677.
[38]e.soderman,m.hjellstrom,j.fahleson,p.engstrom,thehd-zipgeneathb6inarabidopsisisexpressedindevelopingleaves,rootsandcarpelsandup-regulatedbywaterdeficitconditions,plantmol.biol.40(1999)1073-1083.
[39]e.c.rueda,c.a.dezar,d.h.gonzalez,r.l.chan,hahb-10,asunflowerhomeobox-leucinezippergene,isregulatedbylightqualityandquantity,andpromotesearlyfloweringwhenexpressedinarabidopsis,plantcellphysiol.46(2005)1954-1963.
[40]k.ohashi-ito,h.fukuda,hd-zipiiihomeoboxgenesthatincludeanovelmember,zehb-13(zinnia)/athb-15(arabidopsis),areinvolvedinprocambiumandxylemcelldifferentiation,plantcellphysiol.44(2003)1350-1358.
[41]s.p.grigg,c.galinha,n.kornet,c.canales,b.scheres,m.tsiantis,repressionofapicalhomeoboxgenesisrequiredforembryonicrootdevelopmentinarabidopsis,curr.biol.19(2009)1485-1490.
[42]b.shen,k.w.sinkevicius,d.a.selinger,m.c.tarczynski,thehomeoboxgeneglabra2affectsseedoilcontentinarabidopsis,plantmol.biol.60(2006)377-387.
[43]]c.gu,l.wang,w.wang,h.zhou,b.ma,h.zheng,t.fang,c.ogutu,s.vimolmangkang,y.han,copynumbervariationofageneclusterencodingendopolygalacturonasemediatesfleshtextureandstoneadhesioninpeach,j.exp.bot.67(2016)1993-2005.
[44]j.garcía-andrade,v.ramírez,v.flors,p.vera,arabidopsisocp3mutantrevealsamechanismlinkingabaandjatopathogen-inducedcallosedeposition,plantj.67(2011)783-794.
[45]v.ramírez,a.coego,a.lópez,a.agorio,v.flors,p.vera,droughttoleranceinarabidopsisiscontrolledbytheocp3diseaseresistanceregulator,plantj.58:(2009a)578-591.
[46]v.ramírez,s.vanderent,j.garcía-andrade,a.coego,c.m.pieterse,p.vera,ocp3isanimportantmodulatorofnpr1-mediatedjasmonicacid-dependentinduceddefensesinarabidopsis,bmcplantbiol.10(2009b)199.
序列表
<110>江苏省农业科学院
<120>桃转录因子pphbg7基因、蛋白、其重组表达载体及应用
<160>10
<170>siposequencelisting1.0
<210>1
<211>936
<212>dna
<213>碧桃(prunuspersica)
<400>1
atgagttttgatgaagcttgtaacaccggccttggtctcagtctaggctgccgtgacagc60
ccagatcatgaccatacaaatttgcaggctcctcctgatcatcatcatcaccatcaccaa120
aacacgtacaagaacaagcaaatacagttgaaatatgatcacctattgccttctcttacg180
ctcggcccatcggctgcttcaaagattgaggaggccgagtccactgatttgcaccaggca240
gtagccgcacaggaacaggactcttctccctgcagcggcgccttttcatctttttctaac300
tcgtcaagtttcaagagggacagagagttgggaggtgaagagatagaggtagaggtagag360
gtagaggtagaggtagaggaagagagagtggtgaatattacttcctcaagagcaagcgag420
gagttatatgaacaagatcatgagggcagccccagaaagaagctcaggctttcaaaagaa480
caatctgccactttggaagacagcttcagagaacacaccaccctcaatcccaagcaaaag540
caagacctggcaagaaagctaaatctacggccgcgacaagtggaagtctggttccaaaac600
aggagggccaggaccaagttgaagcaaactgaagcggattgtgagttgttgaaaaagtgt660
tgtgagacgctgaaagaagagaacagaaggctacacaaggagctgcaagagctcaagtta720
atgaaacaaacggcaacagcagcagcagcagtaccacccttttacatgcagtttccaaca780
gccactctcaccatgtgcccttcttgtgagaaaatctgcaacggcggtgaccaccataat840
catcatgggtcgtcgacgagttcttttttgattggatcgaagccggctcacttgatcttc900
aacccctttagcacccatccatctacagcttgctaa936
<210>2
<211>311
<212>prt
<213>碧桃(prunuspersica)
<400>2
metserpheaspglualacysasnthrglyleuglyleuserleugly
151015
cysargaspserproasphisasphisthrasnleuglnalapropro
202530
asphishishishishishisglnasnthrtyrlysasnlysglnile
354045
glnleulystyrasphisleuleuproserleuthrleuglyproser
505560
alaalaserlysilegluglualagluserthraspleuhisglnala
65707580
valalaalaglngluglnaspserserprocysserglyalapheser
859095
serpheserasnserserserphelysargasparggluleuglygly
100105110
glugluilegluvalgluvalgluvalgluvalgluvalglugluglu
115120125
argvalvalasnilethrserserargalaserglugluleutyrglu
130135140
glnasphisgluglyserproarglyslysleuargleuserlysglu
145150155160
glnseralathrleugluaspserphearggluhisthrthrleuasn
165170175
prolysglnlysglnaspleualaarglysleuasnleuargproarg
180185190
glnvalgluvaltrppheglnasnargargalaargthrlysleulys
195200205
glnthrglualaaspcysgluleuleulyslyscyscysgluthrleu
210215220
lysglugluasnargargleuhislysgluleuglngluleulysleu
225230235240
metlysglnthralathralaalaalaalavalproprophetyrmet
245250255
glnpheprothralathrleuthrmetcysprosercysglulysile
260265270
cysasnglyglyasphishisasnhishisglyserserthrserser
275280285
pheleuileglyserlysproalahisleuilepheasnpropheser
290295300
thrhisproserthralacys
305310
<210>3
<211>34
<212>dna
<213>人工序列(artificialsequence)
<400>3
tttaagcttagagagagatgagttttgatgaagc34
<210>4
<211>34
<212>dna
<213>人工序列(artificialsequence)
<400>4
ttttctagatttagcaagctgtagatggatgggt34
<210>5
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>5
accgaattcacaccaccctcaatcccaagc30
<210>6
<211>34
<212>dna
<213>人工序列(artificialsequence)
<400>6
ctcggtacctttagcaagctgtagatggatgggt34
<210>7
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>7
tgggtacctgcagtatgtccgttccttggc30
<210>8
<211>34
<212>dna
<213>人工序列(artificialsequence)
<400>8
tcaagcttggttccaaagaatactcacacacaag34
<210>9
<211>36
<212>dna
<213>人工序列(artificialsequence)
<400>9
tgggtaccaagagacatatcaggtgatgaaagaacg36
<210>10
<211>29
<212>dna
<213>人工序列(artificialsequence)
<400>10
tcaagcttgtgaagtggagtttggtgtgg29
- 还没有人留言评论。精彩留言会获得点赞!