一种N端序列元件在调控枯草芽孢杆菌表达蛋白中的应用的制作方法
本发明涉及一种可静态和动态调控枯草芽孢杆菌目的蛋白表达量的n端序列元件及应用,属于基因工程领域。
背景技术:
枯草芽孢杆菌(bacillussubtilis)是革兰氏阳性菌的代表,其不产生内毒素,是非致病的微生物,被美国食品药品监督管理局(fda)认证是安全的(generallyrecognizedassafe,gras)食品级微生物。作为实验室中最常用的出发菌株,也作为在工业酶制剂生产、高附加产物的合成以及科学研究中广泛应用的菌株,枯草芽孢杆菌具有很多优势,包括:生长速度快,培养时间短,培养要求低;分泌信号肽的系统十分高效,使得其分泌蛋白能力强,这对后续蛋白提取工艺提供了很大便利;其分子伴侣系统十分高效,使得其在表达大部分真核来源的蛋白时,可以使得蛋白仍然保持其原有构象和活性。枯草芽孢杆菌的优势使得其在发酵行业应用十分广泛,其表达系统在分泌和表达活性蛋白上的优点逐渐引起人们的关注,并有可能成为继大肠杆菌之后的重要的原核表达系统。
然而,目前已开发的调控枯草芽孢杆菌目的蛋白表达的标准元件尚不完善。例如,启动子和终止子序列相对较长,增加了遗传操作的难度和dna序列合成的成本。此外,大多数标准元件侧重于转录水平的调节,翻译水平调控工具和元件较少。同时,目前翻译水平的调控元件仅注重于调控目的基因的表达量,而没有发现其在动态调控中的作用。动态调节具有重要意义,可以最大限度地提高底物的利用效率,从而最大限度地提高代谢工程中高附加值产品的产量。因此,开发翻译水平上易于使用的动态控制工具也很重要。
技术实现要素:
本发明的第一个目的是提供一种n端序列元件在调控枯草芽孢杆菌目的蛋白表达中的应用,是将具有调控功能的n端编码前15个氨基酸的特定核苷酸序列克隆并添加至目的蛋白的n端,构建到重组表达质粒中,再转化到枯草芽孢杆菌中。
在本发明的一种实施方式中,所述特定核苷酸序列包括seqidno.1~97中的任意一种。将这些n端序列元件按调控功能分为四类,包括生长偶联型(seqidno.1~15,n端序列元件调控目的蛋白主要在稳定期之前表达)、迟滞表达型(seqidno.16~17,n端序列元件调控目的蛋白主要在对数生长末期及稳定期表达)、持续表达型(seqidno.17~90,n端序列元件调控目的蛋白的表达贯穿于细胞生长的所有时期)、强烈抑制型(seqidno.91~97,n端序列元件调控目的蛋白表达被强烈抑制)。
在本发明的一种实施方式中,所述目的蛋白包括绿色荧光蛋白(gfp,genbank:af324408.1)。
在本发明的一种实施方式中,以pp43nmk为表达载体,将具有调控功能的n端序列连接在目的蛋白的n端,表达在枯草芽孢杆菌中。
在本发明的一种实施方式中,所述枯草芽孢杆菌为bacillussubtilis168。
在本发明的一种实施方式中,所述pp43nmk已公开于zhangxz,cuizl,hongq,lisp.high-levelexpressionandsecretionofmethylparathionhydrolaseinbacillussubtiliswb800.appliedandenvironmentalmicrobiology.2005;71(7):4101-3中。
本发明的第二个目的是提供一种调控目的蛋白表达的重组枯草芽孢杆菌,其表达如seqidno.1~97所示的任一基因。
本发明还提供了一种应用上述重组枯草芽孢杆菌发酵使目的蛋白表达的方法,将37℃、750rpm、96孔深孔板中培养9h的重组枯草芽孢杆菌以5%的接种量转入装有190μllb培养基的96孔浅孔板,于37℃、750rpm条件下发酵4小时。
本发明是以枯草芽孢杆菌(bacillussubtilis)作为表达宿主,通过在目的蛋白编码基因前添加内源性蛋白n端编码前15个氨基酸的核酸序列,并在质粒中游离表达,使得枯草芽孢杆菌目的蛋白的表达实现在不同生长阶段的表达差异调控与表达量的调控。在不同生长阶段表达差异方面,我们将序列分成了四种类型:生长偶联型(nhbs等)、迟滞表达型(npdhd等)、持续表达型(nglna等)、强烈抑制型(nsat等)。在表达量方面,提高蛋白表达量的n端序列中,蛋白表达量可提高约7倍;降低蛋白表达量的n端序列中,可降低至对照的0.011倍。这为枯草芽孢杆菌目的蛋白高效表达和基因工程改造奠定了基础。
附图说明
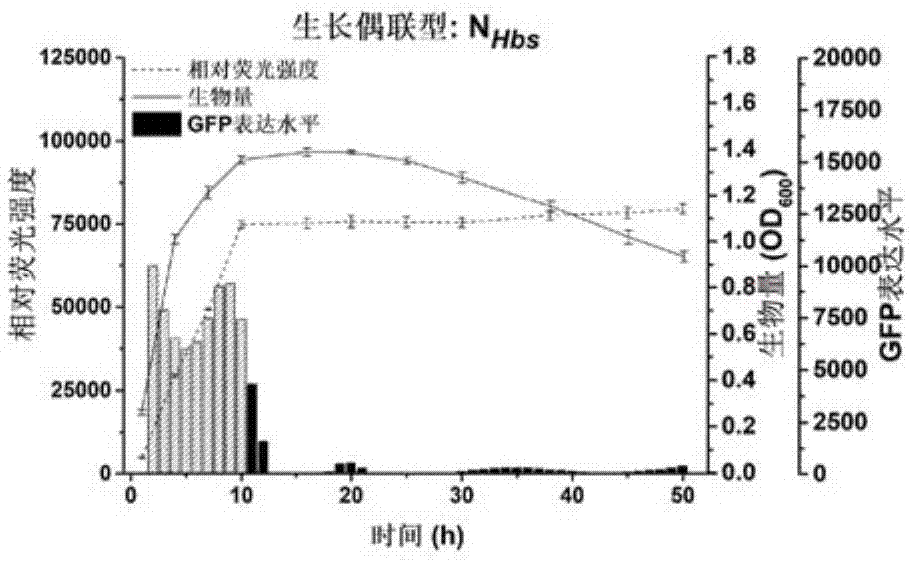
图1:添加如seqidno.1所示的nhbs生长偶联型n端序列对蛋白表达的影响;
图2:添加如seqidno.17所示的npdhd迟滞表达型n端序列对蛋白表达的影响;
图3:添加如seqidno.24所示的nglna持续表达型n端序列对蛋白表达的影响;
图4:添加如seqidno.92所示的nsat强烈抑制型n端序列对蛋白表达的影响。
具体实施方式
(一)重组枯草芽孢杆菌种子培养条件
培养基(g/l):胰蛋白胨10,酵母粉5,nacl10。
培养条件:将37℃、750rpm、700μllb培养基、96孔深孔板中培养9h的种子以5%的接种量转入190μllb培养基,于37℃、750rpm条件下培养4h或20h。
(二)绿色荧光蛋白表达量的测定方法
在96孔板中每孔加入200μl稀释后的发酵液,使用cytation3细胞成像微孔板检测仪(美国伯腾仪器有限公司),激发波长:488nm,发射波长:523nm,增益:60。
实施例1重组质粒的构建
重组质粒的组成是在pp43nmk质粒的p43启动子之后按顺序依次插入如seqidno.1~97所示的任一n端的序列(以glna蛋白的n端序列元件即seqidno.24为例)、绿色荧光蛋白基因(序列如seqidno.98所示)。为了将编码n端的序列引入p43启动子之后,设计引物rh_glna-0.75k_p43nmk-gfp_f(序列如seqidno.99所示)、rh_glna-0.75k_p43nmk-gfp_r(序列如seqidno.100所示)。
以含有绿色荧光蛋白(gfp,genbank:af324408.1)基因的大肠杆菌为模板,通过菌落pcr,得到绿色荧光蛋白片段;
设计引物fx_glna-6.7k_p43nmk-gfp_f(序列如seqidno.101所示);fx_glna-6.7k_p43nmk-gfp_r(序列如seqidno.102所示),以质粒pp43nmk为模板,通过pcr反向扩增得到质粒片段;最后通过gibsonassemblyclongingkit(newenglandbiolabs)构建重组质粒,测序验证,确认重组pp43nmk-glna-gfp质粒构建成功。
实施例2重组pp43nmk-glna-gfp质粒枯草芽孢杆菌的构建
将构建好的pp43nmk-glna-gfp质粒转化枯草芽孢杆菌168野生型菌株。采用yz_zong-p43nmk_f(序列如seqidno.103所示)及yz_zong-p43nmk_r(序列如seqidno.104所示)引物挑选转化子进行菌落pcr,出现1.8kb条带,验证重组枯草芽孢杆菌构建成功。
实施例3添加n端序列对绿色荧光蛋白在工程菌中表达的影响
将37℃、750rpm、700μllb培养基、96孔深孔板中培养9h的种子以5%的接种量转入190μllb培养基,于37℃、750rpm条件下培养4h或20h。相同条件下,若仅以pp43nmk表达,不添加调控的n端序列,最终发酵液中测得荧光强度4小时约为5159,20小时约为48181;而添加glna蛋白的n端序列之后,测得荧光强度4小时平均值为20912.3,20小时平均值为105441.4。分别为对照的4倍、2倍。
添加seqidno.1~97所示的n端序列对绿色荧光蛋白在工程菌中表达的影响概述如下:
(1)调控蛋白表达量的效果:以荧光强度表征蛋白表达量,按照本文所述荧光强度测定方法,n端序列可调控蛋白表达量的范围跨越四个数量级;相比于对照,蛋白表达量可提高约7倍。提高蛋白表达量的n端序列中,以所添加n端序列为ydbp蛋白的n端编码前15个氨基酸的核苷酸序列(即seqidno.18)为例,发酵时间为4h小时左右时,能够使平均荧光强度从约5159提高至35837;发酵时间为20小时左右,能够使荧光强度从约48181提高至138986。分别提高至对照的6.95倍、2.88倍。降低蛋白表达量的n端序列中,以所添加n端序列为rspd蛋白的n端编码前15个氨基酸的核苷酸序列(即seqidno.95)为例,发酵时间为4h小时左右时,能够使平均荧光强度从约5159降低至55;发酵时间为20小时左右,能够使荧光强度从约48181降低至1125。分别降低至对照的0.011、0.023倍。97种不同n端序列的添加对于绿色荧光蛋白表达量调控效果见表1。
(2)调控不同生长阶段蛋白表达差异的效果:以荧光强度表征蛋白表达量,按照本文所述荧光强度测定方法,n端序列可调控目的蛋白在枯草芽孢杆菌的不同生长阶段差异表达,分为生长偶联型、迟滞表达型、持续表达型、强烈抑制型。生长偶联型中,以hbs蛋白的n端序列元件(即seqidno.1)为例,蛋白表达主要集中在10h以内,即生长对数末期之前之前(见图1);迟滞表达型中,以pdhd(即seqidno.17)蛋白的n端序列元件为例,蛋白表达主要集中在15h左右,即生长对数末期(见图2);持续表达型中,以glna蛋白的n端序列元件(即seqidno.24)为例,蛋白表达贯穿于整个生长期的所有阶段(见图3);强烈抑制型中,以sat蛋白的n端序列元件(即seqidno.92)为例,蛋白表达始终受到强烈抑制(见图4);97种不同n端序列的添加对于绿色荧光蛋白表达量调控效果见表1。
表1添加97种不同n端序列对蛋白表达提高情况
对照例1构建无此特定n端序列对照组
重组对照质粒的组成是在pp43nmk质粒的p43启动子之后直接插入绿色荧光蛋白(gfp)基因。设计引物rh_ctr-0.75k_p43nmk-gfp_f(序列如seqidno.105所示),rh_ctr-0.75k_p43nmk-gfp_r(序列如seqidno.106所示)
以含有绿色荧光蛋白(gfp,genbank:af324408.1)基因的大肠杆菌为模板,通过菌落pcr,得到绿色荧光蛋白片段;
设计引物fx_ctr-6.7k_p43nmk-gfp_f(序列如seqidno.101所示)和fx_ctr-6.7k_p43nmk-gfp_r(序列如seqidno.106所示)。
以质粒pp43nmk为模板,通过pcr反向扩增得到质粒片段;最后通过gibsonassemblyclongingkit(newenglandbiolabs)构建重组质粒,测序验证,确认重组pp43nmk-ctr-gfp质粒构建成功后,转化枯草芽孢杆菌168野生型。菌落pcr验证质粒转化成功。
将37℃、750rpm、700μllb培养基、96孔深孔板中培养9h的种子以5%的接种量转入190μllb培养基,于37℃、750rpm条件下培养4h或20h。最终4h发酵液中测得荧光强度是5159,仅为实施例3表达量的四分之一;20h发酵液中测得荧光强度是48181,仅为实施例3表达量的一半。
虽然本发明已以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。
sequencelisting
<110>江南大学
<120>一种n端序列元件在调控枯草芽孢杆菌表达蛋白中的应用
<160>106
<170>patentinversion3.3
<210>1
<400>1
atgaacaaaacagaacttatcaatgcggttgcagaagcaagcgaa45
<210>2
<211>45
<212>dna
<213>bacillussubtilis
<400>2
atgtcacaacaaacaacacccgcagaacaaaaatcacttcaaaga45
<210>3
<211>45
<212>dna
<213>bacillussubtilis
<400>3
atgctgacactgaataatatttcgaagtcgtacaagctgggaaaa45
<210>4
<211>45
<212>dna
<213>bacillussubtilis
<400>4
atgaagcatatttatgagaaaggaacatctgacaacgtacttttg45
<210>5
<211>45
<212>dna
<213>bacillussubtilis
<400>5
atggctacaattcgaattcatgatgaggcaaatacaacgattgaa45
<210>6
<211>45
<212>dna
<213>bacillussubtilis
<400>6
atgatgatgggagtcacaaaaacacctttatacgaaacgttaaat45
<210>7
<211>45
<212>dna
<213>bacillussubtilis
<400>7
atgaaaaatacgatgaaaaggatgttttgcagcatgacggttttg45
<210>8
<211>45
<212>dna
<213>bacillussubtilis
<400>8
atgaatcagcaagacattaaacaaaaagtgcttgatgttctagat45
<210>9
<211>45
<212>dna
<213>bacillussubtilis
<400>9
atgagcttagcaccacacggaggaacattagtaaacagagtagat45
<210>10
<211>45
<212>dna
<213>bacillussubtilis
<400>10
atgtcacagcacgttgaaacgaaattagctcaaattgggaaccgt45
<210>11
<211>45
<212>dna
<213>bacillussubtilis
<400>11
atgttgtttgacactcacgcgcatttaaatgcagaacaatatgat45
<210>12
<211>45
<212>dna
<213>bacillussubtilis
<400>12
atgcttggaattttaaataaaatgtttgatccaacaaaacgtacg45
<210>13
<211>45
<212>dna
<213>bacillussubtilis
<400>13
atgtcagatatggtaaaaatcacatttcctgatggagcagtcaag45
<210>14
<211>45
<212>dna
<213>bacillussubtilis
<400>14
gtgccaacaagtttaaaagatactgtaaagttacataacggagtt45
<210>15
<211>45
<212>dna
<213>bacillussubtilis
<400>15
ttgaaaacaggttactttttattagaagacgggaacaaaattgaa45
<210>16
<211>45
<212>dna
<213>bacillussubtilis
<400>16
atggctaaagaaaaattcgacatgagtaaaggagaagaacttttc45
<210>17
<211>45
<212>dna
<213>bacillussubtilis
<400>17
atggtagtaggagatttccctattgaaacagatactcttgtaatt45
<210>18
<211>45
<212>dna
<213>bacillussubtilis
<400>18
atgaaaaaaatcacaacaaacgaacaatttaatgaactgattcaa45
<210>19
<211>45
<212>dna
<213>bacillussubtilis
<400>19
atggaaatgatgattaaaaaaagaattaaacaagtcaaaaaaggc45
<210>20
<211>45
<212>dna
<213>bacillussubtilis
<400>20
atgagaattaaccacaatattgcagcgcttaacacactgaaccgt45
<210>21
<211>45
<212>dna
<213>bacillussubtilis
<400>21
atgtttatgaaatctactggtattgtacgtaaagttgatgaatta45
<210>22
<211>45
<212>dna
<213>bacillussubtilis
<400>22
atgaatataaatgttgatgtgaagcaaaacgagaatgatatacaa45
<210>23
<211>45
<212>dna
<213>bacillussubtilis
<400>23
atgaactataacatcagaggagaaaatattgaagtgacacccgcg45
<210>24
<211>45
<212>dna
<213>bacillussubtilis
<400>24
atggcaaagtacactagagaagatatcgaaaaattagtaaaagaa45
<210>25
<211>45
<212>dna
<213>bacillussubtilis
<400>25
atggtaaaagtatattataacggtgatatcaaagagaacgtattg45
<210>26
<211>45
<212>dna
<213>bacillussubtilis
<400>26
atgcaaaacggtaaagtaaaatggttcaacaacgaaaaaggattc45
<210>27
<211>45
<212>dna
<213>bacillussubtilis
<400>27
atggaaacgaatgaacaaacaatgccgacgaaatatgatccggca45
<210>28
<211>45
<212>dna
<213>bacillussubtilis
<400>28
atggctaaagaaaaattcgaccgttccaaatcacatgccaatatt45
<210>29
<211>45
<212>dna
<213>bacillussubtilis
<400>29
atgattacgaaaactagcaaaaatgctgctcgtcttaaaagacac45
<210>30
<211>45
<212>dna
<213>bacillussubtilis
<400>30
atgtcagaacagaaaaaagtcgtattagcatactcaggaggtctt45
<210>31
<211>45
<212>dna
<213>bacillussubtilis
<400>31
atgagaaacgaacgcagaaaaaagaaaaaaactttattactgaca45
<210>32
<211>45
<212>dna
<213>bacillussubtilis
<400>32
atgagtataaacataaaagcagtaactgatgataatcgtgctgca45
<210>33
<211>45
<212>dna
<213>bacillussubtilis
<400>33
atgtttaagcacacaaaaatgctgcagcatcctgctaaaccagat45
<210>34
<211>45
<212>dna
<213>bacillussubtilis
<400>34
atggcagacaataacaaaatgagcagagaagaagcaggtagaaaa45
<210>35
<211>45
<212>dna
<213>bacillussubtilis
<400>35
atggctgaatggaaaacaaaacggacatacgatgagatattgtat45
<210>36
<211>45
<212>dna
<213>bacillussubtilis
<400>36
atgttagaaggtaaagtaaaatggttcaactctgaaaaaggtttc45
<210>37
<211>45
<212>dna
<213>bacillussubtilis
<400>37
atgtcattaagagaagaagcattacacctgcataaagtcaaccag45
<210>38
<211>45
<212>dna
<213>bacillussubtilis
<400>38
atggctcaacaaacgaatgttgcaggacaaaaaacagaaaaacaa45
<210>39
<211>45
<212>dna
<213>bacillussubtilis
<400>39
atgtctgattcaaatcttacgaatcctataaaagcattttttcat45
<210>40
<211>45
<212>dna
<213>bacillussubtilis
<400>40
atgaggaaaacagtcattgtaagtgctgcaagaactccatttggc45
<210>41
<211>45
<212>dna
<213>bacillussubtilis
<400>41
atgagaagctatgaaaaatcaaaaacggcttttaaagaagcgcaa45
<210>42
<211>45
<212>dna
<213>bacillussubtilis
<400>42
ttgaatcaaaaagctgtcattctcgacgaacaggcaattagacgg45
<210>43
<211>45
<212>dna
<213>bacillussubtilis
<400>43
atggccaaaataaaagatgattgtatagaacttgaattaacaccg45
<210>44
<211>45
<212>dna
<213>bacillussubtilis
<400>44
atgaccattaaacgtgcattaatcagtgtttcagataaaacaaat45
<210>45
<211>45
<212>dna
<213>bacillussubtilis
<400>45
atggcagacacattagagcgtgtaacgaaaatcatcgtagatcgc45
<210>46
<211>45
<212>dna
<213>bacillussubtilis
<400>46
atggcactatttacagcaaaagtaaccgcgcgaggcggacgagca45
<210>47
<211>45
<212>dna
<213>bacillussubtilis
<400>47
atgggacttttagaagatttgcaaagacaggtgttaatcggtgac45
<210>48
<211>45
<212>dna
<213>bacillussubtilis
<400>48
atggcaggattaattcgtgtcacacccgaagagctaagagcgatg45
<210>49
<211>45
<212>dna
<213>bacillussubtilis
<400>49
atgactaaacaaacaattcgcgttgaattgacatcaacaaaaaaa45
<210>50
<211>45
<212>dna
<213>bacillussubtilis
<400>50
atgacccattcatttgctgttccacgttctgttgaatggaaagaa45
<210>51
<211>45
<212>dna
<213>bacillussubtilis
<400>51
atggaacctttgaaatcacatacggggaaagcagccgtattaaat45
<210>52
<211>45
<212>dna
<213>bacillussubtilis
<400>52
atgaaaaaaattccggttaccgtactgagcggttatctcggtgcg45
<210>53
<211>45
<212>dna
<213>bacillussubtilis
<400>53
atgtgccaatccaatcaaattgtcagccattttttatcccatcga45
<210>54
<211>45
<212>dna
<213>bacillussubtilis
<400>54
atgtctttaatcggtaaagaagtacttccattcgaagcaaaagca45
<210>55
<211>45
<212>dna
<213>bacillussubtilis
<400>55
atggctgcaaaacaagaacgctggcgagagctcgctgaagtaaaa45
<210>56
<211>45
<212>dna
<213>bacillussubtilis
<400>56
atgtcgtttttcagaaatcaattagcgaatgtagtagagtgggaa45
<210>57
<211>45
<212>dna
<213>bacillussubtilis
<400>57
atgtttcaaaatagtatgaaacaacgaatgaattgggaagatttt45
<210>58
<211>45
<212>dna
<213>bacillussubtilis
<400>58
atggcagcaaaatttgaagtgggcagtgtttacactggtaaagtt45
<210>59
<211>45
<212>dna
<213>bacillussubtilis
<400>59
atggtgaccaaaattctaaaagcaccggacggctctccaagtgat45
<210>60
<211>45
<212>dna
<213>bacillussubtilis
<400>60
atgaccaaaggaatcttaggaagaaaaattggtatgacgcaagta45
<210>61
<211>45
<212>dna
<213>bacillussubtilis
<400>61
atgacaaccatcaaaacatcgaatttaggatttccgagaatcgga45
<210>62
<211>45
<212>dna
<213>bacillussubtilis
<400>62
ttgatgtcgaaccagactgtataccagttcattgccgaaaatcaa45
<210>63
<211>45
<212>dna
<213>bacillussubtilis
<400>63
atggctttaaatatcgaagaaatcattgcttccgttaaagaagca45
<210>64
<211>45
<212>dna
<213>bacillussubtilis
<400>64
atgagaatgcgccacaagccttgggctgatgactttttggctgaa45
<210>65
<211>45
<212>dna
<213>bacillussubtilis
<400>65
ttgaggaaagatgaaatcatgcatatcgtatcatgcgcagatgat45
<210>66
<211>45
<212>dna
<213>bacillussubtilis
<400>66
atggatgcgcttattgaggaagttgatggcatttcaaatcgtact45
<210>67
<211>45
<212>dna
<213>bacillussubtilis
<400>67
atggcacatagaattttaattgtagatgacgcagcatttatgcga45
<210>68
<211>45
<212>dna
<213>bacillussubtilis
<400>68
atgggtcttattgtacaaaaattcggaggcacttccgtcggctca45
<210>69
<211>45
<212>dna
<213>bacillussubtilis
<400>69
atgagcagcttgtttcaaacctacggccgttgggatattgacatc45
<210>70
<211>45
<212>dna
<213>bacillussubtilis
<400>70
atggcaaaagtattatatatcactgctcatccacatgacgaagca45
<210>71
<211>45
<212>dna
<213>bacillussubtilis
<400>71
atgattatctgtaaaaccccacgtgaacttggtatcatgcgggaa45
<210>72
<211>45
<212>dna
<213>bacillussubtilis
<400>72
atgaaacgagataaggtgcagaccttacatggagaaatacatatt45
<210>73
<211>45
<212>dna
<213>bacillussubtilis
<400>73
atgtctatgcataaagcactcaccattgccggctcagattccagc45
<210>74
<211>45
<212>dna
<213>bacillussubtilis
<400>74
atggtgacaacggtgcagcgtacgttccgaaaggaagttctacat45
<210>75
<211>45
<212>dna
<213>bacillussubtilis
<400>75
ttgaagaaacgtattgctctattgcccggagacgggatcggccct45
<210>76
<211>45
<212>dna
<213>bacillussubtilis
<400>76
atgaacgaccaatcctgtgtaagaatcatgacagaatgggatatt45
<210>77
<211>45
<212>dna
<213>bacillussubtilis
<400>77
gtggcacaaggtgaaaaaattacagtctctaacggagtattaaac45
<210>78
<211>45
<212>dna
<213>bacillussubtilis
<400>78
atgatacgaagtatgacaggcttcggcagtgcaagcaaaacacaa45
<210>79
<211>45
<212>dna
<213>bacillussubtilis
<400>79
gtgacaaatcgcgatattgtatggcatgaagcctctatcacaaaa45
<210>80
<211>45
<212>dna
<213>bacillussubtilis
<400>80
ttgttatttaaaaaagacagaaaacaagaaacagcttacttttca45
<210>81
<211>45
<212>dna
<213>bacillussubtilis
<400>81
ttgaaaataggaattgtaggtgctacaggatatggaggcaccgaa45
<210>82
<211>45
<212>dna
<213>bacillussubtilis
<400>82
ttgagtaaacacaattggacgctggaaacccagctcgtgcacaat45
<210>83
<211>45
<212>dna
<213>bacillussubtilis
<400>83
gtgaagttttcagaagaatgccgcagtgcagccgcagaatggtgg45
<210>84
<211>45
<212>dna
<213>bacillussubtilis
<400>84
atgtacatatttcaagctgatcagcttagtgccaaagacacatac45
<210>85
<211>45
<212>dna
<213>bacillussubtilis
<400>85
gtgaaaaataaatggctgtcttttttttcgggtaaggtccagctt45
<210>86
<211>45
<212>dna
<213>bacillussubtilis
<400>86
atgaaaacagactggtggaaggatgcagtggtgtaccaaatttac45
<210>87
<211>45
<212>dna
<213>bacillussubtilis
<400>87
atgagaaagtacgaagttatgtacattatccgcccaaacattgac45
<210>88
<211>45
<212>dna
<213>bacillussubtilis
<400>88
gtggaagttactgacgtaagattacgccgcgtgaataccgatggt45
<210>89
<211>45
<212>dna
<213>bacillussubtilis
<400>89
atggcgcaaatgacaatgattcaagcaatcacggatgcgttacgc45
<210>90
<211>45
<212>dna
<213>bacillussubtilis
<400>90
atggaaaaaaaaccgttaactcctagacagattgtagatcggtta45
<210>91
<211>45
<212>dna
<213>bacillussubtilis
<400>91
ttgttaaagccattaggtgatcgcgttgtcattgaactcgtagaa45
<210>92
<211>45
<212>dna
<213>bacillussubtilis
<400>92
ttgaatggaaacgaaccccacggaggggtattaatcaaccgctgt45
<210>93
<211>45
<212>dna
<213>bacillussubtilis
<400>93
ttggcaaagaaagacgaacacctcagaaagcccgaatggcttaaa45
<210>94
<211>45
<212>dna
<213>bacillussubtilis
<400>94
atgcaagcctttgatgagcttctgacggttgagcagctcagcttc45
<210>95
<211>45
<212>dna
<213>bacillussubtilis
<400>95
atggctcgctatacaggtccatcttggaaactgtcccgccgtcta45
<210>96
<211>45
<212>dna
<213>bacillussubtilis
<400>96
atgacaatattagcaattgatacatcaaattatacattgggcatt45
<210>97
<211>45
<212>dna
<213>bacillussubtilis
<400>97
gtgaaaggcacaaaaggaaaggtttttcgtgtttttactgctttt45
<210>98
<211>717
<212>dna
<213>azotobactervinelandii
<400>98
atgagtaaaggagaagaacttttcactggagttgtcccaattcttgttgaattagatggt60
gatgttaatgggcacaaattttctgtcagtggagagggtgaaggtgatgcaacatacgga120
aaacttacccttaaatttatttgcactactggaaaactacctgttccatggccaacactt180
gtcactactttctcttatggtgttcaatgcttttcaagatacccagatcatatgaaacgg240
catgactttttcaagagtgccatgcccgaaggttatgtacaggaaagaactatatttttc300
aaagatgacgggaactacaagacacgtgctgaagtcaagtttgaaggtgatacccttgtt360
aatagaatcgagttaaaaggtattgattttaaagaagatggaaacattcttggacacaaa420
ttggaatacaactataactcacacaatgtatacatcatggcagacaaacaaaagaatgga480
atcaaagttaacttcaaaattagacacaacattgaagatggaagcgttcaactagcagac540
cattatcaacaaaatactccaattggcgatggccctgtccttttaccagacaaccattac600
ctgtccacacaatctgccctttcgaaagatcccaacgaaaagagagaccacatggtcctt660
cttgagtttgtaacagctgctgggattacacatggcatggatgaactatacaaatag717
<210>99
<211>76
<212>dna
<213>人工合成
<400>99
gtacactagagaagatatcgaaaaattagtaaaagaaatgagtaaaggagaagaactttt60
cactggagttgtccca76
<210>100
<211>70
<212>dna
<213>人工合成
<400>100
cgatatcttctctagtgtactttgccatgtgtacattcctctcttacctataatggtacc60
gctatcactt70
<210>101
<211>39
<212>dna
<213>人工合成
<400>101
agtgatgaaagcttggcgtaatcatggtcatagctgttt39
<210>102
<211>83
<212>dna
<213>人工合成
<400>102
cctttttcgttgttgaaccattttactttaccgttttgcatgtgtacattcctctcttac60
ctataatggtaccgctatcactt83
<210>103
<211>22
<212>dna
<213>人工合成
<400>103
ttcttgctgagtctggctttcg22
<210>104
<211>22
<212>dna
<213>人工合成
<400>104
cggctcgtatgttgtgtggaat22
<210>105
<211>43
<212>dna
<213>人工合成
<400>105
acacatgagtaaaggagaagaacttttcactggagttgtccca43
<210>106
<211>59
<212>dna
<213>人工合成
<400>106
tacgccaagctttcatcactatttgtatagttcatccatgccatgtgtaatcccagcag59
<210>1
<211>58
<212>dna
<213>人工合成
<400>107
cttctcctttactcatgtgtacattcctctcttacctataatggtaccgctatcactt
- 还没有人留言评论。精彩留言会获得点赞!