一种用于辅助鉴定身高的方法及其专用引物组与流程
本发明涉及一种用于辅助鉴定身高的方法及其专用引物组。
背景技术:
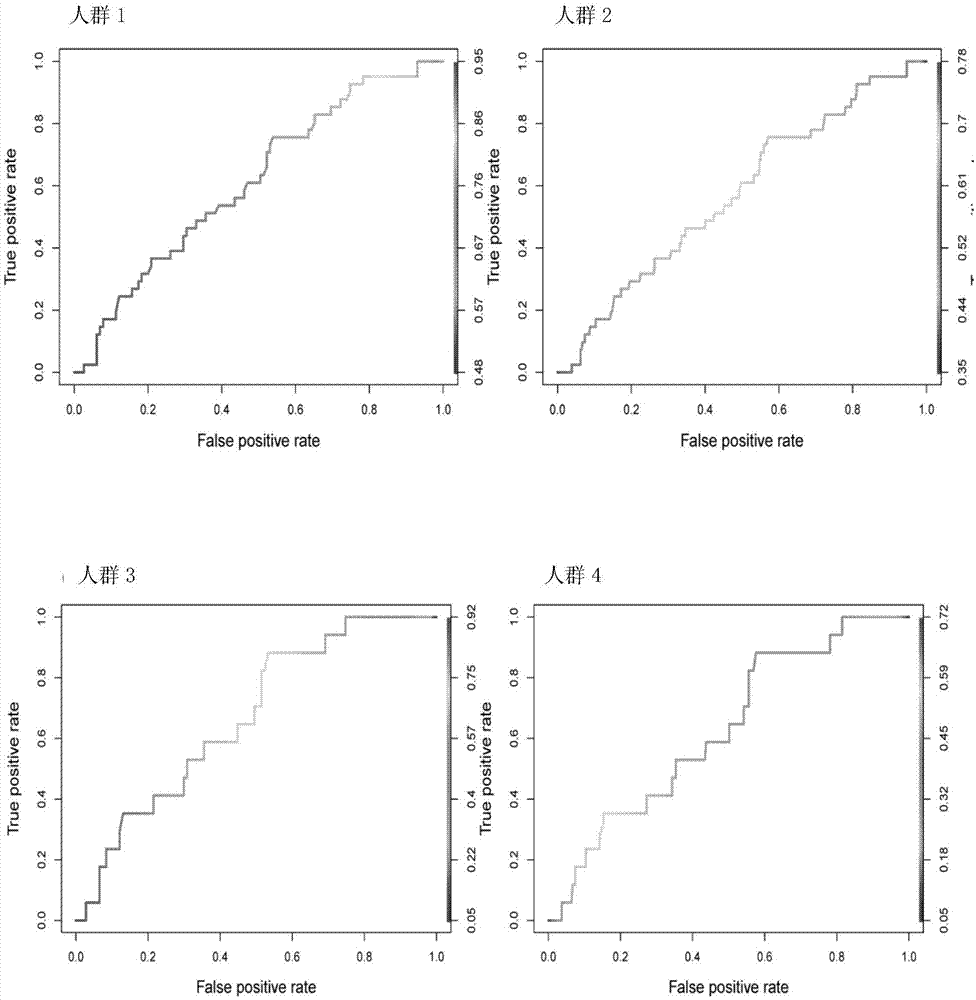
:人类身高是典型的多基因遗传复杂性状。galton等首先发现亲代与子代身高具有一定关联性,fisher等提出身高在遗传表型上的差异是由许多遗传因子共同作用而产生;大家系和双生子研究结果进一步表明,人类身高主要受遗传因素影响,约占80%以上。近年来,随着基因分型技术和dna测序技术的快速发展,尤其是全基因组关联分析(genome-wideassociationstudy,gwas)的应用,人类身高的遗传学研究取得了很多突破性成就。2008年,针对欧洲人群的研究发现了54个与身高呈显著关联性的snp位点。在2010年、2014年及2017年,分别针对18万、25万、71万大样本欧洲人群的gwas分析中共发现约780个身高关联性变异位点,能够解释身高遗传变异的27.4%。这些研究表明身高变异具有多基因性,并且与调节身高的一些遗传学通路相关联。基于身高关联的snp位点推测成年人的身高,对儿科学及法医学具有实践应用意义。身高在不同人群之间存在遗传差异,这种差异表现在关联位点的不同和同一位点对身高表型的效应不同方面。近年来,亚洲人群的身高遗传研究取得了一定的进展,但是由于样本量较小等原因,与欧洲研究相比新发现及验证的身高关联位点有限,且缺少有潜在应用价值的身高推测模型。对身高关联位点进行更为深入的验证并建立相应的推测模型,对我国人群身高遗传学研究具有重要的探索意义。技术实现要素:本发明的目的是提供一种用于辅助鉴定身高的方法及其专用引物组。本发明保护一种特异引物组,由引物组甲-1、引物组甲-2、引物组乙-1和引物组乙-2组成;引物组甲-1由如下24种引物组成:序列表的序列1所示的引物、序列表的序列2所示的引物、序列表的序列4所示的引物、序列表的序列5所示的引物、序列表的序列7所示的引物、序列表的序列8所示的引物、序列表的序列10所示的引物、序列表的序列11所示的引物、序列表的序列13所示的引物、序列表的序列14所示的引物、序列表的序列16所示的引物、序列表的序列17所示的引物、序列表的序列19所示的引物、序列表的序列20所示的引物、序列表的序列22所示的引物、序列表的序列23所示的引物、序列表的序列25所示的引物、序列表的序列26所示的引物、序列表的序列28所示的引物、序列表的序列29所示的引物、序列表的序列31所示的引物、序列表的序列32所示的引物、序列表的序列34所示的引物、序列表的序列35所示的引物;引物组甲-2由如下12种引物组成:序列表的序列3所示的引物、序列表的序列6所示的引物、序列表的序列9所示的引物、序列表的序列12所示的引物、序列表的序列15所示的引物、序列表的序列18所示的引物、序列表的序列21所示的引物、序列表的序列24所示的引物、序列表的序列27所示的引物、序列表的序列30所示的引物、序列表的序列33所示的引物、序列表的序列36所示的引物;引物组乙-1由如下20种引物组成:序列表的序列37所示的引物、序列表的序列38所示的引物、序列表的序列40所示的引物、序列表的序列41所示的引物、序列表的序列43所示的引物、序列表的序列44所示的引物、序列表的序列46所示的引物、序列表的序列47所示的引物、序列表的序列49所示的引物、序列表的序列50所示的引物、序列表的序列52所示的引物、序列表的序列53所示的引物、序列表的序列55所示的引物、序列表的序列56所示的引物、序列表的序列58所示的引物、序序列表的序列59所示的引物、序列表的序列61所示的引物、序列表的序列62所示的引物、序列表的序列64所示的引物、序列表的序列65所示的引物;引物组乙-2由如下10种引物组成:序列表的序列39所示的引物、序列表的序列42所示的引物、序列表的序列45所示的引物、序列表的序列48所示的引物、序列表的序列51所示的引物、序列表的序列54所示的引物、序列表的序列57所示的引物、序列表的序列60所示的引物、序列表的序列63所示的引物、序列表的序列66所示的引物。本发明还保护一种特异引物组,由如下66种引物组成:序列表的序列1所示引物至序列表的序列66所示的引物。所述特异引物组可用于鉴定或辅助鉴定身高。本发明还保护所述特异引物组在制备试剂盒中的应用;所述试剂盒的功能为鉴定或辅助鉴定身高。本发明还保护所述特异引物组的应用,为鉴定或辅助鉴定身高。本发明还保护一种试剂盒,包括所述特异引物组;所述试剂盒的功能为鉴定或辅助鉴定身高。以上任一所述鉴定或辅助鉴定身高具体为鉴定或辅助鉴定待测者(又称受试者)高身高概率或低身高概率。所述试剂盒还包括记载有式ⅰ和/或式ⅱ和/或式ⅲ和/或式ⅳ和/或式ⅴ和/或式ⅵ和/或式ⅶ和/或式ⅷ的载体。式ⅰ:p=exp(0.409+2.117×was)/[1+exp(0.409+2.117×was);式ⅰ为适于男性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥180cm,低身高的含义为身高≤160cm。式ⅱ:p=exp(-0.314+1.241×was)/[1+exp(-0.314+1.241×was)];式ⅱ为适于男性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥180cm,低身高的含义为身高≤165cm。式ⅲ:p=exp(-2.117+3.772×was)/[1+exp(-2.117+3.772×was)];式ⅲ为适于四川男性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥180cm,低身高的含义为身高≤160cm。式ⅳ:p=exp(-2.405+2.628×was)/[1+exp(-2.405+2.628×was)];式ⅳ为适于四川男性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥180cm,低身高的含义为身高≤165cm。式ⅴ:p=exp(0.486+1.697×was)/[1+exp(0.486+1.697×was)];式ⅴ为适于四川女性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥160cm,低身高的含义为身高≤150cm。式ⅵ:p=exp(-0.661+0.897×was)/[1+exp(-0.661+0.897×was)];式ⅵ为适于四川女性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥156cm,低身高的含义为身高<156cm。式ⅶ:p=exp(-0.303+1.261×was)/[1+exp(-0.303+1.261×was)];式ⅶ为适于内蒙男性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥180cm,低身高的含义为身高≤165cm。式ⅷ:p=exp(-0.434+1.103×was)/[1+exp(-0.434+1.103×was)];式ⅷ为适于内蒙男性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥171cm,低身高的含义为身高<171cm。所述身高为亚洲人的身高。所述身高为中国人的身高。所述身高为中国汉族人的身高。本发明还保护一种鉴定待测者身高的方法,包括如下步骤:(1)检测待测者基于snp组合中各个snp的基因型;snp组合为如下22个snp:rs111980222、chr1_103380393、rs17346452、rs2629046、rs12612930、rs7689420、rs11021504、rs3816804、rs11170624、rs3751599、rs11082671、rs6030712、rs992100、rs2166898、rs10037512、rs3823418、rs12413361、rs17152411、rs3781426、rs7909670、rs8052560、rs4821083;(2)根据22个snp的基因型,获得was;was=b1x1+b2x2+b3x3+b4x4+b5x5+b6x6+b7x7+b8x8+b9x9+b10x10+b11x11+b12x12+b13x13+b14x14+b15x15+b16x16+b17x17+b18x18+b19x19+b20x20+b21x21+b22x22;rs111980222的效应等位基因为a,x1为待测者该snp的基因型中效应等位基因的数量,b1=-0.214;chr1_103380393的效应等位基因为a,x2为待测者该snp的基因型中效应等位基因的数量,b2=-0.292;rs17346452的效应等位基因为t,x3为待测者该snp的基因型中效应等位基因的数量,b3=-0.040;rs2629046的效应等位基因为t,x4为待测者该snp的基因型中效应等位基因的数量,b4=0.024;rs12612930的效应等位基因为c,x5为待测者该snp的基因型中效应等位基因的数量,b5=0.100;rs7689420的效应等位基因为t,x6为待测者该snp的基因型中效应等位基因的数量,b6=-0.073;rs11021504的效应等位基因为a,x7为待测者该snp的基因型中效应等位基因的数量,b7=0.083;rs3816804的效应等位基因为c,x8为待测者该snp的基因型中效应等位基因的数量,b8=0.123;rs11170624的效应等位基因为t,x9为待测者该snp的基因型中效应等位基因的数量,b9=0.037;rs3751599的效应等位基因为g,x10为待测者该snp的基因型中效应等位基因的数量,b10=0.187;rs11082671的效应等位基因为g,x11为待测者该snp的基因型中效应等位基因的数量,b11=0.107;rs6030712的效应等位基因为a,x12为待测者该snp的基因型中效应等位基因的数量,b12=-0.021;rs992100的效应等位基因为t,x13为待测者该snp的基因型中效应等位基因的数量,b13=-0.220;rs2166898的效应等位基因为a,x14为待测者该snp的基因型中效应等位基因的数量,b14=-0.052;rs10037512的效应等位基因为t,x15为待测者该snp的基因型中效应等位基因的数量,b15=0.032;rs3823418的效应等位基因为a,x16为待测者该snp的基因型中效应等位基因的数量,b16=0.026;rs12413361的效应等位基因为t,x17为待测者该snp的基因型中效应等位基因的数量,b17=0.020;rs17152411的效应等位基因为a,x18为待测者该snp的基因型中效应等位基因的数量,b18=-0.019;rs3781426的效应等位基因为t,x19为待测者该snp的基因型中效应等位基因的数量,b19=0.019;rs7909670的效应等位基因为t,x20为待测者该snp的基因型中效应等位基因的数量,b20=-0.028;rs8052560的效应等位基因为a,x21为待测者该snp的基因型中效应等位基因的数量,b21=0.025;rs4821083的效应等位基因为t,x22为待测者该snp的基因型中效应等位基因的数量,b22=0.007;(3)根据was鉴定待测者身高。所述鉴定待测者身高具体为鉴定待测者(又称受试者)高身高的概率或低身高的概率。具体将was代入式ⅰ、式ⅱ、式ⅲ、式ⅳ、式ⅴ、式ⅵ、式ⅶ或式ⅷ鉴定待测者身高。式ⅰ:p=exp(0.409+2.117×was)/[1+exp(0.409+2.117×was);式ⅰ为适于男性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥180cm,低身高的含义为身高≤160cm。式ⅱ:p=exp(-0.314+1.241×was)/[1+exp(-0.314+1.241×was)];式ⅱ为适于男性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥180cm,低身高的含义为身高≤165cm。式ⅲ:p=exp(-2.117+3.772×was)/[1+exp(-2.117+3.772×was)];式ⅲ为适于四川男性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥180cm,低身高的含义为身高≤160cm。式ⅳ:p=exp(-2.405+2.628×was)/[1+exp(-2.405+2.628×was)];式ⅳ为适于四川男性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥180cm,低身高的含义为身高≤165cm。式ⅴ:p=exp(0.486+1.697×was)/[1+exp(0.486+1.697×was)];式ⅴ为适于四川女性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥160cm,低身高的含义为身高≤150cm。式ⅵ:p=exp(-0.661+0.897×was)/[1+exp(-0.661+0.897×was)];式ⅵ为适于四川女性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥156cm,低身高的含义为身高<156cm。式ⅶ:p=exp(-0.303+1.261×was)/[1+exp(-0.303+1.261×was)];式ⅶ为适于内蒙男性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥180cm,低身高的含义为身高≤165cm。式ⅷ:p=exp(-0.434+1.103×was)/[1+exp(-0.434+1.103×was)];式ⅷ为适于内蒙男性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥171cm,低身高的含义为身高<171cm。“检测待测者基于snp组合中各个snp的基因型”的方法如下:①以待测者的基因组dna为模板进行两个体系的pcr扩增;一个体系的pcr扩增采用所述引物组甲-1,命名为pcr体系甲;另一个体系的pcr扩增采用所述引物组乙-1,命名为pcr体系乙;②完成步骤①后,在体系中加入核酸内切酶和虾碱性磷酸酶进行反应;③完成步骤②后纯化并回收pcr扩增产物,进行单碱基延伸反应;pcr体系甲得到的pcr扩增产物采用所述引物组甲-2进行单碱基延伸反应;pcr体系乙得到的pcr扩增产物采用所述引物组乙-2进行单碱基延伸反应;④完成步骤③后,体系中加入虾碱性磷酸酶进行反应;⑤采用遗传分析仪进行电泳,然后进行基因分型,得到待测者基于snp组合中各个snp的基因型。步骤①中反应体系具体可为(5μl):2μl模板(含10ngdna),ddh2o1.05μl,mgcl2(25mmmg2+)0.7μl,10×pcrbuffer(15mmmg2+)0.5μl,hotstartaqplus(5u/μl)0.2μl,dntp(10mm)0.05μl,pcr引物混合液0.5μl。步骤①中反应程序具体可为:95℃5min;94℃30s、55℃30s、72℃1min,40个循环;72℃10min。步骤②中,核酸内切酶在体系中的浓度为1u/μl,虾碱性磷酸酶在体系中的浓度为0.3u/μl。步骤②中,反应程序具体可为:37℃60min;96℃10min。步骤③中,反应体系具体可为(5μl):snapshotmultiplexreadyreactionmix1.5μl,ddh2o1.7μl,纯化并回收的pcr扩增产物1.5μl,单碱基引物混合液0.3μl。步骤③中,反应程序具体可为:96℃10s、50℃5s、60℃30s,25个循环。步骤④中,虾碱性磷酸酶在体系中的浓度为0.16u/μl,然后进行反应。步骤④中,反应程序:37℃60min;85℃15min。采用引物组甲-1时,反应体系中,用于检测rs111980222snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.0012μm/μl,用于检测chr1_103380393snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs17346452snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.002μm/μl,用于检测rs2629046snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs12612930snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs7689420snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs11021504snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs3816804snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs11170624snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.0015μm/μl,用于检测rs3751599snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs11082671snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.0025μm/μl,用于检测rs6030712snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl。采用引物组乙-1时,反应体系中,用于检测rs992100snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs2166898snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs10037512snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs3823418snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs12413361snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.0004μm/μl,用于检测rs17152411snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs3781426snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs7909670snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs8052560snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs4821083snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.004μm/μl。采用引物组甲-2时,反应体系中,用于检测rs111980222snp的单碱基引物的浓度为0.00468μm/μl,用于检测chr1_103380393snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs17346452snp的单碱基引物的浓度为0.0072μm/μl,用于检测rs2629046snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs12612930snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs7689420snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs11021504snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs3816804snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs11170624snp的单碱基引物的浓度为0.00468μm/μl,用于检测rs3751599snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs11082671snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs6030712snp的单碱基引物的浓度为0.0036μm/μl。采用引物组乙-2时,反应体系中,用于检测rs992100snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs2166898snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs10037512snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs3823418snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs12413361snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs17152411snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs3781426snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs7909670snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs8052560snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs4821083snp的单碱基引物的浓度为0.0036μm/μl。所述待测者为亚洲人。所述待测者为中国人。所述待测者为中国汉族人。本发现初步筛选出31个身高关联性snp位点,利用其中22个snp位点构建身高推测模型,评估其在中国汉族人群中的身高推测效力,对于儿科学及法医学具有实践应用意义。附图说明图1为不同分组人群roc曲线。图2为不同分组人群roc曲线。图3为was值密度曲线图。图4为was值密度曲线图。具体实施方式以下的实施例便于更好地理解本发明,但并不限定本发明。下述实施例中的实验方法,如无特殊说明,均为常规方法。下述实施例中所用的试验材料,如无特殊说明,均为自常规生化试剂商店购买得到的。以下实施例中的定量试验,均设置三次重复实验,结果取平均值。实施例中的身高均为成人身高。实施例1、模型的建立从现有技术中获取数目极其庞大的与身高具有显著关联性的snp位点,通过高通量测序和关联分析挖掘数目极其庞大的与身高具有显著关联性的snp位点。在中国汉族人群中,进行各个snp位点的分型以及身高测量,将各个snp位点的基因型与身高性状进行关联分析,初步筛选出31个snp位点。对31个snp位点进行进一步筛选和建模。31个snp位点的信息见表1。chr1_103380393snp及其周边核苷酸如序列表的序列67所示,其中第73位为snp位点。表1snp染色体位置基因多态形式rs1420367012219924961ihhg/trs1488335595172755066stc2c/ars10978953998583622znp510(9q22)t/grs10119466998591446znp510(9q22)g/ars10124911998607205znp782(9q23)a/crs7859940998624701znp782(9q25)a/grs1489344121684902472crispld2g/ars169667031736755924krt33aa/grs1378525912366941751arc/grs1119802221102914930col11a1a/gchr1_1033803931103380393col11a1a/grs173464521172084147dnm3t/crs26290462224183027serpine2t/crs12612930271298655znf638c/trs76894204144647200hhipt/crs110215041196262284maml2(11q21)a/trs38168041256286961cs(12q13.3)c/trs111706241253636454atf7t/grs37515991551281336cyp19a1c/trs110826711848339226zbtb7cg/ars60307122037008995rbl1a/grs9921001102913989col11a1t/ars21668982120855084gli2a/grs10037512589058858mef2ct/crs3823418631133165psors1c1a/grs124133611030838237znf438t/grs1715241110124960947zranb1a/grs378142610125014780ctbp2a/grs79096701012876764ccdc3t/crs80525601688710834ctu2/galnsa/crs48210832232660355syn3t/c1、使用plinkv1.90进行多元线性回归分析和logistic回归分析,线性回归分析时以身高为因变量,snp位点分型为自变量;logistic回归分析时身高为二值变量,snp位点分型为自变量;年龄、性别等因素作为协变量,不同人群做人群分层校正分析。身高呈正态分布。2、身高关联性分析使用plinkv1.90软件对样本基因分型数据进行质量控制,控制条件为:(1)剔除基因分型成功率(callrate)小于90%的样本及snp位点;(2)剔除maf<0.01的snp位点;(3)对人群进行hardy-weinberg平衡检测。经过这一过程后:(1)剔除分型成功率(callrate)小于90%的样本1个;(2)31个snp位点基因分型成功率大于90%;(3)剔除maf<0.01的snp位点4个,该低频位点在本研究人群中未发现多态性;(4)对人群进行hardy-weinberg平衡检测,0个snp位点(p<10-6)被剔除。质控后剩余snp位点27个。数据采用3种方案进行关联性分析,年龄、性别及地域作为校正因素。(1)以身高数值作为连续因变量,snp位点分型为自变量;(2)将身高数值按照百分位点和平均值进行划分,身高数值小于等于第20百分位数(p20)设为1,第20百分位到第30百分位(p30)设为2,第30百分位到平均身高(mean)设为3,平均身高到第70百分位(p70)设为4,第70百分位到第80百分位(p80)设为5,大于第80百分位设为6,做线性回归分析。(3)将身高数值分别以第30百分位数和第80百分位数为阈值进行二分类,做logisitic回归分析。样本分组信息见表2。3组方案关联分析结果见表3。3种关联性分析方案分别发现3个、4个、6个与身高呈关联性snp位点(p<0.05)。其中rs3823418、rs2166898在3种方案中与身高均呈关联性(p<0.05),rs4821083、rs11021504在其中2种方案中达到关联性水平(p<0.05),rs3816804、rs3751599及rs6030712在其中一种方案中达到关联性水平(p<0.05)。表2样本分组信息(sd:标准差)表33组方案关联分析结果eaf(effectallelefrequency):效应等位基因频率;or:比值比。3、建立身高预测模型数据质控后去掉4个无多态性snp位点,去掉5个无beta值的snp位点,剩余22个snp位点。在样本中建立身高二元分类预测模型。根据样本身高的分布情况,每个群体采用两种方式分类,每种方式包含相应的高、低2组。采用等位基因加权求和(weightedallelesums,was)法建立身高预测模型。式ⅰ:p=exp(0.409+2.117×was)/[1+exp(0.409+2.117×was);式ⅰ为适于男性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥180cm,低身高的含义为身高≤160cm。式ⅱ:p=exp(-0.314+1.241×was)/[1+exp(-0.314+1.241×was)];式ⅱ为适于男性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥180cm,低身高的含义为身高≤165cm。式ⅲ:p=exp(-2.117+3.772×was)/[1+exp(-2.117+3.772×was)];式ⅲ为适于四川男性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥180cm,低身高的含义为身高≤160cm。式ⅳ:p=exp(-2.405+2.628×was)/[1+exp(-2.405+2.628×was)];式ⅳ为适于四川男性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥180cm,低身高的含义为身高≤165cm。式ⅴ:p=exp(0.486+1.697×was)/[1+exp(0.486+1.697×was)];式ⅴ为适于四川女性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥160cm,低身高的含义为身高≤150cm。式ⅵ:p=exp(-0.661+0.897×was)/[1+exp(-0.661+0.897×was)];式ⅵ为适于四川女性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥156cm,低身高的含义为身高<156cm。式ⅶ:p=exp(-0.303+1.261×was)/[1+exp(-0.303+1.261×was)];式ⅶ为适于内蒙男性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥180cm,低身高的含义为身高≤165cm。式ⅷ:p=exp(-0.434+1.103×was)/[1+exp(-0.434+1.103×was)];式ⅷ为适于内蒙男性的身高预测公式;p值为受试者为高身高的概率,(1-p)值为受试者为低身高的概率;高身高的含义为身高≥171cm,低身高的含义为身高<171cm。实施例2、模型的应用和评估样本人群总量为1221例(其中,四川男性773例、四川女性191例,内蒙古男性257例)。样本人群年龄分布区间为18-59岁,均为汉族。样本人群发育均正常,无甲状腺疾病、骨和软骨发育障碍、糖尿病等影响生长发育的疾病。所有参与者均签署了知情同意书。一、测量受试者的实际身高。采用专业的测量工具及经过培训的人员进行身高测量,测量方法:被测者赤脚,“立正”姿势站在身高计的底板上,脚跟、骶骨部及两肩胛间紧靠身高计的立柱上。测量人员站在被测人的左边,将其头部调整到耳屏上缘与眼眶下缘的最低点齐平,再移动身高计的水平板至被测量人的头顶,使其松紧度适当,测量出身高。二、设计用于检测22个snp位点的引物组。每个引物组由三条引物(pcr扩增上游引物、pcr扩增下游引物和单碱基延伸引物)组成,pcr扩增上游引物和pcr扩增下游引物组成pcr引物对。用于检测rs111980222snp的引物组如下:pcr扩增上游引物(序列表的序列1):5’-acgttggatgattaaccttggcacactggg-3’;pcr扩增下游引物(序列表的序列2):5’-acgttggatgccactacactccaacttggt-3’;单碱基延伸引物(序列表的序列3):5’-ctctctctctctctctctctctctctctctctctctctctctctctcttggtgacaaagtgatatt-3’。用于检测chr1_103380393snp的引物组如下:pcr扩增上游引物(序列表的序列4):5’-acgttggatgctttgggaccctaaacaatg-3’;pcr扩增下游引物(序列表的序列5):5’-acgttggatgccagtgtgccaaggttaata-3’;单碱基延伸引物(序列表的序列6):5’-ctctctctctctctctctctctctctctctctctctctcaactcttctttccttcttctt-3’。用于检测rs17346452snp的引物组如下:pcr扩增上游引物(序列表的序列7):5’-acgttggatgctttgggttttggcaaggtg-3’;pcr扩增下游引物(序列表的序列8):5’-acgttggatgtcctgaaaggtatacaagac-3’;单碱基延伸引物(序列表的序列9):5’-tctctctctctctctctctctggtgctcagcctttc-3’。用于检测rs2629046snp的引物组如下:pcr扩增上游引物(序列表的序列10):5’-acgttggatgagtgcatgctggatgcaaac-3’;pcr扩增下游引物(序列表的序列11):5’-acgttggatggggtatagaagctcaaatgc-3’;单碱基延伸引物(序列表的序列12):5’-ctctctctctctctctctctctctctctctctctctctctctctctctgaggacctacgaaat-3’。用于检测rs12612930snp的引物组如下:pcr扩增上游引物(序列表的序列13):5’-acgttggatgaactgtatatgctacgcctg-3’;pcr扩增下游引物(序列表的序列14):5’-acgttggatgtacatgcaaccatgttgccc-3’;单碱基延伸引物(序列表的序列15):5’-ctctctctctctctctctctctctctctctctctctctctctcctctctctgtaagggtttttcatggatta-3’。用于检测rs7689420snp的引物组如下:pcr扩增上游引物(序列表的序列16):5’-acgttggatggctgtgcaaacttgcctatc-3’;pcr扩增下游引物(序列表的序列17):5’-acgttggatgcacactgttcatcgcgattc-3’;单碱基延伸引物(序列表的序列18):5’-ctctctctctctctctctctctctctctcctatctctgaaaacagacg-3’。用于检测rs11021504snp的引物组如下:pcr扩增上游引物(序列表的序列19):5’-acgttggatgcagagttcagtcacagattc-3’;pcr扩增下游引物(序列表的序列20):5’-acgttggatggctaactgagggtcaaatta-3’;单碱基延伸引物(序列表的序列21):5’-ctctctctctctctctctctctctctctctctctctctttctccaaatgtgctgatt-3’。用于检测rs3816804snp的引物组如下:pcr扩增上游引物(序列表的序列22):5’-acgttggatgatttttgagtgaccccaagg-3’;pcr扩增下游引物(序列表的序列23):5’-acgttggatgaggtgcaggagagatcttac-3’;单碱基延伸引物(序列表的序列24):5’-tctctctctctctctctctctctctctctctctctaggaaaacaaatgaggaaa-3’。用于检测rs11170624snp的引物组如下:pcr扩增上游引物(序列表的序列25):5’-acgttggatggaaaagacaaaagaagagacc-3’;pcr扩增下游引物(序列表的序列26):5’-acgttggatggacaaaaaagcactgaaagtc-3’;单碱基延伸引物(序列表的序列27):5’-ctctctctctctctctctctctctctctctctctctctctctctcaaagtcaaaaattgttactagaaa-3’。用于检测rs3751599snp的引物组如下:pcr扩增上游引物(序列表的序列28):5’-acgttggatggactaggtaagattagaggc-3’;pcr扩增下游引物(序列表的序列29):5’-acgttggatgactggctgagcttctacttg-3’;单碱基延伸引物(序列表的序列30):5’-tctctctctctctctctctctctctacctccagagagtgacc-3’。用于检测rs11082671snp的引物组如下:pcr扩增上游引物(序列表的序列31):5’-acgttggatgtgcatctctgagcaggcac-3’;pcr扩增下游引物(序列表的序列32):5’-acgttggatgagtcacagccaggtgggta-3’;单碱基延伸引物(序列表的序列33):5’-ctctctctctctctccccaggagcctactc-3’。用于检测rs6030712snp的引物组如下:pcr扩增上游引物(序列表的序列34):5’-acgttggatgctgagaagtactgcagttgg-3’;pcr扩增下游引物(序列表的序列35):5’-acgttggatggggaagagagactatacac-3’;单碱基延伸引物(序列表的序列36):5’-tctctctctctctctctctcttaattccatacttcccca-3’。用于检测rs992100snp的引物组如下:pcr扩增上游引物(序列表的序列37):5’-acgttggatgtaaggaagaaagaagatgt-3’;pcr扩增下游引物(序列表的序列38):5’-acgttggatgctcttctataatgcaaatcc-3’;单碱基延伸引物(序列表的序列39):5’-ctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctgaatcatttcaccatgaaca-3’。用于检测rs2166898snp的引物组如下:pcr扩增上游引物(序列表的序列40):5’-acgttggatgggaatgaccctgatttcctc-3’;pcr扩增下游引物(序列表的序列41):5’-acgttggatgtcctctgctcatttgctgtg-3’;单碱基延伸引物(序列表的序列42):5’-ctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctcgaggaggtttatttgggca-3’。用于检测rs10037512snp的引物组如下:pcr扩增上游引物(序列表的序列43):5’-acgttggatggagcagtgcctattagactc-3’;pcr扩增下游引物(序列表的序列44):5’-acgttggatgatgttatattggtcatgtc-3’;单碱基延伸引物(序列表的序列45):5’-ctctctctctctctctctctctctctctctctctctctggtcatgtcaaaatgag-3’。用于检测rs3823418snp的引物组如下:pcr扩增上游引物(序列表的序列46):5’-acgttggatggagtgatttagcactctggg-3’;pcr扩增下游引物(序列表的序列47):5’-acgttggatggactgtgttctacgcatttc-3’;单碱基延伸引物(序列表的序列48):5’-ctctctctctctctctctctctctctctctctctctctctctctctctctctctctcacgcatttcttcttgaac-3’。用于检测rs12413361snp的引物组如下:pcr扩增上游引物(序列表的序列49):5’-gcacatgaacacatagaggg-3’;pcr扩增下游引物(序列表的序列50):5’-aagcctatagtaccctttag-3’;单碱基延伸引物(序列表的序列51):5’-ctctctctctctctctctctctctctctctctctctctctctctctctcccaccctccagtaaac-3’。用于检测rs17152411snp的引物组如下:pcr扩增上游引物(序列表的序列52):5’-acgttggatggatttcagtggtgaatgtgg-3’;pcr扩增下游引物(序列表的序列53):5’-acgttggatgctcttaggttccaaaagtag-3’;单碱基延伸引物(序列表的序列54):5’-ctctctctctctctctctctctctctctatgtggttttggcatac-3’。用于检测rs3781426snp的引物组如下:pcr扩增上游引物(序列表的序列55):5’-acgttggatgtcccctaaagtctcgggaag-3’;pcr扩增下游引物(序列表的序列56):5’-acgttggatgcgtggtcactggtcttgtc-3’;单碱基延伸引物(序列表的序列57):5’-ctctctctctctctctctctctctctctctctctctctctctctccacccagttctgagc-3’。用于检测rs7909670snp的引物组如下:pcr扩增上游引物(序列表的序列58):5’-acgttggatgaggagataacacacgatcac-3’;pcr扩增下游引物(序列表的序列59):5’-acgttggatgtgttctcagatgtctggtcc-3’;单碱基延伸引物(序列表的序列60):5’-ctctctctctctctctctctctctctctctctctcagctggctccaacac-3’。用于检测rs8052560snp的引物组如下:pcr扩增上游引物(序列表的序列61):5’-acgttggatgaaagggagcagcgtttgagg-3’;pcr扩增下游引物(序列表的序列62):5’-acgttggatgagccctgtgtgcagcctaa-3’;单碱基延伸引物(序列表的序列63):5’-ctctctctctctctctctctctctctctctctctctctctctctctctctctctccctgcgcgggccgcc-3’。用于检测rs4821083snp的引物组如下:pcr扩增上游引物(序列表的序列64):5’-acgttggatgccaaacctggtttctttggg-3’;pcr扩增下游引物(序列表的序列65):5’-acgttggatgtttcgcaactttgctgcacc-3’;单碱基延伸引物(序列表的序列66):5’-ctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctcttggcttccaggtcag-3’。三、检测22个snp位点的基因型1、采集样本人群中各个样本的外周血,提取基因组dna。2、采用微测序技术进行基因分型。(1)以步骤1得到的基因组dna为模板进行pcr扩增。反应体系(5μl):2μl模板(含10ngdna),ddh2o1.05μl,mgcl2(25mmmg2+)0.7μl,10×pcrbuffer(15mmmg2+)0.5μl,hotstartaqplus(5u/μl)0.2μl,dntp(10mm)0.05μl,pcr引物混合液0.5μl。反应程序:95℃5min;94℃30s、55℃30s、72℃1min,40个循环;72℃10min。(2)完成步骤(1)后,在体系中加入核酸内切酶(exo-i)和虾碱性磷酸酶(sap),核酸内切酶在体系中的浓度为1u/μl,虾碱性磷酸酶在体系中的浓度为0.3u/μl,然后进行反应。反应程序:37℃60min;96℃10min。(3)完成步骤(2)后,纯化并回收pcr扩增产物,进行单碱基延伸反应。反应体系(5μl):snapshotmultiplexreadyreactionmix1.5μl,ddh2o1.7μl,纯化并回收的pcr扩增产物1.5μl,单碱基引物混合液0.3μl。反应程序:96℃10s、50℃5s、60℃30s,25个循环。(4)完成步骤(3)后,在体系中加入虾碱性磷酸酶,虾碱性磷酸酶在体系中的浓度为0.16u/μl,然后进行反应。反应程序:37℃60min;85℃15min。(5)完成步骤(4)后,取1μl反应产物与9μl含有liz120的甲酰胺混合,使用abi3130自动遗传分析仪进行电泳,在genemapperidv3.2软件上进行基因分型。以genescan-120lizsizestandard(abi)作内标。每个样本均获得了基于22个特异snp的基因型。pcr引物混合液为pcr引物混合液甲或pcr引物混合液乙。pcr引物混合液甲中的有效成分为:用于检测rs111980222snp的pcr引物对、用于检测chr1_103380393snp的pcr引物对、用于检测rs17346452snp的pcr引物对、用于检测rs2629046snp的pcr引物对、用于检测rs12612930snp的pcr引物对、用于检测rs7689420snp的pcr引物对、用于检测rs11021504snp的pcr引物对、用于检测rs3816804snp的pcr引物对、用于检测rs11170624snp的pcr引物对、用于检测rs3751599snp的pcr引物对、用于检测rs11082671snp的pcr引物对和用于检测rs6030712snp的pcr引物对。pcr引物混合液乙中的有效成分为:用于检测rs992100snp的pcr引物对、用于检测rs2166898snp的pcr引物对、用于检测rs10037512snp的pcr引物对、用于检测rs3823418snp的pcr引物对、用于检测rs12413361snp的pcr引物对、用于检测rs17152411snp的pcr引物对、用于检测rs3781426snp的pcr引物对、用于检测rs7909670snp的pcr引物对、用于检测rs8052560snp的pcr引物对和用于检测rs4821083snp的pcr引物对。单碱基引物混合液为单碱基引物混合液甲或单碱基引物混合液乙。单碱基引物混合液甲中的有效成分为:用于检测rs111980222snp的单碱基引物、用于检测chr1_103380393snp的单碱基引物、用于检测rs17346452snp的单碱基引物、用于检测rs2629046snp的单碱基引物、用于检测rs12612930snp的单碱基引物、用于检测rs7689420snp的单碱基引物、用于检测rs11021504snp的单碱基引物、用于检测rs3816804snp的单碱基引物、用于检测rs11170624snp的单碱基引物、用于检测rs3751599snp的单碱基引物、用于检测rs11082671snp的单碱基引物和用于检测rs6030712snp的单碱基引物。单碱基引物混合液乙中的有效成分为:用于检测rs992100snp的单碱基引物、用于检测rs2166898snp的单碱基引物、用于检测rs10037512snp的单碱基引物、用于检测rs3823418snp的单碱基引物、用于检测rs12413361snp的单碱基引物、用于检测rs17152411snp的单碱基引物、用于检测rs3781426snp的单碱基引物、用于检测rs7909670snp的单碱基引物、用于检测rs8052560snp的单碱基引物和用于检测rs4821083snp的单碱基引物。步骤(1)中采用pcr引物混合液甲时,步骤(3)中采用单碱基引物混合液甲。步骤(1)中采用pcr引物混合液乙时,步骤(3)中采用单碱基引物混合液乙。步骤(1)中,采用pcr引物混合液甲时,反应体系中,用于检测rs111980222snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.0012μm/μl,用于检测chr1_103380393snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs17346452snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.002μm/μl,用于检测rs2629046snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs12612930snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs7689420snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs11021504snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs3816804snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs11170624snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.0015μm/μl,用于检测rs3751599snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs11082671snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.0025μm/μl,用于检测rs6030712snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl。步骤(1)中,采用pcr引物混合液乙时,反应体系中,用于检测rs992100snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs2166898snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs10037512snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs3823418snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs12413361snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.0004μm/μl,用于检测rs17152411snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs3781426snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs7909670snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs8052560snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.001μm/μl,用于检测rs4821083snp的pcr扩增上游引物和pcr扩增下游引物的浓度均为0.004μm/μl。步骤(3)中,采用单碱基引物混合液甲时,反应体系中,用于检测rs111980222snp的单碱基引物的浓度为0.00468μm/μl,用于检测chr1_103380393snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs17346452snp的单碱基引物的浓度为0.0072μm/μl,用于检测rs2629046snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs12612930snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs7689420snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs11021504snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs3816804snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs11170624snp的单碱基引物的浓度为0.00468μm/μl,用于检测rs3751599snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs11082671snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs6030712snp的单碱基引物的浓度为0.0036μm/μl。步骤(3)中,采用单碱基引物混合液乙时,反应体系中,用于检测rs992100snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs2166898snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs10037512snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs3823418snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs12413361snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs17152411snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs3781426snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs7909670snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs8052560snp的单碱基引物的浓度为0.0036μm/μl,用于检测rs4821083snp的单碱基引物的浓度为0.0036μm/μl。四、根据22个snp的基因型对每个样本的身高进行预测1、根据22个snp的基因型,获得各个样本的was。was=b1x1+b2x2+b3x3+b4x4+b5x5+b6x6+b7x7+b8x8+b9x9+b10x10+b11x11+b12x12+b13x13+b14x14+b15x15+b16x16+b17x17+b18x18+b19x19+b20x20+b21x21+b22x22。具体见表4。x值为效应等位基因数目。表4snp多态形式效应等位基因b值x值rs111980222a/gab1=-0.214x1=2、1或0chr1_103380393a/gab2=-0.292x2=2、1或0rs17346452t/ctb3=-0.040x3=2、1或0rs2629046t/ctb4=0.024x4=2、1或0rs12612930c/tcb5=0.100x5=2、1或0rs7689420t/ctb6=-0.073x6=2、1或0rs11021504a/tab7=0.083x7=2、1或0rs3816804c/tcb8=0.123x8=2、1或0rs11170624t/gtb9=0.037x9=2、1或0rs3751599g/agb10=0.187x10=2、1或0rs11082671g/agb11=0.107x11=2、1或0rs6030712a/gab12=-0.021x12=2、1或0rs992100t/atb13=-0.220x13=2、1或0rs2166898a/gab14=-0.052x14=2、1或0rs10037512t/ctb15=0.032x15=2、1或0rs3823418a/gab16=0.026x16=2、1或0rs12413361t/gtb17=0.020x17=2、1或0rs17152411a/gab18=-0.019x18=2、1或0rs3781426t/ctb19=0.019x19=2、1或0rs7909670t/ctb20=-0.028x20=2、1或0rs8052560a/cab21=0.025x21=2、1或0rs4821083t/ctb22=0.007x22=2、1或0以rs111980222为例,对表1进行示例性解释如下:多态形式为a/g,效应等位基因为a,b1=-0.214,当基因型为aa时、x1=2,当基因型为ag时、x1=1,当基因型为gg时、x1=0。2、将was代入实施例1建立身高预测模型。所有男性样本中,41位身高≥180cm(高身高),115位身高≤160cm(低身高),这些组成人群1(样本数量为:41+115)。将人群1的各个样本的was分别代入式ⅰ,得到各个样本的p值。所有男性样本中,41位身高≥180cm(高身高),338位身高≤165cm(低身高),这些组成人群2(样本数量为:41+338)。将人群2的各个样本的was分别代入式ⅱ,得到各个样本的p值。所有四川男性样本中,17位身高≥180cm(高身高),107位身高≤160cm(低身高),这些组成人群3(样本数量为:17+107)。将人群3的各个样本的was分别代入式ⅲ,得到各个样本的p值。所有四川男性样本中,17位身高≥180cm(高身高),297位身高≤165cm(低身高),这些组成人群4(样本数量为:17+297)。将人群4的各个样本的was分别代入式ⅳ,得到各个样本的p值。所有四川女性样本中,47位身高≥160cm(高身高),39位身高≤150cm(低身高),这些组成人群5(样本数量为:47+39)。将人群5的各个样本的was分别代入式ⅴ,得到各个样本的p值。所有四川女性样本中,99位身高≥156cm(高身高),92位身高<156cm(低身高),这些组成人群6(样本数量为:99+92)。将人群6的各个样本的was分别代入式ⅵ,得到各个样本的p值。所有内蒙男性样本中,24位身高≥180cm(高身高),41位身高≤165cm(低身高),这些组成人群7(样本数量为:24+41)。将人群7的各个样本的was分别代入式ⅶ,得到各个样本的p值。所有内蒙男性样本中,138位身高≥171cm(高身高),119位身高<171cm(低身高),这些组成人群8(样本数量为:138+119)。将人群8的各个样本的was分别代入式ⅷ,得到各个样本的p值。各个群体,根据每个样本的p值和在群体中的真实分组信息(高身高组、低身高组)制作roc曲线。通过r语言绘制受试者工作特征曲线(receiveroperatingcharacteristic,roc)并计算曲线下面积(areaundercurve,auc),评估预测模型的准确性。auc值分布为0.5-1.0,0.5表示没有预测能力,数值越接近1.0表示模型的预测能力越准确。不同分组人群roc曲线见图1和图2,横轴为假阳性率,纵轴为真阳性率。auc值见表5。预测准确性最高的为四川男性样本,auc值(1组)为0.67(95%ci0.55-0.79)。在四川男性样本中,180cm/160cm的身高分类预测准确度高于180cm/165cm身高分类(auc=0.67vs0.63)。但在全部男性样本中,180cm/160cm的身高分类低于相同分类的四川男性样本(auc=0.61vs0.67),说明在族群、地域来源同一的群体中身高预测准确度更高,进一步提示了群体差异性对身高遗传的影响。表5aucl95u95人群10.610.520.71人群20.570.490.66人群30.670.550.79人群40.630.510.75人群50.620.50.74人群60.580.490.66人群70.570.420.71人群80.570.50.64绘制was值密度曲线图。was值密度曲线图见图3和图4,横轴为was分布,纵轴为密度。图中两条曲线重合的面积越少,说明预测模型对高身高及低身高的区分能力越强。图中显示,高身高与低身高密度曲线重合面积较大,表明低身高到高身高的变化趋势不明显。sequencelisting<110>公安部物证鉴定中心<120>一种用于辅助鉴定身高的方法及其专用引物组<130>gncyx182057<160>67<170>patentinversion3.5<210>1<211>30<212>dna<213>artificialsequence<400>1acgttggatgattaaccttggcacactggg30<210>2<211>30<212>dna<213>artificialsequence<400>2acgttggatgccactacactccaacttggt30<210>3<211>66<212>dna<213>artificialsequence<400>3ctctctctctctctctctctctctctctctctctctctctctctctcttggtgacaaagt60gatatt66<210>4<211>30<212>dna<213>artificialsequence<400>4acgttggatgctttgggaccctaaacaatg30<210>5<211>30<212>dna<213>artificialsequence<400>5acgttggatgccagtgtgccaaggttaata30<210>6<211>60<212>dna<213>artificialsequence<400>6ctctctctctctctctctctctctctctctctctctctcaactcttctttccttcttctt60<210>7<211>30<212>dna<213>artificialsequence<400>7acgttggatgctttgggttttggcaaggtg30<210>8<211>30<212>dna<213>artificialsequence<400>8acgttggatgtcctgaaaggtatacaagac30<210>9<211>36<212>dna<213>artificialsequence<400>9tctctctctctctctctctctggtgctcagcctttc36<210>10<211>30<212>dna<213>artificialsequence<400>10acgttggatgagtgcatgctggatgcaaac30<210>11<211>30<212>dna<213>artificialsequence<400>11acgttggatggggtatagaagctcaaatgc30<210>12<211>63<212>dna<213>artificialsequence<400>12ctctctctctctctctctctctctctctctctctctctctctctctctgaggacctacga60aat63<210>13<211>30<212>dna<213>artificialsequence<400>13acgttggatgaactgtatatgctacgcctg30<210>14<211>30<212>dna<213>artificialsequence<400>14acgttggatgtacatgcaaccatgttgccc30<210>15<211>72<212>dna<213>artificialsequence<400>15ctctctctctctctctctctctctctctctctctctctctctcctctctctgtaagggtt60tttcatggatta72<210>16<211>30<212>dna<213>artificialsequence<400>16acgttggatggctgtgcaaacttgcctatc30<210>17<211>30<212>dna<213>artificialsequence<400>17acgttggatgcacactgttcatcgcgattc30<210>18<211>48<212>dna<213>artificialsequence<400>18ctctctctctctctctctctctctctctcctatctctgaaaacagacg48<210>19<211>30<212>dna<213>artificialsequence<400>19acgttggatgcagagttcagtcacagattc30<210>20<211>30<212>dna<213>artificialsequence<400>20acgttggatggctaactgagggtcaaatta30<210>21<211>57<212>dna<213>artificialsequence<400>21ctctctctctctctctctctctctctctctctctctctttctccaaatgtgctgatt57<210>22<211>30<212>dna<213>artificialsequence<400>22acgttggatgatttttgagtgaccccaagg30<210>23<211>30<212>dna<213>artificialsequence<400>23acgttggatgaggtgcaggagagatcttac30<210>24<211>54<212>dna<213>artificialsequence<400>24tctctctctctctctctctctctctctctctctctaggaaaacaaatgaggaaa54<210>25<211>31<212>dna<213>artificialsequence<400>25acgttggatggaaaagacaaaagaagagacc31<210>26<211>31<212>dna<213>artificialsequence<400>26acgttggatggacaaaaaagcactgaaagtc31<210>27<211>69<212>dna<213>artificialsequence<400>27ctctctctctctctctctctctctctctctctctctctctctctcaaagtcaaaaattgt60tactagaaa69<210>28<211>30<212>dna<213>artificialsequence<400>28acgttggatggactaggtaagattagaggc30<210>29<211>30<212>dna<213>artificialsequence<400>29acgttggatgactggctgagcttctacttg30<210>30<211>42<212>dna<213>artificialsequence<400>30tctctctctctctctctctctctctacctccagagagtgacc42<210>31<211>29<212>dna<213>artificialsequence<400>31acgttggatgtgcatctctgagcaggcac29<210>32<211>29<212>dna<213>artificialsequence<400>32acgttggatgagtcacagccaggtgggta29<210>33<211>30<212>dna<213>artificialsequence<400>33ctctctctctctctccccaggagcctactc30<210>34<211>30<212>dna<213>artificialsequence<400>34acgttggatgctgagaagtactgcagttgg30<210>35<211>29<212>dna<213>artificialsequence<400>35acgttggatggggaagagagactatacac29<210>36<211>39<212>dna<213>artificialsequence<400>36tctctctctctctctctctcttaattccatacttcccca39<210>37<211>29<212>dna<213>artificialsequence<400>37acgttggatgtaaggaagaaagaagatgt29<210>38<211>30<212>dna<213>artificialsequence<400>38acgttggatgctcttctataatgcaaatcc30<210>39<211>90<212>dna<213>artificialsequence<400>39ctctctctctctctctctctctctctctctctctctctctctctctctctctctctctct60ctctctctctgaatcatttcaccatgaaca90<210>40<211>30<212>dna<213>artificialsequence<400>40acgttggatgggaatgaccctgatttcctc30<210>41<211>30<212>dna<213>artificialsequence<400>41acgttggatgtcctctgctcatttgctgtg30<210>42<211>80<212>dna<213>artificialsequence<400>42ctctctctctctctctctctctctctctctctctctctctctctctctctctctctctct60cgaggaggtttatttgggca80<210>43<211>30<212>dna<213>artificialsequence<400>43acgttggatggagcagtgcctattagactc30<210>44<211>29<212>dna<213>artificialsequence<400>44acgttggatgatgttatattggtcatgtc29<210>45<211>55<212>dna<213>artificialsequence<400>45ctctctctctctctctctctctctctctctctctctctggtcatgtcaaaatgag55<210>46<211>30<212>dna<213>artificialsequence<400>46acgttggatggagtgatttagcactctggg30<210>47<211>30<212>dna<213>artificialsequence<400>47acgttggatggactgtgttctacgcatttc30<210>48<211>75<212>dna<213>artificialsequence<400>48ctctctctctctctctctctctctctctctctctctctctctctctctctctctctcacg60catttcttcttgaac75<210>49<211>20<212>dna<213>artificialsequence<400>49gcacatgaacacatagaggg20<210>50<211>20<212>dna<213>artificialsequence<400>50aagcctatagtaccctttag20<210>51<211>65<212>dna<213>artificialsequence<400>51ctctctctctctctctctctctctctctctctctctctctctctctctcccaccctccag60taaac65<210>52<211>30<212>dna<213>artificialsequence<400>52acgttggatggatttcagtggtgaatgtgg30<210>53<211>30<212>dna<213>artificialsequence<400>53acgttggatgctcttaggttccaaaagtag30<210>54<211>45<212>dna<213>artificialsequence<400>54ctctctctctctctctctctctctctctatgtggttttggcatac45<210>55<211>30<212>dna<213>artificialsequence<400>55acgttggatgtcccctaaagtctcgggaag30<210>56<211>29<212>dna<213>artificialsequence<400>56acgttggatgcgtggtcactggtcttgtc29<210>57<211>60<212>dna<213>artificialsequence<400>57ctctctctctctctctctctctctctctctctctctctctctctccacccagttctgagc60<210>58<211>30<212>dna<213>artificialsequence<400>58acgttggatgaggagataacacacgatcac30<210>59<211>30<212>dna<213>artificialsequence<400>59acgttggatgtgttctcagatgtctggtcc30<210>60<211>50<212>dna<213>artificialsequence<400>60ctctctctctctctctctctctctctctctctctcagctggctccaacac50<210>61<211>30<212>dna<213>artificialsequence<400>61acgttggatgaaagggagcagcgtttgagg30<210>62<211>29<212>dna<213>artificialsequence<400>62acgttggatgagccctgtgtgcagcctaa29<210>63<211>70<212>dna<213>artificialsequence<400>63ctctctctctctctctctctctctctctctctctctctctctctctctctctctccctgc60gcgggccgcc70<210>64<211>30<212>dna<213>artificialsequence<400>64acgttggatgccaaacctggtttctttggg30<210>65<211>30<212>dna<213>artificialsequence<400>65acgttggatgtttcgcaactttgctgcacc30<210>66<211>85<212>dna<213>artificialsequence<400>66ctctctctctctctctctctctctctctctctctctctctctctctctctctctctctct60ctctctctcttggcttccaggtcag85<210>67<211>90<212>dna<213>homosapiens<400>67ccaggtggaccagcttcccctttctctcctctttctcctttgggaccctaaacaatgtta60aaaaaaaaaaaagaagaagaaggaaagaag90当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1