一种大豆脂类转运蛋白基因GmABCA9及其应用的制作方法
本发明属于基因工程领域,具体涉及一种大豆脂类转运蛋白基因及其应用。
背景技术:
大豆是我国重要的油料作物,也是人类优质蛋白的主要来源。既是我国主要作物之一,也是我国进口量最大的农产品。近年来,国内大豆作物的产油量已经不能满足日益增加的植物油需求量,国内市场上豆油均以进口油为主。因此,培育国内高油大豆品种,提高国产豆油产量迫在眉睫。
为培育高产高油大豆品种,研究人员通过基因工程手段调控油脂代谢途径中的脂肪酸合成酶和关键转录因子,但是对油脂含量的提升仅维持在10%~20%左右。
油脂的合成在叶绿体中进行,之后运输到内质网进行加工。目前,关于油脂在叶绿体和内质网间运输系统的研究较少,在大豆中尚未见报道。
技术实现要素:
本发明所要解决的技术问题为:如何进一步的提高大豆油脂含量。
本发明的技术方案为:大豆gmabca9基因,核苷酸序列如seqidno.1所示。
如seqidno.1所示的大豆gmabca9基因在调控植物种子油脂含量上的应用。
进一步地,所述植物为豆科植物。
进一步地,所述植物为大豆。
进一步地,所述植物为拟南芥。
含有如seqidno.1所示的大豆gmabca9基因的表达载体在植物种子脂质调控上的应用。
seqidno.1所示的大豆gmabca9基因编码的蛋白质,其氨基酸序列如seqidno.2所示。
gmabca9在植株叶片、茎、根、种子中均可以表达。在大豆成熟的种子发育后期,该基因的表达量较高。
gmabca9在烟草叶片中瞬时表达后,利用显微镜对转化后的烟草叶片和原生质体进行荧光信号观察,发现gmabca9定位在内质网。
拟南芥abca9突变体种子干瘪,粒小。当gmabca9在拟南芥中超量表达时,回复突变体种子恢复了和野生型一样的表型。在无蔗糖培养基上生长时,abca9突变体幼苗生长缓慢,发育不良,当gmabca9在拟南芥中超量表达时,回复突变体幼苗发育正常。这些结果表明gmabca9调控转基因拟南芥的种子发育过程。
过量表达gmabca9提高了种子油脂和c16:0、c18:1、c18:2和c18:3脂肪酸含量。表明gmabca9通过调控种子脂肪酸含量,促进油脂的积累。
与现有技术相比,本发明具有以下有益效果:
本发明明确了大豆gmabca9基因在大豆种子中的特异性表达和内质网定位的特性,该基因的表达调控拟南芥种子的发育,影响种子脂肪酸含量,表明该基因参与内质网脂质的转运系统,进而调控种子油脂的合成。
附图说明
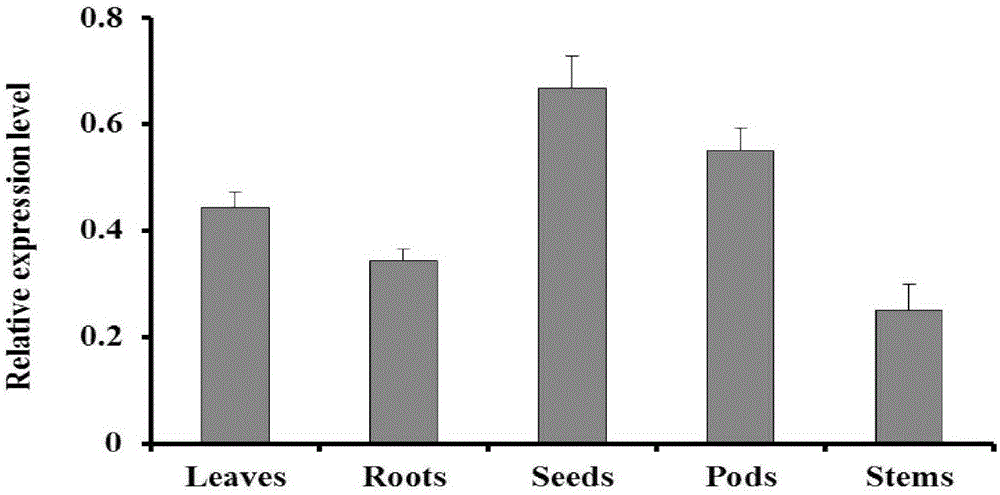
图1:gmabca9在大豆不同组织中的表达水平;leaves:叶片;roots:根;seeds:种子;pods:果荚;stems:茎;叶片、根和茎为播种30d的大豆幼苗植株的组织,种子和果荚为开花后30d的大豆组织,内参基因为大豆actin。
图2:35s:gmabca9-gfp载体构建;图中a,载体构建示意图。图中b,1、2分别代表样品1和样品2;m5:marker5000。35s:gmabca9-gfp经bamhi消化后出现的片段:1523bp和840bp。
图3:gmabca9在烟草叶片中荧光信号;图中a、b、c、d为烟草叶片;图中e、f、g、h为烟草叶片的原生质体。
图4:35s:gmabca9-gfp转基因拟南芥植株pcr鉴定;m5:分子标记marker5000;—:以dh2o为模板的阴性对照;wt:野生型拟南芥植株;1~15:转基因拟南芥植株。
图5:gmabca9在拟南芥不同组织中的表达水平;roots:根;siliques:角果;flowers:花;stems:茎;rosetteleaves:莲座叶;caulineleaves:茎生叶。角果为播种45d的拟南芥植株的组织,根、花、茎、莲座叶、茎生叶为播种30d的拟南芥,内参基因为拟南芥actin2。
图6:拟南芥abca9回复突变体植株的筛选;m5:分子标记marker5000;—1:以dh2o为模板的阴性对照;—2:以abca9突变体基因组dna为模板的阴性对照;wt:野生型拟南芥植株;1~14:拟南芥abca9回复突变体植株。
图7:拟南芥abca9突变体和回复突变体表型;wt:野生型拟南芥植株;oe-2:超量表达株系2;2-c1:abca9-1回复突变体株系2;2-c2:abca9-2回复突变体株系2;2-c3:abca9-3回复突变体株系2。
图8:转基因、突变体和回复突变体植株种子的油脂含量;图中a,超表达植株种子油脂含量;图中b,突变体和回复突变体植株种子油脂含量。
图9:转基因、突变体植株种子的脂肪酸含量;图中a,转基因植株种子脂肪酸含量;图中b,abca9突变体植株种子脂肪酸含量。
图10:超表达、突变体和回复突变体植株种子的千粒重;图中a,转基因种子千粒重;图中b,突变体和回复突变体种子千粒重。
具体实施方式
gmabca9基因的克隆
以大豆黑农44品种rna为模板,合成cdna后,通过pcr扩增,扩增引物对为:
5'-tgctctagaatggcaaccactctcacc-3'
5'-tgctctagaccactgaaaagctactgaact-3',
得到2904bp的基因序列,如seqidno.1所示,命名为大豆gmabca9基因;共编码967个氨基酸,如seqidno.2所示。
gmabca9在大豆组织中的表达分析
基因的时空表达在一定程度上为目的基因功能的验证提供了重要依据。为了分析gmabca9在大豆不同组织中的表达水平,我们提取了播种30d的黑农44大豆叶片、根、茎、开花后30d种子和果荚的rna,通过实时定量pcr检测gmabca9在大豆不同组织中的表达水平(如图1)。
对实时定量pcr结果进行分析后发现,gmabca9在播种30d的大豆组织中表达量均比开花后30d的种子、果荚中低。而在开花30d的种子和果荚中,该基因在种子中表达量较高。播种30d的大豆正处于营养生长时期,而gmabca9在根、茎、叶中的表达可能为植株营养生长提供营养物质。从开花到种子成熟是大豆植株的生殖生长阶段,与果荚相比,开花后30d的种子中该基因的表达量较高,表明gmabca9可能在开花后30d的大豆种子的成熟过程中发挥功能,调控种子的发育过程。
gmabca9的亚细胞定位
为了分析gmabca9蛋白的亚细胞定位情况,我们将大豆中克隆的gmabca9基因,在去除终止密码子后,与pcambia1300的gfp标记融合,构建了35s:gmabca9-gfp载体。使用限制性内切酶bamhi进行酶切验证,结果显示均出现1523bp和840bp的目的片段(如图2),表明载体构建成功。
为了分析gmabca9亚细胞定位情况,我们将构建好的35s:gmabca9-gfp载体转入农杆菌gv3101中,利用农杆菌侵染幼嫩的烟草叶片,在激光共聚焦显微镜下观察gmabca9的荧光信号(如图3)。在烟草叶片的显微图中,使用空载侵染烟草叶片后,在图3中a中可以观察到很强的绿色荧光信号,由于35s强启动子在植物中的组成性表达,可以观察到多处荧光信号,主要分布在细胞核、内质网和细胞膜等。当由35s强启动子启动gmabca9基因的表达时,在35s:gmabca9-gfp侵染的烟草叶片中(图3中b)发现绿色荧光信号环绕在细胞核的周围,而且在细胞质中可以观察到很明显的丝状信号,与图3中c中内质网marker的红色荧光信号一致。将该基因绿色荧光信号与内质网marker红色信号重叠之后,在图3中d中可以观察到黄色信号,表明gmabca9定位在内质网。
为了排除植物细胞壁杂质和其它物质对目的基因荧光信号的干扰,我们提取了被侵染过的烟草叶片原生质体,在显微镜下进行观察。和烟草叶片中的信号相似,与对照图3中e相比,图3中f和g的重叠表明gmabca9定位在内质网。
gmabca9在转基因拟南芥中的表达
为了验证gmabca9的功能,我们通过浸花法侵染野生型拟南芥,将目的基因转入拟南芥中。将所获得的t1转基因植株种子在含有hyg和cef的1/2ms的培养基上进行筛选,共筛选得到25株阳性苗。移栽待其长成健壮幼苗后,提取叶片基因组dna进行pcr验证。目的条带为672bp,最终筛选到23株阳性植株(部分扩增结果如图4)。
gmabca9在拟南芥中的表达
在大豆中已经证实了gmabca9在成熟的种子中较高的表达,暗示该基因可能参与调控种子的发育过程。为验证该基因在拟南芥植株中的表达模式,我们分析了gmabca9在转基因拟南芥不同组织中的表达水平。提取播种30d的拟南芥莲座叶、茎生叶、根、茎、花和播种40d角果的rna,通过实时定量pcr检测了gmabca9在转基因拟南芥不同组织中的表达水平(如图5)。
对实时定量pcr结果进行分析发现,gmabca9在拟南芥角果中表达量最高。在根、茎、花、莲座叶和茎生叶中该基因表达量较低,尤以茎生叶最为显著,表达量几乎为0;而在根、茎、花三种组织中该基因的表达量并没有明显的区别。在拟南芥种子发育过程中,角果源源不断地为种子提供所需营养物质。因此,gmabca9在角果中的大量表达,表明该基因可能参与了转基因种子成熟的发育过程。
大豆gmabca9基因回复拟南芥abca9突变体植株的筛选
将构建好的35s:gmabca9-gfp载体转入农杆菌gv3101中,利用农杆菌侵染拟南芥突变体植株。将目的基因gmabca9转入拟南芥abca9突变体中,收取t1代回复突变体种子,在含有hyg和cef的1/2ms的培养基上进行筛选。移栽待其长成健壮幼苗后,提取回复突变体植株叶片基因组dna进行pcr验证,目的条带为672bp的株系即为阳性植株(部分扩增结果如图6)。结果表明,在abca9-1-c植株中,除株系11外,其它株系均为回复突变体植株。abca9-2-c和abca9-3-c植株的株系1~14均为回复突变体植株。按此方法继续进行筛选至t3代,3种回复突变体植株每种共计筛选到20个株系。
拟南芥abca9突变体和回复突变体种子表型分析
研究报道,拟南芥atabca9的表达会影响种子油脂的产量。为了验证该基因的表达是否会影响拟南芥种子的表型,我们观察了拟南芥abca9突变体和回复突变体的种子表型。
在显微镜下观察三种abca9突变体种子的表型(如图7),发现与wt相比,abca9-1和abca9-2突变体的种子干瘪,粒小,而abca9-3种子表型与wt几乎一致。当gmabca9在野生型和突变体植株中表达时,转基因植株种子与wt相比种子饱满且粒大;而abca9-1和abca9-2回复突变体种子明显比突变体种子更加饱满,种粒变大,尤以abca9-1回复突变体种子表型最为显著;但是abca9-3回复突变体种子的表型和突变体并无明显的差异,可能是因为该突变体的t-dna插入位点位于该基因的内含子区域,并没有完全抑制该基因的表达。结果表明,abca9的突变影响了种子的饱满程度和种粒大小。
拟南芥植株种子油脂含量测定
通过对三种拟南芥突变体和回复突变体种子表型和无蔗糖培养基上的生长情况研究分析后,我们选取了表型比较显著的突变体abca9-1和回复突变体abca9-1-c进行后续的功能研究。为了验证gmabca9的功能,我们检测了gmabca9转基因植株、突变体和回复突变体植株种子油脂的含量(如图8)。与wt植株相比,转基因植株株系oe-1、oe-2、oe-4的种子油脂含量显著增加(如图8中a),分别增加了15%、17%、11%。abca9-1突变体种子油脂含量与野生型植株相比急剧下降。而当gmabca9在abca9-1突变体中过量表达时,与突变体相比,回复突变体株系2-c1和3-c1种子的油脂含量显著增加(如图8中b),分别增加了12%和9%。结果表明,gmabca9基因的表达增加了拟南芥植株种子油脂的含量。
拟南芥植株种子脂肪酸含量测定
为了验证转基因植株种子油脂含量的增加是否由某种脂肪酸的增加而导致的,我们检测了种子中不同脂肪酸的相对含量(如图9)。与野生型植株相比,突变体植株种子c18:2和c18:3的相对含量虽然增加,但是c16:0和c18:1的含量并没有发生变化(如图9中b)。而abca9-1突变体种子总脂含量却呈现急剧下降的趋势,表明abca9的突变可能上调或下调了其他脂肪酸的水平,使脂质总量整体降低。在转基因植株oe-1、oe-2、oe-3和oe-4种子中,c16:0、c18:1、c18:2和c18:3的相对含量均显著增加(如图9中a)。在oe-2种子中,c16:0、c18:1、c18:2和c18:3的相对含量分别增加了3.4mg/g、11.4mg/g、15.4mg/g和10.2mg/g。结果表明,gmabca9通过调控种子脂肪酸的含量,促进种子油脂的积累。
拟南芥植株种子千粒重分析
gmabca9基因的表达影响种子脂肪酸和糖类的含量,为了验证该基因的表达是否会影响到种子的重量,我们选取了1000粒转基因、突变体和回复突变体成熟的种子,对其重量进行检测。结果发现(如图10)。与对照18mg相比,转基因oe-2种子千粒重为21mg,显著增加了18%,而三种突变体和回复突变体种子的千粒重和野生型一致。结果表明,gmabca9过量表达增加种子的重量,是否通过调控种子脂肪酸和葡萄糖含量来影响种子重量还有待进一步研究。
通过上述实验验证大豆转运蛋白gmabca9调控油脂的合成。荧光实时定量pcr分析显示,gmabca9在大豆种子成熟期表达量较高,种子成熟期是种子油脂积累的重要时期。gmabca9亚细胞定位显示该基因定位在内质网,和atabca9具有相同的定位。拟南芥abca9突变体种子干瘪,粒小,并且在无蔗糖培养基上生长时,幼苗黄化,发育不正常。而gmabca9回复突变体植株种子在无蔗糖培养基生长时,表型均正常。拟南芥abca9突变体油脂含量急剧下降,组成油脂的部分脂肪酸组分含量在abca9突变体种子中没有明显变化,可能是突变体种子中另外的脂肪酸我们暂未分析到。gmabca9转基因种子油脂和脂肪酸组分含量均显著增加,表明gmabca9基因的表达通过增加种子脂肪酸的量增加油脂的合成。
序列表
<110>申请人名称西南大学
<120>一种大豆脂类转运蛋白基因gmabca9及其应用
<130>20181127
<160>2
<170>siposequencelisting1.0
<210>1
<211>2904
<212>dna
<213>大豆(glycinemax)
<400>1
atggcaaccactctcaccggcatctcgctggtggcgctgcaattcaaggccttgctgaag60
aaaaatctcttgctgtcatggcggaacaagagagccagcctcttacagttactctcccct120
ctcatattcattttcctcatcttcgcaatcgacaaggccatcaaggcacagacctcgacc180
tcctcctcctacaagagcgtcactgaccctcccatggaaccttctccccccatcactccc240
tgcgaggacaagttcttcatcaaactcccctgctacgacttcgtctggagcggccacgcc300
agccccacgttccagaccatcgtcgcccgcatcatgaacaacaaccccggccgccccatc360
cctccctccaaggtgaagtcttttaaggaaaagagtgaagtggatgcgtggcttctcagc420
aaccctatgcggtgccctggagcgcttcatttttcggagaggaacgacaccgttataagc480
tacggtcttcagaccaattccaccagtcttcagcggcgagggaagtacgaggatcccacg540
gcttcgtttcagctccctcttcagcttgctgccgagagggaaattgccagataccttatt600
ggagatgcggatttcagttggaatgtgtttctgagggaattcgcgcacccttccatgaat660
cctttctctgctgtggcttcaattggtcctgcatttttcctcgcgatcgccatgtttaat720
tttgttcttcagattagctctttggtgacggagaaagagcttaaacttcgccaggcaatg780
aatatgatggggctttacgactttgcttactggttttcctggctcatctgggaggcagtt840
gtcacaatcctatcgtccctcctcatagttctctttggaatgatgttccagtttcgcttt900
ttcttggataacagttttgtggttctgttcgttctgttcttcctgtttgaacttaatatg960
actggcttggccttcatgatatctgctttcattcggaaatcatcttcggcaacaacagtg1020
gggttctctatatttattgtcggctttgtgactcagcttgtggtacaacaaggatttcct1080
tacacagatagcttctccaaaactatacgaaacgtgtggtcgttattccctcccaatctt1140
tttgctcaaggtataaaagtgctttcagatgcagttgcaacttctgaagacaaaggcatt1200
agctggagtaaacggggagaatgtgctcttaatgattcagactgtgtgataactattgat1260
gacatttataaatggcttgcggctactttctttctctggtttgttctggccatttacttt1320
gacaatataatcccaaatgcatcaggtgtgaggaaatcaatattgtactttctaaatccc1380
agttattggatgggaaaaggaggtcaaaaagtgaaagagggtggggtttgtagctgtata1440
ggttcagccccacgtcaagaacaaagtacaccagatgatgaggatgtccttgaagaagaa1500
aacaaagtgaaacagcaactaacagaaggtcttcttgatgcaaatgttgctgttcagata1560
cgtggccttgcaaagacatatcctggcacacgtagcattggttgctgctttaaatgtaaa1620
agaacttcaccttacaatgcagttaagggtttgtgggtgaactttgcaaaggatcagtta1680
ttttgtcttctaggacccaatggagctggaaaaactacagctattaattgcttgacaggg1740
gtaactccagtaactgatggagatgcgttgatttatgggcattcaatcagaagttctact1800
ggcatgtcaaacattcgaaagcttataggagtctgtccacagtttgatatcctatgggat1860
gcactgtctggtcaggaacacctccaactctttgctactattaaaggcctgtccccagct1920
tccataaaatcaattacccagacttcattagcagaggtaagactcacggatgcagccaaa1980
gtgagagctggaagttacagcggaggaatgaaacgccgtctcagtgttgcaattgccctt2040
attggtgaccctaagttagtcattctggatgaaccgactactggtatggatccaataaca2100
agaaggcatgtgtgggacataattgaaaatgcaaagagagggcgtgccattgttcttaca2160
acgcactctatggaagaagctgacattctaagtgaccgtataggaatcatggcaaaagga2220
agtcttcgctgcattggcacctcaatcagattgaagtcccgctttggtactggttttatt2280
gctaatatcagcttcaatggaaacaatatcgaacatagtcctgccaatggagatgcaata2340
tctacagaacgtcgtgaggcagtgaagaaattctttaagaatcatttagatgtagtgcca2400
aaagaggagaaccataacttcctaacttttgtgattcctcatgacagagaggcgctcatg2460
acgaatttcttttcagagctccaagatagagaagaagaatttggcatatctgatatccag2520
cttggtctaacaacgctcgaagaagttttcttgaatattgctagacaagcagagctagaa2580
agtgctgcagctgaagggagactagtgaccctgaccttaacatctggggaatctgtgcag2640
attcctataggagctaggtttgtgggaattccaggaacagagtctgctgaaaaccctact2700
gggtttatggtagaagtatactgggaacaagatgatactggtgccttatgcattgctggc2760
cactcacagaaggttcctattcctaatggcgttcaactatcttcttctccatctgtaaga2820
caccgtagatatttaggccggtcaggaacagttcatggggttgtgattgatccaagtcaa2880
gttagttcagtagcttttcagtga2904
<210>2
<211>967
<212>prt
<213>大豆(glycinemax)
<400>2
metalathrthrleuthrglyileserleuvalalaleuglnphelys
151015
alaleuleulyslysasnleuleuleusertrpargasnlysargala
202530
serleuleuglnleuleuserproleuilepheilepheleuilephe
354045
alaileasplysalailelysalaglnthrserthrsersersertyr
505560
lysservalthraspproprometgluproserproproilethrpro
65707580
cysgluasplysphepheilelysleuprocystyraspphevaltrp
859095
serglyhisalaserprothrpheglnthrilevalalaargilemet
100105110
asnasnasnproglyargproileproproserlysvallysserphe
115120125
lysglulyssergluvalaspalatrpleuleuserasnprometarg
130135140
cysproglyalaleuhisphesergluargasnaspthrvalileser
145150155160
tyrglyleuglnthrasnserthrserleuglnargargglylystyr
165170175
gluaspprothralaserpheglnleuproleuglnleualaalaglu
180185190
arggluilealaargtyrleuileglyaspalaaspphesertrpasn
195200205
valpheleuarggluphealahisprosermetasnpropheserala
210215220
valalaserileglyproalaphepheleualailealametpheasn
225230235240
phevalleuglnileserserleuvalthrglulysgluleulysleu
245250255
argglnalametasnmetmetglyleutyraspphealatyrtrpphe
260265270
sertrpleuiletrpglualavalvalthrileleuserserleuleu
275280285
ilevalleupheglymetmetpheglnpheargphepheleuaspasn
290295300
serphevalvalleuphevalleuphepheleuphegluleuasnmet
305310315320
thrglyleualaphemetileseralapheilearglysserserser
325330335
alathrthrvalglypheserilepheilevalglyphevalthrgln
340345350
leuvalvalglnglnglypheprotyrthraspserpheserlysthr
355360365
ileargasnvaltrpserleupheproproasnleuphealaglngly
370375380
ilelysvalleuseraspalavalalathrsergluasplysglyile
385390395400
sertrpserlysargglyglucysalaleuasnaspseraspcysval
405410415
ilethrileaspaspiletyrlystrpleualaalathrphepheleu
420425430
trpphevalleualailetyrpheaspasnileileproasnalaser
435440445
glyvalarglysserileleutyrpheleuasnprosertyrtrpmet
450455460
glylysglyglyglnlysvallysgluglyglyvalcyssercysile
465470475480
glyseralaproargglngluglnserthrproaspaspgluaspval
485490495
leugluglugluasnlysvallysglnglnleuthrgluglyleuleu
500505510
aspalaasnvalalavalglnileargglyleualalysthrtyrpro
515520525
glythrargserileglycyscysphelyscyslysargthrserpro
530535540
tyrasnalavallysglyleutrpvalasnphealalysaspglnleu
545550555560
phecysleuleuglyproasnglyalaglylysthrthralaileasn
565570575
cysleuthrglyvalthrprovalthraspglyaspalaleuiletyr
580585590
glyhisserileargserserthrglymetserasnilearglysleu
595600605
ileglyvalcysproglnpheaspileleutrpaspalaleusergly
610615620
glngluhisleuglnleuphealathrilelysglyleuserproala
625630635640
serilelysserilethrglnthrserleualagluvalargleuthr
645650655
aspalaalalysvalargalaglysertyrserglyglymetlysarg
660665670
argleuservalalailealaleuileglyaspprolysleuvalile
675680685
leuaspgluprothrthrglymetaspproilethrargarghisval
690695700
trpaspileilegluasnalalysargglyargalailevalleuthr
705710715720
thrhissermetgluglualaaspileleuseraspargileglyile
725730735
metalalysglyserleuargcysileglythrserileargleulys
740745750
serargpheglythrglypheilealaasnileserpheasnglyasn
755760765
asnilegluhisserproalaasnglyaspalaileserthrgluarg
770775780
argglualavallyslysphephelysasnhisleuaspvalvalpro
785790795800
lysglugluasnhisasnpheleuthrphevalileprohisasparg
805810815
glualaleumetthrasnphephesergluleuglnasparggluglu
820825830
glupheglyileseraspileglnleuglyleuthrthrleugluglu
835840845
valpheleuasnilealaargglnalagluleugluseralaalaala
850855860
gluglyargleuvalthrleuthrleuthrserglygluservalgln
865870875880
ileproileglyalaargphevalglyileproglythrgluserala
885890895
gluasnprothrglyphemetvalgluvaltyrtrpgluglnaspasp
900905910
thrglyalaleucysilealaglyhisserglnlysvalproilepro
915920925
asnglyvalglnleuserserserproservalarghisargargtyr
930935940
leuglyargserglythrvalhisglyvalvalileaspprosergln
945950955960
valserservalalaphegln
965
- 还没有人留言评论。精彩留言会获得点赞!