一种谷氨酸脱羧酶重组菌及其构建方法和应用与流程
本发明涉及基因工程领域,尤其涉及一种谷氨酸脱羧酶重组菌及其构建方法和应用。
背景技术:
目前,γ-氨基丁酸(γ-aminobutyricacid,简称gaba)是哺乳动物神经中枢的一种抑制性递质,具有调节血压、促进精神安定、促进脑部血流、增进脑活力、营养神经细胞、增加生长激素分泌、健肝利肾、预防肥胖、促进乙醇代谢、改善更年期综合征等多种功效。γ-氨基丁酸正被广泛应用于医药、食品保健、化工及农业等行业。gaba的生产可以通过化学合成和生物合成。由于化学合成反应条件剧烈,化学原料和溶剂具有毒性和腐蚀性,副产物多,缺乏安全性,主要应用于化工行业,不适宜作为食品添加剂和医药。生物合成条件温和,专一性强、副产物少、安全性高,因此以生物合成法生产食品或者医药级的gaba是一条较为理想的途径。
谷氨酸脱羧酶(glutamatedecarboxylase,gad)是生物体内催化谷氨酸发生α-脱羧生成gaba的唯一的酶。目前已经在多种微生物中发现gad的存在,利用微生物中的gad催化谷氨酸生产γ-氨基丁酸不受资源、环境和空间的限制,具有显著的有点。目前利用基因工程中常利用过量表达谷氨酸脱羧酶基因,来进一步提高gaba的产量,工业上用的gad大多是从各种微生物源中分离出来的,包括大肠杆菌、乳酸菌等,存在产量低,操作繁琐,经济效益等不足。
技术实现要素:
为了克服现有技术的不足,本发明的目的之一在于提供一种适合大规模工业化生产的谷氨酸脱羧酶重组菌,采用将来源于不同链霉菌的ssgad、stgad、scgad克隆后在大肠杆菌e.colibl21(de3)进行过量表达,生产谷氨酸脱羧酶。
本发明的目的之二在于提供了一种谷氨酸脱羧酶重组菌的构建方法。
本发明的目的之三在于提供了一种谷氨酸脱羧酶重组菌的应用。
本发明的目的之一采用如下技术方案实现:
一种谷氨酸脱羧酶重组菌,该重组菌为导入了谷氨酸脱羧酶基因的大肠杆菌,其中所述的谷氨酸脱羧酶基因来源于弓形链霉菌nrrl15443的stgad、链霉菌mj654-nf4的ssgad和嗜铬链霉菌atcc49982的scgad,其核苷酸序列如seqidno:1-3所示。
本发明的目的之二采用如下技术方案实现:
上述谷氨酸脱羧酶重组菌的构建方法,包括以下步骤:
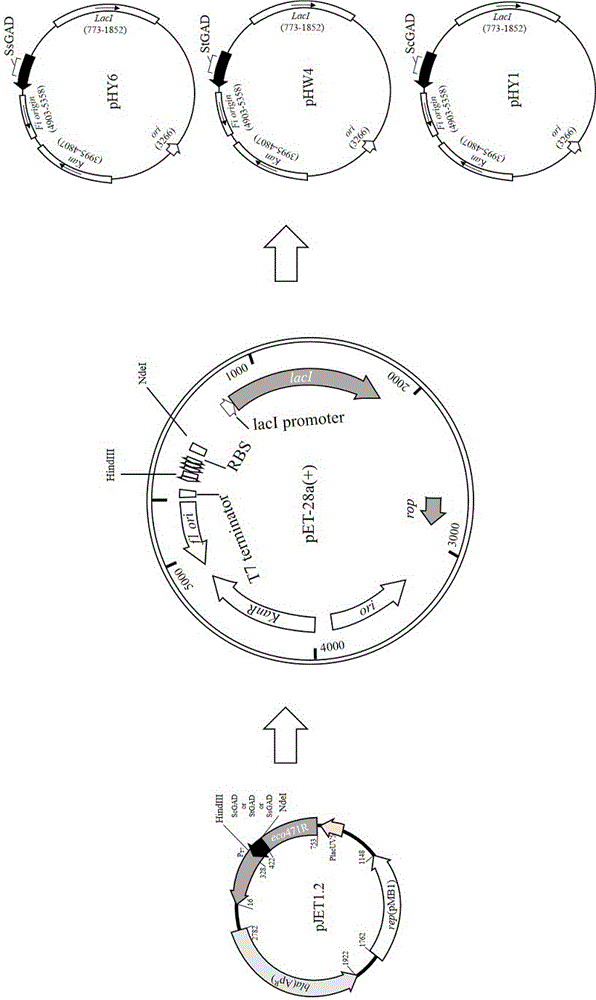
(1)构建质粒:将分别将ssgad、stgad、scgad经pcr扩增后,以pjet1.2作为基因克隆载体,将pcr扩增后的基因序列连接在其上,采用限制性内切酶针对酶切位点ndei和hindiii进行酶切处理,分别将ssgad结合物、stgad结合物、scgad结合物连接到相应制粒上;
(2)构建重组菌:分别将上述步骤(1)中的目标质粒导入相应菌种中即得所述重组菌。
进一步地,上述步骤(1)的制粒为大肠表达质粒pet-28a(+),分别得到目标制粒phy6、phw4、phy1;上述步骤(2)将目标制粒phy6、phw4、phy1导入大肠杆菌e.colibl21(de3)中即的所述重组菌。
进一步地,该基因序列也可以用于其他菌种的表达,选择不同的质粒时,可在酵母菌、乳酸菌、双歧杆菌等中进行表达。
进一步地,上述步骤(1)中pcr扩增中ssgad基因的引物序列为:
5′-aacatatggccttgtacaagggcaccg
3′-aaaagcttttagtggtggaagccggcgcggacc;
stgad基因的引物序列为:
5′-aacatatggctctccacaagacgaagga
3′-aaaagcttttagtggtggaagccggagcgggga;
scgad基因的引物序列为:
5′-aacatatgccactccaccaaggcgcggaca
3′-aaagcttttagtggtggaaggcggtggcggcc。
进一步地,上述步骤(1)中pcr扩增过程的反应条件为:98℃初始变性处理5分钟。随后在相同温度下,每30秒循环变性、退火、延长处理,完成20个循环;持续上述循环,并在每个循环后降温0.5℃,直至75℃;72℃下延伸2分钟,再依次在98℃、65℃下完成变性、退火、延长处理各30秒,72℃延长处理12分钟,即完成。
本发明的目的之三采用如下技术方案实现:
上述谷氨酸脱羧酶重组菌的应用,包括将谷氨酸脱羧酶重组菌进行扩大培养以诱导其大量表达谷氨酸脱羧酶。
进一步地,所述重组菌的扩大培养中还包括向培养基中添加iptg作为诱导剂的步骤。
进一步地,上述扩大培养过程为:将所述重组菌采用含有50μg/ml卡那霉素的lb培养液,37℃,250rpm培养12小时,然后转入含有相同浓度卡那霉素的lb肉汤培养基中,37℃,250rpm培养至od600达到0.4-0.6,添加iptg诱导目标蛋白的表达,28℃,250rpm下持续培养16小时,收集培养液,离心,将获得的细胞体通过超声裂解后再次进行离心,取上清液即的目标蛋白。
进一步地,将其或其产生的谷氨酸脱羧酶应用于生产γ-氨基丁酸。
进一步地,本发明还提供一种氨基酸序列,由上述核苷酸序列形成。
进一步地,所述氨基酸序列为seqidno.10-12所示序列、及其多聚物中的任意一种或几种。
相比现有技术,本发明的有益效果在于:本发明采用弓形链霉菌nrrl15443、链霉菌mj654-nf4和嗜铬链霉菌atcc49982作为菌株,通过pcr扩增技术从上述三种菌株的基因组上扩增得到了谷氨酸脱羧酶的编码基因:ssgad、stgad、scgad,利用大肠杆菌e.colibl21(de3)作为宿主,成功构建了能够高效表达谷氨酸脱羧酶的重组菌,该工程菌经历至少10次的重复利用依然保持极高的转化效率,大大提高了生产效率。本发明还提供了上述重组菌的构建方法及该重组菌在制备gaba的应用,上述方法生产易操作,易放大,稳定性好,适于大规模工业化生产。重组菌表达的gad可对谷氨酸直接进行脱羧,便于合成gaba,进一步降低生产成本。
附图说明
图1为本发明得到的重组蛋白电泳图;
图2为本发明得到的重组蛋白在一定ph范围的酶活性;
图3为本发明得到的重组蛋白在一定温度范围内的酶活性;
图4为本发明得到的重组蛋白在催化l-谷氨酸生产γ-氨基丁酸过程中,l-谷氨酸的浓度和γ-氨基丁酸产量之间的关系图。
图5为本发明得到的重组蛋白酶在催化l-谷氨酸生产γ-氨基丁酸过程中,l-谷氨酸的浓度和γ-氨基丁酸产量之间的关系图;
图6为本发明实施例制粒构建的流程图。
具体实施方式
下面,结合附图以及具体实施方式,对本发明做进一步描述,需要说明的是,在不相冲突的前提下,以下描述的各实施例之间或各技术特征之间可以任意组合形成新的实施例。
实施例1
一种谷氨酸脱羧酶重组菌的构建方法,包括以下步骤:
(1)构建质粒:分别将来源于弓形链霉菌nrrl15443的stgad、链霉菌mj654-nf4的ssgad或嗜铬链霉菌atcc49982的scgad,进行pcr扩增,所用引物序列如表1所示,20μlpcr体系的组成为:前端引物0.2μl,后端引物0.2μl,基因组dna0.2μl,dmso0.4μl,phusiondna聚合酶0.2μl,phusiondna聚合酶缓冲液4μl,100mmdntps0.4μl,水14.4μl;pcr扩增过程的反应条件为:98℃初始变性处理5分钟。随后在相同温度下,每30秒循环变性、退火、延长处理,完成20个循环;持续上述循环,并在每个循环后降温0.5℃,直至75℃;72℃下延伸2分钟,再依次在98℃、65℃下完成变性、退火、延长处理各30秒,72℃延长处理12分钟,即完成,以pjet1.2作为基因克隆载体,将pcr扩增后的基因序列连接在其上,采用限制性内切酶针对酶切位点ndei和hindiii进行酶切处理,分别将ssgad结合物、stgad结合物、scgad结合物连接到大肠表达质粒pet-28a(+)上,形成目标质粒phy6、phw4、phy1,制粒构件流程如图6所示;
(2)构建重组菌:分别将上述步骤(1)中的目标质粒phy6、phw4、phy1导入大肠杆菌e.colibl21(de3)中即的所述重组菌。
表1
实施例2
上述谷氨酸脱羧酶重组菌的应用:采用含有50μg/ml卡那霉素的lb培养液,37℃,250rpm培养12小时,转入含有相同浓度卡那霉素的lb肉汤培养基中,37℃,250rpm培养至od600达到0.4-0.6。添加iptg诱导目标蛋白的表达。28℃,250rpm下持续培养16小时。收集培养液,4℃下12000×g离心10分钟,获得的细胞体通过超声裂解后再进行12000×g离心10分钟,目标蛋白位于上清液中。
试验例1
重组蛋白的确认:将实施例2得到的目标蛋白进行电泳分析,结果如附图1所示,其中,gadb是已知的大肠杆菌gad酶,说明构建的重组菌均正常产生gad酶。
试验例2
最佳酶活性ph测定:采用200mm磷酸氢二钠-柠檬酸缓冲液,37℃下处理30分钟,测定重组蛋白在2.6-6.0的ph范围下的活力,结果如图2所示。由图2可以看出,不同来源的蛋白其最佳酶活ph不同。
试验例3
最佳酶活性温度的测定:采用在稳定ph3.5-5.0下,于18-60℃温度下处理30分钟,测定重组蛋白的最适活性温度,结果如图3所示。
试验例4
酶活力测定:将gadb和本申请的ssgad、stgad、scgad在相同条件下培养,统计对谷氨酸的转化效率,结果如图4所示,可以看出无论在ph4.6条件下或者是水环境下,ssgad、scgad对谷氨酸的转化效率均远高于gadb,stgad对谷氨酸的转化效率和gadb基本持平。
试验例5
本申请的重组蛋白酶在催化l-谷氨酸生产γ-氨基丁酸过程中,l-谷氨酸的浓度和γ-氨基丁酸产量之间的关系如图5所示,可以看出本申请的三种重组蛋白均有较好的转化效率。
本发明采用弓形链霉菌nrrl15443、链霉菌mj654-nf4和嗜铬链霉菌atcc49982作为菌株,通过pcr扩增技术从上述三种菌株的基因组上扩增得到了谷氨酸脱羧酶的编码基因:ssgad、stgad、scgad,利用大肠杆菌e.colibl21(de3)作为宿主,成功构建了能够高效表达谷氨酸脱羧酶的重组菌,该工程菌经历至少10次的重复利用依然保持极高的转化效率,大大提高了生产效率。本发明还提供了上述重组菌的构建方法及该重组菌在制备gaba的应用,上述方法生产易操作,易放大,稳定性好,适于大规模工业化生产。重组菌表达的gad可对谷氨酸直接进行脱羧,便于合成gaba,进一步降低生产成本。
上述实施方式仅为本发明的优选实施方式,不能以此来限定本发明保护的范围,本领域的技术人员在本发明的基础上所做的任何非实质性的变化及替换均属于本发明所要求保护的范围。
序列表
<110>杭州唯铂莱生物科技有限公司
<120>一种谷氨酸脱羧酶重组菌及其构建方法和应用
<141>2018-12-29
<160>12
<170>siposequencelisting1.0
<210>1
<211>1395
<212>dna
<213>人工序列(artificialsequence)
<400>1
atggctctccacaagacgaaggacgtcaccggcggcgacaccgggacggacgtcttcacc60
accgccctgagcggccgcgtcctccccaagtaccggatgccggaggagcattcaccgtcg120
gaggcggtctacgccctgctccgcaacgagctcctcctcgacggcaacgccgcccagaac180
ctggccaccttctgcaccacctggtccgacgacggcgtccaccgcctcatgaacgagtgc240
cttgacaagaacatggtcgacaaggacgagtacccgcagaccgcggagatcgaggcccgc300
tgcgtcaacatcctggcccacctgtggcacgccccggccgaggacggccccgccaccggc360
tgctccaccaccggctccagcgaggccgccatgctcggcggcctcgccctcaaatggcgc420
tggcgggcccgccgccgcgcccagggcctgccggccgaccggcccaacctggtgtgcggg480
cccgtccaggtctgctgggagaagttcgcccgctacttcgacgtcgaactgcgccagatc540
cccctggaggaggacgccaccggcctgcggccccaccagctcgcggagtacgtcgacgag600
aacaccatcggcgtcgtcgccatcctcggcgtcacctacacctgcgactacgagcccgtc660
gccgacatcgcggccgccctcgacgcgatccagcaggagcacggctgggacgtcccgatc720
cacgtcgacggcgccagcggcgggctcgtcgcccccttcctccaccccgacgtggtgtgg780
gacttcgccctcccccgcgtcgcctccatcaacacctccgggcacaagtacgggctcgca840
cccctcggcgtcggctgggtcgtctggcgcaccgcggacctcctgccccccgagctcgtc900
ttcgacgtggactacctgggcggcgacatgccgaccttcgccctgaacttctcccggccc960
ggcggcgaggccatcgcccagtactacctcttcctccggctcggccgcagcggctaccgg1020
agcgtccaccagtcctgcgcggacaccgcccggttcctcgccggcgagatcgccgcgatg1080
ggccccttcaccctgctgtacgacggccagggcgccctccccgccgtctcctaccggctc1140
accgaccccgcggccgccggcttcaccctctacgacctctccgaccggctgcggatgcgc1200
ggctggcaggtccccgcgtacccgctgcccgcccgccgcgacgacaccgtcatccagcgc1260
gtcctgatccgccacggcgtgaccctggaccagatcgccctcctggcggaggacatgcgc1320
cgcgccttggaccacctccgcgcggcgcccccgacccagcccccgacggctccccgctcc1380
ggcttccaccactga1395
<210>2
<211>1401
<212>dna
<213>人工序列(artificialsequence)
<400>2
atggccttgtacaagggcaccggcagcgaccgtcggctgaacgtcagtccgttactggtc60
accaacccgctcgccgcgatggagctcgccccgcccgtccaccggctcggcgacggcgcg120
gtctcggccgacatcgcgtaccagctggtccacgacgagctgatgctggacggcagcgcc180
aagctcaacctggcgaccttcgtcaccacctcgatggaggtgcaggccacccggctgatg240
accgagtgcctggacaagaacatgatcgacaaggacgagtacccgcagaccgccgagctg300
gagcggcgctgcgtggccatcctcgccgacctctggcacgcccccgacccggacaccgcg360
gtcggctgctccaccaccggctccagcgaggcctgcatgctcgccgggatggccctcaag420
cgccgctggatgcgccgcaaccccgagcgctacgcggccggggcccggcccaacctggtg480
atgggcgtcaacgtccaggtgtgctgggagaagttctgcaacttctgggaggtcgaggcc540
cgcaccgtccccatggacggcgagcgcttccacctcggcgccgagcaggcggtcgcgctg600
tgcgacgagaacaccatcggcgtggtcggcgtcctcggctccaccttcgacgggtcgtac660
gagccgatcgcggagatctgcgcggccctggacgagctccaggagcgcaccggcctggac720
atcccggtgcacgtggacggcgcctccggcggcatggtggcgcccttcctcgacccggag780
ctggtctgggacttccggctgccccgggtggcctcgatcaacacctccgggcacaagtac840
ggcctggtctacccgggcgtcggctgggcgctgtggcgggaccaggagtcgctgccggag900
gagctggtcttccgggtcaactacctgggcggcgagatgccgaccttcgcgctcaacttc960
tcccggcccggcgctcaggtggcggcgcagtactacatcttcctgcggctcggccgggag1020
ggcttcaaggcggtccagggcgccagccgggatgtcgccgtctacctggccggggagatc1080
gggaagctgggctgtttccggctgctgacccacggcgaccagctcccggtgttcgcgttc1140
accaccaccgaggacgtgcccttcgacgtcttcgacgtctcgcggcggctgcgcgagcgc1200
ggctggcaggtacccgcgtacaccttccccgcgaaccgggaggacctctcagcactccgg1260
gtggtctgccgcaacggcttctcccgggacctcgccgacatgctgctggccgacctcaac1320
cggctgctgccggagctgaagcgccagcccgccccgctcaaggaactcggggtgccggtc1380
cgcgccggcttccaccactag1401
<210>3
<211>1437
<212>dna
<213>artificialsequence
<400>3
atgccactccaccaaggcgcggacagggcagccgacattcccgaggaggaccgcgccgaa60
ctgcggcggctgtcgctcaaccccttctacggggaggcggatccggtcagcgagatgacc120
tcggcgccgcccacccagcggcttcccgaagggccgaccccgccgcacaccgcctatcag180
ctcgtccacgacgagctgatgctcgacggcaactcacggctcaacctggcgaccttcgtc240
accacctggatggagccgcaggccggggtgctgatggcggagtgccaggacaagaacatg300
atcgacaaggacgagtacccgcgcaccgccgaactggagcggcgctgcgtggcgatgctc360
gccgatctgtggaacgccccggacccggcgaccgccgtcggctgctcgaccaccgggtcc420
agcgaggcgtgcatgctcgccgggctggcgctcaagcgccgctgggccaagcggaacgcg480
gcgcgctatccggcgaccgcccggcccaatctggtcatgggcgtcaacgtccaggtctgc540
tgggagaagttctgcaccttctgggaggtggaggcgcggctcgtccccatggagggcgac600
cgcttccacctcgacccgcaggccgccgccgacctctgcgacgaggacaccatcggggtc660
gtggccgtcctgggctccaccttcgacggatcgtacgaaccggtcgccgagctgtgcgcg720
gtgctggacgccctccaggagcgtacggggctcgatgtccccgtccatgtcgacggggcg780
tcgggcgccatgatcgcgcccttcctcgacgaggacctcgtctgggacttccggctgccg840
cgcgtctcctccatcaacacctcgggccacaagtacgggctggtgtacccgggcgtcggc900
tgggcgctgtggcggtcgcccgccgaactccccgaagagctggtcttccgcgtcaactac960
ctgggcggcgacatgcccaccttcgcgctgaacttctcccggccgggcgcccaggtcgtc1020
gcgcagtactacaccttcctgcggctcggcagggacggctaccgcgccgtccagcagacc1080
gcgcgggacatcgccacgaccctctccgcgcgcatcgagcggctgggcggcttccggctg1140
ctcacccgcggggaccaactgcccgtgttcgccttcaccaccgcctccgatgtgacgaac1200
ttcgatgtcttcgacgtctcgcggcggctgcgcgaacacggctggctggtgcccgcgtac1260
accttccccgcccaccgcgaggacctgtccgtcctgcgcgtggtctgccgcaacggcttc1320
tccgcggacctggcggcgatgctggtggccgacctgaagcgggtgctgcccgaactgcgc1380
gcccaggagcgccccatggaccgcgacccgtcggccgccaccgccttccaccactga1437
<210>4
<211>27
<212>dna
<213>artificialsequence
<400>4
aacatatggccttgtacaagggcaccg27
<210>5
<211>33
<212>dna
<213>artificialsequence
<400>5
aaaagcttttagtggtggaagccggcgcggacc33
<210>6
<211>28
<212>dna
<213>artificialsequence
<400>6
aacatatggctctccacaagacgaagga28
<210>7
<211>33
<212>dna
<213>artificialsequence
<400>7
aaaagcttttagtggtggaagccggagcgggga33
<210>8
<211>30
<212>dna
<213>artificialsequence
<400>8
aacatatgccactccaccaaggcgcggaca30
<210>9
<211>32
<212>dna
<213>artificialsequence
<400>9
aaagcttttagtggtggaaggcggtggcggcc32
<210>10
<211>464
<212>prt
<213>人工序列(artificialsequence)
<400>10
metalaleuhislysthrlysaspvalthrglyglyaspthrglythr
151015
aspvalphethrthralaleuserglyargvalleuprolystyrarg
202530
metproglugluhisserproserglualavaltyralaleuleuarg
354045
asngluleuleuleuaspglyasnalaalaglnasnleualathrphe
505560
cysthrthrtrpseraspaspglyvalhisargleumetasnglucys
65707580
leuasplysasnmetvalasplysaspglutyrproglnthralaglu
859095
ileglualaargcysvalasnileleualahisleutrphisalapro
100105110
alagluaspglyproalathrglycysserthrthrglyserserglu
115120125
alaalametleuglyglyleualaleulystrpargtrpargalaarg
130135140
argargalaglnglyleuproalaaspargproasnleuvalcysgly
145150155160
provalglnvalcystrpglulysphealaargtyrpheaspvalglu
165170175
leuargglnileproleuglugluaspalathrglyleuargprohis
180185190
glnleualaglutyrvalaspgluasnthrileglyvalvalalaile
195200205
leuglyvalthrtyrthrcysasptyrgluprovalalaaspileala
210215220
alaalaleuaspalaileglnglngluhisglytrpaspvalproile
225230235240
hisvalaspglyalaserglyglyleuvalalapropheleuhispro
245250255
aspvalvaltrpaspphealaleuproargvalalaserileasnthr
260265270
serglyhislystyrglyleualaproleuglyvalglytrpvalval
275280285
trpargthralaaspleuleuproprogluleuvalpheaspvalasp
290295300
tyrleuglyglyaspmetprothrphealaleuasnpheserargpro
305310315320
glyglyglualailealaglntyrtyrleupheleuargleuglyarg
325330335
serglytyrargservalhisglnsercysalaaspthralaargphe
340345350
leualaglygluilealaalametglyprophethrleuleutyrasp
355360365
glyglnglyalaleuproalavalsertyrargleuthraspproala
370375380
alaalaglyphethrleutyraspleuseraspargleuargmetarg
385390395400
glytrpglnvalproalatyrproleuproalaargargaspaspthr
405410415
valileglnargvalleuilearghisglyvalthrleuaspglnile
420425430
alaleuleualagluaspmetargargalaleuasphisleuargala
435440445
alaproprothrglnproprothralaproargserglyphehishis
450455460
<210>11
<211>466
<212>prt
<213>人工序列(artificialsequence)
<400>11
metalaleutyrlysglythrglyseraspargargleuasnvalser
151015
proleuleuvalthrasnproleualaalametgluleualapropro
202530
valhisargleuglyaspglyalavalseralaaspilealatyrgln
354045
leuvalhisaspgluleumetleuaspglyseralalysleuasnleu
505560
alathrphevalthrthrsermetgluvalglnalathrargleumet
65707580
thrglucysleuasplysasnmetileasplysaspglutyrprogln
859095
thralagluleugluargargcysvalalaileleualaaspleutrp
100105110
hisalaproaspproaspthralavalglycysserthrthrglyser
115120125
serglualacysmetleualaglymetalaleulysargargtrpmet
130135140
argargasnprogluargtyralaalaglyalaargproasnleuval
145150155160
metglyvalasnvalglnvalcystrpglulysphecysasnphetrp
165170175
gluvalglualaargthrvalprometaspglygluargphehisleu
180185190
glyalagluglnalavalalaleucysaspgluasnthrileglyval
195200205
valglyvalleuglyserthrpheaspglysertyrgluproileala
210215220
gluilecysalaalaleuaspgluleuglngluargthrglyleuasp
225230235240
ileprovalhisvalaspglyalaserglyglymetvalalaprophe
245250255
leuaspprogluleuvaltrpasppheargleuproargvalalaser
260265270
ileasnthrserglyhislystyrglyleuvaltyrproglyvalgly
275280285
trpalaleutrpargaspglngluserleuproglugluleuvalphe
290295300
argvalasntyrleuglyglyglumetprothrphealaleuasnphe
305310315320
serargproglyalaglnvalalaalaglntyrtyrilepheleuarg
325330335
leuglyarggluglyphelysalavalglnglyalaserargaspval
340345350
alavaltyrleualaglygluileglylysleuglycyspheargleu
355360365
leuthrhisglyaspglnleuprovalphealaphethrthrthrglu
370375380
aspvalpropheaspvalpheaspvalserargargleuarggluarg
385390395400
glytrpglnvalproalatyrthrpheproalaasnarggluaspleu
405410415
seralaleuargvalvalcysargasnglypheserargaspleuala
420425430
aspmetleuleualaaspleuasnargleuleuprogluleulysarg
435440445
glnproalaproleulysgluleuglyvalprovalargalaglyphe
450455460
hishis
465
<210>12
<211>478
<212>prt
<213>人工序列(artificialsequence)
<400>12
metproleuhisglnglyalaaspargalaalaaspileprogluglu
151015
aspargalagluleuargargleuserleuasnprophetyrglyglu
202530
alaaspprovalserglumetthrseralaproprothrglnargleu
354045
progluglyprothrproprohisthralatyrglnleuvalhisasp
505560
gluleumetleuaspglyasnserargleuasnleualathrpheval
65707580
thrthrtrpmetgluproglnalaglyvalleumetalaglucysgln
859095
asplysasnmetileasplysaspglutyrproargthralagluleu
100105110
gluargargcysvalalametleualaaspleutrpasnalaproasp
115120125
proalathralavalglycysserthrthrglyserserglualacys
130135140
metleualaglyleualaleulysargargtrpalalysargasnala
145150155160
alaargtyrproalathralaargproasnleuvalmetglyvalasn
165170175
valglnvalcystrpglulysphecysthrphetrpgluvalgluala
180185190
argleuvalprometgluglyaspargphehisleuaspproglnala
195200205
alaalaaspleucysaspgluaspthrileglyvalvalalavalleu
210215220
glyserthrpheaspglysertyrgluprovalalagluleucysala
225230235240
valleuaspalaleuglngluargthrglyleuaspvalprovalhis
245250255
valaspglyalaserglyalametilealapropheleuaspgluasp
260265270
leuvaltrpasppheargleuproargvalserserileasnthrser
275280285
glyhislystyrglyleuvaltyrproglyvalglytrpalaleutrp
290295300
argserproalagluleuproglugluleuvalpheargvalasntyr
305310315320
leuglyglyaspmetprothrphealaleuasnpheserargprogly
325330335
alaglnvalvalalaglntyrtyrthrpheleuargleuglyargasp
340345350
glytyrargalavalglnglnthralaargaspilealathrthrleu
355360365
seralaargilegluargleuglyglypheargleuleuthrarggly
370375380
aspglnleuprovalphealaphethrthralaseraspvalthrasn
385390395400
pheaspvalpheaspvalserargargleuarggluhisglytrpleu
405410415
valproalatyrthrpheproalahisarggluaspleuservalleu
420425430
argvalvalcysargasnglypheseralaaspleualaalametleu
435440445
valalaaspleulysargvalleuprogluleuargalaglngluarg
450455460
prometaspargaspproseralaalathralaphehishis
465470475
- 还没有人留言评论。精彩留言会获得点赞!