隐性代谢产物和用于激活多种微生物中沉默的生物合成基因簇的方法与流程
相关申请的交叉引用本申请要求2017年7月31日提交的美国临时申请no.62/539,263的优先权,其全部内容通过引用合并于此。对以电子方式提交的序列表的引用以电子方式提交的序列表也通过引用将其全部内容并入本文(文件名:prin-57276_st25.txt;创建日期:2018年7月13日;文件大小:524字节)。本文总体上涉及隐性代谢产物,更具体地,涉及一组隐性代谢产物,以及用于激活多种微生物中沉默的生物合成基因簇的无遗传学方法。
背景技术:
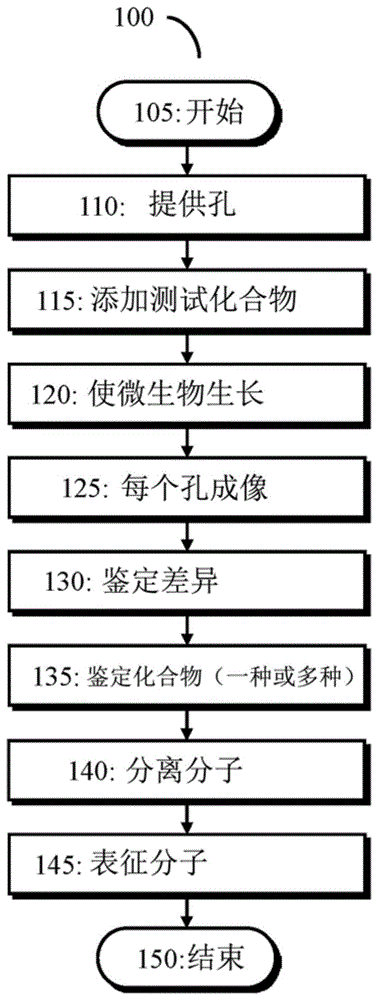
:没有天然产物,现代医学是不可想象的。这些分子主要从微生物和植物中分离出来,也被称为次级代谢产物,形成了>70%的抗生素、>50%的抗癌剂的基础,并且总体上在过去35年中超过一半的药物在美国被批准。在开采了近一个世纪的次级代谢产物之后,微生物似乎已成为一种枯竭的资源。但是,最近微生物基因组序列的爆发指出了新代谢产物的大量、尚未开发的宝藏。具体而言,几个细菌门的成员通常隐藏25种或更多种的生物合成基因簇(bgc)——指导天然产物的生物合成的基因的集合——其在标准实验室条件下并不活跃表达或仅弱表达。这些所谓的“沉默”或“隐性”bgc数量比组成型活跃的bgc数量高5-10倍。因此,寻找获取其产物的新方法可以显著提高我们的新天然产物的全部功能,从而加快并帮助药物发现。诱导沉默的bgc的重要性已被研究界所认识,并且已经开发出几种方法来鉴定和表征其小分子产物,包括bgc在异源宿主中的表达、共培养筛选、核糖体工程、组成型或诱导型启动子的插入、报道分子引导的突变体选择以及调控蛋白的内源性过表达。尽管这些方法已经共同地开始阐明细菌的隐蔽次级代谢组,但是它们都需要挑战性的培养、分子生物学和/或遗传程序,这显著减慢了天然产物发现的进度和通量。获取各种微生物中隐性代谢产物的确定方法尚待开发。因此,期望一种廉价、可靠的系统,用于准确地检测给定位置中的占有者数量。技术实现要素:本公开还涉及用于快速激发隐性代谢产物的方法。该方法需要提供多个适于使微生物生长的孔(例如96个孔),其中每个孔包含至少一种化合物。有利地,每种化合物是小分子库的子集。在每个孔中使微生物生长之后,用质谱法对孔进行成像,并鉴定成像孔和成像对照孔之间的差异。还鉴定激发那种差异的化合物(一种或多种)。有利地,质谱法是激光烧蚀电喷雾电离质谱法(laesi-ms)。同样有利地,该方法还包括分离分子,其有时可以涉及提供包含第二用量的培养基(比上述孔中使用的更大)和来自第一物种的至少一种微生物。然后,分离工艺可以涉及将鉴定的化合物(一种或多种)添加到较大的培养物中,使微生物生长,并通过例如离心去除细胞。该分离工艺还可包括通过例如hplc纯化。该分子也可以被表征,其可以包括利用例如hplc-esi-ms或nmr。通常对微生物没有限制,但有利地微生物是革兰氏阴性细菌、革兰氏阳性细菌或真菌,如拟无枝菌酸菌属(amycolatopsissp.)b24117。在某些情况下,微生物的基因组序列可能未知。本公开涉及隐性代谢产物的集合或其药学上可接受的盐。本公开还涉及利用隐性代谢产物的方法,通过使微生物与隐性代谢产物之一接触来抑制生长。有利地,微生物可以是革兰氏阳性细菌,如金黄色葡萄球菌(staphylococcusaureus)、无乳链球菌(streptococcusagalactiae)、化脓链球菌(streptococcuspyogenes)、肺炎链球菌(streptococcuspneumoniae)、艰难梭菌(clostridiumdifficile)、粪肠球菌(enterococcusfaecalis)或枯草杆菌(bacillussubtilis)。附图说明图1是描述公开的方法的实施方式的流程图。图2a是根据m/z和ms强度将防御假单胞菌(p.protegens)代谢组与每个激发子相关的3d图。图2b是orfamidea和b的结构的图解。图2c是来自未经处理的和caf处理的培养物的orfamidea的hr-ms提取离子色谱图的图,为清楚起见,微量物质被偏移了。图3是orfamidea-b、canucina-b、角蛋白(keratinimicin)a-d和角蛋白环素(keratinicyclin)a-c的hr-ms数据的表。图4a是根据m/z和ms强度将暗灰链霉菌(s.canus)的次级代谢组与502个激发子相关的3d图。图4b是3d图的2d组分,着重于canucina(m/z1579)合成,强调了肯帕罗酮(kenpaullone)是最有效的激发子。图4c描绘了烧瓶培养物中通过ken的canucina(左)和b(右)的hr-ms提取离子色谱图的图,为清楚起见,微量物质被偏移了。图4d是canucina拓扑学的图解,其中h12和f13提供空间锁。图4e是使用nmrnoesy约束和cyana算法叠加canucina的前10个计算结构的图解,显示了套索拓扑学。图4f是使用nmrnoesy约束和cyana算法叠加canucinb的前10个计算结构的图解,显示了套索拓扑学。图4g是通过生物信息学研究鉴定的针对canucin(can)的bgc的描绘。示出了cana的c末端序列以及剪裁酶的预测功能。图5a是cd3oh中canucina的nmr赋值表。图5b示出了针对canucina的编号方案的图解。图5c是总结来自canucina中asp14的cβ处的r和s立体异构体的计算的cyana衍生参数的表。图6a-6e是cd3oh中canucina的nmr光谱,包括1hnmr(图6a)、tocsy(图6b)、noesy(图6c)、hsqc(图6d)和hmbc(图6e)。图7a是cd3oh中canucinb的nmr赋值表。图7b示出了针对canucinb的编号方案的图解。图8是注释canucin生物合成基因簇(can)的表。图9a是根据m/z和ms强度,将keratiniphila拟无枝菌酸菌(a.keratiniphila)的次级代谢组与502个激发子相关的3d图。图9b是3d图中的2d组分,着重于角蛋白a(m/z1811)合成,强调了双氢麦角汀(dhe)和长春胺(vin)是最有效的激发子。图9c描绘了烧瓶培养物中通过dhe和vin的角蛋白a(左)和角蛋白环素b(右)的hr-ms提取离子色谱图的图,为清楚起见,微量物质被偏移了。图9d描绘了用于解析角蛋白a和角蛋白环素a的结构的有关的nmr相关性。图9e描绘了具有所示不同取代模式的四种角蛋白衍生物的结构;示出了鉴定糖肽中不同环的命名法。图9f描绘了具有所示不同取代模式的三种角蛋白环素衍生物的结构。图9g描绘了在对keratiniphila拟无枝菌酸菌的基因组测序之后通过生物信息学分析鉴定的kerbgc。图9h是注释角蛋白生物合成基因簇(ker)的表。图10a是3d图的2d组分,着重于来自keratiniphila拟无枝菌酸菌(m/z1286)的隐性代谢产物,强调有效的激发子。图10b是3d图的2d组分,着重于来自keratiniphila拟无枝菌酸菌的隐性代谢产物(m/z1425),强调有效的激发子。图11a是dmso-d6中角蛋白a的nmr赋值表。图11b是示出了角蛋白a的编号方案的图解。图12a-12e是dmso-d6中角蛋白a的nmr光谱,包括1hnmr(图12a)、gcosy(图12b)、roesy(图12c)、hsqc(图12d)和hmbc(图12e)。图13a是dmso-d6中角蛋白b的nmr赋值表。图13b是示出角蛋白b的编号方案的图解。图14a是dmso-d6中角蛋白c的nmr赋值表。图14b是示出角蛋白c的编号方案的图解。图15a是dmso-d6中角蛋白d的nmr赋值表。图15b是示出角蛋白d的编号方案的图解。角蛋白环素a图16a是dmso-d6中角蛋白环素a的nmr赋值表。图16b是示出角蛋白环素a的编号方案的图解。图17a是dmso-d6中角蛋白环素b的nmr赋值表。图17b是示出角蛋白环素b的编号方案的图解。图18a是dmso-d6中角蛋白环素c的nmr赋值表。图18b是示出角蛋白环素c的编号方案的图解。图19是示出角蛋白和角蛋白环素针对选择的致病菌和病毒的mic和ic50值(以μm计)的表。图20是使用laesi-ms检测的来自高通量激发子筛选的3d图的2d组分的一种实施方式。图21a-21f是公开的隐性代谢产物的分子结构。具体实施方式除非以上另外定义,否则本文中使用的所有技术和科学术语具有与本发明所属领域的普通技术人员通常所理解的相同含义。在以单数形式提供术语的情况下,发明人还考虑了该术语的复数形式。除非上下文另外明确指出,否则单数形式“一个/种(a)”、“一个/种(an)”和“该(the)”包括复数形式。术语“包含(comprise)”和“包含(comprising)”以包括性、开放性的含义使用,意味着可以包括额外元素。公开了一种用于快速激发隐性代谢产物的廉价且非劳动密集的方法。沉默的bgc是新的次级代谢产物的宝库。dna技术的进步和广泛的基因组测序已编辑了一个庞大的基因组数据库,需要挖掘该数据库以收获数十年来的创新成果。这样做的理想方法是不必挑战典型大的bgc的遗传或克隆程序。此外,该方法应以高通量的方式优选在内源宿主中激活沉默的bgc,以避免与异源表达相关的困难。最后,单一培养方法是优选的,以消除有时会困扰混合培养或共培养筛选的不可重复相互作用的可能性。本文公开了一种满足所有这些标准的方法。可以通过例如,将它用于测序和未测序的细菌而看到它的效用。例如,它可以应用于稀有放线菌,其成员难以进行遗传操作。该方法的典型输出是响应~500个条件的给定细菌的总体次级代谢组的图片,其以高通量方式证明了沉默bgc的激活。利用这些优点,本文公开了具有前所未有的翻译后修饰的套索肽,具有与万古霉素相似或更好的生物活性的新的糖肽抗生素,以及新型糖肽化学型,其结合了gpas的特征与恶唑烷酮抗生素的特征并展现了比目前使用的针对rsw的药物利巴韦林更好的抗病毒活性。与初级代谢相反,任何给定的多产细菌的完整次级代谢组尚不清楚。基于maldi-tof和desi的ims在评估细菌培养物或物种间相互作用中是开创性的,但ims在探寻响应数百种条件的隐性代谢组中的应用却尚未被报道。因此,除了发掘出新的隐性代谢产物外,本文还公开了一种高通量激发子筛选(hites)-ims方法,该方法可用于描绘总体次级代谢组,即,选定细菌物种的全部合成能力。这种方法的优势在于,bgc的最终产物,即次级代谢产物,提供了读出而不是转录或翻译测定法。另外,通过将新的隐性代谢产物与激发子连接,可以研究激发的机制,从而可以鉴定响应毒素的亚抑制浓度而启动次级代谢的调节回路。公开的方法是迄今为止激活沉默bgc的最通用的,它有望同时揭示多种细菌中的隐性代谢的产物和调节。参考图1,快速激发隐性代谢产物(100)的方法通常开始于将微生物和生长培养基引入一系列孔(110)中。一般而言,孔的阵列是优选的。在某些实施方式中,利用48孔、96孔、384孔或1536孔的细胞培养板。在某些实施方式中,利用了超过1500个孔的阵列。更优选的实施方式利用50至500个孔。每个孔必须包含微生物,并且通常地,适于该微生物生长的培养基。尽管可以利用任何微生物,但是在优选的实施方式中,微生物是细菌或真菌的物种。在优选的实施方式中,微生物是革兰氏阴性细菌、革兰氏阳性细菌或真菌。注意在某些实施方式中,微生物的基因组序列是未知的。实际上,利用所公开的方法不需要遗传程序。此外,该方法不限于建立了遗传程序的微生物。某些实施方式使用不能被遗传操作的微生物,其可以包括稀有放线菌、尚不能进行遗传操作的新菌株以及细菌混合物或聚生体。可以使用哪些培养基没有限制;任何期望的培养基均可在孔中使用。通常,将至少一个孔用作对照,将其余的用作测试孔。在每个测试孔中,将添加一种或多种额外的化合物。在一些实施方式中,添加到测试孔中的每种化合物都是小分子库的一部分。再次参考图1,一旦提供了具有培养基和微生物的孔,就添加测试化合物(115)。在这些方法中,通常一个孔被视为“对照”孔,并且不包含任何测试化合物。其他孔除微生物和培养基外,还可包含一种或多种测试化合物。可以利用哪些测试化合物没有限制。在一些实施方式中,测试化合物可以包括抗生素或其他已知抑制微生物生长的化合物。在优选的实施方式中,至少一种化合物中的每种都是小分子库的子集。在某些实施方式中,一部分孔利用两种或更多种测试化合物。然后使微生物在每个孔中生长(120)。生长的确切条件将基于本领域技术人员已知的多种因素。在一个实例中,将防御假单胞菌(p.protegens)从冷冻培养物原液划线到lb-琼脂板上,并在30℃下生长过夜。菌落用于在14ml无菌培养管中接种5ml的lb,将其在30℃/250rpm下培养12-13h。然后过夜培养物用于接种在无菌锥形瓶中的550mllb至在600nm的初始光密度(od600nm)为0.01。使用multiflo微孔板分配器(biotek)将培养物分配到六个无菌的深孔96孔板(每孔0.9ml)中。随后,使用cybi-well自动液体转移机器人(cybio)从市售的502个成员的天然产物库(enzoscientific,目录号bml-2865)添加激发子。每个孔接收2.5μl的激发子(来自10mm的原液浓度)。用透气膜密封板,并在25℃/200rpm下生长。44h后,将板稍微离心,将上清液上样到96孔stratac8树脂(phenomenex)上,将物质用600μl50%和600μl100%的mecn洗脱到新的96孔板中。然后将洗脱液在speedvac(真空离心浓缩仪)中干燥,并重悬于30μl40%的mecn(水中)。在其他实例中,将新鲜收集的暗灰链霉菌(s.canus)或keratiniphila拟无枝菌酸菌(~107)转移至配有不锈钢弹簧的250ml锥形瓶中的50mlyeme培养基(3%(w/v)酵母提取物、5%蛋白胨、3%麦芽提取物、1%葡萄糖、34%蔗糖和5mmmgcl26h2o),并在30℃/250rpm下培养3天。然后通过离心(10min,3000g,rt)收集菌丝体,并稀释到300ml的r4培养基中,使其终浓度为0.05%(w/v)。r4培养基1由0.5%(w/v)葡萄糖、0.1%酵母提取物、0.5%mgcl2·6h2o、0.2%cacl22h2o、0.15%脯氨酸、0.118%缬氨酸、0.28%tes、50mg/l酪蛋白氨基酸、100mg/lk2so4和1×痕量元素溶液,其含有40mg/lzncl2、200mg/lfecl3·6h2o、10mg/lcucl22h2o、10mg/lmncl24h2o、10mg/lna2b4o7·10h2o和10mg/l(νη4)6μο7o24·4η2o)。随后,使用multiflo微孔板分配器将300μl分配到六个深孔96孔板中。借助cybi-well自动液体转移机器人,在孔中补充了来自上面使用的相同的502个成员天然产物库的候选激发子。每个孔接收0.85μl的激发子(来自10mm的原液浓度)。将板用透气膜密封并在30℃/250rpm下生长。5天后,如上文针对防御假单胞菌所述将样品脱盐。一旦微生物在每个孔中生长,就使用至少一种成像质谱法技术快速测定响应来自库的每种分子的代谢组。在某些实施方式中,质谱法技术是激光烧蚀电喷雾电离质谱法(laesi-ms)。对每个孔成像(125),并且鉴定该图像与来自对照孔的图像之间的差异(130)。然后鉴定与一种或多种差异相关的测试化合物(135)。在某些实施方式中,这三个步骤中的每一个针对整个阵列孔发生,然后进行下一步。即,拍摄所有图像,然后测定所有孔的差异,然后鉴定激发这些差异的任何测试化合物。在其他实施方式中,拍摄第一孔的图像,将其与对照孔的图像进行比较,如果存在差异,则鉴定与该孔相关的化合物。然后拍摄第二孔的图像,重复该工艺,直到所有孔都已完成。取决于系统的确切需求和配置,其他方法也可能是有利的。通常按照制造商的说明对孔进行成像。为了鉴定差异,通常使用软件来分析收集的图像,查找由激发子诱导且未处理样品中不存在的质量离子。在一个实例中,与ltqxl质谱仪(thermo)偶联的激光烧蚀电喷雾电离(laesi)dp1000系统(proteabioscience)被用于ims分析。连接两个仪器的延长管用外部加热器保持在130℃,分析过程中样品台保持在10℃。鞘气流量设定为2.0l/h。使用80%的激光能量设置(~850μj)和10hz的频率将八十个激光脉冲施加到每个孔,以烧蚀样品。将2:1的mecn/水与0.1%乙酸(v/v)溶液通过以1μl/min的流速运行的注射泵供给,作为电喷雾溶液。在正离子检测模式下,将发射器连接到在+4000v或+4500v下运行的高压电源上。所有数据均在proteaplot软件中可视化。一旦知道差异,就可以典型地通过使用计算机或专用处理器将描述该孔或添加到该孔中的化合物的一些鉴定信息与数据库或查找表中的数据进行比较,来鉴定测试化合物(一种或多种)。这可以手动或自动完成。优选地,仅鉴定最佳少数激发子,通常不超过前4-5个激发子。在一个实例中,数据收集后,使用gmsu-laesi软件(gubbs,inc)提取各孔中观察到的信号,该软件给出了所有的m/z值和每孔(即,每个激发子)的对应强度。数据以1m/z单位进行分箱以进行3d绘图。强度低于设定阈值的信号未包括在3d图中。使用bar3功能在matlab中绘制数据。从matlab中相应的3d数据中提取组分2d图,并在excel中绘制。将2d图归一化为最高强度峰。每个信号都与激发子相关。在另一个实例中,为了开始筛选,拟无枝菌酸菌属(amycolaptopsissp.)b-24117孢子原液用于在500ml锥形瓶中接种100ml的r4培养基(约106个孢子)。将它们生长过夜,然后使用biotekmultiflotm微孔板分配器分配到5×9596孔细胞培养板中。将150μl分配到每个孔中,然后从naturalproducts小分子库(enzoscientific)向每个孔中补充1μl。将板在30℃和250rpm下再次生长4天,然后使用激光烧蚀电喷雾质谱仪(laesi-ms)进行成像,该仪由与thermoscientifictmltq质谱仪偶联的proteabiosciencesdp-1000模块组成。按照制造商的说明使用1:1mecn/水以1μl/min的流速进行每个板的成像,以将样品烧蚀到质谱仪中。每孔收集光谱8秒。然后使用proteaplot软件分析数据,查找由激发子诱导且未处理样品中不存在的质量离子。参考图20,这导致了质量离子的鉴定(参考230,m/s1810,角蛋白a)。最佳激发子被测定为色胺酮(231)和双氢麦角汀(232),每个的浓度均为30μμ。在一些实施方式中,激发子的鉴定是足够的。然而,在其他实施方式中,也可以分离被激发的分子(140)。为了分离分子(140),一种方法是利用较大的培养物,其包含比孔中使用的用量更大的培养基的用量,和靶标微生物。然后将至少一种鉴定的激发子添加到该较大的培养物中。然后使微生物在激发子的精确范围内生长,然后将细胞和分子分开。通常,通过离心去除细胞,但根据系统的确切需求,其他方法也可以接受。可以使用通常的纯化技术,包括但不限于使用hplc,来进一步纯化产出。该分子也可以被表征(145)。在一些实施方式中,使用hplc-esi-ms或nmr进行表征,尽管也可以利用其他方法。用成像质谱法代替遗传学。hites方法由两个部分组成,由激发子筛选激活沉默或低表达的bgc和对该工艺的读出,其迄今为止都依靠遗传报道分子测定法。检测步骤限制了hites的通量,因为取决于菌株,创建合适的遗传构建体通常很费时,如果不是不可能的话。为了避免该缺点,工作流可以由以下组成:使用ims对wt微生物进行激发子筛选,然后对所得的例如500-1000个代谢组进行成像,作为库中每个分子的功能。例如,在hites-ims工作流的一种实施方式中,将细菌培养物部署到96孔板中,并进行高通量激发子筛选。在合适的孵育期之后,通过laesi-ims以96孔形式评估培养物。在3d图中描绘了观察到的总体代谢组,该图将每个激发子与代谢产物联系起来,以其m/z和ms强度值为特征。具有激发子的大规模培养物有助于新的隐性代谢产物的分离和表征。然后可以使用计算方法和适当的可视化来准确定位隐性代谢产物。作为另一个实例,orfamide是从防御假单胞菌(pseudomonasprotegens)pf-5(以下称为防御假单胞菌(p.protegens))分离的低表达代谢产物。以前使用基因组同位素方法来鉴定并最终解析其结构。将wt假单胞菌在96孔板中培养,并使用502个成员的天然产物库进行hites。使用激光烧蚀耦合电喷雾电ms(laesi-ms)分析所得的代谢组,laesi-ms是一种新兴方法,其中中ir激光束产生被电喷雾电离并引入质谱仪的代谢产物的烧蚀羽流。与其他ims方法相比,laesi-ms的优势在于它可以在环境压力下只需最少的样品制备即可应用于液体或固体表面以及活细菌培养物。众多参数的优化促进了502个孔中每个孔内代谢组的快速表征,允许我们在不到一个小时的时间内对96孔板液体培养物进行成像。数据在描绘由给定激发子诱导的每种代谢产物的强度和m/z的3d图中给出(见图2a)。在该实例中,响应502个成员天然产物库,收集了m/z范围为1200-2000的ms数据以聚焦orfamide的产生。在m/z1250以下和m/z1650以上均未检测到信号。ms强度被颜色编码,并且鉴定5-计数阈值以下的信号,因此其未包括在图中。标记了orfamide,为orfamide合成的最佳激发子即咖啡醇(caf)和长春瑞滨(vrl)。图2b表示orfamidea和b的结构。通过计算地检查合并的502个代谢组,本领域技术人员可以容易地看到在不同程度上诱导orfamide——特别是类似物a、b和几种未知的衍生物。轻度的细胞毒素咖啡醇和抗癌剂长春瑞滨——以前未知会激发次级代谢的化合物——触发最佳生产。咖啡醇的刺激活性进一步用hplc-ms确认,从而验证了hites-ims在诱导沉默或低表达的bgc中的应用(见图2c、图3)。图2c是验证caf作为烧瓶培养物中orfamidea的诱导剂的图。图2示出了来自未经处理的(201)和caf处理的(202)培养物的orfamidea的hr-ms提取离子色谱图。图3是示出orfamidea-b、canucina-b、角蛋白a-d和角蛋白环素a-c的hr-ms数据的表。这种方法的简单性允许它被广泛的应用。hites-ims应用于链霉菌(streptomycetes)。该hites-ims方法也适用于链霉菌属(streptomycesspp.),已知的细菌次级代谢产物生产者的最多产的属。暗灰链霉菌(streptomycescanus)nrrlb3980被选择,其与安福霉素生产者有关,并包含超过20个还没有与天然产物联系起来的bgc。再次使用502个成员天然产物库的用暗灰链霉菌的hites-ims结果在3d表示中示出。(见图4a)使用1000-2000的m/z范围来简化ms输出,并同时优化找到新的代谢产物的机会。似乎激发了代表三个化合物家族的三个峰簇:检测到化合物,m/z范围为1260-1295,其进一步被分析鉴定为安福霉素。安福霉素的最佳激发子是黄酮醇桑色素和抗疟疾奎宁。还检测到第二集合诱导的化合物,m/z范围为1220-1245。最后,观察到与其他两个不同的第三化合物家族的诱导,m/z为1563和1579,由细胞周期蛋白依赖性激酶抑制剂肯帕罗酮(kenpaullone)激发(见图4a、4b)。图4b是3d图的2d组分,着重于canucina(m/z1579)合成。在该实例中,肯帕罗酮是最有效的激发子。这些发现突出了所公开的方法以并行方式诱导多个隐性bgc的能力。当前的方法提供了监视一个实验中可以通过检测方法捕获的所有bgc的能力。从在该实例中激发的隐性代谢产物,进一步的努力集中在具有m/z1579和1563的化合物上,这些化合物分别被赋予了通俗名称canucina和b(见图3)。通过观察96孔板并发现肯帕罗酮对两种化合物的强诱导作用来验证结果(图4c)。在图4c中,示出了烧瓶培养物中ken对canucina和b的诱导,突出了未处理的(203,205)和ken处理的(204,206)培养物。为清楚起见,微量物质在x轴和y轴上被偏移了。以肯帕罗酮为诱导剂的大规模生产培养物允许我们分离足够的物质以通过1d/2dnmr解析canucin的结构。1h、gcosy和tocsynmr分析表明,canucina是具有14种可识别的α-1hs的肽。hsqc和hmbc分析显示其中13种为规范的氨基酸,而一种为被修饰的β-羟基-asp(见图5a-5b)。图5a示出了cd3oh中canucina的nmr赋值。canucina的编号方案示出在图5b中。通过nmr和hr-ms的进一步分析表明,canucina在残基glyl和asp8之间隐藏异肽键,即套索肽典型的特征。确实,c末端尾部残基与围绕异肽键的那些残基之间的noesy相关性表明,canucina包含套索拓扑学(见图6a-6e)。图6a-6e提供了cd3oh中canucina的nmr光谱,包括1hnmr(图6a)、tocsy(图6b)、noesy(图6c)、hsqc(图6d)和hmbc(图6e)。为了验证,收集了不同的混合时间的高分辨率noesy光谱,并使用cyana算法解析了canucina的3d结构,该算法利用肽的扭转角空间中的分子动力学模拟来计算与noesy数据最吻合的结构。十个最佳结构在套索拓扑学上收敛,其中his12和phe13分别在环的上方和下方提供空间锁(图4d、4e)。图4d提供了canucina的拓扑学的图解,其中h12和f13提供了空间锁。图4e示出了使用nmrnoesy约束和cyana算法的canucina和b的前10个计算结构的叠加。两者都显示套索拓扑学。在14个氨基酸处,canucina是迄今为止发现的最小的套索肽之一。见图5b。重复尝试通过mosher分析确定c末端asp的β碳的立体化学的努力失败了,这可能是由于空间位阻所致。随后,cyana用于计算哪种立体异构体最适合观察到的noesy相关性。s-立体异构体给出较低的f函数,表明所计算的结构与约束集合之间具有更好的匹配,以及较低的主链rmsd(见图5c)。图5c是根据canucina中asp14的cβ处的r和s立体异构体的计算得出的cyana衍生参数的总结。建议了在asp的β-碳处的s-立体构型。第二类似物——canucinb——也被鉴定。通过hr-ms和nmr的结构阐明将其鉴定为canucina的去羟基变体(参见图3、7a、7b)。图7a提供了cd3oh中canucinb的nmr赋值,而图7b显示了canucinb的编号方案。cyana的计算证实了canucinb隐藏套索拓扑学,再次揭示了his12和phe13为类似于canucina的拓扑学中的空间锁(见图4f)。这些结果表明,在建立带螺纹的套索基序后,canucina中c末端asp发生了羟基化反应。翻译后修饰的套索肽是罕见的,并且在该化合物家族中从未观察到β-羟基化的氨基酸。为了深入了解canucina的生物合成,检查了暗灰链霉菌的基因组序列。bgc被鉴定,将其注释为can,具有典型的套索肽同线性和前体肽,其c-末端序列(gvgfpladffihfd(seqidno.001))与canucin的完全匹配(图4g)。除了前体肽,canbgc还包含典型的蛋白酶和asn合成酶,其分别去除前体肽并形成带螺纹的套索基序。另外,对cane进行了注释,编码一种依赖α-kg依赖的单核fe酶,该酶的成员已显示出羟基化未活化的碳位置。(见图8)。图8提供了canucin生物合成基因簇(can)的注释。cane可能参与了β-oh-asp的合成。canucin的发现表明,hites-ims可用于链霉菌以揭示新的隐性代谢产物。hites-ims应用于稀有放线菌。所公开的方法也被应用于稀有放线菌——一组细菌,其解释为在结构上有些令人着迷且功能上有效的代谢产物,包括抗生素杀手锏万古霉素以及抗癌剂加利车霉素。稀有放线菌是hites-ims的理想测试例,因为除了其丰富的沉默bgc外,它们还难以进行遗传操作,因此几乎排除转录报告基因测定法。keratiniphila拟无枝菌酸菌(amycolatopsiskeratiniphila)nrrlb24117被选择作为测试例。它的基因组尚未测序,但是使用pcr寻找编码糖肽类抗生素(gpas)的其他bgc的努力表明,它含有类万古霉素bgc,尽管产物尚未被报道。keratiniphila拟无枝菌酸菌(a.keratiniphila)进行hites-ims并得到所示的代谢组,着重于典型的gpas的质量范围(图9a)。同样,沉默bgc的诱导以高通量方式发生,因为许多代谢产物被观察到只有当被来自小分子库的化合物激发时才被诱导(图9a、10a、10b):高良姜素(galangin)和含吲哚的生物碱吴茱萸碱和西萝芙木碱诱导m/z为1286的化合物(图10a)。另外,m/z为1425的化合物由包括高良姜素和吴茱萸碱在内的类似激发子诱导(图10b)。最后,在诱导的代谢组中观察到了具有m/z1654和1811的化合物,主要是通过双氢麦角汀(dhe)和长春胺(vin,图9a、9b)诱导。keratiniphila拟无枝菌酸菌似乎通过激活众多、或者沉默的、导致隐性代谢产物产生的bgc,对不同来源的含吲哚的生物碱进行响应。在检测到的化合物中,一种化合物似乎包含ci原子,如由hr-ms同位素分布图所判断的,这是gpas中的常见修饰(见图3)。后续实验验证了dhe和vin的强刺激活性,表明该化合物的明显的激发(图9c)。图9c示出了表明在不受控制的(207、210),vin处理的(208、211)和dhe处理的(209、212)的培养物中角蛋白a(左)和角蛋白环素b(右)的激发的图。由于怀疑可能是推定的糖肽,因此在dhe存在下从大规模生产培养物中分离出七种类似物。hr-ms分析表明,这些化合物分为两个家族,一个被称为角蛋白,第二个被称为角蛋白环素。1d/2dnmr分析表明角蛋白a包含六个高度修饰的氨基酸,可识别的α-1hs在4.41、4.52、4.45、5.62、4.48、4.23和4.42ppm(图9d(左)、11a、11b)。图9d图解了用于解析角蛋白a(左)和角蛋白环素a(右)的结构的相关nmr相关性。图11a提供了dmso-d6中角蛋白a的nmr赋值。图11b提供了角蛋白a的编号方案。tocsy、hsqc和hmbc分析鉴定这些为β-oh-tyr、phe(两个交联的hpg残基)、糖基化的β-oh-3-cl-tyr和交联的3,5-二羟苯甘氨酸(dpg)(见图12a-12e)。图12a-12e提供了dmso-d6中角蛋白a的nmr光谱。具体而言,1hnmr(图12a)、gcosy(图12b)、roesy(图12c)、hsqc(图12d)和hmbc(图12e)。特征性地,如通过hmbc和noesy光谱所阐明的,交联通过碳-碳键发生在a-b环之间,以及通过芳基醚键发生在c-d和d-e环之间(图9e)。在图9e中,指示了具有不同取代模式的四种角蛋白衍生物的结构。示出了鉴定糖肽中不同环的命名法。通过nmr分析鉴定了四个糖基基团,分别在残基a、c和d处的甘露糖、肌动胺(actinosamine)和葡萄糖-鼠李糖二糖,从而完成了角蛋白a的二维结构。为了指定手性中心,选择了组合的光谱和生物信息学方法。keratiniphila拟无枝菌酸菌的整个基因组进行鸟枪法测序允许我们使用先前描述的规范同线性来准确定位gpa簇,该簇被注释为ker。图9g提供了如在对keratiniphila拟无枝菌酸菌的基因组测序之后通过生物信息学分析所鉴定的kerbgc。每个nrps的预测域组成显示为bgc中其余酶的预测功能。tyr*表示修饰的tyr。看来ker是报道的ii类gpa的第一个bgc。生物信息学分析揭示了与原型i类gpas相同的域组织。图9h提供了角蛋白生物合成基因簇(ker)的注释。提出了如所示的d-氨基酸和l-氨基酸的模式(从c末端到n末端:l-l-d-d-l-d-d,图9e)。最后,通过nmrnoesy相关性将β-oh基团的立体构型指定为r,从而完成所提出的三维结构。角蛋白a类似于放线菌素a,除了它包含在g环的氯化物,并且在残基d处携带不同的糖。三种另外的角蛋白被鉴定,并在结构上进行了阐明。相对于变体a,角蛋白b仅包含三个糖基部分:分别在残基c和d处的肌动胺和葡萄糖-acosamine二糖,而在残基a处的甘露糖缺失(见图13a和13b)。图13a提供了dmso-d6中角蛋白b的nmr赋值。图13b提供了角蛋白b编号方案。角蛋白c的n末端由不寻常的2-酮-间-氯-邻-羟基苯乙酸部分封端,并有不同束的糖取代基(见图14a和14b)。图14a提供了dmso-d6中角蛋白c的nmr赋值。图14b提供了角蛋白c的编号方案。角蛋白d也含有2-酮-间-氯-邻-羟基苯乙酸残基,与角蛋白c相比,具有在残基d处不同的糖组合(见图15a和15b)。图15a提供了dmso-d6中角蛋白d的nmr赋值。图15b提供了角蛋白d的编号方案。角蛋白环素a的结构通过导致所示的结构(图9d(右),9f)的光谱数据的广泛分析来表征。图9f提供了具有不同取代模式的三种角蛋白环素衍生物的结构;鉴定糖肽中不同环的命名法在图9e中示出。它由6mer肽主链组成,该肽主链含有与角蛋白相同的序列,但没有n末端3-c1-hpg残基。它带有与角蛋白相同的芳族交联,具有a-b联芳基键以及c-o-d和d-o-e芳基醚交联。还通过如上所述的nmr分析鉴定了糖基基团(见图16a、16b、9e、9f)。图16a提供了dmso-d6中角蛋白环素a的nmr赋值,而图16b提供了角蛋白环素a的编号方案。最值得注意的是,角蛋白环素含有n末端2-恶唑烷酮,其是临床使用的抗生素利奈唑胺和泰地唑利中存在的官能团。因此,角蛋白环素将gpas的特有特征与恶唑烷酮抗生素的那些特征结合在一起,是迄今为止报道的首次此类结合。在gpas内,角蛋白环素代表一种新的化学型。解析了2种另外的角蛋白环素的结构。相对于衍生物a,角蛋白环素b含有在残基d处的葡萄糖-acosamine二糖,而不是葡萄糖-鼠李糖(见图17a和17b)。图17a提供了dmso-d6中角蛋白环素b的nmr赋值。图17b提供了角蛋白环素b的编号方案。角蛋白环素c也含有在残基d处的葡萄糖-acosamine二糖,但缺乏残基a处的甘露糖(见图18a和18b)。图18a提供了dmso-d6中角蛋白环素c的nmr赋值。图18b提供了角蛋白环素c的编号方案。最初的内部测定法显示对角蛋白的强的抗微生物活性,但对角蛋白环素则没有。角蛋白a和c已提交用于针对细菌病原体的广泛生物活性测试。由于一些gpas已被证明隐藏抗病毒特性,所以针对一组病原性人类病毒评估了角蛋白环素b和c。角蛋白显示对多种革兰氏阳性病原体的有效的抗菌活性,mic(最小抑制浓度)类似于万古霉素对链球菌(streptococci)、艰难梭菌(clostridiumdifficile)和粪肠球菌(enterococcusfaecalis)的那些mic(见图19)。图19示出了角蛋白和角蛋白环素针对选择的致病菌和病毒的mic和ic50值(以μm计)。显著的生物活性(mic或ic50<10μm)以粗体显示。它们对耐万古霉素的肠球菌(enterococci)无效,表明与万古霉素相似的作用方式。角蛋白环素没有表现出显著的抗菌活性,但却是针对呼吸道合胞病毒(rsv)的有效抑制剂。实际上,针对rsv测得的半数最大抑制浓度(ic50)比目前使用的药物利巴韦林的效力多约10倍。因此,角蛋白和角蛋白环素可用于各种治疗选择,使微生物与用角蛋白或角蛋白环素或其药学上可接受的盐接触或用角蛋白或角蛋白环素或其药学上可接受的盐治疗病毒感染。在一些实施方式中,微生物可以是革兰氏阳性细菌,包括但不限于金黄色葡萄球菌(staphylococcusaureus)、无乳链球菌(streptococcusagalactiae)、化脓链球菌(streptococcuspyogenes)、肺炎链球菌(streptococcuspneumoniae)、艰难梭菌(clostridiumdifficile)、粪肠球菌(enterococcusfaecalis)和枯草杆菌(bacilllussubtilis)。这些隐性的抗菌和抗病毒剂的发现突出了hites-ims在挖掘具有潜在治疗的生物活性的新的代谢产物中的实用性。在一个实施例中,为了验证在高通量筛选中鉴定的激发子和隐性代谢产物的诱导,使每种条件的烧瓶培养物生长并随后通过hplc-ms进行分析。在另一个实例中,为了验证orfamide的产生,如上所述制备了5ml假单胞菌的过夜培养物。然后将培养物稀释到2×125ml锥形瓶中,每个锥形瓶均包含20ml的lb,以使od600nm的初始值为0.01。一个烧瓶补充有终浓度为22μμ的咖啡醇;另一个补充有相同体积的dmso(无咖啡醇对照)。两种培养物均在25℃/150rpm下孵育。44h后,通过离心去除细胞,并在c8prepsep固相提取柱(fisher)上将10ml的每种上清液脱盐。上样后,用水洗涤柱,并对结合的物质用50%mecn(在h2o中)和100%mecn的两步梯度洗脱。馏分在真空中干燥,在100μlmeoh中再溶解,并通过hplc-qtof-ms分析。在另一个实例中,为了验证canucin产生,如上所述制备暗灰链霉菌(s.canus)的种子培养物。将培养物稀释到2×250ml锥形瓶中,每个锥形瓶包含50ml的r4培养基至终菌丝体浓度为0.05%(w/v)。将肯帕罗酮添加到一个烧瓶中(终浓度为17μm),而另一个用作对照并接收相同体积的dmso。然后使培养物在30℃/200rpm下生长5天。将所得的上清液用30ml的乙酸乙酯萃取两次。将有机相合并,在真空中干燥,在200μlmeoh中再溶解,并将两个样品通过hplc-qtof-ms进行分析。在另一个实例中,为了验证角蛋白和角蛋白环素产生,如上所述制备了keratiniphila拟无枝菌酸菌的种子培养物。将培养物稀释到含有0.9ml的r4培养基的深孔96孔板中的3×1ml孔中,最终菌丝体浓度为0.05%(w/v)。在96孔板中补充有终浓度为7.8μμ的长春胺(孔1)、2μμ的dhe(孔2)和相同体积的dmso作为对照(孔3)。然后使板在30℃/200rpm下生长5天。通过离心去除细胞和菌丝体块,并且如上文针对96孔筛选所述建立孔,并通过hplc-qtof-ms进行分析。可替换地,将种子培养物稀释到3×250ml锥形瓶中,该锥形瓶含有50ml的r4培养基,菌丝体浓度为0.05%(w/v)。将烧瓶补充有7.8μμ长春胺(烧瓶1)、2μμdhe(烧瓶2)和相同体积的dmso作为对照(烧瓶3),并随后在30℃/200rpm下生长5天。通过离心去除细胞和菌丝体,并将10ml的每种上清液上样到c18spe柱(phenomenex,100mg)上,用水洗涤,并用50%和100%mecn洗脱。将馏分在真空中干燥,在100μlmeoh中再溶解,并通过hplc-qtof-ms分析。深孔平板法导致角蛋白和角蛋白环素的更好诱导。在其他实施方式中,按照与上述用于小规模发酵的相似程序进行暗灰链霉菌和keratiniphila拟无枝菌酸菌的大规模发酵。如上所述制备3天种子培养物。然后将它们用于接种数个2l锥形瓶,该锥形瓶含有200ml具有分离的菌丝体的r4培养基,终浓度为0.01%(w/v)。然后在培养物中补充有最佳浓度的激发子。通常,8-12l的总培养物用于化合物分离。将烧瓶在30℃/250rpm下孵育7天,此时纯化所需产物。在一些实例中,在配备有自动液体进样器、二极管阵列检测器和6120系列esi质谱仪的agilent1260infinity系列hplc系统上使用反相lunac18柱(phenomenex,5μm,150×4.6mm)进行hplc-ms分析。流动相由水和mecn(均含有0.1%甲酸)组成。注射后,用5%mecn(在水中)等度(isocratically)洗脱3min,然后在20min内用5%mecn到70%mecn(在水中)的梯度洗脱,然后在5min内用70%到100%的梯度洗脱,流速为0.6ml/min。高分辨率(hr)hplc-ms和hr串联hplc-ms在agilent6540accuratemassqtoflc-ms上进行,其由1260infinity系列hplc系统、自动液体进样器、二极管阵列检测器、jetstreamesi源和6540系列qtof组成。样品在lunac18柱(phenomenex,5μm,100×4.6mm)上分解。流动相由水和mecn(+0.1%甲酸)组成。orfamide的洗脱用10%mecn(在水中)等度进行(3min),然后在8min内用10%-95%mecn的梯度进行,然后是在25min内在95%mecn的等度洗脱,流速为0.4ml/min。canucin、角蛋白和角蛋白环素的洗脱用5%mecn(在水中)等度进行(3min),然后在15min内用5%-95%mecn(在水中)的梯度进行,流速为0.4ml/min。在一些实例中,在配备有1260infinity系列二元泵、二极管阵列检测器和自动馏份收集器的agilent制备型hplc系统上进行hplc纯化。在包含a1260infinity系列二元泵或1290infinity四元泵的agilenthplc系统上进行半制备型或分析规模的纯化。每个系统都配备有自动液体进样器、温控柱室、二极管阵列检测器和自动馏份收集器。所用的流动相由水和mecn组成,有或没有0.1%甲酸,取决于样品。在一些实例中,在肯帕罗酮(终浓度为17μμ)的存在下从暗灰链霉菌的12l发酵纯化canucin。发酵7天后,将所得上清液用等体积的乙酸乙酯萃取两次。合并的有机相在na2so4之上干燥,随后在真空中干燥,重悬于45mlmeoh中,并在agilent制备型hplc上纯化。样品通过重复注射至以12ml/min的流速运行、流动相由水和mecn(+0.1%甲酸)组成的制备型lunac18柱(phenomenex,5μm,21.2x250mm)上而被分解,。注射后,洗脱用20%mecn等度进行2min,然后在25min内用20%-100%mecn的梯度进行。汇集如通过hplc-ms所判断的含canucina和b的峰,真空干燥,重悬于meoh中,并在半制备/分析agilenthplc系统上进一步纯化。将肽在半制备型xdb-c8柱(agilent,5μm,10×250mm)上纯化,在2.5ml/min的流速运行,在30min内梯度为30%-50%mecn(在水中),然后在5min内梯度为50%-100%mecn。合并含有纯canucinb的峰,并冻干至干燥。汇集含有canucina的峰,真空中干燥,重悬于meoh中,并在半制备型lunac18柱(phenomenex,5μm,10×250mm)上进一步纯化,在30min内梯度为33%-55%mecn(在水中),然后在5min内梯度为55%-100%mecn。合并含有纯canucina的峰,并冻干至干燥。该过程得到3.6mgcanucina和1.7mgcanucinb。在一些实例中,在2μμ的dhe存在下从keratiniphila拟无枝菌酸菌的8l发酵中纯化得到角蛋白和角蛋白环素。发酵7天后,将得到的上清液上样到预填充的c18柱(phenomenex,50μm,10g)上,并用20%、50%和100%mecn(在水中)逐步洗脱。将含有角蛋白和角蛋白环素的20%馏分在真空中干燥,重悬于50mlmeoh中,并使用lunac18柱(phenomenex,5μm,21.2×250mm)在agilent1260infinity系列制备型hplc上进一步纯化,在12ml/min的流速运行,流动相由水和mecn(+0.1%甲酸)组成。注射后,先用5%mecn(在水中)等度洗脱2min,然后是在20min内5%-40%mecn(在水中)的梯度洗脱,然后是在5min内40%-100%mecn的梯度洗脱。在5-25min的时间范围内,以1min为间隔收集馏分。汇集如通过hplc-ms分析所判断的含有角蛋白a-d的峰,在真空中干燥,重悬于meoh中,并在半制备型/分析的agilenthplc系统上进一步纯化。将样品施加至rpamide-cl6柱(supelco,5μm,10×250mm),在2.5ml/min的流速运行,流动相与上述相同,在30min内为8%-16%mecn(在水中)的梯度,然后在5min内为16%-100%mecn的梯度。合并含有纯角蛋白c和d的峰,并冻干至干燥。汇集含有角蛋白a和b的峰,在真空中干燥,重悬于meoh中,并在半制备xdb-c8柱(agilent,5μm,10×250mm)上进一步纯化,在30min内为5%-15%mecn(在水中)的梯度,然后在5min内为15%-100%mecn的梯度。合并含有纯角蛋白a和b的峰,并冻干至干燥。该过程得到8.5mg角蛋白a、1.6mg角蛋白b、5.1mg角蛋白c和0.8mg角蛋白d。汇集来自制备型lunac18柱的含有角蛋白环素a-c的峰,在真空中干燥,重悬于meoh中,并在半制备型/分析的agilenthplc系统上进一步纯化。将样品施加到rp酰胺-c16柱(supelco,5μm,10×250mm),在2.5ml/min的流速运行,流动相与上述相同,在30min内为10%-20%mecn(在水中)的梯度,然后在5min内为20%-100%mecn的梯度。合并含有纯角蛋白环素a和c的峰,并冻干至干燥。汇集含角蛋白环素b的峰,在真空中干燥,重悬于meoh中,并在半制备xdb-c8柱(agilent,5μm,10×250mm)上进一步纯化,在30min内用5%-15%mecn(在水中)的梯度,然后在5min内用15%-100%mecn的梯度。此过程产生了2.7mg角蛋白环素a、5.3mg角蛋白环素b和1.2mg角蛋白环素c。在一些实例中,为了结构阐明,在具有三共振冷冻探针的a8avanceiiihd800mhznmr光谱仪(bruker)上获取1d/2dnmr光谱。在dmso-d6中制备了角蛋白a-d、角蛋白环素a-c的nmr样品,在cd3oh中制备了canucina和b的nmr样品。在一些实例中,在295k的cd3oh中以500ms的混合时间获得的canucina的noesy光谱表现出最大数量的相关性,同时避免了自旋扩散,因此被用于结构计算。在mestrenova中测量该光谱中的交叉峰位置和体积,并手动分配。这些作为计算的初始输入数据给出,在linux集群上的cyana2.1中执行。通过显式的距离约束,针对glyl的n和asp8的cy之间的n-c键,将异肽键并入。具体而言,n-cy键长度的上限和下限均设置为加权因子为1.00。这些距离基于酰胺键的平均键长。使用cylib软件2将canucina中非天然氨基酸□-oh-asp编码进cyana残基库中。进行了结合的noesy赋值和结构计算的七个循环,然后进行了最终结构计算。用于从跨峰体积中提取距离约束的校准参数被自动确定。对于每个循环以及对于最终的计算,生成100个初始构象子,并将由10000个扭转角动态步长组成的模拟退火方案应用于每个构象子。为具有最低最终靶标功能的10个构象子生成了统计信息(见表s3)。计算出的构象子在pymol中进行可视化。为了确定抗菌和抗病毒性能,进行了测定法。在一个实例中,由micromyx,llc按照来自clinicalandlaboratorystandardsinstitute(临床实验室标准化协会)的方法进行抗菌测定法。用以下菌株确定最小抑制浓度:金黄色葡萄球菌atcc29213、金黄色葡萄球菌mmx2011、肺炎链球菌atcc49619、化脓链球菌mmx6253、无乳链球菌mmx6189、粪肠球菌atcc29212、粪肠球菌mmx486、枯草杆菌atcc6633、大肠杆菌(e.coli)atcc25922、肺炎克雷伯菌(k.pneumoniae)mmx214、铜绿假单胞菌(p.aeruginosa)atcc27853、鲍曼不动杆菌(a.baumannii)atcc19606、霍乱弧菌(v.cholerae)baa-2163、艰难梭菌atcc700057和脆弱拟杆菌(b.fragilis)atcc25285。在该实例中,由virapur按照来自clinicalandlaboratorystandardsinstitute(临床实验室标准化协会)的方法进行抗病毒测定法。用以下病毒和宿主细胞(在表1中列出)测定了最低抑制浓度:mdck细胞中的a型流感/perth/16/2009、mdck细胞中的b型流感/wisconsin/1/2010、vero细胞中的单纯疱疹1株macintyre、vero细胞中的单纯疱疹2株g、vero细胞中的痘苗病毒wr、hela细胞中的鼻病毒8和hep2细胞中的呼吸道合胞病毒。在另一个实例中,从拟无枝菌酸菌属b24117的大规模生产培养物纯化角蛋白和角蛋白环素。在6×4l锥形瓶中培养总共6l,每个烧瓶包含1l的r4培养基。用拟无枝菌酸菌属b24117的过夜培养物接种烧瓶,补充有30μm的双氢麦角汀,并培养6天。6天后,通过离心(30min,8000g)去除细胞,并将上清液上样到预包装sep-pak柱(5g)中。用水洗涤角蛋白和角蛋白环素,然后用10%、25%、50%和100%mecn(在h2o中)的梯度洗脱。如通过hplc-ms所测定的,糖肽在25%馏分中洗脱。使用配备有光电二极管阵列检测器和自动馏分收集器的agilent1200系列分析型hplc系统,在以2.5ml/min运行的c18半制备型柱上通过hplc进一步纯化它们。该柱在10%mecn(在水中)中被平衡,mecn和水均包含0.1%甲酸。注入粗物质后,在35min内以10-60%mecn(在水中)的梯度进行洗脱。收集如通过hplc-ms所判断的含有糖肽的馏分,在真空中干燥,重悬于小体积的meoh中,然后再施加到同一柱上。在1小时内以13%mecn(在水中)进行等度洗脱。在agilent6540uhdaccuratemassq-toflcms系统上通过高分辨率hplc-esi-ms(hr-ms)对纯角蛋白和角蛋白环素进行表征,该系统由1260infinity系列hplc系统、自动液体进样器和二极管阵列检测器、jetstreamesi源和6540系列q-tof组成。hr-ms检测被校准到0.5ppm以内。将样品在c18的eclipse柱(agilent,2.7μm,2×50mm)上分解,在13min内以0.4ml/min、10%mecn(在h2o中)(和0.1%甲酸)到95%mecn(在h2o中)(和0.1%甲酸)的梯度下运行。还通过nmr表征化合物:1h、13c和2dnmr光谱记录在配备有冷冻探针的brukeravanceiii800mhz光谱仪上。光谱常规地在dmso-d6中获得,化学位移参考残留溶剂峰。对这些数据的分析允许各种化合物的结构阐明,图21a-21f所示。化合物包括:角蛋白a(图21a)、角蛋白b(图21b)、角蛋白c(图21c)、角蛋白环素a(图21d)、角蛋白环素b(图21e)和角蛋白环素c(图21f)。这些化合物有生物活性。在某些情况下,化合物显示出对革兰氏阳性细菌的活性。例如,尽管它们也可以表现出对革兰阴性靶标的活性,但是角蛋白针对各种革兰氏阳性靶标测试了角蛋白b和c的生物活性(图21b和21c)。结果如下表1所示。表1.角蛋白a和c的生物活性靶标角蛋白b角蛋白c金黄色葡萄球菌mssa8μg/ml8μg/ml金黄色葡萄球菌mrsa48肺炎链球菌0.251化脓链球菌0.52无乳链球菌14粪肠球菌vse48粪肠球菌vre>64>64艰难梭菌0.50.5。序列表<110>普林斯顿大学(thetrusteesofprincetonuniversity)<120>隐性代谢产物和用于激活多种微生物中沉默的生物合成基因簇的方法<130>ppi20003250us<150>us62/539,263<151>2017-07-31<160>1<170>patentinversion3.5<210>1<211>14<212>prt<213>暗灰链霉菌(streptomycescanus)<400>1glyvalglypheproleualaaspphepheilehispheasp1510当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1