一种促进侧根形成的大豆低磷响应基因及其蛋白与应用的制作方法
本发明涉及基因工程技术领域,更具体地,涉及一种促进侧根形成的大豆低磷响应基因及其蛋白与应用。
背景技术:
根系是陆生植物在土壤中固定和有效吸收水和矿物质营养的关键,在植物适应各种逆境胁迫方面起着至关重要的作用。根系构型(rsa)是根系统各个部分的整体空间布局,是决定植物有效地获取资源的一个重要因素。植物的生产力受根系结构性状的影响较大,根系结构性状是农业改良的重要目标。根系结构特征的改变可以改变植物根系对异质土壤中不同层的接触,从而使植物有效地吸收更多的养分和水分。这是因为像水和氮这样的资源通常在较深的土层中发现,而磷等其他营养物质在较浅的土层中更为丰富。因而植物根系构型和形态对植物生长发育及适应各种逆境胁迫具有非常重要的作用。
目前研究显示:磷信号参与了改变植物根系构型和形态。植物在缺磷条件下,会做出一系列的应答反应以维持自身磷稳态,其中一个重要的应答反应是根构型的重塑。
研究表明,在低磷环境中,根系会改变其形态特征,提高对土壤中难溶性磷的吸收能力,帮助植物在低磷环境中更好的吸收、利用养分,增强植物在逆境中的适应能力。如低磷胁迫促使大豆根长变长、根表面积增加;磷胁迫下,豇豆会通过增加根长度以及侧根数量来扩大根系的吸收范围;拟南芥在低磷胁迫下,主根的生长受抑制,而侧根数、根毛数变多,从而增加植株与磷的接触面积,提高对磷的吸收;苜蓿通过增加根毛的数量和长度,以应对低磷胁迫,另外也可通过建立菌根组织或形成排根,从而从土壤中获取更多的磷供植物生长;玉米和蚕豆在缺磷条件下,茎的生长显著地受抑制,而根系生长和根系总长度则增加。综上,磷胁迫会参与植物根系构型和形态的改变。
大豆(glycinemax)是人类和动物重要的蛋白质和油脂资源,也是高磷需求品种,因此对低磷胁迫的敏感性,是大豆生产的主要制约因素。大豆作为豆科作物,能够与根瘤菌共生,形成根瘤而进行生物固氮反应,该反应具有减肥增产、培肥土壤、改善土壤质量等作用。低磷胁迫对大豆生长有多方面的影响,如大豆根瘤发育和固氮减少,花荚脱落增多,影响植株整体生长发育,从而影响产量和种子质量。
nf-ya是一类广泛的存在于真核生物的转录因子,近年来研究发现,其广泛的参与了植物胚胎发育、光合作用、开花时间、逆境胁迫响应等众多生理过程,并发挥了重要的调控作用。目前,对大豆已有一定的研究,但对大豆适应低磷胁迫,改变根系构型和形态的机制,及nf-ya转录调控因子是参与相关的调控过程的尚不清楚。
技术实现要素:
本发明的目的是为了克服现有技术尚不清楚大豆适应低磷胁迫,改变根系构型和形态的机制,及nf-ya转录调控因子是参与相关的调控过程的不足,提供一种促进侧根形成的大豆低磷响应基因及其蛋白与应用。
本发明的第一个目的是提供gmnfya7蛋白或其激活剂在促进植物根系生长中的应用。
本发明的第二个目的是提供gmnfya7基因或其激活剂在促进植物根系生长中的应用。
本发明的第三个目的是提供gmnfya7蛋白或其激活剂在促进抗低磷胁迫中的应用。
本发明的第四个目的是提供gmnfya7基因或其激活剂在促进抗低磷胁迫中的应用。
本发明的第五个目的是提供一种促进植物根系生长、增加植物侧根数量、和/或促进侧根原基突破表皮的方法。
为了实现上述目的,本发明是通过以下技术方案予以实现的:
本发明发明人发现一个受低磷诱导表达的nf-ya基因,命名为gmnfya7,我们推测其参与大豆低磷信号转导。利用转基因技术过量表达gmnfya7基因的拟南芥材料,研究发现过量表达gmnfya7基因的拟南芥材料与哥伦比亚野生型拟南芥(col-0)相比,在低磷条件下,显著促进侧根原基突破表皮,提高侧根数量,可能促进磷的吸收。
因此本发明要求保护以下内容:
gmnfya7蛋白或其激活剂在促进植物根系生长中的应用,其特征在于,gmnfya7蛋白氨基酸序列如seqidno:4所示。
gmnfya7基因或其激活剂在促进植物根系生长中的应用,其特征在于,gmnfya7基因核苷酸序列如seqidno:1、seqidno:2或seqidno:3所示。
其中,gmnf-ya7基因组序列的核苷酸序列如seqidno:1所示、gmnfya7转录本序列的核苷酸序列如seqidno:2所示、gmnfya7cds序列的核苷酸序列如seqidno:3所示、gmnf-ya7蛋白序列的氨基酸序列如seqidno:4所示。
优选地,所述促进植物根系生长为增加侧根数量。
优选地,所述增加侧根数量是由于侧根原基突破表皮引起的。
优选地,所述植物为双子叶植物。
gmnfya7蛋白或其激活剂在促进抗低磷胁迫中的应用;gmnfya7基因或其激活剂在促进抗低磷胁迫中的应用,也属于本发明的保护范围。
同时本发明要求保护一种促进植物根系生长、增加植物侧根数量、和/或促进侧根原基突破表皮的方法,在植物中过表达gmnfya7基因和/或gmnfya7蛋白。
本发明选用哥伦比亚野生型拟南芥作为转基因受体材料,但是以上所述植物包括并不限于拟南芥、大豆、或其他豆科植物在内的各种植物。
与现有技术相比,本发明具有如下有益效果:
本发明为包括大豆在内的豆科作物,以至于双子叶植物,在nf-ya调控因子参与植物响应低磷胁迫及改变根系构型和形态的机制上,提供理论基础。同时本发明为植物通过改变根系构型和形态、适应低磷胁迫,提供了一种有效的方法,对农作物具很大的商业价值,值得推广和应用。
附图说明
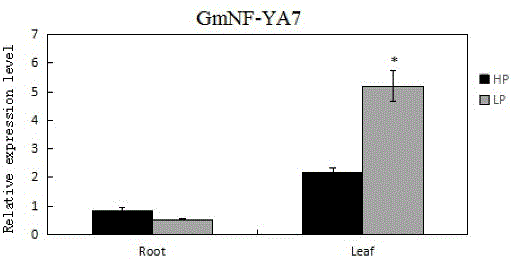
图1为gmnfya7基因在不同磷水平的表达模式;+p表示含正常磷营养,250μmkh2po4;-p表示低磷处理,12.5μmkh2po4;root表示从大豆的根提取的rna;leaf表示从大豆的叶提取的rna;图中数据是3个生物学重复的平均值和标准误,星号代表2个磷水平间差异(student’st-test),*表示差异显著(ρ<0.05),**表示差异极显著(ρ<0.01),***表示差异极其显著(ρ<0.001)。
图2为过表达gmnfya7基因的转基因拟南芥gmnfya7基因相对表达量;
图3为过表达gmnfya7基因对拟南芥转基因植株根系的影响;col-0为哥伦比亚野生型拟南芥,oe-gmnfya7#5、oe-gmnfya7#20为过表达gmnfya7株系;a为侧根数,b为主根长,c为侧根密度;hp为正常营养条件,lp为缺磷处理,处理7天;图中数据为12个样本的平均值和标准差(se),星号代表同一处理下不同植株与野生型间的差异(student’st-test),*表示差异显著(ρ<0.05),**表示差异极显著(ρ<0.01),***表示差异极其显著(ρ<0.001)。
图4为wt和oe-gmnfya7#5、oe-gmnfya7#20在低磷处理上的侧根数;col-0为哥伦比亚野生型拟南芥,oe-gmnfya7#5、oe-gmnfya7#20为过表达gmnfya7株系;图中数据为12个生物学重复的平均值和标准差(se),星号代表同一处理下不同植株与野生型间的差异(student’st-test),*表示差异显著(ρ<0.05),**表示差异极显著(ρ<0.01),***表示差异极其显著(ρ<0.001)
图5为低磷条件下过表达gmnfya7对转基因拟南芥侧根数影响实物图;col-0为哥伦比亚野生型拟南芥,oe-gmnfya7#5、oe-gmnfya7#20为过表达gmnf-ya7株系;低磷条件下,处理7天。
具体实施方式
下面结合说明书附图和具体实施例对本发明作出进一步地详细阐述,所述实施例只用于解释本发明,并非用于限定本发明的范围。下述实施例中所使用的试验方法如无特殊说明,均为常规方法;所使用的材料、试剂等,如无特殊说明,为可从商业途径得到的试剂和材料。
实施例1gmnfya7基因定量实验
一、实验方法
1、定量pcr引物设计
从网站下载gmnfya7基因组序列(核苷酸序列如seqidno:1所示)、gmnfya7转录本序列(核苷酸序列如seqidno:2所示)、gmnfya7cds序列(核苷酸序列如seqidno:3所示)、gmnf-ya7蛋白序列(氨基酸序列如seqidno:4所示)。
设计得到其特异的定量扩增引物:
gmnfya7.qf:catgacctctcagataatgaagc;
gmnfya7.qr:tgcaagtatggcttccgat。
2、大豆材料的种植和缺磷处理
a、种子消毒
将成熟、饱满的大豆种子单层排列在培养皿中,放入干燥器中,并将培养皿打开,盖子紧挨着培养皿。将100ml的次氯酸钠加至干燥器中的250ml烧杯中,然后沿着杯壁缓缓加入4.2ml浓盐酸(hcl),立即密闭干燥器,静置10小时。从干燥器中拿出后除去过多的氯气;
b、催芽
将消毒后的种子点入湿润的石英砂中催芽萌发;
c、移苗
一周后两叶一心时选择正常、长势一致的幼苗移栽至水培系统,于温室中用1/2大豆营养液培养;
d、处理
一周左右待第一片三出复叶完全展开后进行处理:低磷(lp:25μm)、高磷(hp:500μm),每个处理5个生物学重复。每周换一次营养液,每3天调一次ph。
e、采样
对处理后14天的根、叶采样,液氮冷冻,-80℃保存。
(3)总rna的提取
参照trizo1一步法分离总rna。取0.2g样品放入预冷的研钵中磨成粉末状,转至1.5ml离心管,加入1mltrizo1提取液,剧烈震荡混匀,室温静止5分钟;加入0.2ml氯仿,大力摇动,室温静止2~5分钟,4℃下12000rpm离心15分钟;取上清转入新管,加入0.5ml异丙醇,室温放置10分钟,4℃下12000rpm离心5分钟,倒掉乙醇,风干沉淀,加入depc水溶液;最后测其od值,来确定rna的纯度和浓度。
(4)实时荧光定量pcr样品的制备及分析
用dnasei处理总rna以移除基因组dna的污染,按逆转录酶说明书将rna反转成第一条链。将所得第一链稀释100倍作为定量pcr反应模板。将适量cdna原液做梯度稀释为标准曲线的模板。反应体系及反应条件分别见表1和表2。
表1定量pcr反应体系:
表2定量pcr反应条件:
用rotor-gene的real-timeanalysissoftware6.0计算每个样品的表达量。以大豆看家基因gmef1a为参照基因。相对表达量为目的基因的表达量与看家基因表达量的比值。
二、实验结果
gmnfya7在不同磷水平的表达模式如图1所示,gmnfya7基因在大豆叶部或根部,在低磷处理下都被诱导表达。
实施例2gmnfya7基因过表达载体构建(gateway方法)
一、实验方法
1、引物设计
从网站下载gmnfya7基因cds序列(核苷酸序列如seqidno:3所示),设计其特异的扩增引物,
oe-gmnfya7.f:
gggacaagtttgtacaaaaaagcaggcttctcatgtgtgtagattgggacactg;
oe-gmnfya7.r:
ggggaccactttgtacaagaaagctgggtccttaggatgttctatctgatggtgc。
2、片段扩增及纯化
以大豆yco3-3的cdna为模板,用oe-gmnfya7的特异引物扩增相应片段,反应体系及反应条件见下表3和表4;将pcr反应产物跑琼脂糖凝胶电泳,并将位置相符的亮带切下回收,再使用琼脂糖凝胶dna回收试剂盒回收pcr产物,得到目的片段。
表3pcr反应体系:
表4pcr反应条件:
3、中间载体pdonr207的连接转化
将回收目的片段与pdonr207载体连接后转化大肠杆菌dh10b,挑单克隆后,小管摇菌进行pcr检测后,送测序公司测序,中间载连接体系见下表5;
表5中间载连接体系
4、目的载体pmdc32连接转化
测序正确,用质粒小量快速提取试剂盒提取质粒,然后将带有目的片段的质粒与目的载体pmdc32进行连接,然后转化到大肠杆菌dh10b,挑单克隆后,小管摇菌进行pcr检测后,送测序公司测序。测序正确后将菌液和质粒分别保存备用。同时将重组的目的载体转化农杆菌菌株gv3101,检测无误后保存菌液备用,目的载连接体系见下表6。
表6目的载连接体系
二、实验结果
成功构建了gmnfya7基因过表达载体。
实施例3拟南芥遗传转化
一、实验方法
1、野生型拟南芥(col-0)的种植
将野生型拟南芥种子(col-0)均匀播到装有基质的营养钵表面,并喷一些水,然后用塑料膜盖住保湿,并放到4℃黑暗条件下2~3天以打破种子休眠。
然后放置在人工气候室(16小时光照24℃/8小时黑暗24℃)培养,等长一个星期后,然后分别移栽到营养钵中,每盆3~4株,在第一次抽薹1cm时,将主茎剪掉使分枝增多,待下次拟南芥盛花时,可用农杆菌液进行转化。
2、农杆菌液对拟南芥花絮转化
将携带目的载体(实施例2构建的gmnfya7基因过表达载体)的农杆菌gv3101保存液进行划板活化,首先接种到50ml的yep(kan+rif)培养板上,在28℃条件下培养2天;待长菌后挑单克隆,小管摇菌2mlyep(kan+rif)液体,然后将农杆菌液在500mlyep(kan+rif)液体中进行扩大培养,28℃下200rpm培养20小时。4000rpm下将菌液离心10分钟,弃上清,然后用一倍体积的新鲜浸润液进行重悬农杆菌,然后转移到500ml烧杯。将盛花的拟南芥地上部分,浸没在农杆菌液中10秒,并轻轻搅拌,然后将浸泡的植物拿出,用塑料盖子盖住转化后的拟南芥,并侧放16~24h以确保较高的湿度,第二天将塑料盖子去掉,放拟南芥室进行生长待成熟收t0代种子;浸润液配方见下表7。
表7浸润液配方:
3、转基因拟南芥后代鉴定
将成熟的t0代种子进行消毒,先用75%的酒精进行消毒,然后再用100%酒精进行消毒,最后将酒精吹干;将消毒的种子播到1/2ms培养基(含潮霉素×1000)上,进行t1代转基因拟南芥筛选,拟南芥幼苗主根长的,说明抗潮霉素,有可能是转基因材料;然后将主根长的拟南芥幼苗转到基质中生长,收获t1代转基因材料种子,然后又重复上一步骤,用潮霉素进行筛选,看拟南芥幼苗主根长短,是否符合3:1(单拷贝插入),如果符合则可以确定是转基因材料,待成熟收获t2代拟南芥种子。
在t2代转基因拟南芥生长期间,对过表达转基因株系采样提取rna,反转录成cdna后利用荧光定量pcr检测基因相对表达量。具体步骤如下:将在基质上生长14天的拟南芥莲座叶,采叶片0.1g,液氮冷冻,-80℃保存。然后用rna提取试剂盒提取rna,进一步反转录成cdna,最后用荧光定量pcr检测基因相对表达量(具体操作同时实施例1)。
二、实验结果
gmnfya7基因的相对表达量如图2所示,过表达gmnfya7#5、#6、#20的株系中gmnfya7的表达水平明显高于野生型,其中oe-gmnfya7#5、oe-gmnfya7#6、oe-gmnfya7#20株系分别为野生型的1192倍、5602倍和4875倍,统计分析显示差异达到显著(*,ρ<0.05),极显著水平(**,ρ<0.01),极其显著水平(***,ρ<0.001)。
实施例4过量gmnf-ya7功能分析实验
一、实验方法
(1)种子消毒
将成熟、饱满的拟南芥种子放入2ml管中,在超净台上,用75%的酒精1ml吸入到2ml管中,覆盖种子并搅拌均匀后吸出酒精,操作时间不超过1分钟,然后用100%的酒精进行第二次消毒,重复上一步骤,然后放到超净台让种子吹干。
(2)种子萌发
将消毒后的种子撒播到正常营养的1/2ms板上,然后放到4℃黑暗条件下2-3天以打破种子休眠,然后放置在人工气候室(16小时光照24℃/8小时黑暗24℃)培养。
(3)移苗及磷营养处理
种子萌发三天后,分别移20株长势一致的拟南芥幼苗到高磷、低磷处理的1/2ms板上,然后放置在人工气候室(16小时光照24℃/8小时黑暗24℃)培养。其中高磷(p:0.625mm),低磷(p:0mm),每个处理20个样本,三次生物学重复,生长7天。测定指标:侧根数、主根长、侧根密度、突破表皮的侧根数。
二、实验结果
图3到5为拟南芥生长7天后,过量gmnfya7对转基因拟南芥植株根系的影响。如图所示,在正常和低磷条件下,oe-gmnfya7株系(#5、#20)的侧根数与野生型相比明显增加;而主根长、侧根密度与野生型相比没有显著差异;在低磷胁迫下,oe-gmnfya7株系(#5、#20)突破表皮的侧根数显著高于对照。结果显示,gmnfya7基因在低磷条件下显著促进侧根原基突破表皮,提高侧根数量。
序列表
<110>华南农业大学
<120>一种促进侧根形成的大豆低磷响应基因及其蛋白与应用
<160>4
<170>siposequencelisting1.0
<210>1
<211>5679
<212>dna
<213>glycinemax
<400>1
gttaagtttgcctttatttgtgtgggtgggtgtggtgtggataggtggcagctacgacac60
aacacaacacaacacaacacaacacagtcctaagttgtaagaaacactctcttctccttt120
ctcactattgttctgttactgttttttgcagcaacacttcagttcaattaacgaactaca180
ccactttctttctcttcttcgactgctctgtaaccgaaaacctccctttcccagtttcga240
atcttttgtttctgcctttggttactgtttttccgagccatgctattcattattgtcctt300
cgaatcggattggtaacaaggaatttatgtgtgtaggtgtgtgtataaatatataagttg360
tgcttgctagcttgatttgaacttctttctttgtattattggttggttatgcttacgagt420
ctttgaatttgaatcctatggttttactattgggctaagaattggaaaaggtagcttgca480
aatttataattaccagcttggtaacttggttagttgtgaatatgggtctgcataattaaa540
atggtattcagatgtgattttaggagatggttatgcactgaatttaagatgttaggttaa600
acctcaaaatgaggaatagtgtcaatttttttacattggataggttaaacaacccttttc660
cttgatttgttgttttaattaaattttatcatatcttaaatggattagtagtttattttg720
gtggttccattatattccaatccaaaatccttatgtttgtcccccaaatcctattcttag780
agctatgattaataggtgtttctaaatcaatttgatatacaattgaattgtcttctgcag840
acccttttagtggactcttttaataatccatagaatatgaaaattgaattacttgtatgc900
tctctactcccttggctgcagtaattggcggtacagtaaatgattgactataagtttagt960
gggactgtgggaggcacttcatttctaccttgggtgaaaattggagaccatgcctaacca1020
caagtcacatggttaagctatcttgagttcttcatacaaaatgatggagaattaaagggg1080
aatgtagaccataggattcaaatggggtagacaaaattgaagtgtgccttaagtgttgtt1140
tgagatagaaaggcacgacttaatcttaaggggaaattttacctcacaagtcacaactat1200
aagacctgtgatgttgtatgtaaatggaatttatttttgttttatgcctacatctatgtt1260
ttaacatataatatccaaaagataacttcttgagttttattaactatgtgttctgcttcc1320
agattgggacactgtattgcatgtaaatcaggaaatcatgacttctactcatgacctctc1380
aggtatttacattgaaaaaacccttttccaattatcttttaagctacctttttaatttgt1440
tataagtgcataacatttatttctcagtagtatacaatttatgggagaatttcatggttt1500
gttgtctgatttgatcacccatgtaagaacatttgttcaacagaaatgatcaccttgaca1560
tgcattctaaatgcaatagttgtggtgatactagggtgttcgccagaacttatgtgctac1620
ggtgaaagttttattgaattgcagctagacttgcaatgatgagtggactgaaatctgggt1680
attgagctgcaatcaaaatggcacaaaaatcaggaatgttgtcattgatgagtgggcaaa1740
gttgagatggaaaaccattcaagatggcttgggaatgtaagacgaatggatgatgaccta1800
cattggtccttgtaaatgtggtattgtggtattgctgaatatgcccctttaaaaagagat1860
gacgagacaaaaaggacatggtggaagactatttagaaggatggggagtacctagggaga1920
cccacatattgtcttatttcaagccttaaacctattattttatatttcataaactcaact1980
ttggaacttatttgttctaattttagctagtcccttgttagcctatttgacattcatttt2040
aattttggccaattcatgcaatagttacctttctgacctactaccatattgtttttattt2100
tctgtttgtttgagtaagaatgtcacccttgacaaatataatgtgaatatgaaatctata2160
ttctaaaagattatggtgtgaagtgctgagctgtaataatgtgcataaaataatgcttat2220
aaatattttcctaaaactattgaagcataaatgtctatttgaagctttttgtctctcaat2280
gaaaggtacacctgtgcaggatggaattggggttggaatttgaatatcttattaatattc2340
atctagaaattgaaacatatcttctttatctaattccctgtctgttcatctgttttttcg2400
ttggaaaaatgccagagaaaatgatgttagtaaacaataaatccctaacatatataaaaa2460
gctaataattgcaggatgtacagaaattaaccctaaattcttttttttcccaagtttctt2520
cgtaccccattgcccagaggctcttcgctatgcgaaggtatgggggagggatgttgtacg2580
cagccttacccttacatatacaaagaggctgtttccggattcgaacccatgaccaacagg2640
tcaccaaggcacaactttaccactgcaccagggctcgccctctgtataataactaagtaa2700
aaactaagataatttttttttgtggttgctgagaaaaattaattacactatctctattta2760
ttaatattaagcaaattgcaaattctgtttttatgggctactatttttacttatggtgta2820
caattcttactggattacttcgtgagattctcactgtattactggattcagtagtatatg2880
ttctcagttttacagtctgtctatttcttgttctgcaatttattattaattaaattctgt2940
ttattttctctccttttttcttctcttatttttatttagatatctctattcttttttatt3000
gctgtattgaatagctgtccttcagtgattttaaatgactgaaagtcgttagtttaagat3060
tgaaggaatttgaaacaacacaaactcctgttctttactctgtagcagcgtaagttatct3120
tttaaccaaagtagtgtgcatgaataataaatcttcaacatcctaaacaaaccactaact3180
cgtaccttctccccccaataaagcctcacttcatgcccaaagaaaataaaataagtaaat3240
tacaaagaagaaattataaaagctcactttatttgacagattaggcataaaagttccatt3300
caaagaatatccctttctcttctgtttgtttgaaactaaatattttcggcagcctctgtt3360
tctcttaaaacttacaaactgaacatacatccctttcaaactgacagattgggcataaag3420
ttccattcaaagaatatcccttttttctctttgtttcttccaaactaaatatattcagca3480
tcttctgtttcttttagaactcgtaactaaacattgatctggagcttttgtgttccgagt3540
ggagtagatgcttctgcattctggttatggtacatgagtagaaaattatttcttgcttag3600
cttcgtggccctatttggttgactttcaattatttatcttaatatttttttatttttttg3660
agttagtctgccatgctatctaaagtggtgtttttcttaaagtgatgcatttgactagtc3720
caatcaccccccaaaaaaatgtacagaagtaatggctaatcctgaagttattcaactcat3780
tggtgtctagataatgaagctgatgaccagcagcagtcggaatcacaaatggagccttta3840
tctgcaaatggaatttcttatgcaggtattgctactcagaatgttcagtatgcaacacct3900
tcacagcttggaactgggcatgctgtggtatgaacttcacattttaatgggccatcttat3960
cattgtcttagtaaacaatatatgttttggtttgccatcctcagtgctctcattggagct4020
ggtatctcagtaaatgcttaggaatgttttgttttgtattggtcataggctcagcctttt4080
gatgtcatcaaataatgaagttttgtccgaattttgctgttttccttggaggttttgttt4140
gatgatgaaattattttctttttaaataaccctagttttttttacctgacaattgaaggt4200
tagtgaatgattgcctgagcaagtactggtgccagttgttatcatgttatgttgatgtaa4260
tataaagaagtaagaagtggccgctcagacttggtcaggcaattgaattacttctttgtc4320
ccattggccctcctggattttcttatctcattgctgttggtttttctgtcctgtcatatt4380
ttagaaaataaactgtgtttgaactttactgatttttgttgcttggtgtatctaattgaa4440
ggtaccgcccacttacccatatccagatccatactacagaagtatctttgctccctatga4500
tgcacaaacttatcccccacaaccctatggtggaaatccaatggttcattctaatctttt4560
gttgttgatgtttgcaatatagtgtttcttgttatgttcctcactattgaatcgtttcct4620
gtgccaggtccaccttcagttaatgggaattcaacaagcaggtgttcctttgccaactga4680
tacagttgaggagcctgtgtttgtcaatgcaaaacagtatcatggtatattaagacgcag4740
acagtcccgtgctaaagctgaatcagaaaaaaaggctgcaaggaatcggaaggtatatcc4800
ctacccctatctgttctcatgttcctctttaataatgcgaaaagatgattaagaaaaact4860
aattgttaaaaaaggaaaacgaagggaagaaggttcctggaagttgtgttgttagctgct4920
aacttctagatgttgatatttttgtgtgtgcctacatggatttgtttgactcacaaactt4980
ccatgcatccttcatggatttacttgatccttaaacaaatgtttcagccatacttgcatg5040
aatctcgacatttgcatgcactgagaagagcaagaggatgtggaggtcggtttttgaatt5100
caaagaaagatgagaatcaacaggatgaggttgcatcaactgacgaatcacagtccacta5160
tcaatctcaattctgataaaaatgagcttgcaccatcagatagaacatcctaaaactaca5220
gaaatggtgatgctgtagattgcagggatctgttgtgtatatctatattgggagatgaat5280
ctccaaccaacagtatcctcagatatctccctattattcattctgtcgtacaacgccata5340
ggtataagtataggttgtgtagtaggtatgttaggaggttgcaaaataaaacaagtaaaa5400
tgtaaattgaagtgattcaactaagtctatccccaatgtggtcctttcttgcctttttag5460
gtatttttattgtgtgggcttttctttgtattatttggtgcctctgagggaaagagaaga5520
gattatccgaatgcgtgatggtttggttggatcatgtttatttagcttgtgagaattaat5580
tgttgtagcactcttttgatttgttgttataaacgaggatgaaaagtttaaaccaggatt5640
ggtcaagttccgcacagttgcagatagaaggaagaggtt5679
<210>2
<211>1452
<212>dna
<213>glycinemax
<400>2
gttaagtttgcctttatttgtgtgggtgggtgtggtgtggataggtggcagctacgacac60
aacacaacacaacacaacacaacacagtcctaagttgtaagaaacactctcttctccttt120
ctcactattgttctgttactgttttttgcagcaacacttcagttcaattaacgaactaca180
ccactttctttctcttcttcgactgctctgtaaccgaaaacctccctttcccagtttcga240
atcttttgtttctgcctttggttactgtttttccgagccatgctattcattattgtcctt300
cgaatcggattggtaacaaggaatttatgtgtgtagattgggacactgtattgcatgtaa360
atcaggaaatcatgacttctactcatgacctctcagataatgaagctgatgaccagcagc420
agtcggaatcacaaatggagcctttatctgcaaatggaatttcttatgcaggtattgcta480
ctcagaatgttcagtatgcaacaccttcacagcttggaactgggcatgctgtggtaccgc540
ccacttacccatatccagatccatactacagaagtatctttgctccctatgatgcacaaa600
cttatcccccacaaccctatggtggaaatccaatggtccaccttcagttaatgggaattc660
aacaagcaggtgttcctttgccaactgatacagttgaggagcctgtgtttgtcaatgcaa720
aacagtatcatggtatattaagacgcagacagtcccgtgctaaagctgaatcagaaaaaa780
aggctgcaaggaatcggaagccatacttgcatgaatctcgacatttgcatgcactgagaa840
gagcaagaggatgtggaggtcggtttttgaattcaaagaaagatgagaatcaacaggatg900
aggttgcatcaactgacgaatcacagtccactatcaatctcaattctgataaaaatgagc960
ttgcaccatcagatagaacatcctaaaactacagaaatggtgatgctgtagattgcaggg1020
atctgttgtgtatatctatattgggagatgaatctccaaccaacagtatcctcagatatc1080
tccctattattcattctgtcgtacaacgccataggtataagtataggttgtgtagtaggt1140
atgttaggaggttgcaaaataaaacaagtaaaatgtaaattgaagtgattcaactaagtc1200
tatccccaatgtggtcctttcttgcctttttaggtatttttattgtgtgggcttttcttt1260
gtattatttggtgcctctgagggaaagagaagagattatccgaatgcgtgatggtttggt1320
tggatcatgtttatttagcttgtgagaattaattgttgtagcactcttttgatttgttgt1380
tataaacgaggatgaaaagtttaaaccaggattggtcaagttccgcacagttgcagatag1440
aaggaagaggtt1452
<210>3
<211>660
<212>dna
<213>glycinemax
<400>3
atgtgtgtagattgggacactgtattgcatgtaaatcaggaaatcatgacttctactcat60
gacctctcagataatgaagctgatgaccagcagcagtcggaatcacaaatggagccttta120
tctgcaaatggaatttcttatgcaggtattgctactcagaatgttcagtatgcaacacct180
tcacagcttggaactgggcatgctgtggtaccgcccacttacccatatccagatccatac240
tacagaagtatctttgctccctatgatgcacaaacttatcccccacaaccctatggtgga300
aatccaatggtccaccttcagttaatgggaattcaacaagcaggtgttcctttgccaact360
gatacagttgaggagcctgtgtttgtcaatgcaaaacagtatcatggtatattaagacgc420
agacagtcccgtgctaaagctgaatcagaaaaaaaggctgcaaggaatcggaagccatac480
ttgcatgaatctcgacatttgcatgcactgagaagagcaagaggatgtggaggtcggttt540
ttgaattcaaagaaagatgagaatcaacaggatgaggttgcatcaactgacgaatcacag600
tccactatcaatctcaattctgataaaaatgagcttgcaccatcagatagaacatcctaa660
<210>4
<211>219
<212>prt
<213>glycinemax
<400>4
metcysvalasptrpaspthrvalleuhisvalasnglngluilemet
151015
thrserthrhisaspleuseraspasnglualaaspaspglnglngln
202530
sergluserglnmetgluproleuseralaasnglyilesertyrala
354045
glyilealathrglnasnvalglntyralathrproserglnleugly
505560
thrglyhisalavalvalproprothrtyrprotyrproaspprotyr
65707580
tyrargserilephealaprotyraspalaglnthrtyrproprogln
859095
protyrglyglyasnprometvalhisleuglnleumetglyilegln
100105110
glnalaglyvalproleuprothraspthrvalglugluprovalphe
115120125
valasnalalysglntyrhisglyileleuargargargglnserarg
130135140
alalysalagluserglulyslysalaalaargasnarglysprotyr
145150155160
leuhisgluserarghisleuhisalaleuargargalaargglycys
165170175
glyglyargpheleuasnserlyslysaspgluasnglnglnaspglu
180185190
valalaserthraspgluserglnserthrileasnleuasnserasp
195200205
lysasngluleualaproseraspargthrser
210215
- 还没有人留言评论。精彩留言会获得点赞!