一种用于肿瘤基因变异检测的方法、引物、探针和检测剂与流程
本发明属于分子生物学领域,涉及医学及生物技术,具体地涉及一种用于肿瘤基因变异检测的方法、引物、探针和检测剂。
背景技术:
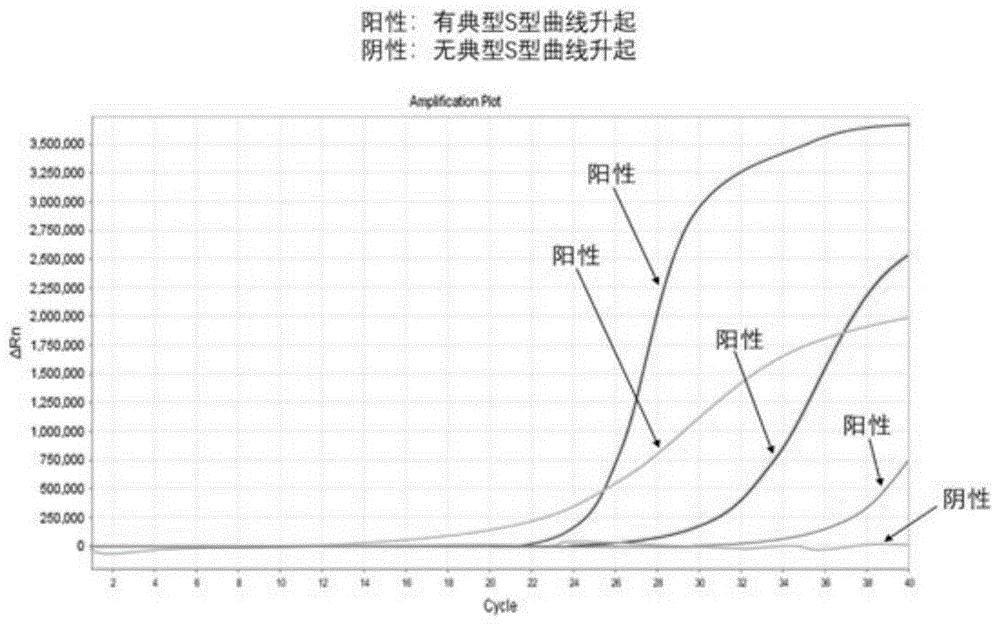
:肿瘤是一种高发病率和高死亡率的疾病,严重危害公众健康。近年来,临床医生在肿瘤治疗中发现,人体肿瘤千差万别,即使是同一个部位的肿瘤,治疗效果和方法也应因人而异。因此在癌症治疗过程中,只有同病异治,因人而异,实施个体化治疗,才能针对不同类型的病人选择合适他们的药物。随着基因分子水平研究的不断深入,越来越多的肿瘤细胞信号通路被发现,大量临床研究表明,通路中的特定基因的突变/扩增/表达状态与靶向、化疗药物的有效性密切相关。因此,临床上检测这些通路中肿瘤相关基因的突变、扩增、表达情况,能针对性地为每位患者“量身定做一套最适合的治疗方案,从而最大程度地提高治疗的有效率,减少药物的毒副作用,避免用药不当贻误治疗时机。在肿瘤临床治疗中,化疗总体有效率仅占30-40%,而通过基因检测筛选出获益病人,有效率可以提高到80%。分子检测为癌症治疗模式带来了翻天覆地的变化,肿瘤治疗开始迈入个性化治疗的新天地。目前,fda(美国食品药品监督管理局)已经强制要求用药前进行egfr、kras等基因检测。此外,nccn(美国癌症综合治疗网络)已经将egfr、kras、braf、her2等基因检测纳入到癌症治疗指南中。肿瘤基因检测主要针对一些实体瘤,如肺癌、乳腺癌、胃癌、食管癌、结直肠癌、肝癌、胆管胆囊癌、胰腺癌、胃肠道间质瘤、头颈部癌、宫颈癌、卵巢癌、子宫内膜癌、前列腺癌、膀胱癌、肾癌、黑色素瘤等。目前,肿瘤基因的检测有的采用第二代高通量基因测序技术对所述文库分子进行测序,例如申请号为cn201810069455.7的专利文献:kras基因检测引物组、试剂盒及检测方法;有的采用数字pcr方法,例如申请号为cn201711128933.9的专利文献:eml4-alk融合基因无创检测试剂盒;有的是基于阴离子卟啉-碳纳米管修饰电极检测her2基因特定序列的方法及应用(cn201810258860.3),还有的是采用一种放射性c-met靶向亲和小分子化合物及其应用(cn201610402431.x)。这些方法不是操作繁琐、对仪器要求高、价格贵、耗费时间长,就是灵敏度欠缺、定性判读主观、不准确。除了以上检测方法,目前肿瘤基因检测主要技术是实时荧光定量pcr,它具有准确率高、特异性强、灵敏度高、操作简单、应用范围广、高通量等特点。目前国内市场已获批上市的有关egfr、kras、braf、nras、pik3ca、alk、ros1等基因变异检测试剂盒,几乎都是采用arms-pcr检测方法。传统的arms-pcr检测原理是将突变位点置于引物的3′端,利用耐热taqdna聚合酶缺乏3′→5′外切校正活性的特点,引物3′端的特异碱基分别互补于突变型等位基因的相对碱基,若此碱基对形成错配,链延伸反应就会因3′-5′-磷酸二酯键形成障碍而受阻,导致野生型模板阻碍,突变型引物放大,从而导致突变型扩增较强、野生型扩增较弱。通过比较野生型和突变型扩增曲线的ct差值来判断阴阳性,而ct值通过人为调整基线(threshold)来确定,然而野生型和突变型扩增曲线往往是不平行的,基线调整值不同则会导致ct差值的不同。因此,arms-pcr检测方法的结果判读的主观性太强,很容易造成人为误判,尤其是针对弱阳性样本,误判可能性极大。此外,传统的armspcr不能避免核酸抽提差异造成的外控和样本dna在基因组dna中的比例发生变化而导致的结果误判。除了在结果判读上缺乏客观、准确性,由于引物设计原理的局限性,传统的armspcr针对一些特殊基因变异的检测缺乏可行性,例如:大片段缺失(一般>100bp)、短片段(一般为8-20bp)重复性插入、相邻突变位点的顺式和反式突变(egfr-t790m/c797s)。近年来一些临床研究发现,基因突变也存在大片段碱基缺失(一般>100bp),如:乳腺癌相关基因brca1/2大片段缺失。由于检测原理的局限性,传统的armspcr无法对该类型的基因突变进行检测。此外,ngs测序方法由于捕获片段大多在100-200bp以内,也很难检测该类型的基因突变。目前brca1/2基因突变检测的中国专家共识中,指出ngs也不能作为brca1/2大片段缺失的最终确诊方法。经ngs检出的brca1/2大片段缺失样本仍需要多重连接探针扩增技术(multiplexligation-dependentprobeamplification,mlpa)方法进行确认。在非小细胞肺癌(nsclc),her2基因突变的发生率约占2-4%,在egfr/kras/alk阴性的nsclc中,her2基因突变发生率约占6%。her2基因突变主要发生于20外显子插入突变,该类型的突变占nsclc中her2基因突变的83-100%。her2基因20外显子插入突变主要包括一些短片段(一般为8-20bp)重复性插入,例如:her2-c.2324_2325insatacgtgatggc、her2-c.2325_2326instacgtgatggct、her2-c.2322_2323insgcatacgtgatg、her2-c.2340_2341insggctcccca。此外,egfr-20ins也是nsclc重要的突变用药靶点,该突变也包括短片段重复性插入(egfr-c.2307_2308insgccagcgtg)。这些短片段重复性插入类型的基因突变不能通过armspcr引物设计原理达到检测目的,目前只能采用ngs测序方法进行检测,众所周知,短片段重复性插入突变并不是ngs检测所擅长的。奥希替尼是第三代egfr-tki,治疗一代egfr-tki耐药的egfr-t790m阳性非小细胞肺癌(nsclc)疗效非常显著。但奥希替尼本身也会出现耐药,其耐药时21%患者出现c797s突变,绝大多数c797s阳性患者,t790m突变仍保持阳性,即t790m/c797s双突变阳性。而且绝大多数表现为t790m/c797s为双突变还分为顺式突变,t790m/c797s和反式突变只占10%。目前的研究显示,虽然t790m/c797s顺式突变对靶向药物的疗效都欠佳缺乏有效的治疗药物,但t790m/c797s反式突变的耐药可以被可采用第一代和第三代egfr-tki联合治疗所克服,疗效显著。因此,在检测出t790m/c797s突变的同时,还能判断出其是顺式或反式突变对临床医生决定治疗药物非常有用。目前临床上采用的arms-pcr方法并不能检测区分t790m/c797s的顺式及反式突变,故只能多采用ngs测序方法进行顺式或反式的区分,耗时长、费用贵。技术实现要素:本发明的目的是克服现有技术的缺陷,提供一种用于肿瘤基因变异检测的方法,该方法可检测的突变类型多样,定性客观准确,且灵敏度高。为了达到上述目的,本发明提供的用于肿瘤基因变异检测的方法,其包含以下步骤:(1)设计一对靶序列特异性扩增引物,用于扩增含有突变碱基的核酸片段;(2)设计能够与所述突变碱基配对的dna探针,该dna探针上带有在所述核酸片段扩增过程中能发荧光的修饰基团;(3)在所述引物和/或dna探针的任意位置引入颈环结构,所述颈环结构为2-12个不与所述核酸片段配对的核苷酸,在所述引物和/或dna探针与所述核酸片段结合时,所述颈环结构凸起形成环状或呈单链状态;(4)利用所述引物对所述核酸片段进行pcr扩增,并通过所述dna探针收集荧光数据。较佳地,步骤(2)中,所述dna探针为taqman探针。较佳地,步骤(3)中,所述颈环结构为1个或2个以上。较佳地,步骤(4)中,采用下列反应体系中的任意一种,对含有所述突变碱基的核酸片段进行pcr扩增,并收集荧光数据;a.以样本dna为模板的pcr反应体系:b.以样本rna为模板的pcr反应体系:较佳地,上述的以样本dna为模板的pcr反应体系的反应条件为:50℃ung酶消化2分钟,1个循环;95℃预变性2分钟,1个循环;95℃变性10秒,60℃退火及延伸40秒,40个循环;在60℃时收集荧光信号;上述的以样本rna为模板的pcr反应体系的反应条件为:50℃逆转录25分钟,1个循环;95℃预变性2分钟,1个循环;95℃变性10秒,60℃退火及延伸40秒,40个循环;在60℃时收集荧光信号。本发明还提供了一种用于肿瘤基因变异检测的检测剂,其包含:缓冲液、mg2+、dntps、pcr反应所需的酶、及引物和探针;pcr反应所需的酶包含taq酶和ung酶;该检测剂中的引物和探针选自上述的引物和探针。本发明还提供了一种用于肿瘤基因变异检测的引物,所述引物的任意位置设有上述的用于肿瘤基因变异检测的方法所采用的颈环结构。本发明还提供了一种用于肿瘤基因变异检测的dna探针,所述dna探针的任意位置设有上述的用于肿瘤基因变异检测的方法所采用的颈环结构。本发明具有以下有益效果:(1)本发明的方法根据荧光pcr扩增中扩增曲线的有无判断结果,避免了传统armspcr检测方法在结果判读中极易造成的人为误判,其结果判读具有客观性,在检测结果的准确性方面远胜于armspcr方法。(2)本发明的方法在肿瘤基因检测应用范围上优于armspcr,能应用于检测大片段缺失(一般>100bp)、短片段(一般为8-20bp)重复性插入、相邻突变位点的顺式和反式突变等各种基因变异类型的检测。(3)本发明的方法在操作难易程度、检测时长、检测成本方面优于一代测序、ngs、数字pcr等方法。附图说明图1为本发明的masspcr技术的数据判读的示例。图2为利用本发明的masspcr技术进行点突变检测的技术原理的示意图。图3为利用armspcr技术进行点突变检测的技术原理的示意图。图4为armspcr结果判读的理想化情况的示例。图5为armspcr中当外控扩增曲线和样本扩增曲线不平行导致人为误判的示例。图6为armspcr中当外控扩增片段和突变扩增片段在样本中的比例发生改变导致人为误判的示例。图7为实施例1的灵敏度检测结果图。图8为实施例2的灵敏度检测结果图。图9为实施例3的灵敏度检测结果图。图10为实施例4的灵敏度检测结果图。具体实施方式以下结合具体实施例,对本发明进一步阐述。应理解,这些实施例仅用于本发明而不用于限制本发明的范围。除非另有定义或说明,本专利所述的科学术语与本领域普通技术人员所理解具有相同的含义。本发明提供的肿瘤基因变异检测技术,命名为突变选择特异性扩增系统(mutation-selectedamplificationspecificsystem,mass)pcr技术。masspcr技术通过独特的引物设计,根据突变位点的碱基序列特征在引物或探针的两端或中间位置设计颈环结构(即2-12个核苷酸不与互补链配对而呈单链状态,从而凸起形成环状结构),并在探针的两端或中间增加修饰基团,从而大大增加突变型和野生型的退火温度差,通过控制退火温度,使引物或荧光探针只针对突变型扩增,完全消除野生型扩增,最后通过扩增曲线的有无来进行结果判读,将突变型和野生型的扩增结果从根本上区别开来,判断结果具有客观性。本方法针对颈环结构的设计位置和修饰基团的设计位置并不唯一,需综合考虑引物或探针本身的碱基序列、gc含量、tm值,使之针对突变型和野生型的结合差异尽可能的拉大,从而达到区分突变型和野生型的目的。引物上的颈环结构增加了引物的tm值,直接影响pcr反应中的退火温度,探针上的颈环结构也是为了增加探针的特异性。这些颈环结构的设计使得pcr反应的特异性很高,可以高敏感性地在野生型dna背景中检测出突变型dna。本技术适用于各种肿瘤基因变异的检测,应用范围广。本发明提供的肿瘤基因变异检测技术包含以下步骤:(1)采用常规方法设计一对靶序列特异性扩增引物,用于扩增含有突变碱基的核酸片段;核酸片段中碱基的突变类型可以是大片段缺失(一般>100bp)、短片段(一般为8-20bp)重复性插入、相邻突变位点的顺式和反式突变(例如非小细胞肺癌中egfr的t790m/c797s突变)等。(2)设计能够与所述突变碱基配对的dna探针,该dna探针选择taqman探针,其两端或中间带有在所述核酸片段扩增过程中能发荧光的修饰基团。(3)在所述引物和/或dna探针的任意位置引入颈环结构,所述颈环结构为2-12个不与所述核酸片段配对的核苷酸,在所述引物和/或dna探针与所述核酸片段结合时,所述颈环结构凸起形成环状或呈单链状态;颈环结构的数量可以是1个或2个,或者是多个。颈环结构增加了退火温度,加大了了突变型和野生型样本的退火温度差,通过控制退火温度,并以未发生突变的野生型样本作为对照,根据荧光pcr过程中扩增曲线的有无,可判断引物或dna探针是否只针对突变型样本进行扩增。(4)利用所述引物和dna探针,对样品进行pcr扩增,并收集荧光数据,当含有突变碱基的核酸片段检测到荧光数据,而未发生突变的野生型样本的核酸片段未检测到荧光数据,则检测出肿瘤基因发生变异。上述步骤(4)中,首次利用所述引物和dna探针进行肿瘤基因变异检测时,需要对设计得到的引物和dna探针进行实验验证,以确认其可用。例如,对pcr扩增后的产物进行检测,以确认产物中为目的片段,而不是非特异性扩增获得的产物。在肿瘤等位基因点突变应用中,masspcr技术基于taqman探针法,通过独特的引物设计,使引物或探针在pcr过程中特异性地结合突变dna模板而不结合野生型dna模板,从而使突变型扩增结果产生荧光信号(有典型s型曲线升起),而野生型扩增结果不产生荧光信号(无典型s型曲线升起),最终判读结果为突变型扩增结果有ct值,而野生型扩增结果无ct值。masspcr技术的数据判读的示例如图1所示。如图2所示,为利用masspcr技术进行点突变检测的技术原理的示意图。图2的(a)中,根据masspcr技术原理设计的探针与突变dna模板匹配,因此探针能够被切断,从而产生荧光信号。图2的(a’)中,探针与野生型dna模板不匹配,因此探针不能够被切断,无荧光信号产生。目前肿瘤基因变异的检测方法常用的有armspcr检测方法。如图3所示,为armspcr技术原理的示意图。如图3的(b)所示,传统armspcr检测方法将突变位点设在引物的3'端,使引物序列跟突变型完全配对,突变型pcr扩增效率较好,荧光较强;而如图3的(b’)所示,引物跟野生型部分碱基不配对,结果导致野生型pcr扩增效率较差,荧光较弱。传统armspcr检测方法就是使引物序列跟突变型完全配对而跟野生型部分碱基不配对,结果导致突变型pcr扩增效率较好而野生型pcr扩增效率较差,从而导致突变型和野生型的pcr扩增曲线ct值产生差异,再以最理想化(相当于100%突变频率)的扩增结果(外控)为参照,将实际样本的扩增曲线ct值与外控ct值进行比较(δct=外控ct-样本ct),最终根据δct的大小来区别突变型和野生型。armspcr结果判读的理想化情况如图4所示。基于传统armspcr检测原理,其扩增灵敏度不高,而且在实际情况下很容易对扩增结果产生误判,例如:当外控扩增曲线和样本扩增曲线不平行的时候,调节扩增曲线基线值的高低不同则导致δct不同,从而很可能导致人为误判(如图5所示);当制备的dna样本质量不理想的时候,部分核酸片段损失较大,造成外控扩增片段和突变扩增片段在样本中的比例发生改变,导致扩增结果的δct发生改变,从而导致检测结果的误判(如图6所示)。此外,传统armspcr不适用于检测大片段缺失(一般>100bp)、短片段(一般为8-20bp)重复性插入、相邻突变位点的顺式和反式突变(egfr-t790m/c797s),其应用范围较为局限。masspcr技术将突变型和野生型pcr扩增曲线的差异直接升华为有跟无的区别,打破了传统armspcr通过比较δct来区别突变型和野生型的缺陷,避免了主观性的人为误判;并且masspcr扩增结果为野生型扩增结果不产生荧光信号(扩增曲线无ct值),不比较δct,即使制备的dna样本质量不理想导致外控扩增片段和突变扩增片段在样本中的比例发生改变,因masspcr技术只涉及突变扩增片段而无需设置外控,只需要看样本扩增结果是否产生荧光信号(有无典型s型曲线升起),从而避免了dna抽提质量差异导致的结果误判。此外,masspcr技术还能应用于检测大片段缺失(一般>100bp)、短片段(一般为8-20bp)重复性插入、相邻突变位点的顺式和反式突变(egfr-t790m/c797s)。根据基因突变类型的不同,在引物或探针的不同位置设计颈环结构,在探针的两端或中间增加修饰基团,从而使该方法可应用于各种基因变异类型的检测。以下选用突变临床组织样本为阳性样本,样本通过一代测序/ngs/qpcr等方法鉴定为明确突变阳性;同时,以野生型组织样本为对照,利用本发明的检测肿瘤基因变异的方法进行检测。突变由fam信号指示,内控由vic信号指示,内控作为结果判读的一部分,选择的是人类β-actin基因相对保守的区段,用于监控样本核酸质量和pcr反应过程。下列引物或探针序列下方的下划线表示设计的颈环结构。实施例1针对braf基因的v600e突变位点进行检测,实施例1的样品为点突变。采用的引物序列如下所示:primer-f1:cttcatgaagacctcacagtaaaaataggprimer-r1:acaaaatggatccagacaactgttc采用的探针序列如下所示:primer-p1:fam-tctaggaacagagaacc-mgb。检测步骤如下:(1)阳性和野生型组织样本dna/rna的提取:采用allprepdna/rna/mirnauniversalkit(qiagencat.no.80224)进行提取,具体提取操作步骤按试剂盒说明书操作。提取的dna稀释到5ng/μl,rna稀释到10ng/μl。(2)将已确定没有发生突变的野生型样本作为阴性对照。(3)按照表1的dnapcr反应体系和表2的rnapcr反应体系配制pcr反应液:表1dnapcr反应体系表2rnapcr反应体系名称终浓度10xbuffer1xbuffermg2+300μmdntps20μmtaq酶3uung酶0.2u逆转录酶30u各突变引物0.4μm各突变探针0.2μm样本rna100ng补纯化水至25μl(4)将配好的反应液加入乳白色8联管,然后放入荧光定量pcr仪(7300plus)。(5)按照以下dna/rna扩增程序分别设置扩增程序,然后开始荧光pcr扩增:dna扩增程序:50℃反应2分钟,1个循环;95℃预变性2分钟,1个循环;95℃变性10秒,60℃退火及延伸40秒,40个循环。在60℃时收集fam和vic荧光信号。rna扩增程序:50℃逆转录25分钟,1个循环;95℃预变性2分钟,1个循环;95℃变性10秒,60℃退火及延伸40秒,40个循环。在60℃时收集fam和vic荧光信号。(6)结果处理a.ct值的确定:确认未选择校正荧光参照,需要同时选择阴性对照反应管和样品反应管;手动设置荧光本底信号(baseline),即样本的荧光背景值和阴性对照的荧光值,根据样本的荧光值曲线,选择进入指数期之前背景荧光值达到平稳期的循环数区域;手动设置荧光信号阈值(threshold),原则要大于样本的荧光背景值和阴性对照的荧光最高值,同时要尽量选择进入指数期的最初阶段,根据实际情况确定扩增曲线升起的拐点处,得到ct值。b.阴性结果:ct值确定以后的检测结果为fam信号无ct值,vic信号有ct值。c.阳性结果:ct值确定以后的检测结果为fam信号有ct值,vic信号有ct值。实验结果:实施例1抽提到的dna样品和rna样品的基因突变均能够被检测出,与一代测序结果一致。灵敏度分析:通过数字pcr确定突变型组织样本的突变频率,取同一份野生型组织样本对突变型组织样本进行不同梯度的稀释,每次反应加入10ngdna模板进行扩增反应。结果表明,实施例1的检测灵敏度能够达到2.5%,即目标基因突变频率为2.5%及以上的样本都能够被检测到(请参阅图7)。实施例2针对egfr基因的v769_d770insasv突变位点进行检测。实施例2的样品突变类型为短片段重复性插入。采用的引物序列如下所示:primer-f2:gccacactgacgtgcctctcprimer-r2:gtccaggaggcagccgaagg采用的探针序列如下所示:primer-p2:fam-ccagcgacagcagcgtg-bhq。检测步骤参照实施例1。实施例2的样品检测结果与一代测序结果一致。实施例2的检测灵敏度能够达到2.0%(请参阅图8)。实施例3针对her2基因的a775_g776insyvma突变位点进行检测。实施例3的样品突变类型为短片段重复性插入。采用的引物序列如下所示:primer-f3:cgtgatggcatacgtgatggcprimer-r3:ccagctgcaccgtggatgtcag采用的探针序列如下所示:primer-p3:fam-ctctatgtctcccgccttctg-bhq。检测步骤参照实施例1。实施例3的样品检测结果与一代测序结果一致。实施例3的检测灵敏度能够达到0.8%(请参阅图9)。实施例4针对brca2基因的asp23_leu105del突变位点进行检测。实施例4的样品突变类型为大片段缺失。采用的引物序列如下所示:primer-f4:gaatggattaatgatcttgtttaaprimer-r4:cctatgagacattattttcatcgtctcca采用的探针序列如下所示:primer-p4:fam-ttttaaatagatatctaatgc-bhq。检测步骤参照实施例1。实施例4的样品检测结果与一代测序结果一致。实施例4的检测灵敏度能够达到0.8%(请参阅图10)。检测结果表明,本发明的检测方法可用于肿瘤基因各种变异类型的基因检测,且检测灵敏度可达到0.8%。综上所述,本发明的用于肿瘤基因变异检测的方法通过在引物和/或dna探针的任意位置引入颈环结构,使引物或探针只针对突变型样品进行扩增,对肿瘤基因变异的定性客观、准确,并且检测灵敏度高;本发明的检测系统并不仅限于对所举例的基因突变类型的检测,所有已知的肿瘤基因突变类型均可用本发明的方法进行检测。尽管本发明的内容已经通过上述优选实施例作了详细介绍,但应当认识到上述的描述不应被认为是对本发明的限制。在本领域技术人员阅读了上述内容后,对于本发明的多种修改和替代都将是显而易见的。因此,本发明的保护范围应由所附的权利要求来限定。序列表<110>上海桐树生物科技有限公司<120>一种用于肿瘤基因变异检测的方法、引物、探针和检测剂<160>12<170>siposequencelisting1.0<210>1<211>29<212>dna<213>人工序列(artificialsequence)<400>1cttcatgaagacctcacagtaaaaatagg29<210>2<211>25<212>dna<213>人工序列(artificialsequence)<400>2acaaaatggatccagacaactgttc25<210>3<211>17<212>dna<213>人工序列(artificialsequence)<400>3tctaggaacagagaacc17<210>4<211>20<212>dna<213>人工序列(artificialsequence)<400>4gccacactgacgtgcctctc20<210>5<211>20<212>dna<213>人工序列(artificialsequence)<400>5gtccaggaggcagccgaagg20<210>6<211>17<212>dna<213>人工序列(artificialsequence)<400>6ccagcgacagcagcgtg17<210>7<211>21<212>dna<213>人工序列(artificialsequence)<400>7cgtgatggcatacgtgatggc21<210>8<211>22<212>dna<213>人工序列(artificialsequence)<400>8ccagctgcaccgtggatgtcag22<210>9<211>21<212>dna<213>人工序列(artificialsequence)<400>9ctctatgtctcccgccttctg21<210>10<211>24<212>dna<213>人工序列(artificialsequence)<400>10gaatggattaatgatcttgtttaa24<210>11<211>29<212>dna<213>人工序列(artificialsequence)<400>11cctatgagacattattttcatcgtctcca29<210>12<211>21<212>dna<213>人工序列(artificialsequence)<400>12ttttaaatagatatctaatgc21当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1