一种过表达羟酸还原异构酶的葡萄汁酵母菌株及其应用的制作方法
技术领域:
本发明属于生物工程和基因工程技术领域,涉及工业微生物的育种及其应用,具体是涉及一种低产双乙酰、过表达羟酸还原异构酶的葡萄汁酵母菌株及其在制备葡萄酒中的应用。
背景技术:
:
葡萄酒是世界上最古老的具有浓厚文化底蕴的绿色保健饮料,也是现代生活中不可或缺的饮品。葡萄酒中的香气成分复杂多样,相对含量从每升几纳克到几百毫克不等,在葡萄酒的呈香过程中起到重要作用。双乙酰是葡萄酒中重要的风味物质,适量的双乙酰,会增加葡萄酒的香气,改善葡萄酒的风味,而当双乙酰含量过高时,就可能产生令人生厌的“馊饭味”,葡萄酒表现变质迹象。因此,葡萄酒中的双乙酰的含量必须控制在合适的范围内。
目前对于双乙酰调控的研究主要集中在啤酒发酵过程中。刘光诚等人发明了七日酿酒法,即在糖化和发酵过程中加入具有超强的电子还原性和离子交换功能的陨水速成剂,大幅降低了双乙酰指标,改善啤酒的品质(中国专利,cn107619733a,2018-01-23)。赵云财等人制作一种富含α-氨基氮的啤酒专用麦芽糖浆,并用该麦芽糖浆作辅料生产啤酒,可以大幅度降低双乙酰含量,提高啤酒的非生物稳定性(中国专利,cn103571672a,2014-02-12)。李红等人采用含有适当浓度d-缬氨酸的13°p麦汁在适当条件下驯化酵母,从酵母泥中筛选出对d-val敏感的菌株并进行发酵实验,检测敏感菌株双乙酰含量,其降低率可达到34.08%(中国专利,cn104877922a,2015-09-02)。但上述工艺和菌株主要用于啤酒上,并未将其应用于葡萄酒,而且菌株通过筛选突变而来,重复性差。
基因ilv5编码羟酸还原异构酶,是支链氨基酸代谢合成途径的相关蛋白,目前对它的研究主要集中在该基因对高级醇的影响,大部分研究认为羟酸还原异构酶会提高高级醇中的含量(低产高级醇酿酒酵母突变菌株的差异蛋白组分析及高级醇合成相关蛋白的差异表达,王国正等,食品与发酵工业,2003-02-28)。也观察到羟酸还原异构酶对联二酮的前驱体物质的影响,经该酶作用后啤酒的风味和发酵度都很好(通过啤酒酵母控制联二酮含量ilv5型基因和ilv3型基因的扩增对联二酮含量和ilv酶活性的影响,祁明霞等,啤酒科技,2002-07-15)。然而这些基因对双乙酰,尤其是葡萄酒中的双乙酰的影响未见报道。
在酿酒过程中酿酒酵母(saccharomycescerevisiae)是传统普遍应用的酵母菌种,但研究发现,在现代葡萄酒发酵中的非酿酒酵母数量较高(每毫升106-108个细胞数量)且持续存在,其与酿酒酵母一样在产生酒精的同时对葡萄酒的风味和香气等感官特征有着重要的影响,这导致人们又重新看待这些酵母在酿酒中的作用。随着分子生物学技术的发展,利用基因工程改造非酿酒酵母获得具有新的理想特征的各种酵母菌种,酿造具有复杂口感、风格独特的葡萄酒将会成为现代葡萄酒产业发展的重要方向。
技术实现要素:
:
本申请发现,葡萄汁酵母(saccharomycesuvarum)是一种具有潜在的酿酒特性的非酿酒酵母,其在高产甘油和低产乙醇的同时,也能比酿酒酵母产生更多的芳香物质。利用分子育种技术构建调控双乙酰葡萄汁酵母工业菌株,对提高葡萄酒的风味质量具有重要意义。因此,
本发明的第一方面,提供一种低产双乙酰的葡萄汁酵母(saccharomycesuvarum)菌株,所述菌株过表达羟酸还原异构酶。
优选的,所述菌株还部分敲除编码α-乙酰乳酸合成酶的ilv2基因,优选的,所述菌株还低产高级醇。
更优选的,所述菌株还过表达二羟异戊酸脱水酶。
更优选的,所述菌株过表达羟酸还原异构酶和部分敲除编码α-乙酰乳酸合成酶的ilv2基因,或者,所述菌株过表达羟酸还原异构酶和二羟异戊酸脱水酶。
更优选的,所述菌株过表达二羟异戊酸脱水酶、羟酸还原异构酶和部分敲除编码的α-乙酰乳酸合成酶的ilv2基因。
优选的,所述ilv2基因部分敲除为至少敲除一个等位基因,例如,敲除一个等位基因,敲除两个等位基因,敲除三个等位基因。
所述葡萄汁酵母是在出发葡萄汁酵母菌株中部分敲除编码α-乙酰乳酸合成酶的ilv2基因,同时过表达分别由ilv3和ilv5编码的二羟异戊酸脱水酶和羟酸还原异构酶所得,该菌株低产双乙酰和高级醇。
所述ilv2基因其geneid为:855135,核苷酸序列如表中seqno:1所示;所述ilv3基因其geneid为:853473,核苷酸序列如表中seqno:2所示;所述ilv5基因其geneid为:851069,核苷酸序列如表中seqno:3所示。所述启动子pgk1其geneid为:850370,核苷酸序列如表中seqidno:4所示。
优选的,所述出发酵母菌株为葡萄汁酵母(saccharomycesuvarum)cicc1465。
本发明还提供一种低产双乙酰的葡萄汁酵母菌株的构建方法,包括如下步骤:
1)过表达ilv5的重组菌株的构建;优选的,构建ilv5基因多拷贝表达载体构建过表达ilv5的重组菌株;
或者,所述构建方法还包括,
2)ilv2基因的部分敲除菌株的构建;优选的,采用多重基因中断技术阻碍α-乙酰乳酸合成酶的表达构建ilv2基因的部分敲除菌株;
和/或,
3)过表达ilv3的重组菌株的构建;优选的,构建ilv3基因多拷贝表达载体构建过表达ilv3的重组菌株。
更优选的,所述构建方法包括:
1):将基因ilv3和/或ilv5通过同源重组原理分别整合至质粒上构建获得携带基因ilv3、ilv5、或者,ilv3和ilv5的重组质粒;
2):ilv2基因的部分敲除菌株的构建:采用缩进式的引物设计方法,pcr扩增筛选标记基因,结合多重基因中断技术将标记基因整合至ilv2的等位基因,且应用cre重组酶去除,构建部分敲除ilv2等位基因的菌株,优选的,所述部分敲除ilv2等位基因包括敲除一个、两个或者三个ilv2等位基因;
3):通过醋酸锂转化法将步骤1)的重组质粒分别导入出发菌株得到过表达ilv3基因的重组菌株、过表达ilv5基因的重组菌株,或者,同时过表达ilv3和ilv5基因的重组菌株;
4):将步骤1)的重组质粒分别导入步骤2)中敲除ilv2等位基因的菌株中,得到部分敲除ilv2基因且过表达ilv3基因的重组菌株、部分敲除ilv2基因且过表达ilv5基因的重组菌株和部分敲除ilv2基因且同时过表达ilv3和ilv5基因的重组菌株。
更优选的,所述构建方法包括:
1):将启动子基因pgk1连接于质粒yep352的bamhi和sali酶切位点处,构建质粒yep-p,将基因片段ilv3和ilv5通过同源重组原理分别整合至质粒yep-p上基因pgk1的xhoi位点构建质粒yep-p3和yep-p5,将质粒yep-p3上的pgkp-ilv3-pgkt基因片段和质粒yep-p5分别通过smai酶切而连接构建质粒yep-p35,将基因片段kanmx通过同源重组原理分别整合至质粒yep-p3、yep-p5和yep-p35的apai位点构建重组质粒yep-kp3、yep-kp5和yep-kp35;
2):采用缩进式的引物设计方法,以质粒puc6为模板,pcr扩增筛选标记kanmx基因,结合多重基因中断技术将kanmx基因应用醋酸锂转化法依次整合至ilv2的第1、2、3条等位基因,且应用cre重组酶去除,构建敲除ilv2一条等位基因的菌株v21,敲除ilv2两条等位基因的菌株v22,和敲除ilv2三条等位基因的菌株v23;
3):通过醋酸锂转化法将步骤1)的重组质粒yep-kp3、yep-kp5和yep-kp35分别导入出发菌株得到过表达ilv3基因的重组菌株wy-3、过表达ilv5基因的重组菌株wy-5和同时过表达ilv3和ilv5基因的重组菌株wy-35;
4):将步骤1)的重组质粒yep-kp3、yep-kp5和yep-kp35分别导入步骤2)中敲除ilv2三条等位基因的菌株中,得到部分敲除ilv2基因且过表达ilv3基因的重组菌株v23-3、部分敲除ilv2基因且过表达ilv5基因的重组菌株v23-5和部分敲除ilv2基因且同时过表达ilv3和ilv5基因的重组菌株v23-35。
更具体的,所述构建方法包括:
(1)过表达ilv3和/或ilv5的重组菌株的构建
①以质粒ppgk1为模板,pcr扩增pgk1基因片段,将质粒yep352和片段pgk1同时进行bamhi和sali双酶切后进行连接,构建质粒yep-p。
②以出发酵母菌株基因组为模板,分别pcr扩增ilv3和ilv5基因,用限制性内切酶xhoi对质粒yep-p进行酶切,将基因片段ilv3和ilv5分别与质粒yep-p连接,构建质粒yep-p3和yep-p5。
③以质粒yep-p3为模板,pcr扩增pgk1p-ilv3-pgk1t片段。用限制性内切酶smai同时对质粒yep-p5和pgk1p-ilv3-pgk1t进行酶切,并将两者连接,构建质粒yep-p35。
④以质粒pug6为模板,pcr扩增kanmx基因,用限制性内切酶apai分别对质粒yep-p3、yep-p5和yep-p35进行酶切后,分别与kanmx基因片段进行连接,构建质粒yep-kp3、yep-kp5和yep-kp35。
⑤将质粒yep-kp3、yep-kp5和yep-kp35分别导入出发菌株中,得到过表达ilv3基因的重组菌株wy-3、过表达ilv5基因的重组菌株wy-5和同时过表达ilv3和ilv5基因的重组菌株wy-35
(2)ilv2基因的部分敲除菌株的构建
①以质粒puc6为模板,用带有ilv2上下游60bp同源序列的引物pcr扩增kanmx基因;
②将步骤(2)-①得到的pcr产物导入出发菌株中,获得敲除ilv2一条等位基因的重组菌株v21
③用pgapza质粒去除步骤(2)-②获得菌株中的kanmx基因,获得不含kanmx基因的重组菌株v21’;
④采用缩进式的引物设计方法,以质粒puc6为模板,pcr扩增kanmx基因,结合多重基因中断技术敲除葡萄汁酵母v21’基因组上ilv2基因的第二个等位基因,获得重组菌株v22;
⑤用pgapza质粒去除步骤(2)-④获得菌株中的kanmx基因,获得不含kanmx基因的重组菌株v22’,结合方法(2)-④敲除ilv2基因的第三个等位基因获得重组菌株v23;
⑥用pgapza质粒去除步骤(2)-⑤获得菌株v23中的kanmx基因,获得不含kanmx基因的重组菌株v23’。
(3)ilv2基因的部分敲除同时过表达ilv3或/和ilv5基因菌株的构建
将步骤(1)-④得到的质粒yep-kp3、yep-kp5和yep-kp35分别导入步骤(2)-⑥所得重组菌株v23’中,获得部分敲除ilv2基因且同时过表达ilv3基因的重组菌株v23-3、部分敲除ilv2基因且同时过表达ilv5基因的重组菌株v23-5和部分敲除ilv2基因且同时过表达ilv3和ilv5基因的重组菌株v23-35。
本发明还提供一种上述菌株在制备葡萄酒中的应用。
优选的,所述葡萄酒为低含量双乙酰,或,低含量双乙酰和高级醇的葡萄酒。
更优选的,所述葡萄酒的双乙酰含量低于2.2mg/l,进一步优选的,所述葡萄酒的双乙酰含量低于1.5mg/l、2mg/l,最为优选的,所述葡萄酒的双乙酰含量低于1.0mg/l。
优选的,所述葡萄酒的高级醇含量低于290mg/l,进一步优选的,所述葡萄酒的高级醇含量低于260mg/l,最为优选的,所述葡萄酒的高级醇含量低于240mg/l。
葡萄酒发酵:葡萄经过筛选、除梗和压榨后,将葡萄汁调至糖浓度21brix,ph3.5,且so2添加量为60-120mg/l,量取调配好的的葡萄汁于三角瓶进行发酵,接菌量为5×107-5×108cfu/ml,25℃培养发酵。
所述高级醇俗称杂醇油,是指具有3个及3个以上碳链骨架的一价醇类的统称。本文测定葡萄酒中的主要高级醇包括异丁醇、异戊醇和活性戊醇。
本发明的有益效果:
1、本发明提供的低产双乙酰含量的葡萄汁酵母在保持良好发酵性能的前提下,单独过表达ilv5基因编码的羟酸还原异构酶,可以降低双乙酰的含量,如实施例所示,重组菌株wy-5发酵后双乙酰含量为2.17mg/l,相对于出发菌株降低了16.86%。
2、本发明所述的葡萄汁酵母重组菌株对降低双乙酰和高级醇都有较好的效果,在其它发酵性能不受影响的情况下,葡萄酒发酵5天后,测得各实施例的重组菌株双乙酰含量在0.85-2.17mg/l之间,最优技术方案中,双乙酰含量为0.85mg/l,与出发菌株相比降低了67.55%,且发酵后异丁醇分在30.81-55.17mg/l之间,异戊醇含量在172.84-213.12mg/l之间,最优技术方案中,异丁醇和异戊醇含量分别为31.08mg/l和172.84mg/l,与出发菌株相比分别降低了25.46%和21.05%,且主要高级醇(异丁醇、异戊醇、活性戊醇)总含量降低了21.15%。
3、本发明所述的葡萄汁酵母重组菌株羟酸还原异构酶和二羟异戊酸脱水酶过表达后,在发酵中对降低葡萄酒中的双乙酰含量有相互促进作用,如实施例所示菌株wy-3和wy-5发酵后双乙酰含量均为2.17mg/l,而菌株wy-35发酵后双乙酰含量为2.13mg/l,基因双重过表达后可以进一步降低双乙酰的含量。
4、本发明所述的葡萄汁酵母重组菌株部分敲除α-乙酰乳酸合成酶编码基因ilv2与二羟异戊酸脱水酶和羟酸还原异构酶过表达后,在发酵中对降低葡萄酒中的双乙酰含量有协同作用,如实施例所示菌株wy-5和wy-35发酵后双乙酰含量分别为2.17mg/l和2.13mg/l,而菌株v23-5和v23-35发酵后双乙酰含量分别为1.32mg/l和0.85mg/l。
本发明的葡萄汁酵母重组菌株克服了普通酵母由于双乙酰和高级醇含量较高导致的风味不协调,调控双乙酰和/或高级醇的目的,为酿造出风味优良且更有利于健康的葡萄酒奠定了理论基础,提高了葡萄酒的风味质量,具有广阔的市场前景。
以上只是概括了本发明的一些方面,不是也不应该认为是在任何方面限制本发明。
本说明书提到的所有专利和出版物都是通过参考文献作为整体而引入本发明的。本领域的技术人员应认识到,对本发明可作某些改变并不偏离本发明的构思或范围。下面的实施例进一步详细说明本发明,不能认为是限制本发明或本发明所说明的具体方法的范围。
附图说明:
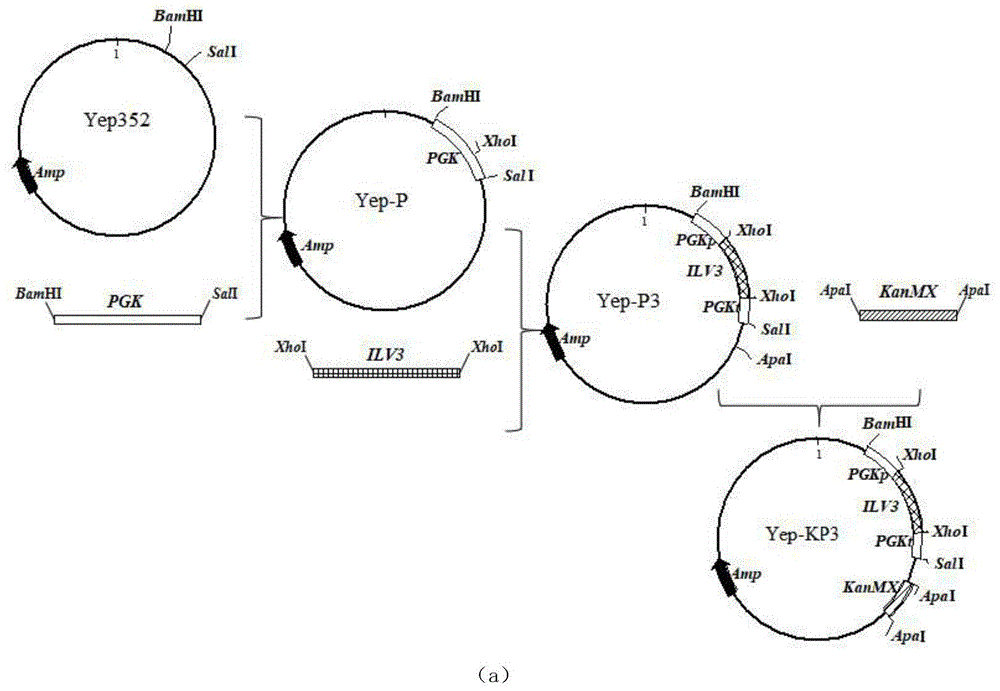
图1为yep-kp3、yep-kp5和yep-kp35质粒构建过程;
其中(a)为yep-kp3质粒构建过程;(b)为yep-kp5质粒构建过程;(c)yep-kp35质粒构建过程;
图2为质粒yep-kp3、yep-kp5和yep-kp35和重组菌株wy-3、wy-5和wy-35的验证:
其中:(a)中m为marker;1为以yep-p为模板,yp-f/yp-r为引物pcr扩增的pgk1基因片段;2为以yep352为模板,yp-f/yp-r为引物pcr扩增结果;3为以yep-p3为模板,yilv3-f/yilv3-r为引物pcr扩增的ilv3基因片段;4为以yep-p为模板,yilv3-f/yilv3-r为引物pcr扩增结果;5为以yep-kp3为模板,yk-f/yk-r为引物pcr扩增的kanmx基因片段;6为以yep-p3为模板,yk-f/yk-r为引物pcr扩增结果;
(b)中m为marker;1为以yep-kp5为模板,yk-f/yk-r为引物pcr扩增的kanmx基因片段;2为以yep-p5为模板,yk-f/yk-r为引物pcr扩增结果;3为以yep-p5为模板,yilv5-f/yilv5-r为引物pcr扩增的ilv5基因片段;4为以yep-p为模板,yilv5-f/yilv5-r为引物pcr扩增结果;
(c)中m为marker;1为以yep-p35为模板,smai-f/smai-r为引物pcr扩增的片段pgkp-ilv3-pgkp;2为以yep-v5为模板,smai-f/smai-r为引物pcr扩增结果;3为以yep-kp35为模板,yk-u/yk-d为引物pcr扩增kanmx基因片段;d4:以yep-p35为模板,yk-u/yk-d为引物pcr扩增结果
(d)中m为marker;1为以菌株wy-3酵母质粒为模板,ka-f和ka-r为引物pcr扩增验证片段;2为以菌株wy-5酵母质粒为模板,ka-f和ka-r为引物pcr扩增验证片段;3为以菌株wy-35酵母质粒为模板,ka-f和ka-r为引物pcr扩增验证片段;4为以出发菌株cicc1465酵母质粒为模板,ka-f和ka-r为引物pcr扩增结果。
图3为ilv2基因的敲除重组菌株的验证:
其中:(a)中m为marker;1和2分别为以出发菌株cicc1465和重组菌株v21的dna为模板,以yv2-f和ykx-r为引物,pcr扩增验证片段;3和4分别为出发菌株cicc1465和重组菌株v21的dna为模板,yks-f和yv2-r为引物,pcr扩增验证片段;
(b)中m为marker;1和2分别为以重组菌株v21和v21’的dna为模板,以yk-f/yk-r为引物,pcr验证结果;
(c)中m为marker;1和3分别为以出发菌株cicc1465和重组菌株v22的dna为模板,以yv2-f和ykx-r为引物,pcr扩增验证结果;2和4分别为以出发菌株cicc1465和重组菌株v22的dna为模板,yks-f和yv2-r为引物,pcr验证结果;
(d)中m为marker;1和2分别为以重组菌株v22’和v22的dna为模板,以yk-f/yk-r为引物,pcr验证结果;
(e)中m为marker;1和2分别为以出发菌株cicc1465和重组菌株v23的dna为模板,以yv2-f和ykx-r为引物,pcr验证结果;3和4分别为以出发菌株cicc1465和重组菌株v23的dna为模板,yks-f和yv2-r为引物,pcr验证结果;
(f)中m为marker;1和2分别为以重组菌株v23’和v23的dna为模板,以yk-f/yk-r为引物,pcr验证结果;
(g)中m为marker;1和2分别为以出发菌株cicc1465和重组菌株v23’的dna为模板,以yv2-f/yv2-r为引物,pcr验证结果;
图4为ilv2基因敲除ilv3和/或ilv5基因过表达的重组菌株的验证:
其中:图4中m为marker;1-4分别以重组菌株v23-3、v23-5、v23-35和v23’酵母质粒为模板,以ka-f/ka-r为引物,pcr验证结果。
具体实施方式:
下面通过具体的实施方案叙述本发明。除非特别说明,本发明中所用的技术手段均为本领域技术人员所公知的方法。另外,实施方案应理解为说明性的,而非限制本发明的范围,本发明的实质和范围仅由权利要求书所限定。对于本领域技术人员而言,在不背离本发明实质和范围的前提下,对这些实施方案中的物料成分和用量进行的各种改变或改动也属于本发明的保护范围。
本实例所用的出发菌株cicc1465。所述大肠杆菌dh5a购自takara公司。
所述ypd培养基为通用的完全培养基,固体培养基含2%进口琼脂粉。
所述ilv2基因其geneid为:855135,核苷酸序列如表中seqidno:1所示;所述ilv3基因其geneid为:853473,核苷酸序列如表中seqidno:2所示;所述ilv5基因其geneid为:851069,核苷酸序列如表中seqidno:3所示。所述启动子pgk1其geneid为:850370,核苷酸序列如表中seqidno:4所示。
根据genebank中的酵母基因组数据和整合质粒序列,设计了下述引物。
表1本实施例中所用到的引物
注:下划线部分为酶切位点。
表2本实施例中所用的pcr扩增体系
实施例1:过表达羟酸还原异构酶的葡萄汁酵母的构建
(1)重组质粒yep-kp5的构建
重组质粒yep-kp5的构建流程如图1(b)所示;
以葡萄汁酵母cicc1465菌株基因组为模板,使用引物对ilv5-f/ilv5-r(seqidno:9和10)pcr扩增得到ilv5基因片段(seqidno:3),pcr反应条件:95℃5min;94℃40s,56℃1min,72℃72s,30个循环;72℃10min。将ilv5基因片段与经xhoi酶切后的yep-p质粒通过同源重组而连接,构建质粒yep-p5;用限制性内切酶apai对质粒yep-p5酶切后与kanmx基因片段通过同源重组连接,构建质粒yep-kp5。
pcr验证结果如图2(b)所示,其中m为marker;1为以yep-kp5为模板,yk-f(seqidno:29)和yk-r(seqidno:30)为引物pcr扩增的kanmx基因片段;2为以yep-p5为模板,yk-f(seqidno:29)和yk-r(seqidno:30)为引物pcr扩增结果;3为以yep-p5为模板,yilv5-f(seqidno:25)和yilv5-r(seqidno:26)为引物pcr扩增的ilv5基因片段;4为以yep-p为模板,yilv5-f(seqidno:25)和yilv5-r(seqidno:26)为引物pcr扩增结果。如图2(b)泳道1和2所示,质粒yep-kp5可pcr扩增出kanmx基因片段,而yep-p5不能,说明kanmx基因片段成功连入质粒yep-p5;泳道3和4显示质粒yep-p5可pcr扩增出ilv5基因片段,而yep-p不能,说明ilv5基因片段成功连入质粒yep-p,表明重组质粒yep-kp5构建成功。
(2)重组菌株wy-5的构建
用醋酸锂转化法将重组质粒yep-kp5转入出发菌株cicc1465中,通过g418抗性筛选重组菌株wy-5,提取该重组菌株wy-5和出发菌株cicc1465的酵母质粒,分别以各酵母质粒为模板,ka-f(seqidno:31)和ka-rka-f(seqidno:32)为引物进行pcr,其中重组菌株酵母质粒可扩增出大小约为1650bp片段,且pcr扩增出的片段与预期的目的产物大小一致,而出发菌株则不能扩增得到该片段,pcr验证结果如图2(d)第2、4泳道所示。
实施例2:过表达二羟异戊酸脱水酶的葡萄汁酵母的构建
(1)重组质粒yep-kp3的构建
重组质粒yep-kp3的构建流程如图1(a)所示;
以质粒ppgk1为模板,pgk-f(seqidno:5)和pgk-r(seqidno:6)为引物,pcr扩增pgk1基因片段(seqidno:4),pcr反应条件:95℃5min;94℃40s,56℃1min,72℃108s,30个循环;72℃10min。用限制性内切酶bamhi和sali对质粒yep532和pgk1基因片段进行酶切,将两者连接构建质粒yep-p。
以葡萄汁酵母cicc1465菌株基因组为模板,使用引物对ilv3-f/ilv3-r(seqidno:7和8)pcr扩增得到片段ilv3(seqidno:2),pcr反应条件:95℃5min;94℃40s,56℃1min,72℃108s,30个循环;72℃10min。将该片段与经xhoi酶切后的yep-p质粒通过同源重组而连接,构建质粒yep-p3;以质粒pug6为模板,k-f(seqidno:11)和k-r(seqidno:12)为引物,pcr扩增kanmx基因片段,pcr反应条件:95℃5min;94℃40s,57℃1min,72℃100s,30个循环;72℃10min。用限制性内切酶apai对质粒yep-p3进行酶切后与kanmx基因片段通过同源重组连接,构建质粒yep-kp3。
pcr验证结果如图2(a)所示,其中m为marker;1为以yep-p为模板,yp-f(seqidno:21)和yp-r(seqidno:22)为引物pcr扩增的pgk1基因片段;2为以yep352为模板,yp-f(seqidno:21)和yp-r(seqidno:22)为引物pcr扩增结果;3为yep-p3为模板,yilv3-f(seqidno:23)和yilv3-r(seqidno:24)为引物pcr扩增的ilv3基因片段;4为以yep-p为模板,yilv3-f(seqidno:23)和yilv3-r(seqidno:24)为引物pcr扩增结果;5为以yep-kp3为模板,yk-f(seqidno:29)和yk-r(seqidno:30)为引物pcr扩增的kanmx基因片段;6为以yep-p3为模板,yk-f(seqidno:29)和yk-r(seqidno:30)为引物pcr扩增结果。如图2(a)泳道1和2所示,质粒yep-p可pcr扩增出pgk1基因片段,而yep352不能,说明pgk1基因片段成功连入质粒yep352;泳道3和4显示质粒yep-p3可pcr扩增出ilv3基因片段,而yep-p不能,说明ilv3基因片段成功连入质粒yep-p;泳道5和6显示质粒yep-kp3可pcr扩增出kanmx基因片段,而yep-p3不能,说明kanmx基因片段成功连入质粒yep-p3,表明重组质粒yep-kp3构建成功。
(2)重组菌株wy-3的构建
用醋酸锂转化法将重组质粒yep-kp3转入出发菌株cicc1465中,通过g418抗性筛选重组菌株wy-3,提取该重组菌株和出发菌株cicc1465的酵母质粒,分别以各酵母质粒为模板,ka-f(seqidno:31)和ka-r(seqidno:32)为引物进行pcr,其中重组菌株wy-3酵母质粒可扩增出大小约为1650bp片段,且pcr扩增出的片段与预期的目的产物大小一致,而出发菌株则不能扩增得到该片段,pcr验证结果如图2(d)第1、4泳道所示。
实施例3:过表达二羟异戊酸脱水酶和羟酸还原异构酶的葡萄汁酵母的构建
(1)重组质粒yep-kp35的构建
以质粒yep-p5为模板,pgk(smai)-f(seqidno:13)和pgk(smai)-r(seqidno:14)为引物,pcr扩增片段pgk1p-ilv5-pgk1t片段,pcr反应条件:95℃5min;94℃40s,56℃1min,72℃180s,30个循环;72℃10min。用限制性内切酶smai分别对质粒yep-p3和pgk1p-ilv5-pgk1t进行酶切,并将两者连接,构建质粒yep-p35。用限制性内切酶apai对质粒yep-p35酶切后与kanmx基因片段通过同源重组连接,构建质粒yep-kp35。
pcr验证结果如图2(c)所示,其中m为marker;1为以yep-p35为模板,smai-f(seqidno:27)和smai-r(seqidno:28)为引物pcr扩增的片段pgkp-ilv3-pgkp;2为以yep-p5为模板,smai-f(seqidno:27)和smai-r(seqidno:28)为引物pcr扩增结果;3为以yep-kp35为模板,yk-u(seqidno:29)和yk-d(seqidno:30)为引物pcr扩增kanmx基因片段;d4:以yep-p35为模板,yk-u(seqidno:29)和yk-d(seqidno:30)为引物pcr扩增结果。如图2(c)泳道1和2所示,质粒yep-p35可pcr扩增出pgkp-ilv3-pgkp基因片段,而yep-p5不能,说明pgkp-ilv3-pgkp基因片段成功连入质粒yep-p5;泳道3和4显示质粒yep-kp35可pcr扩增出kanmx基因片段,而yep-p35不能,说明kanmx基因片段成功连入质粒yep-p35,表明重组质粒yep-kp35构建成功。
(2)重组菌株wy-35的构建
用醋酸锂转化法将重组质粒yep-kp35转入出发菌株cicc1465中,通过g418抗性筛选重组菌株wy-35,提取该重组菌株wy-35和出发菌株cicc1465的酵母质粒,分别以各酵母质粒为模板,ka-f(seqidno:31)和ka-r(seqidno:32)为引物进行pcr,其中重组菌株酵母质粒可扩增出大小约为1650bp大小片段,且pcr扩增出的片段与预期的目的产物大小一致,而出发菌株则不能扩增得到该片段,pcr验证结果如图2(d)第3、4泳道所示。
实施例4:部分敲除ilv2基因的葡萄汁酵母的构建
以质粒puc6为模板,用引物ia1(seqidno:15)和ib1(seqidno:16)pcr扩增得到带有ilv2上下游60bp同源序列的kanmx片段,pcr反应条件:95℃5min;94℃40s,57℃1min,72℃100s,30个循环;72℃10min。通过醋酸锂转化法将其转化到葡萄汁酵母基因组,通过g418抗性筛选重组菌株v21并进行pcr验证,分别在ilv2基因上游外部和下游外边设计引物yv2-f(seqidno:33)和yv2-r(seqidno:34),在kanmx基因内部分别设计引物yks-f(seqidno:35)和ykx-r(seqidno:36),用于验证ilv2基因敲除,以yv2-f和ykx-r为引物,可扩增出大小为1988bp的片段,以yks-f和yv2-r为引物,可扩增出大小为2015bp的片段,而出发菌株均不能扩增出相应的片段,pcr验证结果如图3(a)所示。通过醋酸锂转化的方法将带有cre重组酶的pgapza质粒导入重组菌株v21中获得重组菌株v21’;挑取单菌落于半乳糖培养基中诱导4-5h,稀释106倍涂布于yepd平板上,挑出单菌落于yepd平板上,再影印到g418抗性平板上;挑出在yepd平板上生长而在g418抗性平板上不生长的菌株,提取基因组,以重组菌株v21’的基因组为模板扩增kanmx片段,无法得到1613bp左右的条带,而重组菌株v21则能扩增得到该片段,pcr验证结果如图3(b)所示。
采用缩进式的引物设计方法,结合多重基因中断技术敲除ilv2的第二条等位基因,以质粒puc6为模板,用引物ia2(seqidno:17)和ib2(seqidno:18)pcr扩增得到带有ilv2上下游60bp同源序列的kanmx片段,pcr反应条件:95℃5min;94℃40s,57℃1min,72℃100s,30个循环;72℃10min。通过醋酸锂转化法将其转化到葡萄汁酵母基因组,通过g418抗性筛选重组菌株v22并进行pcr验证,以yv2-f和ykx-r为引物,可扩增出大小为2062bp的片段,以yks-f和yv2-r为引物,可扩增出大小为2082bp的片段,而出发菌株均不能扩增出相应的片段,pcr验证结果如图3(c)所示。通过醋酸锂转化的方法将带有cre重组酶的pgapza质粒导入重组菌株v22中获得重组菌株v22’;挑取单菌落于半乳糖培养基中诱导4-5h,稀释106倍涂布于yepd平板上,挑出单菌落于yepd平板上,再影印到g418抗性平板上;挑出在yepd平板上生长而在g418抗性平板上不生长的菌株,提取基因组,以重组菌株v22’的基因组为模板扩增kanmx片段,无法得到1613bp左右的条带,而重组菌株v22则能扩增得到该片段,pcr验证结果如图3(d)所示。
同理,以质粒puc6为模板,用引物ia3(seqidno:19)和ib3(seqidno:19)pcr扩增得到带有ilv2上下游60bp同源序列的kanmx片段,pcr反应条件:95℃5min;94℃40s,57℃1min,72℃100s,30个循环;72℃10min。通过醋酸锂转化法将其转化到葡萄汁酵母基因组,通过g418抗性筛选重组菌株v23并进行pcr验证,以yv2-f和ykx-r为引物,可扩增出大小为2371bp的片段,以yks-f和yv2-r为引物,可扩增出大小为2305bp的片段,而出发菌株均不能扩增出相应的片段,pcr验证结果如图3(e)所示。通过醋酸锂转化的方法将带有cre重组酶的pgapza质粒导入重组菌株v23中获得重组菌株v23’;经过相同处理方法后,以重组菌株v23’的基因组为模板扩增kanmx片段,无法得到1613bp左右的条带,而重组菌株v23则能扩增得到该片段,pcr验证结果如图3(f)所示。为了验证ilv2基因敲除情况,以yv2-u/yv2-d为引物进行pcr,突变菌株v23’基因组可扩增出大小分别约为1200bp、1350bp、1880bp和3239bp四条带,出发菌株可扩增出大小为3239bp的一条带,说明突变菌株v23’中ilv2基因是部分敲除,pcr验证结果如图3(g)所示。
实施例5:部分敲除ilv2基因同时过表达ilv3和/或ilv5基因的葡萄汁酵母的构建
应用醋酸锂转化法将重组质粒yep-kp3、yep-kp5和yep-kp35分别转入ilv2三条等位基因缺失的重组菌株v23’中,获得葡萄汁酵母重组菌株v23-3、v23-5和v23-35。分别提取菌株v23-3、v23-5、v23-35和v23’的酵母质粒,分别以各酵母质粒为模板,以ka-f(seqidno:31)和ka-r(seqidno:32)为引物进行pcr,其中重组菌株v23-3、v23-5和v23-35酵母质粒可扩增出大小约为1650bp片段,且pcr扩增出的片段与预期的目的产物大小一致,而菌株v23’则不能扩增得到该片段,pcr验证结果如图4。
实施例6:低产双乙酰或/和高级醇葡萄汁酵母菌株发酵实验
(1)重组菌株与出发菌株的葡萄酒发酵实验
①发酵工艺路线图:
葡萄原料→筛选、清洗、晾干、除梗→破碎→调糖、调酸→加亚硫酸、灭菌→接菌→前发酵→皮渣分离→测定指标
②工艺条件:糖度:20.45brix;酸度:ph3.5;so2添加量:80mg/l,4℃静置12h;装液量:250ml的三角瓶装190ml葡萄汁;接菌量:1×108cfu/ml;发酵温度和时间:25℃,5d;蒸酒条件:100ml发酵液,加100ml水,蒸出100ml酒样。
按上述发酵工艺对葡萄汁酵母出发菌株cicc1465和选育实施例1-5的菌株wy-3、wy-5、wy-35、v21、v22、v23、v23-3、v23-5和v23-35进行葡萄酒发酵实验;发酵期间每隔12h振荡并称重,记录失重;发酵结束后,停止培养并称重;用温度计和酒度计测定馏出液的温度和酒精度,并将该温度下的酒精度换算成20℃下对应的酒精度,应用斐林试剂法测定葡萄酒中的还原糖含量,结果如表3。表3表明:在葡萄酒发酵实验中,本发明所获得的葡萄汁酵母重组菌株wy-3、wy-5、wy-35、v21、v22、v23、v23-3、v23-5和v23-35与出发菌株cicc1465相比,基本发酵性能没有太大变化。
表3亲本菌株和重组菌株的发酵性能测定
注:所示数据为三个平行试验结果的平均值。
(2)双乙酰和高级醇含量测定
发酵后葡萄酒中双乙酰和高级醇含量分别以高效液相色谱(hplc)和气相色谱(gc)方法进行测定,hplc分析:葡萄酒发酵液经0.22μm的纤维滤膜过滤后进行高效液相色谱分析。色谱条件为:色谱柱为bio-radhpx-87h,300×7.8mm;检测器为示差折光检测器(rid);流动相为5mmol/l硫酸,流速为0.6ml/min;检测器温度为45℃,柱温为65℃,进样量为20μl;gc分析:发酵液经蒸馏后,酒样进行高效气相色谱分析,色谱条件为:气相色谱仪为agilent7890c,并且配置有agilentg4512a自动进样器,色谱柱agilent1909n-213,30m×0.32mm×0.5μm毛细血管柱,检测器为fid。进样口的温度设置为200℃,检测器的温度为200℃。进样量条件:1μl的进样量,并设置为5:1的分流比。载气为高纯度的氮气,流速设置为2.0ml/min。升温程序:起始柱温设置为50℃,保持8min,再以5℃/min的升温速度上升至120℃,保持5min。测定结果见表4:
表4亲本菌株和重组菌株的双乙酰和高级醇含量(单位mg/l)
注:所示数据为三个平行试验结果的平均值。
表4表明:出发菌株cicc1465双乙酰的生成量是2.61mg/l,而本发明得到重组菌株wy-3、wy-5、wy-35、v21、v22、v23、v23-3、v23-5和v23-35的双乙酰生成量达到了2.17mg/l、2.17mg/l、2.13mg/l、1.85mg/l、1.83mg/l、1.43mg/l、1.09mg/l、1.32mg/l和0.85mg/l,相比较出发菌株降低了16.86%、16.86%、18.39%、20.18%、30.13%、45.21%、58.24%、49.43%和67.55%。说明单独过表达ilv5基因编码的羟酸还原异构酶,可以降低双乙酰的含量;联合过表达二羟异戊酸脱水酶后,在发酵中对降低葡萄酒中的双乙酰含量有相互促进作用;部分敲除α-乙酰乳酸合成酶编码基因ilv2在发酵中对降低葡萄酒中的双乙酰含量有协同作用。
另外,与出发菌株相比,重组菌株v21、v22、v23、v23-3、v23-5和v23-35发酵后葡萄酒异丁醇分别为35.22mg/l、33.42mg/l、30.81mg/l、37.25mg/l、36.04mg/l、31.08mg/l,比出发菌株分别降低了15.55%、19.84%、26.12%、10.68%、13.58%和25.46%,且异戊醇含量分别为212.15mg/l、183.84mg/l、176.15mg/l、190.28mg/l、173.52mg/l和172.84mg/l,降低了3.09%、16.03%、19.54%、13.09%、20.74%和21.05%,主要高级醇(异丁醇、异戊醇、活性戊醇)总含量分别为280.81mg/l、248.76mg/l、237.00mg/l、257.60mg/l、238.43mg/l和231.84mg/l,相比于出发菌株分别降低了4.50%、15.40%、19.40%、12.39%、18.91%和21.15%。说明本发明的重组菌株,在部分敲除ilv2基因后,可以进一步降低葡萄酒中的高级醇。
以上数据表明,本发明得到的菌株可以在很大程度上降低葡萄酒中双乙酰和高级醇含量,丰富了葡萄酒口感,提高葡萄酒的风味质量。
实施例7:葡萄汁酵母菌株培养
接种量的大小直接影响发酵周期和发酵性能,接种量过大会引起菌体大量繁殖使单位体积内的养料和溶氧供应不足而使得菌体得不到充分发育,干扰正常代谢。接种量过小,发酵前期生长缓慢,发酵的整个时间长,菌体浓度小,发酵效果差,因此,接种量一般要求以适量为原则。选择出发菌株,参照实施例6的步骤,单因素菌株接种量分别为1×107cfu/ml、5×107cfu/ml、1×108cfu/ml、5×108cfu/ml、1×109cfu/ml(分别为s1、s2、s3、s4、s5组),研究不同接种量对co2累计失重、酒精度、发酵液残糖,测定结果见表5,综合比较,选择接种量为5×107-5×108cfu/ml为宜。
表5不同接种量的发酵结果
以上详细描述了本发明的优选实施方式,但是,本发明并不限于上述实施方式中的具体细节,在本发明的技术构思范围内,可以对本发明的技术方案进行多种简单变型,这些简单变型均属于本发明的保护范围。
序列表
<110>天津科技大学
<120>一种过表达羟酸还原异构酶的葡萄汁酵母菌株及其应用
<160>38
<170>siposequencelisting1.0
<210>1
<211>1758
<212>dna
<213>葡萄汁酵母(saccharomycesuvarum)
<400>1
atgggcttgttaacgaaagttgctacatctagacaattctctacaacgagatgcgttgca60
aagaagctcaacaagtactcgtatatcatcactgaacctaagggccaaggtgcgtcccag120
gccatgctttatgccaccggtttcaagaaggaagatttcaagaagcctcaagtcggggtt180
ggttcctgttggtggtccggtaacccatgtaacatgcatctattggacttgaataacaga240
tgttctcaatccattgaaaaagcgggtttgaaagctatgcagttcaacaccatcggtgtt300
tcagacggtatctctatgggtactaaaggtatgagatactcgttacaaagtagagaaatc360
attgcagactcctttgaaaccatcatgatggcacaacactacgatgctaacatcgccatc420
ccatcatgtgacaaaaacatgcccggtgtcatgatggccatgggtagacataacagacct480
tccatcatggtatatggtggtactatcttgcccggtcatccaacatgtggttcttcgaag540
atctctaaaaacatcgatatcgtctctgcgttccaatcctacggtgaatatatttccaag600
caattcactgaagaagaaagagaagatgttgtggaacatgcatgcccaggtcctggttct660
tgtggtggtatgtatactgccaacacaatggcttctgccgctgaagtgctaggtttgacc720
attccaaactcctcttccttcccagccgtttccaaggagaagttagctgagtgtgacaac780
attggtgaatacatcaagaagacaatggaattgggtattttacctcgtgatatcctcaca840
aaagaggcttttgaaaacgccattacttatgtcgttgcaaccggtgggtccactaatgct900
gttttgcatttggtggctgttgctcactctgcgggtgtcaagttgtcaccagatgatttc960
caaagaatcagtgatactacaccattgatcggtgacttcaaaccttctggtaaatacgtc1020
atggccgatttgattaacgttggtggtacccaatctgtgattaagtatctatatgaaaac1080
aacatgttgcacggtaacacaatgactgttaccggtgacactttggcagaacgtgcaaag1140
aaagcaccaagcctacctgaaggacaagagattattaagccactctcccacccaatcaag1200
gccaacggtcacttgcaaattctgtacggttcattggcaccaggtggagctgtgggtaaa1260
attaccggtaaggaaggtacttacttcaagggtagagcacgtgtgttcgaagaggaaggt1320
gcctttattgaagccttggaaagaggtgaaatcaagaagggtgaaaaaaccgttgttgtt1380
atcagatatgaaggtccaagaggtgcaccaggtatgcctgaaatgctaaagccttcctct1440
gctctgatgggttacggtttgggtaaagatgttgcattgttgactgatggtagattctct1500
ggtggttctcacgggttcttaatcggccacattgttcccgaagccgctgaaggtggtcct1560
atcgggttggtcagagacggcgatgagattatcattgatgctgataataacaagattgac1620
ctattagtctctgataaggaaatggctcaacgtaaacaaagttgggttgcacctccacct1680
cgttacacaagaggtactctatccaagtatgctaagttggtttccaacgcttccaacggt1740
tgtgttttagatgcttga1758
<210>2
<211>1188
<212>dna
<213>葡萄汁酵母(saccharomycesuvarum)
<400>2
atgttgagaactcaagccgccagattgatctgcaactcccgtgtcatcactgctaagaga60
acctttgctttggccacccgtgctgctgcttacagcagaccagctgcccgtttcgttaag120
ccaatgatcactacccgtggtttgaagcaaatcaacttcggtggtactgttgaaaccgtc180
tacgaaagagctgactggccaagagaaaagttgttggactacttcaagaacgacactttt240
gctttgatcggttacggttcccaaggttacggtcaaggtttgaacttgagagacaacggt300
ttgaacgttatcattggtgtccgtaaagatggtgcttcttggaaggctgccatcgaagac360
ggttgggttccaggcaagaacttgttcactgttgaagatgctatcaagagaggtagttac420
gttatgaacttgttgtccgatgccgctcaatcagaaacctggcctgctatcaagccattg480
ttgaccaagggtaagactttgtacttctcccacggtttctccccagtcttcaaggacttg540
actcacgttgaaccaccaaaggacttagatgttatcttggttgctccaaagggttccggt600
agaactgtcagatctttgttcaaggaaggtcgtggtattaactcttcttacgccgtctgg660
aacgatgtcaccggtaaggctcacgaaaaggcccaagctttggccgttgccattggttcc720
ggttacgtttaccaaaccactttcgaaagagaagtcaactctgacttgtacggtgaaaga780
ggttgtttaatgggtggtatccacggtatgttcttggctcaatacgacgtcttgagagaa840
aacggtcactccccatctgaagctttcaacgaaaccgtcgaagaagctacccaatctcta900
tacccattgatcggtaagtacggtatggattacatgtacgatgcttgttccaccaccgcc960
agaagaggtgctttggactggtacccaatcttcaagaatgctttgaagcctgttttccaa1020
gacttgtacgaatctaccaagaacggtaccgaaaccaagagatctttggaattcaactct1080
caacctgactacagagaaaagctagaaaaggaattagacaccatcagaaacatggaaatc1140
tggaaggttggtaaggaagtcagaaagttgagaccagaaaaccaataa1188
<210>3
<211>2064
<212>dna
<213>葡萄汁酵母(saccharomycesuvarum)
<400>3
atgatcagacaatctacgctaaaaaacttcgctattaagcgttgctttcaacatatagca60
taccgcaacacacctgccatgagatcagtagctctcgcgcagcgcttttatagttcgtct120
tcccgttattacagtgcgtctccattaccagcctctaaaaggccagagcctgctccaagt180
ttcaatgttgatccattagaacagcccgctgaaccttcaaaattggctaagaaactacgc240
gctgagcctgacatggatacctctttcgtcggtttaactggtggtcaaatatttaacgaa300
atgatgtccagacaaaacgttgatactgtatttggttatccaggtggtgctatcctacct360
gtttacgatgccattcataacagtgataaattcaacttcgttcttccaaaacacgaacaa420
ggtgccggtcacatggcagaaggctacgccagagcttctggtaaaccaggtgttgtcttg480
gttacttctgggccaggtgccaccaatgtcgttactccaatggcagatgcctttgcagac540
gggattccaatggttgtctttacagggcaagtcccaactagtgctatcggtactgatgct600
ttccaagaggctgacgtcgttggtatttctagatcttgtacgaaatggaatgtcatggtc660
aagtccgtggaagaattgccattgcgtattaacgaggcttttgaaattgccacgagcggt720
agaccgggaccagtcttggtcgatttaccaaaggatgttacagcagctatcttaagaaat780
ccaattccaacaaaaacaactcttccatcaaacgcactaaaccaattaaccagtcgcgca840
caagatgaatttgtcatgcaaagtatcaataaagcagcagatttgatcaacttggcaaag900
aaacctgtcttatacgtcggtgctggtattttaaaccatgcagatggtccaagattacta960
aaagaattaagtgaccgtgctcaaatacctgtcaccactactttacaaggtttaggttca1020
ttcgaccaagaagatccaaaatcattggatatgcttggtatgcacggttgtgctactgcc1080
aacctggcagtgcaaaatgccgacttgataattgcagttggtgctagattcgacgaccgt1140
gtcactggtaatatttctaaattcgctccagaagctcgtcgtgcagctgccgagggtaga1200
ggtggtattattcatttcgaggttagtccaaaaaacataaacaaggttgttcaaactcaa1260
atagcagtggaaggtgatgctacgaccaatctgggcaaaatgatgtcaaagattttccca1320
gttaaggagaggtctgaatggtttgctcaaataaataaatggaagaaggaatacccatac1380
gcttatatggaggagactccaggatctaaaattaaaccacagacggttataaagaaacta1440
tccaaggttgccaacgacacaggaagacatgtcattgttacaacgggtgtggggcaacat1500
caaatgtgggctgctcaacactggacatggagaaatccacatactttcatcacatcaggt1560
ggtttaggtacgatgggttacggtctccctgccgccatcggtgctcaagttgcaaagcca1620
gaatctttggttattgacattgatggtgacgcatcctttaacatgactctaacggaattg1680
agttctgccgttcaagctggtactccagtgaagattttgattttgaacaatgaagagcaa1740
ggtatggttactcaatggcaatccctgttctacgaacatcgttattcccacacacatcaa1800
ttgaaccctgatttcataaaactagcggaggctatgggtttaaaaggtttaagagtcaag1860
aagcaagaggaattggacgctaagttgaaagaattcgtttctaccaagggcccagttttg1920
cttgaagtggaagttgataaaaaagttcctgttttgccaatggtggcaggtggtagcggt1980
ctagacgagttcataaattttgacccagaagttgaaagacaacagactgaattacgtcat2040
aagcgtacaggcggtaagcactga2064
<210>4
<211>1613
<212>dna
<213>人工序列(artificialsequence)
<400>4
cagctgaagcttcgtacgctgcaggtcgacaacccttaatataacttcgtataatgtatg60
ctatacgaagttattaggtctagagatctgtttagcttgcctcgtccccgccgggtcacc120
cggccagcgacatggaggcccagaataccctccttgacagtcttgacgtgcgcagctcag180
gggcatgatgtgactgtcgcccgtacatttagcccatacatccccatgtataatcatttg240
catccatacattttgatggccgcacggcgcgaagcaaaaattacggctcctcgctgcaga300
cctgcgagcagggaaacgctcccctcacagacgcgttgaattgtccccacgccgcgcccc360
tgtagagaaatataaaaggttaggatttgccactgaggttcttctttcatatacttcctt420
ttaaaatcttgctaggatacagttctcacatcacatccgaacataaacaaccatgggtaa480
ggaaaagactcacgtttcgaggccgcgattaaattccaacatggatgctgatttatatgg540
gtataaatgggctcgcgataatgtcgggcaatcaggtgcgacaatctatcgattgtatgg600
gaagcccgatgcgccagagttgtttctgaaacatggcaaaggtagcgttgccaatgatgt660
tacagatgagatggtcagactaaactggctgacggaatttatgcctcttccgaccatcaa720
gcattttatccgtactcctgatgatgcatggttactcaccactgcgatccccggcaaaac780
agcattccaggtattagaagaatatcctgattcaggtgaaaatattgttggtgcgctggc840
agtgttcctgcgccggttgcattcgattcctgtttgtaattgtccttttaacagcgatcg900
cgtatttcgtctcgctcaggcgcaatcacgaatgaataacggtttggttgatgcgagtga960
ttttgatgacgagcgtaatggctggcctgttgaacaagtctggaaagaaatgcataagct1020
tttgccattctcaccggattcagtcgtcactcatggtgatttctcacttgataaccttat1080
ttttgacgaggggaaattaataggttgtattgatgttggacgagtcggaatcgcagaccg1140
ataccaggatcttgccatcctatggaactgcctcggtgagttttctccttcattacagaa1200
acggctttttcaaaaatatggtattgataatcctgatatgaataaattgcagtttcattt1260
gatgctcgatgattttttctaatcagtactgacaataaaaagattcttgttttcaagaac1320
ttgtcatttgtatagtttttttatattgtagttgttctattttaatcaaatgttagcgtg1380
atttatattttttttcgcctcgacatcatctgcccagatgcgaagttaagtgcgcagaaa1440
gtaatatcatgcgtcaatcgtatgtgaatgctggtcgctatactgctgtcgattcgatac1500
taacgccgccatccagtgtcgaaaacgagctctcgagaacccttaatataacttcgtata1560
atgtatgctatacgaagttattaggtgatatcagatccactagtggcctatgc1613
<210>5
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>5
cgcggatcctctaactgatctatccaaaactg32
<210>6
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>6
acgcgtcgactaacgaacgcagaattttcgag32
<210>7
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>7
gaattccagatctcctcgagatgggcttgttaacgaaagttgcta45
<210>8
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>8
tctatcgcagatccctcgagtcaagcatctaaaacacaaccgttg45
<210>9
<211>43
<212>dna
<213>人工序列(artificialsequence)
<400>9
gaattccagatctcctcgagatgttgagaactcaagccgccag43
<210>10
<211>43
<212>dna
<213>人工序列(artificialsequence)
<400>10
tctatcgcagatccctcgagttattggttttctggtctcaact43
<210>11
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>11
ccgctaacaatacctgggccccagctgaagcttcgtacgc40
<210>12
<211>42
<212>dna
<213>人工序列(artificialsequence)
<400>12
gcacacggtgtggtgggcccgcataggccactagtggatctg42
<210>13
<211>29
<212>dna
<213>人工序列(artificialsequence)
<400>13
cggcccgggtctaactgatctatccaaaa29
<210>14
<211>29
<212>dna
<213>人工序列(artificialsequence)
<400>14
cggcccgggtaacgaacgcagaattttcg29
<210>15
<211>89
<212>dna
<213>人工序列(artificialsequence)
<400>15
tagaaagtattttacaaaatctaaaccctttgagctaagaggagataaatacaacagaat60
caattttcaacagctgaagcttcgtacgc89
<210>16
<211>89
<212>dna
<213>人工序列(artificialsequence)
<400>16
acgattaaataataataaagtctgcattttttactgaaaatgcttttgaaataaatgttt60
ttgaaatgcataggccactagtggatctg89
<210>17
<211>89
<212>dna
<213>人工序列(artificialsequence)
<400>17
atgatcagacaatctacgctaaaaaacttcgctattaagcgttgctttcaacatatagca60
taccgcaacacagctgaagcttcgtacgc89
<210>18
<211>89
<212>dna
<213>人工序列(artificialsequence)
<400>18
tcagtgcttaccgcctgtacgcttatgacgtaattcagtctgttgtctttcaacttctgg60
gtcaaaagcataggccactagtggatctg89
<210>19
<211>80
<212>dna
<213>人工序列(artificialsequence)
<400>19
caaaacgttgatactgtatttggttatccaggtggtgctatcctacctgtttacgatgcc60
atagctgaagcttcgtacgc80
<210>20
<211>82
<212>dna
<213>人工序列(artificialsequence)
<400>20
agcctccgctagttttatgaaatcagggttcaattgatgtgtgtgggaataacgatgttc60
gcataggccactagtggatctg82
<210>21
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>21
tctaactgatctatccaaaactga24
<210>22
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>22
taacgaacgcagaattttc19
<210>23
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>23
atgggcttgttaacgaaagttgcta25
<210>24
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>24
tcaagcatctaaaacacaaccgttg25
<210>25
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>25
atgttgagaactcaagccgccag23
<210>26
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>26
ttattggttttctggtctcaact23
<210>27
<211>17
<212>dna
<213>人工序列(artificialsequence)
<400>27
ttcgagctcggtacccg17
<210>28
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>28
agttagaggatccccggg18
<210>29
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>29
cagctgaagcttcgtacgc19
<210>30
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>30
gcataggccactagtggatctg22
<210>31
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>31
cctgcttcaaaccgctaacaata23
<210>32
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>32
cgaatgcacacggtgtggt19
<210>33
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>33
tgtaaacagaactttgccacta22
<210>34
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>34
ttcatttatggcttcgtcgga21
<210>35
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>35
ccgtacatttagcccatac19
<210>36
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>36
cagatgatgtcgaggcgaa19
<210>37
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>37
atcgtcgaccccacacaccatagcttca28
<210>38
<211>29
<212>dna
<213>人工序列(artificialsequence)
<400>38
gcggtcgacagcttgcaaattaaagcctt29
- 还没有人留言评论。精彩留言会获得点赞!