一种拟禾本科根结线虫钙网蛋白基因及其应用的制作方法
本发明属于生物
技术领域:
,更具体地说,涉及一种拟禾本科根结线虫钙网蛋白基因及其应用。
背景技术:
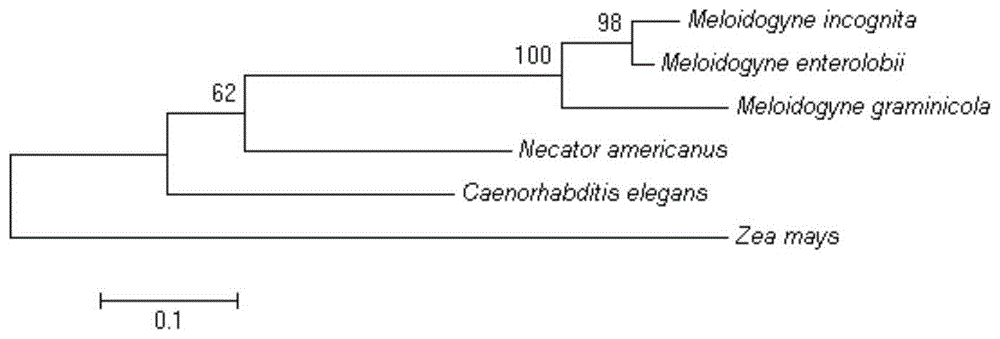
:根结线虫(meloidogynespp.)是一类固着型内寄生植物病原线虫。根结线虫寄主范围广,适应性强,对农业生产危害严重,造成巨大经济损失。轮作、生物熏蒸、生物防治及化学农药是防治线虫的主要方法,但是存在防治效果差、污染环境等问题。存在特异性差、副作用大、防效有限等突出问题。钙网蛋白在人类和多种动物寄生线虫中被发现并被证实与线虫寄生和人类疾病相关,特别是发现肠道寄生线虫在取食时会分泌钙网蛋白,因此推测其在线虫的侵染前后均扮演重要的角色。钙网蛋白的功能在秀丽隐杆线虫(caenorhabditiselegans)中已被大量研究,这些研究表明钙网蛋白有多种重要的生物学功能。在植物寄生线虫中,已报道在南方根结线虫(meloidogyneincognita)上分离得到钙网蛋白基因micrt,该基因是直接通过分离口针分泌物后测序获得的。jaubert等在2005年进一步经原位杂交、蛋白荧光免疫组织定位并将南方根结线虫接种于拟南芥对该基因进行组织定位,证实micrt不仅在南方根结线虫侵染型2龄幼虫的亚腹食道腺中表达,而且在取食位点和2龄幼虫在拟南芥组织中的迁移过程中也有该基因表达产物,暗示该基因与南方根结线虫侵染寄主相关。技术实现要素:针对现有技术存在的上述问题,本发明所要解决的技术问题在于提供一种拟禾本科根结线虫钙网蛋白基因。本发明所要解决的另一技术问题在于提供所述拟禾本科根结线虫钙网蛋白基因的应用。为了解决上述技术问题,本发明所采用的技术方案如下:一种拟禾本科根结线虫钙网蛋白基因,其核苷酸序列如seqidno.1所示。所述的拟禾本科根结线虫钙网蛋白基因的表达蛋白,其氨基酸序列如seqidno.2所示。所述的拟禾本科根结线虫钙网蛋白基因在阻止拟禾本科根结线虫侵染农作物中的应用。所述的应用,是以拟禾本科根结线虫钙网蛋白基因作为靶标,使用小分子rna干扰其表达。一种靶向所述的拟禾本科根结线虫钙网蛋白基因的dsrna,所述的dsrna包括正义链和反义链,所述的正义链的核苷酸序列如seqidno.3所示;所述的反义链的核苷酸序列如seqidno.4所示。所述靶向拟禾本科根结线虫钙网蛋白基因的dsrna在阻止拟禾本科根结线虫侵染农作物中的应用。所述靶向拟禾本科根结线虫钙网蛋白基因的dsrna在阻止拟禾本科根结线虫侵染农作物中的应用的具体方法为:使拟禾本科根结线虫浸泡于所述靶向拟禾本科根结线虫钙网蛋白基因的dsrna的溶液中。所述的应用中,所述的农作物为水稻。制备所述的靶向拟禾本科根结线虫钙网蛋白基因的dsrna的引物:mgcrt-pf:5′-attgactgcggtggtggtt-3′,mgcrt-t7r:5′-taatacgactcactataggggttcatcgtccactttgtattc-3′,mgcrt-pr:5′-gttcatcgtccactttgtattc-3′,mgcrt-t7f:5′-taatacgactcactatagggattgactgcggtggtggtt-3′。相比于现有技术,本发明的有益效果为:本发明首次获得拟禾本科根结线虫(meloidogynegraminicola)钙网蛋白基因mgcrt,其核苷酸序列为seqidno.1所示,其表达蛋白的氨基酸序列为seqidno.2所示。通过实验证实该基因在拟禾本科根结线虫亚腹食道腺中表达。进一步设计并合成该基因的dsrna(包括正义链和反义链,正义链的核苷酸序列如seqidno.3所示,反义链的核苷酸序列如seqidno.4所示),将拟禾本科根结线虫幼虫浸在该dsrna的溶液中,洗涤后接种水稻,发现采用此方法使拟禾本科根结线虫mgcrt基因沉默后,侵染水稻的线虫数量显著减少,表明拟禾本科根结线虫的mgcrt基因与其侵染寄主相关,也表明可以利用mgcrt基因作为靶标使用rna干扰等技术防治拟禾本科根结线虫侵染农作物。附图说明图1为crt基因的进化树图;图2为mgcrt基因在拟禾本科根结线虫中表达定位分析结果图;图中,a为正/反义探针电泳图;b为反义链探针杂交结果图;c为正义链探针杂交结果图;图3为rnai处理后的拟禾本科根结线虫侵染水稻结果图;图中dsrna-crt为对mgcrt基因进行dsrna沉默处理组,ck为对照组;knot表示拟禾本科线虫根结,j2表示拟禾本科根结线虫二龄幼虫。具体实施方式下面结合具体实施例对本发明进一步进行描述。材料:拟禾本科根结线虫(meloidogynegraminicola)来自浙江金华水稻根部;主要试剂:trizolreagent购自invitrogen公司;primescripttm1ststrandcdnasynthesiskit、takaraminibestagarosegeldnaextractionkitver.4.0、3′-fullracecoresetwithprimescripttmrtase购自takara公司;杂交试剂盒(digdnalabelingkit)为roche公司产品;t7rnaitranscriptionkit为南京诺唯赞公司产品。实施例1:拟禾本科根结线虫dna提取1、总rna的提取采用trizolreagent提取总rna,步骤如下:1)rna提取过程中所用ddh2o、离心管和枪头等用0.1%的depc处理。2)将拟禾本科根结线虫收集于1.5ml离心管中,液氮速冻,加入200μltrizol,用微量组织匀浆器充分研磨后,再加入800μltrizol,充分混匀后室温静置5min;3)加入200μl氯仿,充分振荡,室温放置3min,4℃12000rpm离心15min;4)吸取上清至另一1.5ml离心管中,加入550μl异丙醇,混匀,室温静置10min,4℃12000rpm离心10min;5)弃上清,每管中加入1ml75%乙醇洗涤沉淀,4℃7500rpm离心5min;6)弃上清,待管中酒精挥发后,加入20μldepch2o溶解rna,rna于-80℃冰箱保存。2、反转录合成cdna第一链cdna的合成使用primescripttm1ststrandcdnasynthesiskit(takara)试剂盒合成cdna,步骤如下:1)rnasefree的pcr管置于冰上,管中依次加入下面的反应物:2μl总rna,1μldntpsmixture(10mm),1μl引物oligo(dt),rnasefreedh2oupto10μl2)65℃温浴混合液5min,冰上迅速冷却;3)依次加入:4μl5×primerscriptbuffer,0.5μlrnaseinhibitor(40u/μl),1μlprimerscriptrtase(200),rnasefreedh2oupto20μl4)混匀,短暂离心收集至管底;5)50℃30min,85℃5min终止反应。反转录合成的cdna可用于后续的pcr。实施例2:拟禾本科根结线虫钙网蛋白基因(mgcrt)的克隆对拟禾本科根结线虫转录组数据进行拼接处理并一一比对,得到一段1712bp的序列与南方根结线虫ctr序列相似性很高,且该序列已包含crt基因完整的5’端和orf序列,3’序列不全,根据这段序列设计验证引物,见表1,用实施例1得到的cdna进行pcr扩增。表1引物序列引物序列mgcrt-f-575′-acgaagattaagaatggccg-3′mgcrt-r-6565′-ctttctgccttttcgccatc-3′mgcrt-orf-f5′-tctaaacataagagcgactacgg-3′mgcrt-orf-r5′-gtgcggtatttggtagggag-3′pcr反应体系:1μlcdna,1μl引物f(10μm),1μl引物r(10μm),2.5μl10×buffer,2μldntp(2.5mm),0.2μltaq,16.3μlddh2o。pcr反应条件:94℃预变性5min;94℃变性30s,55℃退火30s,72℃延伸1min,循环35次;72℃延伸10min。配制1%琼脂糖凝胶,pcr产物经电泳验证片段大小后,用takaraminibestagarosegeldnaextractionkitver.4.0凝胶回收试剂盒进行切胶回收。pcr纯化产物可直接与pucm-t载体(takara)连接,采用10μl反应体系,依次加入:7μlpcr扩增产物,1μlpucm-t,1μl10×连接缓冲液,1μlt4dna连接酶,10μl终体积。12000rpm离心10秒,置4℃连接过夜或16℃连接4小时。将连接后的载体转入大肠杆菌dh5α感受态并通过菌落pcr和dna测序确定阳性菌落,序列测定由上海生物工程有限公司完成。测序得到的序列与转录组拼接得到序列完全匹配,确定为拟禾本科根结线虫钙网蛋白基因的部分序列。实施例3:race技术克隆mgcrt基因的全长序列根据已获得拟禾本科根结线虫钙网蛋白基因的序列,设计3’端race所需的特异性引物mgcrt-r3’(5′-acatttgcggacctggaac-3′)与3’端接头引物进行巢式pcr反应,扩增目的基因的3’端序列。按照3′-fullracecoresetwithprimescripttmrtase(takara)操作说明,使用3’raceadaptor引物进行反转录反应,合成1ststrandcdna;使用特异性引物和3’raceouterprimer进行pcr反应得到目的片段进行测序,得到序列与已知序列进行拼接,获得拟禾本科根结线虫钙网蛋白(mgcrt)cdna全长序列。mgcrt基因cdna全长1715bp,其核苷酸序列如seqidno.1所示,包含一个1248bp开放阅读框(orf)。cdna开放阅读框编码415个氨基酸,其氨基酸序列如seqidno.2所示。实施例4:序列分析dnastar软件进行序列拼接和序列一致性比较,primer5.0软件进行引物设计,mega5.0软件进行多重序列比对和构建neighbor-joining系统进化树。blast比对发现mgcrt与南方根结线虫(meloidogyneincognita)、象耳豆根结线虫(meloidogyneenterolobii)来源的crt蛋白相似度分别为86.5%、87.7%。用mega5.0软件用neighbor-joining方法进行聚类分析,构建了crt基因的进化树(图1)。结果显示拟禾本科根结线虫mgcrt基因与南方根结线虫、象耳豆根结线虫的紧密聚在一起。实施例5:mgcrt基因表达定位分析选用roche的digdnalabelingkit杂交试剂盒,对mgcrt基因进行组织表达定位分析。根据mgcrt基因序列设计探针引物,如下表2所示;表2引物序列引物序列mgcrt-lab-f5′-gtggggaactggaaactgat-3′mgcrt-lab-r-t75′-taatacgactcactatagggttgcttcatcaatactgtccgtc-3′mgcrt-lab-f-t75′-taatacgactcactataggggtggggaactggaaactgat-3′mgcrt-lab-r5′-ttgcttcatcaatactgtccgtc-3′以第一链cdna为模板,按照roche杂交试剂盒要求,进行不对称pcr合成杂交探针。探针pcr反应程序为:94℃预变性2min;94℃变性30s,56℃退火30s,72℃延伸2min,40个循环;72℃延伸10min。pcr产物经1.2%凝胶电泳检测后,-80℃储存,备用。收集不同龄期新鲜的线虫,用2%的多聚甲醛溶液浸48℃泡16h,室温固定处理4h;用lysisbuffer、蛋白酶k、甲醛、丙酮等溶液,对线虫进行消化和通透处理;用杂交液重悬虫体,将预处理的线虫分别与标记好的正/反义链探针杂交,37℃黑暗处理16h,进行显色反应(显色抗体选用带fitc的抗体),最后用0.01%吐温20停止染色;用leicadm5000显微镜观察、拍照。所得结果如图2,反义链探针杂交处理的线虫,在亚腹食道腺附近显色,说明该基因在拟禾本科根结线虫亚腹食道腺中表达。实施例6:mgcrt基因rna干扰分析采用南京诺唯赞的t7rnaitranscriptionkit试剂盒进行dsrna体外转录合成。根据mgcrt基因序列,设计引物:mgcrt-pf(5′-attgactgcggtggtggtt-3′)和mgcrt-t7r(5′-taatacgactcactataggggttcatcgtccactttgtattc-3′),引物mgcrt-pr(5′-gttcatcgtccactttgtattc-3′)和mgcrt-t7f(5′-taatacgactcactatagggattgactgcggtggtggtt-3′),根据试剂盒说明书,以cdna为模板,分别进行正义链ssrna及反义链ssrna的pcr合成。随后,将上述ssrnapcr产物按1∶1混合,94℃解链处理10min,冷却至25℃退火合成dsrna。凝胶电泳检测合成的dsrna质量。所合成dsrna包括正义链和反义链,正义链的核苷酸序列如seqidno.3所示;反义链的核苷酸序列如seqidno.4所示。采用体外浸泡dsrna法,对mgcrt基因进行rnai沉默处理:将拟禾本科根结线虫幼虫用depc-h2o清洗3次,离心后吸取10μl线虫悬浮液,浸泡至40μl的dsrna溶液(终质量浓度为3μg/μl)中。用移液枪轻轻混匀后,置于25℃、200r/min条件下浸泡处理24h(室温黑暗条件下旋转培养24h)后,洗涤后接种水稻,每株水稻接种150条处理的二龄幼虫,重复4次,将同等数量的线虫浸泡至40μlddh2o中,作对照组。接种50h后,分别记录水稻根部二龄幼虫(j2)和根结(knot)个数,取平均值绘制图表。所得结果如图3,图中dsrna-crt为对mgcrt基因进行dsrna沉默处理组,ck为对照组,knot表示拟禾本科根结线虫根结,j2为拟禾本科根结线虫二龄幼虫,可以发现,对mgcrt基因沉默后,侵染水稻的线虫的根结和幼虫数量均显著减少,表明mgcrt基因的表达蛋白在拟禾本科根结线虫侵染寄主过程中发挥重要作用。也表明使用rna干扰技术沉默mgcrt基因作为防治拟禾本科根结线虫侵染水稻的方法是非常有效的。序列表<110>苏州市外来有害生物防控技术中心<120>一种拟禾本科根结线虫钙网蛋白基因及其应用<130>100<160>17<170>siposequencelisting1.0<210>1<211>1715<212>dna<213>meloidogynegraminicola<400>1ccctatttgagatattaaataatattttaattcaaatttttattgtttatttttcaacga60agattaagaatggccgaaaggttttctcttctatcaacactgcttattgcaggctttttg120accttcacgtacggggaagttttcttcaaggaggaattcacagatgagtcatgggttgat180cgatgggttcaatctaaacataagagcgactacggaaaatttgaactttctcatgggaaa240ttttttggggacaaggaacgcgatcaaggacttaaaacttctcaagatgctcgcttctac300agcatttctgctaaattccctgagaaatttagcaacaaaggaaaaacattagtagtccaa360tttaccattaaacatgaacaggatattgactgcggtggtggttatcttaagctaatggcc420tcaactattaaccaagaagactttcatggagaaaccccttaccatttaatgtttggcccg480gacatttgcggacctggaacgaagaaggttcatgttatcatcaactacaaaggaaagaac540aatcttattaaaaaagacattcgatgcaaggatgatgtgcttacacacctctatactctg600atcctcaacccagacaatacttatgaagttcaaattgatggcgaaaaggcagaaagtggg660gaactggaaactgattgggagttgttgcctgaaaagaaaattaaagatcctgatgccaaa720aagcctgacgattgggatgagaccgagtatatagacgatcctgaggataaaaaaccagaa780gactgggactccacacctgagacgattcctgatccagatgcaaaaaagcctgaggactgg840gatgacgaaatggacggcgaatgggaggcaccaaaaattgataatccgaattataaagga900gaatttaaaccaaagcagatcaaaaatccaaattataaaggcaaatggattcatcctgaa960attgacaatccggaatacaaagtggacgatgaactctatatgcgtgatgattggggtgct1020gttggtattgatatttggcaggttaaatccggttctatctttgacaatattatcgtgacg1080gacagtattgatgaagcaaagtctcatgcaaaagaaacatttgaaccattacgcgatgct1140gagaaaaaacagaaagaagctgctgatgaggaggagaggaagaagtttgaagaggaagag1200aagaaacgtaaggaggaggaagaatcaaaaaagaaagacgaggataaagaagatgaggac1260gaagaggaaaaggaaggaggtgaaaagaaggaggaagatgagcatgatgagctttaagga1320aggtgtgtaataaaaacgttcacttttcttctaataaaaaatatattatggggactctgc1380cggcaccttttctttgttttttgtgggttttttctcttttttaggtaatgatatttttta1440atcgtcatttaaatattaaatttatttgttctctcctccctaccaaataccgcacttttt1500gactataattggttttaaattattttttgcttaaaataattgtgtcttcattattttttg1560ttttgtttttccttaattcgcgctatacttttttcttttattatcggcttctttgtttct1620gaagctcataattaaagaaaaaaatggaatatttttgtcttttatttaaaataaatttgt1680tatttcttcaaaaaaaaaaagaaaaaaaaacaaaa1715<210>2<211>415<212>prt<213>meloidogynegraminicola<400>2metalagluargpheserleuleuserthrleuleuilealaglyphe151015leuthrphethrtyrglygluvalphephelysglugluphethrasp202530glusertrpvalaspargtrpvalglnserlyshislysserasptyr354045glylysphegluleuserhisglylysphepheglyasplysgluarg505560aspglnglyleulysthrserglnaspalaargphetyrserileser65707580alalyspheproglulyspheserasnlysglylysthrleuvalval859095glnphethrilelyshisgluglnaspileaspcysglyglyglytyr100105110leulysleumetalaserthrileasnglngluaspphehisglyglu115120125thrprotyrhisleumetpheglyproaspilecysglyproglythr130135140lyslysvalhisvalileileasntyrlysglylysasnasnleuile145150155160lyslysaspileargcyslysaspaspvalleuthrhisleutyrthr165170175leuileleuasnproaspasnthrtyrgluvalglnileaspglyglu180185190lysalagluserglygluleugluthrasptrpgluleuleuproglu195200205lyslysilelysaspproaspalalyslysproaspasptrpaspglu210215220thrglutyrileaspaspprogluasplyslysprogluasptrpasp225230235240serthrprogluthrileproaspproaspalalyslysprogluasp245250255trpaspaspglumetaspglyglutrpglualaprolysileaspasn260265270proasntyrlysglygluphelysprolysglnilelysasnproasn275280285tyrlysglylystrpilehisprogluileaspasnproglutyrlys290295300valaspaspgluleutyrmetargaspasptrpglyalavalglyile305310315320aspiletrpglnvallysserglyserilepheaspasnileileval325330335thraspserileaspglualalysserhisalalysgluthrpheglu340345350proleuargaspalaglulyslysglnlysglualaalaaspgluglu355360365gluarglyslysphegluglugluglulyslysarglysglugluglu370375380gluserlyslyslysaspgluasplysgluaspgluaspglugluglu385390395400lysgluglyglyglulyslysglugluaspgluhisaspgluleu405410415<210>3<211>610<212>rna<213>dsrnaf(artificial)<400>3auugacugcgguggugguuaucuuaagcuaauggccucaacuauuaaccaagaagacuuu60cauggagaaaccccuuaccauuuaauguuuggcccggacauuugcggaccuggaacgaag120aagguucauguuaucaucaacuacaaaggaaagaacaaucuuauuaaaaaagacauucga180ugcaaggaugaugugcuuacacaccucuauacucugauccucaacccagacaauacuuau240gaaguucaaauugauggcgaaaaggcagaaaguggggaacuggaaacugauugggaguug300uugccugaaaagaaaauuaaagauccugaugccaaaaagccugacgauugggaugagacc360gaguauauagacgauccugaggauaaaaaaccagaagacugggacuccacaccugagacg420auuccugauccagaugcaaaaaagccugaggacugggaugacgaaauggacggcgaaugg480gaggcaccaaaaauugauaauccgaauuauaaaggagaauuuaaaccaaagcagaucaaa540aauccaaauuauaaaggcaaauggauucauccugaaauugacaauccggaauacaaagug600gacgaugaac610<210>4<211>610<212>rna<213>dsrnar(artificial)<400>4guucaucguccacuuuguauuccggauugucaauuucaggaugaauccauuugccuuuau60aauuuggauuuuugaucugcuuugguuuaaauucuccuuuauaauucggauuaucaauuu120uuggugccucccauucgccguccauuucgucaucccaguccucaggcuuuuuugcaucug180gaucaggaaucgucucagguguggagucccagucuucugguuuuuuauccucaggaucgu240cuauauacucggucucaucccaaucgucaggcuuuuuggcaucaggaucuuuaauuuucu300uuucaggcaacaacucccaaucaguuuccaguuccccacuuucugccuuuucgccaucaa360uuugaacuucauaaguauugucuggguugaggaucagaguauagagguguguaagcacau420cauccuugcaucgaaugucuuuuuuaauaagauuguucuuuccuuuguaguugaugauaa480caugaaccuucuucguuccagguccgcaaauguccgggccaaacauuaaaugguaagggg540uuucuccaugaaagucuucuugguuaauaguugaggccauuagcuuaagauaaccaccac600cgcagucaau610<210>5<211>20<212>dna<213>mgcrt-f-57(artificial)<400>5acgaagattaagaatggccg20<210>6<211>20<212>dna<213>mgcrt-r-656(artificial)<400>6ctttctgccttttcgccatc20<210>7<211>23<212>dna<213>mgcrt-orf-f(artificial)<400>7tctaaacataagagcgactacgg23<210>8<211>20<212>dna<213>mgcrt-orf-r(artificial)<400>8gtgcggtatttggtagggag20<210>9<211>19<212>dna<213>mgcrt-r3'(artificial)<400>9acatttgcggacctggaac19<210>10<211>20<212>dna<213>mgcrt-lab-f(artificial)<400>10gtggggaactggaaactgat20<210>11<211>43<212>dna<213>mgcrt-lab-r-t7(artificial)<400>11taatacgactcactatagggttgcttcatcaatactgtccgtc43<210>12<211>40<212>dna<213>mgcrt-lab-f-t7(artificial)<400>12taatacgactcactataggggtggggaactggaaactgat40<210>13<211>23<212>dna<213>mgcrt-lab-r(artificial)<400>13ttgcttcatcaatactgtccgtc23<210>14<211>19<212>dna<213>mgcrt-pf(artificial)<400>14attgactgcggtggtggtt19<210>15<211>42<212>dna<213>mgcrt-t7r(artificial)<400>15taatacgactcactataggggttcatcgtccactttgtattc42<210>16<211>22<212>dna<213>mgcrt-pr(artificial)<400>16gttcatcgtccactttgtattc22<210>17<211>39<212>dna<213>mgcrt-t7f(artificial)<400>17taatacgactcactatagggattgactgcggtggtggtt39当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1