靶RNA检测
本公开涉及在检测样品中的靶rna序列中应用的产品和方法。
背景技术:
1、2020年3月11日,covid-19被宣布为全球大流行,114个不同的国家报告了超过118,000例病例。三个月内,188个国家的病例数已增至近650万例。为了减少其在人群中的传播,准确有效的病毒检测策略是必要的。该策略的关键部分是以减少试验时间并提高样品通量为目的,进行持续的试验开发。鉴于sars-cov-2感染的临床症状可能类似于普通感冒和流感,因此明确识别病毒本身对于有效诊断至关重要。在疾病的早期阶段,这只能通过检测病毒rna来真正实现。
2、诊断行业迅速响应,开发了一系列检测平台,cdc、赛默飞世尔(thermo fisher)和英国公共卫生局(public health england)等都发布了测试来应对这场前所未有的危机。1用于sars-cov-2检测的最常见试验方法是基于qpcr的检测方法,每个样品需要多于60分钟的时间,并且通常涉及两步过程。首先,使用逆转录酶将病毒rna转化为互补dna(cdna),这一过程可能需要多达30分钟。2然后进行定量聚合酶链式反应(qpcr)以扩增cdna,并使用荧光染料检测所述cdna,该过程需要多达一个小时。3–5为了缩短试验时间,过去几个月的文献中出现了大量sars-cov-2rna检测的新方法。可以说,其中最成功的方法是将等温方法应用于dna扩增,大大加快了扩增速度并因此减少了试验时间。6,7最常见的等温扩增系统是环介导等温扩增(lamp)。已经针对sars-cov-2开发了lamp方法,但它们很难在<10分钟内给出结果。7
3、另一种常用于诊断试验的技术是指数扩增反应(expar)。expar反应仅限于dna,因为反应所需的切口核酸内切酶仅对dna双链体有活性。这意味着若要使用expar检测rna,需要rna->dna步骤。这通常由逆转录酶(rt)反应提供。除了经常需要rt步骤外,expar比其他类似技术更快,因为它经过优化,只需要非常短的单链序列即可扩增。expar试验所需的时间通常无法进一步优化,因为得到的短片段不具有期望温度下的结合能量以形成引发聚合酶所需的双链体。可以通过升高温度来提高速率,但这也意味着需要更长的序列以产生双链体。这意味着提高expar速度的方法(例如采用rt步骤的基于expar的试验)是受限的。此外,进行试验所需的任何附加反应都将进一步增加总试验时间。
技术实现思路
1、本公开的基础是鉴定和利用能够使rna/dna双链体的dna链产生切口的酶,并且利用带切口的dna以产生扩增产物。在一方面教导中,扩增产物可以使用指数扩增反应(expar)产生。根据本文的教导,发明人显示他们可以在10分钟内(例如在5分钟内)准确地识别样品(例如,covid-19患者样品)rna。
2、在第一方面教导中,提供了一种用于检测样品中的靶rna序列的方法,所述方法包括:
3、a)在第一温度,使样品与结合物dna分子和限制性核酸内切酶接触,所述限制性核酸内切酶能够在特异性识别序列处使rna/dna复合物的dna链产生切口,以产生带切口的dna分子,
4、其中所述结合物dna分子能够在第一温度与靶rna序列特异性结合,从而形成rna/dna双链体:结合物dna分子包含:在第一温度不与靶rna序列结合且包含与模板dna分子互补的序列的部分序列;和,能够在第一温度特异性结合至靶rna序列且包括特异性识别序列的另一部分序列,
5、其中所述限制性核酸内切酶在识别序列处使结合的结合物dna分子产生切口,使得带切口的dna分子能够从rna/dna双链体释放;或
6、a')在第一温度,使样品与引物,逆转录酶和限制性核酸内切酶接触,所述限制性核酸内切酶能够在特异性识别序列处使rna/dna复合物的dna链产生切口,以产生带切口的dna分子,
7、其中所述引物能够在第一温度特异性结合至靶rna序列,从而形成rna/dna双链体并在dntp和逆转录酶存在下起始链延伸并产生延伸的rna/dna双链体,所述延伸的rna/dna双链体包含特异性识别序列,
8、其中所述限制性核酸内切酶在识别序列处使延伸的rna/dna双链体产生切口,使得带切口的dna分子能够从rna/dna双链体释放;和
9、b)在第二温度,使带切口的dna分子与模板dna分子接触,使得带切口的dna分子的至少3'部分结合模板dna分子并在dntp和dna聚合酶存在下起始链延伸并产生扩增产物;
10、以及检测任何所述扩增产物,其中若检测到所述扩增产物则指示样品包含待检测的靶rna序列。
11、如本文将更详细地描述的,步骤a)和b)在单一腔(receptacle)/容器(container)或两个分开的腔/容器中进行。
12、在一些教导中,第一和第二温度是相同的温度。
13、靶rna序列可以是任何合适的rna序列,包括人rna序列,并且可以包括mrna、rrna、sirna、hnrna、pirna、arna、mirna、来自感染原的rna和合成的rna分子等。在一教导中,靶rna序列来自感染原,并且可以采用本文描述的方法来确定样品是否含有感染原或来自感染原的rna。因此,在一些教导中,例如,所述方法可以用于检测对象是否被感染原感染,或者某一基质是否被感染原污染。
14、此外,对于一些整合型病毒(能整合至宿主基因组的病毒),例如人乳头瘤病毒(hpv),检测转录的mrna可能比检测病毒dna更好。这是因为,就hpv而言,高危hpv的e6/e7致癌基因的表达对于发育异常表型的发展和维持是必需的。因此,e6/e7 mrna的检测可提供比简单检测hpv dna更好的预后评估。因此,根据本公开,可检测在细胞中转录的hpv和其它整合型病毒的mrna。实例包括爱泼斯坦-巴尔病毒(ebv);肝炎,例如乙型肝炎病毒(hbv)和丙型肝炎病毒(hcv);腺相关病毒-2(aav-2);和逆转录病毒,例如hiv和内源性逆转录病毒(erv1)
15、感染原可以是例如细菌、真菌或病毒。在某些教导中,感染原是单链或双链rna病毒、或基因组整合型病毒,其中其一个或多个基因由宿主细胞转录。这样的教导尤其适合于单链rna病毒的检测,例如包括正链和负链rna病毒。
16、正链rna病毒分为黄色病毒门(kitrinoviricota)、光滑裸露病毒门(lenarviricota)和小核糖病毒门(pisuviricota)(具体分类为小南嵌套病毒纲(pisoniviricetes)和(stelpavirictes)),所有这些病毒都属于正核糖病毒界(orthornavirae)和核糖病毒域(riboviria)。它们是单系的,源自共同的rna病毒祖先。
17、正义rna病毒占已知病毒的很大一部分,包括许多病原体,例如丙型肝炎病毒、西尼罗河病毒、登革热病毒以及sars、mers和sars-cov-2冠状病毒,以及临床上较不严重的病原体,例如引起普通感冒的鼻病毒。
18、示例性正链rna病毒包括:
19、1.大麦黄花叶病毒属(bymoviruses)、豇豆花叶病毒属(comoviruses)、线虫传多面体病毒属(nepoviruses)、诺达病毒(nodaviruses)、微小核糖核酸病毒(picornaviruses)、马铃薯y病毒属(potyviruses)、南方菜豆花叶病毒(sobemoviruses)和变黄病毒属(luteoviruses)(甜菜黄化病毒和马铃薯卷叶病毒)的子集——微小核糖核酸样组(picornavirata)。
20、2.香石竹斑驳病毒属(carmoviruses)、香石竹环斑病毒属(dianthoviruses)、黄病毒(flaviviruses)、瘟病毒属(pestiviruses)、(statoviruses)、番茄丛矮病毒属(tombusviruses)、单链rna噬菌体、丙型肝炎病毒和黄体病毒的一个子集(大麦黄矮病毒)——黄病毒样组(黄病毒属)。
21、3.甲病毒属(alphaviruses)、卡拉病毒属(carlaviruses)、糠病毒属(furoviruses)、大麦病毒属(hordeiviruses)、马痘病毒属(potexviruses)、红病毒属(rubiviruses)、托布拉病毒属(tobraviruses)、三角病毒属(tricornaviruses)、芜青黄花叶病毒属(tymoviruses)、苹果褪绿叶斑病毒(apple chlorotic leaf spot virus)、甜菜黄化病毒和戊型肝炎病毒——α样组(rubivirata)。
22、示例性负链rna病毒包括:丁型肝炎病毒和负核糖病毒门(negarnaviricorta)病毒,包括简单病毒亚门(haploviricotina)和复杂病毒亚门(polyploviricotina)病毒。众所周知的简单病毒亚门病毒包括例如埃博拉病毒、马尔堡病毒、麻疹病毒、流行性腮腺炎病毒、rsv病毒和狂犬病病毒。众所周知的复杂病毒亚门包括拉沙病毒和流感病毒。
23、本发明在本文中可以具体地在检测冠状病毒和covid-19方面进行例证,但这不应被解释为形成限制,并且本领域技术人员容易理解且能够将本文教导应用于其它rna靶标的检测。
24、包含靶rna序列的任何类型的样品均可以用于本文公开的方法或与本文公开的试剂盒一起使用。因此,含有或疑似含有靶rna序列的样品不具体限于并且包括,例如源自活体对象的生物样品,例如全血、血清、血沉棕黄层、尿液、粪便、脑脊液、精液、唾液、组织(例如肺组织)、细胞培养物(例如哺乳动物细胞培养物或细菌培养物);含有rna的样品,例如类病毒、病毒、细菌、真菌、酵母、植物和动物;含有病毒或细菌等微生物或被病毒或细菌感染的样品(例如食品和生物制剂);含有生物物质的样品,例如土壤、工业过程和制造设备和废水;和源自各种水源(例如饮用水)的样品。样品还可以是拭过固体表面的拭子,例如疑似含有感染原或被感染原污染的固体表面。
25、此外,可以通过任何已知的方法处理样品以制备在本文公开的方法中使用的含有rna的组合物。此类制备的示例包括细胞破裂物(例如细胞裂解物和提取物)和样品分级分离物。样品可以是单一样品,或者可以是多个样品(例如2、3、4、5、6、8、10或更多个样品),其被汇集在一起并在单一测试中进行测试。
26、如本文进一步描述,发明人已鉴定到某些dna限制性核酸内切酶能够识别dna/rna双链体(即使它们的正常/常规靶标是dna/dna双链体),并且能够使dna/rna双链体的dna链产生切口。本发明的结合物dna和/或引物分子必须能够在第一温度特异性结合靶rna序列,并且该限制性核酸内切酶能够在识别序列(recognition sequence)处使dna/rna双链体的dna链产生切口。结合物dna/引物分子与靶rna序列互补,或包含与靶rna序列互补的序列部分,使得结合物dna/引物能够在第一温度结合靶rna。然而,结合物dna/引物在第一温度不能结合模板dna,或者至少不能结合模板dna并由此起始链延伸。
27、在另一方面中,提供了用于特异性结合rna模板的结合物dna分子,所述结合物dna分子包含:
28、与靶rna分子互补且包括dna限制性核酸内切酶识别位点的部分(portion),该位点在结合物dna存在于dna/rna双链体中时能被限制性内切核酸酶产生切口;当与靶rna分子互补的上述部分在第一温度与靶rna分子结合时,不与靶rna分子互补且不与靶rna分子结合的另一部分(further portion)。
29、如本文中述及,在一项教导中,本公开涉及covid-19的检测,并且在这方面,该教导提供了结合物dna序列,其根据本教导为covid-19检测应用而特别设计。
30、因此,在covid-19检测应用的一项教导中,结合物dna分子可以基本上由以下序列组成,或由以下序列组成:
31、5’-agg gta aac caa ata cct ggt gta tac gtt-3’,
32、5’-agg gtt aaa cca ccg cct gga gat caa ttt-3',或
33、5’-agg gtc ctt aac ttg cct ggt tgt gat ggt-3’。
34、在一项教导中,结合物dna分子基本由以下序列组成,或由以下序列组成:
35、5’-agg gta aac caa ata cct ggt gta tac gtt–3’。
36、在本文中,基本由...组成指一个或多个确定的核苷酸,例如3'核苷酸,可经化学修饰,只要这本质上不影响该序列结合至其靶rna序列的能力即可。
37、在一项教导中,结合物dna分子包含能在第一温度结合靶rna序列的部分,和在第一温度不结合靶rna的另一部分。结合靶rna序列的部分还包括dna限制性核酸内切酶的识别序列,该dna限制性核酸内切酶能够识别dna/rna双链体并在其识别序列处使dna链产生切口。选择第一温度以限制或防止结合物dna/引物分子与非靶rna序列的任何非特异性结合,并因此确保结合物dna/引物仅结合其特异性靶标。就此可能希望在一些情况下,结合靶rna序列的所述部分从与其靶rna序列结合的角度而言可以是完全互补的。然而,在一些情况下,结合至靶rna的结合物dna/引物的部分可能是不完全互补的,并且可以容许多个错配(例如在第一温度能够结合靶rna的结合物dna/引物序列的部分的长度上,有1、2、3或4个错配)。此类错配可以在包括dna限制性核酸内切酶的识别序列的区域中、在不包括dna限制性核酸内切酶的识别序列的区域中,或在这两种区域中。在一项教导中,所有错配可仅存在于不包括识别序列的区域中。
38、结合物dna/引物分子的长度通常可以是20–50个核苷酸,例如25–40个核苷酸。当存在时,不结合靶rna序列的区域的长度可以是,例如2–8个核苷酸,例如3–6个核苷酸。不结合靶rna分子的区域可位于结合物dna的5’末端。然而,结合物dna分子及其靶rna结合区和非结合区的长度也可以以功能性方式确定。就此而言,结合物dna必须能够在第一温度结合至其靶rna序列,由其衍生的带切口的dna分子在第一温度从靶rna分子释放。因此,结合物dna/引物分子在第一温度结合至靶rna分子,而带切口的dna分子不结合靶rna分子。
39、结合物dna可包含化学修饰,例如本领域通常已知的化学修饰。在一些实施方式中,例如,结合物dna可包含经化学修饰的核苷酸(例如,2'-o甲基衍生物、硫代磷酸酯等)、3’末端修饰、5’末端修饰或其任何组合。在一些实施方式中,结合物dna的3’末端可被修饰,使得延伸反应不会或不能从结合物dna的3’末端发生(例如,在结合至靶rna序列或另一非靶标序列时,可作为聚合酶延伸的引物)。从结合物dna的3’末端开始的任何复制都可能导致检测错误(例如假阳性)。因此,在一些教导中,结合物dna包含3’末端修饰,其可以减少或消除任何不期望的延伸反应的发生,例如上述讨论的那些。3'-末端修饰的非限制性实例包括tamra、dabcyl和fam的添加。修饰的其他非限制性实例包括,例如生物素化、荧光染色、磷酸化、硫醇化、胺化或经修饰/倒置核苷酸,其在利用dna聚合酶的常规dna延伸反应中不太可能让其它核苷酸添加至结合物dna分子的3'末端。
40、使用本领域已知的技术(例如,参见参考文献8),本领域技术人员能够计算相应靶rna序列的任何特定dna序列的解链温度,以及带切口的dna序列的解链温度。选择的第一温度是预期使结合物dna结合的温度,第一温度低于结合物dna与靶rna的解链温度,而且使限制性核酸内切酶和适当时逆转录酶能够发挥作用。温度过高,限制性核酸内切酶/逆转录酶可能无法发挥作用(例如,酶可能会变性),温度过低,切口反应和/或链式延伸反应可能会变得过慢。
41、在其中结合物dna包含靶rna结合部分和不结合靶rna的部分的教导中,所选择的第一温度将低于结合物dna与靶rna序列的解链温度,但高于带切口的dna分子的解链温度,使得在产生切口后,所述带切口dna分子能从靶rna分子释放。本领域读者能够容易地考虑到跨越结合物dna长度的任何错配和不结合靶rna分子的区域对解链温度的影响,并应用本领域读者的常识而相应地设置第一温度。
42、通过对结合物dna分子的适当设计,考虑其长度、错配、识别序列的位置和不结合靶rna分子的区域的长度,可以设计结合物dna分子以使得其对靶rna分子的解链温度为/可以在45-70℃的范围内,并且带切口的dna分子的解链温度为/可以在30-45℃的范围内。选择的第一温度低于结合物dna分子的解链温度,但高于带切口的dna分子的解链温度。因此,例如,如果结合物dna分子与互补核酸分子的解链温度经计算为60℃并且带切口的dna分子的解链温度经计算为35℃,那么第一温度可以例如在45℃–55℃之间,以确保结合物dna分子结合靶rna分子,而带切口的dna不结合且从靶rna分子释放。
43、带切口的dna序列包含不结合至(因此非互补于)靶rna序列的序列区域,以及与rna靶标互补并且在发生切口反应之前结合至rna靶标的区域。
44、根据本文所述的方法,其利用引物和逆转录酶,所述引物与其靶rna序列互补,因此不包括不不期望结合其靶rna序列的部分。然而,不同于涉及利用包含靶rna结合区域和非结合区域的结合物dna并且不利用逆转录酶的教导,带切口的dna序列可能不会简单地因为第一温度高于带切口dna分子与其靶rna序列的解链温度而从其靶rna序列释放。在这种情况下,逆转录酶和/或其它加工酶可以促进带切口的dna分子从靶rna序列释放。
45、如上所述,带切口的单链dna分子的长度可以取决于结合物dna分子的设计。然而,带切口的单链dna分子的长度通常是10-30个核苷酸,例如长度是12-24个核苷酸,或14-20个核苷酸,或在上限值和下限值之间的任何整数或范围,例如长度是10-20个,或14-30个核苷酸。
46、根据本公开内容设计结合物dna的通用方法可以涉及首先寻找dna序列中的所有bstni、avrii或phoi限制性位点,所述dna序列将与潜在的靶rna互补。一旦确认具有此类限制性位点的结合物dna分子可能会结合的合适的靶区域,则进行以下步骤中的一个或多个(例如至少两个、三个或四个):
47、1)从任何可能的结合物dna序列中,排除具有多个bstni、avrii或phoi限制性位点(例如两个、三个、四个或更多个限制性位点)的序列以及包含多聚a'的序列;
48、2)设计在5'末端带有aggg的结合物dna序列;
49、3)如果鉴定到多于一个可能的结合物dna序列,则确定可能的结合物dna序列的tm值并去除具有最高和最低tm值的序列;
50、4)评估结合物dna分子形成切口后将产生的带切口dna/触发序列(triggersequence)的3'末端,以查看碱基ccac是否存在。选择除结合物dna序列外还带有ccac最优匹配的序列,其在步骤3后具有最高和最低的tm。
51、dna限制性核酸内切酶可以是能够使dna/rna双链体的dna链产生切口或优先使dna/rna双链体的dna链产生切口的任何限制性核酸内切酶。“优先产生切口”是指在dna/rna双链体中,相比rna链,以更大程度使dna链产生。更大程度是指相对于rna链而言,大于60%、70%、80%、90%或95%。bstni就是一种此类dna限制性核酸内切酶。murray等nucleic acids research,2010年,第38卷,第22期8257–8268表明一些限制性核酸内切酶可以切割rna:dna异源双链体。例如,bstni(切割位点cc/wgg,其中w=a或t)仅有效切割rna/dna异源双链体的dna链。这意味着根据本教导,酶(如bstni)可用于切割dna/rna异源双链体以产生切口的dna分子。至关重要的是,限制性核酸内切酶不具有进行性,因此只需要一次反应即可产生片段。例如,在生成用于扩增反应的dna片段之前,需要逆转录酶(rt)催化形成多个磷酸二酯键。这使得核酸内切酶反应(例如bstni反应),本质上比rt反应更快。能够切割rna/dna双链体的dna链的其他限制酶包括avrii、ndel和phoi。参见例如:http://rebase.neb.com/cgi-bin/hybridlist和参考文献14。
52、在带切口的dna分子从靶rna分子释放后,带切口的dna分子结合至/结合更长的模板dna分子,并且能够在dntp和dna聚合酶存在的情况下起始链延伸并产生扩增产物。设计模板分子使得带切口的dna分子能够与其特异性结合并起始链延伸。在一种教导中,扩增产物通过本领域已知的expar技术产生。9-11
53、示例性扩增反应可以快速合成由模板dna分子内的序列指定的短寡核苷酸(例如,8-16个碱基)。本领域技术人员已知的反应形式可以用线性或指数扩增反应(expar)进行。另一个实例是滚环扩增(rca)和环介导等温扩增(lamp)。这两个反应都利用简单、稳定的等温(即非热循环)条件。也可以采用热循环反应,例如聚合酶链式反应(pcr)及其形式。扩增的速率完全取决于控制反应中分子相互作用的分子参数。该方法的指数形式是分子链式反应,其可以利用各线性反应的寡核苷酸产物以产生更多相同带切口的dna分子,以用于后续轮数扩增并产生扩增产物。
54、带切口的dna分子可以结合至模板dna的3'末端或末端区域并扩增模板dna分子的5'末端或末端区域。模板dna分子的长度通常小于100个核苷酸,例如长度小于70、60或50个核苷酸,以及其间的任何整数。在一些情况下,如本文将更详细描述的,模板dna分子比带切口的dna分子更长,并且其长度可以是带切口的缩短的单链dna分子长度的至少两倍。
55、至少在一个腔/容器实施方式中,其中步骤a)和b)在单一腔/容器中进行,例如eppendorf管、多孔板的孔等,带切口的dna分子与模板dna分子的解链温度将高于对于靶rna分子的解链温度。因此,可选择第二温度,使带切口dna分子将能够结合模板dna分子并能够起始链延长,但不能结合靶rna分子。在一项教导中,第一温度和第二温度相同,但情况不总是如此。例如,如果第一温度为55℃,并且带切口的dna分子与靶dna分子的解链温度在30℃–45℃之间,那么带切口的dna分子与模板dna分子的解链温度可以例如在55℃–65℃之间。既然如此,第二温度可以是例如低于计算出的模板dna与带切口dna的解链温度的温度(例如40℃-50℃)。
56、在一个实施方式中,扩增产物的产生是通过用于扩增dna的等温反应进行的,例如lamp或expar。等温扩增方法以简化、指数方式提供核酸靶序列的检测,并且不受热循环的限制,例如当采用聚合酶链式反应(pcr)时。许多等温技术对于本领域技术人员来说是已知的,但是它们都有一些共同的特征。例如,由于dna链不会热变性,所有等温方法都依赖于另一种手段以实现引物结合并起始扩增反应:具有链置换活性的聚合酶。一旦反应开始,聚合酶必须同样分离仍与感兴趣序列退火的链。
57、等温方法通常采用独特的dna聚合酶来分离双链体dna。具有这种能力的dna聚合酶包括用于中等温度反应(25–40℃)的klenow exo-、bsu大片段和phi29以及用于较高温度(50–65℃,例如50℃、55℃或60℃)反应的bst dna聚合酶的大片段。根据本教导的方法可以在等温或基本恒定的温度条件下进行。在涉及在基本恒定的温度下执行该方法的实施方式中,允许有一些温度的波动。例如,在一些实施方式中,基本恒定的温度可以在期望的或确认的目标温度范围内波动(例如,约+/-2℃或约+/-5℃)。在实施方式中,基本恒定的温度可以包括不包括热循环的温度,例如在进行pcr时采用的温度。在一些实施方式中,第二温度等于或低于模板dna分子的计算/预测或实验确定的最佳杂交或解链温度,例如5℃-10℃或更低于模板dna分子的大约计算/预测或实验确定的最佳杂交温度或的解链温度。在一些实施方式中,为了方便起见,第一温度和第二温度相同或基本相同(例如,约+/-2℃或约+/-5℃)。
58、在一个实施方式中,本教导利用链置换扩增(sda)或切口酶扩增反应(near)。这两种技术都依赖于链置换dna聚合酶,通常是来自大肠杆菌的bst dna聚合酶、bsu大片段或klenow片段,以在模板dna分子所含位点上由链限制的限制性核酸内切酶或切口酶产生的切口处起始。各聚合酶置换步骤都会再生成切口位点,导致指数扩增。near极其快速且灵敏,能够在数分钟内检测到低量的靶标。
59、一种此类合适的切口酶是nt.bstnbi。nt.bstnbi是仅切割双链dna底物中一条dna链的位点特异性核酸内切酶。切口核酸内切酶在识别序列3'侧外4个碱基处催化单链断裂。合适的切口酶的其他非限制性实例包括nb.bbvci、nt.alwi、nt.bbvci、nb.bsrdi、nb.btsl、nt.bspqi、nb.bsmi、nt.cvipii和nt.bsmai。
60、本文描述的方法可以用任何合适的形式进行,例如包括在单一腔中,例如eppendorf或其他管,或者以多重形式进行,例如多孔板。可以设想与或不与相关读取器一起应用的另外的管盒(cartridge)形式,例如基于侧流或基于微流体的管盒。
61、扩增产物可以通过本领域已知的技术检测,例如发光、uv或可见光谱或度谱术、荧光光谱或度谱术、质谱、液相色谱、荧光偏振、电化学、电泳、酶标记(例如过氧化物酶或碱性磷酸酶)、荧光标记(例如荧光素或罗丹明)、化学发光、生物发光、表面等离子共振(spr)或荧光团修饰的探针dna(例如taqman探针),裸眼视检,通过合适的发色/颜色变化或生成(例如通过使用合适的标签或染料修饰的探针或核酸)或上述技术的组合。
62、通常,从使结合物dna与靶rna序列接触到检测足够的(例如,与基线值(通常获自试验开始时,在任何信号产生之前)相比大于或等于5、6、7、8、9或10标准偏差的可检测信号)扩增产物的总时间可少于15分钟、少于10分钟、少于9、8、7、6甚至5分钟。
63、expar速度的关键有两个:首先,扩增在一个温度下进行,从而避免了冗长的加热和冷却步骤,其次扩增的序列相对较小(通常为15-20个碱基长度)。这两个因素导致一旦引发expar,只需几分钟即可产生约109dna链产物。9,12根据本文教导,带切口的单链dna分子(触发物)通过与模板dna分子相互作用来启动expar反应。然后在等温循环(涉及用以延伸序列的dna聚合酶,和用以切割序列的切口核酸内切酶)中生成大量短双链dna序列,同时保持模板完整(参见方案1a)。与qpcr试验一样,可以通过光谱监测双链体形成,例如通过使用荧光染料,例如sybr green ii。13
64、如本文所述的结合物dna和模板dna分子可以经合成制备,并且可以使用本领域已知的亚磷酰胺法、磷酸三酯法、h-膦酸酯法或硫代膦酸酯法来合成。合成后,可以纯化(例如使用离子交换hplc)结合物dna和模板dna分子。
65、术语“3’(末)端”和“5’(末)端”在本文中用于描述单链核酸内特定位点的位置。当核酸中的位置是参考核苷酸或参考核苷酸序列的“3'”或“3'处”时,这意味着该位置在参考核苷酸或参考核苷酸序列的3'末端与该核酸链的3'羟基之间。同样地,当核酸中的位置是参考核苷酸或参考核苷酸序列的“5'”或“5'处”时,这意味着该位置在参考核苷酸或参考核苷酸序列的5'末端与该核酸链的5'端磷酸之间。
66、如本文所用,“切口”是指在相对于产生切口的酶所识别的核苷酸序列的特定位置上,仅切割完全双链核酸分子的一条链或部分双链核酸分子的双链部分的一条链。核酸产生切口的特定位置被称为“切口位点”(ns)。
67、例如,示例性双链dna切口核酸内切酶n.bstnb i的识别序列和切口位点如下所示,“▼”表示切割位点,n表示任何核苷酸:
68、▼
69、5'-g a g t c n n n n n-3'
70、3'-c t c a g n n n n n-5'
71、nt.bstnbi识别序列的正义链序列是5’-gagtc-3’,而反义链序列是5’-gactc-3’。
72、“起始链延伸”,是指带切口的dna分子结合至模板dna结合时,带切口的dna分子充当引物,并向带切口的dna分子的3’端添加核苷酸,该核苷酸与存在于模板dna上的下一个碱基互补。以这种方式,带切口dna的长度随着每次向带切口dna分子延伸的3’端添加其它的核苷酸而增长或延伸。这也适用于引物结合至靶rna序列并且使用逆转录酶以产生靶rna的反向互补dna序列的教导。
73、术语“扩增产物的产生”是指使用带切口的dna分子作为引物,其包含与模板dna的5'末端或区域互补的序列,制作多于一个拷贝的模板dna分子或其部分的过程。
74、如本文所用,短语“扩增产物”是指包含特定核酸分子的一个或多个拷贝的产物。通常,扩增产物包含核酸分子的至少103、104、105、106、107、108、109或更多个拷贝。
75、在另一方面,提供了一种试剂盒,其包含,基本由以下组成或由以下组成:
76、如本文所定义的结合物dna分子;和
77、限制性核酸内切酶,其在结合至靶rna分子时能够在限制性核酸内切酶的识别序列处使结合物dna序列产生切口。
78、在covid-19检测应用的一项教导中,结合物dna分子基本由以下序列组成,或由以下序列组成:
79、5’-agg gta aac caa ata cct ggt gta tac gtt-3’,
80、5’-agg gtt aaa cca ccg cct gga gat caa ttt-3',或
81、5’-agg gtc ctt aac ttg cct ggt tgt gat ggt-3’。
82、在一项教导中,结合物dna分子基本由以下序列组成,或由以下序列组成:
83、5’-agg gta aac caa ata cct ggt gta tac gtt–3’。
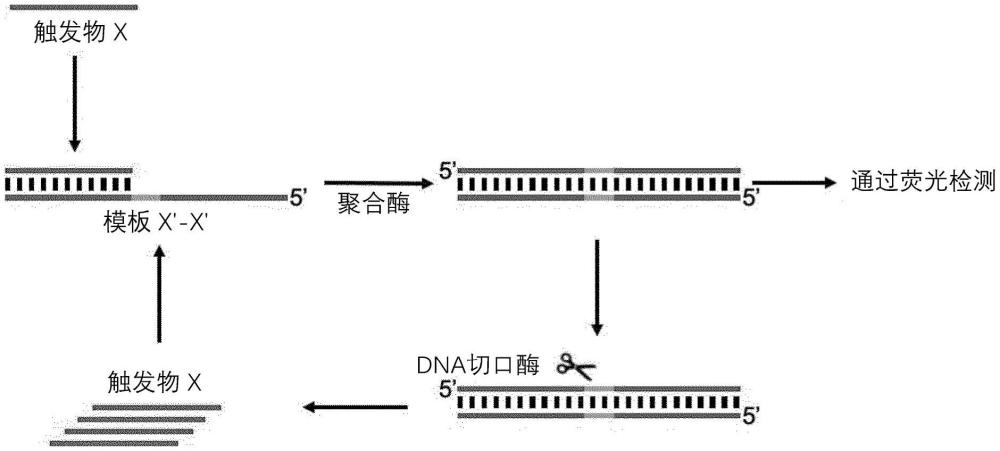
84、在本文中,基本由...组成指一个或多个确定的核苷酸,例如3’核苷酸,可以被化学修饰,只要这本质上不影响该序列结合至其靶rna序列的能力即可。
85、在一项教导中,限制性核酸内切酶是bstni。
86、所述试剂盒还可以任选地包括:
87、模板dna分子,其能与带切口的dna分子结合并起始链延伸,以在(任选地加标签或染料标记的)dntp和dna聚合酶存在下产生扩增产物。该试剂盒进一步包括(任选地加标签或染料标记的)dntp和/或dna聚合酶,例如bst dna聚合酶、大片段或klenow片段(3’-5’exo–)和任选的dna切口酶,例如nt.bstnbi。
88、在一些实施方式中,方法和/或试剂盒可进一步包含其它试剂。根据本文的教导可使用的其它试剂的一些非限制性实例,包括金属盐,例如氯化钠、氯化镁、乙酸镁和硫酸镁;底物,例如dntp混合物;缓冲液,例如tris-hcl缓冲液、n-三(羟甲基)甲基甘氨酸(tricine)缓冲液、磷酸钠缓冲液和磷酸钾缓冲液。同样地,去污剂、氧化剂和还原剂也可用于实施本文公开的方法和/或dna嵌入染料,例如sybr green i,或可以进一步提供被设计为结合至任何扩增产物的加标签或染料标记的探针。
89、现在将通过示例并参考附图的方式来描述本公开的实施方式,其中:
90、图1示意性地显示了(a)指数扩增反应。触发物x与模板x’-x’退火,并通过dna聚合酶(bst 2.0聚合酶)延伸。然后,当前双链体dna的顶链被切口酶(nt.bstnbi)切割。释放的dna与触发物x相同,因此能够引发另一个模板x’-x’。(b)无逆转录酶expar。结合物dna与病毒rna退火。dna:rna异源双链体的dna被限制性核酸内切酶bstni切割,bstni作为一种切割酶,只切割dna。释放的dna链是触发物x,并被expar放大;(c)逆转录酶引物与病毒rna退火,并通过逆转录酶(superscript iv逆转录酶)转化为cdna。rna:dna异源双链体的形成为限制性核酸内切酶(bstni)生成了识别位点以切割dna链。切割的cdna链包含触发物x序列,该序列引发模板x’-x’且该序列被延伸以新形成完整双链体。然后,当前双链体dna的顶链被切口酶(nt.bstnbi)切割。释放的dna是触发物x,并因此能够引发另一个模板x’-x’。
91、图2更详细地显示了图1的实例,其具有covid-19靶rna序列、能结合靶rna的结合物dna序列、带切口的dna序列和模板dna序列的特定序列;
92、图3是显示扩增触发物x和空白后,标准化荧光单位相对于时间的曲线。
93、图4是显示扩增触发物a和空白后,标准化荧光单位相对于时间的曲线。
94、图5是显示扩增触发物b和空白后,标准化荧光单位相对于时间的曲线。
95、图6是显示扩增浓度范围为10nm–100fm的触发物x和空白后,标准化荧光单位相对于时间的曲线。
96、图7是显示了扩增浓度范围为10nm-100fm的触发物x和空白后,荧光信号大于基线上10标准偏差的时间的柱状图。
97、图8是显示使用模板x’-x’在扩增触发物x、触发物a、触发物b和空白后,标准化荧光单位随时间变化的曲线。
98、图9是显示使用模板x’-x’在扩增触发物x、触发物a、触发物b和空白后,荧光信号大于基线上10标准偏差的时间的柱状图。
99、图10是显示在两锅式逆转录酶expar后,患者阳性和阴性样品的的标准化荧光单位相对于时间的曲线。
100、图11是显示在两锅式逆转录酶expar之后,患者阳性和阴性样品的荧光信号大于基线上10标准偏差的时间的柱状图。
101、图12是显示患者阳性和阴性样品的一锅式逆转录酶expar后的标准化荧光单位相对于时间的曲线。
102、图13是显示在一锅式逆转录酶expar之后,患者阳性和阴性样品的荧光信号大于基线上10标准偏差的时间的柱状图。
103、图14是显示在两锅式逆转录酶expar之后,患者阳性和阴性样品的标准化荧光单位相对于时间的曲线。
104、图15是显示在两锅式逆转录酶expar之后,患者阳性和阴性样品的荧光信号大于基线上10标准偏差的时间的柱状图。
105、图16是显示在一锅式逆转录酶expar后,患者阳性和阴性样品的标准化荧光单位相对于时间的曲线。
106、图17是显示在一锅式逆转录酶expar之后,患者阳性和阴性样品的荧光信号大于基线上10标准偏差的时间的柱状图。
107、图18显示了来自观察患者样品的covid19 orf1ab基因的结果a)患者验证样品的rtf-expar试验数据,显示了rtf-expar产生信号的时间。红色柱代表阳性样品,蓝色柱代表阴性样品,黄色虚线代表6分钟的截止时间。各运行时间计算为荧光信号大于基线信号上10标准偏差的点(10-σ时间);和b)显示患者样品验证研究结果的混淆矩阵。
108、图19显示了来自针对患者样品的covid19 n基因的结果a)患者验证样品的rtf-expar试验数据,显示了rtf-expar产生信号的时间。红色柱代表阳性样品,蓝色柱代表阴性样品,黄色虚线代表6分钟的截止时间。各运行时间计算为荧光信号大于基线信号上10标准偏差的点(10-σ时间);和b)显示患者样品验证研究结果的混淆矩阵。
109、图20是显示在一锅式逆转录酶expar后,2ng/μl hpv mrna和阴性对照的标准化荧光单位相对于时间的曲线。
110、材料
111、所有实验均使用经millipore elix-gradient a10系统(电阻率>18μω.cm,toc≤5ppb,密理博公司(millipore),法国)纯化的milli-q水。nt.bstnbi、bstni和bst 2.0聚合酶获得自新英格兰生物实验室公司(new england biolabs)(希钦,英国),缓冲液也是如此,10x等温扩增缓冲液(200mm tris-hcl,100mm(nh4)2so4,500mm kcl,20mm mgso4,1%tween 20,ph 8.8),其用于所有实验。superscript iv逆转录酶获得自赛默飞世尔公司(thermofisher)(佩斯利,英国),dmso(>=99%)获自飞世尔科技公司(fisherscientific)(拉夫堡,英国),dsgreen 100x(sybr green i的类似物)获自lumiprobe公司(汉诺威,德国)。牛血清白蛋白(bsa,用水稀释至4mg/ml)和单链结合蛋白(ssb,在20mmtris-hcl中的0.5mg的溶液,ph 8.0,0.5m nacl,0.1mm edta,0.1mm dtt,50%甘油)购自西格玛奥里奇公司(sigma-aldrich)(多塞特郡,英国)。所有三磷酸核苷酸和寡核苷酸序列(脱盐)均获得自西格玛奥里奇公司(多塞特郡,英国)。sars-cov-2rna和cdna患者样品(在magna pure洗脱缓冲液中)获得自英国公共卫生局(phe),并保存在-80℃。
112、phe样品:所有临床试样均在2级生物安全实验室(containment level2laboratory)中处理。为了制备各样品,将来自鼻咽拭子的病毒运输培养基(vtm,300μl,medical wire virocult)以1:1的比率添加至缓冲液al(恰根)(qiagen)中,并在校准的加热模块中加热60℃,持续30分钟。然后在magnapure96(罗氏)(roche)自动提取系统上提取样品,然后在abbott m2000 rt-qpcr测试上运行以进行sars-cov-2rna检测。对于expar试验开发,将来自sars-cov-2rna试验的阳性和阴性样品分开合并在magna pure洗脱缓冲液中(使合并的阳性样品得到29,080个rna拷贝/μl)。从phe收到后,各样品都稀释400倍,等分到50μl小瓶中并储存在-80℃。使用前,将各样品浸入冰中并使其缓慢融化;一旦熔化,样品立即使用,然后再次冷却并储存在-80℃。
113、表1:用于expar期间的寡核苷酸。
114、
115、
116、要素:
117、–非结合片段
118、高亮–bstni识别位点(与rna靶标中所需的5’-ccagg-3’序列互补)
119、粗体–触发物x序列
120、数据分析与分类
121、为了分析expar实时荧光扩增曲线,我们开发了一个c#程序来进行荧光数据分析。该程序分析前20个数据点并计算平均值和标准偏差作为基线。生成这两个值后,将分析各后续数据点以确定其值减去平均值是否大于平均值的10标准偏差。将满足该条件的循环换算成时间,作为最小扩增时间。如果该时间少于20分钟,则输出为true,表明存在sars-cov-2rna,20分钟之后将生成false输出,表明不存在sars-cov-2rna。
122、实施例
123、开发成功的expar试验的关键要素是确认靶基因组中的最佳核苷酸序列。
124、qian等于先前发现expar中使用的特定触发物序列在决定其成功方面发挥着至关重要的作用。12我们采用他们的手段,为expar设计了一个17聚体dna触发物序列(触发物x,图1a)以与sars-cov-2基因组中的orf1ab14序列互补。
125、结合物dna序列的设计必须使其能够:
126、1)具有足够的碱基配对相互作用,以与rna上的相关序列产生稳定的复合物
127、2)生成能被相关酶识别并产生切口的序列
128、3)包含非互补序列(例如在5’末端),使其能够与dna模板x’-x’序列退火以进行下一阶段的反应
129、为了实现这一目标,我们设计了一个结合物dna序列,在5’末端包含“尾部(tail)”,该“尾部”不与病毒rna互补,但与模板x’-x’序列互补。必须设计“尾部”的长度和序列,使其不会形成可能会抑制与rna结合或在没有rna的情况下触发expar反应的任何二级结构。“尾部”序列的序列还必须提供足够的能用于结合至模板x'-x'的结合能量,以确保形成足够稳定的双链体,以允许聚合酶催化与模板x’-x’互补的链延伸(参见图1b)。
130、尾部的序列还需要确保一旦其从dna:rna异源双链体释放出来,expar反应就可以继续进行。示例性尾部序列是根据qian等的教导开发的。然而,简单来说,该过程涉及以下步骤:
131、1)在感兴趣区域中的所有bstni限制性位点均在5’和3’末端发现有10个碱基。
132、2)从这些序列中,消除了具有多个限制性位点和多聚a的序列
133、3)从这些剩余序列中,添加aggg至5’末端(因为这些碱基已被证明对expar很重要(qian等))
134、4)分析这些结合物序列的tm值,并移除具有最高和最低tm值的序列
135、5)我们选择的序列是具有高和低tm的序列以及在3’末端具有最多碱基ccac的序列。(qian等的教导)。
136、更详细地说,图2确认了特定covid-19病毒靶rna序列(a)和设计为结合该靶rna序列的结合物dna序列(b)。如图所示,结合物dna包含与靶rna序列互补的3’端部分,并包含bstni识别序列(cctgg),以及非互补的5’末端。在用bstni使结合物dna产生切口后,带切口的dna(标记为触发物x)从靶rna释放。带切口的dna结合至模板dna(标记为模板x’-x’),并能够起始链延伸并生成扩增的dna/dna双链体模板x’-x’。模板dna的序列设计为在模板两端都有重复序列,中间有间隔区,该间隔区有nt.bstnbi的识别序列。nt.bstnbi的作用在dna/dna双链体的顶链上产生切口,聚合酶分子能够从形成切口的位点继续延伸,置换可结合模板x’-x’分子的触发物x分子的拷贝,并起始另一轮扩增。以这种方式,可以发生扩增的dna/dna双链体模板x’-x’分子的指数生成,并且可以通过例如使用能够结合dna/dna双链体分子的染料来检测增加数量的此类dna/dna双链体分子的生成。
137、在图1c中,确认了特定covid-19rna靶序列,引物序列退火并使用逆转录酶延伸。bstni识别序列(cctgg)上的延伸允许dna链被转录酶切割和被链置换活性置换。切割的dna链与模板x’-x’互补,模板x’-x’由bst聚合酶延伸以形成模板x’-x’的dna/dna双链体。nt.bstnbi的识别序列使dna/dna双链体的顶链能够产生切口,置换触发物x分子的拷贝,该触发物x分子可以结合模板x’-x’分子并起始expar。
138、实施例1:确定最合适的触发物序列。
139、该实验针对其各自的模板序列测试了为此过程设计的三个触发物,以确定提供特异性和非特异性扩增之间最大间隔的序列。
140、方法
141、a部分:
142、1.50μl水,2.50μl 10x等温扩增缓冲液,3.75μl bsa溶液、1.50μl bst 2.0dna聚合酶(1.6u/μl),0.75μl nt.bstnbi(10u/μl)。
143、b部分:
144、5.55μl水,5.00μl 10x等温扩增缓冲液,1.50μl模板(1μm),2.40μlmgso4(100mm),1.50μl dntp(10nm),0.75μl dsgreen(在dmso中1:5稀释,从100x到20x),0.30μl ssb溶液。
145、c部分:
146、100nm(触发物x或触发物a或触发物b)的3μl的一种触发物
147、添加步骤:将b部分(17μl)添加至在4℃冷却的pcr管中。向其中添加c部分并在4℃下保持5分钟,然后添加a部分(10μl)。然后将管密封,接着对内容物进行扩增。
148、扩增步骤:使用设置为55℃恒温的agilent mx3005p实时pcr系统(迪德科特,英国)进行等温孵育和荧光信号测量。在至少20分钟的孵育时间内每10秒进行荧光测量。
149、结果
150、实验结果如图3–5所示。图的x轴显示时间。所有测试均显示含有触发物的样品在2分钟内被扩增。非特异性扩增所需的时间因触发物而异,触发物x需要多于9分钟的时间信号才会增强。基于非特异性样品的荧光扩增,选择触发物x作为最成功的触发物并继续使用。
151、实施例2:确定expar序列的灵敏度。
152、使用触发物x和模板x’-x’进行优化步骤,从而导致阳性扩增和阴性扩增之间的间隔更大。模板浓度从50nm降低至25nm,反应温度从55℃降低至50℃。使用该改良过程,使用一定浓度范围(10nm–100fm)的触发物x观察expar反应的灵敏度。
153、方法
154、a部分:
155、1.50μl水,2.50μl 10x等温扩增缓冲液,3.75μl bsa溶液、1.50μl bst 2.0dna聚合酶(1.6u/μl),0.75μl nt.bstnbi(10u/μl)。
156、b部分:
157、6.30μl水,5.00μl 10x等温扩增缓冲液,0.75μl模板x’-x’(1μm),2.40μlmgso4(100mm),1.50μl dntp(10nm),0.75μl dsgreen(在dmso中1:5稀释,从100x到20x),0.30μlssb溶液。
158、c部分:
159、(100nm、10nm、1nm、100pm、10pm、1pm、空白)的3μl触发物x。
160、添加步骤:将b部分(17μl)添加至在4℃冷却的pcr管中。向其中添加c部分并在4℃下保持5分钟,然后添加a部分(10μl)。然后将管密封,接着对内容物进行扩增。
161、扩增步骤:使用设置为50℃恒温的agilent mx3005p实时pcr系统(迪德科特,英国)进行等温孵育和荧光信号测量。在至少20分钟的孵育时间内每10秒进行荧光测量。
162、结果
163、结果示于图6-7。随着触发物x浓度的降低,扩增发生的时间也会增加。这些结果表明更高浓度的触发物x将更快地产生扩增,因此更优选较高浓度的结合物dna。
164、实施例3:确定expar过程的特异性。
165、本实验针对模板x’-x’序列测试多个非特异性触发物序列,以确定是否有任何序列可以产生扩增。
166、方法
167、a部分:
168、1.50μl水,2.50μl 10x等温扩增缓冲液,3.75μl bsa溶液、1.50μl bst 2.0dna聚合酶(1.6u/μl),0.75μl nt.bstnbi(10u/μl)。
169、b部分:
170、6.30μl水,5.00μl 10x等温扩增缓冲液,0.75μl模板x’-x’(1μm),2.40μlmgso4(100mm),1.50μl dntp(10nm),0.75μl dsgreen(在dmso中1:5稀释,从100x到20x),0.30μlssb溶液。
171、c部分:
172、在(触发物x、触发物a、触发物b和触发物c)的3μl的一种触发物序列
173、添加步骤:将b部分(17μl)添加至在4℃冷却的pcr管中。向其中添加c部分并在4℃下保持5分钟,然后添加a部分(10μl)。然后将管密封,接着对内容物进行扩增。
174、扩增步骤:使用设置为50℃恒温的agilent mx3005p实时pcr系统(迪德科特,英国)进行等温孵育和荧光信号测量。在至少20分钟的孵育时间内每10秒进行荧光测量。
175、结果
176、结果示于图8-9。这些数据集表明,完全互补的触发物x能够在8分钟后产生扩增。不同的是,非特异性触发物要到20分钟后才能产生扩增。这表明该反应具有高度序列特异性。
177、实施例4:两锅式逆转录expar。
178、该实验测试两锅式rt-expar过程是否能够在病毒rna产生后检测sars-cov-2cdna的存在。通过使用患者的阳性和阴性样品,评估扩增时间是否存在差异。
179、方法:
180、a部分:
181、1.50μl水,2.50μl 10x等温扩增缓冲液,3.75μl bsa溶液,1.50μl bst 2.0dna聚合酶(1.6u/μl),0.75μl nt.bstnbi(10u/μl)。
182、b部分:
183、6.30μl水,5.00μl 10x等温扩增缓冲液,0.75μl模板x’-x’(1μm),2.40μlmgso4(100mm),1.50μl dntp(10nm),0.75μl dsgreen(在dmso中1/5稀释),0.30μl ssb溶液。
184、c部分:
185、2μl bstni(10u/μl)和3μl病毒cdna。
186、添加步骤:将b部分(17μl)添加至在4℃冷却的pcr管中。向其中添加c部分并在4℃下保持5分钟,然后添加a部分(10μl)。然后将管密封,接着对内容物进行扩增。
187、扩增步骤:使用设置为50℃恒温的agilent mx3005p实时pcr系统(迪德科特,英国)进行等温孵育和荧光信号测量。在至少20分钟的孵育时间内每10秒进行荧光测量。
188、结果:
189、结果示于图10-11。这些数据集显示患者阳性样品在8.33分钟后产生扩增,而患者阴性样品在22.33分钟后产生扩增。这表明样品的扩增时间存在显著差异,并且能检测cdna。
190、实施例5:一锅式逆转录expar。
191、该实验测试一锅式rt-expar过程是否能够检测sars-cov-2rna的存在。通过使用患者的阳性和阴性样品,评估扩增时间是否存在差异。
192、方法:
193、a部分:
194、1.50μl水,2.50μl 10x等温扩增缓冲液,3.75μl bsa溶液,1.50μl bst 2.0dna聚合酶(1.6u/μl),0.75μl nt.bstnbi(10u/μl)。
195、b部分:
196、6.30μl水,5.00μl 10x等温扩增缓冲液,0.75μl模板x’-x’(1μm),2.40μlmgso4(100mm),1.50μl dntp(10nm),0.75μl dsgreen(在dmso中1/5稀释),0.30μl ssb溶液。
197、c部分:
198、1μl rt-引物(100nm)、1μl bstni(10u/μl)、3μl病毒rna(72.7拷贝/μl)和0.1μlsuperscript iv逆转录酶(200u/μl),
199、添加步骤:将b部分(17μl)添加至在4℃冷却的pcr管中。向其中添加c部分并在4℃下保持5分钟,然后添加a部分(10μl)。然后将管密封,接着对内容物进行扩增。
200、扩增步骤:使用设置为50℃恒温的agilent mx3005p实时pcr系统(迪德科特,英国)进行等温孵育和荧光信号测量。在至少20分钟的孵育时间内每10秒进行荧光测量。
201、结果:
202、结果示于图12-13。这些数据集显示患者阳性样品在7.50分钟后产生扩增,而患者阴性样品在23.04分钟后产生扩增。这表明样品的扩增时间存在显著差异,并且能检测rna。
203、实施例6:两锅式无逆转录expar。
204、该实验测试两锅式rtf-expar程序是否能够使用结合物dna序列和酶bstni检测sars-cov-2rna的存在。通过使用患者的阳性和阴性样品,评估扩增时间是否存在差异。
205、方法:
206、a部分:
207、1.50μl水,2.50μl 10x等温扩增缓冲液,3.75μl bsa溶液,1.50μl bst 2.0dna聚合酶(1.6u/μl),0.75μl nt.bstnbi(10u/μl)。
208、b部分:
209、6.30μl水,5.00μl 10x等温扩增缓冲液,0.75μl模板x’-x’(1μm),2.40μlmgso4(100mm),1.50μl dntp(10nm),0.75μl dsgreen(在dmso中1:5稀释,从100x到20x),0.30μlssb溶液。
210、c部分:
211、10μl rna:dna异源双链体消化混合物,按以下制备:25μl水,5μl 10x等温扩增缓冲液,5μl bstni(10u/μl),10μl结合物dna(1μm)和5μl病毒rna(72.7拷贝/μl)。然后将混合物在50℃孵育,持续5分钟。
212、添加步骤:将b部分(17μl)添加至在4℃冷却的pcr管中。向其中添加c部分并在4℃下保持5分钟,然后添加a部分(10μl)。然后将管密封,接着对内容物进行扩增。
213、扩增步骤:使用设置为50℃恒温的agilent mx3005p实时pcr系统(迪德科特,英国)进行等温孵育和荧光信号测量。在至少20分钟的孵育时间内每10秒进行荧光测量。
214、结果:
215、结果示于图14-15。这些数据集显示患者阳性样品在9.12分钟后产生扩增,而患者阴性样品在27.29分钟后产生扩增。这表明样品的扩增时间存在显著差异,并且利用结合物dna能检测rna。
216、实施例7:一锅式无逆转录expar。
217、该实验测试一锅式rtf-expar过程是否能够在单一锅中使用结合物dna序列和酶bstni检测sars-cov-2rna的存在。通过使用患者的阳性和阴性样品,评估扩增时间是否存在差异。
218、方法:
219、a部分:
220、1.50μl水,2.50μl 10x等温扩增缓冲液,3.75μl bsa溶液,1.50μl bst 2.0dna聚合酶(1.6u/μl),0.75μl nt.bstnbi(10u/μl)。
221、b部分:
222、6.30μl水,5.00μl 10x等温扩增缓冲液,0.75μl模板x’-x’(1μm),2.40μlmgso4(100mm),1.50μl dntp(10nm),0.75μl dsgreen(在dmso中1:5稀释,从100x到20x),0.30μlssb溶液。
223、c部分:
224、1μl bstni(10u/μl),2μl结合物dna(1μm)和3μl病毒rna(72.7拷贝/μl)。
225、添加步骤:将b部分(17μl)添加至在4℃冷却的pcr管中。向其中添加c部分并在4℃下保持5分钟,然后添加a部分(10μl)。然后将管密封,接着对内容物进行扩增。
226、扩增步骤:使用设置为50℃恒温的agilent mx3005p实时pcr系统(迪德科特,英国)进行等温孵育和荧光信号测量。在至少20分钟的孵育时间内每10秒进行荧光测量。
227、结果:
228、结果示于图16-17。这些数据集显示患者阳性样品在4.69分钟后产生扩增,而患者阴性样品在24.90分钟后产生扩增。这表明样品的扩增时间存在显著差异,并且利用结合物dna能检测rna。
229、总之,我们已经证明通过使用涉及dna选择性限制性核酸内切酶的新型无逆转录酶expar方法,可在少于10分钟的时间内,以少于5分钟的扩增时间,成功检测到sars-cov-2rna。这些速度不仅比qpcr(至少60分钟的试验时间)快得多,目前还优于lamp和30分钟侧流测试。这种rtf-expar方法将完全兼容(可部署),可用于目前用于基于qpcr的covid-19检测的设备。试验的简单性和速度使得该方法能够被调整以检测如上所述的一系列传染病,例如流感、呼吸道合胞病毒和埃博拉病毒(仅举几例)。
230、本文涉及的全部参考文献通过引用其全部内容纳入本文。
231、实施例8:基于orf1ab和n基因检测covi19的患者验证研究。
232、指数扩增反应
233、该方案首先涉及三种溶液的制备:a部分、b部分和c部分,然后是添加步骤,最后是扩增步骤
234、a部分
235、1.50μl水,2.50μl 10x等温扩增缓冲液,3.75μl bsa溶液,1.50μl bst 2.0dna聚合酶(1.6u/μl),然后是0.75μl nt.bstnbi(10u/μl)。
236、b部分
237、6.30μl水,5.00μl 10x等温扩增缓冲液,0.75μl模板x’-x’(1μm),2.40μlmgso4(100mm),1.50μl dntp(10nm),0.75μl dsgreen(在dmso中1:5稀释,从100x到20x),然后是0.30μl ssb溶液。
238、c部分
239、rtf expar试验(一锅式rtf-expar):试剂按如下方式混合在一起:1μlbstni(2u/μl)、2μl结合物dna x(100nm),然后是3μl阳性或阴性样品。
240、添加步骤
241、将b部分(17μl)添加至pcr管中,然后添加c部分(6μl),接着添加a部分(10μl)。然后将管密封,接着对内容物进行扩增。
242、expar扩增
243、使用赛默飞世尔quantstudio 5实时pcr系统,96孔,0.2ml进行等温孵育和荧光信号测量。将温度设置为25℃,持续15秒,然后在试验期间升至50℃,在30分钟的孵育时间内每10秒进行荧光读数测量。
244、结果
245、结果示于图18-19。这些数据集显示截止时间为6分钟。所有扩增时间小于6分钟的样品记录为阳性,而任何超过6分钟的时间记录为阴性。图18a和b显示了orf1ab基因靶标的结果,显示灵敏度为82%,阴性率(negativity)为96%。图19a和b显示n基因靶标的结果,显示灵敏度为96%,阴性率为96%。这表明含有和不含rna样品的扩增时间存在显著差异。
246、实施例9;:hpv实例
247、
248、指数扩增反应
249、该方案首先涉及三种溶液的制备:a部分、b部分和c部分,然后是添加步骤,最后是扩增步骤。
250、a部分
251、1.50μl水,2.50μl 10x等温扩增缓冲液,3.75μl bsa溶液,1.50μl bst 2.0dna聚合酶(1.6u/μl),然后是0.75μl nt.bstnbi(10u/μl)。
252、b部分
253、6.30μl水,5.00μl 10x等温扩增缓冲液,0.75μl模板hpv’-hpv’(1μm),2.40μlmgso4(100mm),1.50μl dntp(10nm),0.75μl dsgreen(在dmso中1:5稀释,从100x到20x),然后是0.30μl ssb溶液。
254、c部分
255、rtf expar试验(一锅式rtf-expar):试剂按如下方式混合在一起:1μlbstni(2u/μl)、2μl结合物dna hpv(100nm),然后是3μl阳性或阴性样品。
256、附加步骤
257、将b部分(17μl)添加至pcr管中,然后添加c部分(6μl),接着添加a部分(10μl)。然后将管密封,接着对内容物进行扩增。
258、使用赛默飞世尔quantstudio 5实时pcr系统,96孔,0.2ml进行expar扩增等温孵育和荧光信号测量。将温度设置为25℃,持续15秒,然后在试验期间升至50℃,在30分钟的孵育时间内每10秒进行荧光读数测量。
259、结果
260、结果示于图20。这些数据集显示mrna阳性样品在4.5分钟后产生扩增,而阴性样品在6分钟后产生扩增。这表明样品的扩增时间存在显著差异,并且利用结合物dna和如本文所述方法能检测mrna。
261、参考文献
262、1.sheridan,c.冠状病毒和分发可靠诊断方法的竞争(coronavirus and therace to distribute reliablediagnostics).nature biotechnology 38,382–384(2020).
263、2.标准反转录方案(两步法).西格玛奥里奇公司https://www.sigmaaldrich.com/technical-documents/protocols/biology/standard-reverse-transcription-protocol-two-step.html.
264、3.notomi,t.dna的环介导等温扩增(loop-mediated isothermal amplificationof dna).nucleic acids research 28,63e–663(2000).
265、4.ali,m.m.等.滚环扩增:用于化学生物学、材料科学和医学的多功能工具(rolling circle amplification:a versatile tool for chemical biology,materialsscience and medicine).chemical society reviews 43,3324(2014).
266、5.hindson,b.j.等,用于dna拷贝数绝对定量的高通量微滴数字pcr系统(high-throughput droplet digital pcr system for absolute quantitation of dna copynumber).anal.chem.83,8604–8610(2011).
267、6.asiello,p.j.&baeumner,a.j.小型化等温核酸扩增的综述(miniaturizedisothermal nucleic acid amplification,a review).lab on a chip 11,1420(2011).
268、7.carter,l.j.等.用于covid-19诊断的检测技术和测试开发(assay techniquesand test development for covid-19diagnosis).acs cent.sci.6,591–605(2020).
269、8.寡核苷酸分析仪.https://eu.idtdna.com/calc/analyzer.
270、9.tan,e.等,等温dna扩增与基于dna纳米球的比色检测相联合(isothermal dnaamplification coupled with dna nanosphere-based colorimetric detection).anal.chem.77,7984–7992(2005)
271、10.reid,m.s.,le,x.c和zhang,h.核酸的指数等温扩增以及蛋白质、细胞、小分子和酶活性的测定:expar实例(exponential isothermal amplification of nucleicacids and assays for proteins,cells,small molecules,and enzyme activities:anexpar example).angewandte chemie international edition 57,11856–11866(2018).
272、11.mok,e.,wee,e.,wang,y.和trau,m.等温指数扩增反应分子增强剂的综合评价(comprehensive evaluation of molecular enhancers of the isothermalexponential amplification reaction).scientific reports 6,(2016).
273、12.qian,j.等.通过expar实现等温dna扩增的序列依赖性(sequence dependenceof isothermal dna amplification via expar).nucleic acids research 40,e87–e87(2012).
274、13.基于green的定量pcr.西格玛奥德里奇https://www.sigmaaldrich.com/life-science/molecular-biology/pcr/quantitative-pcr/sybr-green-based-qpcr.html.
275、14.murray,i等,ii型限制酶对rna进行序列特异性切割(sequence-specificcleavage of rna by type ii restriction enzymes).nucleic acids research 30(22)第8257-8268页(2010)
276、 序列表
277、<110> 伯明翰大学
278、<120> 靶rna检测
279、<130> pb158379wo
280、<150> gb2019059.1
281、<151> 2020-12-03
282、<160> 18
283、<170> patentin版本3.5
284、<210> 1
285、<211> 30
286、<212> dna
287、<213> covid19
288、<400> 1
289、agggtaaacc aaatacctgg tgtatacgtt 30
290、<210> 2
291、<211> 27
292、<212> dna
293、<213> covid19
294、<400> 2
295、agggttaaac caccgcctgg agatcaa 27
296、<210> 3
297、<211> 30
298、<212> dna
299、<213> covid19
300、<400> 3
301、agggtcctta acttgcctgg ttgtgatggt 30
302、<210> 4
303、<211> 10
304、<212> dna
305、<213> 人工序列
306、<220>
307、<223> 核酸内切酶识别序列
308、<220>
309、<221> misc_特征
310、<222> (6)..(10)
311、<223> n是a, c, g,或 t
312、<400> 4
313、gagtcnnnnn 10
314、<210> 5
315、<211> 10
316、<212> dna
317、<213> 人工序列
318、<220>
319、<223> 核酸内切酶识别序列
320、<220>
321、<221> misc_特征
322、<222> (6)..(10)
323、<223> n是a, c, g,或 t
324、<400> 5
325、ctcagnnnnn 10
326、<210> 6
327、<211> 17
328、<212> dna
329、<213> 人工序列
330、<220>
331、<223> 寡聚物
332、<400> 6
333、agggtaaacc aaatacc 17
334、<210> 7
335、<211> 17
336、<212> dna
337、<213> 人工序列
338、<220>
339、<223> 寡聚物
340、<400> 7
341、agggttaaac caccgcc 17
342、<210> 8
343、<211> 17
344、<212> dna
345、<213> 人工序列
346、<220>
347、<223> 寡聚物
348、<400> 8
349、agggtcctta acttgcc 17
350、<210> 9
351、<211> 15
352、<212> dna
353、<213> 人工序列
354、<220>
355、<223> 寡聚物
356、<400> 9
357、ccgggattgg ttgat 15
358、<210> 10
359、<211> 44
360、<212> dna
361、<213> 人工序列
362、<220>
363、<223> 模板序列
364、<400> 10
365、ggtatttggt ttaccctgtg agactctggt atttggttta ccct 44
366、<210> 11
367、<211> 30
368、<212> dna
369、<213> 人工序列
370、<220>
371、<223> 结合物 dna
372、<400> 11
373、agggtaaacc aaatacctgg tgtatacgtt 30
374、<210> 12
375、<211> 19
376、<212> dna
377、<213> 人工序列
378、<220>
379、<223> 引物
380、<400> 12
381、ggagcacaaa accagttga 19
382、<210> 13
383、<211> 44
384、<212> dna
385、<213> 人工序列
386、<220>
387、<223> 模板序列
388、<400> 13
389、ggaactaatc agaccctgtg agactctgga actaatcaga ccct 44
390、<210> 14
391、<211> 17
392、<212> dna
393、<213> 人工序列
394、<220>
395、<223> 触发物序列
396、<400> 14
397、agggtctgat tagttcc 17
398、<210> 15
399、<211> 30
400、<212> dna
401、<213> 人工序列
402、<220>
403、<223> 结合物 dna
404、<400> 15
405、agggtctgat tagttcctgg tccccaaaat 30
406、<210> 16
407、<211> 30
408、<212> dna
409、<213> 人工序列
410、<220>
411、<223> hpv 结合物序列
412、<400> 16
413、agggttaagg ggtcggtgga ccggtcgatg 30
414、<210> 17
415、<211> 20
416、<212> dna
417、<213> 人工序列
418、<220>
419、<223> hpv 触发物序列
420、<400> 17
421、agggttaagg ggtcggtgga 20
422、<210> 18
423、<211> 49
424、<212> dna
425、<213> 人工序列
426、<220>
427、<223> hpv 模板序列
428、<400> 18
429、tccaccgacc ccttaaccct gtgagactct ccaccgaccc cttaaccct 49
- 还没有人留言评论。精彩留言会获得点赞!