一套用于枳壳种质资源鉴定的SNP分子标记及其应用
一套用于枳壳种质资源鉴定的snp分子标记及其应用
技术领域
1.本发明涉及植物种质资源鉴定技术领域,涉及枳壳的种质资源鉴定,具体涉及一套用于枳壳种质资源鉴定的snp分子标记及其应用。
背景技术:
2.枳壳为芸香科(rutaceae)柑桔属(citrus)植物酸橙(citrus aurantium l.)及其栽培变种的干燥未成熟果实,为常用中药,具有理气宽中、行滞消胀、化痰等功效。枳壳在长期的人工栽培过程中被不断选择产生了许多栽培品种,由于传统的药材品种识别主要依赖外观形态特征,极易受环境因子的影响,因此,药材市场经常出现优劣品种混杂现象,品种混乱,质量参差不齐。因此,寻找一种经济、高效、准确的种质资源鉴别方法,对于提高枳壳种质资源管理效率和种质资源保护能力以及药材品质均具有重要意义。
3.随着分子生物学的发展,分子标记技术的出现为种质资源鉴定提供了新的手段。分子标记技术具有检测周期短、不受环境影响、可进行高通量测试分析等优点,已经在品种真实性鉴定、种子纯度鉴定等方面得到广泛应用。国际植物新品种保护联盟(upov)于2010 年发布了dna分子标记选择和数据库构建指南(简称bmt指南),指出ssr和snp标记是特别适用于品种鉴定的方法。ssr标记具有多态性高、共显性、实验程序简单、结果重复性好等优点,在作物品种鉴定中得到广泛的应用。但利用ssr标记进行品种鉴定存在着ssr标记数量有限、检测位点少,ssr位点存在一定的突变率、对变异反应比较敏感等缺点,而利用snp标记构建的指纹库则能克服这些问题。相对于传统的ssr标记, snp标记具有数量多、分布广泛、稳定性高、易于实现快速高通量分型的特点。常用的 snp检测方法有基于dna芯片技术、测序及竞争性等位基因特异性pcr等。dna芯片技术是检测snp的最常用方法,但该方法的芯片开发及检测成本较高。
4.kasp(kompetitive allele specific pcr,即竞争性等位基因特异性pcr)技术是基于引物末端碱基的特异匹配来对snp进行分型,该技术具有高度的稳定性与准确性。其适用于多样本、少位点的快速检测,具有高效、灵活、准确、低成本的特点,国内外研究者已利用kasp技术分别构建小麦、玉米、水稻、棉花、甘蓝、黄瓜等作物的核心标记体系,为相应作物的种质资源鉴定和种质资源保护等提供了便利。基于kasp技术开发snp标记进行种质资源鉴定,可大大提高鉴定效率。
技术实现要素:
5.本发明的目的之一是针对上述问题,提供一套用于枳壳种质资源鉴定的snp分子标记,基于所述核心snp标记,可实现对枳壳种质资源的高通量snp分型检测。
6.本发明的一套用于枳壳种质资源鉴定的snp分子标记,是基于kasp技术开发,由4 个snp标记组成,所述snp标记的具体信息如下:
7.标记编号染色体snp物理位置等位基因型ci900011chr0122550627[t/a]
ci900021chr0239351254[c/t]ci900025chr068158400[a/t]ci900018chr085774144[a/g]
[0008]
所述snp标记的物理位置是基于柚子参考基因组序列而确定的,所述柚子参考基因组序列在ncbi中的基因组版本号:ncbi genome assembly asm200692v1。
[0009]
所述枳壳种质资源为绿衣枳壳、香橙、臭橙、代代花。
[0010]
所述香橙为以绿衣枳壳为砧木嫁接的香橙,所述臭橙为以绿衣枳壳为砧木嫁接的臭橙;优选所述绿衣枳壳、香橙、臭橙、代代花均为江西地区的枳壳种质资源材料。
[0011]
本发明的再一目的是提供一套用于扩增上述任一项所述的snp分子标记的引物组合,包含特异性引物primer x、primer y和通用引物primer c;其中,
[0012]
用于扩增snp标记ci900011的引物组中primer x、primer y和primer c的核苷酸序列分别如seq id no.5、seq id no.6、seq id no.7所示;
[0013]
用于扩增snp标记ci900021的引物组中primer x、primer y和primer c的核苷酸序列分别如seq id no.8、seq id no.9、seq id no.10所示;
[0014]
用于扩增snp标记ci900025的引物组中primer x、primer y和primer c的核苷酸序列分别如seq id no.11、seq id no.12、seq id no.13所示;
[0015]
用于扩增snp标记ci900018的引物组中primer x、primer y和primer c的核苷酸序列分别如seq id no.14、seq id no.15、seq id no.16所示。
[0016]
本发明的再一目的是提供一种用于鉴定或辅助鉴定枳壳种质资源的试剂盒,所述试剂盒包括上述的引物组合。
[0017]
在所述试剂盒的技术方案中,每一组引物中的特异性引物primer x和primer y的5’端分别带有荧光标签a序列和荧光标签b序列,所述荧光标签a序列和荧光标签b序列的核苷酸序列互不相同并且与枳壳基因组序列不同源;
[0018]
优选荧光标签a序列为fam荧光标签序列,荧光标签b序列为hex荧光标签序列。
[0019]
本发明的再一目的是提供上述任一项所述的snp分子标记或权上述的引物组合或上述任一项所述的试剂盒在鉴定或辅助鉴定枳壳种质资源,或者在枳壳种质资源基因分型中的应用。
[0020]
本发明的最后一目的是提供一种用于鉴定或辅助鉴定枳壳种质资源的方法,对待测枳壳样品进行snp分型检测,包括以下步骤:
[0021]
(1)提取待测枳壳样品的基因组dna;
[0022]
(2)采用前述引物组合或者前述的试剂盒对步骤(1)中提取的基因组dna进行snp多态性检测,根据基因型检测结果判断待测枳壳样品是否为前述的4份枳壳种质资源之一。
[0023]
在上述技术方案中,所述步骤(2)是采用前述的引物组合中的每一组引物分别对待测枳壳样品的基因组dna进行kasp扩增,荧光检测仪检测pcr产物,确定待测枳壳品种在每一组引物对应的snp分子标记位点的基因型,将检测结果与前述的4份枳壳种质资源的基因型进行比对,从而判断待测枳壳样品是否为前述的4份枳壳种质资源之一。
[0024]
所述kasp扩增的pcr的反应体系为0.8μl:含有样品dna 20ng-50ng,浓度为100μm 的两条特异性引物各0.0013μl,浓度为100μm的通用引物0.0033μl,2
×
kasp master mix 0.3945μl,超纯水0.3996μl;
[0025]
所述pcr的反应条件为:94℃预变性15min;第一步扩增反应,94℃变性20s,65℃~ 57℃退火并延伸60s,10个循环,每个循环退火及延伸的温度降低0.8℃;第二步扩增反应,94℃变性20s,57℃退火并延伸60s,30个循环。
[0026]
本发明的有益效果是:
[0027]
利用基于kasp反应原理及枳壳材料的单碱基差异设计的分子标记,可以对4份枳壳材料进行高通量的snp分析检测和种质资源快速鉴定,结果准确性高,稳定性好,能够显著提高枳壳种质资源鉴定效率。
附图说明
[0028]
图1是本发明实施例实验流程图。
[0029]
图2是标记ci900011在4份材料中分型图,图中红色圆点为纯合tt基因型;蓝色圆点为纯合aa基因型;紫色圆点为杂合基因型ta。
[0030]
图3是标记ci900021在4份材料中分型图,图中红色圆点为纯合cc基因型;蓝色圆点为纯合tt基因型;紫色圆点为杂合基因型ct。
[0031]
图4是标记ci900025在4份材料中分型图,图中红色圆点为纯合aa基因型;蓝色圆点为纯合tt基因型;紫色圆点为杂合基因型at。
[0032]
图5是标记ci900018在4份材料中分型图,图中红色圆点为纯合aa基因型;蓝色圆点为纯合gg基因型;紫色圆点为杂合基因型ag。
具体实施方式
[0033]
下面结合实施例对本发明作进一步说明,但并不因此而限制本发明。
[0034]
下述实施例中所用试剂如无特殊说明,均为本技术领域常规试剂,均可商购获得。未特别说明的实验条件,均为本领域常规实验条件,可参考分子克隆实验手册(sambrookj&russell dw,molecular cloning:a laboratory manual,2001),或按照制造厂商说明书建议的条件。
[0035]
本发明的实施例实验流程如图1所示。
[0036]
实验材料
[0037]
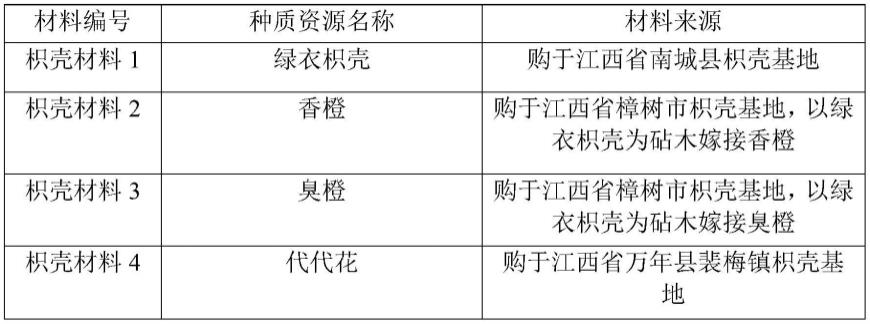
用于snp位点筛选的4份枳壳种质资源及其来源如表1中所示。臭橙、香橙是江西省新干县特产,称为江枳壳,是中国国家地理标志保护产品。
[0038]
表1.实验材料
[0039]
[0040]
*注:香橙、臭橙嫁接苗是从枳壳基地直接购买的已经嫁接好的苗子。
[0041]
实施例1核心snp标记筛选与扩增引物设计
[0042]
对表1的4份江西省枳壳种质资源进行基因组测序,得到重测序数据。由于枳壳目前还没有参考基因组,枳壳的原植物是酸橙,根据目前的报道,柚子和宽皮橘是其原始亲本,因此参照柚子(citrus maxima)参考基因组(基因组版本号为:ncbi genome assemblyasm200692v1)进行变异查找以及snp位点筛选,基于4份枳壳种质资源的基因型结果进行核心snp位点确认。
[0043]
核心snp位点的筛选和确定:利用已完成重测序的4份枳壳种质资源数据与柚子参考基因组序列进行比对获得snp位点。对snp位点进行筛选,筛选标准如下:
①
平均测序深度大于10x;
②
位点无缺失;
③
最小等位基因频率(maf)在0.1-0.5之间;
④
杂合率小于平均杂合率;
⑤
单拷贝的位点;筛选其中多态性较好、在染色体上均匀分布的snp 位点共9个,将其转化为kasp标记。利用kasp平台对4份枳壳种质资源进行基因分型,对每个位点进行筛选分析,去除不符合要求的位点;最终筛选得到多态信息含量高且分型效果理想的核心snp位点,合计4个,相关信息如表2所示,平均杂合率为0.188,平均最小等位基因频率(maf)0.219,平均多态性信息含量(pic)0.242。
[0044]
本发明开发的核心snp标记,分布在基因组的第1、2、6、8号染色体上,这四条染色体上各一个snp标记,共计4个snp标记。
[0045]
表2.用于鉴别4份江西枳壳种质资源鉴定的核心snp标记
[0046]
标记编号染色体snp物理位置等位基因型ci900011chr0122550627[t/a]ci900021chr0239351254[c/t]ci900025chr068158400[a/t]ci900018chr085774144[a/g]
[0047]
将筛选到的4个snp位点转化为kasp标记。提取4个核心snp位点前后各100bp 侧翼序列(序列信息如表3中所示),利用batchprimer3 (http://probes.pw.usda.gov/batchprimer3/)引物设计网站对其进行引物设计,序列中其他位置snp位点用简并碱基代替。每个标记的扩增引物由3条引物组成,包括两条等位基因特异性引物primer x和primer y以及一条通用引物primer c,引物序列如表4中所示。其中2条特异性引物的5’端分别连接适用于英国lgc公司kasp master mix的序列标签,具体地,在本实施例中连接的是英国lgc公司kasp反应试剂的fam和hex荧光接头序列,引物委托invitrogen公司合成。引物合成时,所有基因特异性引物primer x的5’端连接fam荧光接头序列(gaaggtgaccaagttcatgct,seq id no.17),primer y 的5’端连接hex荧光接头序列(gaaggtcggagtcaacggatt,seq id no.18)。
[0048]
如果样品中只检测到fam荧光,则该样品的基因型为纯合等位基因x(allele_x);如果只检测到hex荧光,则该样品的基因型为纯合等位基因y(allele_y);如果同时检测到 fam和hex荧光,则该样品的基因型为杂合(同时带有等位基因x和y)。
[0049]
表3. 4个核心snp标记的序列信息
[0050]
[0051][0052]
表4.用于扩增snp标记的引物序列
[0053][0054]
实施例2snp分子标记的kasp引物验证
[0055]
利用kasp平台对表1所示的4份枳壳种质资源进行基因型分型。
[0056]
一、试验材料的基因组dna提取
[0057]
采用简化ctab法,从枳壳叶片中提取基因组dna。
[0058]
①
取约30mg叶片至1.3ml 96孔板,并置于冻干机中,抽真空12h或以上;
[0059]
②
抽真空结束后,使用分珠器向每孔中加入两粒钢珠,并盖上硅胶膜,置于高通量研磨仪中研磨1min,研磨后置于深孔板离心机中瞬离,将磨碎组织离心到孔底;
[0060]
③
用移液工作站tecan向每孔加ctab提取液700μl,震荡混匀后置于65℃水浴锅中温浴约1-1.5h,每20min将1.3ml 96孔板取出于漩涡振荡器上振荡数次;
[0061]
④
温浴结束后取出1.3ml 96孔板,将其置于深孔板冷冻离心机中,4000rpm离心 10min;
[0062]
⑤
用移液工作站tecan将每孔380μl上清转移到新的1.3ml 96孔板中,并加入等体
积氯仿,颠倒混匀后静置2min,置于深孔板冷冻离心机中,4000rpm,离心10min;
[0063]
⑥
离心后,用移液工作站tecan抽取上清液250μl至预先加好250μl异丙醇的 0.8ml 96孔板中,漩涡振荡混匀,将其置于-20℃冰箱沉淀1h或以上;
[0064]
⑦
取出0.8ml 96孔板置于深孔板冷冻离心机中,4000rpm,离心15min;
[0065]
⑧
弃掉上清,用移液工作站tecan向每孔中加入250μl 70%乙醇,漩涡振荡器上振荡数次,5000rpm,离心15min;
[0066]
⑨
弃掉上清,并置于65℃烘箱中30min将其烘干;
[0067]
⑩
每孔加200μl灭菌超纯水,置于室温过夜溶解备用。
[0068]
二、采用kasp技术进行基因分型
[0069]
用4个标记(ci900011、ci900021、ci900025和ci900018)对表1的4份枳壳材料进行kasp检测验证。以上述步骤中提取的dna为模板,加入实验组1中的snp引物组合中的每一组引物混合液和pcr预混液,采用douglas arraytape基因型分型平台进行pcr扩增体系的自动组装,pcr扩增体系如下表5所示。本发明中使用的douglas arraytape 基因分型平台配套的试剂和耗材均购于英国lgc公司。
[0070]
表5.kasp检测反应体系
[0071] 终浓度实际用量100μm primer c0.42μm0.0033μl100μm primer x0.17μm0.0013μl100μm primer y0.17μm0.0013μl2
×
kasp master mix1
×
0.3945μl超纯水 0.3996μldna(干燥) 20ng-50ng总体积 0.8μl
[0072]
pcr扩增在水浴热循环仪中完成,touchdown pcr反应条件为:94℃预变性15min;第一步扩增反应,94℃变性20s,65℃~57℃退火并延伸60s,10个循环,每个循环退火及延伸的温度降低0.8℃;第二步扩增反应,94℃变性20s,57℃退火并延伸60s,30 个循环。反应完成后利用arraytape扫描系统对kasp反应产物进行荧光数据读取,荧光扫描的结果会自动转化成图形。根据荧光信号的颜色判断待测枳壳种质资源在所用的引物组合对应的snp标记位点的基因型。在snp分子标记基因型分型检测中,样品的基因型分成3簇,分别为x簇(红色)、y簇(蓝色)以及杂合基因型簇(紫色)。
[0073]
分型结果见表6,分型图如图2-图5所示,结果显示ci900011、ci900021、ci900025 和ci900018四个标记分型结果的组合可实现对4份江西枳壳种质资源的区分。
[0074]
表6. 4个标记分型结果
[0075][0076]
实施例3
[0077]
为检测本发明中4个snp标记的特异性及实用性,按照实施例2中的检测方法,将含有表1的4份种质资源的待测枳壳样品9份,进行kasp反应分型。
[0078]
基因型检测结果如表7所示,其中绿衣枳壳(江西南城)、香橙(江西樟树市嫁接香橙)、臭橙(江西樟树市嫁接臭橙)、代代花(江西万年县裴梅镇代代花)基因型区别于其他枳壳样品,并且4份种质资源之间基因型也具备特异性。结果表明,本发明中4个 snp标记分型准确,可用于绿衣枳壳(江西南城)、香橙(江西樟树市嫁接香橙)、臭橙(江西樟树市嫁接臭橙)、代代花(江西万年县裴梅镇代代花)4份种质资源的快速高通量鉴定。
[0079]
表7. 9份枳壳材料的4个标记分型结果
[0080][0081][0082]
*注:嫁接苗是从枳壳基地直接购买的已经嫁接好的苗子。
[0083]
上面结合附图对本发明实施例作了详细说明,但是本发明不限于上述实施例,在所属技术领域普通技术人员所具备的知识范围内,还可以在不脱离本发明宗旨的前提下作出各种变化。此外,在不冲突的情况下,本发明的实施例及实施例中的特征可以相互组合。
[0084]
[0085]
[0086]
[0087]
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1