一种基于核心基因设计的茄科雷尔氏菌检测引物及其应用
1.本发明涉及生物技术领域,具体涉及一种基于核心基因设计的茄科雷尔氏菌检测引物及其应用。
背景技术:
2.茄科雷尔氏菌(ralstonia solanacearum)是一种革兰氏阴性植物病原细菌,由其造成的细菌性青枯病是一种毁灭性的土传病害。茄科雷尔氏菌在全球范围内均有分布,在我国主要分布于南方地区。在无寄主植物时,茄科雷尔氏菌也可在土壤或者水体中存活若干年,在有合适的寄主植物时,茄科雷尔氏菌可以从根部或者茎基部的伤口或自然开口侵染植物,并在木质部大量繁殖,致使植物体内的正常水分运输途径受阻,造成植株缺水萎蔫甚至死亡。茄科雷尔氏菌是一个复合种,可以划分为4个演化型,其中,在我国的分布主要以演化型i为主。茄科雷尔氏菌可以侵染50余科200余种植物,并且其发病速度较快,一旦发病就会影响作物产量,严重影响我国农产品质量与农业经济的发展。因此在发病前或发病早期对茄科雷尔氏菌的种群密度进行检测,通过生物防治或者化学防治的方法进行干预,可以有效防止细菌性青枯病的大面积爆发。
3.随着现代分子生物学的发展,pcr技术已经广泛应用于病原菌种群动态的监控、检测及预警。相比于传统的平板涂布计数法和平板划线分离法,pcr技术具有检测时间短且准确度高的优点,可以大大提高检测效率。目前,该技术应用于实际生产的主要难点在于寻找特异性引物的靶标基因。
技术实现要素:
4.本发明目的在于利用茄科雷尔氏菌中保守的直系同源基因为靶点,设计并验证针对茄科雷尔氏菌快速定量检测所适用的特异性引物,解决农业生产中面临的细菌性青枯病防治问题。
5.orthofinder是目前在比较基因组学研究中一个常用的工具,具有运行快速、全面、准确的优点。它可以找到正交群和直系同源物,并对所有正交群的根基因树进行推断,识别这些基因树中所存在的基因重复事件。直系同源基因又称垂直同源基因,指的是从同一祖先垂直进化而来的基因,一般具有编码序列及功能保守的特点。本发明利用orthofinder分析茄科雷尔氏菌中存在的直系同源基因,寻找序列保守的基因靶标,根据基因的保守核苷酸位点设计茄科雷尔氏菌快速检测的特异性引物,并使用ncbi数据库进行特异性验证,从而提供一种可以应用于实际生产的茄科雷尔氏菌快速定量检测方法,有助于解决我国实际生产中面临的细菌性青枯病防治问题。
6.第一方面,本发明要求保护用于鉴定或者辅助鉴定茄科雷尔氏菌(ralstonia solanacearum)的引物对。
7.本发明所要求保护的引物对是根据如下(i)或(ii)设计得到的:
8.(i)茄科雷尔氏菌基因组中cdc45_rs17435基因;
9.(ii)以茄科雷尔氏菌基因组为模板,采用由seq id no.1和seq id no.2所示两条单链dna组成的引物对进行扩增后所得扩增产物。
10.进一步地,所述引物对由seq id no.1和seq id no.2所示两条单链dna组成。
11.第二方面,本发明要求保护含有前文第一方面所述引物对的试剂或试剂盒。
12.进一步地,所述试剂盒中,除了所述引物对外还可含有其他pcr常规试剂。
13.第三方面,本发明要求保护前文第一方面中所述引物对或前文第二方面中所述试剂或试剂盒在如下任一中的应用:
14.(a1)鉴定或者辅助鉴定茄科雷尔氏菌(ralstonia solanacearum);
15.(a2)鉴定或者辅助鉴定待测菌株是否为茄科雷尔氏菌(ralstonia solanacearum);
16.(a3)检测或辅助检测待测样本中是否含有茄科雷尔氏菌(ralstonia solanacearum);
17.(a4)对青枯病进行预防、监测和/或预警;
18.(a5)对土壤和/或植物样品中的茄科雷尔氏菌种群密度进行定量检测。
19.第四方面,本发明要求保护一种鉴定或辅助鉴定待测菌株是否为茄科雷尔氏菌(ralstonia solanacearum)的方法。
20.本发明要求保护额鉴定或辅助鉴定待测菌株是否为茄科雷尔氏菌(ralstonia solanacearum)的方法,可包括如下步骤:
21.以待测菌株的基因组dna为模板,采用前文第一方面所述引物对(seq id no.1和seq id no.2)进行pcr扩增,得到扩增产物,然后按照如下确定所述待测菌株是否为茄科雷尔氏菌(ralstonia solanacearum):若所述扩增产物中含有141bp目的片段,则所述待测菌株为或候选为茄科雷尔氏菌(ralstonia solanacearum);反之,则所述待测菌株不为或候选不为茄科雷尔氏菌(ralstonia solanacearum)。
22.第五方面,本发明要求保护一种检测或辅助检测待测样本中是否含有茄科雷尔氏菌(ralstonia solanacearum)的方法。
23.本发明要求保护的检测或辅助检测待测样本中是否含有茄科雷尔氏菌(ralstonia solanacearum)的方法,可包括如下步骤:
24.从待测样本中提取总dna,以所述总dna为模板,采用前文第一方面所述引物对(seq id no.1和seq id no.2)进行pcr扩增,得到扩增产物,然后按照如下确定所述待测样本中是否含有茄科雷尔氏菌(ralstonia solanacearum):若所述扩增产物中含有141bp目的片段,则所述待测样本中含有或候选含有茄科雷尔氏菌(ralstonia solanacearum);反之,则所述待测样本中不含有或候选不含有茄科雷尔氏菌(ralstonia solanacearum)。
25.在前文第四和第五方面所述方法中,进行所述pcr扩增时的退火温度可为62℃。
26.进一步地,进行所述pcr扩增时的反应程序为:95℃预变性5min;95℃变性45s,62℃退火45s,72℃延伸1min,共35个循环;最后72℃延伸10min。
27.在(b1)和(c1)中,进行所述pcr扩增时的反应体系中,所述引物对中正向引物(seq id no.1)和反向引物(seq id no.2)的浓度均为10μm。
28.进一步地,进行所述pcr扩增时的反应体系为20μl,包括10μl 2
×
taq pcr master mix(康润生物),1μl正向引物,1μl反向引物,1μl土壤样品dna,7μl ddh2o,其中正向引物与
反向引物浓度均为10μm,土壤样品dna总量在20ng-2μg之间。
29.在本发明中,所述pcr既可以为普通pcr(用于定性检测),也可以为荧光定量pcr(用于定量检测)。
30.在上述相关方面中,所述待测菌株可选自茄科雷尔氏菌(ralstonia solanacearum)、除茄科雷尔氏菌外的其他雷尔氏菌或者非雷尔氏菌。
31.进一步地,所述除茄科雷尔氏菌外的其他雷尔氏菌可选自如下:ralstonia eutropha、ralstonia respiraculi、ralstonia taiwanensis。所述非雷尔氏菌可选自如下:psedomonas syringae、bacillus sp.、pantoea dispersa、xanthomonas sacchari、pseudomonas mediterranea、pseudomonas chlororapghis、pseudomonas fluorescens、ensifer sp.、pseudomonas syringae、chryseobacterium sp.、acidovorax citrulli、pseudomonas fluorescens、pseudomonas fluorescens、bacillus tequilensis、pseudomonas mohnii、pseudomonas moraviensis、pseudomonas putida、streptomyces laurentii、bacillus subtilis。
32.在上述相关方面中,所述待测样本可为环境样本或植物样本;
33.进一步地,所述环境样本可为土壤样本。
34.第六方面,本发明要求保护前文第四方面或第五方面中所述的方法在如下任一中的应用:
35.(b1)对青枯病进行预防、监测和/或预警;
36.(b2)对土壤和/或植物样品中的茄科雷尔氏菌种群密度进行定量检测。
37.本发明利用orthofinder分析茄科雷尔氏菌中存在的直系同源基因,寻找序列保守的基因靶标,根据基因的保守核苷酸位点设计茄科雷尔氏菌快速检测的特异性引物(seq id no.1和seq id no.2),并使用ncbi数据库进行特异性验证,从而提供一种可以应用于实际生产的茄科雷尔氏菌快速定量检测方法,有助于解决我国实际生产中面临的细菌性青枯病防治问题。
38.本发明与现有技术相比,具有以下优势:
39.(1)利用生物信息学方法,对茄科雷尔氏菌种群进行核心基因的挖掘,并发现了仅在该种群中高度保守的基因cdc45_rs17435,发明人在此基础上设计了可用于pcr快速检测的特异性引物,该引物对可以在茄科雷尔氏菌中特异性扩增出141bp的条带,条带清晰且单一,表明本发明的引物特异性良好。
40.(2)本发明设计的特异性引物灵敏度高,将dna模板梯度稀释后,在59
×
10-3
ng/μl水平上特异性引物仍然可以稳定检测到目标条带。
41.(3)本发明设计的特异性引物实用性良好,可在实际大田生产中对茄科雷尔氏菌进行快速鉴定。传统的分离涂板鉴定方法耗时长,成本高,步骤繁琐,不适宜高通量对土壤样品进行定性检测。本发明设计的引物可在含有茄科雷尔氏菌的土壤样品中扩增出预期大小的电泳条带,说明该引物可应用于实际生产中对于青枯病的预防、监测以及预警,进而为青枯病的防治提供科学的理论依据,保护我国农业经济的发展。
42.(4)本发明设计的引物扩增产物大小仅有141bp,pcr反应程序中所需的延伸时间短,目前大多数可通过商业化途径获得的taq酶最多仅需延伸10s就可完成获得该特异性条带的延伸反应,可以大幅度缩短检测所需要的反应时间,提高检测效率;应用本发明所提供
的特异性引物,可以快速简单地完成对样品中茄科雷尔氏菌的定性检测。从提取样品dna到完成琼脂糖凝胶电泳整个过程需要约2.5小时。与传统的平板分离方法相比,可以大大缩短时间,减少所需的人力物力,更符合我国农业生产需求。
43.(5)本发明设计的特异性引物还适用于荧光定量pcr检测,可以对土壤及植物样品中的茄科雷尔氏菌种群密度进行定量检测,可对细菌性青枯病进行风险预警。
附图说明
44.图1为雷尔氏菌属中茄科雷尔氏菌特有的直系同源基因比较分析结果。
45.图2为茄科雷尔氏特有的直系同源基因与假单胞菌直系同源基因比较分析结果。
46.图3为利用ncbi blastp工具在茄科雷尔氏菌中对wp_011001652比对的结果。
47.图4为利用ncbi blastp工具在非茄科雷尔氏菌中对wp_011001652比对的结果。
48.图5为利用ncbi blastp工具在茄科雷尔氏菌中对wp_011002720比对的结果。
49.图6为利用ncbi blastp工具在非茄科雷尔氏菌中对wp_011002720比对的结果。
50.图7为利用ncbi blastp工具在茄科雷尔氏菌中对wp_011001985比对的结果。
51.图8为利用ncbi blastp工具在非茄科雷尔氏菌中对wp_011001985比对的结果。
52.图9为利用ncbi blastp工具在茄科雷尔氏菌中对wp_011003273比对的结果。
53.图10为利用ncbi blastp工具在非茄科雷尔氏菌中对wp_011003273比对的结果。
54.图11为利用ncbi blastp工具在茄科雷尔氏菌中对wp_011000296比对的结果。
55.图12为利用ncbi blastp工具在非茄科雷尔氏菌中对wp_011000296比对的结果。
56.图13为利用ncbi blastp工具在茄科雷尔氏菌中对wp_011001553比对的结果。
57.图14为利用ncbi blastp工具在非茄科雷尔氏菌中对wp_011001553比对的结果。
58.图15为利用ncbi blastp工具在茄科雷尔氏菌中对wp_011002155比对的结果。
59.图16为利用ncbi blastp工具在非茄科雷尔氏菌中对wp_011002155比对的结果。
60.图17为利用ncbi blastp工具在茄科雷尔氏菌中对wp_011000032比对的结果。
61.图18为利用ncbi blastp工具在非茄科雷尔氏菌中对wp_011000032比对的结果。
62.图19为利用ncbi blastp工具在茄科雷尔氏菌中对wp_011000639比对的结果。
63.图20为利用ncbi blastp工具在非茄科雷尔氏菌中对wp_011000639比对的结果。
64.图21为利用ncbi blastp工具在茄科雷尔氏菌中对wp_011002147比对的结果。
65.图22为利用ncbi blastp工具在非茄科雷尔氏菌中对wp_011002147比对的结果。
66.图23为利用ncbi blastn对wp_011000032的编码基因cdc45_rs00335在非茄科雷尔氏菌中的保守性分析结果。
67.图24为利用ncbi blastn对wp_011002155的编码基因cdc45_rs11410在非茄科雷尔氏菌中的保守性分析结果。
68.图25为利用ncbi blastn对wp_011002147的编码基因cdc45_rs11370在非茄科雷尔氏菌中的保守性分析结果。
69.图26为利用ncbi blastn对wp_011003273的编码基因cdc45_rs17435在非茄科雷尔氏菌中的保守性分析结果。
70.图27为cdc45_rs17435基因在茄科雷尔氏菌中的特异性引物设计位置。
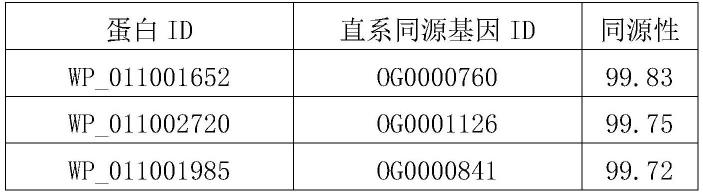
71.图28为本发明所设计引物最适tm值检测结果。
72.图29为本发明所设计引物的特异性检测结果。
73.图30为本发明所设计引物的灵敏度检测。
74.图31为本发明所设计引物在土壤样品中的检测结果。
75.图32为利用本发明所设计引物所构建的qpcr标准曲线。
具体实施方式
76.下面结合具体实施方式对本发明进行进一步的详细描述,给出的实施例仅为了阐明本发明,而不是为了限制本发明的范围。以下提供的实施例可作为本技术领域普通技术人员进行进一步改进的指南,并不以任何方式构成对本发明的限制。
77.下述实施例中的实验方法,如无特殊说明,均为常规方法,按照本领域内的文献所描述的技术或条件或者按照产品说明书进行。下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
78.实施例1、用于设计特异性引物的直系同源基因查找
79.本实例提供特异性引物设计靶点的筛选过程。通过ncbi(https://www.ncbi.nlm.nih.gov/)在线网站,下载包括95株茄科雷尔氏菌在内的106株雷尔氏菌的基因组。通过orthofinder(2.3.7)对上述106株雷尔氏菌进行直系同源基因比较分析,获得共存于106株雷尔氏菌的直系同源基因1035个,共存于95株茄科雷尔氏菌的直系同源基因1250个,venn分析获得茄科雷尔氏菌特异性存在的直系同源基因共259个(图1)。为了进一步验证该259个直系同源基因在茄科雷尔氏菌中的特异性,发明人继续利用orthofinder(2.3.7)对ncbi已公布的与茄科雷尔氏菌亲缘关系较近的88株假单胞菌的共有直系同源基因进行比较分析,发现共存在356个直系同源基因。发明人比较该356个直系同源基因与茄科雷尔氏菌特有的259个直系同源基因发现,它们之间不存在共有基因(图2),进一步说明了上述259个直系同源基因在茄科雷尔氏菌中高度特异性。
80.实施例2、特异性直系同源基因的保守性预测
81.为了寻找更适宜设计引物的靶标基因,发明人将上述具有特异性的259个直系同源基因中过短序列的基因删除,剩余167个直系同源基因。利用danman对这167个直系同源基因对应的单拷贝直系同源序列(single copy orthologue sequences)计算相似性,将软件计算获得的直系同源基因id映射为实际存在的蛋白id,取排名前十的基因进行后续广谱性、保守性验证(表2)。
82.表2、167个直系同源基因中相似性排名前十的蛋白id
83.[0084][0085]
发明人在此基础上利用ncbi网站的blastp工具,对上述10个蛋白在雷尔氏菌属及非雷尔氏菌属中对其保守性进行预测。结果如图3至图22。上述10个基因的蛋白序列在茄科雷尔氏菌中高度保守,而在非茄科雷尔氏菌中,wp_011003273,wp_011002155,wp_011000032,wp_011002147的氨基酸序列保守性较低。发明人继而对此四个基因的核苷酸序列进行了保守性分析。结果如图23至图26。分析发现,wp_011003273的编码基因cdc45_rs17435在非雷尔氏菌属中保守性最低。
[0086]
实施例3、基于cdc45_rs17435在茄科雷尔氏菌中保守位点的特异性引物设计
[0087]
鉴于上述基因cdc45_rs1743在非茄科雷尔氏菌中保守性很低,发明人以此基因为靶点进行茄科雷尔氏菌特异性引物的设计。茄科雷尔氏菌可以分为4个演化型,发明人在4个演化型中选取了具有代表性的菌株,进而比对这些菌株中cdc45_rs17435的同源基因并分析其保守的核苷酸位点。所选取的菌株如表3所示。
[0088]
表3、茄科雷尔氏菌中cdc45_rs17435的同源基因比对所选取菌株
[0089]
gmi 1000和ralstonia solanacearum p380记载于“shen cong et al.diverse interactions of five core type iii effectors from ralstonia solanacearum with plants.j genet genomics.2022may 18;s1673-8527(22)00138-2.doi:10.1016/j.jgg.2022.04.018.”一文,公众可从申请人处获得,仅可用于重复本发明实验使用,不得他用。
[0100]
所选用非茄科雷尔氏菌为:psedomonas syringae dc3000(实验室保藏),bacillus sp.s120(实验室保藏),pantoea dispersa pr834(实验室保藏),xanthomonas sacchari jt1-9(实验室保藏),pseudomonas mediterranea s58(实验室保藏),pseudomonas chlororapghis s1130(实验室保藏),pseudomonas fluorescens 2p24(实验室保藏),ensifer sp.s1279(实验室保藏),pseudomonas syringae s1174(实验室保藏),chryseobacterium sp.s1137(实验室保藏),acidovorax citrulli mh21(实验室保藏),pseudomonas fluorescens s823(实验室保藏),pseudomonas fluorescens s827(实验室保藏),bacillus tequilensis s878(实验室保藏),pseudomonas mohnii s891(实验室保藏),pseudomonas moraviensis s886(实验室保藏),pseudomonas putida s1250(实验室保藏),streptomyces laurentii s1238(实验室保藏),bacillus subtilis s1227(实验室保藏)。其中,acidovorax citrulli mh21记载于“yi-nan ma et al.identification of benzyloxy carbonimidoyl dicyanide derivatives as novel type iii secretion system inhibitors via high-throughput screening.front plant sci.2019sep 5;10:1059.doi:10.3389/fpls.2019.01059.ecollection 2019.”一文,pseudomonas fluorescens 2p24记载于“jing wang et al.characterization of the spi-1type iii secretion system in pseudomonas fluorescens 2p24.front microbiol.2021sep 21;12:749037.doi:10.3389/fmicb.2021.749037.ecollection 2021.”一文,pseudomonas mediterranea s58记载于“yilin gu et al.characterization of a versatile plant growth-promoting rhizobacterium pseudomonas mediterranea strain s58.microorganisms.2020feb 27;8(3):334.doi:10.3390/microorganisms8030334.”一文,psedomonas syringae dc3000记载于“hai-lei wei et al.modular study of the type iii effector repertoire in pseudomonas syringae pv.tomato dc3000 reveals a matrix of effector interplay in pathogenesis.cell rep.2018may 8;23(6):1630-1638.doi:10.1016/j.celrep.2018.04.037.”一文,公众可从申请人处获得,仅可用于重复本发明实验使用,不得他用。
[0101]
pcr扩增体系(20μl):10μl 2
×
taq pcr master mix(康润生物),1μl上游引物(seq id no.1),1μl下游引物(seq id no.2),1μl待测菌株基因组dna,7μl ddh2o,其中上下游引物浓度均为10μm,待测菌株基因组dna总量在20ng-2μg之间。
[0102]
pcr扩增条件为:95℃预变性5min;95℃变性45s,62℃退火45s,72℃延伸1min,共35个循环;最后72℃延伸10min。
[0103]
pcr扩增结束后采用琼脂糖凝胶电泳法进行检测,取5μl扩增产物在2%的琼脂糖凝胶中进行分离,100v电压下电泳30min,经gelred染色后于紫外灯下观察条带的有无以及大小。
[0104]
扩增产物检测如图29所示。由扩增产物检测发现,该引物对在8株茄科雷尔氏菌的
基因组dna中均可以扩增出141bp大小的特异性条带。但是在其余的非茄科雷尔氏菌中均无法扩增得到该特异性条带,说明该引物对特异性良好。
[0105]
实施例6、特异性引物对灵敏性检测
[0106]
利用无菌ddh2o将原始浓度为59ng/μl的茄科雷尔氏菌gmi1000的基因组dna梯度稀释10-1
,10-2
,10-3
,10-4
,10-6
,10-7
,10-8
,10-9
倍,利用特异性引物(seq id no.1和seq id no.2)对稀释后的样品进行pcr扩增,以ddh2o为模板的扩增结果作为阴性对照。扩增体系、反应程序以及扩增产物检测均如实施例5所述。
[0107]
扩增产物检测结果如图30所示。可以发现,在模板浓度为59
×
10-3
ng/μl时该引物对仍然可以扩增出141bp的特异性条带,而当模板浓度在59
×
10-4
ng/μl及其以下时,该引物对无法扩增出预期大小的特异性条带,故该特异性引物的检测阈值在59
×
10-3
ng/μl与59
×
10-4
ng/μl之间,表明该特异性引物对灵敏度良好。
[0108]
实施例7、特异性引物在实际环境样品中应用的特异性检测
[0109]
为了进一步验证本发明所设计引物(seq id no.1和seq id no.2)的实用性,发明人对青枯病土的dna基因了pcr检测。青枯病土dna初始浓度为62.7ng/μl,利用无菌ddh2o对土壤dna梯度稀释10-1
,10-2
,10-3
,10-4
,10-6
倍,利用特异性引物对稀释后的样品进行pcr扩增。扩增体系、反应程序以及扩增产物检测均如实施例5所述。
[0110]
扩增产物检测结果如图31所示。可以发现,在健康植株土壤dna中,在任何一种浓度下特异性引物均无法扩增出141bp大小的条带,在发病植株土壤dna中,在62.7ng/μl与6.27ng/μl浓度条件下该特异性引物均可扩增出141bp大小条带。这表明该特异性引物适用于对土壤样品进行茄科雷尔氏菌的定性检测,且最低检测阈值在0.627ng/μl-6.27ng/μl之间。
[0111]
实施例8、特异性引物应用于荧光定量pcr中进行茄科雷尔氏菌定量检测
[0112]
在现代分子生物学中,荧光定量pcr技术已经广泛用于对各种样品中微生物的定量检测。本发明中的特异性引物(seq id no.1和seq id no.2)设计靶点为单拷贝基因,理论上可适用于荧光定量pcr检测。为了探究本发明所设计引物用于荧光定量pcr检测的可行性,发明人利用茄科雷尔氏菌gmi1000的基因组dna构建了可用于荧光定量pcr检测的标准曲线。将浓度为7ng/μl的gmi1000的基因组dna梯度稀释10-1
,10-2
,10-3
,10-4
倍,利用特异性引物(seq id no.1和seq id no.2)对稀释后的样品进行荧光定量pcr检测。荧光定量pcr体系为20μl,包括10μl 2
×
sybr green(艾科瑞公司),1μl上游引物(seq id no.1),1μl下游引物(seq id no.2),1μl dna(约50ng/μl),7μl ddh2o,其中上下游引物浓度均为10μm。反应条件为:95℃预变性10min;95℃变性10min,62℃退火45s,72℃延伸1min,共40个循环。通过ct值(y)与样品浓度(x,单位:ng/μl)之间的关系构建可得标准曲线y=2.5427x+27.396,r2=0.9999(图32)。该标准曲线相关系数大于0.98,表明该特异性引物可以应用于对茄科雷尔氏菌的定量检测。
[0113]
以上对本发明进行了详述。对于本领域技术人员来说,在不脱离本发明的宗旨和范围,以及无需进行不必要的实验情况下,可在等同参数、浓度和条件下,在较宽范围内实施本发明。虽然本发明给出了特殊的实施例,应该理解为,可以对本发明作进一步的改进。总之,按本发明的原理,本技术欲包括任何变更、用途或对本发明的改进,包括脱离了本技术中已公开范围,而用本领域已知的常规技术进行的改变。按以下附带的权利要求的范围,
可以进行一些基本特征的应用。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1