诊断脑卒中认知障碍的蛋白标志物以及模型的训练方法与流程
本发明涉及脑卒中诊断技术领域,具体而言,涉及诊断脑卒中认知障碍的蛋白标志物以及模型的训练方法。
背景技术:
血管性认知障碍(vascularcognitiveimpairment,vci)据估计占所有确诊认知障碍人群的50%-70%。vci强调是一个连续的过程,是指由脑血管病的危险因素(如高血压、糖尿病等)、或脑血管病引起的从轻度认知损害到失智的一类综合征。我国人群脑血管病发病率显著上升,其中将近一半会出现vci。相对于另一种常见认知障碍——阿尔兹海默病(alzheimer’sdisease,ad),vci的发病机制研究刚起步,临床上缺乏病理学分子影像、外周标志物等有效预测手段。目前对于脑卒中后认知障碍的诊断主要是通过临床认知量表实现,并无客观指标来反映认知障碍情况。现有研究尚未发现临床公认可用的特异性分子标志物,这极大地阻碍了vci病人的早期诊断,并为后续的治疗带来巨大困难。
鉴于此,特提出本发明。
技术实现要素:
本发明的目的在于提供诊断脑卒中认知障碍的蛋白标志物以及模型的训练方法。
本发明是这样实现的:
一方面,本发明提供一种诊断脑卒中患者存在认知障碍的蛋白标志物,所述蛋白标志物选自atp5b蛋白、opcml蛋白和mog蛋白中的至少一种。
在可选的实施方式中,所述蛋白标志物还包括gfap蛋白。
本发明的研究发现,存在认知障碍脑卒中患者和不存在认知障碍脑卒中患者两组群体中存在差异表达蛋白,例如atp5b蛋白、opcml蛋白、mog蛋白和gfap蛋白;说明这些差异表达蛋白可以单独或联合作为诊断脑卒中患者是否存在认知障碍的蛋白标志物;进一步地,本发明以这些差异表达蛋白作为变量构建预测脑卒中患者是否存在认知障碍风险的模型,经验证,该预测模型具有较高的灵敏度和特异度,能够较为准确地进行预测。
本发明为脑卒中发生后是否存在认知障碍的诊断提供了新的标志物和策略。
其中,atp5b蛋白、opcml蛋白、mog蛋白和gfap蛋白的氨基酸序列分别如seqidno.1-4所示,或与其具有至少95%的同源性。
atp5b蛋白序列(seqidno.1)如下,或与如下序列具有至少95%的同源性:
mlgfvgrvaaapasgalrrltpsaslppaqlllraaptavhpvrdyaaqtspspkagaatgrivavigavvdvqfdeglppilnalevqgretrlvlevaqhlgestvrtiamdgteglvrgqkvldsgapikipvgpetlgrimnvigepidergpiktkqfapihaeapefmemsveqeilvtgikvvdllapyakggkiglfggagvgktvlimelinnvakahggysvfagvgertregndlyhemiesgvinlkdatskvalvygqmneppgararvaltgltvaeyfrdqegqdvllfidnifrftqagsevsallgripsavgyqptlatdmgtmqeritttkkgsitsvqaiyvpaddltdpapattfahldattvlsraiaelgiypavdpldstsrimdpnivgsehydvargvqkilqdykslqdiiailgmdelseedkltvsrarkiqrflsqpfqvaevftghmgklvplketikgfqqilageydhlpeqafymvgpieeavakadklaeehss。
opcml蛋白序列(seqidno.2)如下,或与如下序列具有至少95%的同源性:
mgvcgylflpwkclvvvslrllflvptgvpvrsgdatfpkamdnvtvrqgesatlrctiddrvtrvawlnrstilyagndkwsidprviilvntptqysimiqnvdvydegpytcsvqtdnhpktsrvhlivqvppqimnissditvnegssvtllclaigrpeptvtwrhlsvkegqgfvsedeyleisdikrdqsgeyecsalndvaapdvrkvkitvnyppyiskakntgvsvgqkgilsceasavpmaefqwfkeetrlatgldgmrienkgrmstltffnvsekdygnytcvatnklgntnasitlygpgavidgvnsasralaclwlsgtllahffikf。
mog蛋白序列(seqidno.3)如下,或与如下序列具有至少95%的同源性:
maslsrpslpsclcsfllllllqvsssyagqfrvigprhpiralvgdevelpcrispgknatgmevgwyrppfsrvvhlyrngkdqdgdqapeyrgrtellkdaigegkvtlrirnvrfsdeggftcffrdhsyqeeaamelkvedpfywvspgvlvllavlpvlllqitvgliflclqyrlrgklraeienlhrtfdphflrvpcwkitlfvivpvlgplvaliicynwlhrrlagqfleelrnpf。
gfap蛋白序列(seqidno.4)如下,或与如下序列具有至少95%的同源性:
merrritsaarrsyvssgemmvgglapgrrlgpgtrlslarmppplptrvdfslagalnagfketraseraemmelndrfasyiekvrfleqqnkalaaelnqlrakeptkladvyqaelrelrlrldqltansarleverdnlaqdlatvrqklqdetnlrleaennlaayrqeadeatlarldlerkiesleeeirflrkiheeevrelqeqlarqqvhveldvakpdltaalkeirtqyeamassnmheaeewyrskfadltdaaarnaellrqakheandyrrqlqsltcdleslrgtneslerqmreqeerhvreaasyqealarleeegqslkdemarhlqeyqdllnvklaldieiatyrkllegeenritipvqtfsnlqiretsldtksvseghlkrnivvktvemrdgevikeskqehkdvm。
另一方面,本发明提供检测如上所述的蛋白标志物的试剂在制备用于诊断脑卒中患者存在认知障碍的试剂盒中的应用。
基于此本发明的研究发现,检测上述蛋白标志物的试剂具有新的用途,例如制备用于诊断脑卒中患者存在认知障碍的试剂盒。
在可选的实施方式中,所述试剂包括抗所述蛋白标志物的抗体。
在可选的实施方式中,所述抗体包括抗atp5b蛋白抗体、抗opcml蛋白抗体、抗mog蛋白抗体和抗gfap蛋白抗体。
需要说明的是,本领域技术人员可以利用本领域常规的方法例如酶联免疫吸附试验(elisa)或化学发光法等检测上述蛋白标志物的存在或浓度,采用这些方法进行检测所使用的抗体是容易获得的。
再一方面,本发明提供一种用于诊断脑卒中患者存在认知障碍的试剂盒,其包括检测如上所述的蛋白标志物的试剂。
在可选的实施方式中,所述试剂包括抗所述蛋白标志物的抗体。
在可选的实施方式中,所述抗体包括抗atp5b蛋白抗体、抗opcml蛋白抗体、抗mog蛋白抗体和抗gfap蛋白抗体。
在本发明公开了上述蛋白标志物的氨基酸序列的基础上,本领域技术人员通过本领域常规方法容易获得上述蛋白标志物的抗体。
再一方面,本发明提供一种用于预测脑卒中患者是否存在认知障碍风险的模型的训练方法,其包括如下步骤:
步骤s1:获取样本中的蛋白标志物浓度数据,所述样本来自脑卒中患者;所述蛋白标志物为如上所述的蛋白标志物;
步骤s2:利用获得的蛋白标志物浓度数据对预设建立的机器学习模型进行训练,获得用于预测脑卒中患者是否存在认知障碍风险的模型。
在可选的实施方式中,所述蛋白标志物包括atp5b蛋白、opcml蛋白、mog蛋白以及gfap蛋白。
需要说明的是,蛋白标志物浓度数据可以通过本领域常规的方法进行检测例如酶联免疫吸附试验(elisa)的方法检测得到,也可以采用本领域常规的色谱串联质谱的方法进行检测得到。
在可选的实施方式中,所述预先建立的机器学习模型通过以下至少一个算法构建:随机森林算法、贝叶斯分类器、多元非线性回归拟合方法、adaboost算法、支持向量机、k均值算法、决策树算法、k近邻算法、线性回归、逻辑回归、隐马尔可夫模型、神经网络、极限学习机和聚类算法中的至少一种。
在可选的实施方式中,所述样本为血液样本。
血液样本可以是全血样本,血浆样本或血清样本,本领域技术人员可以需要进行合理选择。
在可选的实施方式中,所述样本的数量至少为20份,优选为28份。
本领域技术人员可以根据实际需要合适数量的样本进行模型训练。
附图说明
为了更清楚地说明本发明实施例的技术方案,下面将对实施例中所需要使用的附图作简单地介绍,应当理解,以下附图仅示出了本发明的某些实施例,因此不应被看作是对范围的限定,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他相关的附图。
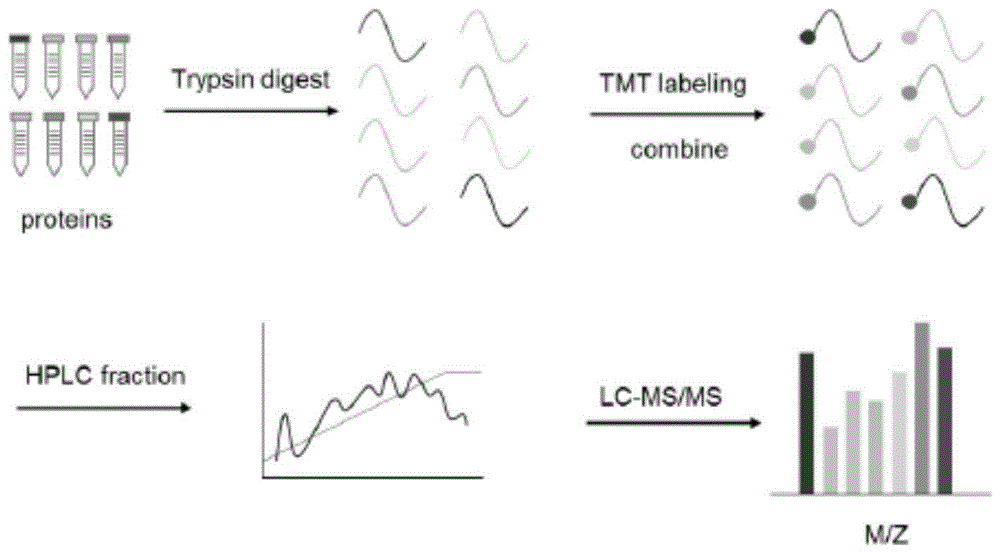
图1:血浆蛋白质组学研究策略;血浆样品除去高丰度蛋白后,通过胰蛋白酶酶解成多肽,tmt试剂标记多肽,等量混合之后,用hplc分离并合并成10分组分,最后经lc-ms/ms检测。
图2:缺血性脑卒中发生后发生认知障碍m1与未发生认知障碍m0的差异蛋白火山图分析;横坐标表示代谢物比值取对数,纵坐标表示两组各蛋白进行t-test检验所得的p-value;其中蓝色表示下调蛋白,橙色表示上调蛋白,灰色表示无显著变化蛋白;蛋白变化比值=缺血性脑卒中发生认知障碍(m1)各蛋白丰度中位数/缺血性脑卒中未发生认知障碍(m0)各蛋白丰度中位数。
图3:缺血性脑卒中发生后发生认知障碍(m1)中下调蛋白kegg通路分析;nfld:non-alcoholicfattyliverdisease(非酒精性脂肪肝);gdm:glyoxylateanddicarboxylatemetabolism(乙醛酸和二羧酸代谢);als:amyotrophiclateralsclerosis(肌萎缩侧索硬化);vlid:valine,leucineandisoleucinedegradation(缬氨酸,亮氨酸和异亮氨酸的降解);cmm:cysteineandmethioninemetabolism(半胱氨酸和蛋氨酸的代谢)。
图4:缺血性脑卒中与认知障碍相关的差异性蛋白;a:缺血性脑卒中发生后发生认知障碍(m1)与未发生认知障碍(m0)的差异蛋白富集热图;b:发生认知障碍(m1)中下调蛋白kegg通路富集;nfld:non-alcoholicfattyliverdiseasegdm:glyoxylateanddicarboxylatemetabolismals:amyotrophiclateralsclerosisvlidvaline,leucineandisoleucinedegradationcmmcysteineandmethioninemetabolism。
图5:缺血性脑卒中发生后发生认知障碍(m1)与未发生认知障碍(m0)的病人血浆差异蛋白生物过程分析;a:缺血性脑卒中发生后发生认知障碍(m1)组中下调蛋白的生物过程分析;b:缺血性脑卒中发生后发生认知障碍(m1)组中上调蛋白的生物过程分析;mdnd:midbraindopaminergicneurondifferentiation(中脑多巴胺能神经元分化);nriasp:negativeregulationofintrinsicapoptoticsignalingpathway(内源性凋亡信号通路的负调控);cccc:cellcommunicationbychemicalcoupling(化学偶联细胞通讯);ccgabaergic:cerebralcortexgabaergicinterneurondevelopment(大脑皮层gaba能中间神经元发育);eobipm:embryonicolfactorybulbinterneuronprecursormigration(胚胎嗅球间神经元前体迁移);prcea:positiveregulationofcysteine-typeendopeptidaseactivityinvolvedinapoptoticprocess(凋亡过程中半胱氨酸型内肽酶活性的正调控);rosm:reactiveoxygenspeciesmetabolicprocess(活性氧代谢过程);ecdiipgd:epithelialcelldifferentiationinvolvedinprostateglanddevelopment(前列腺发育中涉及的上皮细胞分化);protmvt:positiveregulationoftelomeremaintenanceviatelomerase(端粒酶对端粒维持的正调控作用);arcm:auditoryreceptorcellmorphogenesis(听觉受体细胞形态发生);ldlpr:low-densitylipoproteinparticleremodeling(低密度脂蛋白颗粒重塑);rros:responsetoreactiveoxygenspecies(对活性氧的反应)。
图6:缺血性脑卒中发生后发生认知障碍m1与未发生认知障碍m0的差异蛋白细胞成分分析;左侧:缺血性脑卒中发生后发生认知障碍(m1)中下调蛋白的细胞成分分析;右侧:缺血性脑卒中发生后发生认知障碍(m1)中上调蛋白的细胞成分分析;mis:mitochondrialintermembranespace(线粒体膜间隙)。
图7:缺血性脑卒中发生后发生认知障碍m1与未发生认知障碍m0的差异蛋白基因本体论分子功能分析;a:缺血性脑卒中发生后发生认知障碍(m1)中下调蛋白的分子功能;b:缺血性脑卒中发生后发生认知障碍(m1)中上调蛋白的分子功能分析。
图8:随机森林预测模型所纳入变量贡献分类重要性对比。
图9:随机森林模型roc曲线。
具体实施方式
为使本发明实施例的目的、技术方案和优点更加清楚,下面将对本发明实施例中的技术方案进行清楚、完整地描述。实施例中未注明具体条件者,按照常规条件或制造商建议的条件进行。所用试剂或仪器未注明生产厂商者,均为可以通过市售购买获得的常规产品。
以下结合实施例对本发明的特征和性能作进一步的详细描述。
实施例1
蛋白质是生命物质的基础,是构成细胞的基本有机物,是生命活动的主要承担者,在细胞代谢等方面起着重要作用,其具有时空特异性。人的基因组通常情况下是恒定不变的,但是蛋白质在不同的细胞和器官中,或在同一组细胞在不同的环境下或者不同时段其内部蛋白表达水平却千差万别。因此,研究蛋白质结构和功能是生物学的一个核心内容。随着基因组学概念的提出,在1994年由wilkins在siena双向凝胶电泳会议上首次提出蛋白质组学的概念。蛋白质组学主要研究细胞或者组织样品中蛋白质组的表达、修饰水平、细胞定位、相互作用等,系统阐述蛋白质生物学功能。根据不同的研究目的,蛋白质组学一般分为差异蛋白质组学与功能蛋白质组学。差异蛋白质组学是定量比较不同条件下样品全部蛋白质表达水平的差异,描绘出整个蛋白表达图谱,试图找到疾病、药物处理等条件下发生显著变化的蛋白质。功能蛋白质组学的目的则是研究生物体内与某个功能相关或者某个条件下特定一群蛋白质。液相-高分辨质谱联用仪是实现蛋白组学研究的核心仪器,通过分析离子的荷质比(m/z)和拼接碎片化离子,确定多肽和蛋白质序列,实现对蛋白质的高通量检测。蛋白质组学也在生命科学研究中广泛应用,涉及到动物学、植物学、疾病诊断、医药研发、病毒学、肿瘤研究等多个领域。
近年来,蛋白质组学技术在检测通量、定性分析和定量分析的精准度等方面都有了明显的提高。现常用的定量方法包括稳定同位素标记定量(silac)、等重同位素标记定量(如tmt和itraq)等。然而,受到同位素标签数量的限制,在进行大批样品实验时,以labelfree和dia为代表的非标记定量蛋白质组学技术则具有更多优势。目前已经形成了itraq,tmt,labelfree,dia/swath,silac等定量技术为核心的蛋白质组学技术。等重同位素标记定量法是对蛋白质进行体外同位素标记,然后利用质谱在ms2层面对多肽和蛋白进行定量,常用的等重同位素试剂包括tmt[46]和itraq。这些等重同位素标签的结构和原理基本类似,以tmt法为例,tmt试剂由三部分构成,包括反应基团、平衡基团和报告基团,tmt试剂通过反应基团与多肽上赖氨酸侧链或多肽氨基末端相连,多个样品中的同一条tmt标记的蛋白多肽在ms1图谱上有相同的峰,通过二级质谱的碎裂之后,可利用低荷质比区的报告基团丰度实现对多肽和蛋白质进行定量。等重同位素标记蛋白定量方法特点如下:1)样品的适应范围广,基本上不受样品种类限制;2)定量的样品数目较多,根据不同的报告离子,可以实现同时对高达16个样品定性定量分析;3)准确度较高,可以准确反映不同样品间的蛋白差异。但也存在一些问题,如对母离子进行筛选时不可避免的会筛选到共洗脱离子,这些离子也有报告基团,一同经过二级质谱的碎裂之后,共洗脱离子报告基团和母离子的报告基团混合,会对样品本身的定量结果产生一定影响,出现比例压缩现象。现在通过分更多的多肽组分,或者设置小的分离窗口,或者利用ms3碎裂定量,或者利用气相纯化以定量的准确性。
本实施例中,为了得到缺血性脑卒中的蛋白组,选择基于等重同位素标记试剂tmt的定量蛋白质组学技术,可以同时对多组样品准确定量,tmt试剂最高可标记10组样品(图1)。该方法标记效率高、定量精确、灵敏度高且通量高,但容易受到共洗脱的干扰;因此,为了降低共洗脱干扰,采用0.6da的母离子分离窗口。此外,为了提高检测限定量到更多的蛋白,将标记后的混合多肽均在碱性(ph=10)性条件下使用高效液相色谱仪将混合后的样品分成120个组分,再将样品按分级梯度交叉混合成10个样品在酸性(ph=3)条件下除盐,随后进行lc-ms/ms分析,得到的raw文件,再用maxquant(版本1.6)软件,基于swiss-prothumanproteinsequencedatabase(20413序列)进行搜库。其中样品包含缺血性脑卒中组(m)28人;缺血性脑卒中组中12人的moca评分为0;16人的moca评分为1;即缺血性脑卒中组中包含无认知障碍(m0)12人、认知障碍患者(m1)16人。
表1缺血性脑卒中组基本信息
具体实验方法如下:
1血浆白蛋白/igg去除
(1)取血浆样本(病人发病3个月后采集)60μl,加入540μlbindingbuffer(10mm磷酸氢二钠溶液)里,温和混匀。
(2)去掉柱子蓝盖子,倒转去除存储缓冲液,再去除tip。加850μlbindingbuffer,重力作用下流过柱子,丢弃流下的液体。
(3)加入稀释后的上层血浆,重力作用下流过柱子,收集流下的液体。
(4)加入600μl10mmna2hpo4溶液,洗柱子,收集流下的液体;重复一次。
(5)三次收集的样品转移至2mlep管,加蛋白酶抑制剂和磷酸酶抑制剂,测蛋白浓度。
(6)柱子用100mmglyine洗3次;1*pbs洗2-3次;保存于bindingbuffer中。
2蛋白浓度测定
采用bradford法测定蛋白样品浓度,其原理是利用游离状态下的考马斯亮蓝为红色,在488nm波长处有最大吸收;而与蛋白结合后的考马斯亮蓝呈青色,色素-蛋白复合物的最大光吸收在595nm波长处。其光吸收值与蛋白浓度成正相关关系,因此可利用该方法对溶液中的蛋白浓度进行测定。操作流程如下:
(1)开启核酸蛋白浓度测定仪,选择程序protein,设置参数为1μl+1000μl。
(2)空白组取10μl10mmna2hpo4溶液于ep管中;实验组取白蛋白/igg去除的蛋白样品10μl于ep管中。
(3)向上述ep管加入90μlbradford,涡旋混匀后,离心,使液体集中于ep管底部。
(4)取上述反应液10μl,加入990μlbradford,涡旋混匀后,离心,室温放置2min。
(5)先用空白校正后,依次对样品进行浓度测定,记录数值。
3蛋白样品sds-page凝胶电泳定量
根据测定好的浓度,取出50μg蛋白量对应的蛋白溶液体积,加入一定体积的5xloadingbuffer使其稀释成1x,再加入一定体积的1xloadingbuffer定容到50μl将样品制备成1μg/μl,涡旋混匀。样品制好后,将其放在干式恒温金属浴中,95oc煮5min。完成后,冰上冷却后离心。
配置12%分离胶:选择1.0mm的垂直电泳后玻板,配置分离胶,混匀后迅速将其灌注到固定好的玻板中,用无水乙醇压胶消除气泡。待分离胶完全凝固后,倒掉无水乙醇,纯水冲洗净去掉无水乙醇,倒置晾干。配置浓缩胶,混匀后迅速灌注于玻璃板中,插上梳子,待其完全凝固后使用。
电泳:将配置好的凝胶置于电泳槽中,向内槽中倒入一定量的1x电泳液,拔下梳子,按照顺序上样,以蛋白marker作为对照,每个样品的上10μg,补足内外槽电泳液。先用80v的恒定电压跑胶,待溴酚蓝条带完全进入分离胶后,改变电压为120v,继续跑胶。待溴酚蓝条带即将跑出玻板下缘时,停止跑胶。
考马斯亮蓝染色:待电泳完成后,小心拆下凝胶,将其置于干净带盖塑料盒中,加入染料,置于水平摇床上,缓慢摇动室温染色30min。
脱色:将已经染好的胶置于干净带盖塑料盒中,加入脱色液,置于水平摇床上,快速摇动,待条带显现背景完全消除后,倒掉脱色液,换成水,室温继续脱色。
4蛋白样品前处理经过sds-page凝胶电泳检测血浆白蛋白/igg去除效果后,对样品进行前处理:
(1)蛋白样品还原烷基化:取各个样品50μg于1.5mlep管中,用真空冷冻干燥机将样品冻干。用50μl6m尿素将样品溶解,各样品加入2.5μl200mmtcep后,涡旋混匀,瞬时离心,将ep管置于恒温混匀器中,设置转速为850rpm,反应1h后取出置于冰上。按照2.4.12中的配方,避光配置375mmiaa。每个样品避光加入2.5μl375mmiaa,涡旋混匀,瞬时离心后于mt-360震荡混匀器室温避光反应30min。
(2)蛋白酶解:thermomixerc恒温混匀器预热到37℃。将样品稀释至2m终浓度的尿素中,各加入1μl(1μg/μl)胰酶,涡旋混匀,瞬时离心后,置于恒温混匀器,设置转速为1000rpm,反应12~16小时后,取出样品,各样品中加入10%tfa调ph至3左右,灭活未反应完全的胰酶。
5蛋白样品tmt标记
a)将tmt标记试剂从-20℃冰箱中取出,放至室温。每管tmt标记试剂共0.8mg,各加42μl乙腈充分溶解。
b)首先将酶解好的多肽样品各取出12.5μl(即12.5μg)于新的1.5mlep管中。
c)向各样品中加入5μl(即100μg)对应标签的tmt试剂,涡旋混匀并瞬时离心后,置于mt-360震荡混匀器室温反应1h。
d)终止标记:向各反应完全的样品中加入1μl5%羟胺,涡旋混匀并瞬时离心后,置于mt-360震荡混匀器室温反应15min。
e)将同一组反应完全的样品合并至同一ep管中,真空浓缩干燥。
6混合多肽样品脱盐
将浓缩干燥后的混合多肽样品按照如下方法进行脱盐:
a)将样品溶解于300μl0.1%tfa,并用10%tfa调ph为2-3。
b)甲醇活化c18固相萃取柱。萃取柱填料为100mg,总体积为1ml。每次1ml甲醇活化,活化2次。
c)0.1%tfa平衡c18固相萃取柱。每次1ml0.1%tfa,平衡3次。
d)载样。使溶解后的多肽样品自由流过萃取柱,重复载样5次。
e)0.1%tfa脱盐,每次1ml,共计3次。
f)1ml80%acn洗脱。洗脱下来的样品置于真空浓缩仪进行干燥,干燥完成后保存于-80℃冰箱备用。
7hplc分离混合多肽样品
使用的仪器是agilent-1260高效液相色谱,分离柱选用的是agilent4.6x250mmc18反向色谱柱,进样环的规格是500μl载样量。
首先配置所需的流动相buffera和bufferb,同时准备hplc级别的水和甲醇。使用前,利用非接触式超声仪超声4瓶液体各15min,排除液体中的气泡。完成后,将各缓冲液装配到对应的泵上,a泵对应甲醇,b泵对应水,c泵对应buffera,d泵对应bufferb。
随后用300μlbuffera溶解上述干燥后的混合多肽样品,并于常温离心机10000g离心10min。所有准备工作完成后,打开仪器控制软件并开启仪器。待仪器完全开启后,设定仪器柱温为37℃,检测波长为214nm,流速为1ml/min。
准备工作完成后,首先在线排除4个泵对应管道中的气泡,然后用甲醇活化c18反向色谱柱,直至基线稳定。活化完成后,用buffera平衡c18反向色谱柱,直至基线均稳定。冲洗色谱柱的同时,用hplc级的甲醇和buffera依次冲洗进样环三次。
以上步骤完成后,进样溶解好的混合多肽样品,并按照预先设定的hplc梯度洗脱程序(表2)分离多肽样品。按照编号从小到大依次收集洗脱的样品,每管1ml,共收集120管。收集完成后,保存分离样品的信号谱图,并先用95%(5%甲醇)冲洗c18反向色谱柱,至基线稳定,然后用95%甲醇冲洗c18反向色谱柱,直至基线稳定,并将c18反向色谱柱保存于95%甲醇中。
表2hplc梯度洗脱条件
8多肽样品质谱进样前处理
将分离后的多肽样品用真空浓缩仪去除有机溶剂,再每间隔10个合并成一个组分,共计10个。利用真空冷冻干燥机冻干每个组分。随后对每个样品用ziptip脱盐柱脱盐。本实验中选用的ziptip最大载样量为5μg多肽。脱盐流程如下:
a)20μl质谱级0.1%fa溶解各个样品,并用质谱级5%fa调ph为2-3。
b)质谱级乙腈活化ziptip脱盐柱3次,每次20μl,舍弃。
c)质谱级50%乙腈活化ziptip脱盐柱3次,每次20μl,舍弃。
d)质谱级0.1%fa平衡ziptip脱盐柱6次,每次20μl,舍弃。
e)载样:反复吸打溶解好的多肽样品,每次20μl,总共20次以上,保留。
f)脱盐:质谱级0.1%fa脱盐,每次20μl,共计10次,舍弃。
g)洗脱:30μl50%乙腈洗脱ziptip脱盐柱上的多肽样品,反复吸打,每次20μl,共计10次,保留。
h)洗脱后的样品,于真空浓缩仪中干燥,完成后保存于-20℃冰箱,待质谱进样。
9液质联用多肽样品分析
(1)使用的是100μm(内径)×2cm(长度)的预柱和75μm(内径)×12cm(长度)的分析柱。预柱和分析柱均装载dikma公司的c18填料。
(2)首先配置nano-lcbuffera和nano-lcbufferb,并将除盐后的多肽样品复溶于buffera中。配置好的buffera/b置于非接触式超声仪中超声20min,排除液体中的气泡。将bufffera置于a泵中,bufferb置于b泵中,在线排除系统管道中的气泡。随后用已知质荷比的标准品对质谱检测器进行校准,误差满足在10ppm范围内。
(3)设置质谱参数。一级扫描和二级扫描质谱参数如表3。
表3质谱参数
此外,选择信号在2e5以上谱图做二级质谱,除电荷为+1、+8、>+8价态之外的母离子被筛选进行二级质谱分析,多肽自动匹配天然同位素峰,动态排除(dynamicexclude)时间为50.0ms。离子传输管温度为320℃,离子源电压为2.50千伏(kvs透镜的无线电频率为55.0。
(4)设定液相流速为300nl/min,参数如表4。此外,载样最大压力设定为250.0bar,流动相为buffera,总体积12μl。预柱的平衡压力为250.0bar流动相为buffera,总体积为6μl,分析柱的平衡压力为300.0bar,流动相为buffera,总体积为4μl。每次采样完成后,液相系统自动洗针,按照标准每次取100μl0.1%甲酸溶液。
表4easy-nanolc洗脱梯度设置
(5)样品准备:除盐后的样品先用10μl含2%乙腈、0.1%甲酸水溶液充分溶解,10000g、4℃离心10min,取上清9.5μl加入到样品瓶中,依次放置于液相的样品盘。
(6)在控制质谱的电脑上按照样品放置的位置提交上样的序列,按照设定好的液相和质谱参数进行液质联用分析。
10原始数据搜库
同一个样品的10个组分的raw文件合并在一起搜库。搜库软件为maxquant(版本1.6),参考的数据库为swiss-prothumanproteinsequencedatabase(20413序列,2017/01/14更新)。母离子质量偏差为10ppm范围内,碎片离子质量偏差在0.02da以内。允许最大有2个胰酶(trypsin)漏切位点,设置半胱氨酸的脲基甲基化(carbamidomethyl_c)为固定修饰,蛋氨酸的氧化oxidation_m为动态修饰。选择二级离子定量tmt-10plex,蛋白和多肽的错误发现率均小于1%。
11数据处理过程
定量蛋白筛选条件:搜库完成后,得到的txt文件用excel打开,去除表中被标注为reverse和contaminant的蛋白,peptidecounts为1的蛋白,报告基团在各样品中丰度均为零的蛋白后,余下的蛋白即为本次实验中的定量蛋白。取56个样本中均定量到的蛋白数的并集为本次实验中总共鉴定到的蛋白数;取56个样本中均定量到的蛋白数的交集为共同定量的蛋白数。取28个样本(即50%的比例)中共同定量到的蛋白进行功能蛋白的分析。
12结果:
(1)经t检验后(p<0.05),m0和m1蛋白变化比例大于1.2倍为显著变化的蛋白质。统计结果显示,有166个蛋白(包括有atp5b蛋白、opcml蛋白、mog蛋白和gfap蛋白)在发生认知障碍(m1)中下调,有13个蛋白质在发生认知障碍(m1)中上调,表明发病3个月后认知障碍组和非认知障碍组的血浆蛋白存在巨大的差异(图2)。
(2)对这些差异蛋白进行kegg富集分析,发现发生认知障碍(m1)的缺血性脑卒中患者中下调的蛋白主要富集在碳代谢、帕金森病、阿尔茨海默病、氧化磷酸化、非酒精性脂肪肝(nafld)、柠檬酸循环(tca循环)、糖酵解等通路,下调蛋白参与个代谢通路的基因详见图3。发生认知障碍患者血浆中(m1)上调蛋白因数目较少而未富集出显著的通路(图4),上调的蛋白信息详见表5。
表5认知障碍患者血浆中上调蛋白
(3)为了研究这些差异蛋白参与的生物过程、分子功能以及细胞成分,我们利用david6.8进行了go功能注释分析。首先是差异蛋白生物过程分析,如图4所示。可以发现在缺血性脑卒中发生后发生认知障碍(m1中下调的蛋白主要参与的生物过程有中脑多巴胺能神经元分化(midbraindopaminergicneurondifferentiation)、成纤维细胞迁移的调控regulationoffibroblastmigration)、糖原生物合成过程(glycogenbiosyntheticprocess)、蛋白质折叠(proteinfolding)、内源性凋亡信号通路的负调控(negativeregulationofintrinsicapoptoticsignalingpathway)、三羧酸循环(tricarboxylicacidcycle)、活性氧代谢过程(reactiveoxygenspeciesmetabolicprocess)、细胞氧化还原稳态(cellredoxhomeostasis)、碳水化合物代谢过程(carbohydratemetabolicprocess)、轴突形成(axonogenesis)等(见图5中a),这里值得注意的是钙离子流动也是下调。
在缺血性脑卒中发生后发生认知障碍(m1)中上调的蛋白主要参与的生物过程有脂蛋白分解代谢过程(lipoproteincatabolicprocess)、脂蛋白生物合成过程(lipoproteinbiosyntheticprocess低密度脂蛋白颗粒重塑(ow-densitylipoproteinparticleremodeling、胆固醇流出(cholesterolefflux)、甘油三酯分解代谢过程triglyceridecatabolicprocess)、脂蛋白代谢过程(lipoproteinmetabolicprocess)、胆固醇稳态(cholesterolhomeostasis)、胆固醇代谢过程(cholesterolmetabolicprocess)、脂质转运(lipidtransport等(图5中b)。
差异蛋白组成分析发现在缺血性脑卒中发生后发生认知障碍(m1中下调的蛋白主要富集到组成成分除了与胞外体、胞浆、膜、微管等各个细胞都包含的主成分外,其它大部分都是线粒体、线粒体呼吸链复合体、线粒体内膜、丙酮酸脱氢酶复合物、线粒体膜间隙、线粒体类核仁等能量代谢相关的主成分,这说明发生认知障碍与能量代谢相关(图6)。
缺血性脑卒中发生后发生认知障碍(m1中上调的蛋白主要富集到除了常规的细胞外区、细胞外间隙、胞外体、细胞外基质、质膜等,我们还可以发现中密度脂蛋白颗粒、低密度脂蛋白颗粒、乳糜微粒、极低密度脂蛋白颗粒等与脂蛋白相关的成分表达量也相对升高这提示我们血液中脂蛋白的含量与是否发现认知障碍有关,这一结论也与前期代谢物研究结果具有一致性。
(4)分子功能分析发现在缺血性脑卒中发生认知障碍(m1患者血浆中下调的蛋白主要的分子功能是与nad、nadh结合相关(图7中a。此外,与钙离子结合相关的蛋白也相应的发生了下调。钙离子在机体内扮演重要角色,如促进神经递质分泌、心机细胞收缩等。在发生认知障碍(m1患者血浆中上调的蛋白主要的分子功能仍是与脂质转运、胆固醇转运、低密度脂蛋白颗粒受体结合、胆固醇结合、磷脂结合、脂质结合等与脂质代谢相关的蛋白(图7中b)。这与前面代谢物的结果,以及蛋白与生物进程、细胞成分的分析结果都是一致的即能量代谢下调与脂质堆积和认知障碍的发生密切相关。
实施例2
建立随机森林预测模型脑卒中发生后是否存在认知障碍
从以上差异分析,发现脑卒中发生后认知障碍组和非认知障碍组的血浆蛋白存在巨大的差异。为了验证缺血性脑卒中后是否存在与认知障碍相关的蛋白标志物,选取fdr<0.05且在28个病人中均被鉴定到的差异蛋白gfap、atp5b、opcml和mog,建立随机森林预测模型来验证这些蛋白是否可作为缺血性脑卒中后是否发生认知障碍的蛋白标志物。
使用筛选出来的变量建立预测模型及采用三折交叉验证的方式(将样本随机分为三组,两组作为验证集,一组作为训练集,依次进行训练和验证)进行评估。在选择进入模型构建的变量中不同蛋白进行分类时产生的平均减少基尼系数如图8所示,可以看出gfap、atp5b、opcml、mog这四个蛋白平均减少基尼系数依次递减。随机森林预测模型结果如表6和图9所示。
表6基于随机森林模型对验证数据集的分类
从表6中可以看出,所构建的随机森林预测模型灵敏度:16/17=94.12%;特异度:11/11=100%;阳性预测值:16/16=100%;阴性预测值:11/12=91.67%;诊断准确率:(16+11)/28=96.43%。通过绘制受试者特征曲线(roc),求得曲线下面积(areaundercurve,auc)为0.9635(图9),这说明筛选出的上述差异蛋白,可以作为缺血性脑卒中发生后是否发生认知障碍的生物标志物。
以上所述仅为本发明的优选实施例而已,并不用于限制本发明,对于本领域的技术人员来说,本发明可以有各种更改和变化。凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
序列表
<110>四川大学华西医院
<120>诊断脑卒中认知障碍的蛋白标志物以及模型的训练方法
<160>4
<170>siposequencelisting1.0
<210>1
<211>529
<212>prt
<213>人工序列
<400>1
metleuglyphevalglyargvalalaalaalaproalaserglyala
151015
leuargargleuthrproseralaserleuproproalaglnleuleu
202530
leuargalaalaprothralavalhisprovalargasptyralaala
354045
glnthrserproserprolysalaglyalaalathrglyargileval
505560
alavalileglyalavalvalaspvalglnpheaspgluglyleupro
65707580
proileleuasnalaleugluvalglnglyarggluthrargleuval
859095
leugluvalalaglnhisleuglygluserthrvalargthrileala
100105110
metaspglythrgluglyleuvalargglyglnlysvalleuaspser
115120125
glyalaproilelysileprovalglyprogluthrleuglyargile
130135140
metasnvalileglygluproileaspgluargglyproilelysthr
145150155160
lysglnphealaproilehisalaglualaprogluphemetglumet
165170175
servalgluglngluileleuvalthrglyilelysvalvalaspleu
180185190
leualaprotyralalysglyglylysileglyleupheglyglyala
195200205
glyvalglylysthrvalleuilemetgluleuileasnasnvalala
210215220
lysalahisglyglytyrservalphealaglyvalglygluargthr
225230235240
arggluglyasnaspleutyrhisglumetilegluserglyvalile
245250255
asnleulysaspalathrserlysvalalaleuvaltyrglyglnmet
260265270
asngluproproglyalaargalaargvalalaleuthrglyleuthr
275280285
valalaglutyrpheargaspglngluglyglnaspvalleuleuphe
290295300
ileaspasnilepheargphethrglnalaglysergluvalserala
305310315320
leuleuglyargileproseralavalglytyrglnprothrleuala
325330335
thraspmetglythrmetglngluargilethrthrthrlyslysgly
340345350
serilethrservalglnalailetyrvalproalaaspaspleuthr
355360365
aspproalaproalathrthrphealahisleuaspalathrthrval
370375380
leuserargalailealagluleuglyiletyrproalavalasppro
385390395400
leuaspserthrserargilemetaspproasnilevalglyserglu
405410415
histyraspvalalaargglyvalglnlysileleuglnasptyrlys
420425430
serleuglnaspileilealaileleuglymetaspgluleuserglu
435440445
gluasplysleuthrvalserargalaarglysileglnargpheleu
450455460
serglnpropheglnvalalagluvalphethrglyhismetglylys
465470475480
leuvalproleulysgluthrilelysglypheglnglnileleuala
485490495
glyglutyrasphisleuprogluglnalaphetyrmetvalglypro
500505510
ilegluglualavalalalysalaasplysleualaglugluhisser
515520525
ser
<210>2
<211>345
<212>prt
<213>人工序列
<400>2
metglyvalcysglytyrleupheleuprotrplyscysleuvalval
151015
valserleuargleuleupheleuvalprothrglyvalprovalarg
202530
serglyaspalathrpheprolysalametaspasnvalthrvalarg
354045
glnglygluseralathrleuargcysthrileaspaspargvalthr
505560
argvalalatrpleuasnargserthrileleutyralaglyasnasp
65707580
lystrpserileaspproargvalileileleuvalasnthrprothr
859095
glntyrserilemetileglnasnvalaspvaltyraspgluglypro
100105110
tyrthrcysservalglnthraspasnhisprolysthrserargval
115120125
hisleuilevalglnvalproproglnilemetasnileserserasp
130135140
ilethrvalasngluglyserservalthrleuleucysleualaile
145150155160
glyargprogluprothrvalthrtrparghisleuservallysglu
165170175
glyglnglyphevalsergluaspglutyrleugluileseraspile
180185190
lysargaspglnserglyglutyrglucysseralaleuasnaspval
195200205
alaalaproaspvalarglysvallysilethrvalasntyrpropro
210215220
tyrileserlysalalysasnthrglyvalservalglyglnlysgly
225230235240
ileleusercysglualaseralavalprometalaglupheglntrp
245250255
phelysglugluthrargleualathrglyleuaspglymetargile
260265270
gluasnlysglyargmetserthrleuthrphepheasnvalserglu
275280285
lysasptyrglyasntyrthrcysvalalathrasnlysleuglyasn
290295300
thrasnalaserilethrleutyrglyproglyalavalileaspgly
305310315320
valasnseralaserargalaleualacysleutrpleuserglythr
325330335
leuleualahisphepheilelysphe
340345
<210>3
<211>247
<212>prt
<213>人工序列
<400>3
metalaserleuserargproserleuprosercysleucysserphe
151015
leuleuleuleuleuleuglnvalsersersertyralaglyglnphe
202530
argvalileglyproarghisproileargalaleuvalglyaspglu
354045
valgluleuprocysargileserproglylysasnalathrglymet
505560
gluvalglytrptyrargpropropheserargvalvalhisleutyr
65707580
argasnglylysaspglnaspglyaspglnalaproglutyrarggly
859095
argthrgluleuleulysaspalaileglygluglylysvalthrleu
100105110
argileargasnvalargpheseraspgluglyglyphethrcysphe
115120125
pheargasphissertyrglngluglualaalametgluleulysval
130135140
gluaspprophetyrtrpvalserproglyvalleuvalleuleuala
145150155160
valleuprovalleuleuleuglnilethrvalglyleuilepheleu
165170175
cysleuglntyrargleuargglylysleuargalagluilegluasn
180185190
leuhisargthrpheaspprohispheleuargvalprocystrplys
195200205
ilethrleuphevalilevalprovalleuglyproleuvalalaleu
210215220
ileilecystyrasntrpleuhisargargleualaglyglnpheleu
225230235240
glugluleuargasnprophe
245
<210>4
<211>432
<212>prt
<213>人工序列
<400>4
metgluargargargilethrseralaalaargargsertyrvalser
151015
serglyglumetmetvalglyglyleualaproglyargargleugly
202530
proglythrargleuserleualaargmetproproproleuprothr
354045
argvalasppheserleualaglyalaleuasnalaglyphelysglu
505560
thrargalasergluargalaglumetmetgluleuasnaspargphe
65707580
alasertyrileglulysvalargpheleugluglnglnasnlysala
859095
leualaalagluleuasnglnleuargalalysgluprothrlysleu
100105110
alaaspvaltyrglnalagluleuarggluleuargleuargleuasp
115120125
glnleuthralaasnseralaargleugluvalgluargaspasnleu
130135140
alaglnaspleualathrvalargglnlysleuglnaspgluthrasn
145150155160
leuargleuglualagluasnasnleualaalatyrargglngluala
165170175
aspglualathrleualaargleuaspleugluarglysilegluser
180185190
leugluglugluileargpheleuarglysilehisgluglugluval
195200205
arggluleuglngluglnleualaargglnglnvalhisvalgluleu
210215220
aspvalalalysproaspleuthralaalaleulysgluileargthr
225230235240
glntyrglualametalaserserasnmethisglualagluglutrp
245250255
tyrargserlysphealaaspleuthraspalaalaalaargasnala
260265270
gluleuleuargglnalalyshisglualaasnasptyrargarggln
275280285
leuglnserleuthrcysaspleugluserleuargglythrasnglu
290295300
serleugluargglnmetarggluglnglugluarghisvalargglu
305310315320
alaalasertyrglnglualaleualaargleugluglugluglygln
325330335
serleulysaspglumetalaarghisleuglnglutyrglnaspleu
340345350
leuasnvallysleualaleuaspilegluilealathrtyrarglys
355360365
leuleugluglyglugluasnargilethrileprovalglnthrphe
370375380
serasnleuglnilearggluthrserleuaspthrlysservalser
385390395400
gluglyhisleulysargasnilevalvallysthrvalglumetarg
405410415
aspglygluvalilelysgluserlysglngluhislysaspvalmet
420425430
- 还没有人留言评论。精彩留言会获得点赞!