用于分析体细胞可动因子的方法及其用途与流程
本申请要求于2014年2月27日提交的美国临时申请序列号61/945,791的权益,该申请通过引用全文并入本文。
援引并入
本说明书中提到的所有出版物、专利和专利申请均通过引用而并入本文,其程度犹如特别地且单独地指出每个单独的出版物、专利或专利申请通过引用而全文并入。
技术实现要素:
一些实施方案涉及鉴定可动因子插入(MEI)标记的细胞增殖的方法,该方法包括以下步骤:定量测量第一核酸样品中第一MEI插入位点处的MEI水平,定量测量第二核酸样品中第一MEI插入位点处的MEI水平,以及如果第一核酸样品中第一MEI插入位点处的MEI水平与第二核酸样品中第一MEI插入位点处的MEI水平显著(substantially)不同,则将所述第一MEI插入位点鉴定为标记MEI标记的细胞增殖。在所述方法的一些方面,所述第一核酸样品和所述第二核酸样品包含基本相似量的核酸。在所述方法的一些方面,所述第一核酸样品和所述第二核酸样品中存在基本相似量的对照核酸。所述方法的一些方面包括鉴定与所述第一MEI插入位点相邻的序列。所述方法的一些方面包括选择与应对与所述第一MEI插入位点相邻的序列中的缺陷的功效相关的治疗。在所述方法的一些方面,所述第一核酸样品和所述第二核酸样品在第一时间点和第二时间点从相同(common)个体获得。在所述方法的一些方面,所述第一时间点和第二时间点被施用于所述个体的治疗隔开。在所述方法的一些方面,所述治疗包括癌症疗法。在所述方法的一些方面,所述第一时间点和第二时间点相隔至少6个月。在所述方法的一些方面,所述第一时间点和第二时间点相隔至少1年。在所述方法的一些方面,所述第一时间点和第二时间点相隔至少2年。在所述方法的一些方面,所述第一时间点和第二时间点相隔至少5年。在所述方法的一些方面,所述第一核酸样品和所述第二核酸样品从血液中提取。在所述方法的一些方面,所述第一核酸样品和所述第二核酸样品包含循环游离核酸。在所述方法的一些方面,所述第一核酸样品和所述第二核酸样品包含循环游离基因组DNA。在所述方法的一些方面,所述第一核酸样品从个体的第一位置获得,而所述第二核酸样品从该个体的第二位置获得。在所述方法的一些方面,所述第一位置包含第一癌性组织。在所述方法的一些方面,所述第二位置包含健康组织。在所述方法的一些方面,所述第二位置包含第二癌性组织。在所述方法的一些方面,所述第二癌性组织和所述第一癌性组织来源于相同的癌。所述方法的一些方面包括生成报告,该报告披露第一核酸样品中第一MEI插入位点处的MEI水平和第二核酸样品中第一MEI插入位点处的MEI水平。在所述方法的一些方面,所述报告提供给所述个体。在所述方法的一些方面,所述报告提供给医疗保健专业人员。在所述方法的一些方面,所述报告是保密的。
一些实施方案涉及可动因子插入(MEI)监测方案,其包括以下步骤:从个体获得包含多个MEI插入边界的基因组序列信息,考察所述多个MEI插入边界以鉴定与癌基因相邻的边界,以及随时间监测与所述癌基因相邻的MEI边界的定量丰度。在所述方法的一些方面,所述随时间监测与所述癌基因相邻的MEI边界的定量丰度包括在第一时间点获得第一血液样品,确定在所述第一时间点所述第一血液样品中所述MEI边界的定量丰度,在第二时间点获得第二血液样品,以及确定所述在第二时间点所述第二血液样品中所述MEI边界的定量丰度。在所述方法的一些方面,所述随时间监测与所述癌基因相邻的MEI边界的定量丰度包括在第一时间点获得第一组织样品,确定在所述第一时间点所述第一组织样品中所述MEI边界的定量丰度,在第二时间点获得第二组织样品,以及确定在所述第二时间点所述第二组织样品中所述MEI边界的定量丰度。在所述方法的一些方面,所述第一组织样品和所述第二组织样品包含肿瘤组织。所述方法的一些方面包括选择应对与所述癌基因中的缺陷有关的癌症的治疗。所述方法的一些方面包括如果从所述第一时间点到所述第二时间点,样品中MEI插入位点的定量丰度增加高于阈值,则施用所述应对与所述癌基因中的缺陷有关的癌症的治疗。在所述方法的一些方面,所述阈值为10%的增加。在所述方法的一些方面,所述阈值为20%的增加。在所述方法的一些方面,所述阈值为30%的增加。在所述方法的一些方面,所述阈值为50%的增加。所述方法的一些方面包括在第一时间点前施用第一剂量的所述应对与所述癌基因中的缺陷有关的癌症的治疗,并且如果从所述第一时间点到所述第二时间点,样品中MEI插入位点的定量丰度未能降低至阈值以下,则增加剂量。在所述方法的一些方面,所述阈值为所述第一时间点的量的90%。在所述方法的一些方面,所述阈值为所述第一时间点的量的80%。在所述方法的一些方面,所述阈值为所述第一时间点的量的70%。在所述方法的一些方面,所述阈值为所述第一时间点的量的60%。在所述方法的一些方面,所述阈值为所述第一时间点的量的50%。在所述方法的一些方面,所述阈值为所述第一时间点的量的10%。在所述方法的一些方面,所述治疗包括化疗。在所述方法的一些方面,所述治疗包括放疗。在所述方法的一些方面,所述治疗包括针对与MEI插入相邻的序列中的缺陷的药物。在所述方法的一些方面,所述治疗包括针对由与MEI插入位点相邻的序列编码的蛋白质参与的途径的误调节的药物。在所述方法的一些方面,所述治疗包括特异性结合MEI插入接合点(junction)的核酸。在所述方法的一些方面,所述核酸包括piRNA。在所述方法的一些方面,所述核酸包括siRNA。在所述方法的一些方面,所述核酸包括CRISPR核酸。在所述方法的一些方面,所述核酸引导MEI插入边界的甲基化。
一些实施方案涉及用于癌组织的体内可视化的组合物,该组合物包含与检测元件偶联的、跨越与癌基因相邻的MEI边界的核酸探针。在所述组合物的一些方面,所述检测元件包含荧光团。在所述组合物的一些方面,所述检测元件包含光可激发的部分。在所述组合物的一些方面,所述探针穿过细胞膜。在所述组合物的一些方面,所述探针穿过细胞核膜。在所述组合物的一些方面,探针荧光依赖于探针与包含与癌基因相邻的MEI边界的靶核酸序列的结合。在所述组合物的一些方面,所述探针通过手持荧光团激发装置可视化。
一些实施方案涉及用于监测基因组老化的方法,该方法包括以下步骤:在第一时间段定量测量第一核酸样品中MEI插入位点的数目,在第一时间段定量测量第一核酸样品中MEI插入位点的数目,以及将MEI插入边界的增加与基因组老化的增加相关联。在所述方法的一些方面,MEI插入位点数目的10%的增加表明基因组老化。在所述方法的一些方面,MEI插入位点数目的20%的增加表明基因组老化。在所述方法的一些方面,MEI插入位点数目的30%的增加表明基因组老化。在所述方法的一些方面,MEI插入位点数目的50%的增加表明基因组老化。所述方法的一些方面包括如果表明基因组老化,则推荐抗老化方案。在所述方法的一些方面,该抗老化方案包括卡路里限制。在所述方法的一些方面,该抗老化方案包括NTHE的施用。在所述方法的一些方面,该抗老化方案包括DNA甲基化酶的施用。在所述方法的一些方面,该抗老化方案包括小调节eRNA的施用。在所述方法的一些方面,该抗老化方案包括逆转录酶抑制剂的施用。在所述方法的一些方面,该抗老化方案包括逆转录病毒抑制剂的施用。在所述方法的一些方面,该抗老化方案包括HIV抑制剂的施用。在所述方法的一些方面,该抗老化方案包括AZT的施用。在所述方法的一些方面,该抗老化方案包括HBV抑制剂的施用。在所述方法的一些方面,该抗老化方案包括三氮唑核苷(ribavirin)的施用。在所述方法的一些方面,该抗老化方案包括转座酶抑制剂的施用。
一些实施方案涉及比较第一核酸样品与第二核酸样品的方法,该方法包括以下步骤:获得所述第一核酸样品的多个MEI边界的可动因子插入(MEI)边界序列,分析所述第二核酸样品中是否存在所述多个MEI边界,以及如果所述第二核酸样品缺少在所述第一核酸样品中存在的MEI边界序列,则将所述第二核酸样品鉴定为与所述第一核酸样品不同。所述方法的一些方面包括如果所述第二核酸样品包含在所述第一核酸样品中不存在的MEI边界序列,则将所述第二核酸样品鉴定为与所述第一核酸样品不同。在所述方法的一些方面,获得所述第一核酸样品的多个MEI边界的可动因子插入(MEI)边界序列包括进行所述第一核酸样品的全基因组测序。在所述方法的一些方面,获得所述第一核酸样品的多个MEI边界的可动因子插入(MEI)边界序列包括对所述第一核酸样品的多个MEI边界进行靶向测序。在所述方法的一些方面,分析所述第二核酸样品中是否存在所述多个MEI边界包括进行所述第二核酸样品的全基因组测序。在所述方法的一些方面,分析所述第二核酸样品中是否存在所述多个MEI边界包括进行对所述第二核酸样品的多个MEI边界进行靶向测序。在所述方法的一些方面,对所述第二核酸样品的多个MEI边界进行靶向测序包括使所述第二核酸样品与引物组接触,该引物组包含特异性扩增所述第一核酸样品的每个MEI插入位点的引物。在所述方法的一些方面,对所述第二核酸样品的多个MEI边界进行靶向测序包括使所述第二核酸样品与探针组接触,该探针组包含与所述第一核酸样品的每个MEI插入位点特异性退火的探针。在所述方法的一些方面,所述探针组包含至少一种与荧光团结合的探针,使得与底物结合的探针相对于未与底物结合的探针可被差异性地可视化。在所述方法的一些方面,所述第二样品包括法医样品。在所述方法的一些方面,所述第二样品包括植物样品。在所述方法的一些方面,所述植物样品是植物作物样品。在所述方法的一些方面,所述第二样品包含生物危害性物质。
一些实施方案涉及用于在延缓年龄相关的基因组退化中使用的组合物,该组合物包含可动因子插入抑制药物。在所述组合物的一些方面,所述组合物包含逆转录酶抑制剂。在所述组合物的一些方面,所述组合物包含逆转录病毒抑制剂。
附图说明
本发明的新颖特征在所附的权利要求书中具体阐述。通过参考以下对利用本发明原理的说明性实施方案加以阐述的详细描述以及附图,将会获得对本发明的特征和优点的更好的理解,在这些附图中:
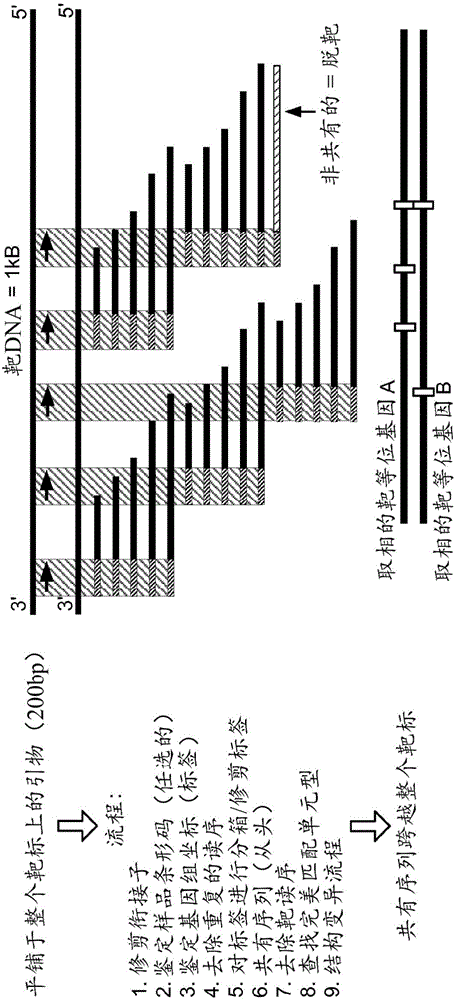
图1示出了使用靶向测序来探测和/或检测复杂变体。
图2显示了使用冗余度和标签来确认和/或定量插入事件。
具体实施方式
可动因子插入(MEI)也被称为转座因子,其占人类基因组的三分之二。存在数百种由于古老MEI活性而进化的人类基因。一些MEI在人类基因组中仍具有活性,包括现代Alu序列。神经元细胞具有高MEI活性,并且病毒MEI的效应在全基因组范围内在癌症中发挥作用。MEI随机无偏倚地出现在基因组的蛋白质编码区和非编码区二者中。MEI影响人类宿主转录和细胞活性,因此在破坏宿主基因的功能时是非常有害的。针对这些有害事件的种系传递会发生强阴性选择。MEI与癌症及其他遗传病症有关,但体细胞MEI的规模和范围尚未得到很好的研究或证明。新的DNA测序技术正在难以解释该作用,因为,例如,样品制备和分析方法缺乏对活性MEI在疾病中的作用进行定量的必要灵敏度。由于有偏倚的扩增,许多这样的方法错报了MEI的活性。用于准确检测MEI的方法需要能够确定被体细胞MEI影响的关键基因并定量其随疾病进展的活性。用于检测和定量关键基因功能破坏的一种非侵入性试验是细胞健康的通用试验,并且已经涉及成年发作疾病的几乎所有领域。
可动DNA因子是进化和遗传病的主要驱动力。可动因子占人类基因组的近三分之二。MEI的主要类型包括但不限于Alu、LINE、SVA、I型逆转录转座子、ERV(内源性逆转录病毒),并且它们被统称为Mobilome。
下一代测序技术增加了我们对MEI在人类基因组中的普遍性的了解。Alu、SINE和SVA因子现今在人类基因组中具有活性。MEI的特定家族在其插入位点处具有共同的序列特征,由此允许产生合成寡核苷酸来探询这些插入事件的诊断性序列。对遗传的MEI的分析及其在1000个基因组计划的可公开获得的数据内的群体频率表明,在研究群体中发现的几乎所有MEI都被认为是罕见的并且以小于10%的频率出现。大多数遗传的MEI都不编码蛋白质编码,表明MEI对基因功能具有高度破坏性,因此通过自然选择而被去除。特别地,体细胞MEI是组织特异性的。例如,Alu和SVA MEI是常见的肿瘤特异性事件,特别是在上皮癌中,但不太可能在血液或脑癌中发现。这表明对MEI活性的环境效应。应激诱导的MEI活性的进一步证据来自于许多转座因子具有与热休克TF结合位点相似的启动子序列这一事实。ME的激活与甲基化的减少之间存在相关性,已经提出这是MEI活性的控制机理。体细胞MEI活性在胚胎发生、肿瘤细胞系和神经元祖细胞中是丰富的,但关于MEI在正常体细胞组织中的活性是鲜为人知的。
数十年来癌症研究的一种主要材料(staple)——HeLa细胞系,具有在c-Myc基因上游的HPV插入位点,这可能是无限细胞分裂的原因。
MEI可以通过破坏开放阅读框或通过在人类基因中提供选择性剪接位点、备选的启动子位点或备选的polyA信号来改变人的转录。根据2001年的人类基因组草图,许多人惊讶于人类基因组仅编码约20,000个基因,特别是相比于>100,000种翻译的蛋白质。现在认为MEI通过引入新的剪接位点(例如,L1、Alu)而与此现象关联。大多数Alu衍生的基因被选择性地剪接,并且许多选择性剪接是组织特异性的。大多数人类基因利用选择性剪接位点,受到基因的备选末端的MEI加工的影响。例如,ATRN基因在内含子中具有L1因子。选择性剪接的基因编码可溶形式的引蛋白(Attractin),这是炎症应答的一部分。该备选形式充当色素沉着和能量代谢的受体。超过120种逆转录转座子序列已进化成功能性的人类基因。对于Alu、L1和SVA因子,估计的从头种系MEI突变的比例分别为20个初生儿中1例到100个中1例到1000个中1例。DNA甲基化被证明是宿主防御机制,缺少甲基转移酶的小鼠表现出高的染色体不稳定性,最终变为灾难性的。小RNA也是MEI活性和作用的调节机制。这些小RNA(包括piRNA和siRNA的类别)也是MEI衍生的。MEI对转录的影响可以通过选择性剪接、备选的启动子或备选的polyA位点而在单个基因座处。在整体水平上,转录网络由MEI启动子活性控制。胚胎干细胞显示出基因网络的连接。ES细胞显示出内源性逆转录病毒长串联重复序列的网络,该内源性逆转录病毒长串联重复序列引发通过甲基化控制的基因表达的网络。在多能性状态下,ERV被甲基化阻抑。哺乳动物妊娠途径通过MEI活性进化。该基因网络在孕酮应答中被MER20因子激活。例如,催乳素启动子来源于MER39可动因子。用于胎儿-母体交换的合胞素(Synctin)基因也来源于ERV基因。ERV的侧翼为约300至约1200个核苷酸的LTR。对于更常见的因子,许多MEI的大小可以在例如约200bp至约10kb的范围内。
ME扫描可从人类基因组中最具活性的常见MEI——AluYb8/9因子中鉴定遗传的MEI。然而该方法在本质上不是定量的。当研究比较非洲与欧洲人群时,这些Alu因子模拟SNP的多样性特征。Alu拷贝在数量上远超过人类基因组中的编码基因,并且据估计,由于在肿瘤和衰老细胞中发生MEI活性的脱阻抑,体细胞MEI事件在数量上远超过种系事件。这些因子的丰度和重复性质对此时的全基因组研究造成了问题。测序深度越大,检测MEI的灵敏度越高,但在文库制备步骤期间,可能由于嵌合分子的产生而引起假阳性,并且由于在诸如PCR等过程中的偏倚,某些因子可能被过度表示。
尽管上述实例示出了遗传的MEI的作用、其破坏性作用和阴性选择,但对体细胞MEI的普遍性也有了更多的了解。MEI的镶嵌性在神经元细胞中是丰富的。神经元具有水平升高的非整倍性和逆转录转座,这可能有助于人脑中的功能多样性。
有证据暗示,体细胞组织中MEI的活化对老化有影响。在正常老化过程中,体细胞MEI转录开始变得活化。这些因子的活化逆转录转座在小鼠的晚期衰老中发生,这对应于随细胞年龄升高的基因组不稳定性。MEI位置和丰度调节衰老的速率,并且缺乏维持复杂DNA结构的能力导致组织的功能紊乱以及生物体的最终死亡。这些转座事件可以被多种应激相关因素如炎症加速。在一些情况下,逆转录转座通过逆转录酶的抑制如用于乙肝病毒(HBV)和人类免疫缺陷病毒(HIV)感染的药物来介导。小鼠中自然发生的癌症具有增加的MEI活性。
由于癌症被认为是基因组的疾病,因此癌症是并且将很可能继续是最流行的体细胞MEI研究领域。可采用高覆盖度全基因组测序(WGS)分析HBV整合事件的影响及其在肝细胞癌(HCC)中的作用。从技术角度来看,由于插入事件的数目与测序的深度成正比,因此测序深度增加会导致许多更加确定的体细胞插入事件。与来自相同个体的正常样品相比,肝细胞癌(HCC)肿瘤中的克隆扩充导致更高频率的相同事件。平均来说,在肿瘤来源的DNA中,对于宿主人类基因组的每个拷贝均可发现病毒基因组的两个拷贝。在插入位点附近发现了破坏性事件,包括直接的基因破坏、病毒启动子驱动的人类基因转录、病毒-人类转录物融合以及DNA拷贝数变化。有证据支持插入事件的随机模型,提示插入大多数是随机的,其中可能仅有的影响是未束缚在染色质中的DNA的可接近性。由于扩增的偏倚,先前基于PCR的方法通常过高地估计了某些插入事件的普遍性。整合广泛存在于整个肿瘤和正常肝组织中,但由于功能性影响限于肿瘤细胞的肿瘤抑制基因和癌基因在肿瘤中的插入事件存在不同模式,并且丰度是这些细胞的克隆扩充的结果。插入“端”映射(map)至HBV基因组的不同区域,这可用于插入位点的检测目的。例如,DR1和DR2位点是在HBV线性病毒的HBx基因的末端处发现的直接重复元件。随机模型显示,来自转录物的融合产物可映射至基因组中的任何位置,因此在插入点存在来自病毒的共同位点和来自人类基因组的无偏倚位点。
大多数HBV插入位点不是频繁发生的,但经历克隆扩充的肿瘤中的主要插入位点的丰度显著升高。肿瘤中的大多数事件出现在蛋白质编码基因附近,提示未在染色质中的暴露的DNA使其易于插入。肿瘤中的插入事件存在对于启动子和外显子的阳性选择。在癌基因普查数据库中,大多数插入可能似乎是中性的,不插入基因中。肿瘤中整合位点的数目可能与结果或其他医疗指标如存活率对应。例如,具有>3个插入事件的肿瘤可能对存活具有更大的负面影响。
可以使用来自各种癌症类型的肿瘤的RNA-Seq数据来研究宿主/病毒融合,特别是已知的致癌病毒。例如,可将NGS RNA-Seq读序映射至HPV、HBV、HCV、EBV和HHV的常见病毒株以观察其对宫颈癌、肝癌和伯基特淋巴瘤的影响。使用从头组装,可以重新诊断具有新型HPV株的阴性宫颈癌肿瘤。PCR测定可能错报HPV整合的丰度。不论涉及的肿瘤类型或组织如何,HPV阳性整合均可显示出肿瘤聚类(clustering)。病毒MEI可通过病毒癌基因的表达或通过整合从而改变癌基因或肿瘤抑制基因的活性来引起细胞转化。
为了MEI检测的临床应用及其诊断意义,必须采用靶向策略。MEI是镶嵌性的,由某种形式的环境因素如应激诱导的MEI活性或病毒诱导的MEI活性引起。已证明,MEI对进化有显著的影响,并且当其在基因组的功能区中被发现时具有非常有害的影响。SNP的遗传模式模型以及对体细胞MEI的影响是一个刚刚开始被解开的谜。观察插入位点以及定量其在所有组织类型(可能使用血液或血液中的无细胞DNA作为替代品)中的丰度的能力是用于确定细胞健康的有用工具。这些活跃可动的或侵入性的基因组因子的靶标可用于诊断目的,以及通过小RNA、甲基化或甚至对逆转录酶抑制对特定个体的合理治疗干预。了解这些因子对个体以及特别是个体疾病的影响将能够提供新的治疗和诊断选择。例如,引起癌症的MEI事件扰乱癌基因或肿瘤抑制基因。使用在活组织内通过各种手段激活的荧光探针来靶向用于在手术中提取的那些细胞。跨越可动因子与其扰乱的人类宿主基因序列的接合点、仅对特定接合事件具有结合效率的探针将为手术中的提取提供标志物或信标。
MEI的起源可能是非人类的,其随时间进化或通过病毒感染而引入。从病毒的角度来看,其主要目的是存活。由感染、炎症、毒素如酒精、物理压力、溃疡等诱导的细胞应激全都影响细胞的存活,因此使MEI活性增加。活性MEI的失调(Deregulation)可能只是偶然地发生。然后活性MEI使染色体重排或改变细胞转录。这些影响可能是温和的或灾难性的。一个细胞变得脱阻抑,并最终克隆扩充。被活性MEI破坏的基因决定扩充的速率。如果细胞生长基因或调节基因被破坏,则其分裂速率上升,导致肿瘤生长。相反,非癌症相关基因也可被扰乱,并且激活/灭活关键的细胞机制(例如,凋亡、坏死、增殖、细胞分裂)。例如,如果凋亡途径被灭活,则细胞可能继续分裂,增加在器官中的普遍性,并开始负面地影响器官功能。最终,这可导致功能丧失。被扰乱的基因以及插入事件的数目均可充当细胞健康或疾病进展的诊断性指标。在一些情况下,这些细胞中的一些可能死亡,并且来自这些细胞的DNA将会以无细胞DNA的形式在血液中被发现。采用具有足以检测这些罕见事件的灵敏度以及足以定量这些罕见事件的精确度的技术监测这些无细胞分子的增加将是分子医学的主要研究内容。从早期基线开始的事件的数目以及其扰乱的基因都可以将全部人类器官的细胞功能分类并监测个体在其一生中的细胞健康。这些诊断试验可能导致受MEI扰乱影响的几乎所有成人发作疾病和病症的早期检测和预防。大脑中体细胞活性的增加可导致癌症、神经变性病症如阿尔茨海默病或帕金森病,或其他病症如孤独症。MEI既含有遗传的组分也含有体细胞活性。它们之间的关系可以解释这些病症中的许多病症的遗传力缺失以及应激或环境诱导的这些因子的激活。这可能是这些复杂病症的合理解释。
MEI代表了我们DNA中仅有的真正个体基因组标志物。对于携带几乎相同DNA的双胞胎,由于错误率,使用目前的测序仪无法检测出绝对差异。相反,寻找MEI谱可以确定在两个几乎相同的基因组(例如,双胞胎)之间的致病的基因组差异。此外,MEI的真正独特的遗传组成构成仅有的真正独特的法医鉴定标志物。最简单的实例是密切相关的个体或双胞胎的MEI。MEI与传统SNP检测或微卫星标志物相结合能够确定地排除甚至最接近的基因组序列。
随着产生的关于个人基因组的序列信息的量增加以及公开共享该信息的意愿增加,伪造基因组身份的能力必定成为现实。在一些情况下,对用于PCR分析的引物具有更好结合亲和力的合成DNA序列从通过公共领域、研究领域或者甚至通过基因检测公司的网络安全性缺乏而获得的个体序列数据生成。个体的血液中掺有这些高度有效的DNA序列不大可能对个体的健康造成影响,或者如果造成了影响,那些个体也可能愿意接受这种风险。当抽取血液用于DNA检测时,这些更加有效的分子可能混淆或彻底掩盖个体的身份或甚至将该样品表示为另一个个体。MEI,特别是最近作为体细胞MEI有活性的那些MEI,将呈现一种完全独特的鉴定策略,由于这些体细胞事件在基因组中的位置以及这样的事件在复杂背景中的量,在一些情况下使用该策略作为基因组鉴定。
这些法医目的还可在农业中用于检测GMO作物。来自GMO农场的种子可轻易地传播至相邻的农场。许多农业公司检测在相邻农场中的这些转座的因子,以确定其不希望的知识产权转移的程度。以极高灵敏度定量这些诊断标志物的方法将允许检测和定量已被其产物污染的生物体的百分比的能力。PCR方法以及其他偏倚策略不提供这一水平的灵敏度。
MEI也可对化妆品工业具有重大的影响。由于MEI激活与细胞老化有关,因此其代表了一种用于研究和确定皱纹或脱发的原因的独特方法。MEI可以是遗传的、体细胞激活的或病毒诱导的。全都导致基因组及其功能的破坏。确定被扰乱的基因代表了用于降低或消除其活性的治疗和美容干预的新目标。监测MEI活性的速率和水平可以是通过自然手段如卡路里限制或通过增加药理干预剂量进行干预的信号。
如本文公开的,从早期MEI活性基线开始的监测细胞健康的试验是针对所有个体的常见检测选择。
在本文的整个说明书中,为了便于理解,本公开内容被分成多个部分。应理解,这些划分是为了方便理解,而并不一定限制说明书的一些部分相对于彼此的适用性。因此,说明书的任何一个部分中的公开内容在一些情况下不仅与该部分有关,而且与其他部分有关,并且在一些情况下与作为整体的公开内容有关。
用于体细胞MEI检测和定量的方法
当前用于MEI检测的全基因组方法包括全基因组测序和生物信息学分析。MEI事件导致“分离读序(split-reads)”,其中序列的一部分映射至人类参考基因组而另一部分未恰当地映射。配双或配对端读序提供了使用一个读序的全部或一部分来锚定DNA分子的未映射或连接部分的位置的能力。大规模平行测序通过更多的采样允许冗余的探询和置信水平增加。然而,该增加的采样导致成本急剧增加。更深的测序深度与MEI检测的灵敏度成比例。全基因组测序(WGS)方法造成了成本增加以及不想要的数据和伦理考虑方面的问题,但在一些情况下具有无偏倚检测整个样品中的MEI插入位点的优势。在一些情况下,这些方法引入序列特异性扩增偏倚,这将抑制定量一些MEI事件的能力,该能力对确定中性MEI与致病MEI之间的差异是关键的。
针对MEI的一些先前的靶向方法通常涉及半特异性PCR的变化形式。如先前讨论的,由于序列特异性偏倚,这些方法在一些情况下不是定量的,由于序列扩增效率而极大地过度呈现一些MEI位置相对于其他位置的量。无法确定体细胞MEI事件是否为中性的,因此无法确定该事件是否是随机呈现的,或者无法确定其是否已在诸如癌症中克隆扩充。此外,对于MEI的插入端,基因座特异性引物的设计的灵活性有限。如果序列发生突变或差异足以导致不发生扩增或导致不那么有效的扩增,则对该特异性事件的量进行定量是不可能的。因此在使用这些方法时必须小心,以确保序列结果定量地反映原始核酸样品中的模板量。
由于MEI的插入端通常是重复或改变的,因此一些适当的靶向体细胞MEI检测方法能够提供冗余度。活性体细胞MEI是现代的,与古老的无活性MEI相比不大可能被截短,但就诊断性序列而言,活性体细胞MEI可以是突变或最小化的。因此,针对MEI的插入端,如TSR或在HBV的情况下在Hbx基因附近的DR 1/2诊断区域,设计了多个冗余的基因座特异性引物。这些多个不同的起始点还允许MEI的确认,因为MEI事件的多次独立采样允许事件的内部确认以及更高的灵敏度和特异性。此外,应该对NGS文库分子产生天然标签或替代的3'端。由于NGS文库分子的替代3'端引起的冗余引物位点与天然标签的组合显示了DNA模板的独立采样,从而保证了任何局部性的插入事件均可通过在扩增步骤期间去除克隆矫作物(artifact)而得到确认和定量。此外,这样的方法需要在制备过程中避免片段化和连接,因为嵌合分子可在这些制备步骤中产生并导致假阳性。
本文设想了其他定量方法,并且本文公开的与MEI位点有关的方法不受任一种定量方法的限制。各种方法在本文中均作为替代方案呈现,突出了其各自呈现的挑战和优势,以及为了使每种方法均适用于本文公开的方法而将要采取的防范措施。
本文公开内容的各个实施方案包括一个或多个MEI事件相对于其插入相邻的基因组序列的定量。定量通过多种方法实现。MEI,有时被称为MEI及其插入相邻的基因组序列,最初通过非靶向方法中的全基因组测序,或通过特异性或半特异性PCR或本领域已知的其他方法来鉴定。在一些实施方案中,使用了TAIL-PCR或本领域中已知用于确定插入相邻序列的其他方法。在许多实施方案中,对于映射至插入相邻边界的初始MEI,全基因组测序或其他非靶向方法是优选的。在后续测定中,在一些实施方案中使用了全基因组方法,而针对特异性MEI和插入相邻序列的靶向测定在备选的后续测定中使用或与全基因组测定组合使用。
核酸样品中MEI插入相邻序列接合点的丰度的定量通过多种备选或协调的方法实现。通过将跨越给定MEI及其插入相邻序列的读序的数目或独特读序的数目或独立衍生的读序的数目与以下任一项或多项进行比较来定量特异性MEI插入边界:样品中核酸的量;映射至核酸样品中的已知单拷贝序列的读序的数目或独特读序的数目或独立衍生的读序的数目,映射至单独MEI及其插入相邻序列的读序的数目或独特读序的数目或独立衍生的读序的数目;或在不同时间点映射至相同MEI及其插入相邻序列的读序的数目或独特读序的数目或独立衍生的读序的数目。在一些情况下,通过相对于输入核酸的总量测量跨越其插入位点的独立读序的数目来定量特异性MEI插入位点。在一些情况下,通过相对于映射至核酸样品的已知独特基因座的独立读序的数目测量跨越其插入位点的独立读序的数目来定量特异性MEI插入位点。在一些情况下,通过相对于映射至已知拷贝数的多拷贝基因座的独立读序的数目测量跨越其插入位点的独立读序的数目来定量特异性MEI插入位点。在一些情况下,通过相对于来自第二时间点的样品的跨越其插入位点的独立读序的数目测量来自第一时间点的样品的跨越其插入位点的独立读序的数目来定量特异性MEI插入位点。组合地或作为替代方案考虑备选的定量方法,如通过与具有可定量的荧光水平的荧光探针杂交而定量。
图2呈现了用于MEI插入位点定量的多个独立读序的实例。每个读序均包含MEI和插入相邻序列,并且每个读序均具有5’端、3’端和插入长度的独特组合。因此,每个读序均可被鉴定,并且是MEI和插入相邻序列而不是克隆扩增的PCR产物的独立表示。
设计
每个MEI家族均在MEI的插入端具有相似的序列。例如,在Alus中,重复序列的侧翼可以有7bp诊断性序列。末端的长度可以变化,并且/或者可具有一些重复序列。在Alu序列中,还可以有polyA序列的延伸。可以部分地靶向polyA序列。还可以靶向具有序列同源性的直接重复区,如DR1和DR2。使用更长的读取长度(例如,MiSeq 2x350读序)、用于配对末端测序的更长插入片段(例如,500bp插入片段)以及由于ddNTP掺入引起的可控的片段长度,可为可动因子的DR1和DR2区的每条链设计多种引物。例如,为了靶向可动因子(例如,Alus、LINE、SVA、病毒MEI等)的每个末端处的1kb区域,可设计多个非重叠引物,以从最末端(接近末端重复序列)跨越通过更加复杂的序列,从而提供更高的特异性。在一些情况下,对于MEI的每个侧翼元件,可以使用至少约三种引物。与PCR不同,由于使用链置换聚合酶的线性引物延伸,多个引发位点将不会互相干扰。因子的每个家族均可具有足够的序列差异,以通过生成的合成序列经由测序立即鉴定因子类型。可鉴定每个因子家族内的多种引物,并将其分箱(bin)在一起以供自组装。在一些情况下,读序可以以足够的确定性进行映射,以确定是否存在关键基因的中断。然后可通过简单地比较从嵌合分子产生的非MEI序列,使用相同MEI的多种引物作为相同MEI破坏事件的独立确认。随着多个引发事件(例如,每个MEI约3个至约10个),每一种引物将从基因组的多个拷贝产生相同事件的多个拷贝。天然标签和3'合成标签可用来确定模板的独立采样,并进一步确认该事件。有趣的是,相同的方法可用来确定事件的相对年龄。更加古老的MEI事件倾向于在MEI序列自身内具有截短的末端或突变,并且这些事件因为缺乏剪切和粘贴或拷贝和粘贴活性所需的插入序列而通常被显示为无活性的。
本公开内容还提供了用于检测遗传疾病中的MEI的方法。在全长MEI的存在下,可使用其他的数据来源进一步确定MEI是否是体细胞的或是否仍然具有活性。通常,截短事件可以表明无活性的MEI,该MEI可能是遗传的并且可能在大部分分子中找到。另一方面,在较小百分比的分子中发现新的体细胞MEI活性(可以是细胞老化的指示),这是需要极深的序列深度的原因。通过反算,体细胞活性的比率为25次细胞分裂中接近约一次。在杂合的群体中,这相当于来自给定组织或活检物的50个DNA分子中约一个。对于50分之一的事件,在每个个体事件三个读序的情况下,需要150x的测序深度。鉴于潜在的高异质性和其中许多是单例(singleton)事件的事实,可能需要提供平均至少1000倍的覆盖来分析肿瘤,并且相比于分析古老MEI如Alus的内源活性的覆盖或许甚至更高(例如,约1百万倍的覆盖)。
其他的数据来源来自其扰乱的一个或多个基因,并且如果事件是克隆扩增的,其还来自事件的数目。例如,病毒如HPV或HBV将会自身随机地插入到基因组的许多区域。这是导致每个个体事件中同等水平的标准化覆盖的随机事件。如果该事件命中细胞生长基因(例如,癌基因或肿瘤抑制基因),则可以观察到那些细胞类型的克隆扩充。因此与背景单例(singleton)或双例(doubleton)体细胞事件的数目相比,特定MEI事件的数目充当疾病诊断的指示。对于单细胞工作,如已经在肿瘤中显示的,相同细胞中的多个事件可以是结果的指示。由于每个肿瘤可具有受感染的但非肿瘤的细胞的集合,因此甚至观察异质肿瘤也可以提供另一水平的数据。背景事件与引起肿瘤的事件的比例可通过对每个事件中的测序深度覆盖取平均值来计算。例如,3倍或更大的增加将是引起肿瘤的事件相对于良性事件的截止值。这可用作治疗期间血液中特定肿瘤的监测目标,或用于确定疾病进展,或作为探针(跨越该事件)用于在手术期间提取以保证肿瘤的去除是完全的。例如,在肝细胞中HBV感染的情况下,可利用针对HBV序列的特异性引物,使用靶向接近线性病毒中Hbx基因的DR1或DR2区的插入末端的3种不同引物来鉴定所有读序。通过计算来自三种引物(其从基因组中的给定位置产生数据)中的每一种的平均覆盖深度,并将给定事件的平均深度与其他随机插入事件的平均深度进行比较,可将更高平均覆盖的事件突出显示为可导致克隆扩充的主要插入位点。在一些实例中,给定事件的平均深度可以是其他随机插入事件的平均深度的超过约1.2倍、约1.4倍、约1.6倍、约1.8倍、约2倍、约3倍、约4倍、约5倍、约10倍、约20倍、约50倍或约100倍。可使用三种不同引物从更加有效的序列(例如,较低GC含量的区域)中去除扩增矫作物。可使用天然和随机合成的标签去除任一事件的克隆扩增。总的来说,可以存在多种用于确认和定量每个事件的信息来源。
本公开内容可提供一种包含分子文库的组合物,其中每个分子均代表MEI事件。该文库可以为多重(multiplex)的形式。
本公开内容可进一步提供一种用于测试所有已知致癌病毒和/或在种系中传代的所有已知活性ALU及MEI的方法。该方法可用于诸如癌基因破坏、细胞老化、每个组织特异性MEI事件中关键基因的破坏(例如,老化大脑中的阿尔茨海默症)和/或针对细胞健康和老化的测试等应用。
本公开内容提供了一种从已知插入位点序列生成基因组中的未知序列的方法。可使用该未知序列确定基因的破坏。可使用来自读序的合成引物序列确定经测序的MEI类型,可使用基因组序列鉴定被破坏的基因,并且可使用天然和合成的标签确定每个事件的定量数量。因此,事件的位置和丰度以及总活性(事件的总数)均可在成年发作的疾病和细胞健康中具有诊断或预后意义。
针对插入区域的引物设计出现在MEI的5'或3'插入位点处的已知诊断性序列中。通过分解成20、50、100个碱基对的窗口,考虑TM、简并位置和重复位置,设计了独特或有些独特的引物序列。由于设计了从插入端开始的多种引物,因此引物设计是冗余的。开发、合成并以等摩尔比合并了针对所有已知MEI病毒性和内源性MEI序列设计的单引物文库。
引物在与所使用的测序平台的衔接子互补物(compliment)对应的5'端包含分子“尾”。在一些情况下,为了样品多重化在合成步骤中包含任选的分子条形码。
引物延伸通过在均匀温度下使用链置换聚合酶或通过使用热稳定聚合酶以及使引物延伸反应循环而发生。该聚合酶必须具有在掺入修饰的碱基或具有缺少羟基的末端3'端的碱基的同时进行延伸的能力。
在反应混合物中使用天然dNTP和生物素化的ddNTP的组合。ddNTP与天然dNTP之比决定了延伸分子的片段长度。例如,使用1%比例的ddNTP将产生1/100的在任何给定碱基处掺入终止分子的几率。通常的结果显示,1%的ddNTP比例产生约500bp的片段峰。这可能是因为天然NTP相对于改变的NTP的掺入效率差异。
所得到的分子是嵌合体,其由在5'端的合成序列和在3'端的来源于患者的序列组成。该分子以终止的、生物素化的核苷酸结束。
通过使用亲和反应,从基因组背景中纯化所述分子。该步骤使用链霉亲和素涂覆的磁珠。珠子上的每个链霉亲和素分子结合四个生物素化的分子,而剩余的ddNTP、dNTP和未使用的引物被去除。
使用随机引物进行第二引物延伸反应,该随机引物由在3'端的8个核苷酸和与测序仪平台对应的B-衔接子互补物组成。随机引发在整个分子上发生,但通过使用链置换聚合酶,只有最远端的随机引物及其延伸产物将保持与链霉亲和素珠子氢键结合。来自B反应的拷贝分子将一直运行通过前一条链上的A引物,并产生具有5'B衔接子、8bp合成随机序列、MEI插入的人类宿主基因组序列位点、MEI基因座特异性引物的合成序列以及在3端的A衔接子互补物的单链分子。如果需要样品多重化,则使这些分子从链霉亲和素结合的分子上变性并PCR扩增以掺入全长测序仪衔接子和任选的外部条形码。
这种嵌合读序结构及其特征在数据分析中具有许多优势。使用引物的合成基因座特异性序列来确定在读序中靶向哪个MEI。对于相同MEI种类导致不同延伸起始点的冗余引物位点可用作插入事件的内部确认。这也避免了因基因座特异性引物不太有效或设计不当而退出(drop out)。该基因座特异性引物可用于所有已知的MEI,包括Alus、LINE以及病毒MEI。将在单个文库中设计已知病毒的全谱,多种病毒在相同样品中的可能性较低。有可能许多病毒引物在任何给定的样品中将不产生数据。
3'片段化和改变的3'序列充当内部分子标签或天然条形码。如果两个读序具有不同的天然标签(3'序列),则它们肯定是模板DNA的独立读出,而非克隆错误。
来自B衔接子反应的合成序列的随机8bp也可充当随机标签。可以组合使用随机3'序列与来自随机8-mer的随机标签的组合,以进一步保证读序是独立的而非克隆扩增的。
在数据分析过程中,首先对来自给定MEI的读序修剪去(trim of)衔接子序列。如果测序仪运行含有多重化的条形码化样品,则鉴定分子条形码。鉴定与合成基因座特异性引物对应的前5-25个碱基以确定被靶向的MEI事件。然后从读序中修剪这些碱基以供映射和组装。将剩余的序列相对于人类参考基因组映射并跨越重叠读序进行组装,以提供人类基因组中的插入位置的证据。重复读序基于其3'端和随机标签而被去除。对于配对端读序,如果未单独地映射,则使用减小的插入大小募集第二个读序,以针对插入位点提供重叠读序。通过使用MiSeq系统,采用300bp的插入大小(优先于ILMN聚类产生),生成了约400-500bp的累积序列以供位置映射。在去除全部克隆读序后,对每个位置定量事件的位置和数目。
因此,本文公开了与可动因子插入(MEI)插入位点序列和可动因子活性有关的方法、组合物及使用方法,例如其涉及人类健康。可将人类可动因子分类为DNA转座子或逆转录转座子。DNA转座子通过剪切-粘贴机制移动。逆转录转座子通过经由RNA中间体的拷贝-粘贴机制(被称为逆转录转座的过程)调动。
与人类疾病有关的可动因子是本领域已知的。示例性的可动因子包括但不限于L1、Alu、SINE-R/VNTR/Alu(SVA)、经加工的假基因和人类内源性逆转录病毒(HERV)。位于蛋白质编码基因座的5’侧的逆转录转座子常常充当备选启动子。例如,如通过帽分析基因表达和焦磷酸测序所评价的,位于基因的3’UTR(非翻译区)中的逆转录转座子显示出降低相应基因表达的有力证据。已知逆转录转座子的低甲基化影响该逆转录转座子自身或附近基因的转录。例如,已知与MET(肝细胞生长因子受体)癌基因相关的L1中启动子的增加的甲基化诱导携带肿瘤的膀胱的尿路上皮内的备选MEI转录物。
类似地,由于其对人类基因组序列的影响,本文设想多种构成‘可动因子’的人类逆转录病毒。多种人类逆转录病毒是本领域已知的。已知逆转录病毒以两种形式存在:作为其染色体DNA中的正常遗传元件(内源性逆转录病毒)和作为从人到人传播的水平传播传染性含RNA病毒(外源性逆转录病毒,例如HIV和人T细胞白血病病毒HTLV)。已知由于人类逆转录病毒插入导致的DNA异常变化与疾病的发作有关。插入到人DNA中的示例性人类逆转录病毒包括但不限于HIV1、HIV2、HTLV1、HTLV2和HSRV。
本文公开了鉴定可动因子插入(MEI)标记的细胞增殖的方法。在一些情况下,这些方法包括以下步骤:定量测量第一核酸样品中第一MEI插入位点处的MEI水平;定量测量第二核酸样品中第一MEI插入位点处的MEI水平;以及如果第一核酸样品中第一MEI插入位点处的MEI水平与第二核酸样品中第一MEI插入位点处的MEI水平显著不同,则将所述第一MEI插入位点鉴定为标记MEI标记的细胞增殖。
在一些情况下对样品的核酸量进行归一化,而在备选的情况下,通过例如测量已知在健康个体中将在每个单倍体基因组以单拷贝存在的一种或多种核酸的水平来对核酸量进行归一化。在一些情况下,当样品的核酸丰度或核酸相对丰度或归一化的核酸丰度相差5%、10%、15%、20%、25%、30%、35%、40%、45%、50%或大于50%时发生‘显著不同’。在一些情况下,‘显著不同’是指相差5%。在一些情况下,‘显著不同’是指相差10%。在一些情况下,‘显著不同’是指相差15%。在一些情况下,‘显著不同’是指相差20%。在一些情况下,‘显著不同’是指相差25%。在一些情况下,‘显著不同’是指相差30%。在一些情况下,‘显著不同’是指相差35%。在一些情况下,‘显著不同’是指相差40%。在一些情况下,‘显著不同’是指相差45%。在一些情况下,‘显著不同’是指相差50%。在一些情况下,‘显著不同’是指相差大于50%。
在一些情况下确定了与MEI插入位点相邻的序列。在一些情况下使用与MEI插入位点相邻的序列来选择治疗,例如,如果相对于其他MEI或在一个时间点相对于之前的时间点,该MEI插入与过度增殖有关。
例如,如果MEI相邻序列与已知癌基因对应,则选择与应对与该癌基因相关的癌症相关的治疗,施用于在时间上或空间上表现出该MEI的过度增殖的个体。
多种与癌症发作有关的基因是本领域已知的。这些基因有不同名称,包括但不限于,癌症驱动基因(driver)、癌基因、肿瘤抑制基因和肿瘤易感基因。已知这些基因的异常DNA变化有助于癌症进展。变化时与驱动癌症有关的示例性基因包括但不限于abl1、acvr1b、af4/hrx、akt1、akt-2、alk、alk/npm、aml1、aml1/mtg8、apc、ar、arid1a、arid1b、arid2、asxl1、atm、atrx、axin1、axl、b2m、bap1、bcl2、blc-3、bcl-6、bcor、bcr/abl、braf、brca1、brca2、card11、casp8、c-myc、cbl、cdc73、cdh1、cdkn2a、cebpa、cic、crebbp、crlf2、csf1r、ctnnb1、cyld、daxx、dbl、del/can、dnmt1、dnmt3a、e2a/pbx1、egfr、enl/hrx、ep300、erbB、erbB-2、erg/TLS、ets-1、ews/fli-、ezh2、fam123b、fbxw7、fgfr2、fgfr3、flt3、fms、fos、foxl2、fps、fubp1、gata1、gata2、gata3、gli、gna11、gnaq、gnas、gsp、her2/neu、h3f3a、hist1h3b、hnf1a、hras、hox11、hst、idh1、idh2、il-2、int-2、jak1、jak2、jak3、jun、kit、ks3、K-sam、kdm5c、kdm6a、kit、klf4、kras、lbc、lck、lmo1、lmo2、l-myc、lyl-1、lyt-10C alpha-1、mas、mdm-2、mos、map2k1、map3k1、med12、men1、met、mlh1、mll2、mll3、mpl、msh2、msh6、myd88、myb、myh11/cbfb、ncor1、neu、n-myc、nf1、nf2、nfe2l2、notch1、notch2、npm1、nras、ost、pax5、pbx1/e2a、pbrm1、pdgfra、phf6、pik3ca、pik3r1、pim-1、prad-1、ppp2r1a、prdm1、ptch1、pten、ptpn11、raf、rar/pml、rasH、rasN、rb1、rel/nrg、ret、rhom1、rehom2、ros、rnf43、runx1、ski、sis、set/can、srcret、setd2、setbp1、sf3b1、smad2、smad4、smarca4、smarcb1、smo、socs1、sox9、spop、srsf2、stag2、stk11、tal1、tal2、tan-1、tiam1、tsc2、trk、tet2、tnfaip3、traf7、tp53、tsc1、tshr、u2af1、vhl和wt1。例如,映射至该列表中的基因的MEI插入相邻序列在一些情况下提示,将选择与所述基因或与所述基因的基因产物参与的信号途径有关的治疗以并入治疗方案中。
类似地,多种基因组重排被鉴定为与癌症有关。本领域已知,癌症中的基因重排主要由DNA双链断裂(DSB)引起。导致基因重排的示例性机制包括但不限于合成依赖性末端连接(SDEJ)、通过断裂-融合-桥循环的姐妹染色单体融合导致的基因扩增、V(D)J重组活化(RAG)蛋白介导的易位以及活化诱导的胞苷脱氨酶(AID)类别转换重组。
示例性的基因重排包括但不限于ACSL3/ETV1、ACTB/GLI1、AFF3/BCL2、AGTRAP/BRAF、AHRR/NCOA2、AKAP9/BRAF、ALK/PTPN3、ANKRD28/NUP98、ARHGAP6/PRCC、ASPSCR1/TFE3、ATIC/ALK、BACH2/BCL2L1、BCL11B/TCR、BCL2/Ig、BCOR/RARA、BCR/ABL1、BCR/FGFR1、BCR/JAK2、BCR/PDGFRA、BIRC3/MALT1、BRD3/C15orf55、BRWD3/ARHGAP2、BRWD3/ARHGAP20、C11orf95/MKL2、C15orf21/ETV1、C15orf55/BRD4、C6orf204/PDGFRB、CACNA2D4/WDR43、CANT1/ETV4、CAPRIN1/PDGFRB、CARS/ALK、CBFB/MYH11、CCDC6/PDGFRB、CCDC6/RET、CCDC88C/PDGFRB、CCND1/FSTL3、CD44/SLC1A2、CD74/ROS1、CDH11/USP6、CDK5RAP2/PDGFRA、CDK6/MLL、CEP110/FGFR1、CHCHD7/PLAG1、CHIC2/ETV6、CIC/DUX4、CLTC/ALK、CLTC/TFE3、CNBP/USP6、CNTRL/KIT、COL1A1/PDGFB、COL1A1/USP6、COL1A2/PLAG1、COL6A3/CSF1、CREB3L2/PPARG、CRTC1/MAML2、DGKB/MIPOL1、EML1/ABL1、EML4/ALK、EPC1/PHF1、ERC1/PDGFRB、ESRP1/RAF1、ETV6/ABL1、ETV6/ABL2、ETV6/ACSL6、ETV6/ARNT、ETV6/BAZ2A、ETV6/CDX2、ETV6/FGFR3、ETV6/FLT3、ETV6/GOT1、ETV6/ITPR2、ETV6/JAK2、ETV6/LYN、ETV6/MDS2、ETV6/MECOM、ETV6/NKAIN2、ETV6/NTRK3、ETV6/PDGFRA、ETV6/PDGFRB、ETV6/PER1、ETV6/PRDM16、ETV6/RUNX1、ETV6/SYK,EWSR1/ATF1、EWSR1/CREB1、EWSR1/DDIT3、EWSR1/ERG、EWSR1/ETV1、EWSR1/ETV4、EWSR1/FEV、EWSR1/FLI1、EWSR1/NFATC2、EWSR1/NR4A3、EWSR1/PATZ1、EWSR1/PBX1、EWSR1/POU5F1、EWSR1/SMARCA5、EWSR1/SP3、EWSR1/WT1、EWSR1/ZNF444、EXOC2/IGH、FCHSD1/BRAF、FGFR1OP/FGFR1、FGFR1OP/FGFR1、FGFR1OP2/FGFR1、FIP1L1/PDGFRA、FIP1L1/RARA、FOXO1/PAX3、FOXP1/ABL1、FUS/ATF1、FUS/CREB3L1、FUS/CREB3L2、FUS/DDIT3、FUS/ERG、FUS/FEV、FZD6/SDC2、GAPDH/BCL6、GIT2/PDGFRB、GOLGA4/PDGFRB、GOLGA5/RET、GOPC/ROS1、HAS2/PLAG1、HELIOS/BCL11B、ERVK-17/ETV1、HIP1/PDGFRB、HIST1H4I/BCL6、HMGA1/LAMA4、HMGA2/CCNB1IP1、HMGA2/COG5、HMGA2/COX6C、HMGA2/FHIT、HMGA2/LPP、HMGA2/NFIB、HMGA2/RAD51L1、HMGA2/WIF1、HMGN2P46/ETV1、HNRNPA2B1/ETV1、HOOK3/RET、HPR/MRPS10、HSP90AA1/BCL6、HSP90AB1/BCL6、IKZF1/BCL6、IL2/DEXI、IL2/TNFRSF17、IL21R/BCL6、INPP5D/ABL1、ITK/SYK、Ig/BCL11B、Ig/BCL3、Ig/BCL6、Ig/BCL7A、Ig/CCND1、Ig/CCND3、Ig/CDKN2A、Ig/FCGR2B、Ig/FCRL4、Ig/FOXP1、Ig/IL3、Ig/KDSR、Ig/LHX4、Ig/LHX4、Ig/MUC1、Ig/MYC、Ig/PAFAH1B2、Ig/WHSC1、Ig/WWOX、JAZF1/PHF1、JAZF1/SUZ12、KIAA1549/BRAF、KIF5B/ALK、KIF5B/PDGFRA、KIF5B/RET、KLK2/ETV4、KTN1/RET、LCK/TCR、LCP1/BCL6、LEO1/SLC12A1、LIFR/PLAG1、LRRFIP1/FGFR1、LYL1/TCR、MALAT1/ACAT2、MALAT1/TFEB、MALT1/MAP4、MEF2D/DAZAP1、MIR142/MYC、MLL/ABI1、MLL/ABI2、MLL/ACACA、MLL/AFF1、MLL/AFF3、MLL/AFF4、MLL/ARHGAP26、MLL/ARHGEF12、MLL/CASC5、MLL/CASP8AP2、MLL/CBL、MLL/CREBBP、MLL/DAB2IP、MLL/EEFSEC、MLL/ELL、MLL/EP300、MLL/EPS15、MLL/FLNA、MLL/FOXO3、MLL/GAS7、MLL/GMPS、MLL/GPHN、MLL/KIAA0284、MLL/KIAA1524、MLL/LASP1、MLL/LPP、MLL/MAML2、MLL/MAPRE1、MLL/MLLT1、MLL/MLLT10、MLL/MLLT11、MLL/MLLT3、MLL/MLLT4、MLL/MLLT6、MLL/MYO1F、MLL/NCKIPSD、MLL/NEBL、MLL/PICALM、MLL/PDS5A、MLL/SACM1L、MLL/SEPT11、MLL/SEPT2、MLL/SEPT5、MLL/SEPT6、MLL/SEPT9、MLL/SH3GL1、MLL/SORBS2、MLL/TET1、MLL/ZFYVE19、MN1/ETV6、MSI2/HOXA9、MSN/ALK、MYB/GATA1、MYB/NFIB、MYC/Ig、MYC/ZBTB5、MYH9/ALK、MYO18A/FGFR1、MYST3/ASXL2、MYST3/CREBBP、MYST3/NCOA2、MYST3/NCOA3、MYST4/CREBBP、NAV2/TCF7L1、NCOA4/RET、NDE1/PDGFRB、NDRG1/ERG、NDRG1/ERG、NFKB2/INA、NFKB2/TBXAS1、NIN/PDGFRB、NONO/TFE3、NOTCH1/TCR、NPM1/ALK、NPM1/MLF1、NPM1/RARA、NSD1/ANKRD28、NUMA1/RARA、NUP214/ABL1、NUP214/DEK、NUP98/ADD3、NUP98/CCDC28A、NUP98/DDX10、NUP98/HHEX、NUP98/HMGB3、NUP98/HOXA11、NUP98/HOXA13、NUP98/HOXA9、NUP98/HOXC11、NUP98/HOXC13、NUP98/HOXD11、NUP98/HOXD13、NUP98/IQCG、NUP98/KDM5A、NUP98/LNP1、NUP98/MLL、NUP98/NSD1、NUP98/PRRX1、NUP98/PRRX2、NUP98/PSIP1、NUP98/RAP1GDS1、NUP98/SETBP1、NUP98/TOP1、NUP98/WHSC1L1、OMD/USP6、P2RY8/CRLF2、PAX3/NCOA1、PAX3/NCOA2、PAX5/AUTS2、PAX5/BRD1、PAX5/C20orf112、PAX5/DACH1、PAX5/ELN、PAX5/ETV6、PAX5/FOXP1、PAX5/HIPK1、PAX5/JAK2、PAX5/PML、PAX5/POM121、PAX5/SLCO1B3、PAX5/ZNF521、PAX8/PPARG、PCM1/JAK2、PCM1/RET、PDE4DIP/PDGFRB、PEX5/LPL、PICALM/MLLT10、PIM1/BCL6、PML/RARA、POU2AF1/BCL6、PPP2R2A/CHEK2、PRKAR1A/RARA、PRKAR1A/RET、PRKG2/PDGFRB、PVRL2/TCR、RABEP1/PDGFRB、RANBP17/TCR、RANBP2/ALK、RBM15/MKL1、RBM6/CSF1R、RCSD1/ABL1、RNF213/ALK、RPN1/MECOM、RUNX1/AFF3、RUNX1/CBFA2T3、RUNX1/CLCA2、RUNX1/LPXN、RUNX1/MACROD1、RUNX1/RUNX1T1、RUNX1/SH3D19、RUNX1/TRPS1、RUNX1/USP42、RUNX1/YTHDF2、RUNX1/ZNF687、RYK/ATP5O、SEC31A/ALK、SEC31A/JAK2、SENP6/NKAIN2、SET/NUP214、SFPQ/ABL1、SFPQ/TFE3、SFRS3/BCL6、SLC34A2/ROS1、SLC45A3/BRAF、SLC45A3/ELK4、SLC45A3/ERG、SLC45A3/ETV1、SLC45A3/FLI1、SNX2/ABL1、SPECC1/PDGFRB、SPTBN1/FLT3、SQSTM1/ALK、SRGAP3/RAF1、SS18/SSX1、SS18/SSX2、SS18/SSX4、SS18L1/SSX1、SSBP2/JAK2、STAT5B/RARA、STRN/PDGFRA、TAF15/NR4A3、TAF15/ZNF384、TAL1/RHOA、TAL1/TCR、TCEA1/PLAG1、TCF12/NR4A3、TCF3/HLF、TCF3/NOP2、TCF3/PBX1、TCF3/TFPT、TCF3/ZNF384、TCR/LMO1、TCR/LMO2、TCR/MTCP1NB、TFG/ALK、TFG/NR4A3、TFG/NTRK1、TFRC/BCL6、THRAP3/USP6、TLX1/TCR、TMPRSS2/ERG、TMPRSS2/ERG、TMPRSS2/ETV1、TMPRSS2/ETV4、TMPRSS2/ETV5、TP53BP1/PDGFRB、TPM3/PDGFRB、TPM4/ALK、TPR/NTRK1、TRIM24/FGFR1、TRIM27/RET、TRIM33/RET、TRIP11/PDGFRB、VTI1A/TCF7L2、WDR48/PDGFRB、WWTR1/CAMTA1、ZBTB16/RARA、ZMIZ1/ABL1、ZMYM2/FGFR1、RUNX1/KIAA1549L、YAP1/TFE3、GTF2I/NCOA2、EWS/FLI1、SLC44A1/PRKCA、NAB2/STAT6、CUX1/AGR3、FGFR3/BAIAP2L1、FGFR3/TACC3、FGFR3/TACC3和NABP1/RARA。因此,对应于与癌基因重排有关的基因的MEI插入相邻序列提示,与该重排相关的治疗在针对该个体的治疗方案中将是有效的。
在一些情况下,根据从基因组分析获得的信息施用抗癌剂。化疗抗癌剂的实例包括:氮芥类(Nitrogen Mustards),如苯达莫司汀、苯丁酸氮芥、氮芥(chlormethine)、环磷酰胺、异环磷酰胺、美法仑、泼尼氮芥、氯乙环磷酰胺;烷基磺酸盐,如白消安、甘露舒凡、苏消安;乙烯亚胺,如卡波醌、塞替派、三亚胺醌;亚硝基脲,如卡莫司汀、福莫司汀、洛莫司汀、尼莫司汀、雷莫司汀、司莫司汀、链脲菌素;环氧化物,如依托格鲁;其他烷化剂,如达卡巴嗪、二溴甘露醇、哌泊溴烷、替莫唑胺;叶酸类似物,如甲氨蝶呤、培美曲塞、普拉曲沙、雷替曲塞;嘌呤类似物,如克拉屈滨、氯法拉滨、氟达拉滨、巯嘌呤、奈拉滨、硫鸟嘌呤;嘧啶类似物,如阿扎胞苷、卡培他滨、卡莫氟、阿糖胞苷、地西他滨、氟尿嘧啶、吉西他滨、替加氟;长春花生物碱,如长春碱、长春新碱、长春地辛、长春氟宁、长春瑞滨;鬼臼毒素衍生物,如依托泊苷、替尼泊苷;秋水仙素衍生物,如脱羰秋水仙碱;紫杉烷,如多西他赛、紫杉醇、聚谷氨酸紫杉醇;其他植物生物碱和天然产物,如曲贝替定;放线菌素,如更生霉素;蒽环霉素,如阿柔比星、柔红霉素、阿霉素、表阿霉素、伊达比星、米托蒽醌、吡柔比星、戊柔比星、佐柔比星;其他细胞毒性抗生素,如博来霉素、伊沙匹隆、丝裂霉素、普卡霉素;铂化合物,如卡铂、顺铂、奥沙利铂、赛特铂;甲基肼,如甲基苄肼;致敏剂,如氨基酮戊酸、乙丙昔罗、氨基酮戊酸甲酯、卟吩姆钠、替莫卟吩;蛋白激酶抑制剂,如达沙替尼、厄洛替尼、依维莫司、吉非替尼、伊马替尼、拉帕替尼、尼洛替尼、pazonanib、索拉非尼、舒尼替尼、西罗莫司;其他抗肿瘤剂,如阿利维甲酸、六甲蜜胺、安吖啶、阿那格雷、三氧化二砷、天冬酰胺酶、贝沙罗汀、硼替佐米、塞来考昔、地尼白介素(denileukin diftitox)、雌氮芥、羟基脲、伊立替康、氯尼达明、马索罗酚、米替福新、米托胍腙、米托坦、奥利默森(oblimersen)、培门冬酶、喷司他丁、罗米地辛、塞西马集(sitimagene ceradenovec)、噻唑呋林、拓扑替康、维甲酸、伏立诺他;雌激素,如二乙基二苯乙烯醇、炔雌醇、磷雌酚、磷酸聚雌醇;孕激素,如孕诺酮、甲羟孕酮、甲地孕酮;促性腺激素释放激素类似物,如布舍瑞林、戈舍瑞林、亮丙瑞林、曲普瑞林;抗雌激素,如氟维司群、他莫昔芬、托瑞米芬;抗雄激素,如比卡鲁胺、氟他胺、尼鲁米特;酶抑制剂,如氨鲁米特、阿那曲唑、依西美坦、福美坦、来曲唑、伏氯唑;其他激素拮抗剂,如阿巴瑞克、地加瑞克;免疫刺激剂,如组胺二盐酸盐、米伐木肽、匹多莫德、普乐沙福、罗喹美克、胸腺五肽;免疫抑制剂,如依维莫司、胍立莫司、来氟米特、霉酚酸、西罗莫司;钙调磷酸酶抑制剂,如环孢素、他克莫司;其他免疫抑制剂,如硫唑嘌呤、来那度胺、甲氨蝶呤、沙利度胺;以及放射性药物,如碘苄胍。
在一些实施方案中,抗癌剂是毒素,例如白喉毒素。在某些实施方案中,使生物相容性水凝胶聚合物负载有治疗有效量的一种或多种毒素以形成生物相容性水凝胶聚合物。毒素的实例包括外毒素,如白喉毒素、肉毒杆菌毒素、溶细胞素、溶血素(例如,金黄色葡萄球菌(Staphyllococcus aureus)的α毒素或α溶血素)、霍乱毒素、百日咳毒素、志贺毒素;来自大肠杆菌(E.coli)的热稳定肠毒素;箭毒;α-眼镜蛇毒素;维罗毒素-1(Verotoxin-1);以及来自百日咳博德特氏菌(Bordetella pertussis)的腺苷酸环化酶(AC)毒素。
在一些情况下,治疗包括施用特异性地以包含MEI插入相邻连续序列的核酸序列的降解为目标的组合物。
除了使用MEI边界来选择与与被上述MEI插入相邻序列标记的基因产物相关的途径或基因产物或基因有关的治疗之外,在一些情况下还使用MEI插入边界序列来开发直接靶向跨越MEI和插入相邻序列的序列的核酸靶向药物。本文设想包含跨越MEI和插入相邻边界序列的核酸序列的多种组合物。在一些情况下,这样的组合物的一个共同方面是,它们包含对跨越MEI边缘序列和插入相邻基因组序列二者的序列具有特异性并且长度不足以靶向单独的MEI序列或插入相邻序列的核酸组分。
也就是说,本文在许多情况下设想和公开的组合物在不存在插入相邻序列的情况下不与MEI结合,并且在不存在相邻MEI的情况下不与插入相邻序列结合;确切地说,本文公开的组合物包含与含有MEI和相邻基因组序列两者的序列进行特异性结合的核酸组分。因此,在用这样的组合物治疗后,只有与MEI插入相邻序列对应的核酸,例如本文公开的已经在时间或空间测定(例如,如上文公开的)中被鉴定为明显过度表示的核酸,才会被该组合物所靶向,而其他MEI和包含插入相邻序列但不含MEI序列的未插入的等位基因不被该组合物结合。在一些情况下,该组合物的核酸组分包含MEI序列的3、4、5、6、7、8、9、10个或超过10个碱基,以及插入相邻序列的3、4、5、6、7、8、9、10个或超过10个碱基,使得该组合物与单独的MEI之间或者该组合物与单独的插入相邻序列之间的结合能不足以保证结合。
例如,如本文公开的组合物包含与指导包含MEI和插入相邻序列的靶序列的内切核苷酸切割的部分组合的、具有如上所述的特征的引导核酸。
在一些实施方案中,所述引导核酸分子为引导RNA分子。在一些情况下,例如通过募集具有内切核酸酶活性的蛋白质如Cas9蛋白,该引导RNA分子或其他引导核酸分子指导与其结合的DNA分子的内切核苷酸切割。锌指核酸酶(ZFN)、转录激活物样效应物核酸酶和基于成簇规律间隔短回文重复序列/Cas的RNA指导的DNA核酸酶(CRISPR/Cas9)等与本公开内容的一些实施方案相匹配。
引导RNA分子或其他引导核酸分子包含与将从测序中去除的靶序列发生碱基配对的序列(靶序列区内的非靶序列)。在一些实施方案中,该碱基配对是完全的,而在一些实施方案中,该碱基配对是部分的,或包含未配对的碱基以及与非靶序列配对的碱基。
引导RNA分子或其他引导核酸分子可包含一个或多个形成‘发夹’结构的区域。这样的一个或多个区域包含部分或完全回文的序列,使得该区域的5’和3’端可彼此杂交形成双链‘茎'结构,在一些实施方案中该结构被非回文环加帽,从而将双链环中的每条单链彼此拴住。
在一些实施方案中,引导RNA分子或其他引导核酸分子包含茎环,如tracrRNA茎环。茎环如tracrRNA茎环可与核酸内切核酸酶如Cas9DNA内切核酸酶复合或结合。或者,茎环可与除Cas9之外的内切核酸酶复合,或与除内切核酸酶之外的核酸修饰酶如碱基切除酶、甲基转移酶或具有干扰一种或多种DNA聚合酶的其他核酸修饰活性的酶复合。
tracrRNA/CRISPR/内切核酸酶系统被鉴定为真细菌和古细菌原核生物中的适应性免疫系统,细胞借由该系统获得对具有已知序列的病毒的反复感染的抗性。参见,例如,Deltcheva E,Chylinski K,Sharma CM,Gonzales K,Chao Y,Pirzada ZA等人(2011)"CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III"Nature 471(7340):602–7.doi:10.1038/nature09886.PMC 3070239.PMID 21455174;Terns MP,Terns RM(2011)"CRISPR-based adaptive immune systems"Curr Opin Microbiol 14(3):321–7.doi:10.1016/j.mib.2011.03.005.PMC 3119747.PMID 21531607;Jinek M,Chylinski K,Fonfara I,Hauer M,Doudna JA,Charpentier E(2012)"A Programmable Dual-RNA-Guided DNA Endonuclease in Adaptive Bacterial Immunity"Science 337(6096):816–21.doi:10.1126/science.1225829.PMID 22745249;以及Brouns SJ(2012)"A Swiss army knife of immunity"Science 337(6096):808–9.doi:10.1126/science.1227253.PMID 22904002。该系统已被调适用于在真核细胞中引导靶向诱变。参见,例如,Wenzhi Jiang,Huanbin Zhou,Honghao Bi,Michael Fromm,Bing Yang和Donald P.Weeks(2013)"Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification in Arabidopsis,tobacco,sorghum and rice"Nucleic Acids Res.Nov 2013;41(20):e188,2013年8月31日在线发表.doi:10.1093/nar/gkt780,以及其中的参考文献。
如本文所设想的,在一些实施方案中使用引导RNA分子或其他引导核酸分子来提供对DNA内切核酸酶如Cas9内切核酸酶的序列特异性。在这些实施方案中,引导RNA分子或其他引导核酸分子包含与内切核酸酶如Cas9结合或被内切核酸酶如Cas9结合的发夹结构(在一些实施方案中设想其他内切核酸酶作为替代物或添加物),并且引导RNA分子或其他引导核酸分子进一步包含与将从测序文库或测序反应中去除的序列结合或特异性结合或排他性地结合的识别序列。引导RNA分子或其他引导核酸分子中的识别序列的长度可根据序列消除过程中期望的特异性程度而变化。如上文所讨论的,核酸特异性在许多情况下被解释为以下情况:RNA分子或其他引导核酸分子与MEI-插入相邻序列接合点特异性结合,但不与单独的MEI也不与单独的插入相邻序列结合。包含在样品中频繁出现的序列或包含差异丰富序列(富含AT的基因组样品中的AT丰度或富含GC的基因组样品中的GC丰度)的短识别序列有可能鉴定相对大量的位点,并因此引导频繁的核酸修饰,如内切核酸酶活性、碱基切除、甲基化或干扰至少一种DNA聚合酶活性的其他活性。包含在样品中不频繁出现的序列或包含表示不足的碱基组合(富含AT的基因组样品中的GC丰度或富含GC的基因组样品中的AT丰度)的长识别序列有可能鉴定相对少量的位点,并因此引导不频繁的核酸修饰,如内切核酸酶活性、碱基切除、甲基化或干扰至少一种DNA聚合酶活性的其他活性。因此,如本文公开的,在一些实施方案中,通过对识别序列长度的修饰,可以调节从测序反应中去除序列的频率,以便特异性地靶向单个MEI-插入相邻序列。
引导RNA分子或其他引导核酸分子可通过多种符合本公开内容的方法合成。可使用标准合成技术来产生大量的引导RNA分子或其他引导核酸分子。双链DNA分子可包含RNA分子或其他引导核酸分子位点特异性结合序列、引导RNA分子或针对Cas9蛋白和T7启动子位点的其他引导核酸分子序列。在一些情况下,该双链DNA分子的长度可小于约100bp。可使用T7聚合酶来创建单链RNA分子,该单链RNA分子可包含靶RNA序列和针对Cas9蛋白的引导RNA序列。
例如,本文公开的组合物包含具有如上所述的MEI插入相邻序列结合特征的引导核酸,该引导核酸引导插入相邻序列中的基因的沉默,使得基因产物如癌基因产物、导致例如细胞周期调节、细胞生长调节或细胞分裂调节缺陷的基因产物的截短或以其他方式突变的等位基因在插入相邻序列处在被引导核酸结合时得以沉默。在一些情况下,引导核酸包含siRNA部分、piRNA部分或参与基因沉默、基因产物的转录调节或转录后调节的其他核酸部分。
siRNA和piRNA是与基因沉默有关的小RNA分子。向生物体中引入dsRNA可导致基因表达的特异性干扰。该现象被称为RNA干扰(RNAi),由特异性靶向mRNA以便被植物、无脊椎动物和哺乳动物细胞中的细胞机制降解而导致。本领域已知的示例性RNAi技术包括但不限于siRNA、shRNA和piRNA。RNAi机制的组分包括靶向靶基因的dsRNA(siRNA或shRNA)、Dicer、Argonaute蛋白家族(特别是Ago-2)、Drosha、RISC、TRBP和PACT。小干扰RNA(siRNA)通常被认为是具有2nt 3’端突出端的dsRNA,其激活RNAi,导致mRNA以依赖于靶mRNA的互补结合的序列特异性方式降解。shRNA通常被认为是含有环结构的短发夹RNA(shRNA),其被加工成siRNA并且也导致mRNA以依赖于靶mRNA的互补结合的序列特异性方式降解。Drosha通常被认为是在细胞核中加工pri-miRNA和shRNA的RNA酶III酶。Dicer通常被认为是将dsRNA加工成20-25bp的siRNA,而在3'端留下2nt突出端的核糖核酸酶(RNA酶)III酶。果蝇(Drosophila)Dicer-2切割长dsRNA,而Dicer-1对miRNA加工是重要的。RISC通常被认为是由Argonaute蛋白质和缔合的siRNA组成的最小RNA诱导的沉默复合物(RISC)。其还可含有PACT、TRBP和Dicer。应注意,RISC的确切组成尚未得到描述。TRBP通常被认为是Dicer对dsRNA的切割以及随后向RISC的传递所需的。蛋白R(PKR)活化蛋白(PACT)通常被认为与用于dsRNA切割的Dicer和TRBP相关。与单链siRNA一起,argonaute蛋白家族组装形成RISC,结合21–35nt RNA(包括miRNA和siRNA)以及其关联的靶mRNA,随后通过其核酸内切功能将它们切割。
小干扰RNA(siRNA)——有时被称为短干扰RNA或沉默RNA,是一类双链RNA分子,通常为20-25个碱基对的长度。siRNA是RNA干扰(RNAi)途径中最值得注意的,其中其干扰具有互补核苷酸序列的特定基因的表达。siRNA通过使mRNA在转录后分解来起作用,导致不发生翻译。siRNA还在RNAi相关途径中起作用,例如,充当抗病毒机制,或在塑造基因组的染色质结构中起作用。
当在siRNA或shRNA之间进行选择时,要考虑的重要因素是治疗的长度。siRNA在细胞中瞬时表达,而shRNA可通过病毒介导的转导而被稳定地整合。siRNA设计的指导方针包括:(1)通常推荐19–29nt的siRNA序列以避免非特异性沉默,(2)包含AA二核苷酸的靶向位点,和(3)具有3’dUdU或dTdT二核苷酸突出端的siRNA使有效性增强。通常,siRNA序列应该具有35–55%的G/C含量。
由于不同的细胞类型对核酸的引入具有不同的敏感性,因此用于递送RNAi的方案将取决于细胞类型。转染、电穿孔和某些病毒递送方法是瞬时的。
最常见的核酸递送方法包括转染和电穿孔。转染包括核酸与载体分子的复合物的形成,这允许它们穿过细胞膜。转染方法包括脂质转染,在脂质转染中,具有带有带正电荷的头部基团的长疏水链的阳离子脂质与带负电荷的siRNA相互作用,从而将其包围在脂双层中,该脂双层随后被细胞内吞;基于阳离子聚合物的纳米颗粒,其允许降低的毒性和增加的效率,并且允许递送修饰的siRNA;以及脂质或细胞穿透肽(CPP)缀合,其包括siRNA与疏水部分(例如,胆固醇)或阳离子CCP(例如,转运蛋白或pentatratin)的缀合,这促进了向靶细胞内的递送。
在电穿孔方法中,向由具有带负电荷的头部基团的磷脂分子组成的细胞膜施加电场。电脉冲导致磷脂重新定向,在膜中产生孔,从而允许siRNA进入。电穿孔常用于难以转染的细胞。然而,必须针对每种细胞或组织类型来优化具体的设置(电压、脉冲数和脉冲的长度)。
已知RNAi干预对癌症、神经疾病、病毒感染、黄斑变性、糖尿病性视网膜病和丙型肝炎以及其他病症具有治疗价值。
转座子沉默是以转座子为目标的转录基因沉默的一种形式。转录基因沉默是防止DNA的该区域转录的组蛋白修饰的结果。转座子的转录沉默对基因组的维持至关重要。转座子的“跳跃(jumping)”产生基因组不稳定性并且可以导致非常有害的突变。转座因子插入与包括血友病、重症联合免疫缺陷和癌症倾向在内的多种疾病有关。因此,为阻止转座子突变发展并传递到下一代,转座子的沉默在种系中是非常重要的。
最大的一类小RNA——Piwi相互作用RNA(piRNA)的长度在26到31个核苷酸之间,并且其通过与来自Argonaute蛋白家族的piwi蛋白(基因沉默蛋白质)相互作用而起作用。本领域已知与PIWI蛋白结合的piRNA采用转录后转录物破坏来使转座子沉默。大多数piRNA相对于从沉默的转座子转录的mRNA是反义的,其通常与Piwi和Aubergine(Aub)蛋白相缔合,而相反,有义链piRNA倾向于与Argonaute 3(Ago3)缔合。被称为“乒乓”扩增的循环在有义和反义piRNA之间进行,其包括大量的修剪和加工以产生成熟的piRNA。该过程导致种系中大多数piRNA的产生,并且还可以解释piRNA在种系发育中的起源。Piwi-piRNA复合物通过增加转座子区之内或上游的CpG甲基化,和/或转座子区周围的染色质修饰,或通过直接降解转座子的转录物来抑制转座子表达。
可替代地或组合地,在一些情况下,选择与应对癌症有关的治疗,该癌症与细胞生长、细胞周期或细胞增殖途径(与MEI有关的基因为其编码参与成员)的误调节有关。例如,TOR(雷帕霉素信号传导的靶标)的负调节物如TSC2基因座中的MEI提示采用生长调节抑制剂的治疗,而编码视网膜母细胞瘤抑制基因Rb的基因座中的MEI提示与细胞周期进展有关的治疗。
在一些情况下,在个体中的位置之间或在来自个体的相同样品来源的时间之间比较MEI水平。
在一些情况下,使用血液作为待测定核酸如游离循环核酸的来源,以单独地或与替代监测方法组合地用于MEI水平的持续时间监测。可替代地或组合地,在一些实施方案中使用来自其他来源的循环游离DNA或其他DNA。
提取循环游离核酸的方法是本领域已知的。当核酸在细胞内时,提取程序通常包括细胞裂解(通常通过对样品进行化学和物理法混合、研磨或超声处理而实现),通过添加去污剂或表面活性剂(也用于细胞裂解)去除膜脂质,任选地通过添加蛋白酶去除蛋白质,任选地通过添加RNA酶去除RNA(当DNA为所需靶标时进行)。DNA纯化方法是本领域已知的。示例性的DNA纯化方法包括但不限于乙醇沉淀、酚-氯仿提取和微型柱纯化。可使用冰冷的乙醇或异丙醇完成乙醇沉淀。由于DNA在这些醇中是不可溶的,因此其将聚集在一起,在离心后得到沉淀物。通过增加离子强度(通常通过添加乙酸钠)来改善DNA的沉淀。酚-氯仿提取使样品中的蛋白质变性。在样品离心后,变性的蛋白质留在有机相中,而含有核酸的水相与氯仿混合,从而将酚残留物从溶液中去除。对于微型柱纯化,根据缓冲液的pH和盐含量,核酸与固相(二氧化硅或其他)结合,随后洗脱。
供提取的循环核酸的示例性形式包括但不限于DNA、RNA、mRNA寡聚核小体的、线粒体的、表观遗传学修饰的、单链的、双链的、环状的,质粒、粘粒、酵母人工染色体人工或人造DNA(如包括独特DNA序列)、以及从RNA样品逆转录的DNA如cDNA,及其组合。用于核酸提取的示例性生物来源包括但不限于全血、血清、血浆、脐带血、绒膜绒毛、羊水、脑脊液、脊髓液、灌洗液(例如,支气管肺泡的、胃的、腹膜的、导管的、耳的、关节镜的(athroscopic))活检样品、尿液、粪便、痰液、唾液、鼻粘膜、前列腺液、精液、淋巴液、胆汁、泪液、汗液、乳汁、乳房流体、胚胎细胞和胎儿细胞。该生物样品可以是含有核酸的任何组织或流体。示例性的生物样品包括但不限于石蜡包埋的组织、冷冻的组织、手术细针抽吸物,以下的细胞:皮肤、肌肉、肺、头和颈、食管、肾、胰腺、口、咽喉、咽、喉、食道、筋膜(facia)、脑、前列腺、乳腺、子宫内膜、小肠、血细胞、肝、睾丸、卵巢、子宫、子宫颈、结肠、胃、脾、淋巴结、骨髓或肾。流体样品可包括支气管刷出物、支气管洗液、支气管破坏物、外周血淋巴细胞、淋巴液、腹水、浆液、胸腔积液、痰液、脑脊液、泪液、食道洗液以及粪便或尿液标本如膀胱洗液和尿。
如上文讨论或本领域已知的核酸样品来源以一个或多个时间间隔获得,并且获得核酸以用于MEI插入边界丰度的定量评价。时间点可相隔数日、数周、数月或数年,如1个月、2个月、3个月、4个月、5个月、6个月、1年、2年、3年、4年、5年、10年或超过10年。
在一些情况下,时间点被部分或完全执行治疗方案如肿瘤或其他癌性组织切除而隔开,或被施用以消除肿瘤或癌性组织为目标的治疗如化疗或放疗而隔开。在一些情况下,如上文公开的治疗方案和组合物预期用于治疗方案的时间分析。
因此,使用针对与过度增殖细胞有关的MEI的MEI水平定量,例如,以便监测干预的效力,其中MEI水平的降低表明有效力,或者MEI相对水平的升高速率的降低表明有效力,或者MEI插入边界的相对量稳定在稳定水平表明有效力。
除样品的时间分隔之外,本文还涉及样品的空间分隔。因此,在一些情况下,从第一区域或组织取得与肿瘤或癌活性在表型上不相关的样品,并从疑似具有癌活性或癌前活性或者观察为肿瘤或癌的第二区域或组织取得第二样品。
在一些情况下,从癌或肿瘤内的多个区域如静止区域和有丝分裂活跃或增殖活跃区域取得样品,使得与肿瘤增生、生长、细胞分裂或转移有关的细胞与良性、静止或衰老肿瘤组织相关细胞分开。
在一些情况下,将肿瘤组织在空间上区分,使得例如内部和边缘的细胞群体分开提取。可替代地或组合地,通过表面特征或生物标志物对肿瘤细胞进行分选。
若干细胞分选方法是本领域已知的。示例性的细胞分选的类型包括但不限于荧光激活细胞分选(FACS)、磁性细胞选择和单细胞分选。单细胞分选提供了根据细胞内和细胞外性质分选细胞的不均匀混合物的方法。FACS利用流式细胞术提供细胞内和细胞外性质(不包括形态学)的定量测量,以供分选细胞的不均匀混合物。磁性细胞分选提供了根据细胞外性质(通常是细胞表面蛋白质(即,抗原))富集细胞的不均匀混合物的方法。磁性激活细胞分选(MACS)是基于柱子的分离技术,其中使标记的细胞通过磁柱。SEP系统提供了不使用柱子的细胞分离技术,其中将含有标记的细胞的管放置在磁场内。阳性选择的细胞保留在管中,而阴性选择的细胞存在于液体悬浮液中。细胞分选方法包括特异性结合癌症生物标志物以分选细胞的分选剂(例如,抗体)。
示例性的癌症生物标志物包括但不限于CCR10、CD9、CD13、CD15、CD24、CD26、CD29、CD32、CD46、CD49a、CD49b、CD49c、CD49f、CD51、CD54、CD55、CD56、CD58、CD63、CD66a、CD66c、CD66e、CD71、CD73、CD81、CD82、CD91、CD98、CD99、CD102、CD104、CD105、CD108、CD111、CD117、CD118、CD130、CD131、CD133、CD136、CD141、CD146、CD147、CD148、CD151、CD155、CD157、CD164、CD166、CD167a、CD172a、CD177、CD186、CD196、CD221、CD230、CD234、CD244、CD245、CD262、CD265、CD273、CD275、CD295、CD298、CD299、CD317、CD318、CD324、CD340、BMPR-1B、钙粘蛋白-11、c-Met、密蛋白-3(Claudin-3)、DLL-1、DLL-3、Eph-B2、Eph-B4、FOLR1、Frizzled-3、Glut-1、Glut-2、磷脂酰肌醇聚糖5、HLA-A/B/C、HLA-A2、HER3、IL-15R、IL-20Ra、jagged-2、整联蛋白-a8、整联蛋白a9b1、整联蛋白b5、LAG-3、白三烯-B4R、Lox-1、LDL-R、MCSP、mer、柄蛋白-4(nectin-4)、notch2、NPC、PD-L2、丛蛋白-B1、脑信号蛋白4B、促生长素抑制素-R2(somatostatin-R2)、TROP-2、ULBP2、整联蛋白aVb9和VEGFR2。在单细胞分选和FACS的情况下,生物标志物可以是细胞内或细胞外的。
比较样品之间的MEI水平,以鉴定在第二样品中差异性过度丰富的MEI插入接合点。如本文所述,在一些情况下,一个样品中差异丰富的MEI插入接合点比另一样品丰富10%、20%、30%、40%、50%、70%、100%、2倍、2.5倍、3倍、3.5倍、4倍、5倍或超过5倍。
使用被鉴定为在假定不健康的组织中差异性存在的MEI插入边界来指导如上所述的治疗选择。使用被鉴定为在假定不健康的组织中差异性存在的MEI插入边界来监测疾病进展或治疗效力,使得相对水平的降低,或相对水平的稳定化,或相对水平增加速率的降低表明有治疗效力。
在一些情况下,使用与过度增殖细胞活性有关的MEI-插入相邻序列来监测在鉴定的肿瘤或癌症部位之外的肿瘤或癌症或癌前细胞扩充,使得来源于假定健康组织的样品中MEI插入位点的相对丰度的增加指示该样品所源自的组织为潜在癌前或癌性的风险。
在一些实施方案中,提供详述MEI定量测序分析的结果的报告。例如,该报告包含关于经时程相对于治疗方案的或在一个组织或区域中相对于另一个的MEI相对丰度水平的信息。在一些情况下,该报告随附有治疗推荐,该治疗推荐关联于与MEI插入位点或与过度增殖细胞有关的位点相邻的序列的身份或由该身份获知。在各个实施方案中,这样的治疗推荐包括化疗、放疗、组织切除或其组合。在一些情况下,该治疗针对与MEI插入位点有关的破坏基因的产物,而在一些实施方案中,该治疗针对破坏基因的产物所参与的途径的成员的误调节。例如,如果负调节物被破坏(如通过MEI插入表明的),则治疗可针对由于MEI插入破坏而预期将会上调的下游信号组分。
在一些情况下,将报告提供给个体,而在一些情况下,将报告提供给医疗保健专业人员。在一些情况下,报告以保密形式提供,使得其不被提供给公众,而是仅直接提供给提供样品的个体或该个体及相关的医疗保健专业人员,或保密地提供给医疗保健专业人员。
多种方法可用于MEI-插入相邻序列定量。重复元件如MEI序列如何通过全基因组测序进行定量分析的概念性实例如下。
在一些情况下,此处获得的序列信息用于样品中的核酸序列丰度。如本文公开或如本领域已知的那样生成文库并对文库进行测序。排除重复读序,使得仅包括独特标记的读序。将独特读序映射至基因组序列。对映射至靶区域的独特文库序列读序的数目进行计数并用于表示该序列在样品中的丰度。在一些实施方案中,独特标记的序列读序均映射至样品序列中的单个位点。在一些情况下,独特标记的序列读序映射至整个基因组中的多个位点,如转座子插入位点或重复元件位点。因此,在一些情况下,映射至转录物组‘基因座’或转录物的文库分子的数目与该转录物在产生文库的样品中的累积水平相对应。相对于映射至基因组的给定独特区域的文库分子的数目,映射至重复元件的文库分子的数目指示该重复元件在样品中的相对丰度。映射至给定MEI插入接合点的序列读序用来定量给定样品中的该插入接合点。因此,通过比较跨越MEI插入边界的读序的数目,相对于例如样品中的其他序列,如已知在样品的健康单倍体基因组中为单拷贝的序列,定量了该插入边界。
因此,定量样品中核酸分子序列的相对丰度通过生成包含独特标记的文库片段的序列文库并将该核酸分子序列映射至该文库上而实现,例如该核酸分子序列在该文库中的出现频率与该核酸分子序列在产生该文库的样品中的丰度相对应。在一些情况下,相对于第二核酸分子序列在该文库中的出现频率评价核酸分子序列在该文库中的出现频率,所述第二核酸序列与在转录物组中具有已知丰度或对于基因组样品的每个基因组具有已知拷贝数的基因座或转录物相对应。
下文提供了用于核酸样品中核酸序列定量的更加详细的方案。然而,需要强调的是,本文公开的方法不限于任何一种在核酸样品中的核酸序列定量方法。
从基因组中每个可能的位置生成下一代测序(NGS)文库需要无偏倚的方法,以将基因组DNA(gDNA)模板转化成适当大小的、平台特异性测序衔接子在gDNA侧翼的文库分子。这可以采用如下式所示的具有测序衔接子尾部的随机引物来进行:5’-衔接子序列-NNNNNNNN-3’。
为了使给定基因组的偏倚最小化,可以以半随机方式合成引物的“随机”部分,以应对感兴趣的基因组中的可变内含物。可将给定基因组(例如,人类基因组)分解成具有不同GC含量的100bp窗口。理想地,合成包含代表性“随机性”的引物,该“随机性”针对基因组中1%至100%GC的GC含量的窗口而排列,并且以相对于每个GC%下的基因组含量的比例合成并合并这些引物。
随机引发可允许基因组的每个碱基被表示为测序仪读序的起始位置。为了在基因组中每个可能的碱基处结束每个文库分子,需要用来终止从随机引物开始的聚合的随机/无偏倚方法。要做到这一点,可以使用含有固定比例的四种天然核苷酸中的每一种的ddNTP与固定比例的缺少3’-OH基团的双脱氧核苷酸的混合物。ddNTP与dNTP的比例可以决定在任意给定碱基位置终止的可能性。例如,1%ddNTP混合物(99%dNTP)将给出这样的可能性,即99%的从随机引物延伸的分子将越过第一碱基聚合。该相同的实例将给出50bp的N50(50%的分子将长于N个碱基)。随着相对ddNTP比例降低,N50插入大小增加。因此,在某些条件下,0.8的ddNTP%导致中值插入大小(N50)为62.5,并且包含衔接子和随机引物的全长文库分子的相应N50为198.5,0.4的ddNTP%导致中值插入大小(N50)为125,并且包含衔接子和随机引物的全长文库分子的相应N50为261,0.2的ddNTP%导致中值插入大小(N50)为250,并且包含衔接子和随机引物的全长文库分子的相应N50为386,0.1的ddNTP%导致中值插入大小为500,并且包含衔接子和随机引物的全长文库分子的相应N50为636,而0.05的ddNTP%导致中值插入大小为1000,并且包含衔接子和随机引物的全长文库分子的相应N50为1136。对于低复杂度的区域,如一段AT或GC,该基因组位置中ddNTP的有效浓度将减半,导致在1%ddNTP混合物下,对于在这样的低复杂度基因组基因座中发生的引物延伸反应,N50为100个核苷酸。(未考虑全部8种核苷酸之间的聚合酶掺入效率差异)。
调节反应中的ddNTP%可调节聚合的分子的范围和多样性。ddNTP浓度对片段长度和腺嘌呤-酪氨酸偏倚的影响在图11中示出。ddNTP浓度对产率的影响在图12中示出。在0.4%ddNTP下,300-1000bp(摩尔)的摩尔浓度为27.5;在0.2%ddNTP下,300-1000bp(摩尔)的摩尔浓度为16.1;在0.1%ddNTP下,300-1000bp(摩尔)的摩尔浓度为5.8;而在0.05%ddNTP下,300-1000bp(摩尔)的摩尔浓度为4.9。图13示出了按照大小选择的分子的读取位置。
另一个步骤可以是将衔接子标记的分子与gDNA模板以及任何过量的反应物如引物和过量的NTP分离。这可以通过使用生物素化的ddNTP完成。可使用链霉亲和素涂覆的磁珠来实现该分离。
聚合酶的选择可限于具有链置换以及ddNTP/生物素掺入能力的酶。SEQUENASE和THERMOSEQUENASE(Affymetrix,Santa Clara,CA)是两种这样的酶。如果由于缺少样品资源或强制稀释而需要低输入量,则可通过使用酶混合物如SEQUENASE和Phi29(一种缺乏掺入ddNTP能力的高度持续性聚合酶)优化反应以改善产率。phi 29酶将增加用于在反应中被SEQUENASE加工的模板量。还可以通过优化反应持续时间来提高模板的产率和多样性。
这样的测序反应的产物由下式表示:5’-衔接子-NNNNNNNN-基因组插入片段-ddNTP/生物素。
目前的商用测序仪需要gDNA插入片段的侧翼为2个衔接子序列。可通过第二随机引发反应添加第二衔接子。从磁珠上分离的产物可用作使用随机引物与第二衔接子的第二随机引发反应的模板,如下式所示:5’-衔接子2-NNNNNNNN-3’。置换的产物也可用作使用随机引物与第二衔接子的第二随机引发反应的模板。
用于第二衔接子添加的酶可以不需要掺入ddNTP的能力。链置换可能是必需的。可接受的酶包括SEQUENASE、THERMOSEQUENASE、Phi29、Bst DNA聚合酶和Taq DNA聚合酶。引物的随机部分可以与珠子结合的模板结合并通过该模板分子的末端延伸。在最靠近模板的3'端处结合的引物可以置换在下游结合的引物,使得将产生单拷贝的具有第一和第二衔接子两者的与珠子结合的模板。该拷贝可与磁珠保持氢键键合。可通过珠子洗涤去除过量的引物、NTP、酶和置换产物。可对所得的产物进行热变性(将其从珠子上释放),并测序或通过采用与衔接子互补的引物的PCR进行扩增。由此产生的产物由下式表示(以3’至5’方向示出):3’-衔接子1-NNNNNNNN-gDNA插入片段-NNNNNNNN-衔接子2-5’。
NGS测序中的关键错误模式是文库制备中的错误的克隆扩增。对于无PCR方案,这可能很少受到关注,但任何低输入方案都需要扩增来获得足够的文库以加载到测序仪上。扩增过程中引入的错误可在测序仪中显露出来。这些错误的标准减少是从分析中去除重复。然而,如果给予样品足够的测序能力,则重复读序(具有相同起始和终止位置的读序)可自然发生。因此,去除这些读序将降低该试验的覆盖度和准确性。在分析中使用合成随机引物可允许相对于低频突变准确确定克隆矫作物。PCR重复可在两端上具有相同的随机引物序列,而由于深度测序覆盖导致的重复可具有不同的随机引物序列。由于合成序列总是在每个读序的相同位置处,因此该信息可以很容易地在分析中获得。
非终止性合成测序化学法(如Qiagen和ION Torrent)在对均聚物的长段进行测序时遇到困难。这可以通过复杂文库生成来减轻,这种复杂文库生成通过在本文所述的均聚物的每个碱基处的终止来实现。
因此,与以上公开内容一致,生成了第一链寡核苷酸文库。为了生成随机文库,合成了第一轮合成寡核苷酸的群体。第一链寡核苷酸均包含位于随机寡聚物序列(如2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30聚物或更大的寡聚物)的5’侧的序列衔接子,该序列衔接子之后为发生模板引导的延伸的3’OH。在一些情况下,该序列衔接子被配置为包含可变标识符序列。在替代情况下,该序列衔接子是不变的。在一些情况下,使用序列衔接子作为引物结合位点进行测序衔接子如A衔接子的后续添加,例如通过经由扩增的标准引物引导的序列添加。
在一些情况下,合成了寡核苷酸群体,使得给定随机寡聚物碱基序列(如随机5、6、7、8、9或10聚物)的所有可能的组合都在第一链寡核苷酸群体中呈现。在其他情况下,特别是当选择长随机寡聚物时,但偶尔也在较小寡聚物的情况下,存在给定随机寡聚物碱基序列的部分可能的组合。
在一些情况下,随机寡聚物的碱基以相等的比例呈现核酸碱基的无偏倚的随机分布。在一些情况下,每个碱基在给定位置出现或在随机寡聚物群体中总计出现的可能性相同。然而,在其他情况下,为了增加退火和随后第一链合成的效率,合成了群体,使其包含具有某些碱基或碱基对的偏倚呈现的随机寡聚物(如随机8聚物)的偏倚。例如,据观察,人类基因组具有约40%的GC百分比,而不是从真正随机碱基丰度预期的50%GC组成。参见例如图10。在一些情况下,随机寡聚物分布是偏倚的,使得第一链合成文库中随机寡聚物序列(如8聚物序列)的总体分布反映出偏斜的靶标平均值如靶基因组、靶基因座、靶基因家族、靶基因组元件(例如外显子、内含子或启动子序列)的平均值的总体分布,或在一些实施方案中,匹配作为整体的人类基因组。
使第一链寡核苷酸文库或代表第一链寡核苷酸文库的90%、80%、70%、60%、50%、40%、30%、20%、10%或少于10%的寡核苷酸文库子集与包含核酸如脱氧核糖核酸或核糖核酸的样品接触。核酸如DNA或RNA可以以宽范围的量提供。在一些情况下,基因组DNA样品以等于或约为诸如1ng、2ng、3ng、4ng、5ng、6ng、7ng、8ng、9ng、10ng、11ng、12ng、13ng、14ng、15ng、16ng、17ng、18ng、19ng、20ng、21ng、22ng、23ng、24ng、25ng、26ng、27ng、28ng、29ng、30ng、31ng、32ng、33ng、34ng、35ng、36ng、37ng、38ng、39ng、40ng、41ng、42ng、43ng、44ng、45ng、46ng、47ng、48ng、49ng、50ng、51ng、52ng、53ng、54ng、55ng、56ng、57ng、58ng、59ng、60ng、61ng、62ng、63ng、64ng、65ng、66ng、67ng、68ng、69ng、70ng、71ng、72ng、73ng、74ng、75ng、76ng、77ng、78ng、79ng、80ng、81ng、82ng、83ng、84ng、85ng、86ng、87ng、88ng、89ng、90ng、91ng、92ng、93ng、94ng、95ng、96ng、97ng、98ng、99ng或100ng的量,或在由上述列表限定的范围之外的数值的量提供。从下文可以看出,下游热循环的数目将随着起始模板量的增加而减少。在一些情况下,RNA样品由从少至1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、66、67、68、69、70、71、72、73、74、75、76、77、78、79、80、81、82、83、84、85、86、87、88、89、90、91、92、93、94、95、96、97、98、99、100个细胞或多于100个细胞的细胞群体提取的RNA提供。
还向混合物中添加包含与DNA聚合酶活性一致的试剂的聚合酶缓冲液。多种聚合酶符合本公开内容。在一些情况下,示例性的聚合酶具有链置换活性、ddNTP掺入活性,并且能够掺入生物素标记的核苷酸如生物素标记的ddNTP。示例性的聚合酶是测序酶(Sequenase),而示例性的逆转录酶是HIV逆转录酶。
还向混合物中添加核苷酸群体,如包含dATP、dTTP、dCTP和dGTP的群体,以及在一些情况下还包含ddNTP如ddATP、ddTTP、ddCTP和ddGTP的群体。在一些情况下,仅向dNTP群体中添加单种ddNTP,如单独的ddATP、单独的ddTTP、单独的ddCTP和单独的ddGTP。在一些情况下,添加了ddNTP对,如ddATP和ddTTP,或ddCTP和ddGTP。
在一些情况下,添加至组合物的ddNTP群体如ddATP、ddTTP、ddCTP和ddGTP包含至少一种生物素标记的ddNTP,如生物素标记的ddATP、生物素标记的ddTTP、生物素标记的ddCTP和生物素标记的ddGTP。
dNTP/ddNTP比例的范围符合本公开内容。99.9%/0.1%、99.5%/0.5%、99%/1%、98%/2%的比例和替代的比例符合本公开内容。在一些情况下,选择了99%脱氧NTP与1%双脱氧NTP的相对比例。
在一些情况下通过加热至解链温度以上,如95℃、96℃、97℃、98℃或99℃或更高的温度,使混合物变性。在许多情况下,低于100℃的变性温度是示例性的。
然后将混合物例如在冰上冷却30秒、1分钟、2分钟或多于2分钟,或在4℃下冷却30秒、1分钟、2分钟或多于2分钟,或在足以允许第一链合成寡核苷酸与核酸样品如基因组DNA样品或RNA样品之间反向互补碱基配对的备选冷却温度下冷却。在一些情况下,第一链合成寡核苷酸中的一些或全部在其随机寡核苷酸(如随机8聚物)与其各自结合的核酸样品序列如基因组DNA序列、cDNA序列或RNA序列之间显示出完全的反向互补性。在一些情况下,一些寡核苷酸结合与寡核苷酸的随机寡聚物(如随机8聚物)不完全反向互补的基因组区。在一些情况下,不具有完全反向互补性的碱基配对对随机文库制备过程中的后续步骤无害。
在备选的实施方案中,在任选的变性步骤之前或之后添加聚合酶。将混合物加热至与聚合酶活性如最佳聚合酶活性一致的温度(例如,20℃、21℃、22℃、23℃、24℃、25℃、26℃、27℃、28℃、29℃、30℃、31℃、32℃、33℃、34℃、35℃、36℃、37℃、38℃、39℃、40℃、41℃、42℃,或在一些情况下,比该范围中的数值更高或更低的数值),并温育足以合成第一链文库的一段时间,如5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45分钟或超过45分钟。在一些情况下,在该温育期间的时间点如每10分钟对反应进行搅拌。
延伸从第一链合成寡核苷酸的3’OH进行,导致在每个退火寡核苷酸的退火位点处与模板反向互补的序列被掺入每个退火寡核苷酸的3’端。延伸持续直到掺入生物素标记的ddNTP分子,在该时间点延伸终止。如果dNTP和生物素-ddNTP以99%/1%的比例提供,则发生延伸的第一链寡核苷酸的50%在掺入生物素-ddNTP分子之前显示出超过50个碱基的延伸。在其他参数未同时改变的一些情况下,ddNTP比例降低,代表至少50%延伸产物的长度的N50增加。
在温育期完成时,例如通过在98℃下热灭活五分钟来终止反应。或者,可在另一个温度下,或通过添加螯合剂或dNTP酶来实现灭活。
如上文提到的,在一些情况下,掺入的ddNTP例如被生物素标签标记。在一些情况下,考虑了生物素的替代物,如二硝基苯基。可与ddNTP结合并通过至少一种核酸聚合酶掺入至新生核酸分子中的任何亲和标签都符合本公开内容。类似地,可经由例如ddNTP结合部分递送至核酸分子的ddNTP端的任何亲和标签也都符合本公开内容。在一些情况下,该亲和标签是生物素-ddNTP。
在一些情况下,提供了与本文提供的标记的第一链核酸分子结合的标签结合剂,如在标签生物素的情况下的亲和素或链霉亲和素。在特定情况下,将链霉亲和素与磁珠结合,使得可通过放置在磁场中(如在磁性支架(stand)上)来分离链霉亲和素和任何结合配偶体。
使用标签结合剂例如针对生物素标记的ddNTP核酸末端的链霉亲和素来分离标记的第一链文库。在一些情况下,将珠子/样品混合物在22C下温育并以10分钟的间隔搅拌30分钟。然后将混合物放置在磁性支架上,在珠子沉降后去除上清液。对该管进行搅拌并使其在磁性支架上静置。用200uL TE缓冲液洗涤珠子三次。替代的标签结合剂组合和替代的方案符合本文公开内容。
在一些情况下,在不依赖于标记的情况下,例如通过大小选择如凝胶电泳以及随后对所需大小的核酸的纯化来纯化第一链分子。在一些情况下,分离了大小范围为10-100、10-150、10-200、1-300、10-350、10-400、10-500、10-600、10-700、10-800、10-900或10-1000个碱基的片段。
将如上纯化的第一链文库模板重新引入至反应缓冲液中。例如,在一些情况下,将模板与其纯化标签分离,从链霉亲和素标签上洗脱,并重悬于包含dNTP的核酸合成缓冲液中。在一些情况下,模板保持与其纯化标签附接,洗涤所述模板,并将其重悬于反应缓冲液中。在一些情况下,包括在第一链文库生成后的NaOH洗涤,以去除遗留序列并减少第一链文库产物的自折叠。
如下合成文库第二链分子。添加包含第二链引物群体的第二探针文库。在一些情况下,每个第二链引物包含在随机寡聚物序列如2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30聚物或更大寡聚物(例如8聚物)的5’侧的B-衔接子序列,该B-衔接子序列之后为发生模板引导的延伸的3’OH。在一些情况下,该序列衔接子被配置为包含可变标识符序列。在替代情况下,该序列衔接子是不变的。在一些情况下,使用序列衔接子作为引物结合位点进行测序衔接子如B衔接子的后续添加,例如通过经由扩增的标准引物引导的序列添加。
在一些情况下,随后合成了寡核苷酸群体,使得给定随机寡聚物碱基序列(如随机8聚物)的所有可能的组合都在第二链寡核苷酸群体中呈现。在其他情况下,特别是当选择长随机寡聚物时,但偶尔也在较小寡聚物的情况下,存在给定随机寡聚物碱基序列的部分可能的组合。
在一些情况下,随机寡聚物的碱基以相等的比例呈现核酸碱基的无偏倚的随机分布。在一些情况下,每个碱基在给定位置出现或在随机寡聚物群体中总计出现的可能性相同。然而,在其他情况下,为了增加退火和随后第二链合成的效率,合成了群体,使其包含具有某些碱基或碱基对的偏倚呈现的随机寡聚物(如随机8聚物)的偏倚。例如,据观察,人类基因组具有约40%的GC百分比,而不是从真正随机碱基丰度预期的50%GC组成。参见例如图10。在一些情况下,随机寡聚物分布是偏倚的,使得第二链合成文库中随机寡聚物序列(如8聚物序列)的总体分布反映出偏斜的靶标平均如靶基因组、靶基因座、靶基因家族、靶基因组元件(例如外显子、内含子或启动子序列)的平均的总体分布,或在一些实施方案中,匹配作为整体的人类基因组。
将混合物加热至98℃持续3分钟。该混合物在冰上冷却2分钟,以允许第二链合成寡核苷酸与第一链文库之间的反向互补碱基配对。观察到一些寡核苷酸在其随机8聚物与各自结合的第一链序列之间显示出完全的反向互补性。还观察到一些寡核苷酸结合与寡核苷酸的随机8聚物不完全反向互补的基因组区。不具有完全反向互补性的碱基配对对随机文库制备过程中的后续步骤无害。
将组合物加热至室温并使之持续30分钟。对于输入DNA量更低的样品,可以延长该时间段。
观察到从第一链合成寡核苷酸的3’OH的延伸,导致在每个退火寡核苷酸的退火位点处与模板反向互补的序列被掺入每个退火寡核苷酸的3’端。延伸持续直到到达第一链模板的5’端。观察到远离第一链模板的3’端退火的第二链寡核苷酸经历从其3’端的延伸,但通过由进一步向第一链模板的3’端退火的寡核苷酸引发的延伸反应从第一链被置换。
因此,合成了双链文库分子,其包含两条不同的链:1)第一链,其从5’端开始,具有A衔接子、随机8聚物序列和1-100个核苷酸数量级的靶序列,在生物素标记的ddNTP处终止;和2)第二链,其从5’端开始,具有B衔接子、第二随机8聚物序列、来源于样品的靶序列、与第一链的随机8聚物反向互补的第一随机8聚物序列以及与第一A衔接子反向互补的序列。
在一些情况下,使用磁性链霉亲和素珠子分离生物素标记的双链文库分子。例如,在结合缓冲液中提供磁性链霉亲和素珠子,混合,并使其在磁性支架上静置。然后可将结合缓冲液替换成25uL、50uL、75uL、100uL、125uL、150uL、175uL、200uL、225uL、250uL、275uL、300uL、350uL、400uL、450uL或500uL体积并重复该过程。然后吸出上清液,并可将珠子重悬在5uL、10uL、12uL、14uL、16uL、18uL、20uL、22uL、24uL、26uL、28uL、30uL、31uL、32uL、33uL、34uL、35uL、36uL、37uL、38uL、39uL、40uL、41uL、42uL、43uL、44uL、45uL、46uL、47uL、48uL、49uL 50uL、52uL、54uL、56uL、58uL或60uL的结合缓冲液中。
在一些情况下,然后将生物素标记的双链文库分子添加至重悬的珠子。在一些情况下,将珠子/样品混合物在22℃下温育并以10分钟的间隔搅拌30分钟。然后将混合物放置在磁性支架上,并在珠子沉降后去除上清液。对该管进行搅拌并使其在磁性支架上静置。用200uL TE缓冲液洗涤珠子三次。在一些情况下,这产生链霉亲和素纯化的双链文库分子的群体,该双链文库分子包含两条不同的链:1)第一链,其从5’端开始,具有A衔接子、随机寡聚物(如8聚物)序列和1-100个核苷酸数量级的靶序列、在生物素标记的ddNTP处终止;和2)第二链,其从5’端开始,具有B衔接子、第二随机寡聚物(如8聚物)序列、来源于样品的靶序列、与第一链的随机寡聚物(如8聚物)反向互补的第一随机寡聚物(如8聚物)序列以及与第一A衔接子反向互补的序列。替代的标签结合剂组合和替代的方案符合本文公开内容。
然后,例如,将与双链文库分子群体结合的磁性链霉亲和素珠子重悬在一定量的无核酸酶的水中。该量可以是10uL、12uL、14uL、16uL、18uL、20uL、22uL、24uL、26uL、28uL、30uL、32uL、34uL、36uL、37uL、38uL、39uL、40uL、41uL、42uL、43uL、44uL、45uL、46uL、47uL、48uL、50uL、52uL、54uL、56uL、58uL或60uL无核酸酶的水。向重悬的珠子中添加一定量的衔接子A引物和一定量的衔接子B引物。衔接子A引物的量与衔接子B引物的量可以是相同或不同的。衔接子A引物的量和衔接子B引物的量可独立地为1uL、2uL、3uL、4uL、5uL、6uL、7uL、8uL、9uL或10uL。在一些情况下,衔接子A引物在引物的3’端包含与双链模板的第一衔接子相同的序列,并进一步包含如本文所述的合成测序反应所必需的序列。在其他情况下,衔接子A引物在引物的3’端具有与双链模板的第一衔接子的序列的一个碱基对错配、两个碱基对错配、三个碱基对错配、四个碱基对错配、五个碱基对错配、六个碱基对错配、七个碱基对错配、八个碱基对错配、九个碱基对错配或十个碱基对错配。在一些情况下,衔接子B引物在引物的3’端包含与双链模板的第二链的第二衔接子相同的序列,并进一步包含如本文所述的合成测序反应所必需的序列。在其他情况下,衔接子B引物在引物的3’端具有与双链模板的第二链的第二衔接子的序列的一个碱基对错配、两个碱基对错配、三个碱基对错配、四个碱基对错配、五个碱基对错配、六个碱基对错配、七个碱基对错配、八个碱基对错配、九个碱基对错配或十个碱基对错配。
将2x PCR主混合物以10uL、15uL、20uL、25uL、30uL、35uL、40uL、45uL、50uL、55uL、60uL、65uL、70uL、75uL、80uL、85uL、90uL、95uL或100uL的量添加至珠子和引物的混合物中。在一些情况下,然后使该混合物经历如下的热循环:约98℃持续约2分钟;接着为约98℃持续约20秒、约60℃持续约30秒以及约72℃持续约30秒的约6个循环;在所述约6个循环后,将反应保持在约72℃持续约5分钟,随后在约4℃下储存。本公开内容设想热循环条件的优化,如对于具有较低模板输入的样品增加PCR循环的次数。在一些情况下,在没有PCR的情况下进行扩增。在一个实例中,模板核酸与含有全长测序衔接子的引物一起使用,并且采用随后的大小选择进行第一链合成和第二链合成。这可能需要或不需要使用发夹来避免二聚化。
在一些情况下,观察到由此生成的测序文库具有以下特征。每个双链分子按顺序包含足以进行合成测序的衔接子A序列、第一随机寡聚物序列(如8聚物)、未知长度但可能在1-100个碱基内的靶区域、第二随机寡聚物(如8聚物)序列以及足以进行如本文公开的合成测序的B衔接子序列。
在一些情况下,观察到文库成分具有以下特征。每个分子均包含与文库中其他分子的第一分子标签(如8聚物)相独立的第一分子标签(如8聚物)。每个分子均包含与原始样品的序列对应的靶序列。每个给定分子的靶序列的起点、靶序列的长度和靶序列的终点均与文库中每个其他分子的起点、长度和终点相独立。每个分子均包含与文库中其他分子的第二分子标签(如8聚物)相独立的第二分子标签(如8聚物)。
在一些情况下,观察到文库总体上具有以下特征。基本上所有的样品序列在文库中由多个重叠分子表示。基本上所有的文库分子(罕见事件除外)在通过热循环最终添加A和B衔接子之前都是独特的,就其第一分子标签(如8聚物)序列、靶序列起点、靶序列、靶序列长度、靶序列终点和第二分子标签(如8聚物)序列而言彼此不同。
使此处生成的序列文库经历与其A衔接和B衔接子相容的合成测序,并评价测序结果。独立地,准备第二份原始样品,以便采用标准的基于PCR的文库标记进行测序,其包括对未标记的模板的基于PCR的大量扩增。对文库进行测序并将结果进行比较。
观察到,在传统序列文库测序结果中鉴定了与MEI对应的序列。观察发现MEI单体单元与多个插入相邻边界序列相邻,提示其以多个拷贝存在于样品中。
由于序列读序在每个文库成员中被5’标签、3’标签以及样品序列的独特起点、终点和长度独特地标记,因此序列读序可容易地被分成与独特文库分子对应的组。通过对序列读序群体中呈现的独特文库分子的数目(而不是序列读序的数目)进行计数,可以获得具有经历测序的核酸样品中的给定MEI插入相邻序列的分子的绝对或相对数目的定量量度。
可使用备选的定量方法,并且本文公开的方法不限于单一的定量方法。例如,在一些情况下使用定量PCR来确定一个或多个样品中的MEI插入相邻序列的水平。
通常,在具有用指定波长的光束照射每个样品并检测由激发的荧光团发射的荧光的能力的热循环仪中进行定量PCR。该热循环仪还能够快速加热和冷却样品,由此利用核酸和DNA聚合酶的物理化学性质。PCR过程通常由重复25-40次的一系列温度变化组成。这些循环通常由三个阶段组成:第一阶段,在约95℃下,使双链核酸解链;第二阶段,在约50-60℃的温度下,使引物与DNA模板结合;第三阶段,在68-72℃之间,促进由DNA聚合酶进行的聚合。由于片段大小较小,在这一类型的PCR中通常省略最后一步,这是因为该酶能够在对齐(alignment)阶段与变性阶段之间的变化期间增加其数目。此外,一些热循环仪向每个循环增加仅持续数秒、温度为例如80℃的另一个短的温度阶段,以便降低在使用非特异性染料时由引物二聚体的存在而导致的噪声。每个循环所采用的温度和时间安排依赖于很多参数,如:用于合成DNA的酶、反应中二价离子和dNTP的浓度以及引物的键合温度。
在定量PCR(qPCR)的情况下,DNA结合染料与双链(ds)DNA在PCR中结合,导致该染料的荧光。PCR期间DNA产物的增加导致荧光强度的增加,并在每个循环进行测量,从而允许DNA浓度被定量。定量PCR还可包含荧光报告探针,以仅检测含有该探针序列的DNA,这增加了特异性并使得能够甚至在非特异性DNA扩增的存在下进行定量。
使用qPCR的定量方法包括相对定量和绝对定量。绝对定量通过使用校准曲线与DNA标准比较而给出靶DNA分子的确切数目。相对定量基于内部参考基因来确定靶基因表达的倍数差异。将定量表示为被解释为互补DNA(cDNA,由mRNA的逆转录生成)的mRNA的表达水平变化。
与终点PCR(常规PCR)不同,实时PCR允许通过测量荧光在扩增过程中的任何时间点定量所需产物。常采用的通过定量PCR的DNA定量方法依赖于在对数尺度上将荧光对循环数作图。将基于DNA的荧光的检测阈值设置为稍高于背景。荧光超过该阈值的循环数被称为阈值循环(Ct)或定量循环(Cq)。
商业定量PCR组合物、试剂盒和方法是可获得的,并且其使用符合本文公开的、涉及MEI插入相邻序列定量的一些方法。
本文公开的一些实施方案涉及随时间监测总体体细胞基因组健康。如本文公开的,总体基因组健康涉及由独立MEI事件的丰度反映的体细胞基因组‘健康’状态,在一些情况下与插入位点无关。因此,在一些情况下,方法涉及MEI事件的总数的时间或空间测定。在一些情况下,由于每个插入事件传达了对相关插入位点基因有害的风险,因此MEI事件数目的增加表明‘总基因组健康’的降低。在一些情况下,MEI事件的总数与癌症、衰老、细胞活性丧失或细胞活性降低的风险相关。
例如使用如本文或其他地方公开的定量全基因组测序确定总MEI事件。可替代地或组合地,使用例如本领域已知的Q-PCR或荧光原位杂交法,使用对单个可动因子具有特异性的引物、探针或引物和探针,或使用引物组、探针组或引物组和探针组来测定单独的可动因子,使得多个可动因子(至多且包括已知可动因子的10%、20%、30%、40%、50%、60%、70%、80%、90%、95%,或约100%,或100%)就其在第一时间点或第一组织中的丰度得到定量。
在一些情况下,使用该定量作为基因组健康的基线,尤其当样品取自基因组健康预期较高(如在青年或成年早期)时的组织或第一时间段时。
第二样品在第二时间点,如在第一时间点后不到1年、1年、2年、3年、4年、5年、10年或超过10年的时间点取得。测量总MEI水平,并将其与初始时间点的水平或客观上与患者的基因组健康总体相关的水平进行比较。
如果相比于第一样品或之前的样品,第二样品中的MEI事件总数在丰度上升高10%、20%、30%、40%、50%、70%、100%、2倍、2.5倍、3倍、3.5倍、4倍、5倍或大于5倍,则将该样品中的核酸确定为‘衰老的’或基因组健康不佳。多个治疗选择可用于被确定为体细胞核酸样品的基因组健康不佳的个体。在一些情况下,选择卡路里限制。在一些情况下,推荐NSAID作为治疗方案的一部分。NSAID的部分列表包括以下药物:阿司匹林、塞来昔布(Celebrex)、双氯芬酸(Cambia、Cataflam、Voltaren-XR、Zipsor、Zorvolex)、二氟尼柳、依托度酸、布洛芬(Motrin,Advil)、吲哚美辛(Indocin)、酮洛芬、酮咯酸、萘丁美酮、萘普生(Aleve、Anaprox、Naprelan、Naprosyn)、奥沙普秦(Daypro)、吡罗昔康(Feldene)、双水杨酸酯、舒林酸和妥美汀。其他NSAID也予以考虑并且符合本文公开内容。
可动因子活性与逆转录转座酶(retrotransposase)活性有关,并且在一些情况下与抑制性基因组甲基化的缺陷有关。因此,在一些情况下,治疗方案包括施用逆转录酶抑制剂。在一些情况下,治疗包括施用逆转录转座酶抑制剂。在一些情况下,治疗包括施用逆转录病毒抑制剂。可基于从基因组分析获得的信息施用治疗方法。针对遗传异常的治疗方案是本领域已知的。为了治疗逆转录病毒病症而施用的示例性抑制剂包括但不限于核苷类似物、蛋白酶抑制剂、非核苷反转录酶抑制剂(NNRTI)、核苷酸慢转录酶抑制剂(NtRTI)、混合抑制剂或进入抑制剂以及整合酶抑制剂。示例性的NRTI包括齐多夫定(Retrovir)、拉米夫定(Epivir)、地达诺新(Videx)、扎西他滨(Hivid)、司他夫定(Zerit)和阿巴卡韦(Ziagen)。示例性的蛋白酶抑制剂包括沙奎那韦(Invirase)、利托那韦(Norvir)、茚地那韦(Crixivan)、奈非那韦(Viracept)、安普那韦(Agenerase)、洛匹那韦、阿扎那韦(Reyataz)和替拉那韦(Aptivus)。示例性的非核苷反转录酶抑制剂(NNRTI)包括奈韦拉平(Viramune)、地拉韦啶(Rescriptor)、依非韦伦(Sustiva)和依曲韦林(Intelence)。示例性的NtRTI包括替诺福韦(Viread)。示例性的混合抑制剂或进入抑制剂包括马拉维若(Maraviroc)和恩夫韦地。示例性的整合酶抑制剂包括雷特格韦(Isentress)。可替代地或与以上列出的治疗的任意组合联合地,向个体施用甲基转移酶或促进DNA甲基化的组合物。用于治疗HBV的示例性抑制剂包括但不限于干扰素α(IFN-α)、PEG-IFN-α、恩替卡韦和替诺福韦。
在一些情况下,随时间监测治疗对MEI丰度增加的影响。例如,在治疗方案如本文公开的治疗方案开始后的时间点(如第一时间点后的1个月、2个月、3个月、4个月、5个月、6个月、7个月、8个月、9个月、10个月、11个月、不到1年、1年、2年、3年、4年、5年、10年或超过10年的时间点)取得第三样品。测量总MEI水平,并将其与初始时间点的水平、或客观上与患者基因组健康总体相关的水平、或在治疗方案开始前确定的水平进行比较,或者与之前的MEI丰度测量值进行比较。继续进行导致MEI丰度增加的速率降低(直到并且包括使MEI总量稳定在插入前水平)的治疗方案,在一些情况下伴随着对总MEI水平的持续监测。替换、补充不影响总MEI水平增加的治疗方案,或修改或增加剂量方案,使得MEI水平增加可能受到积极影响。
在一些情况下,该测定与显示出随时间特定增加的特定MEI插入相邻位点的监测,或与MEI相邻边界的监测组合进行,以鉴定涉及已知或疑似癌基因(如本文列出的癌基因)或与癌基因活性有关的基因组重排(如本文列出的基因组重排)或这二者的事件,使得特别被怀疑与目前或未来癌症或肿瘤活性有关的MEI插入事件得到早期鉴定,并且例如使用本文公开的组合物和方法来应对。
为了细胞健康,一个或多个试验在早期进行并监测血液中无细胞DNA的插入事件。相同插入事件的增加代表该事件的克隆扩充,并且可将其定量并与疾病进展相关联。该试验可与针对MEI插入的组织特异性试验、种系变体分析(包括外显子组或全基因组测序)或甲基化或定量RNA分析组合使用,以确定细胞健康或疾病进展。
此外,本公开内容的一些实施方案涉及具有MEI插入边界的组织的可视化,该MEI插入边界例如是与过度增殖(如癌症或肿瘤细胞群体中的过度增殖)有关的MEI插入相关边界。在一些情况下,使用具有与包含MEI插入相邻连续序列的核酸序列特异性退火的核苷酸序列的寡核苷酸探针,使得在退火后,该探针对于例如分析癌性或肿瘤组织的成功切除的医师而言是可检测的。
在一些情况下,使用MEI插入边界序列来开发直接将跨越MEI和插入相邻序列的序列可视化的核酸靶向探针。本文设想了包含跨越MEI和插入相邻边界序列的核酸序列的多种组合物。在一些情况下,这样的组合物的一个共同方面是,它们包含对跨越MEI边缘序列和插入相邻基因组序列二者的序列具有特异性并且长度不足以单独靶向MEI序列或插入相邻序列的核酸组分。
也就是说,本文在许多情况下设想和公开的组合物在不存在插入相邻序列的情况下不与MEI结合,并且在不存在相邻MEI的情况下不与插入相邻序列结合;确切地说,本文公开的组合物包含与包含MEI和相邻基因组序列两者的序列进行特异性结合的核酸组分。因此,在用这样的组合物治疗后,只有与MEI插入相邻序列对应的核酸,例如本文公开的已经在时间或空间测定(例如,如上文公开的)中被鉴定为明显过度表示的核酸,才会被该组合物所可视化,而其他MEI和包含插入相邻序列但不含MEI序列的未插入的等位基因不被该组合物结合。在一些情况下,该组合物的核酸组分包含MEI序列的3、4、5、6、7、8、9、10个或超过10个碱基,和插入相邻序列的3、4、5、6、7、8、9、10个或超过10个碱基,使得该组合物与单独的MEI之间或者该组合物与单独的插入相邻序列之间的结合能不足以保证结合。
在一些实施方案中,与核酸结合的还有荧光团或其他可视化部分。在一些情况下,只有在核酸与底物结合时该部分才被可视化。例如,在一些情况下,探针包含荧光团和猝灭剂,使得在不存在与靶MEI插入相邻位点的结合时,该猝灭部分阻止荧光,而在存在与靶MEI插入相邻位点的结合时,该猝灭剂在空间上从荧光团上去除,使得该荧光团能够在被激发剂激发时发射。
在一些情况下,使用探针来分析癌性组织的完全切除。将组织切除并与探针接触。通过例如在经受具有与荧光团的激发谱匹配的电磁能的波长时在切除的组织中存在荧光来确认癌性组织。通过在经受具有与荧光团的激发谱匹配的电磁能的波长时不存在荧光来鉴定非癌性组织。多种激发装置是本领域已知的,如在手术室环境中容易实用的手持激发装置。
本领域已知,荧光团和其他染料的化学反应性衍生物可用作标记分子的报告分子。示例性的DNA结合报告分子包括但不限于:SeTau-380-NHS、羟基香豆素、氨基香豆素、甲氧基香豆素、瀑布蓝(Cascade Blue)、太平洋蓝(Pacific Blue)、太平洋橙(Pacific Orange)、SeTau-405-NHS、SeTau-405-马来酰亚胺、萤光黄、SeTau-425-NHS、NBD、R-藻红蛋白(PE)、Seta-PerCP-680、PE-Cy5缀合物、PE-Cy7缀合物、Red 613、PerCP、TruRed、FluorX、荧光素、BODIPY-FL、Cy2、Cy3、Seta-555-NHS、Seta-555-叠氮化物、Seta-555-DBCO、Seta-R-PE-670、Cy3B、Seta-580-NHS、Cy3.5、SeTau-647-NHS、Cy5、Seta-APC-780、Cy5.5、Seta-680-NHS、Cy7、TRITC、X-罗丹明、丽丝胺罗丹明B、德克萨斯红(Texas Red)、别藻蓝蛋白(APC)、APC-Cy7缀合物、Seta-780-NHS。
荧光团和其他报告分子可用来与结合DNA的探针结合。本领域已知,这样的探针被设计用于增加定量PCR的特异性。例如,TaqMan探针原理依赖于Taq聚合酶的5′至3′核酸外切酶活性,以在与互补靶序列杂交和基于荧光团的检测过程中切割双标记的探针。所得到的荧光信号允许在PCR的指数阶段定量测量产物的积累。
TaqMan探针由与寡核苷酸探针的5’-端共价附接的荧光团和在3’-端的猝灭剂组成。具有不同化学性质的其他探针是本领域已知的,并且包括但不限于6-羧基荧光素或四氯荧光素,和猝灭剂(例如,四甲基罗丹明)。经由FRET(荧光共振能量转移),猝灭剂分子猝灭荧光团在被热循环仪的光源激发时发射的荧光。只要荧光团和猝灭剂是接近的,猝灭就会抑制荧光信号。
在一些情况下,所述探针包含引导探针跨细胞膜、跨核膜或跨细胞膜和核膜二者易位的部分,使得便于接近组织核DNA。
此外,本文公开的一些实施方案涉及通过将样品的MEI插入相邻序列的谱与第二样品的谱或已知参考谱进行比较来鉴定生物样品如人类样品、其他动物样品、植物样品或生物危害性样品。使待确定其谱的样品经历例如通过全基因组测序或其他合适方法的MEI插入相邻序列确定过程,并确定其单个MEI插入相邻谱。在一些情况下,开发了引物组、探针组或引物组及探针组,使得在其他样品中检测样品的MEI插入相邻序列谱而无需依赖于全基因组测序。
获得了与已开发出MEI插入相邻谱的样品具有相同物种和表型的未知来源的样品。在一些情况下,该样品是作物植物如转基因作物植物的样品,并且存在关于该作物植物种系的来源的某一问题。获得了相同物种并且具有相同转基因抗性的市售转基因植物的谱,并将该谱与未知来源的样品的MEI插入相邻谱进行比较。通过将样品的MEI插入相邻序列与参考进行比较,确定样品和参考是否来自最近共同的原种(stock)。
在备选的实施方案中,使用MEI插入相邻谱来确定例如法医样品或者生物危害性材料如炭疽、鼠疫耶尔森氏菌(Yersinia pestris)、耐甲氧西林金黄色葡萄球菌(MRSA)或其他可武器化的生物材料的来源。
在一些实施方案中,将第二核酸样品鉴定为不同于第一或参考核酸样品包括确定所述第二核酸样品是否缺少该第一核酸样品中存在的MEI边界序列。
在一些实施方案中,将所述第二核酸样品鉴定为不同于所述第一核酸样品包括确定所述第二核酸样品是否包含在所述第一核酸样品中不存在的MEI边界序列。
在备选的实施方案中,通过靶向测序或通过全基因组测序或二者确定边界序列。在一些情况下,使样品接触探针如上文讨论的探针,或探针组,并且在一些情况下通过在与探针分子接触后,单独地、连续地或组合地评价探针激发时样品的荧光来实现样品鉴定。
尽管本文中已经示出并描述了本发明的优选实施方案,但对于本领域技术人员显而易见的是,这些实施方案仅以示例的方式提供。本领域技术人员在不脱离本发明的情况下现将想到多种变化、改变和替代。应当理解,本文中所述的本发明实施方案的各种替代方案可用于实施本发明。目的在于以下述权利要求限定本发明的范围,并由此涵盖这些权利要求范围内的方法和结构及其等同项。
实施例
实施例1.时间MEI监测。对来自个体的核酸样品进行全基因组定量测序。鉴定到以每两个单倍体基因组拷贝出现一次的频率出现的MEI插入位点,表明产生MEI插入的事件很可能发生在个体的先祖种系中而不是个体的体细胞中。
鉴定到以每两个单倍体基因组拷贝出现少于一次的频率出现的MEI插入位点,表明该事件已在个体的一些但并非全部的体细胞中发生。检查MEI插入位点,并确定一些MEI插入位点很可能已经破坏了其功能丧失与细胞周期调节、细胞生长调节或细胞分裂调节缺陷有关的基因。
随时间监测MEI插入位点丰度。两年后,来自实施例1的个体的核酸样品从该个体的血液中取出。分析来自该个体的血液的核酸。
鉴定了MEI插入位点。观察到第一MEI插入位点以与先前全基因组测序工作中观察到的频率相当的频率出现。得出以下结论:MEI插入边界本身与细胞周期调节、细胞生长调节或细胞分裂调节缺陷不相关。
观察到第二MEI插入位点出现的频率是先前全基因组测序工作中观察到的频率的10倍。得出以下结论:MEI插入边界与细胞周期调节、细胞生长调节或细胞分裂调节缺陷相关。对该个体进行进一步的观察,以寻找癌症或其他欠调节的细胞增殖缺陷,从中可以获得DNA以确定肿瘤或其他细胞缺陷是否与MEI插入边界对应。
鉴定了假定癌性组织。对来自假定癌性组织的核酸样品进行全基因组定量测序。发现第二MEI插入位点出现的频率是原始全基因组MEI调查中的频率的100倍。
实施例2.时间MEI监测。来自实施例1的个体的核酸样品从该个体的血液中取出。分析了来自该个体的血液的核酸,并确定了相对和绝对的MEI插入位点频率。
将假定癌性肿瘤组织从该个体切除。按照该程序,来自实施例1的个体的第二核酸样品从该个体的血液中取出。分析了来自该个体的血液的核酸,并确定了相对和绝对的MEI插入位点频率。观察到第二MEI插入位点的频率已回到原始全基因组MEI调查中的频率。
实施例3.时间MEI监测。在切除假定癌性肿瘤组织后两年,从个体的血液中取出来自实施例1和2的个体的核酸样品。分析了来自该个体的血液的核酸,并确定了相对和绝对的MEI插入位点频率。观察到第二MEI插入位点的频率保持在原始全基因组MEI调查中的频率。
在切除假定癌性肿瘤组织后四年,来自实施例1和2的个体的核酸样品从个体的血液中取出。分析了来自该个体的血液的核酸,并确定了相对和绝对的MEI插入位点频率。观察到第二MEI插入位点的频率是原始全基因组MEI调查中的频率的5倍。
对该个体进行进一步的观察,以寻找癌症或其他欠调节的细胞增殖缺陷,从中可以获得DNA以确定肿瘤或其他细胞缺陷是否与MEI插入边界对应。
鉴定了假定癌性组织。对来自假定癌性组织的核酸样品进行全基因组定量测序。发现第二MEI插入位点出现的频率是原始全基因组MEI调查中的频率的100倍。
将假定癌性肿瘤组织从该个体切除。按照该程序,从该个体的血液中取出核酸样品。分析了来自该个体的血液的核酸,并确定了相对和绝对的MEI插入位点频率。观察到第二MEI插入位点的频率已回到原始全基因组MEI调查中的频率。
实施例4.空间MEI监测。对来自患有肿瘤的个体的表型健康组织的第一核酸样品进行全基因组定量测序。鉴定到以每两个单倍体基因组拷贝出现少于一次的频率出现的MEI插入位点,表明该事件已在个体的一些但并非全部的体细胞中发生。检查了MEI插入位点,并确定一些MEI插入位点很可能已经破坏了其功能丧失与细胞周期调节、细胞生长调节或细胞分裂调节缺陷有关的基因。
对来自患有肿瘤的个体的肿瘤组织的第二核酸样品进行全基因组定量测序。鉴定到以每两个单倍体基因组拷贝出现少于一次的频率出现的MEI插入位点,表明该事件已在个体的一些但并非全部的肿瘤细胞中发生。检查了MEI插入位点,并确定一些MEI插入位点很可能已经破坏了其功能丧失与细胞周期调节、细胞生长调节或细胞分裂调节缺陷有关的基因。
检查了插入位点的相对和绝对丰度。观察到一些MEI插入位点以与在来源于个体的表型健康组织的核酸样品中发现的频率相当的相对和绝对频率出现。得出以下结论:这些位点与细胞周期调节、细胞生长调节或细胞分裂调节缺陷不相关。
鉴定了对肿瘤组织核酸样品独特的MEI位点。一些肿瘤特异性MEI插入位点以低丰度出现在肿瘤组织核酸样品中。得出以下结论:这些MEI插入与肿瘤活性不相关。
在整个肿瘤组织核酸样品中发现一些MEI插入位点。得出以下结论:这些MEI插入位点对于细胞周期调节、细胞生长调节或细胞分裂调节缺陷的显现是必需的。然而,它们在非肿瘤核酸样品中的相对丰富的存在表明,其本身不表明与肿瘤活性相关的细胞周期调节、细胞生长调节或细胞分裂调节缺陷的存在。
一些MEI插入位点在整个肿瘤组织核酸样品中以非常高的频率被发现,而在非肿瘤核酸样品中以非常低的频率被发现。得出以下结论:这些MEI插入位点指示与肿瘤活性相关的细胞周期调节、细胞生长调节或细胞分裂调节缺陷的显现。
实施例5.特异性MEI插入边界靶向。将来自实施例2-3的MEI插入边界用作药物干预的源头。开发了包含MEI插入序列和插入相邻基因组序列的核酸分子。将该分子封装到CRISPR核酸靶向复合物中,该复合物特异性地引导内切核酸酶切割与MEI插入序列和插入相邻基因组序列相邻的核酸,而不切割其他MEI插入位点。
实施例6.排除具有与假定癌性组织相关的MEI插入边界的细胞的治疗干预。在切除假定癌性肿瘤组织后两年,来自实施例1和2的个体的核酸样品从个体的血液中取出。分析了来自该个体的血液的核酸,并确定了相对和绝对的MEI插入位点频率。观察到第二MEI插入位点的频率保持在原始全基因组MEI调查中的频率。
在切除假定癌性肿瘤组织后四年,来自实施例1和2的个体的核酸样品从个体的血液中取出。分析了来自该个体的血液的核酸,并确定了相对和绝对的MEI插入位点频率。观察到第二MEI插入位点的频率是原始全基因组MEI调查中的频率的5倍。
对该个体进行进一步的观察,以寻找癌症或其他欠调节的细胞增殖缺陷,从中可以获得DNA以确定肿瘤或其他细胞缺陷是否与MEI插入边界对应。
鉴定了假定癌性组织。对来自假定癌性组织的核酸样品进行全基因组定量测序。发现第二MEI插入位点出现的频率是原始全基因组MEI调查中的频率的100倍。
用包含实施例5的靶向MEI插入边界的药物的治疗方案治疗该个体。观察到假定癌性组织经历特异性细胞死亡。
按照该程序,从该个体的血液中取出核酸样品。分析了来自该个体的血液的核酸,并确定了相对和绝对的MEI插入位点频率。观察到第二MEI插入位点的频率已回到原始全基因组MEI调查中的频率。
实施例7.排除具有与假定癌性组织相关的MEI插入边界的细胞的治疗干预。在切除假定癌性肿瘤组织后两年,来自实施例1和2的个体的核酸样品从个体的血液中取出。分析了来自该个体的血液的核酸,并确定了相对和绝对的MEI插入位点频率。观察到第二MEI插入位点的频率保持在原始全基因组MEI调查中的频率。
在切除假定癌性肿瘤组织后四年,来自实施例1和2的个体的核酸样品从个体的血液中取出。分析了来自该个体的血液的核酸,并确定了相对和绝对的MEI插入位点频率。观察到第二MEI插入位点的频率是原始全基因组MEI调查中的频率的5倍。
对该个体进行进一步的观察,以寻找癌症或其他欠调节的细胞增殖缺陷,从中可以获得DNA以确定肿瘤或其他细胞缺陷是否与MEI插入边界对应。
没有鉴定出假定癌性组织。
用包含实施例5的靶向MEI插入边界的药物的治疗方案治疗该个体。
按照该程序,从该个体的血液中取出核酸样品。分析了来自该个体的血液的核酸,并确定了相对和绝对的MEI插入位点频率。观察到第二MEI插入位点的频率已回到原始全基因组MEI调查中的频率。
实施例8.年龄特异性基因组衰老的监测。对来自个体的核酸样品进行全基因组定量测序。鉴定到以每两个单倍体基因组拷贝出现一次的频率出现的MEI插入位点,表明产生MEI插入的事件很可能发生在个体的先祖种系中而不是个体的体细胞中。
鉴定到以每两个单倍体基因组拷贝出现少于一次的频率出现的MEI插入位点,表明该事件已在个体的一些但并非全部的体细胞中发生。检查了MEI插入位点,并确定一些MEI插入位点很可能已经破坏了其功能丧失与细胞周期调节、细胞生长调节或细胞分裂调节缺陷有关的基因。
随时间监测MEI插入位点丰度。五年后,从个体的血液中取出该个体的核酸样品。分析来自该个体的血液的核酸。
观察到MEI插入位点以与在初始全基因组定量测序后观察到的相当的相对频率和相对丰度出现。
十年后,从个体的血液中取出该个体的核酸样品。分析来自该个体的血液的核酸。
观察到MEI插入位点以与在初始全基因组定量测序后观察到的丰度相当的相对丰度出现。然而,观察到已发生新的MEI插入事件,使插入位点的总数上升至2倍。
推荐包含卡路里限制的抗老化方案。
15年后,从个体的血液中取出该个体的核酸样品。分析来自该个体的血液的核酸。
观察到MEI插入位点以与十年时观察到的丰度相当的相对丰度出现,表明MEI插入位点频率的增加没有持续。
实施例9.年龄特异性基因组衰老的监测。对来自个体的核酸样品进行全基因组定量测序。鉴定到以每两个单倍体基因组拷贝出现一次的频率出现的MEI插入位点,表明产生MEI插入的事件很可能发生在个体的先祖种系中而不是个体的体细胞中。
鉴定到以每两个单倍体基因组拷贝出现少于一次的频率出现的MEI插入位点,表明该事件已在个体的一些但并非全部的体细胞中发生。检查了MEI插入位点,并确定一些MEI插入位点很可能已经破坏了其功能丧失与细胞周期调节、细胞生长调节或细胞分裂调节缺陷有关的基因。
随时间监测MEI插入位点丰度。五年后,从个体的血液中取出该个体的核酸样品。分析来自该个体的血液的核酸。
观察到MEI插入位点以与在初始全基因组定量测序后观察到的相当的相对频率和相对丰度出现。
十年后,从个体的血液中取出该个体的核酸样品。分析来自该个体的血液的核酸。
观察到MEI插入位点以与在初始全基因组定量测序后观察到的丰度相当的相对丰度出现。然而,观察到已发生新的MEI插入事件,使插入位点的总数上升至2倍。
随后是包含逆转录酶抑制剂治疗的抗老化方案。
15年后,从个体的血液中取出该个体的核酸样品。分析来自该个体的血液的核酸。
观察到MEI插入位点以与十年时观察到的丰度相当的相对丰度出现,表明MEI插入位点频率的增加没有持续。
实施例10.年龄特异性基因组衰老的监测。对来自个体的核酸样品进行全基因组定量测序。鉴定到以每两个单倍体基因组拷贝出现一次的频率出现的MEI插入位点,表明产生MEI插入的事件很可能发生在个体的先祖种系中而不是个体的体细胞中。
鉴定到以每两个单倍体基因组拷贝出现少于一次的频率出现的MEI插入位点,表明该事件已在个体的一些但并非全部的体细胞中发生。检查了MEI插入位点,并确定一些MEI插入位点很可能已经破坏了其功能丧失与细胞周期调节、细胞生长调节或细胞分裂调节缺陷有关的基因。
随时间监测MEI插入位点丰度。五年后,从个体的血液中取出该个体的核酸样品。分析来自该个体的血液的核酸。
观察到MEI插入位点以与在初始全基因组定量测序后观察到的相当的相对频率和相对丰度出现。
十年后,从个体的血液中取出该个体的核酸样品。分析来自该个体的血液的核酸。
观察到MEI插入位点以与在初始全基因组定量测序后观察到的丰度相当的相对丰度出现。然而,观察到已发生新的MEI插入事件,使插入位点的总数上升至2倍。
随后是包含逆转录病毒抑制剂治疗的抗老化方案。
15年后,从个体的血液中取出该个体的核酸样品。分析来自该个体的血液的核酸。
观察到MEI插入位点以与十年时观察到的丰度相当的相对丰度出现,表明MEI插入位点频率的增加没有持续。
- 还没有人留言评论。精彩留言会获得点赞!