一种环氧琥珀酸水解酶编码基因及其应用的制作方法
本发明属于生物化工技术领域,具体涉及一种具有顺式环氧琥珀酸水解酶活性的编码基因,同时涉及将该基因在大肠杆菌工程菌株中进行分泌型表达,使重组菌有氧培养后的发酵液能够直接进行酶转化,催化顺式环氧琥珀酸水解为D-酒石酸。
背景技术:
D-酒石酸是L-酒石酸的同型异构体,在自然界中含量稀少,但其用途广泛,尤其在制药行业可作为手性源及拆分剂,用于D-葡萄糖-甘油-1,5-内酸胺、D-半乳糖-甘油-1,5-内酰胺、肿瘤活性天然产物以及C2位邻二胺等多种产品的合成。
目前发现的可以产D-酒石酸的微生物包括恶臭假单胞菌(Pseudomonas putida)、产碱杆菌(Alcaligenes)和博德特氏菌(Bordetella)。2000年Ikuta等分离获得一株产碱杆菌,其底物转化率可达96%以上,已应用于工业化生产,但其转化时间较长,且菌株培养过程难度大,培养成本高。
随着分子克隆技术的兴起,重组的基因工程菌能大大提高目的蛋白的表达量,从而大幅度提高细胞的生物学活性。目前已经报道了产L-酒石酸的诺卡氏菌和红球菌的CESH基因序列,并将其基因工程菌应用于工业化生产,工程菌的活力是野生菌活力的20倍。另外还报道了产D-酒石酸的产碱杆菌和博德特氏菌的CESH酶基因序列,其工程菌的酵活是野生菌活力的50多倍。
E.coli BL21来源于B株,为Lon蛋白水解酶、ompT外膜蛋白水解酶缺陷型,因此,表达的外源蛋白在该体系中有更高的稳定性,常用于表达外源蛋白。然而,在E.coli中表达的外源蛋白常常会发生各种问题,例如,形成不溶的包涵体等,原因大多是采用高效的pET表达载体,由于较快的蛋白质表达速率,导致没有进行正确的折叠。
本发明针对D-酒石酸合成中存在的问题以及大肠杆菌中蛋白表达的问题,公开了一种新的环氧琥珀酸水解酶编码基因及应用与D-酒石酸高效合成的方法。
技术实现要素:
本发明的目在于针对现有D-酒石酸合成以及大肠杆菌中蛋白表达中存在的问题,提供一种可用于D-酒石酸高效合成的新型环氧琥珀酸水解酶编码基因。
为实现上述技术目的,本发明采用如下技术方案:
一种环氧琥珀酸水解酶编码基因cesH,其编码的氨基酸序列如SEQ ID NO:2所示。其核苷酸序列如SEQ ID NO:1所示。
优选的,在环氧琥珀酸水解酶编码基因起始密码子和第二个密码子之间插入一段信号肽,该信号肽的核苷酸序列如SEQ ID NO:3所示,形成新的环氧琥珀酸水解酶编码基因序列。
本发明的第二目的是提供含有上述编码基因的表达载体及基因工程菌。
所述表达载体选用质粒;质粒选用PET系列表达质粒;优选含有pelB信号肽的pET-(X)b系列,其中X代表数字。
选用E.coli BL21(DE3)宿主菌构建所述基因工程菌,所述的基因工程菌能够分泌型表达环氧琥珀酸水解酶,具有低包涵体生成。
本发明的第三目的在于提供所述环氧琥珀酸水解酶在D-酒石酸合成中的应用。
本发明的第四目的在于提供一株催化顺式环氧琥珀酸制备D-酒石酸的基因工程菌。所述基因工程菌能够分泌表达本发明的环氧琥珀酸水解酶,基因工程菌有氧培养后的发酵液能够直接进行酶转化,催化顺式环氧琥珀酸水解为D-酒石酸。
为实现上述技术目的,本发明采用如下技术方案:
一株催化顺式环氧琥珀酸制备D-酒石酸的基因工程菌,其分类命名为大肠埃希氏菌Escherichia coli CM-TA-122,已于2016年9月5日保藏于中国典型培养物保藏中心CCTCC,保藏编号为:CCTCC NO:M 2016456。
上述基因工程菌CM-TA-122,在发酵制备D-酒石酸中的应用在本发明的保护范围之内。
本发明提供了一种基因工程菌CM-TA-122在发酵制备D-酒石酸中的具体应用方法,具体如下:
其中,发酵液的培养过程如下:
(S1)将重组菌按体积分数为1~2%从冻存管转接到LB培养基中,有氧培养10~12h;
(S2)将S1中的培养液按体积分数为1~2%转接到种子发酵罐的LB培养基中,有氧培养至OD600在2.5~4;
(S3)将S2中的培养液按体积分数为5~10%接种发酵培养基,所述发酵培养基的配方为:0.02%MgSO4·7H2O,0.1%KH2PO4,0.5%NaCl,1.0%环氧琥珀酸钠,2.0%玉米浆干粉,1.0%乳糖,有氧培养时间12~16h。
步骤(S1)和(S2)中,培养温度控制在32~35℃,步骤(S3)的温度控制在25~28℃。
步骤(S1)和(S2)不控制溶氧,步骤(S3)中溶解氧需控制在5~40%,且培养过程pH用氨水调节为7.0~7.5。
另一方面,利用发酵液进行酶转化的过程如下:
通过(S3)步骤有氧培养获得含菌发酵液,将含菌发酵液按1:10~15的体积比添加到1~1.2mol/L的环氧琥珀酸钠溶液中,控制pH在6.6~6.8,温度控制在40~45℃,转化时间控制在1~2h。
pH的控制可采用环氧琥珀酸钠溶液在配置时采用pH为6.6~6.8的缓冲液,也可利用在线pH检测及反馈系统,通过自动补加NaOH或KOH控制pH在6.6~6.8。
本发明利用挖掘的新顺式环氧琥珀酸水解酶编码基因,建立了适合工业化操作的酶生产及酶催化工艺,该路线高效、低成本。
附图说明
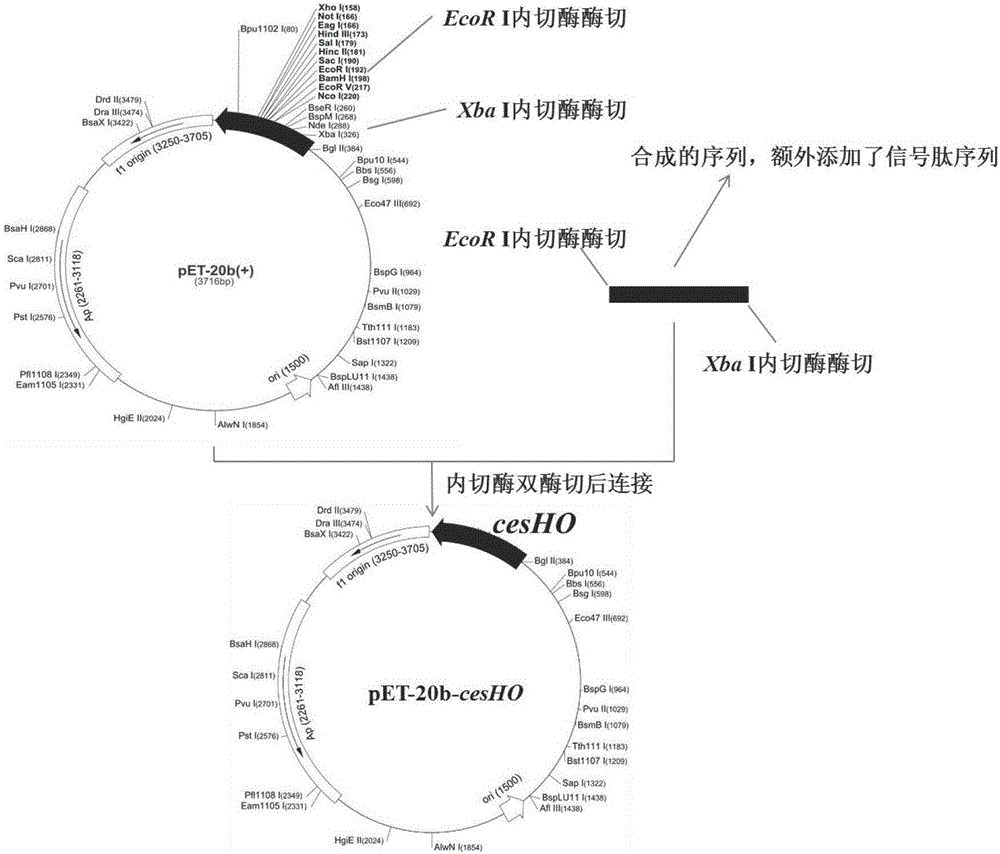
图1是重组质粒pET-20b-cesH构建过程示意图。
图2是重组质粒pET-20b-cesHO构建过程示意图。
图3是SDS-PAGE电泳图;其中Lane M指蛋白marker;Lane 1为CM-TA-122可溶性蛋白电泳图;Lane 2大肠杆菌BL21(DE3)-pET-20b-cesH可溶性蛋白电泳图。
图4是顺式环氧琥珀酸钠对酶催化合成D-酒石酸效率的影响曲线图。
本发明所述的生物材料,其分类命名为大肠埃希氏菌(Escherichia coli)CM-TA-122,已于2016年9月5日保藏于中国典型培养物保藏中心CCTCC,保藏编号为:CCTCC NO:M 2016456,保藏地址为:中国.武汉.武汉大学。
具体实施方式
根据下述实施例,可以更好地理解本发明。然而,本领域的技术人员容易理解,实施例所描述的内容仅用于说明本发明,而不应当也不会限制权利要求书中所详细描述的本发明。
实施例1
本实施例说明构建表达顺式环氧琥珀酸水解酶的表达质粒,并将其导入BL21(DE3)宿主中,构建获能重组大肠杆菌BL21(DE3)-pET-20b-cesH的过程。
1、构建表达顺式环氧琥珀酸水解酶的表达质粒,其过程包括:
(1)人工设计并合成一段基因序列,即顺式环氧琥珀酸水解酶编码基因(SEQ ID NO:1),同时最终合成的基因在其两端带有Xba I和EcoR I酶切位点序列。
(2)将质粒pET-20b及上述人工合成的基因用Xba I和EcoR I进行双酶切,并连接获得重组质粒pET-20b-cesH。酶切和连接过程参考TAKARA公司限制性内切酶和连接酶的使用说明书。重组质粒的构建过程及载体图谱如图1所示,其测序结果与设计的基因序列100%一致。
2、将重组质粒pET-20b-cesH导入BL21(DE3),获得的阳性转化子即为本发明的新构建菌株BL21(DE3)-pET-20b-cesH。
实施例2
本实施例说明构建超量表达顺式环氧琥珀酸水解酶的表达质粒,并将其导入BL21(DE3)宿主中,构建获得重组大肠杆菌BL21(DE3)-pET-20b-cesHO的过程,即CM-TA-122的构建过程。
1、构建超量表达顺式环氧琥珀酸水解酶的表达质粒,其过程包括:
(1)人工设计并合成一段基因序列,具体序列为在顺式环氧琥珀酸水解酶编码基因(SEQ ID NO:1)的起始密码子和第二个密码子之间插入了一段信号肽序列SEQ ID NO:3,同时最终合成的基因在其两端带有Xba I和EcoR I酶切位点序列。
(2)将质粒pET-20b及上述人工合成的基因用Xba I和EcoR I进行双酶切,并连接获得重组质粒pET-20b-cesHO。酶切和连接过程参考TAKARA公司限制性内切酶和连接酶的使用说明书。重组质粒的构建过程及载体图谱如图2所示,其测序结果与设计的基因序列100%一致。Ps:
2、将重组质粒pET-20b-cesHO导入BL21(DE3),获得的阳性转化子即为本发明的新构建菌株CM-TA-122。
实施例3
本实施例说明新构建的重组大肠杆菌BL21(DE3)-pET-20b-cesH与CM-TA-122发酵产顺式琥珀酸水解酶的能力,及其酶的分布情况,考察信号肽添加对于产酶的影响。
重组菌按体积分数为1~2%从-80℃保存的甘油管(含15%甘油,菌体密度在4~5g/gDCW)转接到LB培养基中,有氧培养10~12h,收集的细胞破碎后其SDS-PAGE的鉴定图,如图3所示。结果表明两株菌的酶的大小均在33kDa左右,但添加信号肽的菌株的表达量明显更高。
为进一步分析两株菌的酶活力和分布情况,进行了5L罐发酵,发酵过程如下:
(a)将重组菌按体积分数为1~2%从冻存管转接到LB培养基中,有氧培养10~12h,32~35℃,不控制pH和溶氧;(b)将按照步骤(1)处理后的培养液按体积分数为1~2%转接到种子发酵罐的LB培养基中,32~35℃,不控制pH和溶氧,培养至OD600在2.5~4;(c)将按步骤(2)处理后的培养液按体积分数为5~10%接种至发酵培养基,所述发酵培养基的配方为:0.02%MgSO4·7H2O,0.1%KH2PO4,0.5%NaCl,1.0%环氧琥珀酸钠,2.0%玉米浆干粉,1.0%乳糖,25~28℃,溶解氧需控制在5~40%,且培养过程pH用氨水调节为7.0~7.5,有氧培养时间12~16h。
结果如表1所示,该结果表明为使用信号肽时顺式琥珀酸水解酶表达量略低,总酶活为添加信号肽重组菌株的85.1%,且大部分在胞内,而添加了信号肽的重组菌不仅总酶活提高了17.5%,且有80%以上的酶分泌到胞外,酶活的总量达到每克细胞干重1600U。
表1重组菌产酶情况
实施例4
本实施例说明新构建的重组大肠杆菌CM-TA-122培养后的发酵液直接转化顺式环氧琥珀酸盐合成D-酒石酸的能力。
按实施例3中的发酵条件培养细胞,将步骤(c)有氧培养获得含菌发酵液,按不同的体积比添加到1mol/L的环氧琥珀酸钠溶液中,自动控制pH在6.6~6.8,温度控制在40~45℃,转化时间控制在1h。
酶催化结果见表2:
表2不同比例重组大肠杆菌CM-TA-122发酵液对酶转化效率影响
根据表2可以看出,在发酵液与底物(顺式环氧琥珀酸钠)添加比例为1:5~15时,顺式环氧琥珀酸钠无残留,转化率为100%。
实施例5
本实施例说明新构建的重组大肠杆菌CM-TA-122培养后的发酵液直接转化顺式环氧琥珀酸盐合成D-酒石酸的能力。
按实施例3中的发酵条件培养细胞,将步骤(c)有氧培养获得含菌发酵液,按1:10的体积比添加到0.4~1.2mol/L的环氧琥珀酸钠溶液中,自动控制pH在6.6~6.8,温度控制在40~45℃,转化时间控制在1h。
酶催化结果见图4,从图4可以看出,酶转化效率随着环氧琥珀酸钠溶液浓度的增加而提高,在环氧琥珀酸钠溶液浓度高于0.7mol/L时,酶转化效率可超过90%。
序列表
<110> 南京工业大学
<120> 一种环氧琥珀酸水解酶编码基因及其应用
<130> xb16112203
<160> 3
<170> PatentIn version 3.5
<210> 1
<211> 876
<212> DNA
<213> Artificial Sequence
<220>
<223> 1
<220>
<221> CDS
<222> (1)..(876)
<400> 1
atg acc cga acc ccc ttg ata ctt gaa gct cgt atc aat gaa tac atg 48
Met Thr Arg Thr Pro Leu Ile Leu Glu Ala Arg Ile Asn Glu Tyr Met
1 5 10 15
ccg cgt cgc ggt aat ccc cat gtg ccg tgg acg cca aag gag atc ggt 96
Pro Arg Arg Gly Asn Pro His Val Pro Trp Thr Pro Lys Glu Ile Gly
20 25 30
gag gct gcg gca cag gca cgg gaa gcg ggt gca tcg att gtc cat ttc 144
Glu Ala Ala Ala Gln Ala Arg Glu Ala Gly Ala Ser Ile Val His Phe
35 40 45
cat gcc cgt caa gct gac ggt tct ccc agc cat gac tat gaa act tat 192
His Ala Arg Gln Ala Asp Gly Ser Pro Ser His Asp Tyr Glu Thr Tyr
50 55 60
gca gaa tcg atc cgt gag att cgc gca gac gtt cta gta cat ccg aca 240
Ala Glu Ser Ile Arg Glu Ile Arg Ala Asp Val Leu Val His Pro Thr
65 70 75 80
ctg ggc cag atc agt ctt gga ggg agg gaa tca cga ctg gca cac att 288
Leu Gly Gln Ile Ser Leu Gly Gly Arg Glu Ser Arg Leu Ala His Ile
85 90 95
gag cga ctg tgc ctt gac cca gca cta aaa ccg gac ttt gca ccg gta 336
Glu Arg Leu Cys Leu Asp Pro Ala Leu Lys Pro Asp Phe Ala Pro Val
100 105 110
gac ctg ggt agc acg aac atc gat cgt tat gac gat gtg gag aag cgc 384
Asp Leu Gly Ser Thr Asn Ile Asp Arg Tyr Asp Asp Val Glu Lys Arg
115 120 125
tac gag aca ggc gac cgc gtg tac ttg aac aac atc gac acg ctg cag 432
Tyr Glu Thr Gly Asp Arg Val Tyr Leu Asn Asn Ile Asp Thr Leu Gln
130 135 140
cac ttc agc aag aga cta cga gaa ctc ggg gtt aag cct gcg ttg gaa 480
His Phe Ser Lys Arg Leu Arg Glu Leu Gly Val Lys Pro Ala Leu Glu
145 150 155 160
gct tgg acg gtc ccc ttc acc cga aca ctt gac gcc ttt atg gat atg 528
Ala Trp Thr Val Pro Phe Thr Arg Thr Leu Asp Ala Phe Met Asp Met
165 170 175
ggt ttg gtc gac gac ccg gca tat ctg ctg ttt gag ctc acc gac tgc 576
Gly Leu Val Asp Asp Pro Ala Tyr Leu Leu Phe Glu Leu Thr Asp Cys
180 185 190
ggc atc cgc ggt gga cat ccg gga acg ata cgg ggg ctg cgt gca cat 624
Gly Ile Arg Gly Gly His Pro Gly Thr Ile Arg Gly Leu Arg Ala His
195 200 205
acc gac ttc ctg cca ccc gga cga cag atc caa tgg acc gtc tgc aac 672
Thr Asp Phe Leu Pro Pro Gly Arg Gln Ile Gln Trp Thr Val Cys Asn
210 215 220
aag atc ggc aac cta ttt ggc cct gct gct gcg gcc att gag gaa gga 720
Lys Ile Gly Asn Leu Phe Gly Pro Ala Ala Ala Ala Ile Glu Glu Gly
225 230 235 240
gga cac gtc ccg att ggc cta ggc gat tac ctc tat ccg gaa ttg ggt 768
Gly His Val Pro Ile Gly Leu Gly Asp Tyr Leu Tyr Pro Glu Leu Gly
245 250 255
aca ccc acc aat ggg gaa gtc gtt cag acc gta gcg aat atg gca cgt 816
Thr Pro Thr Asn Gly Glu Val Val Gln Thr Val Ala Asn Met Ala Arg
260 265 270
gcg atg ggt cgt gag att gca acg cca gca gag aca aag gaa ctg ggt 864
Ala Met Gly Arg Glu Ile Ala Thr Pro Ala Glu Thr Lys Glu Leu Gly
275 280 285
att agc aac tag 876
Ile Ser Asn
290
<210> 2
<211> 291
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 2
Met Thr Arg Thr Pro Leu Ile Leu Glu Ala Arg Ile Asn Glu Tyr Met
1 5 10 15
Pro Arg Arg Gly Asn Pro His Val Pro Trp Thr Pro Lys Glu Ile Gly
20 25 30
Glu Ala Ala Ala Gln Ala Arg Glu Ala Gly Ala Ser Ile Val His Phe
35 40 45
His Ala Arg Gln Ala Asp Gly Ser Pro Ser His Asp Tyr Glu Thr Tyr
50 55 60
Ala Glu Ser Ile Arg Glu Ile Arg Ala Asp Val Leu Val His Pro Thr
65 70 75 80
Leu Gly Gln Ile Ser Leu Gly Gly Arg Glu Ser Arg Leu Ala His Ile
85 90 95
Glu Arg Leu Cys Leu Asp Pro Ala Leu Lys Pro Asp Phe Ala Pro Val
100 105 110
Asp Leu Gly Ser Thr Asn Ile Asp Arg Tyr Asp Asp Val Glu Lys Arg
115 120 125
Tyr Glu Thr Gly Asp Arg Val Tyr Leu Asn Asn Ile Asp Thr Leu Gln
130 135 140
His Phe Ser Lys Arg Leu Arg Glu Leu Gly Val Lys Pro Ala Leu Glu
145 150 155 160
Ala Trp Thr Val Pro Phe Thr Arg Thr Leu Asp Ala Phe Met Asp Met
165 170 175
Gly Leu Val Asp Asp Pro Ala Tyr Leu Leu Phe Glu Leu Thr Asp Cys
180 185 190
Gly Ile Arg Gly Gly His Pro Gly Thr Ile Arg Gly Leu Arg Ala His
195 200 205
Thr Asp Phe Leu Pro Pro Gly Arg Gln Ile Gln Trp Thr Val Cys Asn
210 215 220
Lys Ile Gly Asn Leu Phe Gly Pro Ala Ala Ala Ala Ile Glu Glu Gly
225 230 235 240
Gly His Val Pro Ile Gly Leu Gly Asp Tyr Leu Tyr Pro Glu Leu Gly
245 250 255
Thr Pro Thr Asn Gly Glu Val Val Gln Thr Val Ala Asn Met Ala Arg
260 265 270
Ala Met Gly Arg Glu Ile Ala Thr Pro Ala Glu Thr Lys Glu Leu Gly
275 280 285
Ile Ser Asn
290
<210> 3
<211> 69
<212> DNA
<213> Artificial Sequence
<220>
<223> 1
<400> 3
atgttgaatc cgaaggttgc ctacatggtc tggatgacgt gcctgggttt aacgttgccc 60
agccaggca 69
- 还没有人留言评论。精彩留言会获得点赞!