口腔鳞状细胞癌生物标记物及其应用的制作方法
本发明属于生物医药领域,涉及一种在口腔鳞状细胞癌生物标记物及其应用,具体的涉及在口腔鳞状细胞癌显著上调的XIRP2的应用。
背景技术:
口腔恶性肿瘤是一类严重威胁人类健康的疾病。在口腔恶性肿瘤中,口腔鳞状细胞癌(oral squamous cell carcinoma,OSCC)比例约占90%。与其他肿瘤的发生一样,OSCC的发生发展是一个多基因,多分子呈网络结构,多步骤、多阶段共同作用的长期复杂过程。各肿瘤基因、分子之间相互协同作用促使肿瘤细胞形成和发展。目前肿瘤的发生机制及治疗均未取得重大突破,如何早期检测肿瘤病损及根治是现代肿瘤工作者难题及机遇。
肿瘤是一种分子水平的疾病,从基因蛋白水平进行肿瘤的研究是最终途径。近年在整体水平上以高通量分子扫描手段为基础的基因组学、蛋白质组学以及计算机辅助设计等技术的整合及相互关联的“技术链”的应用等为肿瘤的研究提供了新的契机,并且已经在乳腺癌、肺癌、胃癌、结肠癌、卵巢癌、黑色素瘤等的研究中取得了丰硕的成果。其中基因表达谱芯片是最典型的代表,通过基因表达谱芯片可以检测和筛选肿瘤组织与正常组织的差异基因以及癌基因、抑癌基因的变化及变化关系,从中寻找病因,为早期诊断及根治提供线索和依据。
技术实现要素:
为了弥补现有技术的不足,本发明的目的在于提供一种口腔鳞状细胞癌的生物标记物,灵敏和特异性的实现口腔鳞状细胞癌的诊断和治疗。
为了实现上述目的,本发明采用如下技术方案:
本发明提供了XIRP2基因在制备诊断口腔鳞状细胞癌的产品中的应用。
进一步,所述产品包括通过芯片、印迹法、RT-PCR、实时定量PCR、FISH法、CGH法或阵列CGH法、亚硫酸氢盐测序法、COBRA法检测XIRP2基因的变化以诊断口腔鳞状细胞癌的产品。
其中,印迹法包括Northern、Southern、Western印迹法。Southern印迹法是将从样本得到的基因组DNA分离并固定,通过检测DNA与XIRP2基因的杂交来测定样本中的XIRP2基因;Northern印迹法是一种将由样本中获得的mRNA分离、固定,通过检测mRNA与XIRP2基因的杂交来检测该基因的mRNA;Western印迹法是一种将样本中的蛋白分离、固定,通过检测抗体与XIRP2蛋白的免疫反应来分析基因的表达程度。
其中,所述用RT-PCR诊断口腔鳞状细胞癌的产品至少包括一对特异扩增XIRP2基因的引物;所述用实时定量PCR诊断口腔鳞状细胞癌的产品至少包括一对特异扩增XIRP2基因的引物。
进一步,所述用实时定量PCR诊断管鳞癌的产品至少包括的一对特异扩增XIRP2基因的引物序列如SEQ ID NO.3和SEQ ID NO.4所示。
本发明提供了一种诊断口腔鳞状细胞癌的产品,所述产品能够通过检测样本中XIRP2基因的表达水平来诊断口腔鳞状细胞癌。
进一步,所述产品包括芯片、试剂盒或制剂。其中,所述芯片包括基因芯片、蛋白质芯片;所述基因芯片包括固相载体以及固定在固相载体的寡核苷酸探针,所述寡核苷酸探针包括用于检测XIRP2基因转录水平的针对XIRP2基因的寡核苷酸探针;所述蛋白质芯片包括固相载体以及固定在固相载体的XIRP2蛋白的特异性抗体;所述试剂盒包括基因检测试剂盒和蛋白免疫检测试剂盒;所述基因检测试剂盒包括用于检测XIRP2基因转录水平的试剂;所述蛋白免疫检测试剂盒包括XIRP2蛋白的特异性抗体。
本发明所述的基因芯片或基因检测试剂盒可用于检测包括XIRP2基因在内的多个基因(例如,与口腔鳞状细胞癌相关的多个基因)的表达水平。所述蛋白芯片或蛋白免疫检测试剂盒可用于检测包括XIRP2蛋白在内的多个蛋白质(例如与口腔鳞状细胞癌相关的多个蛋白质)的表达水平。将口腔鳞状细胞癌的多个标志物同时进行检测,可大大提高口腔鳞状细胞癌诊断的准确率。
本发明提供了XIRP2基因在制备治疗口腔鳞状细胞癌的药物组合物中的应用。
进一步,所述药物组合物包括XIRP2基因和/或其表达产物的抑制剂。所述抑制剂包括抑制XIRP2基因表达的物质、抑制XIRP2基因表达产物稳定性的物质、和/或抑制XIRP2基因表达产物活性的物质。
进一步,所述抑制剂是针对XIRP2基因的siRNA、针对XIRP2蛋白的抗体;优选的,所述抑制剂为siRNA。
在本发明的具体实施方式中,所述针对XIRP2基因的siRNA的序列如SEQ ID NO.9和SEQ ID NO.10所示。
本发明还提供了一种治疗口腔鳞状细胞癌的药物组合物,所述药物包含XIRP2基因和/或其表达产物抑制剂。所述抑制剂包括抑制XIRP2基因表达的物质、抑制XIRP2基因表达产物稳定性的物质、和/或抑制XIRP2基因表达产物活性的物质。
本发明还提供了一种抑制细胞增殖的方法,所述方法是将针对XIRP2基因的siRNA、shRNA、反义寡核苷酸或功能缺失型基因在体外导入到肿瘤细胞中。
本发明提供了一种组合药物,所述组合药物包括上述的药物组合物、和含抗肿瘤剂的药物组合物。
进一步,抗肿瘤剂的药物组合物包括但不限于免疫治疗剂如西妥昔单克隆抗体,化学治疗剂或化学放射性治疗剂如卡波铂或者是一种相关类别的铂类药物、紫杉烷或者是一种类别的紫衫烷,或者是两者。
本发明的优点和有益效果:
本发明首次发现了与口腔鳞状细胞癌发生发展相关的生物标记物—XIRP2,通过检测受试者XIRP2的变化,来实现口腔鳞状细胞癌的早期诊断。
本发明提供了治疗口腔鳞状细胞癌的分子靶标,通过靶向于分子标志物来治疗疾病具有敏感性和特异性。
本发明对口腔鳞状细胞癌的机制研究提供了一定的理论基础。
附图说明
图1显示利用QPCR检测XIRP2基因在口腔鳞状细胞癌组织中的表达情况;
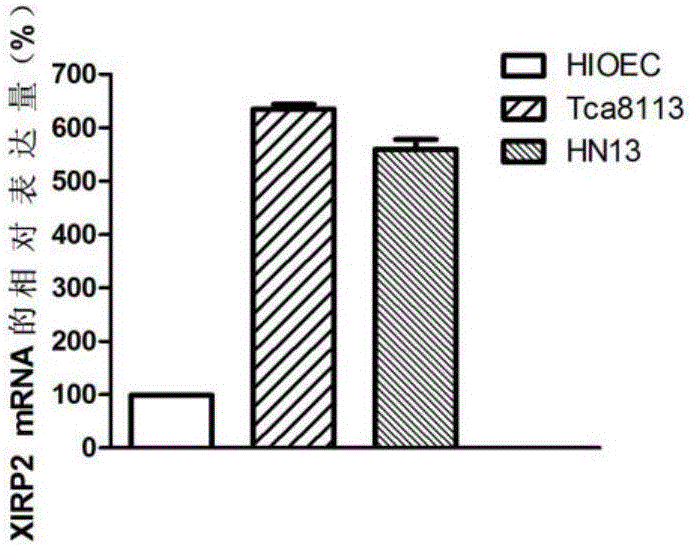
图2显示利用QPCR检测XIRP2基因在口腔鳞状细胞癌细胞中的表达情况;
图3显示利用QPCR检测siRNA对XIRP2基因表达的影响;
图4显示MTT法检测XIRP2对口腔鳞状细胞癌细胞增殖活性的影响;
图5显示利用transwell小室检测XIRP2基因对口腔鳞状细胞癌细胞侵袭的影响。
具体的实施方式
本发明首次发现了XIRP2与口腔鳞状细胞癌的发生发展相关,并验证了XIRP2在口腔鳞状细胞癌中高表达。XIRP2可作为口腔鳞状细胞癌的独立预测因子,也可以和其他的基因标志物联合应用。
本发明中术语“生物标记物”是其在组织或细胞中的表达水平与正常或健康细胞或组织的表达水平相比发生改变的任何基因或蛋白。
本领域技术人员将认识到,本发明的实用性并不局限于对本发明的标志物基因的任何特定变体的基因表达进行定量。作为非限制性的实例,标志物基因可具有SEQ ID NO.1或SEQ ID NO.2指定的编码序列或氨基酸序列。在一些实施方案中,其具有与所列的序列至少85%相同或相似的cDNA序列或氨基酸序列,诸如上述所列的序列至少90%、91%、92%、93%、94%、95%、96%、97%、98%或至少99%相同或相似的cDNA序列或氨基酸序列。
本文所述的生物标记物包括基因和蛋白。此类生物标记物包括含有编码生物标记物的核酸序列或此序列的互补序列的完整或部分序列的DNA。生物标记物核酸还包括含有所关注的任何核酸序列的完整或部分序列的RNA。生物标记物蛋白是由本发明的DNA生物标记物编码的或对应于本发明的DNA生物标记物的蛋白。生物标记物蛋白包含任何生物标记物蛋白或多肽的完整或部分氨基酸序列。生物标记物基因和蛋白的片段和变体也包括在本发明的范围内。所谓“片段”是指多核苷酸的一部分或氨基酸序列并因而编码的蛋白的一部分。为生物标记物核苷酸序列的片段的多核苷酸通常包含至少10、15、20、50、75、100、150、200、250、300、350、400、450、500、550、600、650、700、800、900、1,000、1,100、1,200、1,300或1,400个连续的核苷酸,或最多存在于本文所公开的全长生物标记物多核苷酸中的核苷酸个数。生物标记物多核苷酸的片段将通常编码至少15、25、30、50、100、150、200或250个连续氨基酸,或存在于本发明的全长生物标记物蛋白中的氨基酸的总数。“变体”旨在表示基本上相似的序列。一般来讲,本发明的特定生物标记物的变体将具有通过序列比对程序测定的与所述生物标记物至少约40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更高的序列同一性。
本发明可以利用本领域内已知的任何方法测定基因表达。本领域技术人员应当理解,测定基因表达的手段不是本发明的重要方面。可以在转录或翻译(即蛋白)水平上检测生物标记物的表达水平。
在一些实施方案中,在转录水平上检测生物标记物的表达水平。利用核酸杂交技术进行具体DNA和RNA测量的多种方法是本领域技术人员已知的。一些方法涉及电泳分离(例如,用于检测DNA的Southern印迹和用于检测RNA的Northern印迹),但是也可以在不利用电泳分离的情况下进行DNA和RNA的测量(例如,通过斑点印迹)。基因组DNA(例如,来自人)的Southern印迹可用于筛选限制性片段长度多态性(RFLP),以检测影响本发明多肽的遗传病症的存在。可以检测所有形式的RNA,包括但不限于信使RNA(mRNA)、微RNA(miRNA)、核糖体RNA(rRNA)和转运RNA(tRNA)。
本发明中的药物组合物还包括药学上可接受的载体,这类载体包括(但并不限于):稀释剂、赋形剂如乳糖、氯化钠、葡萄糖、尿素、淀粉、水等、填充剂如淀粉、蔗糖等;粘合剂如单糖浆、葡萄糖溶液、淀粉溶液、纤维素衍生物、藻酸盐、明胶和聚乙烯吡咯烷酮;湿润剂如甘油;崩解剂如干淀粉、海藻酸钠、海带多糖粉末、琼脂粉末、碳酸钙和碳酸氢钠;吸收促进剂季铵化合物、十二烷基硫酸钠等;表面活性剂如聚氧化乙烯山梨聚糖脂肪酸酯、十二烷基硫酸钠、硬脂酸单甘油酯、十六烷醇等;致湿剂如甘油、淀粉等;吸附载体如淀粉、乳糖、斑脱土、硅胶、高岭土和皂粘土等;润滑剂如滑石粉、硬脂酸钙和镁、聚乙二醇、硼酸粉末等。
本发明的药物组合物可以使用不同的添加剂进行制备,例如缓冲剂、稳定剂、抑菌剂、等渗剂、螯合剂、pH控制剂及表面活性剂。
缓冲剂可以包括硼酸、磷酸、乙酸、柠檬酸、谷氨酸及相应的盐(它们的碱金属或碱性稀土金属盐,例如钠盐、钾盐、钙盐及镁盐)。等渗剂包括氯化钾、氯化钠、糖及甘油。螯合剂包括乙二胺四乙酸钠及柠檬酸。抑菌剂包括但不限于有效浓度(例如<1%w/v)的苄醇、苯酚、间甲酚、氯丁醇、对羟基苯甲酸甲酯和/或对羟基苯甲酸丙酯。稳定剂包括人类血清蛋白、L-氨基酸、糖及纤维素衍生物。L-氨基酸还可以包括甘氨酸、半胱氨酸及谷氨酸中的任意一个。糖类包括单糖,例如葡萄糖、甘露糖、半乳糖、果糖等;糖醇,例如甘露醇、纤维醇、木糖醇等;二糖,例如蔗糖、麦芽糖、乳糖等;多聚糖,例如葡聚糖、羟丙基淀粉、硫化软骨素、透明质酸等及它们的衍生物。纤维素衍生物包括甲基纤维素、乙基纤维素、羟乙基纤维素、羟丙基纤维素、羟丙甲基纤维素及羟甲基纤维素钠。表面活性剂包括离子或非离子表面活性剂,例如聚氧化乙烯烷基酯、山梨聚糖单酰基酯、脂肪酸甘油酯。
本发明的药物还可包括药学上可接受的涂层材料包括(但不限于),快速分解涂层材料、染色剂、肠溶性聚合物、增塑剂、水溶性聚合物、水不溶性聚合物、染料、色素、其他崩散剂。常见快速分解涂层材料包括OPADRY;肠溶性聚合物包括甲基内烯酸聚合物、磷羟丙甲基纤维素苯二甲酸酯、羟丙甲基纤维素乙酸酯、羟丙甲基纤维素琥珀酸酯、羟甲乙基纤维素、乙酰磷苯二甲酸纤维素;增塑剂包括聚乙二醇(PEG)、丙二醇等。
本发明所述药物组合物可口服给药、非胃肠道给药、通过吸入喷雾给药、局部给药、直肠给药、鼻给药、颊给药、阴道给药或通过植入的贮药装置给药。优选口服给药或注射给药。本发明药物组合物可含有任何常用的无毒可药用载体、辅料或赋形剂。在某些情况下,药用酸、碱或缓冲剂可用来调节制剂的pH以提高所配制的化合物或其给药剂型的稳定性。本文所用术语非胃肠道包括皮下、皮内、静脉内、肌内、关节内、动脉内、滑膜内、胸骨内、鞠内、损伤部位内、和颅内注射或输注技术。只要能达到目标组织,本发明所述药物组合物可以通过任何途径给予受体。
本发明药物组合物可以以任何口服剂型的形式口服给药,包括但不限于胶囊、片剂、乳剂和水悬浮液、分散剂和溶液。对于口服片剂,常用载体包括乳糖和玉米淀粉。一般还加入润滑剂例如硬脂酸镁。为了以胶囊形式口服给药,适用的稀释剂包括乳糖和无水玉米淀粉。当口服施用水悬浮液和/或乳液时,可将活性组分悬浮或溶解在油相中,并与乳化剂和/或悬浮剂合并。如果需要的话,可加入一些甜味剂和/或矫味剂和/或着色剂。适当时,可将用于口服给药的剂量单位制剂包微囊。例如,通过在聚合物、蜡等中将颗粒物质包衣或包埋,也可制备所述制剂已延长或维持释放。本发明的药物组合物可以用于补充内源性的XIRP2蛋白的缺失或不足,通过提高XIRP2蛋白的表达或增强XIRP2蛋白的功能,从而治疗因XIRP2蛋白减少导致的口腔鳞状细胞癌。
本发明的药物还可与其他治疗口腔鳞状细胞癌的药物联用,其他治疗性化合物可以与主要的活性成分同时给药,甚至在同一组合物中同时给药。还可以以单独的组合物或与主要的活性成分不同的剂量形式单独给予其它治疗性化合物。主要成分的部分剂量可以与其它治疗性化合物同时给药,而其它剂量可以单独给药。在治疗过程中,可以根据症状的严重程度、复发的频率和治疗方案的生理应答,调整本发明药物组合物的剂量。
本发明所述的药物组合物也可以脂质体输送系统形式给药,如小单层囊泡、大单层囊泡和多层囊泡。脂质体可有多种磷脂形成,如胆甾醇、硬脂基胺或磷脂酰胆碱。
本发明药物组合物可以局部给药的药物组合物,可以配制成软膏、乳膏剂、混悬剂、洗剂、散剂、溶液剂、糊剂、凝胶剂、喷雾剂、气雾剂或油剂。
本发明所述携带基因的载体是本领域已知的各种载体,如市售的载体、包括质粒、粘粒、噬菌体、病毒等。
本发明中术语“探针”指能与另一分子的特定序列或亚序列或其它部分结合的分子。除非另有指出,“探针”通常指能通过互补碱基配对与另一多核苷酸(往往称为“靶多核苷酸”)结合的多核苷酸探针。根据杂交条件的严谨性,探针能和与该探针缺乏完全序列互补性的靶多核苷酸结合。探针可作直接或间接的标记,其范围包括引物。杂交方式,包括,但不限于:溶液相、固相、混合相或原位杂交测定法。
作为探针,可以使用荧光标记、放射标记、生物素标记等对癌检测用多核苷酸进行了标记的标记探针。多核苷酸的标记方法本身是公知的。可通过如下方法检查试样中是否存在受试核酸:固定受试核酸或者其扩增物,与标记探针进行杂交,洗涤,以及然后测定与固相结合的标记。备选地,还可固定癌检测用多核苷酸,使受试核酸与其杂交,然后应用标记探针等检测结合于固相上的受试核酸。在这种情况下,结合于固相上的癌检测用多核苷酸也称为探针。使用多核苷酸探针测定受试核酸的方法在本领域也是公知的。可以如下进行该方法:在缓冲液中使多核苷酸探针与受试核酸在Tm或者其附近(优选在±4℃以内)接触用于杂交,洗涤,然后测定杂交的标记探针或者与固相探针结合的模板核酸。
在作为探针使用的多核苷酸的大小优选为18个或更多个核苷酸、更优选为20个或更多个核苷酸,以及编码区域的全长或更少。作为引物使用时,该多核苷酸大小优选为18个或更多个核苷酸,以及50个或更少核苷酸。
术语“芯片”也称为“阵列”,指包含连接的核酸或肽探针的固体支持物。阵列通常包含按照不同的已知位置连接至基底表面的多种不同的核酸或肽探针。这些阵列,也称为“微阵列”,通常可以利用机械合成方法或光引导合成方法来产生这些阵列,所述光引导合成方法合并了光刻方法和固相合成方法的组合。阵列可以包含平坦的表面,或者可以是珠子、凝胶、聚合物表面、诸如光纤的纤维、玻璃或任何其它合适的基底上的核酸或肽。可以以一定的方式来包装阵列,从而允许进行全功能装置的诊断或其它方式的操纵。
在本发明中,术语“抗体”是指选择性结合目标抗原的天然或合成抗体。该术语包括多克隆和单克隆抗体。除了完整的免疫球蛋白分子外,那些免疫球蛋白分子的片段或聚合物以及选择性结合目标抗原的免疫球蛋白分子的人类或人源化形式也包括在术语“抗体”的范围内,只要它们展现出期望的生物学活性即可。“单克隆抗体”指从一群基本上同质的抗体获得的抗体,即构成群体的各个抗体相同和/或结合相同表位,除了生产单克隆抗体的过程中可能产生的可能变体外,此类变体一般以极小量存在。此类单克隆抗体典型的包括包含结合靶物的多肽序列的抗体,其中靶物结合多肽序列是通过包括从众多多肽序列中选择单一靶物结合多肽序列在内的过程得到的。
单克隆抗体还包括“嵌合”抗体,其中重链和/或轻链的一部分与衍生自特定物种或属于特定抗体类别或亚类的抗体中的相应序列相同或同源,而链的剩余部分与衍生自另一物种或属于另一抗体类别或亚类的抗体中的相应序列相同或同源,以及此类抗体的片段,只要它们展现出期望的生物学活性即可。
多克隆抗体包含对产生人抗体的动物(例如,小鼠)免疫XIRP2蛋白质而得的抗体。当制备了嵌合抗体或人源化抗体之后,可以将可变区(例如,FR)和/或恒定区中的氨基酸用其他氨基酸替换等。
氨基酸的替换例如是小于15个、小于10个、8个以下、7个以下、6个以下、5个以下、4个以下、3个以下、或2个以下的氨基酸、优选1~5个氨基酸、更优选1或2个氨基酸的替换,替换抗体应与未替换抗体在功能上等同。期望替换是保守的氨基酸替换,这是电荷、侧链、极性、芳香族性等性质类似的氨基酸之间的替换。性质类似的氨基酸例如可以分类为碱性氨基酸(精氨酸、赖氨酸、组氨酸)、酸性氨基酸(天冬氨酸、谷氨酸)、无电荷极性氨基酸(甘氨酸、天冬酰胺、谷氨酰胺、丝氨酸、苏氨酸、半胱氨酸、酪氨酸)、无极性氨基酸(亮氨酸、异亮氨酸、丙氨酸、缬氨酸、脯氨酸、苯丙氨酸、色氨酸、蛋氨酸)、支链氨基酸(亮氨酸、缬氨酸、异亮氨酸)、芳香族氨基酸(苯丙氨酸、酪氨酸、色氨酸、组氨酸)等。
本发明中术语“治疗”是指以治愈、改善、稳定或预防疾病、病理状态或病症为目的对患者进行的医学管理。该术语包括积极疗法,即专门以改善疾病、病理状态或病症为目的的治疗,并且还包括病因治疗,即以除去相关疾病、病理状态或病症的病因为目的的治疗。此外,该术语还包括姑息治疗,即设计用于缓解症状而非治愈疾病、病理状态或病症的治疗;预防性治疗,即以最大程度降低或者部分或完全抑制相关疾病、病理状态或病症的发展为目的的治疗;以及支持性治疗,即用于补充另一种以改善相关疾病、病理状态或病症为目的的特定疗法的治疗。
下面结合附图和实施例对本发明作进一步详细的说明。以下实施例仅用于说明本发明而不用于限制本发明的范围。实施例中未注明具体条件的实验方法,通常按照常规条件,例如Sambrook等人,分子克隆:实验室手册(New York:Cold Spring HarborLaboratory Press,1989)中所述的条件,或按照制造厂商所建议的条件。
实施例1筛选与口腔鳞状细胞癌相关的基因标志物
1、样品收集
各收集6例周围正常粘膜组织和口腔鳞状细胞癌组织,均经病理学诊断证实,所有患者术前未接受任何形式的治疗。手术切下的样本于液氮中冻存,患者均知情同意,上述所有标本的取得均通过组织伦理委员会的同意。
2、RNA样品的制备(利用QIAGEN的组织RNA提取试剂盒进行操作)
取出冻存于液氮中的组织样本,把组织样本放入已预冷的研钵中进行研磨,按照试剂盒中的说明书提取分离RNA。具体如下:
1)加入Trizol,室温放置5min;
2)加入氯仿0.2ml,用力振荡离心管,充分混匀,室温下放置5-10min;
3)12000rpm离心15min,将上层水相移到另一新的离心管中(注意不要吸到两层水相之间的蛋白物质),加入等体积的-20℃预冷的异丙醇,充分颠倒混匀,置于冰上10min;
4)12000rpm高速离15min后小心弃掉上清液,按1ml/ml Trizol的比例加入75%DEPC乙醇洗涤沉淀(4℃保存),洗涤沉淀物,振荡混匀,4℃,12000rpm离心5min;
5)弃去乙醇液体,室温下放置5min,加入DEPC水溶解沉淀;
6)用Nanodrop2000紫外分光光度计测量RNA纯度及浓度,冻存于-70℃冰箱。
3、逆转录和标记
用Low RNA Input Linear Amplification Kit将mRNA逆转录成cDNA,同时用Cy3分别标记实验组和对照组。
4、杂交
基因芯片采用Aglient公司的人全基因组表达谱芯片,每张芯片包括45015个寡核苷酸,其中有43376个人基因探针和1639个实验控制探针。按芯片使用说明书的步骤进行,温度在65℃,经17h 10r/min滚动杂交,37℃洗片。
5、数据处理
杂交后芯片用Agilent扫描仪扫描,分辨率为5μm,扫描仪自动以100%和10%PMT各扫描1次,2次结果Agilent软件自动合并。扫描图像数据采用Feature Extraction进行处理分析,得到的原始数据应用Bioconductor程序包进行后续数据处理。最后Ratio值为实验组和对照组。差异基因筛选标准:ratio≥4为上调基因,ratio≤0.25为下调基因。
6、结果
与正常粘膜组织相比,XIRP2基因在口腔鳞状细胞癌组织中的表达量显著上调。
实施例2 QPCR测序验证XIRP2基因的差异表达
1、对XIRP2基因差异表达进行大样本QPCR验证。按照实施例1中的样本收集方式选择正常粘膜组织和口腔鳞状细胞癌组织各80例。
2、RNA提取步骤如实施例1所述。
3、逆转录:
1)反应体系:
表1逆转录反应体系
2)逆转录反应条件
按照RNA PCR Kit(AMV)Ver.3.0中逆转录反应条件进行。
42℃60min,99℃2min,5℃5min。
3)聚合酶链反应
1)引物设计
根据Genebank中XIRP2基因和GAPDH基因的编码序列设计QPCR扩增引物,由博迈德生物公司合成。具体引物序列如下:
XIRP2基因:
正向引物为5’-GCAGCCTTATCTACAGTC-3’(SEQ ID NO.3);
反向引物为5’-TCTTCCTTCCTTCCTTCT-3’(SEQ ID NO.4)。
管家基因GAPDH的引物序列为:
正向引物:5’-CTCTGGTAAAGTGGATATTGT-3’(SEQ ID NO.5)
反向引物:5’-GGTGGAATCATATTGGAACA-3’(SEQ ID NO.6)
2)按照表1配制25μl PCR反应体系:
表2 PCR反应体系
3)PCR反应条件:94℃4min,(94℃20s,60℃30s、72℃30s)×30个循环。以SYBR Green作为荧光标记物,在Light Cycler荧光定量PCR仪上进行PCR反应,通过融解曲线分析和电泳确定目的条带,ΔΔCT法进行相对定量,每个样进行3次重复实验。
5、统计学方法
以GAPDH为内参,计算口腔鳞状细胞癌组织与正常粘膜组织荧光定量RT-PCR的实验结果,采用SPSS18.0统计软件来进行统计分析,两者之间的差异采用t检验,以P<0.05具有统计学差异。
6、结果
结果如图1所示,与周围正常粘膜组织相比,XIRP2基因在口腔鳞状细胞癌组织中表达上调,差异具有统计学意义(P<0.05),同RNA-sep结果一致。
实施例3 XIRP2基因在口腔鳞状细胞癌细胞系中的差异表达
1、细胞培养
口腔鳞状细胞癌细胞系Tca8113、HN13,正常粘膜上皮细胞系HIOEC购自于上海交通大学附属第九人民医院。HIOEC的培养基为K-SFM;Tca8113、HN13的培养基为DMEM;以含10%胎牛血清和1%P/S的培养基在37℃、5%CO2、相对湿度为90%的培养箱中培养。2-3天换液1次,使用0.25%含EDTA的胰蛋白酶常规消化传代。
2、细胞总RNA的提取
1)当细胞达80-90%融合时终止培养,0.25%胰蛋白酶消化收集细胞于1.5m1EP管中,每管中加入lm1Trizol缓慢摇动破碎细胞,冰上放置10min。
2)去蛋白,去DNA:每个1.5m1EP管加入0.2ml三氯甲烷,摇晃15s,室温放置10min。4℃,12000rpm离心15min。
剩余操作步骤同组织中RNA提取过程。
3、逆转录
具体步骤同实施例2.
4、统计学方法
实验都是按照重复3次来完成的,结果数据都是以平均值±标准差的方式来表示,采用SPSS18.0统计软件来进行统计分析的,两者之间的差异采用t检验,认为当P<0.05时具有统计学意义。
5、结果
结果如图2所示,与正常粘膜上皮细胞相比,XIRP2基因在口腔鳞状细胞癌细胞Tca8113、HN13中表达均上调,差异具有统计学意义(P<0.05),同RNA-sep结果一致。
实施例4 XIRP2基因的沉默
1、细胞培养
人口腔鳞状细胞癌细胞株Tca8113,以含10%胎牛血清和1%P/S的培养基DMEM在37℃、5%CO2、相对湿度为90%的培养箱中培养。2-3天换液1次,使用0.25%含EDTA的胰蛋白酶常规消化传代。
2、siRNA设计
针对XIRP2基因的siRNA序列:
阴性对照siRNA序列(siRNA-NC):
正义链为5’-UUCUCCGAACGUGUCACGU-3’(SEQ ID NO.7),
反义链为5’-ACGUGACACGUUCGGAGAA-3’(SEQ ID NO.8);
siRNA1-XIRP2:
正义链为5’-UGUUGAUAUUGGUAUUGAGCA-3’(SEQ ID NO.9),
反义链为5’-CUCAAUACCAAUAUCAACAUC-3’(SEQ ID NO.10);
siRNA2-XIRP2:
正义链为5’-AUAACAUCCUCUUUCUGACAA-3’(SEQ ID NO.11),
反义链为5’-GUCAGAAAGAGGAUGUUAUAG-3’(SEQ ID NO.12);
将细胞按4×104/孔接种到六孔细胞培养板中,在37℃、5%CO2培养箱中细胞培养24h,在无双抗、含10%FBS的DMEM培养基中,转染按照脂质体转染试剂2000(购自于Invitrogen公司)的说明书转染。
将实验分为三组:对照组(Tca8113)、阴性对照组(siRNA-NC)和实验组(siRNA1-XIRP2、siRNA2-XIRP2),其中阴性对照组siRNA与XIRP2基因的序列无同源性,浓度为20nM/孔,同时分别进行转染。
3、QPCR检测XIRP2基因的转录水平
3.1细胞总RNA的提取
具体步骤同实施例3。
3.2逆转录步骤同实施例2。
3.3 QPCR扩增步骤同实施例2。
4、统计学方法
实验都是按照重复3次来完成的,结果数据都是以平均值±标准差的方式来表示,采用SPSS18.0统计软件来进行统计分析的,干扰XIRP2基因表达组与对照组之间的差异采用t检验,认为当P<0.05时具有统计学意义。
5、结果
结果如图3显示,相比TCA8113、转染空载siRNA-NC、siRNA2-XIRP2组,siRNA1-XIRP2组能够显著降低XIRP2基因的表达,差异具有统计学意义(P<0.05)。
实施例5 ELISA检测Tca8113细胞中XIRP2的蛋白表达
应用双抗体夹心酶标免疫(Enzyme-Linked Immunosorbent Assay,ELISA)分析法测定Tca8113细胞上清中XIRP2蛋白水平。RNA干扰后第六天,分别收集三组Tca8113细胞的上清液,按照ELISA试剂盒操作流程定量检测肿瘤细胞上清液中XIRP2的浓度。
1、配置浓度为70000pg/ml的标准品,10倍稀释后,再进行2倍倍比稀释,共有7个稀释度。
2、加样:分别设空白孔、标准孔、待测样品孔。空白孔加样品稀释液50μl,余孔分别加不同浓度梯度的标准品和待测样品各50μl。轻轻晃动混匀,酶标板加上盖,37℃反应2h。
3、弃去液体,晾干。每孔加200μl VEGF-C结合物。37℃,120min后,弃去孔内液体,晾干,PBS洗板3次。
4、依序每孔加底物溶液200μl,37℃避光显色30min。
5、依序每孔加终止溶液50μl,终止反应。
6、用酶联仪在450nm波长依序测量各孔的光密度(OD值)。所有标准品和待测样品的OD值均需减去零孔的OD值以得到校正值。
7、计算样品的实际浓度。
8、结果如下表3所示,siRNA沉默Tca8113细胞的XIRP2基因,XIRP2的蛋白含量也相应降低,说明沉默XIRP2基因可以抑制XIRP2基因的表达。
表3 ELISA检测不同组别细胞中的XIRP2蛋白的表达
实施例6 MTT法检测Tca8113细胞增殖活性
应用MTT(Methyl thiazolyl tetrazolium,甲基噻唑基四唑)法检测XIRP2基因沉默后对Tca8113细胞增殖活性的影响。
1、细胞培养将Tca8113细胞按1×103/孔接种于96孔板,每孔100μ1,37℃,5%CO2孵箱内孵育培养。
2、细胞转染步骤同实施例3。
3、MTT检测
1)分别于转染1~7天时,弃掉各孔培养基,加入MTT(5mg/ml)20μl。继续常规培养4h。
2)吸去混合液,每孔加入DMSO 200μl,震荡10min使结晶充分溶解。在酶联免疫仪上测490nm处的光吸收值,记录结果。
4、以时间为横轴,光吸收值(OD)为纵轴绘制细胞生长曲线。
5、结果:
结果如图4所示,与对照相比,转染siRNA1-XIRP2组的细胞增殖显著降低。
实施例7 Transwell细胞体外侵袭实验
RNA干扰后第六天收集不同组别的Tca8113细胞,重悬于培养液中,使细胞终浓度为2×106/ml,吸取100μl细胞悬液加入Transwell小室中。应用Transwell小室方法观察XIRP2基因沉默对Tca8113细胞侵袭性的影响。
1、将Matrigel(4μg/μl)放入4℃融化,准备冰盒(冰浴环境)。Matrigel用DMEM稀释8倍后使用。在Transwell小室滤膜(8μm孔径)的外表面涂8μg人纤粘连蛋白,置超净台内风干。
2、在6孔Transwell小室滤膜的内表面铺以100μ1/孔的Matrigel胶,37℃、5%CO2孵箱内孵育1h,形成一个基质屏障层备用。
3、在6孔板每孔内加入含20%FBS的DMEM培养液2.5m1。
4、收集对数生长期的细胞,重悬于培养液中,终浓度为2×106/ml。
5、将细胞悬液加入Transwell小室中,每孔100μ1,将小室浸于6孔板的条件培养基中,37℃、5%CO2孵箱内孵育24h。
6、将Transwell小室取出,滤膜用甲醇固定1分钟。
7、HE染色:苏木精染色3min,水洗;伊红染色10~30s,水洗。并用棉签擦掉未穿过膜的细胞。
8、显微镜下观察、照相并计数侵袭细胞数,每膜计数上下左右中5个不同视野的透过细胞数,计算平均值。每组平行设3个滤膜。
9、数据处理
用SPSS18.0软件对数据进行统计学分析。计量资料用均数±标准差表示。多个样本均数比较采用单因素方差分析,P<0.05为差异有统计学意义。
10、结果
结果如图5所示,Tca8113、siRNA1-XIRP2、siRNA-NC组细胞在transwell小室中培养24h后,siRNA1-XIRP2组聚碳酸酯膜下室面的细胞数显著减少。
上述实施例的说明只是用于理解本发明的方法及其核心思想。应当指出,对于本领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以对本发明进行若干改进和修饰,这些改进和修饰也将落入本发明权利要求的保护范围内。
daSEQUENCE LISTING
<110> 北京泱深生物信息技术有限公司
<120> 口腔鳞状细胞癌生物标记物及其应用
<160> 12
<170> PatentIn version 3.5
<210> 1
<211> 2817
<212> DNA
<213> 人源
<400> 1
atgttcccaa tgcagaaggg ctccctcaac ctcctgaggc agaaatggga atcttgtgat 60
tatcagagaa gtgagtgtca tcccagggac agccattgta caattttcca gcctcaggaa 120
agcaaattgc ttgcgcctga aggagaggta gtatcagcac ctcaatcttt ggatcccaca 180
agtctgccct acagtacagg ggaagagatg tggagttcga agccggaaga gaaggattct 240
gtggacaaga gtaacaacac cagggaatat ggtcggccag aagtgctgaa ggaggattcc 300
ctgagcagtc ggcgcaggat tgaacgcttt tccattgccc ttgatgagct gaggagtgtg 360
tttgaggctc ctaagagtgg aaacaaacca gctgagtacg gtggaaagga agtggaaatt 420
gagcgaagtt tgtgctcgcc agcttttaag agtcaccctg ggagccagct ggaggattct 480
gtgaaagatt cagacaagaa aggcaaggaa acatcttttg acaagatgtc acctgaaagt 540
ggtcacagcc gcatctttga agcgactgct ggccctaata agcctgagag tggatttgca 600
gaagacagtg ctgctcgggg cgagggtgtg tcagacctcc acgaagtggt ctccctgaag 660
gagcggatgg cgaggtacca ggcagctgtt tccaggggtg actgccgcag cttctctgct 720
aatatgatgg aagaatcaga aatgtgcgca gtgcctggtg gtttggccaa ggtgaagaaa 780
caatttgagg acgaaattac ttcttcccgt aatacctttg ctcaatacca atatcaacat 840
cagaacagat ctgagcagga ggcaattcat agcagccagg ttggcacttc aagaagcagc 900
caggaaatgg caagaaatga acaagaaggg tccaaagtac agaaaattga tgttcatgga 960
acagaaatgg tctctcatct tgaaaagcac accgaggaag taaaccaagc atctcagttt 1020
catcaatatg ttcaagaaac tgtcattgat acacctgagg atgaagagat tccaaaggtt 1080
tcgactaagt tgttaaaaga gcagtttgaa aagtctgccc aggaaaagat cctttattct 1140
gacaaagaga tgacaacccc agccaagcag attaagaagc tgctgctcca agacaaggaa 1200
atatgtatac tttgtcaaaa gacagtttat ccaatggagt gcctagtggc agacaagcag 1260
aattttcata agtcctgctt ccgatgccac cattgcaaca gtaaactaag tttgggaaat 1320
tatgcatcac ttcatggaca aatatactgt aaacctcact ttaaacaact tttcaaatcc 1380
aaaggaaatt atgatgaagg ttttggacat aagcagcata aagatagatg gaactgcaaa 1440
aaccaaagca gatcagtgga ctttattcct aatgaagaac caaatatgtg taaaaatatt 1500
gcagaaaaca cccttgtacc tggagatcgt aatgaacatt tagatgctgg taacagtgaa 1560
gggcaaagga atgatttgag aaaattaggg gaaaggggaa aattaaaagt catttggcct 1620
ccttccaagg agatccctaa gaaaacctta ccctttgagg aagagctcaa aatgagtaaa 1680
cctaagtggc cacctgaaat gacaaccctg ctatcccctg aatttaaaag tgaatctctg 1740
ctagaagatg ttagaactcc agaaaataaa ggacaaagac aagatcactt tccatttttg 1800
cagccttatc tacagtccac ccatgtttgt cagaaagagg atgttatagg aatcaaagaa 1860
atgaaaatgc ctgaaggaag aaaagatgaa aagaaggaag gaaggaagaa tgtgcaagat 1920
aggccgagtg aagctgaaga cacaaagagt aacaggaaaa gtgctatgga tcttaatgac 1980
aacaataatg tgattgtgca gagtgctgaa aaggagaaaa atgaaaaaac taaccaaact 2040
aatggtgcag aagttttaca ggttactaac actgatgatg agatgatgcc agaaaatcat 2100
aaagaaaatt tgaataagaa taataataac aattatgtag cagtctcata tctgaataat 2160
tgcaggcaga agacatctat tttagaattt cttgatctat tacccttgtc gagtgaagca 2220
aatgacactg caaatgaata tgaaattgag aagttagaaa atacatctag aatctcagag 2280
ttacttggta tatttgaatc tgaaaagact tattcgagga atgtactagc aatggctctg 2340
aagaaacaga ctgacagagc agctgctggc agtcctgtgc agcctgctcc aaaaccaagc 2400
ctcagcagag gccttatggt aaagggggga agttcaatca tctctcctga tacaaatctc 2460
ttaaacatta aaggaagcca ttcaaagagc aaaaatttac actttttctt ttctaacacc 2520
gtgaaaatca ctgcattttc caagaaaaat gagaacattt tcaattgtga tttaatagat 2580
tctgtagatc aaattaaaaa tatgccatgc ttggatttaa gggaatttgg aaaggatgtt 2640
aaaccttggc atgttgaaac aacagaagct gcccgcaata atgaaaacac aggttttgat 2700
gctctgagcc atgaatgtac agctaagcct ttgtttccca gagtggaggt gcagtcagaa 2760
caactcacgg tggaagagca gattaaaaga aacaggtgct acagtgacac tgagtaa 2817
<210> 2
<211> 938
<212> PRT
<213> 人源
<400> 2
Met Phe Pro Met Gln Lys Gly Ser Leu Asn Leu Leu Arg Gln Lys Trp
1 5 10 15
Glu Ser Cys Asp Tyr Gln Arg Ser Glu Cys His Pro Arg Asp Ser His
20 25 30
Cys Thr Ile Phe Gln Pro Gln Glu Ser Lys Leu Leu Ala Pro Glu Gly
35 40 45
Glu Val Val Ser Ala Pro Gln Ser Leu Asp Pro Thr Ser Leu Pro Tyr
50 55 60
Ser Thr Gly Glu Glu Met Trp Ser Ser Lys Pro Glu Glu Lys Asp Ser
65 70 75 80
Val Asp Lys Ser Asn Asn Thr Arg Glu Tyr Gly Arg Pro Glu Val Leu
85 90 95
Lys Glu Asp Ser Leu Ser Ser Arg Arg Arg Ile Glu Arg Phe Ser Ile
100 105 110
Ala Leu Asp Glu Leu Arg Ser Val Phe Glu Ala Pro Lys Ser Gly Asn
115 120 125
Lys Pro Ala Glu Tyr Gly Gly Lys Glu Val Glu Ile Glu Arg Ser Leu
130 135 140
Cys Ser Pro Ala Phe Lys Ser His Pro Gly Ser Gln Leu Glu Asp Ser
145 150 155 160
Val Lys Asp Ser Asp Lys Lys Gly Lys Glu Thr Ser Phe Asp Lys Met
165 170 175
Ser Pro Glu Ser Gly His Ser Arg Ile Phe Glu Ala Thr Ala Gly Pro
180 185 190
Asn Lys Pro Glu Ser Gly Phe Ala Glu Asp Ser Ala Ala Arg Gly Glu
195 200 205
Gly Val Ser Asp Leu His Glu Val Val Ser Leu Lys Glu Arg Met Ala
210 215 220
Arg Tyr Gln Ala Ala Val Ser Arg Gly Asp Cys Arg Ser Phe Ser Ala
225 230 235 240
Asn Met Met Glu Glu Ser Glu Met Cys Ala Val Pro Gly Gly Leu Ala
245 250 255
Lys Val Lys Lys Gln Phe Glu Asp Glu Ile Thr Ser Ser Arg Asn Thr
260 265 270
Phe Ala Gln Tyr Gln Tyr Gln His Gln Asn Arg Ser Glu Gln Glu Ala
275 280 285
Ile His Ser Ser Gln Val Gly Thr Ser Arg Ser Ser Gln Glu Met Ala
290 295 300
Arg Asn Glu Gln Glu Gly Ser Lys Val Gln Lys Ile Asp Val His Gly
305 310 315 320
Thr Glu Met Val Ser His Leu Glu Lys His Thr Glu Glu Val Asn Gln
325 330 335
Ala Ser Gln Phe His Gln Tyr Val Gln Glu Thr Val Ile Asp Thr Pro
340 345 350
Glu Asp Glu Glu Ile Pro Lys Val Ser Thr Lys Leu Leu Lys Glu Gln
355 360 365
Phe Glu Lys Ser Ala Gln Glu Lys Ile Leu Tyr Ser Asp Lys Glu Met
370 375 380
Thr Thr Pro Ala Lys Gln Ile Lys Lys Leu Leu Leu Gln Asp Lys Glu
385 390 395 400
Ile Cys Ile Leu Cys Gln Lys Thr Val Tyr Pro Met Glu Cys Leu Val
405 410 415
Ala Asp Lys Gln Asn Phe His Lys Ser Cys Phe Arg Cys His His Cys
420 425 430
Asn Ser Lys Leu Ser Leu Gly Asn Tyr Ala Ser Leu His Gly Gln Ile
435 440 445
Tyr Cys Lys Pro His Phe Lys Gln Leu Phe Lys Ser Lys Gly Asn Tyr
450 455 460
Asp Glu Gly Phe Gly His Lys Gln His Lys Asp Arg Trp Asn Cys Lys
465 470 475 480
Asn Gln Ser Arg Ser Val Asp Phe Ile Pro Asn Glu Glu Pro Asn Met
485 490 495
Cys Lys Asn Ile Ala Glu Asn Thr Leu Val Pro Gly Asp Arg Asn Glu
500 505 510
His Leu Asp Ala Gly Asn Ser Glu Gly Gln Arg Asn Asp Leu Arg Lys
515 520 525
Leu Gly Glu Arg Gly Lys Leu Lys Val Ile Trp Pro Pro Ser Lys Glu
530 535 540
Ile Pro Lys Lys Thr Leu Pro Phe Glu Glu Glu Leu Lys Met Ser Lys
545 550 555 560
Pro Lys Trp Pro Pro Glu Met Thr Thr Leu Leu Ser Pro Glu Phe Lys
565 570 575
Ser Glu Ser Leu Leu Glu Asp Val Arg Thr Pro Glu Asn Lys Gly Gln
580 585 590
Arg Gln Asp His Phe Pro Phe Leu Gln Pro Tyr Leu Gln Ser Thr His
595 600 605
Val Cys Gln Lys Glu Asp Val Ile Gly Ile Lys Glu Met Lys Met Pro
610 615 620
Glu Gly Arg Lys Asp Glu Lys Lys Glu Gly Arg Lys Asn Val Gln Asp
625 630 635 640
Arg Pro Ser Glu Ala Glu Asp Thr Lys Ser Asn Arg Lys Ser Ala Met
645 650 655
Asp Leu Asn Asp Asn Asn Asn Val Ile Val Gln Ser Ala Glu Lys Glu
660 665 670
Lys Asn Glu Lys Thr Asn Gln Thr Asn Gly Ala Glu Val Leu Gln Val
675 680 685
Thr Asn Thr Asp Asp Glu Met Met Pro Glu Asn His Lys Glu Asn Leu
690 695 700
Asn Lys Asn Asn Asn Asn Asn Tyr Val Ala Val Ser Tyr Leu Asn Asn
705 710 715 720
Cys Arg Gln Lys Thr Ser Ile Leu Glu Phe Leu Asp Leu Leu Pro Leu
725 730 735
Ser Ser Glu Ala Asn Asp Thr Ala Asn Glu Tyr Glu Ile Glu Lys Leu
740 745 750
Glu Asn Thr Ser Arg Ile Ser Glu Leu Leu Gly Ile Phe Glu Ser Glu
755 760 765
Lys Thr Tyr Ser Arg Asn Val Leu Ala Met Ala Leu Lys Lys Gln Thr
770 775 780
Asp Arg Ala Ala Ala Gly Ser Pro Val Gln Pro Ala Pro Lys Pro Ser
785 790 795 800
Leu Ser Arg Gly Leu Met Val Lys Gly Gly Ser Ser Ile Ile Ser Pro
805 810 815
Asp Thr Asn Leu Leu Asn Ile Lys Gly Ser His Ser Lys Ser Lys Asn
820 825 830
Leu His Phe Phe Phe Ser Asn Thr Val Lys Ile Thr Ala Phe Ser Lys
835 840 845
Lys Asn Glu Asn Ile Phe Asn Cys Asp Leu Ile Asp Ser Val Asp Gln
850 855 860
Ile Lys Asn Met Pro Cys Leu Asp Leu Arg Glu Phe Gly Lys Asp Val
865 870 875 880
Lys Pro Trp His Val Glu Thr Thr Glu Ala Ala Arg Asn Asn Glu Asn
885 890 895
Thr Gly Phe Asp Ala Leu Ser His Glu Cys Thr Ala Lys Pro Leu Phe
900 905 910
Pro Arg Val Glu Val Gln Ser Glu Gln Leu Thr Val Glu Glu Gln Ile
915 920 925
Lys Arg Asn Arg Cys Tyr Ser Asp Thr Glu
930 935
<210> 3
<211> 18
<212> DNA
<213> 人工序列
<400> 3
gcagccttat ctacagtc 18
<210> 4
<211> 18
<212> DNA
<213> 人工序列
<400> 4
tcttccttcc ttccttct 18
<210> 5
<211> 21
<212> DNA
<213> 人工序列
<400> 5
ctctggtaaa gtggatattg t 21
<210> 6
<211> 20
<212> DNA
<213> 人工序列
<400> 6
ggtggaatca tattggaaca 20
<210> 7
<211> 19
<212> RNA
<213> 人工序列
<400> 7
uucuccgaac gugucacgu 19
<210> 8
<211> 19
<212> RNA
<213> 人工序列
<400> 8
acgugacacg uucggagaa 19
<210> 9
<211> 21
<212> RNA
<213> 人工序列
<400> 9
uguugauauu gguauugagc a 21
<210> 10
<211> 21
<212> RNA
<213> 人工序列
<400> 10
cucaauacca auaucaacau c 21
<210> 11
<211> 21
<212> RNA
<213> 人工序列
<400> 11
auaacauccu cuuucugaca a 21
<210> 12
<211> 21
<212> RNA
<213> 人工序列
<400> 12
gucagaaaga ggauguuaua g 21
- 还没有人留言评论。精彩留言会获得点赞!