一种检测MET基因14外显子突变的探针库、检测方法和试剂盒与流程
技术领域
本发明涉及基因检测领域,具体而言,本发明涉及一种检测MET基因14外显子突变的探针及检测方法。该方法可以精准地富集MET基因14外显子突变序列,所得DNA样本库可进一步结合下一代测序技术(NGS),精准地判断病人的MET基因14外显子突变情况,并为相关肿瘤的诊断,预后,及临床治疗方案的设计提供指导及理论基础。
背景技术:
原发性肺癌(以下简称肺癌),发生于支气管粘膜上皮,故亦称支气管肺癌,是最常见的肺部原发性恶性肿瘤,也是我国最常见的恶性肿瘤之一,严重危害民众的生命与健康。
在我国,肺癌发病率和死亡率在男性常见恶性肿瘤中均占首位;在女性常见恶性肿瘤中分别占第一、二,仅次于乳腺癌的死亡人数。根据全国肿瘤登记中心2014年发布的数据显示,2010年,我国新发肺癌病例60.59万(男性41.63万,女性18.96万),占恶性肿瘤新发病例的19.59%(男性23.03%,女性14.75%)。肺癌发病率为35.23/10万(男性49.27/10万,女性21.66/10万)。同期,我国肺癌死亡人数为48.66万(男性32.58万,女性16.08万),占恶性肿瘤死因的24.87%(男性26.85%,女性21.32%)。肺癌死亡率为27.93/10万(男性39.79/10万,女性16.62/10万)。从分布来看,上海、北京、东北和沿海地区的几个较大城市的死亡率最高。
目前肺癌的治疗方式主要是手术、化疗、放疗,其中化疗是中晚期肺癌的主要治疗方式,但是其毒副作用较大,且个体间治疗疗效差异较大。随着肿瘤生物学的发展,及肿瘤发生机制的进一步探索,分子靶向治疗因其治疗有效率高、毒副作用小的特点,逐渐成为晚期肺癌的常规临床治疗方案。MET蛋白由原癌基因c-MET编码,是肝细胞生长因子HGF的受体。MET与多种细胞行为相关,参与细胞的信号传导、细胞骨架重排,促进细胞的增殖与分化。通过基因扩增或者过表达的方式激活MET的激酶活性可以促使细胞的癌变(8-10)。MET扩增在未经治疗的非小细胞肺癌(NSCLC)患者中并不常见,发生频率约为2-4%(8,9,11)。
从2014年首次发现MET基因14外显子跳读突变到现在,已经有多个研究针对不同的肿瘤类型对该突变类型进行研究。MET基因14外显子跳读突变在肺腺癌中的发生频率约为4%,而在肺肉瘤样癌中的频率高达22%。该突变的机理是,MET基因14外显子是MET蛋白泛素化降解过程中的结合位点,其丢失会导致MET蛋白无法被泛素化降解,从而引起MET蛋白的持续表达。
在非小细胞肺癌中,携带MET基因14外显子跳读突变的患者对MET抑制剂(克唑替尼、卡博替尼)在临床试验中有较好的应答。并且该突变与EGFR、ALK、KRAS、BRAF等突变不共存,意味着该靶点在肺腺癌中有较高的特异性。所以在NCCN指南中,建议MET基因14外显子跳读的患者选择克唑替尼进行治疗。MET基因14外显子跳读也成为肺腺癌中重要的一类分子分型。
MET基因14外显子跳读突变检测目前只有实时荧光定量PCR的试剂盒,其特点是检测方法简单,易于操作,但是需要提取RNA,对样本要求较高,实验结果判读较复杂,并且一次只能检测MET基因14外显子跳读突变,无法同时检测其它突变类型,所以实际上应用范围有限。
现有的主流基因突变检测方法主要集中于以下3个技术:
1)PCR突变检测
基于PCR片段扩增和凝胶电泳分离的方法,从而分辨出野生型和突变性的差异。其缺点包括:其为单碱基突变类型检测,不能对mRNA进行定量,不能检测基因颠换;敏感性低;耗时长,单次只能检测一个基因突变;不能确定碱基的具体变化;无法进行高通量检测。
2)Q-PCR(定量PCR)检测
基于荧光和PCR片段扩增来对模板mRNA多少来进行定量。其缺点包括:不能检测单碱基类型突变;不能检测基因颠换;只能检测已知突变;耗时长,单次只能检测一个基因突变;不能确定碱基的具体变化。
3)基因芯片技术
用高精度技术将DNA片段打印在芯片上,然后通过DNA杂交结合特性来确定突变性质。其缺点包括:不能检测基因颠换;只能检测已知突变;成本高,通量较小;准确率低,通常需要重复2次以上才能确定结果。
技术实现要素:
针对上述技术问题,本发明人通过大量实验,开发了用于检测MET基因14外显子突变的方法,该方法利用探针液相捕获、二代测序技术,不仅可以实现几千倍地富集的MET基因14外显子及其附近内含子的DNA片段,而且实现了较高的检测特异性和敏感性。
本发明的第一个方面:
一种检测MET基因14外显子突变的探针库,其中包括有核苷酸序列如SEQ ID NO. 1~46所示的任意一条探针,或者与其具有相同功能的探针。
优选的:探针库中包括上述的全部探针。
优选的:所述的具有相同功能的探针,是指将SEQ ID NO. 1~46任意一条探针经过一个或几个核苷酸的取代和/或缺失和/或添加且具有相同杂交捕获功能的探针。
优选的:所述的具有相同功能的探针与原探针具有80%以上相同的碱基,更优选是90%以上相同碱基,再优选是95%以上相同碱基。
本发明的第二个方面:
本发明提供一种富集MET基因14外显子及其附近内含子片段的方法,所述方法包括以下步骤:
1)获得受试者的DNA样本库;
2)获得所述的检测MET基因14外显子突变的探针;
3)使所述DNA探针库与所述DNA样本库进行杂交;和
4)分离步骤3)的杂交产物,然后释放经杂交富集的MET基因14外显子及其附近内含子DNA片段。
其中,所述步骤1)中的DNA样本库由双链DNA片段组成,并且,所述步骤1)包括:
1-1)提取全基因组DNA,然后将其片段化;或者
1-2)提取mRNA,将其片段化,然后以该经片段化的mRNA为模板合成双链cDNA;
其中,所述受试者为哺乳动物,优选人,且从受试者的细胞、组织或体液样本中提取全基因组DNA或mRNA;
优选地,所述DNA片段的长度为150~600bp;
进一步优选地,所述DNA片段的长度为150~200bp。
所述步骤2)中的DNA探针库为如上所述的DNA探针库。具体而言,所述DNA探针库包括一个或多个能够与MET基因14外显子杂交的DNA探针,所述DNA探针如以下序列所示:SEQ ID NO. 1~46。
此外,所述步骤3)包括:
3-1)采用选择性标记标记DNA探针库中的DNA探针;和
3-2)使所述DNA探针库与DNA样本库进行杂交;
优选地,所述步骤3-1)中的选择性标记为生物素;进一步优选地,所述步骤3-2)包括在PCR扩增仪中,在65℃下将所述DNA探针库与DNA样本库孵育24小时。
因此,所述方法的步骤4)中,优选利用DNA探针上的选择性标记分离杂交产物。进一步优选地,所述步骤3-1)中的选择性标记为生物素,所述步骤4)中利用链霉亲和素-生物素的亲和作用分离杂交产物。
本发明的第三个方面:
本发明还提供一种检测MET基因14外显子突变的方法,所述方法包括以下步骤:
1)根据上述方法富集MET基因14外显子及其附近内含子的DNA片段;和
2)检测所述MET基因14外显子的突变。
优选地,所述步骤2)中采用下一代测序技术,通过对富集到的MET基因14外显子片段进行测序而检测所述MET基因14外显子片段的突变。
本发明的第四个方面:
本发明提供一种用于检测MET基因14外显子突变的试剂盒,所述试剂盒包含上述的DNA探针库。
本发明的第五个方面:
上述的试剂盒在用于非治疗与诊断目的的MET基因14外显子突变测序中的应用。
有益效果
本发明开发了基于杂交选择而捕获特定MET基因14外显子及其附近内含子序列的方法,采用该方法可以获得成几千倍富集的MET基因14外显子及其附近内含子DNA片段,该经富集的MET基因14外显子及其附近内含子片段样本可以选择性地应用于各种基因检测技术,特别是可以应用下一代测序技术进行基因突变、缺失、增加、和颠换等方面的检测,以取得高效且准确的结果,对相关症状的后续治疗提供有意义的理论及临床指导。
并且,对于将通过本发明的方法富集得到的MET基因14外显子及其附近内含子片段用于基于下一代测序技术的基因结构突变检测的应用而言,还具有以下有益效果:
使用本发明的基因富集方法及筛选得到的特定DNA探针库,能够成数千倍地富集MET基因14外显子及其附近内含子片段,从而可以应用下一代测序技术、利用该MET基因14外显子及其附近内含子片段的测序,而准确地获得MET基因14外显子的各种突变。并且,由于采用下一代测序技术,因此能够一次性检测多个基因的多种类型基因突变;准确性高,传统技术例如基因芯片技术,通常需要重复两次以上才能确定检测结果,而本发明在一次反应中,对单个碱基进行反复测序,保证了数据的精准度,并且缩短了检测周期;敏感性高,本发明提供的方法和试剂盒能够有效地使对于低突变丰度样本的检测灵敏性得到提高,和传统检测技术相比,本发明产生的数据能够达到碱基级的分辨率。
附图说明
图1为本发明技术方案的示例性工艺流程图,其中富集得到目标基因群,并用于基于下一代测序技术的基因结构突变检测。
图2为本发明的探针设计策略的示意图。
图3为本发明的探针覆盖范围示意图。
图4是上机测序结果示意图。
具体实施方式
下面通过具体实施方式对本发明作进一步详细说明。但本领域技术人员将会理解,下列实施例仅用于说明本发明,而不应视为限定本发明的范围。实施例中未注明具体技术或条件者,按照本领域内的文献所描述的技术或条件或者按照产品说明书进行。所用试剂或仪器未注明生产厂商者,均为可以通过市购获得的常规产品。
在本文中所使用的术语”DNA”为脱氧核糖核酸(英文:Deoxyribonucleicacid,缩写为DNA)是一种由脱氧核糖核苷酸组成的双链分子。可组成遗传指令,引导生物发育与生命机能运行,其碱基排列顺序构成了遗传信息,所以在遗传病的诊断中具有重要的作用。
在本文中所使用的术语“高通量测序技术”指的是第二代高通量测序技术及之后发展的更高通量的测序方法。第二代高通量测序平台包括但不限于Illumina-Solexa(Miseq、Hiseq-2000、Hiseq-2500、Hiseq X ten等)、ABI-Solid和Roche-454测序平台等。随着测序技术的不断发展,本领域技术人员能够理解的是还可以采用
其他方法的测序方法和装置进行本检测。根据本发明的具体示例,可以将根据本发明实施例的核酸标签用于Illumina-Solexa、ABI-Solid和Roche-454测序平台等的至少一种进行测序。高通量测序技术,例如Miseq测序技术具有以下优势:(1)高灵敏度:高通量测序,例如Miseq的测序通量大,目前一个实验流程下来可以产生最多15G碱基数据,高的数据通量可以再测序序列数确定的情况下,使得每条序列获得高的测序深度,所以可以检测到含量更低的突变,同时因其测序深度高,其测序结果也更为可靠。(2)高通量,低成本:利用根据本发明实施例的标签序列,通过一次测序可以检测上万份样本,从而大大降低了成本。
本发明中的“突变”、“核酸变异”、“基因变异”可通用,本发明中的“SNP”(SNV)、“CNV”、“插入缺失”(indel)和“结构变异”(SV)同通常定义,但本发明中对各种变异的大小不作特别限定,这样这几种变异之间有的有交叉,比如当插入/缺失的为大片段甚至整条染色体时,也属于发生拷贝数变异(CNV)或是染色体非整倍性,也属于SV。这些类型变异的大小交叉并不妨碍本领域人员通过上述描述执行实现本发明的方法和/或装置并且达到所描述的结果。
本发明提供一种富集MET基因14外显子及其附近内含子DNA片段的方法。具体而言,本发明的方法包括:从哺乳动物例如人的细胞、体液或组织样本中提取基因组DNA或mRNA,经处理或合成cDNA,从而获得片段化的双链DNA作为DNA样本库;此外,针对要富集的MET基因14外显子片段,设计与该MET基因14外显子及部分内含子杂交的DNA探针,从中筛选出多个探针作为DNA探针库;然后,将该DNA样本库与DNA探针库进行杂交,从而从DNA样本库中富集得到MET基因14外显子及附近内含子DNA片段。根据本发明的具体实施方式,可以先将DNA探针库中的各个探针进行生物素化,然后在杂交后用链霉亲和素磁珠吸附杂交产物,再从磁珠上释放出富集的MET14外显子基因片段。经适应性处理,可以采用下一代测序基因对MET基因14外显子片段进行基因结构突变的检测,以确认MET14外显子基因的各类突变。
下面以富集得到的MET14外显子基因片段用于基于下一代测序技术的基因突变检测为例,示例性地说明本发明。
一、准备mRNA/DNA样本库
1. 准备基因组DNA样本(采用此种方式获得的DNA样本库称为“源自全基因组的DNA样本库”)
1.1 DNA提取
DNA提取,包括新鲜组织,新鲜血液和细胞,固定和石蜡样本,商业化公司提取试剂盒。以上均按说明书指示方法操作。
使用分光光度定量仪以及凝胶电泳系统检测DNA模板质量和浓度。DNA模板260nm吸光率大于0.05以上,吸光率A260/A280比值在1.8到2之间为合格。
1.2 DNA片段化
将3微克高质量的基因组DNA用低TE缓冲液稀释至120微升。按照组织匀浆机使用说明书,将DNA片段化,片段长度为150~200碱基。
DNA过柱纯化,商业化公司纯化试剂盒。
1.3 DNA样本库质量检测
用生物分析仪进行DNA定性定量分析,确认DNA片段长度峰值合理。
2. 准备cDNA样本(采用此种方式获得的DNA样本库称为“源自mRNA的DNA样本库”即cDNA样本库)
2.1 mRNA提取
mRNA提取,包括新鲜组织,新鲜血液和细胞,固定和石蜡样本,商业化公司提取试剂盒。以上均按说明书指示方法操作。
使用分光光度定量仪以及凝胶电泳系统检测mRNA质量和浓度,吸光率A260/A280比值在1.8到2之间为合格。
2.2 mRNA片段化
采用NEBNext RNA Fragmentation系统或者其他商业化公司mRNA片段化试剂盒。
mRNA过柱纯化,商业化公司纯化试剂盒。
2.3 用商业化公司cDNA合成试剂盒进行mRNA合成第一链以及第二链cDNA。
cDNA过柱纯化,商业化公司纯化试剂盒。
3. cDNA/DNA末端修补
将DNA片段进行末端修复可以利用Klenow片段、T4DNA聚合酶和T4多核苷酸激酶进行,其中,所述Klenow片段具有5’-3’’聚合酶活性和3’-5’聚合酶活性,但缺少5’-3’外切酶活性。由此,能够方便准确地对DNA片段进行末端修复。根据本发明的实施例,还可以进一步包括对经过末端修复的DNA片段进行纯化的步骤,由此能够方便地进行后续处理。
利用T4聚合酶及Klenow大肠杆菌聚合酶片断,对于cDNA/DNA 5'突出粘末端补平以及3'突出粘末端打平,产生平末端,用于后续的平端连接。反应在PCR扩增仪中进行,20摄氏度,30分钟。
cDNA/DNA过柱纯化,商业化公司纯化试剂盒。
4. 在cDNA/DNA样本3'末端加上碱基A
在经过末端修复的DNA片段的3’末端添加碱基A,以便获得具有粘性末端A的DNA片段。根据本发明的一个实施例,可以利用Klenow(3’-5’exo-),即具有3’-5’外切酶活性的Klenow,在经过末端修复的DNA片段的3’末端添加碱基A。由此,能够方便准确地将碱基A添加到经过末端修复的DNA片段的3’末端。根据本发明的实施例,还可以进一步包括对具有粘性末端A的DNA片段进行纯化的步骤,由此能够方便地进行后续处理。
反应在PCR扩增仪中进行,37℃,30分钟。
cDNA/DNA过柱纯化,商业化公司纯化试剂盒。
5. 在cDNA/DNA两端加上接头
cDNA/DNA过柱纯化,商业化公司纯化试剂盒。
如使用mRNA→cDNA,进行6和7;
如果是用基因组DNA,直接跳到8。
6. 分离出合适长度的cDNA片段
使用电泳凝胶,对照DNA梯度标准,在凝胶上剪切出150-250碱基cDNA片段。
将含有cDNA样本库的凝胶样本过柱纯化,商业化公司纯化试剂盒。
7. cDNA片段样本库质量检测
使用生物分析仪,进行cDNA定性定量分析,并确认分离出的cDNA片断长度峰值合理。
PCR条件:置于PCR扩增仪中,98℃预变性30秒,98℃变性30秒,65℃退火30秒,72℃延伸30秒,共循环15次(cDNA样本库)或者4-6次(DNA样本库)。最后在72℃延伸5分钟。
PCR扩增产物过柱纯化,商业化公司纯化试剂盒。
8. 扩增DNA模板
在本发明的一个实施例中,样本为含微量游离DNA片段的血浆样本,包含极其微量的目标游离DNA片段,第一扩增使得核酸的量能满足芯片/探针杂交捕获的需求
聚合酶链反应(PCR),在PCR扩增仪中进行。
PCR条件:置于PCR扩增仪中,98℃预变性30秒,98℃变性30秒,65℃退火30秒,72℃延伸30秒,共循环15次(cDNA样本库)或者4-6次(DNA样本库)。最后在72℃延伸5分钟。
PCR扩增产物过柱纯化,商业化公司纯化试剂盒。
9. 扩增后cDNA/DNA样本库质量检测
使用生物分析仪,进行cDNA/DNA定性定量分析,并确认纯化后片段长度峰值合理,约200bp。
对于得到的cDNA/DNA样本库,如果cDNA小于30纳克/微升,DNA浓度小于150纳克/微升,须将样品经过真空浓缩机低温干燥(低于45℃),再用无核酸酶水溶解至所需浓度。
二、探针的设计
针对MET14外显子基因准备DNA探针库。
本领域技术人员知晓:捕获的特异性受各种因素影响,如捕获探针的设计不佳,捕获条件不理想,基因组DNA中重复序列的封闭不充分及基因组DNA与捕获探针的比例不合适等因素都会影响捕获的特异性、敏感性、测序覆盖率等诸多结果。为了实现目标基因的高度富集和低脱靶率,本领域技术人员需要对探针的类型、长度、序列、杂交条件等进行大量实验摸索,需要通过创造性的探索工作才能够获得最佳的参数组合,没有在相应的证据证明下,其是否能够达到相同的效果,是本领域技术人员无法预期的。同时,对于产生突变的样本进行检测时,由于突变样本在组织样本中所占的比例会因个体而不同,因此,如果突变样本的丰度较低时,较容易导致的问题是探针往往无法准确地与突变的片断杂交,而导致检测的灵敏度低,这也需要对探针序列进行试验进行摸索。
由于基因组中的高GC、重复序列等复杂结构的存在,测序最终拼接组装获得的序列往往无法覆盖有所的区域,这部分没有获得的区域就称为Gap。例如一个细菌基因组测序,覆盖率是95%,那么还有5%的序列区域是没有通过测序获得的。
构建一个基于杂交原理的目标序列捕获系统,有较多的设计因素需要考虑,例如:探针的长度和探针的合成成本。一般来说一个8碱基的探针就有了足够的杂交特异性,而探针越长,杂交的特异性就越差。目前商业试剂盒的探针长度都在60nt到200nt之间,这其中的一个重要考虑是,杂交的特异性限定(或者说杂交的错配容忍度)。我们需要研究的是目标DNA片段中发生的突变、插入/缺失等,如果探针特异性太高,在DNA捕获时就会产生有利于参考序列(与探针完全互补的序列)的选择,这在后期数据统计上就会产生明显的偏差,同时太短的探针结合目标DNA片段的结合力度不足以将目标DNA捕获下来;而探针太长,探针合成的效率降低,并且合成成本大大增加。
在探针实际应用的时候,探针覆盖策略(Tiling Strategy),也是一个影响捕获效率的重要因素。具体含义是,目标区域中每个碱基位点被多少个不同的探针覆盖。例如,每个位点有2根不同探针覆盖,则该位点是2倍探针覆盖;每个位点有3根不同探针覆盖,则该位点是3倍探针覆盖。
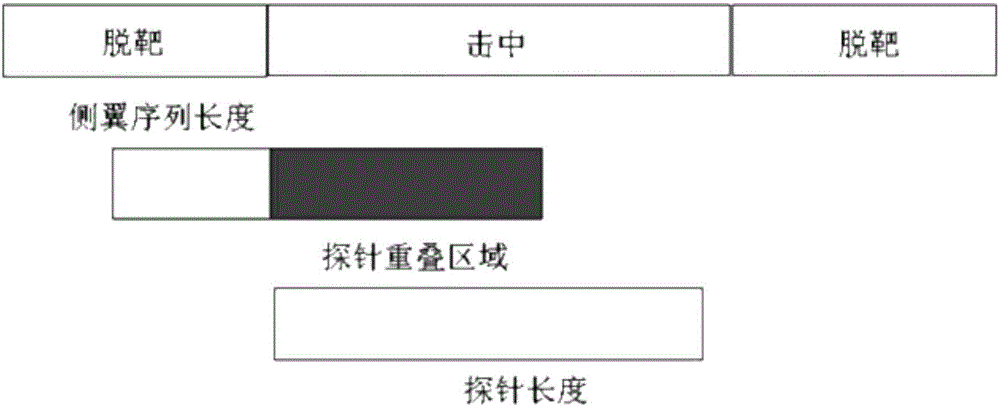
探针设计策略见图2和图3。其中,侧翼序列长度为内含子序列,内含子序列的重复性较高。探针重叠区域为每个探针之间的重叠碱基数目。重叠区域越大,目标区域覆盖率越高;重叠区域越小,甚至探针间隔分布,测序成本则越低。探针长度越长,测序时对于单核苷酸多态性(SNP)的容忍度越高。
使用每个探针分别对全基因组进行了富集和扩增并根据结果进行筛选。通过IDT DNA Technologies,单独合成了每一个探针并用质谱分析保证质量,在5’端有生物素(Biotin)。
三、DNA捕获探针杂交
1. 将DNA样本库与生物素化的DNA探针库杂交
将cDNA/DNA样本库与杂交缓冲液混合,反应条件为95℃ 5分钟,之后保持在65℃。反应在PCR扩增仪中进行。
然后将该混合物与探针库混合,反应条件为65℃ 5分钟。将杂交反应置于PCR扩增仪中,65℃孵育24小时。
四、得到经杂交富集的MET14外显子基因片段
1. 准备链霉亲和素(Streptavidin-Coated)磁珠
使用Dynabeads链霉亲和素磁珠或者其它商业化公司链霉亲和素磁珠。将磁珠置于混匀仪上混匀,每个样本需要50微升磁珠。
磁珠洗涤:混合50微升磁珠和200微升结合缓冲液,在混匀仪上混匀,使用Dynal磁选机或者其它商业化公司磁选机,将磁珠与缓冲液分离纯化,缓冲液弃掉不用。重复三次,每次加入200微升结合缓冲液。
2. 分离杂交产物
混合1中的杂交反应混合物与2中的链霉亲和素磁珠,反复颠倒试管5次。在室温下振摇30分钟。使用Dynal磁选机或者其它商业化公司磁选机,将磁珠分离纯化。
然后向磁珠中加入500微升洗涤缓冲液,在65℃孵育10分钟,每隔5分钟混匀一次。使用Dynal磁选机或者其它商业化公司磁选机,将磁珠分离纯化。
以上步骤重复三次。
3. cDNA/DNA富集样本释放
将磁珠与50微升洗脱缓冲液混合,室温孵化10分钟,每隔5分钟混匀一次。使用Dynal磁选机或者其它商业化公司磁选机,将磁珠分离弃掉。此时上清液中即含有富集过的MET14外显子基因片段cDNA/DNA样本库。
将样本库过柱纯化,商业化公司纯化试剂盒。
五、PCR扩增与纯化
因杂交捕获会损耗一定量的核酸,第二扩增能使捕获下的目标片段获得再次扩增以满足上机测序和质控检测的要求。本发明的这一文库构建方法特别适用于总游离核酸不低于10ng或者常规组织基因组DNA不低于1μg的样本的测序文库构建。
将富集cDNA/DNA样本库进一步扩增,为测序仪器上样做准备。
PCR条件:置于PCR扩增仪中,98℃预变性30秒,98℃变性30秒,65℃退火30秒,72℃延伸30秒,共循环15次(cDNA样本库)或者4-6次(DNA样本库)。最后在72℃延伸5分钟。
PCR扩增产物过柱纯化,商业化公司纯化试剂盒。
六、采用下一代测序技术检测MET基因14外显子的突变
使用下一代商业化的测序仪器进行测序,如Roche 454、Illumina Hiseq等。测序结果用已有的测序软件分析包进行分析。
示例性地,使用TruSeq PE Cluster Kit v3-cBot-HS,使用桥式PCR对DNA样本库模板进行扩增:每个DNA样本片段将会在芯片上形成克隆簇,每条泳道上产生数百万这样的克隆簇。使用Illumina HiSeq2000下一代测序系统。和传统Sanger方法相比,利用“可逆性末端终结反应”技术,四种dNTP碱基末端被保护基团封闭,并分别以不同颜色荧光标记。
经过QC筛选后,对测序结果使用了Bowtie对所得片段进行序列映射,利用Bioconductor软件,成功映射片段进行突变分析。
实施例1 富集并检测MET14外显子基因
一、构建样本库
1. 构建阳性质控质粒及内控质粒
阳性质控质粒:构建常见MET基因14外显子跳读突变D1028N(c.G3082A)的质粒。
内控质粒:与上阳性质粒相对应的同序列的野生型质粒。
将阳性质控质粒和内控质粒按照拷贝数比例混合,获得突变频率10%的质粒样本溶液,然后用Tris-HCl 缓冲液(2.5mM,pH8.5)调整DNA浓度,得到DNA浓度为5μg/μL的溶液。
2. DNA片段化
按照DNA破碎仪使用说明书,将DNA样本片段化,使片段长度为150-200碱基。
使用Beckman Coulter Ampure Beads试剂盒(货号:A63880)将DNA过柱纯化。
3. DNA样本库质量检测
用生物分析仪进行DNA定性定量分析,确认DNA片段长度峰值合理。
4. DNA末端修补
利用T4聚合酶及Klenow大肠杆菌聚合酶片断,对于cDNA/DNA 5'突出粘末端补平以及3'突出粘末端打平,产生平末端,用于后续的平端连接。反应在PCR扩增仪中进行,20摄氏度,30分钟。
使用Beckman Coulter Ampure Beads试剂盒(货号:A63880)将cDNA/DNA过柱纯化。
5. 在DNA样本3'末端加上碱基A
反应在PCR扩增仪中进行,37℃,30分钟。
使用Beckman Coulter Ampure Beads试剂盒(货号:A63880)将cDNA/DNA过柱纯化。
6. 在DNA两端加上接头
使用Beckman Coulter Ampure Beads试剂盒(货号:A63880)将cDNA/DNA过柱纯化。
7. 扩增5获得的DNA片段样本库
聚合酶链反应(PCR),在PCR扩增仪中进行。
PCR条件:置于PCR扩增仪中,98℃预变性30秒,98℃变性30秒,65℃退火30秒,72℃延伸30秒,共循环15次(cDNA样本库)或者4~6次(DNA样本库)。最后在72℃延伸5分钟。
使用Beckman Coulter Ampure Beads试剂盒(货号:A63880)将PCR扩增产物过柱纯化。
8. 扩增后DNA样本库的质量检测
使用生物分析仪,进行DNA定性定量分析,并确认纯化后片段长度峰值合理,约200bp。
对于得到的DNA样本库,如果DNA浓度小于150纳克/微升,须将样品经过真空浓缩机低温干燥(低于45℃),再用无核酸酶水溶解至所需浓度。
二、针对MET14外显子基因准备DNA探针库
根据上述的探针设计方法和思路,设计并合成探针进行试验,在5’端有生物素(Biotin)。
三、将DNA样本库与生物素化的DNA探针库杂交
将DNA样本库与杂交缓冲液(10mM Tris-HCl, 2%牛血清白蛋白, pH8.0)混合(混合后,DNA样本库浓度至多不超过50ng/ul),反应条件为95℃ 5分钟,之后保持在65℃。反应在PCR扩增仪中进行。
然后以DNA样本库:探针库为1:100的摩尔比,将探针库加入上述混合物,反应条件为65℃ 5分钟。将杂交反应置于PCR扩增仪中,65℃孵育24小时。
四、得到经杂交富集的MET14外显子基因片段
1. 准备链霉亲和素磁珠
使用Dynabeads(Life technologies, 货号:11206D)链霉亲和素磁珠或者其它商业化公司链霉亲和素磁珠。将磁珠置于混匀仪上混匀。
磁珠洗涤:混合50微升磁珠和200微升结合缓冲液(10mM Tris-HCl, 2%牛血清白蛋白, pH8.0),在混匀仪上混匀,使用Dynal磁选机或者其它商业化公司磁选机,将磁珠与缓冲液分离纯化,缓冲液弃掉不用。重复三次,每次加入200微升结合缓冲液。
2. 分离杂交产物
混合步骤三中得到的杂交反应混合物与步骤四的1中得到的链霉亲和素磁珠,反复颠倒试管5次。在室温下振摇30分钟。使用Dynal磁选机或者其它商业化公司磁选机,将磁珠分离纯化。
然后向磁珠中加入500微升洗涤缓冲液(磷酸缓冲液,0.1% Tween-20, 0.1% SDS, pH7.4),在65℃孵育10分钟,每隔5分钟混匀一次。使用Dynal磁选机或者其它商业化公司磁选机,将磁珠分离纯化。以上步骤重复三次。
3. DNA富集样本释放
将磁珠与50微升洗脱缓冲液(10mM氢氧化钠溶液)混合,室温孵化10分钟,每隔5分钟混匀一次。使用Dynal磁选机或者其它商业化公司磁选机,将磁珠分离弃掉。此时上清液中即含有富集过的MET14外显子基因片段DNA样本库。
使用Beckman Coulter Ampure Beads试剂盒(货号:A63880)将样本库过柱纯化。
五、PCR扩增与纯化
将富集cDNA/DNA样本库进一步扩增,为测序仪器上样做准备。
PCR条件:置于PCR扩增仪中,98℃预变性30秒,98℃变性30秒,65℃退火30秒,72℃延伸30秒,共循环15次(cDNA样本库)或者4-6次(DNA样本库)。最后在72℃延伸5分钟。
使用Beckman Coulter Ampure Beads试剂盒(货号:A63880)将PCR扩增产物过柱纯化。
六、采用下一代测序技术检测MET14外显子基因的基因结构突变
使用TruSeq PE Cluster Kit v3-cBot-HS,使用桥式PCR对DNA样本库模板进行扩增:每个DNA样本片段将会在芯片上形成克隆簇,每条泳道上产生数百万这样的克隆簇。使用Illumina HiSeq2000下一代测序系统,其原理是边合成边测序。和传统Sanger方法相比,利用“可逆性末端终结反应”技术,四种dNTP碱基末端被保护基团封闭,并分别以不同颜色荧光标记。
根据以上方法,采用不同探针进行测试结果如下:
探针长度对于检测的特异性、中靶率具有较大的影响,因此,在2X覆盖倍数的条件下设计了不同长度的探针,我们设计了3种探针长度,分别是100nt、120nt和140nt,通过中靶率和经济性来来确定什么长度下的探针最适合用于检测。
三次重复检测结果如下:
根据测序结果可以得到,140nt的探针中靶率最高,100nt探针的中靶率最低。其中120nt探针明显优于100nt探针,这两者之间有显著性差异;140nt探针优于120nt探针,这两者之间也有显著性差异。从中靶率来看,140nt探针的效果最好。
最后将探针长度确定为140个碱基,保证目标序列的捕获中靶率。通过对primer软件的改进,对设计的探针进行分析,以准确的知道探针的退火温度、GC成分连续重复单基数量(如CCCCCCC)。
在探针实际应用的时候,探针覆盖倍数也是一个影响捕获效率的重要因素。由于基因组中的高GC、重复序列等复杂结构的存在,测序最终拼接组装获得的序列往往无法覆盖有所的区域,这部分没有获得的区域就称为Gap。例如一个细菌基因组测序,覆盖率是95%,那么还有5%的序列区域是没有通过测序获得的。
探针覆盖倍数的含义是,目标区域中每个碱基位点被多少个不同的探针覆盖。例如,每个位点有2根不同探针覆盖,则该位点是2倍探针覆盖;每个位点有3根不同探针覆盖,则该位点是3倍探针覆盖。对此我们设计了4组实验,采用140nt长度探针,来确定最优化的探针覆盖倍数。实验结果如下:
根据测序结果可以得到,1倍覆盖倍数的探针中靶率最低,1.5倍覆盖倍数的探针中靶率优于1X,并且两者差异有统计学意义;2倍覆盖倍数的探针中靶率优于1.5X,两者有统计学差异;2.5倍覆盖倍数的探针中靶率优于2X,两者没有统计学差异。从中靶率来看,2.5倍覆盖倍数的探针效果最好,但是2.5X的探针价格高于2X,同时2.5X探针与2X探针的差异并不大,所以综合考虑选择2X探针。
并且我们分析了每个探针目标区域DNA序列的测序深度,结果如下:
注:>0.2x平均覆盖占比的意思是大于目标区域DNA序列平均覆盖倍数20%的区域在总区域中的占比,同理0.5x和1x平均覆盖占比的意思是大于目标区域平均覆盖倍数50%/100%的区域在总区域中的占比。
根据测序结果可以得到,随着探针覆盖倍数的增加,目标区域的平均覆盖倍数逐渐增加,并且均一性更好。但是外显子的探针捕获效率与内含子的探针捕获效率不一样,外显子在2X探针覆盖的情况下可以得到较好的目标区域覆盖,而内含子在2.5X探针覆盖的情况下才能得到与2X探针外显子类型的目标区域覆盖。综合中靶率、经济性,最后选择2X的探针作为外显子区域的覆盖倍数,选择2.5X探针作为内含子预期的覆盖倍数。
另外,对于低丰度样本的检测灵敏性也是需要经过试验考察。针对跳读突变D1028N设计了多组探针,并与其它探针共同进行了样本检测,探针如下:
第1组:1X的100nt探针
SEQ ID NO. 47
第2组:1X的120nt探针
SEQ ID NO. 48
第3组:2X的140nt探针
seq1: SEQ ID NO. 49
Seq2: SEQ ID NO. 50
第4组:2X的140nt探针
seq1: SEQ ID NO. 3
Seq2: SEQ ID NO. 26
可以看出,经过优选后的探针在检测时,可以检测到0.5%丰度的突变,具有较高的灵敏性。另外,经过DNA提取、PCR扩增和Sanger测序法,以上突变已被验证。
最终确定的优选探针序列如SEQ ID NO. 1~46所示。
对173例存在MET 14外显子跳跃突变的患者样本和5例阴性样本进行DNA提取,并通过本发明优选的探针和方法进行测序,结果中显示,阳性样本和阴性样本的测序结果与Sanger测序法所得结果相同,证明了本方法的可靠性。
SEQUENCE LISTING
<110> 南京世和基因生物技术有限公司
<120> 一种检测MET基因14外显子突变的探针库、检测方法和试剂盒
<130> 无
<160> 50
<170> PatentIn version 3.5
<210> 1
<211> 140
<212> DNA
<213> 人工序列
<400> 1
agaaagcaaa ttaaaggtgc atttttgtta ctgttcattt ttagaagtta ccttaagaac 60
acagtcatta cagtttaaga ttgtcgtcga ttcttgtgtg ctgtcttata tgtagtccat 120
aaaacccatg agttctgggc 140
<210> 2
<211> 140
<212> DNA
<213> 人工序列
<400> 2
actgggtcaa agtctcctgg ggcccatgat agccgtcttt aacaagctct ttctttctct 60
ctgttttaag atctgggcag tgaattagtt cgctacgatg caagagtaca cactcctcat 120
ttggataggc ttgtaagtgc 140
<210> 3
<211> 140
<212> DNA
<213> 人工序列
<400> 3
ccgaagtgta agcccaacta cagaaatggt ttcaaatgaa tctgtagact accgagctac 60
ttttccagaa ggtatatttc agtttattgt tctgagaaat acctatacat atacctcagt 120
gggttgtgac attgttgttt 140
<210> 4
<211> 140
<212> DNA
<213> 人工序列
<400> 4
atttttggtt ttgcatttat atttttataa aaacctaaag gaagtattta cctctgccaa 60
gtaagtattt gacacaaaat tacatggctc ttaattttaa aagaacccat gtatatatta 120
cattatgatt ttagagtcca 140
<210> 5
<211> 140
<212> DNA
<213> 人工序列
<400> 5
taagctctca tttcacaaaa aggttaattt gagcaaaagt aatttgttta tcatctaagt 60
gcaatagtaa gaaattgcga agctctcttt tacaatccag gaagagttaa gttacaaaat 120
atacttattt aaatgtaagt 140
<210> 6
<211> 140
<212> DNA
<213> 人工序列
<400> 6
tggaactgct acatttttta cctgttgaag cccaaacatt gaaattatac tgttagtaat 60
tcttcgaagt gttttcaatg aactgttagt acacagcctt tttcccacca tattctagga 120
cttgaatgta ttttgagact 140
<210> 7
<211> 140
<212> DNA
<213> 人工序列
<400> 7
tagccaagga aaaccttcaa ttatgccatg aaaaaaagga ggggtcaata tcatcagctt 60
tgtaaaacac tatgcctagt aatgttcagg ttaatcagag ttttcatgtt gttttattta 120
aatctcctgg taaaagcaaa 140
<210> 8
<211> 140
<212> DNA
<213> 人工序列
<400> 8
aggtctgtat tgtatcagct ccattatctt tagaagttac aggatgtgag tcaagtacaa 60
gcatttcctt ggttgaatat ttaccattgg acaaataaaa tgagtcacag atcattgagg 120
atactggaaa agttagaagt 140
<210> 9
<211> 140
<212> DNA
<213> 人工序列
<400> 9
tgctcatcca aacaagttca agagcaatga agcacttaac attttaacat tttcaacact 60
tactacctct tatgttttga agtttatgtt atttctatgg agatacacat agtaaacatt 120
gtctttgccc tgattccatt 140
<210> 10
<211> 140
<212> DNA
<213> 人工序列
<400> 10
cacctttaaa aatccattcg tttaaccgtg tggaaaaatc aaacctagtt tattgttttg 60
aaatttagat ctatttagta ttttatgtgc acatttagtg catctattta gtattttata 120
tgcacatttc atagttctaa 140
<210> 11
<211> 140
<212> DNA
<213> 人工序列
<400> 11
tctgagatca ttaaaattta caaattttct ttgaaaaaaa aacttaccta atcttctttg 60
aacctcctta ctcaccaaag ctctgtcatc attgctaaga aggttgagtt tcacactctt 120
ttctccattg agcctgctcc 140
<210> 12
<211> 140
<212> DNA
<213> 人工序列
<400> 12
ttggagacat gaaaagaaaa caggtaaaag agggtcattt agagagaatg agaaaatagg 60
tgcacagcca aaacctaatg aagaggcaac tgcagagctt tcctctctac atctggtggg 120
gacagcattc tcatcagact 140
<210> 13
<211> 140
<212> DNA
<213> 人工序列
<400> 13
ttttcacgga gacctagagt gctatgtggt gtgacatcag ggtggcacac tgatggtttc 60
aattggtttc tgcacatgtt ggaatttagc tgaagagtca cgttttcatg ccaaagggct 120
tttatccatg tctcaccaag 140
<210> 14
<211> 140
<212> DNA
<213> 人工序列
<400> 14
gatttccctc aatctgtgca cccttaagca tttagagccc tgatctccag atgcaaaggc 60
tttaggaagt gagaatgaaa gacctgagtt tagagaggct gattggcatt cccaatcccc 120
tggggaaggt ttagagaccc 140
<210> 15
<211> 140
<212> DNA
<213> 人工序列
<400> 15
tgactccttg gaattaaggg agcaagtacc cagctaggct ccttccttcc tcactcaccc 60
aacatttcag gtacttcact gatgttccac atccttcttt aaaggttgct cttgtctttt 120
ttctggctag tgtctactat 140
<210> 16
<211> 140
<212> DNA
<213> 人工序列
<400> 16
aactgtaatt gatgcccaac gcttttctgg aaccactttt ggccaagttc atttattatt 60
aatcaaactg tccactgtag aaaatactaa aaatgctcaa gtgggattag gaatagtcaa 120
ggtactaaca gcattctttt 140
<210> 17
<211> 140
<212> DNA
<213> 人工序列
<400> 17
tatgcccttc tctcagattc tgattctcct gcttatttgc aaacaaatga tacattttgg 60
tgctaatgag gaacccccac ataaccttct ccctgtgtta catactaata catttcaata 120
ctatgcctag tttatcttca 140
<210> 18
<211> 140
<212> DNA
<213> 人工序列
<400> 18
tgtcagttgc tgtggctatg atgccccctc cttgatatgt gtgaattccc agtggaaaga 60
gaaagggaaa gtggaaatgc cctatttggc attaagaaat tgactatcag caccatttct 120
tcccctgaaa taaaaaaaaa 140
<210> 19
<211> 140
<212> DNA
<213> 人工序列
<400> 19
aattctcctt gcaaaaggga actttgcctg aggttcttac agagctttgg ttataaagat 60
caacttataa agaatgctta cccctttcat agtgtcctta actaaacaac aaggatggtc 120
cactaaccga gatctaacct 140
<210> 20
<211> 140
<212> DNA
<213> 人工序列
<400> 20
gccttctcta aacaacagta acactaaatc cagtgccatc actgcacagt ggagaattta 60
ccactaatgt gaaaagcttt cagttttggg aatatagcca ttatttattt ctaatcatat 120
gtgtattttt cccttggcca 140
<210> 21
<211> 140
<212> DNA
<213> 人工序列
<400> 21
ggaatccata ggttttgcac aatagtaatt aattccatta acaaatagta gtgtctcaaa 60
aggcatcttt ttcattttct tatatttgag ctggattttt gtgagacgag gcaattgctc 120
aactaccttt gctgctacca 140
<210> 22
<211> 140
<212> DNA
<213> 人工序列
<400> 22
ctgcttccat tcttaaggac atagtatatt caaaaataaa ccataagcat ggctttttgc 60
tattgataaa gagagaaatg tctaaggaaa tgagggtaaa aagctttcaa aattaatact 120
tagtctactt aaatgaaaat 140
<210> 23
<211> 140
<212> DNA
<213> 人工序列
<400> 23
ctgtaaacat ctaatgaaat gcttgtatat ataacttagt atcttttccc aatttattat 60
catttttatc aaactaattc cattataaaa gctcttcctg tttcagtccc cattaaatga 120
ggttttactg ttgttcttta 140
<210> 24
<211> 140
<212> DNA
<213> 人工序列
<400> 24
cttaagaaca cagtcattac agtttaagat tgtcgtcgat tcttgtgtgc tgtcttatat 60
gtagtccata aaacccatga gttctgggca ctgggtcaaa gtctcctggg gcccatgata 120
gccgtcttta acaagctctt 140
<210> 25
<211> 140
<212> DNA
<213> 人工序列
<400> 25
tctttctctc tgttttaaga tctgggcagt gaattagttc gctacgatgc aagagtacac 60
actcctcatt tggataggct tgtaagtgcc cgaagtgtaa gcccaactac agaaatggtt 120
tcaaatgaat ctgtagacta 140
<210> 26
<211> 140
<212> DNA
<213> 人工序列
<400> 26
ccgagctact tttccagaag gtatatttca gtttattgtt ctgagaaata cctatacata 60
tacctcagtg ggttgtgaca ttgttgttta tttttggttt tgcatttata tttttataaa 120
aacctaaagg aagtatttac 140
<210> 27
<211> 140
<212> DNA
<213> 人工序列
<400> 27
ctctgccaag taagtatttg acacaaaatt acatggctct taattttaaa agaacccatg 60
tatatattac attatgattt tagagtccat aagctctcat ttcacaaaaa ggttaatttg 120
agcaaaagta atttgtttat 140
<210> 28
<211> 140
<212> DNA
<213> 人工序列
<400> 28
catctaagtg caatagtaag aaattgcgaa gctctctttt acaatccagg aagagttaag 60
ttacaaaata tacttattta aatgtaagtt ggaactgcta cattttttac ctgttgaagc 120
ccaaacattg aaattatact 140
<210> 29
<211> 140
<212> DNA
<213> 人工序列
<400> 29
gttagtaatt cttcgaagtg ttttcaatga actgttagta cacagccttt ttcccaccat 60
attctaggac ttgaatgtat tttgagactt agccaaggaa aaccttcaat tatgccatga 120
aaaaaaggag gggtcaatat 140
<210> 30
<211> 140
<212> DNA
<213> 人工序列
<400> 30
catcagcttt gtaaaacact atgcctagta atgttcaggt taatcagagt tttcatgttg 60
ttttatttaa atctcctggt aaaagcaaaa ggtctgtatt gtatcagctc cattatcttt 120
agaagttaca ggatgtgagt 140
<210> 31
<211> 140
<212> DNA
<213> 人工序列
<400> 31
caagtacaag catttccttg gttgaatatt taccattgga caaataaaat gagtcacaga 60
tcattgagga tactggaaaa gttagaagtt gctcatccaa acaagttcaa gagcaatgaa 120
gcacttaaca ttttaacatt 140
<210> 32
<211> 140
<212> DNA
<213> 人工序列
<400> 32
ttcaacactt actacctctt atgttttgaa gtttatgtta tttctatgga gatacacata 60
gtaaacattg tctttgccct gattccattc acctttaaaa atccattcgt ttaaccgtgt 120
ggaaaaatca aacctagttt 140
<210> 33
<211> 140
<212> DNA
<213> 人工序列
<400> 33
attgttttga aatttagatc tatttagtat tttatgtgca catttagtgc atctatttag 60
tattttatat gcacatttca tagttctaat ctgagatcat taaaatttac aaattttctt 120
tgaaaaaaaa acttacctaa 140
<210> 34
<211> 140
<212> DNA
<213> 人工序列
<400> 34
tcttctttga acctccttac tcaccaaagc tctgtcatca ttgctaagaa ggttgagttt 60
cacactcttt tctccattga gcctgctcct tggagacatg aaaagaaaac aggtaaaaga 120
gggtcattta gagagaatga 140
<210> 35
<211> 140
<212> DNA
<213> 人工序列
<400> 35
gaaaataggt gcacagccaa aacctaatga agaggcaact gcagagcttt cctctctaca 60
tctggtgggg acagcattct catcagactt tttcacggag acctagagtg ctatgtggtg 120
tgacatcagg gtggcacact 140
<210> 36
<211> 140
<212> DNA
<213> 人工序列
<400> 36
gatggtttca attggtttct gcacatgttg gaatttagct gaagagtcac gttttcatgc 60
caaagggctt ttatccatgt ctcaccaagg atttccctca atctgtgcac ccttaagcat 120
ttagagccct gatctccaga 140
<210> 37
<211> 140
<212> DNA
<213> 人工序列
<400> 37
tgcaaaggct ttaggaagtg agaatgaaag acctgagttt agagaggctg attggcattc 60
ccaatcccct ggggaaggtt tagagaccct gactccttgg aattaaggga gcaagtaccc 120
agctaggctc cttccttcct 140
<210> 38
<211> 140
<212> DNA
<213> 人工序列
<400> 38
cactcaccca acatttcagg tacttcactg atgttccaca tccttcttta aaggttgctc 60
ttgtcttttt tctggctagt gtctactata actgtaattg atgcccaacg cttttctgga 120
accacttttg gccaagttca 140
<210> 39
<211> 140
<212> DNA
<213> 人工序列
<400> 39
tttattatta atcaaactgt ccactgtaga aaatactaaa aatgctcaag tgggattagg 60
aatagtcaag gtactaacag cattcttttt atgcccttct ctcagattct gattctcctg 120
cttatttgca aacaaatgat 140
<210> 40
<211> 140
<212> DNA
<213> 人工序列
<400> 40
acattttggt gctaatgagg aacccccaca taaccttctc cctgtgttac atactaatac 60
atttcaatac tatgcctagt ttatcttcat gtcagttgct gtggctatga tgccccctcc 120
ttgatatgtg tgaattccca 140
<210> 41
<211> 140
<212> DNA
<213> 人工序列
<400> 41
gtggaaagag aaagggaaag tggaaatgcc ctatttggca ttaagaaatt gactatcagc 60
accatttctt cccctgaaat aaaaaaaaaa attctccttg caaaagggaa ctttgcctga 120
ggttcttaca gagctttggt 140
<210> 42
<211> 140
<212> DNA
<213> 人工序列
<400> 42
tataaagatc aacttataaa gaatgcttac ccctttcata gtgtccttaa ctaaacaaca 60
aggatggtcc actaaccgag atctaacctg ccttctctaa acaacagtaa cactaaatcc 120
agtgccatca ctgcacagtg 140
<210> 43
<211> 140
<212> DNA
<213> 人工序列
<400> 43
gagaatttac cactaatgtg aaaagctttc agttttggga atatagccat tatttatttc 60
taatcatatg tgtatttttc ccttggccag gaatccatag gttttgcaca atagtaatta 120
attccattaa caaatagtag 140
<210> 44
<211> 140
<212> DNA
<213> 人工序列
<400> 44
tgtctcaaaa ggcatctttt tcattttctt atatttgagc tggatttttg tgagacgagg 60
caattgctca actacctttg ctgctaccac tgcttccatt cttaaggaca tagtatattc 120
aaaaataaac cataagcatg 140
<210> 45
<211> 140
<212> DNA
<213> 人工序列
<400> 45
gctttttgct attgataaag agagaaatgt ctaaggaaat gagggtaaaa agctttcaaa 60
attaatactt agtctactta aatgaaaatc tgtaaacatc taatgaaatg cttgtatata 120
taacttagta tcttttccca 140
<210> 46
<211> 140
<212> DNA
<213> 人工序列
<400> 46
atttattatc atttttatca aactaattcc attataaaag ctcttcctgt ttcagtcccc 60
attaaatgag gttttactgt tgttctttaa taattttcct tcatcttaca gatcagtttc 120
ctaattcatc tcagaacggt 140
<210> 47
<211> 100
<212> DNA
<213> 人工序列
<400> 47
tgtaagtgcc cgaagtgtaa gcccaactac agaaatggtt tcaaatgaat ctgtagacta 60
ccgagctact tttccagaag gtatatttca gtttattgtt 100
<210> 48
<211> 120
<212> DNA
<213> 人工序列
<400> 48
gtgtaagccc aactacagaa atggtttcaa atgaatctgt agactaccga gctacttttc 60
cagaaggtat atttcagttt attgttctga gaaataccta tacatatacc tcagtgggtt 120
<210> 49
<211> 140
<212> DNA
<213> 人工序列
<400> 49
ttgtaagtgc ccgaagtgta agcccaacta cagaaatggt ttcaaatgaa tctgtagact 60
accgagctac ttttccagaa ggtatatttc agtttattgt tctgagaaat acctatacat 120
atacctcagt gggttgtgac 140
<210> 50
<211> 140
<212> DNA
<213> 人工序列
<400> 50
ctgtagacta ccgagctact tttccagaag gtatatttca gtttattgtt ctgagaaata 60
cctatacata tacctcagtg ggttgtgaca ttgttgttta tttttggttt tgcatttata 120
tttttataaa aacctaaagg 140
- 还没有人留言评论。精彩留言会获得点赞!