MYLK基因作为诊断子宫内膜癌的标志物的制作方法
本发明属于诊断领域,涉及一种子宫内膜癌的诊断工具,还涉及mylk基因在制备子宫内膜癌诊断工具中的应用。
背景技术:
子宫内膜癌(endometrialcarcinoma或carcinomaofendometrium,ec)是发生于子宫内膜的一组上皮性恶性肿瘤,以来源于子宫内膜腺体的腺癌最常见,为女性生殖道三大恶性肿瘤之一,占女性全身恶性肿瘤7%,在全世界范围不同地区其发病率有差异,是西方工业化国家最常见的妇科恶性肿瘤,北美、北欧地区发病率最高,亚洲日本、印度及中南美等地区发病率较低。我国尚缺乏子宫内膜癌比较详尽的流行病学调查资料,估计与日本发病情况相仿。北美及欧洲发达国家高于发展中国家,前者是后者的10倍,其发病率位于乳腺癌、大肠癌、肺癌之后,是女性全身第四位常见恶性肿瘤,位居女性生殖道恶性肿瘤第一位。
虽然子宫内膜癌症状出现较早、诊断相对较容易。但是在实际临床工作中关于子宫内膜癌的诊断及治疗仍存在一些争议。肿瘤的恶性程度及病变范围,包括手术分期、组织学类型、肿瘤分级、肌层浸润深度、淋巴转移及子宫外转移等与子宫内膜癌的预后有关。因此,早期诊断子宫内膜癌非常重要。目前,子宫内膜癌的诊断主要依据病史及临床表现,b超、ct及mri等影像学检查,诊断性刮宫,宫腔镜,肿瘤标志物ca-125等辅助检查。其中ca-125还可以作为疗效观察的指标。但ca-125的缺乏组织敏感性和特异性,在卵巢上皮性肿瘤、子宫内膜癌、子宫内膜异位症、消化系统肿瘤等疾病中均可检测到ca-125水平升高。
近年来,基因诊断成为了体外诊断行业新的发展趋势。基因诊断,又称之为dna诊断或分子诊断,是应用分子生物学方法,检测患者体内遗传物质结构或表达水平的变化,继而做出诊断的技术,它为疾病的预防、预测、诊断、治疗和疾病转归提供更为准确的信息。鉴于目前子宫内膜癌诊断方法存在的以上缺陷,急需开发一种价格便宜,操作简便,诊断灵敏度和特异性高的可用于诊断子宫内膜癌的基因诊断的方法。
技术实现要素:
本发明首次发现mylk基因在子宫内膜癌组织中的含量比正常子宫内膜组织低很多,mylk基因的差异表达可作为诊断子宫内膜癌的一种方法,据此可以开发诊断子宫内膜癌的工具。
具体的,本发明提供了检测mylk基因表达的产品在制备诊断子宫内膜癌的工具中的应用。
进一步,上面所提到的检测产品包括:通过rt-pcr、实时定量pcr、免疫检测、原位杂交、芯片或高通量测序平台检测mylk基因的表达水平以诊断子宫内膜癌的产品。
进一步,所述用rt-pcr诊断子宫内膜癌的产品至少包括一对特异扩增mylk基因的引物;所述用实时定量pcr诊断子宫内膜癌的产品至少包括一对特异扩增mylk基因的引物;所述用免疫检测诊断子宫内膜癌的产品包括:与mylk蛋白特异性结合的抗体;所述用原位杂交诊断子宫内膜癌的产品包括:与mylk基因的核酸序列杂交的探针;所述用芯片诊断子宫内膜癌的产品包括:蛋白芯片和基因芯片;其中,蛋白芯片包括与mylk蛋白特异性结合的抗体,基因芯片包括与mylk基因的核酸序列杂交的探针。
在本发明的具体实施方案中,所述用实时定量pcr诊断子宫内膜癌的产品至少包括一对特异扩增mylk基因的引物的序列如seqidno.3和seqidno.4所示。
优选地,所述诊断工具包括芯片、试剂盒、试纸或高通量测序平台。其中,高通量测序平台是一种特殊的诊断工具,检测mylk基因表达的产品可以应用于该平台实现对mylk基因的表达情况的检测。随着高通量测序技术的发展,对一个人的基因表达谱的构建将成为十分便捷的工作。通过对比疾病患者和正常人群的基因表达谱,容易分析出哪个基因的异常与疾病相关。因此,在高通量测序中获知mylk基因的异常与子宫内膜癌相关也属于mylk基因的用途,同样在本发明的保护范围之内。
本发明还提供了一种诊断子宫内膜癌的工具,所述产品包括芯片、试剂盒、试纸、或高通量测序平台。
其中,所述芯片包括基因芯片、蛋白质芯片;所述基因芯片包括固相载体以及固定在固相载体的寡核苷酸探针,所述寡核苷酸探针包括用于检测mylk基因转录水平的针对mylk基因的寡核苷酸探针;所述蛋白质芯片包括固相载体以及固定在固相载体的mylk蛋白的特异性抗体;所述基因芯片可用于检测包括mylk基因在内的多个基因(例如,与子宫内膜癌相关的多个基因)的表达水平。所述蛋白质芯片可用于检测包括mylk蛋白在内的多个蛋白质(例如与子宫内膜癌相关的多个蛋白质)的表达水平。通过将多个与子宫内膜癌的标志物同时检测,可大大提高子宫内膜癌诊断的准确率。
其中,所述试剂盒包括基因检测试剂盒和蛋白免疫检测试剂盒;所述基因检测试剂盒包括用于检测mylk基因转录水平的试剂;所述蛋白免疫检测试剂盒包括mylk蛋白的特异性抗体。进一步,所述试剂包括使用rt-pcr、实时定量pcr、免疫检测、原位杂交或芯片方法检测mylk基因表达水平过程中所需的试剂。优选度,所述试剂包括针对mylk基因的引物和/或探针。根据mylk基因的核苷酸序列信息容易设计出可以用于检测mylk基因表达水平的引物和探针。
所述高通量测序平台包括检测mylk基因表达水平的试剂。
所述试纸包括试纸载体和固定在试纸载体上的寡核苷酸,所述寡核苷酸能够检测mylk基因的转录水平。
与mylk基因的核酸序列杂交的探针可以是dna、rna、dna-rna嵌合体、pna或其它衍生物。所述探针的长度没有限制,只要完成特异性杂交、与目的核苷酸序列特异性结合,任何长度都可以。所述探针的长度可短至25、20、15、13或10个碱基长度。同样,所述探针的长度可长至60、80、100、150、300个碱基对或更长,甚至整个基因。由于不同的探针长度对杂交效率、信号特异性有不同的影响,所述探针的长度通常至少是14个碱基对,最长一般不超过30个碱基对,与目的核苷酸序列互补的长度以15-25个碱基对最佳。所述探针自身互补序列最好少于4个碱基对,以免影响杂交效率。
进一步,所述mylk蛋白的特异性抗体包括单克隆抗体、多克隆抗体。所述mylk蛋白的特异性抗体包括完整的抗体分子、抗体的任何片段或修饰(例如,,嵌合抗体、scfv、fab、f(ab’)2、fv等。只要所述片段能够保留与mylk蛋白的结合能力即可。用于蛋白质水平的抗体的制备时本领域技术人员公知的,并且本发明可以使用任何方法来制备所述抗体。
在本发明的具体实施方案中,所述针对mylk基因的引物序列如下:正向引物序列如seqidno.3所示,反向引物如seqidno.4所示。
用于诊断子宫内膜癌的mylk基因及其表达产物的来源包括但不限于组织和体液,体液包括血液、组织液等存在dna的体内液体成分。在本发明的具体实施方案中,用于诊断子宫内膜癌的mylk基因及其表达产物的来源是组织。
本发明的mylk基因(nc_000003.12(123612296..123884302))的具体序列可在国际公共核酸序列数据库genebank中查询到。
本发明的mylk基因的mrna序列相对应的dna序列是序列表中seqidno.1所示的dna序列。
本发明的“mylk蛋白”包括mylk蛋白以及mylk蛋白的任何功能等同物。所述功能等同物包括mylk蛋白保守性变异蛋白质、或其活性片段,或其活性衍生物,等位变异体、天然突变体、诱导突变体、在高或低的严紧条件下能与mylk的dna杂交的dna所编码的蛋白质。
优选地,mylk蛋白是具有下列氨基酸序列的蛋白质:
(1)由序列表中seqidno.2所示的氨基酸序列组成的蛋白质;
(2)将seqidno.2所示的氨基酸序列经过一个或几个氨基酸残基的取代和/或缺失和/或添加且与seqidno.2所示的氨基酸序列具有相同功能的由seqidno.2所示的氨基酸序列衍生的蛋白质。取代、缺失或者添加的氨基酸的个数通常为1-50个,较佳地1-30个,更佳地1-20个,最佳地1-10个。
在本发明的具体实施方案中,所述mylk蛋白是具有seqidno.2所示的氨基酸序列的蛋白质。
通常,已知的是,一个蛋白质中一个或多个氨基酸的修饰不会影响蛋白质的功能。本领域技术人员会认可改变单个氨基酸或小百分比的氨基酸或对氨基酸序列的个别添加、缺失、插入、替换是保守修饰,其中蛋白质的改变产生具有相似功能的蛋白质。提供功能相似的氨基酸的保守替换表是本领域公知的。
通过添加一个氨基酸或多个氨基酸残基修饰的蛋白质的例子是mylk蛋白的融合蛋白。对于与mylk蛋白融合的肽或者蛋白质没有限制,只要所得的融合蛋白保留mylk蛋白的生物学活性即可。
本发明的mylk蛋白也包括对seqidno.2所示的氨基酸序列的非保守修饰,只要经过修饰的蛋白质仍然能够保留mylk蛋白的生物学活性即可。在此类修饰蛋白质中突变的氨基酸数目通常是10个或者更少,例如6个或者更少,例如3个或者更少。
在本发明的上下文中,“诊断子宫内膜癌”既包括判断受试者是否已经患有子宫内膜癌、也包括判断受试者是否存在患有子宫内膜癌的风险,还包括预测子宫内膜癌患者的预后,还包括判断患有子宫内膜癌的受试者在经过治疗后是否已经复发。
附图说明
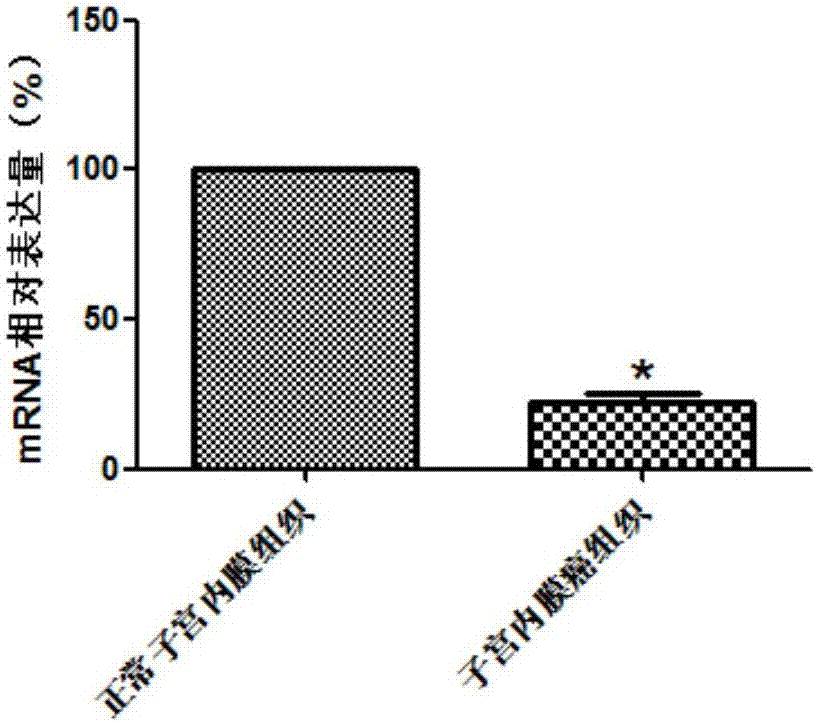
图1显示利用rna-seq检测mylk基因在子宫内膜癌组织和正常子宫内膜组织中的表达差异;
图2显示利用qpcr检测mylk基因在子宫内膜癌组织和正常子宫内膜组织中的表达差异。
具体的实施方式
下面结合附图和实施例对本发明作进一步详细的说明。以下实施例仅用于说明本发明而不用于限制本发明的范围。实施例中未注明具体条件的实验方法,通常按照常规条件,例如sambrook等人,分子克隆:实验室手册(newyork:coldspringharborlaboratorypress,1989)中所述的条件,或按照制造厂商所建议的条件。
实施例1筛选差异表达基因
1、样本收集:
子宫内膜癌组织样本:收集子宫内膜癌患者,全部患者均经手术治疗,手术石蜡标本10例。所有患者均经过病理检查确诊患有子宫内膜癌。其他入组条件为:所有患者入院前未接受过任何治疗:未合并其他恶性肿瘤;未合并其他激素相关性疾病;临床资料完整。
10例子宫内膜癌患者,发病平均年龄57岁。患者主要临床表现为阴道不规则出血、下腹痛、月经紊乱、阴道排液等,亦有部分患者无明显症状而于健康查体发现。子宫内膜癌标本经he染色切片,组织形态学确诊为子宫内膜癌。
正常子宫内膜组织样本:10例正常子宫内膜手术石蜡标本。样本来源的病人所患疾病包括:子宫肌瘤、子宫脱垂、膀胧膨出、直肠膨出。
2、rna提取
将子宫内膜组织分别放在陶瓷研钵中液氮低温环境下碾碎成粉末,加入trizol试剂匀浆,离心后取上清液经1∶1酸性酚-氯仿两次抽提后再经醋酸钠和5∶1酸性酚-氯仿抽提,等体积异丙醇沉淀,离心后用milli-q水溶解沉淀。
3、rna样品的纯度分析(nanodrop1000分光光度计)
nanodrop1000分光光度计检测rna样品,rna-seq测序的样品要求:od260/od280为1.8-2.2。
4、rna样品的质量分析(agilenttechnologies2100bioanalyzer)
agilenttechnologies2100bioanalyzer检测rna样品质量,观察28srrna和18srrna主带明显、无降解、rna完整性指数合格、浓度达到要求的符合rna-seq测序cdna文库构建的要求,可以用于文库构建及测序。
5、高通量转录组测序
(1)rna-seq读段定位
首先将低质量的读段去除得到清洁读段,然后利用tophatv1.3.1将清洁片段与ucsch.sapiens参考基因组(hg19)进行匹配,h.sapiensucschg19版的预先构建的索引从tophat主页下载,并作为参考基因组,利用tophat与基因组匹配时,允许每个读段(默认到20)有多个匹配位点,最多2次错配。tophat根据外显子区域和gt-ag剪切信号建立可能的剪切位点库,根据这些剪切位点库将没有定位到基因组的读段定位到基因组上。我们使用tophat方法的系统默认参数。
(2)转录丰度评估
匹配上的读段文件通过cufflinksv1.0.3处理,cufflinksv1.0.3将rna-seq片段数目进行标准化计算转录本的相对丰度。fpkm值指的是每一百万测序片段中匹配到特定基因1kb长的外显子区域的片段数目。通过贝叶斯推理方法计算fpkm估计值的置信区间。cufflinks使用的参考的gtf注释文件从ensembl数据库下载(homo_sapiens.grch37.63.gtf)。
(3)差异表达基因的检测
将下载的ensemblgtf文件和通过tophat匹配的原始文件传输到cuffdiff,cuffdiff使用原始的匹配文件重新估算gtf文件中列出的转录本的表达丰度,检测差异表达。在cuffidff输出中只有q值<0.01,测试显示成功的比较才被认为是差异表达。
6、结果
rna-seq结果显示,正常子宫内膜组织和子宫内膜癌组织之间存在差异表达基因,其中,上调的基因有374个,下调的基因有268个。其中,子宫内膜癌组织中mylk基因mrna水平显著降低,差异具有统计学意义(p<0.05)。
实施例2差异表达基因在大样本中的验证
1、样本收集
按照实施例1的方法收集子宫内膜癌组织50例,正常子宫内膜组织45例。
2、在mrna水平上进行验证
2.1提取组织rna
步骤同实施例1。
2.2逆转录
使用逆转录试剂盒,用逆转录缓冲液对lμg总rna进行逆反录合成cdna。采用25μl反应体系,每个样品取1μg总rna作为模板rna,在pcr管中分别加入以下组分:depc水,5×逆转录缓冲液,10mmol/ldntp,0.1mmol/ldtt,30μmmol/loligodt,200u/μlmmlvrt,模板rna。42℃孵育1小时,72℃10分钟,短暂离心。
2.3pcr
使用引物设计软件primerpremier5.0设计mrna荧光定量上下游pcr引物,合成引物序列,使用sybrgreenpcrmastermix试剂盒操作,具体步骤按说明书进行操作,采用25μl反应体系,每个样本设置3个平行管,所有扩增反应均重复三次以上以保证结果的可靠性。配制以下反应体系(如表1所示),各项操作均于冰上进行:
表1荧光定量pcr各组分及相应体积
以gapdh为内参,以sybrgreeni作为荧光标记物,在lightcycler荧光定量pcr仪上进行pcr反应,通过融解曲线分析和电泳确定目的条带,δδct法进行相对定量。
mylk基因引物序列如下所示:
上游引物:5’-aagtggaggtgtcagatg-3’(seqidno.3);
下游引物:5’-tcaatgtcgtagaagtcaga-3’(seqidno.4)。
gapdh基因引物序列如下所示:
上游引物:5’-aactctggtaaagtggatattg-3’(seqidno.5);
下游引物:5’-ggtggaatcatattggaaca-3’(seqidno.6)。
2.4结果
结果如图2所示,与正常子宫内膜组织相比,子宫内膜癌组织中mylk基因的mrna水平明显下调,差异具有统计学意义(p<0.05),结果同rna-seq。
实施例3子宫内膜癌诊断试剂盒的制备
根据mylk基因与子宫内膜癌的相关性,可以通过检测mylk基因的表达情况来诊断子宫内膜癌是否发生,据此本发明提供了一种基于检测mylk基因表达来诊断子宫内膜癌的试剂盒,该诊断试剂盒中的组分如下:sybrgreen聚合酶链式反应体系;扩增mylk基因和gapdh基因的引物对。扩增mylk基因的正向序列5’-aagtggaggtgtcagatg-3’,反向序列5’-tcaatgtcgtagaagtcaga-3’;扩增gapdh的正向引物序列为5’-aactctggtaaagtggatattg-3’,反向引物序列为5’-ggtggaatcatattggaaca-3’。sybrgreen聚合酶链式反应体系包含pcr缓冲液、dntps、sybrgreen荧光染料。pcr缓冲液成分为:25mmkcl,2.5mmmgcl2、200mm(nh4)2so4。
实施例4子宫内膜癌诊断试剂盒的制备
根据mylk基因与子宫内膜癌的相关性,可以通过检测mylk基因的表达情况来诊断子宫内膜癌是否发生,据此本发明提供了一种基于检测mylk基因表达来诊断子宫内膜癌的试剂盒,该诊断试剂盒中的组分如下:sybrgreen聚合酶链式反应体系、扩增mylk基因和gapdh基因的引物对、m-mlv逆转录体系。扩增mylk基因的正向序列5’-aagtggaggtgtcagatg-3’,反向序列5’-tcaatgtcgtagaagtcaga-3’;扩增gapdh的正向引物序列为5’-aactctggtaaagtggatattg-3’,反向引物序列为5’-ggtggaatcatattggaaca-3’。sybrgreen聚合酶链式反应体系包含pcr缓冲液、dntps、sybrgreen荧光染料。pcr缓冲液组分为:25mmkcl、2.5mmmgcl2、200mm(nh4)2so4。m-mlv逆转录体系组分为:t重复寡核苷酸oligo(dt)、逆转录反应液、m-mlv逆转录酶、rna酶抑制剂、dntps。逆转录反应液组分为:250mmtris-hcl(ph8.3)、375mmkcl、15mmmgcl2、50mm的dtt。rna酶抑制剂为大肠杆菌表达的非竞争性抑制rna酶的重组蛋白酶。
实施例5子宫内膜癌诊断试剂盒的制备
根据mylk基因与子宫内膜癌的相关性,可以通过检测mylk基因的表达情况来诊断子宫内膜癌是否发生,据此本发明提供了一种基于检测mylk基因表达来诊断子宫内膜癌的试剂盒,该诊断试剂盒中的组分如下:sybrgreen聚合酶链式反应体系、扩增mylk基因和gapdh基因的引物对、rna提取试剂。扩增mylk基因的正向序列5’-aagtggaggtgtcagatg-3’,反向序列5’-tcaatgtcgtagaagtcaga-3’;扩增gapdh的正向引物序列为5’-aactctggtaaagtggatattg-3’,反向引物序列为5’-ggtggaatcatattggaaca-3’。sybrgreen聚合酶链式反应体系包含pcr缓冲液、dntps、sybrgreen荧光染料。pcr缓冲液组分为:25mmkcl、2.5mmmgcl2、200mm(nh4)2so4。rna提取试剂包含trizol、氯仿、异丙醇、75%乙醇。
实施例6子宫内膜癌诊断试剂盒的制备
根据mylk基因与子宫内膜癌的相关性,可以通过检测mylk基因的表达情况来诊断子宫内膜癌是否发生,据此本发明提供了一种基于检测mylk基因表达来诊断子宫内膜癌的试剂盒,该诊断试剂盒中的组分如下:sybrgreen聚合酶链式反应体系、扩增mylk基因和gapdh基因的引物对、m-mlv逆转录体系、rna提取试剂。扩增mylk基因的正向序列5’-aagtggaggtgtcagatg-3’,反向序列5’-tcaatgtcgtagaagtcaga-3’;扩增gapdh的正向引物序列为5’-aactctggtaaagtggatattg-3’,反向引物序列为5’-ggtggaatcatattggaaca-3’。sybrgreen聚合酶链式反应体系包含pcr缓冲液、dntps、sybrgreen荧光染料。pcr缓冲液组分为:25mmkcl、2.5mmmgcl2、200mm(nh4)2so4。m-mlv逆转录体系组分为:t重复寡核苷酸oligo(dt)、逆转录反应液、m-mlv逆转录酶、rna酶抑制剂、dntps。逆转录反应液组分为:250mmtris-hcl(ph8.3)、375mmkcl、15mmmgcl2,50mmdtt。rna酶抑制剂为大肠杆菌表达的非竞争性抑制rna酶的重组蛋白酶。rna提取试剂包含trizol、氯仿、异丙醇、75%乙醇。
上述实施例的说明只是用于理解本发明的方法及其核心思想。应当指出,对于本领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以对本发明进行若干改进和修饰,这些改进和修饰也将落入本发明权利要求的保护范围内。
sequencelisting
<110>上海市长宁区妇幼保健院
<120>mylk基因作为诊断子宫内膜癌的标志物
<160>6
<170>patentinversion3.5
<210>1
<211>7644
<212>dna
<213>人源
<400>1
ggcgctgagcgagctcggagcccgcgctgtgcgcctgcggccggggcgccccgccgagcg60
ccggtgccccggctcccgggccgccttcgccgcgcgggaaggattcttcaaaattaacag120
aaaccaattcgggccagctgaagagaaaaaataaaggtggctcccggctgcctctgctgc180
agttcagagcaacttcaggagcttcccagccgagagcttcaggacgcctttcctgtccca240
ctggcccagttgccacaacaaacaacagagaagacggtgaccatgggggatgtgaagctg300
gttgcctcgtcacacatttccaaaacctccctcagtgtggatccctcaagagttgactcc360
atgcccctgacagaggcccctgctttcattttgccccctcggaacctctgcatcaaagaa420
ggagccaccgccaagttcgaagggcgggaagttttgcgaagcagcttggtcagcctgttg480
tttccaaaaccttaggggatagattttcagctccagcagtggagacccgtcctagcatct540
ggggggagtgcccaccaaagtttgctaccaagctgggccgagttgtggtcaaagaaggac600
agatgggacgattctcctgcaagatcactggccggccccaaccgcaggtcacctggctca660
agggaaatgttccactgcagccgagtgcccgtgtgtctgtgtctgagaagaacggcatgc720
aggttctggaaatccatggagtcaaccaagatgacgtgggagtgtacacgtgcctggtgg780
tgaacgggtcggggaaggcctcgatgtcagctgaactttccatccaaggtttggacagtg840
ccaataggtcatttgtgagagaaacaaaagccaccaattcagatgtcaggaaagaggtga900
ccaatgtaatctcaaaggagtcgaagctggacagtctggaggctgcagccaaaagcaaga960
actgctccagcccccagagaggtggctccccaccctgggctgcaaacagccagcctcagc1020
ccccaagggagtccaagctggagtcatgcaaggactcgcccagaacggccccgcagaccc1080
cggtccttcagaagacttccagctccatcaccctgcaggccgcaagagttcagccggaac1140
caagagcaccaggcctgggggtcctatcaccttctggagaagagaggaagaggccagctc1200
ctccccgtccagccaccttccccaccaggcagcctggcctggggagccaagatgttgtga1260
gcaaggctgctaacaggagaatccccatggagggccagagggattcagcattccccaaat1320
ttgagagcaagccccaaagccaggaggtcaaggaaaatcaaactgtcaagttcagatgtg1380
aagtttccgggattccaaagcctgaagtggcctggttcctggaaggcacccccgtgagga1440
gacaggaaggcagcattgaggtttatgaagatgctggctcccattacctctgcctgctga1500
aagcccggaccagggacagtgggacatacagctgcactgcttccaacgcccaaggccagc1560
tgtcctgtagctggaccctccaagtggaaaggcttgccgtgatggaggtggccccctcct1620
tctccagtgtcctgaaggactgcgctgttattgagggccaggattttgtgctgcagtgct1680
ccgtacgggggaccccagtgccccggatcacttggctgctgaatgggcagcccatccagt1740
acgctcgctccacctgcgaggccggcgtggctgagctccacatccaggatgccctgccgg1800
aggaccatggcacctacacctgcctagctgagaatgccttggggcaggtgtcctgcagcg1860
cctgggtcaccgtccatgaaaagaagagtagcaggaagagtgagtaccttctgcctgtgg1920
ctcccagcaagcccactgcacccatcttcctgcagggcctctctgatctcaaagtcatgg1980
atggaagccaggtcactatgactgtccaagtgtcagggaatccaccccctgaagtcatct2040
ggctgcacaatgggaatgagatccaagagtcagaggacttccactttgaacagagaggaa2100
ctcagcacagcctttgtatccaggaagtgttcccggaggacacgggcacgtacacctgcg2160
aggcctggaacagcgctggagaggtccgcacccaggccgtgctcacggtacaagagcctc2220
acgatggcacccagccctggttcatcagtaagcctcgctcagtgacagcctccctgggcc2280
agagtgtcctcatctcctgcgccatagctggtgacccctttcctaccgtgcactggctca2340
gagatggcaaagccctctgcaaagacactggccacttcgaggtgcttcagaatgaggacg2400
tgttcaccctggttctaaagaaggtgcagccctggcatgccggccagtatgagatcctgc2460
tcaagaaccgggttggcgaatgcagttgccaggtgtcactgatgctacagaacagctctg2520
ccagagcccttccacgggggagggagcctgccagctgcgaggacctctgtggtggaggag2580
ttggtgctgatggtggtggtagtgaccgctatgggtccctgaggcctggctggccagcaa2640
gagggcagggttggctagaggaggaagacggcgaggacgtgcgaggggtgctgaagaggc2700
gcgtggagacgaggcagcacactgaggaggcgatccgccagcaggaggtggagcagctgg2760
acttccgagacctcctggggaagaaggtgagtacaaagaccctatcggaagacgacctga2820
aggagatcccagccgagcagatggatttccgtgccaacctgcagcggcaagtgaagccaa2880
agactgtgtctgaggaagagaggaaggtgcacagcccccagcaggtcgattttcgctctg2940
tcctggccaagaaggggacttccaagacccccgtgcctgagaaggtgccaccgccaaaac3000
ctgccaccccggattttcgctcagtgctgggtggcaagaagaaattaccagcagagaatg3060
gcagcagcagtgccgagaccctgaatgccaaggcagtggagagttccaagcccctgagca3120
atgcacagccttcagggcccttgaaacccgtgggcaacgccaagcctgctgagaccctga3180
agccaatgggcaacgccaagcctgccgagaccctgaagcccatgggcaatgccaagcctg3240
atgagaacctgaaatccgctagcaaagaagaactcaagaaagacgttaagaatgatgtga3300
actgcaagagaggccatgcagggaccacagataatgaaaagagatcagagagccagggga3360
cagccccagccttcaagcagaagctgcaagatgttcatgtggcagagggcaagaagctgc3420
tgctccagtgccaggtgtcttctgaccccccagccaccatcatctggacgctgaacggaa3480
agaccctcaagaccaccaagttcatcatcctctcccaggaaggctcactctgctccgtct3540
ccatcgagaaggcactgcctgaggacagaggcttatacaagtgtgtagccaagaatgacg3600
ctggccaggcggagtgctcctgccaagtcaccgtggatgatgctccagccagtgagaaca3660
ccaaggccccagagatgaaatcccggaggcccaagagctctcttcctcccgtgctaggaa3720
ctgagagtgatgcgactgtgaaaaagaaacctgcccccaagacacctccgaaggcagcaa3780
tgccccctcagatcatccagttccctgaggaccagaaggtacgcgcaggagagtcagtgg3840
agctgtttggcaaagtgacaggcactcagcccatcacctgtacctggatgaagttccgaa3900
agcagatccaggaaagcgagcacatgaaggtggagaacagcgagaatggcagcaagctca3960
ccatcctggccgcgcgccaggagcactgcggctgctacacactgctggtggagaacaagc4020
tgggcagcaggcaggcccaggtcaacctcactgtcgtggataagccagaccccccagctg4080
gcacaccttgtgcctctgacattcggagctcctcactgaccctgtcctggtatggctcct4140
catatgatgggggcagtgctgtacagtcctacagcatcgagatctgggactcagccaaca4200
agacgtggaaggaactagccacatgccgcagcacctctttcaacgtccaggacctgctgc4260
ctgaccacgaatataagttccgtgtacgtgcaatcaacgtgtatggaaccagtgagccaa4320
gccaggagtctgaactcacaacggtaggagagaaacctgaagagccgaaggatgaagtgg4380
aggtgtcagatgatgatgagaaggagcccgaggttgattaccggacagtgacaatcaata4440
ctgaacaaaaagtatctgacttctacgacattgaggagagattaggatctgggaaatttg4500
gacaggtctttcgacttgtagaaaagaaaactcgaaaagtctgggcagggaagttcttca4560
aggcatattcagcaaaagagaaagagaatatccggcaggagattagcatcatgaactgcc4620
tccaccaccctaagctggtccagtgtgtggatgcctttgaagaaaaggccaacatcgtca4680
tggtcctggagatcgtgtcaggaggggagctgtttgagcgcatcattgacgaggactttg4740
agctgacggagcgtgagtgcatcaagtacatgcggcagatctcggagggagtggagtaca4800
tccacaagcagggcatcgtgcacctggacctcaagccggagaacatcatgtgtgtcaaca4860
agacgggcaccaggatcaagctcatcgactttggtctggccaggaggctggagaatgcgg4920
ggtctctgaaggtcctctttggcaccccagaatttgtggctcctgaagtgatcaactatg4980
agcccatcggctacgccacagacatgtggagcatcggggtcatctgctacatcctagtca5040
gtggcctttcccccttcatgggagacaacgataacgaaaccttggccaacgttacctcag5100
ccacctgggacttcgacgacgaggcattcgatgagatctccgacgatgccaaggatttca5160
tcagcaatctgctgaagaaagatatgaaaaaccgcctggactgcacgcagtgccttcagc5220
atccatggctaatgaaagataccaagaacatggaggccaagaaactctccaaggaccgga5280
tgaagaagtacatggcaagaaggaaatggcagaaaacgggcaatgctgtgagagccattg5340
gaagactgtcctctatggcaatgatctcagggctcagtggcaggaaatcctcaacagggt5400
caccaaccagcccgctcaatgcagaaaaactagaatctgaagaagatgtgtcccaagctt5460
tccttgaggctgttgctgaggaaaagcctcatgtaaaaccctatttctctaagaccattc5520
gcgatttagaagttgtggagggaagtgctgctagatttgactgcaagattgaaggatacc5580
cagaccccgaggttgtctggttcaaagatgaccagtcaatcagggagtcccgccacttcc5640
agatagactacgatgaggacgggaactgctctttaattattagtgatgtttgcggggatg5700
acgatgccaagtacacctgcaaggctgtcaacagtcttggagaagccacctgcacagcag5760
agctcattgtggaaacgatggaggaaggtgaaggggaaggggaagaggaagaagagtgaa5820
acaaagccagagaaaagcagtttctaagtcatattaaaaggactatttctctaaaactca5880
aaaaaaaaaaaaaaactcaagatagtaaaagcacctagtgtgatagattatcggttaggt5940
catttgtgggttgattcttcagaaacagcagttgatacctagcagcgttattgatgggca6000
ttaatctatgttagttggcaccttaagatactagtgcagctagatttcatttagggaaat6060
caccagtaacttgactgaccaattgattttagagagaaagtaaccaaaccaaatatttat6120
ctgggcaaagtcataaattctccacttgaatgcgctcatgaaaaataaggccaaaacaag6180
agttctgggccacagctcagcccagagggttcctggggatgggaggcctctctctcccca6240
ccccctgactctagagaactgggttttctcccagtactccagcaattcatttctgaaagc6300
agttgagccactttattccaaagtacactgcagatgttcaaactctccatttctctttcc6360
ccttccacctgccagttttgctgactctcaacttgtcatgagtgtaagcattaaggacat6420
tatgcttcttcgattctgaagacaggtccctgctcatggatgactctggcttccttagga6480
aaatatttttcttccaaaatcagtaggaaatctaaacttatcccctctttgcagatgtct6540
agcagcttcagacatttggttaagaacccatgggaaaaaaaaaatccttgctaatgtggt6600
ttcctttgtaaaccaggattcttatttgtgctgttatagaatatcagctctgaacgtgtg6660
gtaaagatttttgtgtttgaatataggagaaatcagtttgctgaaaagttagtcttaatt6720
atctattggccacgatgaaacagatttcaactgataaagagctggagaactccatgtact6780
ttggaatctcctccaagatagccagagtttaatacatcttcattctcaacactctccaaa6840
gaacttgacctaccttatgggttccatatttttcttcttaaatgtgcatcaatcatgcct6900
tgcccccaacctttaaatatattcttagacctggtaaatgcactcagacttgcgtcttta6960
ggaatttttaactttctttcactacattggcacttaaattttttctttataaagcttttt7020
gaaggtcataaacaaagaccataattgatgatagacctaatacatttcctctgtgtgtgt7080
gtgtaacattccaaatactttttttttcttttccactgtttgtaaggtgcaacaatttaa7140
tatttttaagggactttttaagagttccttaagaaccaatttaaaattacttcagtgcaa7200
tcctacacagtatcaacattagaattttgatattagtcttatgttatcttccattctatt7260
tttatctgctttttgctgctagtttcaaactgccagtatttttccttttgcttttaaaat7320
agttacaatatttttcatgatagccacagtattgccacagtttattataataaagggttt7380
ttatttgatttagcgcattcaaagcttttttctatcacttttgtgttcagaatataacct7440
ttgtgtgcgtgtatgttgtgtgtgtgcatgtgtggcgtatatgtgtgttacaggttaatg7500
ccttcttggaattgtgttaatgttctcttggtttattatgccatcagaatggtaaatgag7560
aacactacaactgtagtcagctcacaatttttaaataaaggataccacagtgcatgctgt7620
ttgttcaaaaaaaaaaaaaaaaaa7644
<210>2
<211>1738
<212>prt
<213>人源
<400>2
metglyargphesercyslysilethrglyargproglnproglnval
151015
thrtrpleulysglyasnvalproleuglnproseralaargvalser
202530
valserglulysasnglymetglnvalleugluilehisglyvalasn
354045
glnaspaspvalglyvaltyrthrcysleuvalvalasnglysergly
505560
lysalasermetseralagluleuserileglnglyleuaspserala
65707580
asnargserphevalarggluthrlysalathrasnseraspvalarg
859095
lysgluvalthrasnvalileserlysgluserlysleuaspserleu
100105110
glualaalaalalysserlysasncysserserproglnargglygly
115120125
serproprotrpalaalaasnserglnproglnproproarggluser
130135140
lysleuglusercyslysaspserproargthralaproglnthrpro
145150155160
valleuglnlysthrserserserilethrleuglnalaalaargval
165170175
glnprogluproargalaproglyleuglyvalleuserprosergly
180185190
glugluarglysargproalaproproargproalathrpheprothr
195200205
argglnproglyleuglyserglnaspvalvalserlysalaalaasn
210215220
argargileprometgluglyglnargaspseralapheprolysphe
225230235240
gluserlysproglnserglngluvallysgluasnglnthrvallys
245250255
pheargcysgluvalserglyileprolysprogluvalalatrpphe
260265270
leugluglythrprovalargargglngluglyserilegluvaltyr
275280285
gluaspalaglyserhistyrleucysleuleulysalaargthrarg
290295300
aspserglythrtyrsercysthralaserasnalaglnglyglnleu
305310315320
sercyssertrpthrleuglnvalgluargleualavalmetgluval
325330335
alaproserpheserservalleulysaspcysalavalileglugly
340345350
glnaspphevalleuglncysservalargglythrprovalproarg
355360365
ilethrtrpleuleuasnglyglnproileglntyralaargserthr
370375380
cysglualaglyvalalagluleuhisileglnaspalaleuproglu
385390395400
asphisglythrtyrthrcysleualagluasnalaleuglyglnval
405410415
sercysseralatrpvalthrvalhisglulyslysserserarglys
420425430
serglutyrleuleuprovalalaproserlysprothralaproile
435440445
pheleuglnglyleuseraspleulysvalmetaspglyserglnval
450455460
thrmetthrvalglnvalserglyasnproproprogluvaliletrp
465470475480
leuhisasnglyasngluileglnglusergluaspphehispheglu
485490495
glnargglythrglnhisserleucysileglngluvalpheproglu
500505510
aspthrglythrtyrthrcysglualatrpasnseralaglygluval
515520525
argthrglnalavalleuthrvalglngluprohisaspglythrgln
530535540
protrppheileserlysproargservalthralaserleuglygln
545550555560
servalleuilesercysalailealaglyasppropheprothrval
565570575
histrpleuargaspglylysalaleucyslysaspthrglyhisphe
580585590
gluvalleuglnasngluaspvalphethrleuvalleulyslysval
595600605
glnprotrphisalaglyglntyrgluileleuleulysasnargval
610615620
glyglucyssercysglnvalserleumetleuglnasnserserala
625630635640
argalaleuproargglyarggluproalasercysgluaspleucys
645650655
glyglyglyvalglyalaaspglyglyglyseraspargtyrglyser
660665670
leuargproglytrpproalaargglyglnglytrpleuglugluglu
675680685
aspglygluaspvalargglyvalleulysargargvalgluthrarg
690695700
glnhisthrgluglualaileargglnglngluvalgluglnleuasp
705710715720
pheargaspleuleuglylyslysvalserthrlysthrleuserglu
725730735
aspaspleulysgluileproalagluglnmetasppheargalaasn
740745750
leuglnargglnvallysprolysthrvalsergluglugluarglys
755760765
valhisserproglnglnvalasppheargservalleualalyslys
770775780
glythrserlysthrprovalproglulysvalproproprolyspro
785790795800
alathrproasppheargservalleuglyglylyslyslysleupro
805810815
alagluasnglyserserseralagluthrleuasnalalysalaval
820825830
gluserserlysproleuserasnalaglnproserglyproleulys
835840845
provalglyasnalalysproalagluthrleulysprometglyasn
850855860
alalysproalagluthrleulysprometglyasnalalysproasp
865870875880
gluasnleulysseralaserlysglugluleulyslysaspvallys
885890895
asnaspvalasncyslysargglyhisalaglythrthraspasnglu
900905910
lysargsergluserglnglythralaproalaphelysglnlysleu
915920925
glnaspvalhisvalalagluglylyslysleuleuleuglncysgln
930935940
valserseraspproproalathrileiletrpthrleuasnglylys
945950955960
thrleulysthrthrlyspheileileleuserglngluglyserleu
965970975
cysservalserileglulysalaleuprogluaspargglyleutyr
980985990
lyscysvalalalysasnaspalaglyglnalaglucyssercysgln
99510001005
valthrvalaspaspalaproalasergluasnthrlysalapro
101010151020
glumetlysserargargprolysserserleuproprovalleu
102510301035
glythrgluseraspalathrvallyslyslysproalaprolys
104010451050
thrproprolysalaalametproproglnileileglnphepro
105510601065
gluaspglnlysvalargalaglygluservalgluleuphegly
107010751080
lysvalthrglythrglnproilethrcysthrtrpmetlysphe
108510901095
arglysglnileglnglusergluhismetlysvalgluasnser
110011051110
gluasnglyserlysleuthrileleualaalaargglngluhis
111511201125
cysglycystyrthrleuleuvalgluasnlysleuglyserarg
113011351140
glnalaglnvalasnleuthrvalvalasplysproasppropro
114511501155
alaglythrprocysalaseraspileargserserserleuthr
116011651170
leusertrptyrglysersertyraspglyglyseralavalgln
117511801185
sertyrserilegluiletrpaspseralaasnlysthrtrplys
119011951200
gluleualathrcysargserthrserpheasnvalglnaspleu
120512101215
leuproasphisglutyrlyspheargvalargalaileasnval
122012251230
tyrglythrsergluproserglnglusergluleuthrthrval
123512401245
glyglulysproglugluprolysaspgluvalgluvalserasp
125012551260
aspaspglulysgluprogluvalasptyrargthrvalthrile
126512701275
asnthrgluglnlysvalseraspphetyraspileglugluarg
128012851290
leuglyserglylyspheglyglnvalpheargleuvalglulys
129513001305
lysthrarglysvaltrpalaglylysphephelysalatyrser
131013151320
alalysglulysgluasnileargglngluileserilemetasn
132513301335
cysleuhishisprolysleuvalglncysvalaspalapheglu
134013451350
glulysalaasnilevalmetvalleugluilevalserglygly
135513601365
gluleuphegluargileileaspgluaspphegluleuthrglu
137013751380
argglucysilelystyrmetargglnilesergluglyvalglu
138513901395
tyrilehislysglnglyilevalhisleuaspleulysproglu
140014051410
asnilemetcysvalasnlysthrglythrargilelysleuile
141514201425
asppheglyleualaargargleugluasnalaglyserleulys
143014351440
valleupheglythrprogluphevalalaprogluvalileasn
144514501455
tyrgluproileglytyralathraspmettrpserileglyval
146014651470
ilecystyrileleuvalserglyleuserprophemetglyasp
147514801485
asnaspasngluthrleualaasnvalthrseralathrtrpasp
149014951500
pheaspaspglualapheaspgluileseraspaspalalysasp
150515101515
pheileserasnleuleulyslysaspmetlysasnargleuasp
152015251530
cysthrglncysleuglnhisprotrpleumetlysaspthrlys
153515401545
asnmetglualalyslysleuserlysaspargmetlyslystyr
155015551560
metalaargarglystrpglnlysthrglyasnalavalargala
156515701575
ileglyargleusersermetalametileserglyleusergly
158015851590
arglysserserthrglyserprothrserproleuasnalaglu
159516001605
lysleugluserglugluaspvalserglnalapheleugluala
161016151620
valalagluglulysprohisvallysprotyrpheserlysthr
162516301635
ileargaspleugluvalvalgluglyseralaalaargpheasp
164016451650
cyslysilegluglytyrproaspprogluvalvaltrpphelys
165516601665
aspaspglnserilearggluserarghispheglnileasptyr
167016751680
aspgluaspglyasncysserleuileileseraspvalcysgly
168516901695
aspaspaspalalystyrthrcyslysalavalasnserleugly
170017051710
glualathrcysthralagluleuilevalgluthrmetgluglu
171517201725
glygluglygluglyglugluglugluglu
17301735
<210>3
<211>18
<212>dna
<213>人工序列
<400>3
aagtggaggtgtcagatg18
<210>4
<211>20
<212>dna
<213>人工序列
<400>4
tcaatgtcgtagaagtcaga20
<210>5
<211>22
<212>dna
<213>人工序列
<400>5
aactctggtaaagtggatattg22
<210>6
<211>20
<212>dna
<213>人工序列
<400>6
ggtggaatcatattggaaca20
- 还没有人留言评论。精彩留言会获得点赞!