一种原位多重核酸检测方法与流程
本发明属于分子生物学
技术领域:
,具体涉及一种原位多重核酸检测方法。
背景技术:
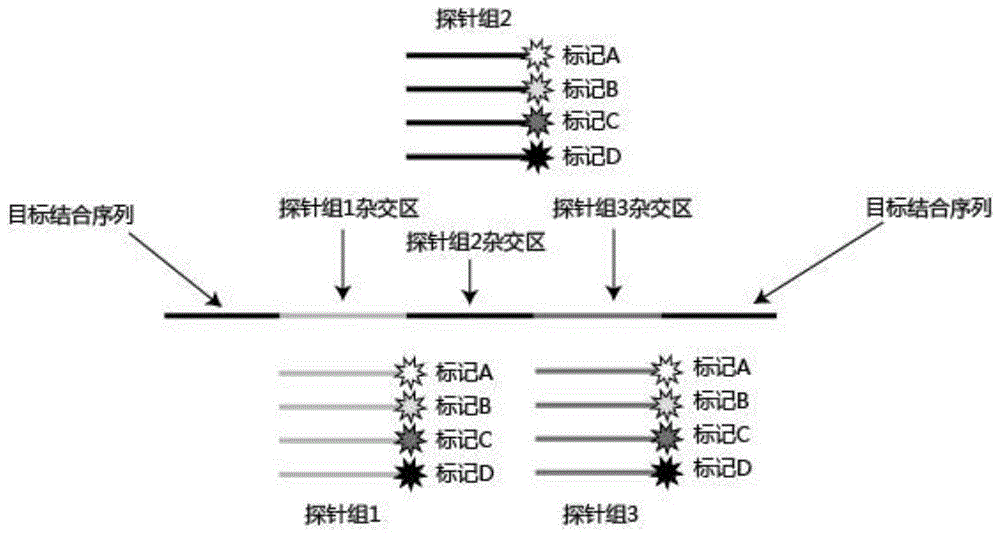
:原位杂交技术是带有标记的检测探针与目标核酸序列通过碱基互补配对实现异杂交,未结合的检测探针被洗去,之后对结合到目标核酸序列上的检测探针进行检测,从而实现对目标核酸的定性、定位和定量等原位检测。原位检测依赖于对检测探针的标记的信号进行检测。目前,检测探针的标记主要有荧光标记和生物酶标记两大类。前者一般是能够产生荧光的化学分子,经过共价交联结合到检测探针上面,进而对荧光信号进行检测;后者则是通过共价交联将具有催化活性的酶分子标记在检测探针上面,通过酶催化底物产生有颜色的产物进行检测。对于荧光标记检测探针来说,利用光谱特征不同的荧光信号,可以实现同时对不同的目标核酸序列进行同时标记检测。而对于生物酶标记检测探针来说,可以通过标记不同的生物酶,催化底物产生不同的颜色来实现多重检测。以上提到的基于传统的检测的原位杂交核酸检测方法由于标记物种类的限制,只能实现同时对几种不同的目标核酸序列进行检测。为了实现更多重检测,可以将同一目标核酸序列同时利用不同荧光标记的检测探针同时杂交,不同组合的荧光标记可以检测更多的目标核酸序列。近年来,以原位测序技术为代表的多重原位rna检测技术,通过多轮的检测,能够实现对同一个目标核酸分子进行编码检测,不同的编码代表检测到不同的目标核酸种类,从而可以实现高度多重的rna原位检测。原位测序技术主要是利用带有碱基编码的锁式探针与目标基因的cdna结合连接成环,之后通过滚环扩增进行信号放大,通过边连接边测序的方法,对探针上的碱基编码进行测序,得到的不同碱基编码标签即代表检测到了不同的rna分子。与原位测序类似的技术还有merfish,多重杂交链式反应(hcr)技术等。merfish是一种能够实现高度多重原位rna检测技术的单分子荧光原位杂交技术,该方法利用多个探针对同一个rna分子多轮杂交,得到一个二进制的编码标签,不同的标签表示不同的rna分子,从而实现高度多重rna原位检测。多重杂交链式反应是通过在同一个rna分子上进行多轮的hcr反应,每次hcr反应可以带有一种荧光标记,几轮反应之后得到一个有顺序的颜色编码标签,从而实现多重rna原位检测。技术实现要素:本发明的目的在于提供一种原位多重核酸检测方法。本发明的技术方案如下:一种原位多重核酸检测方法,包括如下步骤:(1)设计至少一成环探针,至少一成环探针的3’端和5’端的序列与待测样品中的至少一靶序列特异杂交互补,每一成环探针的3’端和5’端之间均包括不同的至少二探针杂交序列,每一探针杂交序列均可以与不同的至少二检测探针特异杂交且每次仅与一检测探针特异杂交;(2)将上述至少一成环探针与待测样品中的至少一靶序列特异结合后,该至少一成环探针的3’端和5’端首尾相邻,通过dna连接酶将其连接形成至少一环状模板;(3)对上述至少一环状模板进行滚环扩增,得到至少一滚环扩增产物;(4)将上述至少一滚环扩增产物与一检测探针进行杂交以进行检测,获得一信号;(5)去除上述检测探针产生的信号,再与不同的另一检测探针进行杂交以进行检测,获得另一信号;重复该步骤,且该重复的过程中的检测探针各不相同;(6)将步骤(4)和(5)获得的信号顺序排列,获得特异对应上述至少一靶序列的至少一信号编码。在本发明的一个优选实施方案中,所述成环探针包括锁式探针、双连接探针和倒置探针。进一步优选的,所述成环探针中含有自然和/或非自然核苷酸。在本发明的一个优选实施方案中,所述检测探针通过修饰不同的荧光标记物、显色标记物、酶标记物、放射性标记物、磁性标记物或发光密度标记物以进行信号的区分。在本发明的一个优选实施方案中,所述至少一靶序列为dna、rna或rna通过逆转录产生的cdna。在本发明的一个优选实施方案中,所述待测样品包括培养的细胞、组织整体中的细胞和组织切片中的细胞。在本发明的一个优选实施方案中,所述步骤(5)中的去除的方法包括酶消化、物理化学反应和光漂白。本发明的有益效果是:本发明能够有效的实现对目标核酸的进行多重检测,并可以实现高度多重检测,特异性好。附图说明图1为本发明实施例1中的带有不同检测探针组合的成环探针设计结构示意图。图2为本发明实施例1的滚环扩增产物生产的原理示意图。图3为本发明实施例1中检测探针多轮杂交后判定不同基因扩增产物的原理示意图。图4为本发明实施例1的检测结果图:经过三轮检测,可以达到同时检测多种基因。图5为本发明实施例1的检测结果分析图:21个基因表达情况如图所示,其中的白色小点为检测到的基因信号。具体实施方式以下通过具体实施方式结合附图对本发明的技术方案进行进一步的说明和描述。实施例1人皮肤成纤维细胞为实验样品考察双连接探针法原位检测细胞actbmrna。(一)细胞培养与固定:乳腺癌细胞系sk-br-3经dmem(含10%fbs)培养24-48h后,用胰蛋白酶处理成悬浮细胞,并种于无菌载玻片上,重新培养12-24h。depc-pbs冲洗,3×3min;用depc-pbs配置的3%pfa常温固定30min;再次用depc-pbs冲洗一次后,分别经梯度乙醇:70%、85%、100%脱水处理,各5min。空气晾干。(二)原位核酸检测,如图1至图3所示,具体包括如下步骤:(1)锁式探针杂交对乳腺癌细胞系sk-br-3的细胞膜进行透膜打孔,在样本上加入0.1mhcl室温下孵育5min。用depc-pbs-tween20洗三次后进行后续反应。加入还有终浓度为2xssc,20%甲酰胺和0.1μm各个锁式探针的杂交混合液,37℃下孵育3小时,使锁式探针的目标结合序列充分与其靶基因上的目标序列充分杂交。上述锁式探针如下表所示:(2)探针连接成环在样品加入50μl含有终浓度为1×splintrbuffer(neb)、0.1u/μlsplintrligase(neb)、1u/μlribolockrna酶抑制剂(fermantas)、和50%甘油连接反应混合液,37℃下孵育2小时。使锁式探针在rna模板介导下连接成环形dna模板。反应完成后用depc-pbs-tween20洗三次。(3)滚环扩增对上述环形dna模板通过滚环扩增,得到滚环扩增产物,具体的:用depc-pbs-tween20洗三次,在样本中加入50uldepc处理过的水中含有终浓度为1×的phi29buffer(fermantas)、1u/μlphi29聚合酶(fermantas)、0.25mmdntp、0.2mg/mlbsa以及5%甘油的滚环扩增反应混合液,反应条件为37℃下扩增2-3h或室温下过夜;(4)检测探针杂交和图像获取将上述滚环扩增产物与第一组检测探针杂交,最后用dapi进行细胞核染色,进行检测,具体的:将样本用depc-pbs-tween20洗三次后加入含有0.1μm检测探针、20%甲酰胺的2xssc缓冲液,37℃下孵育30分钟后,分别经梯度乙醇:70%、85%、100%脱水处理,各5min。空气晾干。最后加入含有100ng/mldapi的goldantifademountan封片剂(fermantas)室温下孵育10min后进行荧光显微镜检测并拍照。检测结果如图4i所示。上述第一组检测探针如下表所示:(均为dna,其中u来自于dutp(2’-deoxyuridine-5’-triphosphate,2’-脱氧尿苷-5’-三磷酸))序号名称序列5’标记seqidno22do-1rugcgucuauuuaguggagcccy3seqidno23do-1gucgcgcuugguauaaucgcufamseqidno24do-1baguagccgugacuaucgacucy5seqidno25do-1pcagugaaugcgaguccgucutexasred(5)第一组检测探针的消除将上述拍照成像后的载玻片置入70%乙醇中孵育,洗去盖玻片和封片剂。之后分别经梯度乙醇:70%、85%、100%脱水处理,各5min。空气晾干。在反应区加入终浓度为1×ung缓冲液(neb),0.1u/μlung(neb)和0.2μg/μlbsa的ung处理液,室温孵育2小时。用depc-pbs-tween20洗三次后,用80%甲酰胺孵育5min,重复三次。最后再用depc-pbs-tween20洗三次。去除消化并去除第一轮杂交的检测探针。(6)检测探针杂交和图像获取将上述去除了第一组检测探针的滚环扩增产物与第二组检测探针杂交,最后用dapi进行细胞核染色,进行检测,具体的:将样本用depc-pbs-tween20洗三次后加入含有0.1μm检测探针、20%甲酰胺的2×ssc缓冲液,37℃下孵育30分钟后,分别经梯度乙醇:70%、85%、100%脱水处理,各5min。空气晾干。最后加入含有100ng/mldapi的goldantifademountan封片剂(fermantas)室温下孵育10min后进行荧光显微镜检测并拍照。检测结果如图4ii所示。上述第二组检测探针如下表所示:(均为dna,其中u来自于dutp(2’-deoxyuridine-5’-triphosphate,2’-脱氧尿苷-5’-三磷酸))序号名称序列5’标记seqdno26do-2rgggccuuauuccggugcuaucy3seqdno27do-2gcuggaccuuaaucgugugcgfamseqidno28do-2bagcagcgaaccauacgugaccy5seqdno29do-2pgagaggccgucgcuauacautexasred(7)第二组检测探针的消除将上述拍照成像后的载玻片置入70%乙醇中孵育,洗去盖玻片和封片剂。之后分别经梯度乙醇:70%、85%、100%脱水处理,各5min。空气晾干。在反应区加入终浓度为1×ung缓冲液(neb),0.1u/μlung(neb)和0.2μg/μlbsa的ung处理液,室温孵育2小时。用depc-pbs-tween20洗三次后,用80%甲酰胺孵育5min,重复三次。最后再用depc-pbs-tween20洗三次。去除消化并去除第二轮杂交的检测探针。(8)第三轮检测探针杂交和图像获取将上述滚环扩增产物与第三组检测探针杂交,最后用dapi进行细胞核染色,进行检测,具体的:将样本用depc-pbs-tween20洗三次后加入含有0.1μm检测探针、20%甲酰胺的2×ssc缓冲液,37℃下孵育30分钟后,分别经梯度乙醇:70%、85%、100%脱水处理,各5min。空气晾干。最后加入含有100ng/mldapi的goldantifademountan封片剂(fermantas)室温下孵育10min后进行荧光显微镜检测并拍照。检测结果如图4iii所示。上述第三组检测探针如下表所示:(均为dna,其中u来自于dutp(2’-deoxyuridine-5’-triphosphate,2’-脱氧尿苷-5’-三磷酸))(9)基因的解析综合三轮检测探针杂交的信号,每个点得到一串信号,对应之前设计的探针信号顺序可以得知该点检测的为何种基因,如图5所示。以上所述,仅为本发明的较佳实施例而已,故不能依此限定本发明实施的范围,即依本发明专利范围及说明书内容所作的等效变化与修饰,皆应仍属本发明涵盖的范围内。序列表<110>华侨大学<120>一种原位多重核酸检测方法<160>33<170>siposequencelisting1.0<210>1<211>96<212>dna<213>artificalsequence<400>1ttggtgggcaggtaggtgcagtgaatgcgagtccgtctgggccttattccggtgctatgt60atctgcttatgtcgcccgaggaaggacaggctggca96<210>2<211>96<212>dna<213>artificalsequence<400>2aagagcaagttaggagcaagtagccgtgactatcgactgagaggccgtcgctatacatct60agtgctggatgatcgtcctttccctggttcctgtcc96<210>3<211>96<212>dna<213>artificalsequence<400>3tcccttaagcagactgactcgcgcttggtataatcgctagcagcgaaccatacgtgacgt60atctgcttatgtcgcccggcaaatccaaagttttct96<210>4<211>93<212>dna<213>artificalsequence<400>4tccctcacttctcaccttgcgtctatttagtggagccctggaccttaatcgtgtgcgaag60ctgatctaagccgcgaggacactgccttcttcc93<210>5<211>92<212>dna<213>artificalsequence<400>5tccaattcgtaggtcacagtgaatgcgagtccgtctgggccttattccggtgctatagcg60atctgcgagaccgtatggcctacatcttcatc92<210>6<211>90<212>dna<213>artificalsequence<400>6ttaaaagctgtggggtgcgtctatttagtggagccctggaccttaatcgtgtgcgctagt60gctggatgatcgtcctcttgcaggtacttc90<210>7<211>92<212>dna<213>artificalsequence<400>7agtgcaccatgtcatatgcgtctatttagtggagccagcagcgaaccatacgtgacctag60tgctggatgatcgtccatttgagaaggaggaa92<210>8<211>92<212>dna<213>artificalsequence<400>8aggcatgatagcgaagtcgcgcttggtataatcgctgagaggccgtcgctatacatagcg60atctgcgagaccgtatagacccagatgaacca92<210>9<211>92<212>dna<213>artificalsequence<400>9ggtggagtggtacagacagtgaatgcgagtccgtctctggaccttaatcgtgtgcgctag60tgctggatgatcgtcccaaaggtagggatata92<210>10<211>92<212>dna<213>artificalsequence<400>10ttggtggagaacaaggtgcgtctatttagtggagccctggaccttaatcgtgtgcggtat60ctgcttatgtcgcccgtctttcttgccttgct92<210>11<211>92<212>dna<213>artificalsequence<400>11tctcccatgactcctgtcgcgcttggtataatcgctgagaggccgtcgctatacatgtat60ctgcttatgtcgcccggaccagggtgttgttt92<210>12<211>92<212>dna<213>artificalsequence<400>12aggaagtctcggtactcagtgaatgcgagtccgtctctggaccttaatcgtgtgcgaagc60tgatctaagccgcgagtgtccattctttgccc92<210>13<211>92<212>dna<213>artificalsequence<400>13catcggccaagtactgcagtgaatgcgagtccgtctctggaccttaatcgtgtgcgagcg60atctgcgagaccgtattcatcagaggtgtcca92<210>14<211>92<212>dna<213>artificalsequence<400>14ttgcacgttgagtttgtcgcgcttggtataatcgctagcagcgaaccatacgtgacaagc60tgatctaagccgcgagtgcaataatccgaggc92<210>15<211>92<212>dna<213>artificalsequence<400>15atccaggatacggtcttcgcgcttggtataatcgctagcagcgaaccatacgtgacagcg60atctgcgagaccgtatttcggatttcaggcgc92<210>16<211>92<212>dna<213>artificalsequence<400>16actgtgtcaccttcctagtagccgtgactatcgactgggccttattccggtgctatagcg60atctgcgagaccgtatcttcagggacaagacc92<210>17<211>92<212>dna<213>artificalsequence<400>17ataagccagactcccatgcgtctatttagtggagccgagaggccgtcgctatacatctag60tgctggatgatcgtccttaactcaggtgctgc92<210>18<211>92<212>dna<213>artificalsequence<400>18acttctgatctggggttgcgtctatttagtggagccagcagcgaaccatacgtgacagcg60atctgcgagaccgtatgtgttgtccatggagt92<210>19<211>92<212>dna<213>artificalsequence<400>19acagcaatttcctcccagtagccgtgactatcgactgggccttattccggtgctataagc60tgatctaagccgcgagcatgtccacgaactga92<210>20<211>92<212>dna<213>artificalsequence<400>20ctccatgcaagagacaagtagccgtgactatcgactgagaggccgtcgctatacatagcg60atctgcgagaccgtatacagtaggtgagggtt92<210>21<211>92<212>dna<213>artificalsequence<400>21ccatgtcagggaggtatgcgtctatttagtggagccagcagcgaaccatacgtgacgtat60ctgcttatgtcgcccggcagcctgatgatgaa92<210>23<211>20<212>dna/rna<213>artificalsequence<400>23ugcgucuauuuaguggagcc20<210>23<211>20<212>dna/rna<213>artificalsequence<400>23ucgcgcuugguauaaucgcu20<210>24<211>20<212>dna/rna<213>artificalsequence<400>24aguagccgugacuaucgacu20<210>25<211>20<212>dna/rna<213>artificalsequence<400>25cagugaaugcgaguccgucu20<210>26<211>20<212>dna/rna<213>artificalsequence<400>26gggccuuauuccggugcuau20<210>27<211>20<212>dna/rna<213>artificalsequence<400>27cuggaccuuaaucgugugcg20<210>28<211>20<212>dna/rna<213>artificalsequence<400>28agcagcgaaccauacgugac20<210>29<211>20<212>dna/rna<213>artificalsequence<400>29gagaggccgucgcuauacau20<210>30<211>20<212>dna/rna<213>artificalsequence<400>30cuagugcuggaugaucgucc20<210>31<211>20<212>dna/rna<213>artificalsequence<400>31aagcugaucuaagccgcgag20<210>32<211>20<212>dna/rna<213>artificalsequence<400>32agcgaucugcgagaccguau20<210>33<211>20<212>dna/rna<213>artificalsequence<400>33guaucugcuuaugucgcccg20当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1