B7-H3抗原结合结构域的分离的结合蛋白、核酸、载体、CAR-T细胞及其应用的制作方法
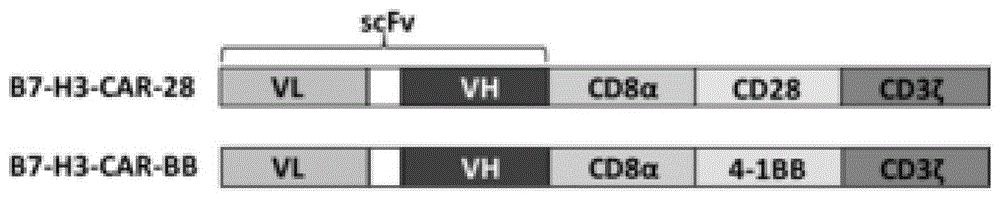
本发明涉及car-t细胞领域,具体而言,涉及b7-h3抗原结合结构域的分离的结合蛋白、核酸、载体、car-t细胞及其应用。
背景技术:
肺癌是全球癌症死亡率第一位的肿瘤,每年可增加大约130万新发病例,大约80-85%的新诊断的肺癌为非小细胞肺癌,如腺癌、鳞癌和大细胞癌等。中国在2017年有约80万新发肺癌病例,70万死亡肺癌病例,发病率和死亡率均占世界肺癌的40%左右,远远高于其它国家。在中国,肺癌也是发病率和死亡率双率第一的癌症,随着近年来空气污染问题的不断加重,预计未来肺癌患者的数量将进一步增加,严重影响着国人的健康和寿命。
虽然早期肺癌可通过手术的方式切除肿瘤,部分患者可实现治愈,但在绝大大多数的病例当中,患者就诊时已经是晚期癌症,属于不可切除的病变或者已经出现转移,属于无法治愈的疾病状态。对于局部进展期的非小细胞肺癌患者可以接受同步放化疗,联合或不联合手术治疗,这样可以获得平均8个月的无疾病进展生存,但是5年总生存率仍小于15%。就诊时已经诊断为晚期肺癌的患者可接受新型的细胞毒治疗药物,如培美曲塞等,中位生存时间约为17个月。
car-t(chimericantigenreceptortcells的英文缩写)是一种基于基因改造的自体t细胞的免疫疗法。该疗法在提取患者的t细胞后,通过基因改造,将一种特定的蛋白----嵌合抗原受体(即car)表达在t细胞的表面,借此识别癌细胞表面的特异抗原,并通过这些t细胞的抗原清除能力杀死癌细胞。car-t治疗在目前开展的多项临床试验中取得了巨大的成功,尤其是靶向cd19和bcma的car-t。虽然car-t治疗淋巴瘤、白血病等血液系统肿瘤已取得非常好的临床治疗效果,但car-t在实体瘤中的研究进展相对缓慢,其中一个重要的原因是没有合适的靶点,因为通常在实体瘤中高表达的抗原也会在一些正常组织表达。
car-t作为最新型的革命性的免疫治疗方法在白血病中已经实现了惊人的治疗效果,如何应用于实体瘤还需要进一步地研发。
有鉴于此,特提出本发明。
技术实现要素:
本发明针对b7-h3抗原得到的scfv片段,设计构建了用cd28或者4-1bb为共刺激分子的第二代b7-h3-car,并且在b7-h3阳性的非小细胞肺癌中验证了其杀伤癌细胞的能力,结果表明当t细胞与癌细胞比例为1:5时,b7-h3-car-t仍能够非常高效的杀伤b7-h3阳性的非小细胞肺癌细胞,并且释放出大量的i型干扰素,如ifnγ和tnfα。
为了实现本发明的上述目的,特采用以下技术方案:
一方面,本发明提供包含b7-h3抗原结合结构域的分离的结合蛋白,其轻链可变区和/或重链可变区的氨基酸序列依次如seqidno.1-2所示以及与seqidno.1-2所示的氨基酸序列具有至少95%的序列同一性的氨基酸序列。
须知,与seqidno.1-2所示的氨基酸序列具有至少95%的序列同一性的氨基酸序列是由本领域技术人员能够推知通过某一个或某几个氨基酸序列变换而仍具有较好的与b7-h3抗原结合的效果。如根据实际情况,至少95%的序列同一性可以为95%、96%、97%、98%、99%等等,氨基酸发生变化的个数可以为1个、2个、3个、4个、5个、6个等等。
进一步地,所述结合蛋白为scfv片段。
进一步地,所述轻链可变区与所述重链可变区通过连接区连接,所述连接区的氨基酸序列如seqidno.3所示。
b7-h3是一个i型膜蛋白,在多种实体瘤中高表达,例如93.7%的胰腺癌、90.6%的乳腺癌、83%的卵巢癌、86%的直肠癌、93.8%的肝癌和70%的非小细胞肺癌,是一个肿瘤免疫治疗的新靶点。但b7-h3并不在正常组织表达,比如心脏、肺、肾脏、肝脏以及大脑等神经细胞。近年来,多个靶向b7-h3的抗体药物,包括fc增强的抗体、同位素标记的抗体和抗体-药物偶联物等,在多个i期临床试验以及临床前的小鼠肿瘤模型研究中表现出了较好的抗肿瘤效果。这些研究结果进一步表明,设计靶向b7-h3的car用于实体瘤治疗是合理的,也是可行的。
本发明经筛选得到抗b7-h3单克隆抗体2d6,其能够稳定分泌抗b7-h3的抗体。经分子生物学手段将该单克隆抗体的重链可变区和轻链可变区以及两者之间的连接区克隆,其核酸序列依次如seqidno.4-6所示,其氨基氨酸序列依次如seqidno.1-3所示。
第二方面,本发明提供了一种分离的核酸,所述核酸编码上述的结合蛋白。
进一步地,所述轻链可变区的核酸序列如seqidno.4所示。
进一步地,所述重链可变区的核酸序列如seqidno.5所示。
进一步地,所述连接区的核酸序列如seqidno.6所示。
进一步地,所述核酸还连接有信号肽核酸序列。
进一步地,所述信号肽的核酸序列如seqidno.7所示。信号肽对应的氨基酸序列如seqidno.8所示。
进一步地,所述信号肽位于所述轻链可变区的氮端。
本发明中的信号肽序列包含在pcr所使用的n-端引物中,这样扩增得到的序列中即含有信号肽。
第三方面,本发明提供一种载体,其包含上述的核酸。
进一步地,所述载体含有cd8α的铰链区、cd8α的跨膜结构域以及cd3ζ的细胞内结构域,并包含cd28或4-1bb共刺激结构域。
进一步地,cd8α的铰链区、cd8α的跨膜结构域的核酸序列如seqidno.9所示,其对应的氨基酸序列如seqidno.10所示。
进一步地,cd3ζ的细胞内结构域的核酸序列如seqidno.11所示,其对应的氨基酸序列如seqidno.12所示。
进一步地,cd28的核酸序列如seqidno.13所示,其对应的氨基酸序列如seqidno.14所示。
进一步地,4-1bb的核酸序列如seqidno.15所示,其对应的氨基酸序列如seqidno.16所示。
进一步地,所述载体为慢病毒载体;所述慢病毒载体优选为pcdh-cmv-car-28或pcdh-cmv-car-bb。
进一步地,所述载体中各核酸序列的连接方式如下:
pcdh-cmv-b7-h3-car-28或pcdh-cmv-b7-h3-car-bb;
其中,b7-h3-car-28的核酸序列依次为信号肽-vl-linker-vh-cd8ɑ-cd28-cd3ζ;
作为优选,b7-h3-car-28的核酸序列如seqidno.17所示,其对应的氨基酸序列如seqidno.18所示。
b7-h3-car-bb的核酸序列依次为信号肽-vl-linker-vh-cd8ɑ-4-1bb-cd3ζ。
作为优选,b7-h3-car-bb的核酸序列如seqidno.19所示,其对应的氨基酸序列如seqidno.20所示。
本发明提供的载体中,构建了cd28或者4-1bb作为共刺激分子的第二代b7-h3-car,并且在b7-h3阳性的非小细胞肺癌中验证了其杀伤癌细胞的能力,能够非常高效的杀伤b7-h3阳性的非小细胞肺癌细胞。
第四方面,本发明提供一种宿主细胞,所述宿主细胞包括上述的核酸或上述的载体。
进一步地,所述宿主细胞用于表达所述核酸或载体。即该宿主细胞用于表达外源基因的细胞株。
进一步地,所述宿主细胞为293细胞。
293细胞是人肾上皮细胞系,有多种衍生株,比如hek293,293t/17等。
本发明提供的该宿主细胞,以进行扩增和富集慢病毒或者是质粒。用于后续的car-t细胞制备。
在实际操作中,质粒转染宿主细胞后,将含有慢病毒的培养上清收集,并用0.45μm过滤器过滤,然后冻存待用。
第五方面,本发明提供了一种car-t细胞的制备方法,包括如下步骤:
上述的宿主细胞制得的含有病毒的上清感染t细胞,分离得到car-t细胞。
t细胞采用以下方法制备:外周血中分离pbmc淋巴细胞,然后加入cd3和cd28抗体包被的培养板中,20-30小时之后,加入il2(10μg/ml),刺激40-60小时后收集t细胞,即得。
进一步地,pbmc淋巴细胞加入cd3和cd28抗体包被的24孔板中,1×106个细胞每孔。
外周血从健康的志愿者获得。
将刺激好的t细胞用于上述重组慢病毒感染。
具体地,t细胞用于上述重组慢病毒感染采用以下方法进行:
在24孔细胞培养板中加入5×105个刺激好的t细胞,然后加入1ml表达b7-h3-car-28或b7-h3-car-bb的慢病毒,2000g离心1.5小时,离心结束后将细胞置于37℃细胞培养箱中培养,24小时之后,将含有慢病毒的培养基去掉,换为新鲜培养基,此后,每两天更换一次培养基,12天后,收集car-t细胞,检测car-t细胞的杀伤功能。
第六方面,本发明提供上述制备方法制得的car-t细胞。
第七方面,本发明提供上述的car-t细胞在制备治疗肿瘤制剂中的应用。
进一步地,所述肿瘤包括所有b7-h3阳性的肿瘤,包括血液瘤和实体瘤,例如但不限于肺癌、胰腺癌、胃癌、乳腺癌、卵巢癌、宫颈癌、脑瘤、头颈癌、结直肠癌、黑色素瘤、肝癌、胃癌、食道癌、睾丸癌、前列腺癌、肾癌、神经胶质瘤、神经母细胞瘤、骨髓瘤、淋巴瘤中的任一种。
本发明验证了提供的car-t细胞对非小细胞肺癌具有高效的杀伤力。显而易见的可推知,对其他实体瘤如胰腺癌、乳腺癌、卵巢癌、直肠癌、肝癌也会有高效的杀伤力。
与现有技术相比,本发明的有益效果为:
(1)本发明针对b7-h3抗原得到的scfv片段,设计构建了用cd28或者4-1bb为共刺激分子的第二代b7-h3-car,并且在b7-h3阳性的非小细胞肺癌中验证了其杀伤癌细胞的能力。
(2)本发明提供的car-t细胞与癌细胞比例为1:5时,仍能够非常高效的杀伤b7-h3阳性的非小细胞肺癌细胞,并且释放出大量的i型干扰素,如ifnγ和tnfα,为实体瘤的治疗提供良好的基础。
(3)本发明提供的car-t细胞可用于治疗实体瘤,实体瘤包括非小细胞肺癌、胰腺癌、乳腺癌、卵巢癌、直肠癌、肝癌等。
附图说明
为了更清楚地说明本发明实施例或现有技术中的技术方案,以下将对实施例或现有技术描述中所需要使用的附图作简单地介绍。
图1为本发明实施例1中载体的结构示意图;
图2为本发明实施例1中b7-h3-car-28或b7-h3car-bb的质粒dna的慢病毒感染激活的t细胞制得的car-t细胞中,b7-h3-car-28和b7-h3-car-bb的表达情况图;
图3为本发明实施例1中不同细胞与不同非小细胞肺癌细胞系共培养后,流式细胞仪检测t细胞(cd3+)和剩余癌细胞的比例(b7-h3+)的结果图;
图4为本发明实施例1中b7-h3抗体感染不同非小细胞肺癌细胞系后b7-h3的表达情况图;
图5为本发明实施例1中不同细胞作用于非小细胞肺癌细胞时分泌ifnγ和tnfα量的柱形图;
图6为本发明实施例2中b7-h3-car-t在小鼠体内清除肺癌肿瘤情况的监测图。
具体实施方式
下面将结合实施例对本发明的实施方案进行详细描述,但是本领域技术人员将会理解,下列实施例仅用于说明本发明,而不应视为限制本发明的范围。实施例中未注明具体条件者,按照常规条件或制造商建议的条件进行。所用试剂或仪器未注明生产厂商者,均为可以通过市售购买获得的常规产品。
实施例1
1、b7-h3抗体筛选
用纯化的b7-h3-his蛋白与弗氏佐剂混合,通过皮下注射免疫balb/c小鼠,每两周免疫一次,共免疫三次。第35天,采血并通过elisa检测血清中抗b7-h3的抗体,当4000倍稀释后的od值大于1.0时,表明血清中含有较高浓度的抗b7-h3抗体。第39天,再次免疫小鼠,第42天收集小鼠脾脏细胞,制备单细胞混悬液,并与骨髓瘤细胞f0通过peg进行融合。培养7天后,通过elisa对融合的杂交瘤克隆的上清进行筛选,检测它们的特异性和敏感性,经过复筛和亚克隆之后,得到单克隆2d6能够稳定分泌抗b7-h3的抗体。
2、质粒构建
为了构建靶向b7-h3的car,我们选择了抗b7-h3的2d6单克隆抗体,将编码抗体可变区的dna序列(scfv)克隆入慢病毒载体,以用包含编码b7-h3-car-28或b7-h3car-bb的质粒dna的慢病毒感染激活的t来制作car-t细胞。
其中,慢病毒载体选用pcdh-cmv-car-28或pcdh-cmv-car-bb,这两个载体都含有cd8α的铰链区和跨膜结构域以及cd3ζ的细胞内结构域,并分别包含cd28或4-1bb共刺激结构域,用来构建b7-h3-car-28和b7-h3-car-bb(图1)。
通过pcr将抗b7-h3单克隆抗体2d6的轻链和重链的可变区片段(vl和vh)扩增出来,信号肽(sp)序列包含在pcr所使用的n-端引物中,然后用xbai和bamhi限制性内切酶酶切后,通过t4连接酶克隆入慢病毒载体pcdh-cmv-car-28(pcdh-cmv-b7-h3-car-28)或pcdh-cmv-car-bb(pcdh-cmv-b7-h3-car-bb)。
其中,2d6单克隆抗体编码抗体可变区的dna序列(scfv)包括轻链可变区(核酸序列如seqidno.4所示)与重链可变区(核酸序列如seqidno.5所示),以及之间的连接区(核酸序列如seqidno.6所示);信号肽的核酸序列如seqidno.7所示,其对应的氨基酸序列如seqidno.8所示;cd8α的核酸序列如seqidno.9所示,其对应的氨基酸序列如seqidno.10所示;cd3ζ的核酸序列如seqidno.11所示,其对应的氨基酸序列如seqidno.12所示;cd28的核酸序列如seqidno.13所示,其对应的氨基酸序列如seqidno.14所示;4-1bb的核酸序列如seqidno.15所示,其对应的氨基酸序列如seqidno.16所示;b7-h3-car-28即b7-h3-car-cd8α-cd28-cd3ζ完整序列如seqidno.17所示,其对应的氨基酸序列如seqidno.18所示;b7-h3-car-bb即b7-h3-car-cd8α-4-1bb-cd3ζ完整序列如seqidno.19所示,其对应的氨基酸序列如seqidno.20所示。
3、表达b7-h3-car的慢病毒制备
将7×106293t细胞接种于10cm的细胞培养皿中,16小时之后,将4μgpcdh-cmv-b7-h3-car-28或pcdh-cmv-b7-h3-car-bb质粒,4μgpspax2质粒和2μgpmd2g质粒混合,然后将质粒混合物与genejuice转染试剂(merckmillipore)混匀,静置10分钟,然后转染293t细胞。转染48小时之后,将含有慢病毒的培养上清收集,并用0.45μm过滤器过滤,然后冻存待用。
4、car-t细胞的制备和体外扩增
从健康的志愿者外周血中分离pbmc淋巴细胞,然后加入cd3和cd28抗体包被的24孔板中,1×106个细胞每孔。24小时之后,加入il2(10μg/ml),刺激两天后收集t细胞,计数待用。
将刺激好的t细胞用上述重组慢病毒感染,具体感染步骤如下:在24孔细胞培养板中加入5×105个刺激好的t细胞,然后加入1ml表达b7-h3-car-28或b7-h3-car-bb的慢病毒,2000g离心1.5小时,离心结束后将细胞置于37℃细胞培养箱中培养,24小时之后,将含有慢病毒的培养基去掉,换为新鲜培养基,此后,每两天更换一次培养基,12天后,收集car-t细胞,检测car-t细胞的杀伤功能。感染4天后,取5×105个细胞,用抗fab的抗体染car-t细胞表面的car,并用流式细胞仪检测b7-h3-car的表达情况,结果如图2所示。
即用包含编码b7-h3-car-28或b7-h3car-bb的质粒dna的慢病毒感染激活的t细胞来制作car-t细胞,结果表明b7-h3-car-28和b7-h3-car-bb可以高效的表达在car-t细胞表面,95%以上的car-t细胞是b7-h3-car阳性(图2)。
5、流式检测
t细胞和癌细胞分别用cd3-apc-h7和b7-h3-apc(bdbioscience)的抗体来标记,b7-h3-car的表达是通过anti-fab(jacksonimmunoresearchlaboratoriesinc.)的抗体来标记,标记的样品通过bdfacscantoii流式细胞仪用bddivasoftware(bdbiosciences)来检测,每个样品最少收集10,000个活细胞,用flowjo10软件分析收集的结果。
nt、b7-h3-car-28-t或b7-h3-car-bb-t细胞与三种非小细胞肺癌细胞系hcc827、nci-h522和nci-h1299共培养,t细胞与癌细胞的比例为1:5,共培养5天后,用流式细胞仪检测t细胞(cd3+)和剩余癌细胞的比例(b7-h3+)。
检测b7-h3-car-28和b7-h3-car-bb杀伤b7-h3+癌细胞的能力,将hcc827、nci-h522和nci-h1299这三种b7-h3+肺癌细胞(图3)与对照t细胞(未被慢病毒感染的t细胞,nt)、b7-h3-car-28-t或b7-h3-car-bb-t细胞共培养,t细胞与癌细胞比例为1:5,共培养5天后,收集悬浮的t细胞和贴壁的癌细胞,将两种细胞合在一起,分别用cd3和b7-h3的抗体标记t细胞和癌细胞,然后用流式细胞仪检测残留癌细胞的比例。结果如图3所示。
从图3可以看出,共培养5天后,b7-h3-car-28-t和b7-h3-car-bb-t细胞均能完全将这三种肺癌细胞清除。验证了b7-h3-car-28-t和b7-h3-car-bb-t细胞能够识别并靶向杀伤这三种肺癌细胞。
用b7-h3的抗体感染非小细胞肺癌细胞系hcc827、nci-h522和nci-h1299,然后用流式细胞仪检测b7-h3的表达,结果如图4所示。
从图4可以看出,非小细胞肺癌均高表达b7-h3。
6、elisa检测
b7-h3-car-t杀伤非小细胞肺癌细胞时分泌大量ifnγ和tnfα。采用elisa检测ifnγ和tnfα的量。具体如下:
将非小细胞肺癌细胞以5×105每孔加入24孔细胞培养板中,16小时之后,加入5×105nt、b7-h3-car-28或b7-h3-car-bbcar-t细胞,t细胞与癌细胞的比例为1:5,每孔2ml细胞培养基。共培养24小时后,收集1ml上清,用ifnγ和tnfα的elisa试剂盒(r&dsystem)来检测共培养上清中这两种i型干扰素的量。结果如图5所示。
从图5可以看出,b7-h3-car-28-t和b7-h3-car-bb-t细胞释放出大量的i型干扰素-ifnγ和tnfα。
实施例2
1、共培养杀伤实验
将非小细胞肺癌细胞以5×105每孔加入24孔细胞培养板中,16小时之后,加入5×105nt、b7-h3-car-28或b7-h3-car-bbcar-t细胞,t细胞与癌细胞的比例为1:5。共培养5天后,将悬浮的t细胞收集和贴壁的癌细胞全部收集,分别用cd3和b7-h3的抗体标记t细胞和癌细胞,然后用流式细胞仪检测残留癌细胞的比例,用zombieaquadye(biolegend)去除死细胞。
2、用h522肺癌转移模型在小鼠体内评估b7-h3-car-t杀伤肿瘤的效果
将过表达萤光素酶的h522肺癌细胞通过尾静脉注射入nsg小鼠体内,每只小鼠注射2×106个h522细胞,以此构建小鼠的肺癌转移模型。14天后,用nt、b7-h3-car-28-t或b7-h3-car-bb-t细胞处理荷瘤小鼠,每只小鼠注射10×106个t细胞,每周通过ivis仪器拍照来监测肿瘤大小的变化。
结果如图6所示,经过一周的治疗之后,在b7-h3-car-28-t或b7-h3-car-bb-t细胞处理组中,小鼠肺部的癌细胞完全消失,但在小鼠靠近尾部仍有部分癌细胞存活,两周之后,小鼠全身的癌细胞被b7-h3-car-28-t或b7-h3-car-bb-t细胞完全清除,在后续60多天的跟踪监测中,所有b7-h3-car-t处理的小鼠,未观察到肺癌复发现象。以上结果表明b7-h3-car-28-t和b7-h3-car-bb-t细胞可以在小鼠体内高效杀伤b7-h3阳性肺癌细胞,并且防止复发。
由于b7-h3几乎高表达于所有的实体瘤癌细胞,例如93.7%的胰腺癌、90.6%的乳腺癌、83%的卵巢癌、86%的直肠癌、93.8%的肝癌和70%的非小细胞肺癌等。因此,靶向b7-h3的car-t可以广泛应用于靶向治疗所有b7-h3+的实体瘤或者骨髓瘤。
尽管已用具体实施例来说明和描述了本发明,然而应意识到,在不背离本发明的精神和范围的情况下可以作出许多其它的更改和修改。因此,这意味着在所附权利要求中包括属于本发明范围内的所有这些变化和修改。
sequencelisting
<110>北京善科生物科技有限公司
<120>b7-h3抗原结合结构域的分离的结合蛋白、核酸、载体、car-t细胞及其应用
<130>2019
<160>20
<170>patentinversion3.3
<210>1
<211>106
<212>prt
<213>musmusculus
<400>1
glnilevalleuthrglnserproalailemetseralaserprogly
151015
glulysvalthrmetthrcysseralaserserservaliletyrmet
202530
histrptyrglnglnlysserglythrserserlysargtrpiletyr
354045
aspthrserlysleualaserglyvalproalaargpheserglyser
505560
alaserglyalasertyrserleuserilesersermetglualaglu
65707580
aspalaalathrtyrtyrcysglnglntrpgluileasnproleuthr
859095
pheglyalaglythrlysleugluleulys
100105
<210>2
<211>118
<212>prt
<213>musmusculus
<400>2
glnvalglnleuglngluserglyproglyleuvalvalhissergln
151015
serleuserilethrcysthrvalserglypheserleuileglytyr
202530
glyvalasntrpvalargglnproproglylysserleuglutrpleu
354045
glymetiletrpcysaspglyargthrasptyrasnserglyleulys
505560
serargleuserileserlysaspasnserlysserglnvalpheleu
65707580
lysmetasnserleuglnthraspaspthralaargtyrtyrcysala
859095
argglytyrglyasnhisalatrpleualatyrtrpcysglnglythr
100105110
leuvalservalserala
115
<210>3
<211>15
<212>prt
<213>homosapiens
<400>3
glyglyglyglyserglyglyglyglyserglyglyglyglyser
151015
<210>4
<211>318
<212>dna
<213>musmusculus
<400>4
caaattgttctcacccagtctccagcaatcatgtctgcatctccaggggagaaggtcacc60
atgacctgcagtgccagctcaagtgtaatttacatgcactggtaccagcagaagtcaggc120
acctcctccaaaagatggatctatgacacatccaaactggcttctggagtccctgctcgc180
ttcagtggcagtgcgtctggcgcctcttactctctctcaatcagcagcatggaggctgaa240
gatgctgccacttattactgccagcagtgggaaattaacccgctcacgttcggtgctggg300
accaagctggagctgaaa318
<210>5
<211>354
<212>dna
<213>musmusculus
<400>5
caggtgcagctgcaggagtcaggacctggcctggtggtgcactcacagagcctgtccatc60
acatgcaccgtttcagggttctcattaatcggctatggtgtaaactgggttcgccagcct120
ccaggaaagagtctggagtggctcggaatgatatggtgtgatggaaggacagactataat180
tcaggtctcaaatccagactgagcatcagcaaggacaactccaagagccaagttttctta240
aaaatgaacagtctgcaaactgatgacacagccaggtattactgtgccagagggtatggt300
aaccacgcctggcttgcttactggtgccaagggactctggtcagtgtctctgca354
<210>6
<211>45
<212>dna
<213>homosapiens
<400>6
ggcggcggaggatctggcggaggcggaagtggcggagggggctct45
<210>7
<211>57
<212>dna
<213>homosapiens
<400>7
atggaattcggcctgagctggctgttcctggtggccatcctgaagggcgtgcagtgc57
<210>8
<211>19
<212>prt
<213>homosapiens
<400>8
metglupheglyleusertrpleupheleuvalalaileleulysgly
151015
valglncys
<210>9
<211>207
<212>dna
<213>homosapiens
<400>9
accacgacgccagcgccgcgaccaccaacaccggcgcccaccatcgcgtcgcagcccctg60
tccctgcgcccagaggcgtgccggccagcggcggggggcgcagtgcacacgagggggctg120
gacttcgcctgtgatatctacatctgggcgcccttggccgggacttgtggggtccttctc180
ctgtcactggttatcaccctttactgc207
<210>10
<211>69
<212>prt
<213>homosapiens
<400>10
thrthrthrproalaproargproprothrproalaprothrileala
151015
serglnproleuserleuargproglualacysargproalaalagly
202530
glyalavalhisthrargglyleuaspphealacysaspiletyrile
354045
trpalaproleualaglythrcysglyvalleuleuleuserleuval
505560
ilethrleutyrcys
65
<210>11
<211>339
<212>dna
<213>homosapiens
<400>11
agagtgaagttcagcaggagcgcagacgcccccgcgtaccagcagggccagaaccagctc60
tataacgagctcaatctaggacgaagagaggagtacgatgttttggacaagagacgtggc120
cgggaccctgagatggggggaaagccgagaaggaagaaccctcaggaaggcctgtacaat180
gaactgcagaaagataagatggcggaggcctacagtgagattgggatgaaaggcgagcgc240
cggaggggcaaggggcacgatggcctttaccagggtctcagtacagccaccaaggacacc300
tacgacgcccttcacatgcaggccctgccccctcgctaa339
<210>12
<211>112
<212>prt
<213>homosapiens
<400>12
argvallyspheserargseralaaspalaproalatyrglnglngly
151015
glnasnglnleutyrasngluleuasnleuglyargarggluglutyr
202530
aspvalleuasplysargargglyargaspproglumetglyglylys
354045
proargarglysasnproglngluglyleutyrasngluleuglnlys
505560
asplysmetalaglualatyrsergluileglymetlysglygluarg
65707580
argargglylysglyhisaspglyleutyrglnglyleuserthrala
859095
thrlysaspthrtyraspalaleuhismetglnalaleuproproarg
100105110
<210>13
<211>123
<212>dna
<213>homosapiens
<400>13
aggagtaagaggagcaggctcctgcacagtgactacatgaacatgactccccgccgcccc60
gggcccacccgcaagcattaccagccctatgccccaccacgcgacttcgcagcctatcgc120
tcc123
<210>14
<211>41
<212>prt
<213>homosapiens
<400>14
argserlysargserargleuleuhisserasptyrmetasnmetthr
151015
proargargproglyprothrarglyshistyrglnprotyralapro
202530
proargaspphealaalatyrargser
3540
<210>15
<211>126
<212>dna
<213>homosapiens
<400>15
aaacggggcagaaagaaactcctgtatatattcaaacaaccatttatgagaccagtacaa60
actactcaagaggaagatggctgtagctgccgatttccagaagaagaagaaggaggatgt120
gaactg126
<210>16
<211>42
<212>prt
<213>homosapiens
<400>16
lysargglyarglyslysleuleutyrilephelysglnprophemet
151015
argprovalglnthrthrglnglugluaspglycyssercysargphe
202530
proglugluglugluglyglycysgluleu
3540
<210>17
<211>1443
<212>dna
<213>人工序列
<400>17
atggaattcggcctgagctggctgttcctggtggccatcctgaagggcgtgcagtgccaa60
attgttctcacccagtctccagcaatcatgtctgcatctccaggggagaaggtcaccatg120
acctgcagtgccagctcaagtgtaatttacatgcactggtaccagcagaagtcaggcacc180
tcctccaaaagatggatctatgacacatccaaactggcttctggagtccctgctcgcttc240
agtggcagtgcgtctggcgcctcttactctctctcaatcagcagcatggaggctgaagat300
gctgccacttattactgccagcagtgggaaattaacccgctcacgttcggtgctgggacc360
aagctggagctgaaaggcggcggaggatctggcggaggcggaagtggcggagggggctct420
caggtgcagctgcaggagtcaggacctggcctggtggtgcactcacagagcctgtccatc480
acatgcaccgtttcagggttctcattaatcggctatggtgtaaactgggttcgccagcct540
ccaggaaagagtctggagtggctcggaatgatatggtgtgatggaaggacagactataat600
tcaggtctcaaatccagactgagcatcagcaaggacaactccaagagccaagttttctta660
aaaatgaacagtctgcaaactgatgacacagccaggtattactgtgccagagggtatggt720
aaccacgcctggcttgcttactggtgccaagggactctggtcagtgtctctgcaaccacg780
acgccagcgccgcgaccaccaacaccggcgcccaccatcgcgtcgcagcccctgtccctg840
cgcccagaggcgtgccggccagcggcggggggcgcagtgcacacgagggggctggacttc900
gcctgtgatatctacatctgggcgcccttggccgggacttgtggggtccttctcctgtca960
ctggttatcaccctttactgcaggagtaagaggagcaggctcctgcacagtgactacatg1020
aacatgactccccgccgccccgggcccacccgcaagcattaccagccctatgccccacca1080
cgcgacttcgcagcctatcgctccagagtgaagttcagcaggagcgcagacgcccccgcg1140
taccagcagggccagaaccagctctataacgagctcaatctaggacgaagagaggagtac1200
gatgttttggacaagagacgtggccgggaccctgagatggggggaaagccgagaaggaag1260
aaccctcaggaaggcctgtacaatgaactgcagaaagataagatggcggaggcctacagt1320
gagattgggatgaaaggcgagcgccggaggggcaaggggcacgatggcctttaccagggt1380
ctcagtacagccaccaaggacacctacgacgcccttcacatgcaggccctgccccctcgc1440
taa1443
<210>18
<211>480
<212>prt
<213>人工序列
<400>18
metglupheglyleusertrpleupheleuvalalaileleulysgly
151015
valglncysglnilevalleuthrglnserproalailemetserala
202530
serproglyglulysvalthrmetthrcysseralaserserserval
354045
iletyrmethistrptyrglnglnlysserglythrserserlysarg
505560
trpiletyraspthrserlysleualaserglyvalproalaargphe
65707580
serglyseralaserglyalasertyrserleuserilesersermet
859095
glualagluaspalaalathrtyrtyrcysglnglntrpgluileasn
100105110
proleuthrpheglyalaglythrlysleugluleulysglyglygly
115120125
glyserglyglyglyglyserglyglyglyglyserglnvalglnleu
130135140
glngluserglyproglyleuvalvalhisserglnserleuserile
145150155160
thrcysthrvalserglypheserleuileglytyrglyvalasntrp
165170175
valargglnproproglylysserleuglutrpleuglymetiletrp
180185190
cysaspglyargthrasptyrasnserglyleulysserargleuser
195200205
ileserlysaspasnserlysserglnvalpheleulysmetasnser
210215220
leuglnthraspaspthralaargtyrtyrcysalaargglytyrgly
225230235240
asnhisalatrpleualatyrtrpcysglnglythrleuvalserval
245250255
seralathrthrthrproalaproargproprothrproalaprothr
260265270
ilealaserglnproleuserleuargproglualacysargproala
275280285
alaglyglyalavalhisthrargglyleuaspphealacysaspile
290295300
tyriletrpalaproleualaglythrcysglyvalleuleuleuser
305310315320
leuvalilethrleutyrcysargserlysargserargleuleuhis
325330335
serasptyrmetasnmetthrproargargproglyprothrarglys
340345350
histyrglnprotyralaproproargaspphealaalatyrargser
355360365
argvallyspheserargseralaaspalaproalatyrglnglngly
370375380
glnasnglnleutyrasngluleuasnleuglyargarggluglutyr
385390395400
aspvalleuasplysargargglyargaspproglumetglyglylys
405410415
proargarglysasnproglngluglyleutyrasngluleuglnlys
420425430
asplysmetalaglualatyrsergluileglymetlysglygluarg
435440445
argargglylysglyhisaspglyleutyrglnglyleuserthrala
450455460
thrlysaspthrtyraspalaleuhismetglnalaleuproproarg
465470475480
<210>19
<211>1446
<212>dna
<213>人工序列
<400>19
atggaattcggcctgagctggctgttcctggtggccatcctgaagggcgtgcagtgccaa60
attgttctcacccagtctccagcaatcatgtctgcatctccaggggagaaggtcaccatg120
acctgcagtgccagctcaagtgtaatttacatgcactggtaccagcagaagtcaggcacc180
tcctccaaaagatggatctatgacacatccaaactggcttctggagtccctgctcgcttc240
agtggcagtgcgtctggcgcctcttactctctctcaatcagcagcatggaggctgaagat300
gctgccacttattactgccagcagtgggaaattaacccgctcacgttcggtgctgggacc360
aagctggagctgaaaggcggcggaggatctggcggaggcggaagtggcggagggggctct420
caggtgcagctgcaggagtcaggacctggcctggtggtgcactcacagagcctgtccatc480
acatgcaccgtttcagggttctcattaatcggctatggtgtaaactgggttcgccagcct540
ccaggaaagagtctggagtggctcggaatgatatggtgtgatggaaggacagactataat600
tcaggtctcaaatccagactgagcatcagcaaggacaactccaagagccaagttttctta660
aaaatgaacagtctgcaaactgatgacacagccaggtattactgtgccagagggtatggt720
aaccacgcctggcttgcttactggtgccaagggactctggtcagtgtctctgcaaccacg780
acgccagcgccgcgaccaccaacaccggcgcccaccatcgcgtcgcagcccctgtccctg840
cgcccagaggcgtgccggccagcggcggggggcgcagtgcacacgagggggctggacttc900
gcctgtgatatctacatctgggcgcccttggccgggacttgtggggtccttctcctgtca960
ctggttatcaccctttactgcaaacggggcagaaagaaactcctgtatatattcaaacaa1020
ccatttatgagaccagtacaaactactcaagaggaagatggctgtagctgccgatttcca1080
gaagaagaagaaggaggatgtgaactgagagtgaagttcagcaggagcgcagacgccccc1140
gcgtaccagcagggccagaaccagctctataacgagctcaatctaggacgaagagaggag1200
tacgatgttttggacaagagacgtggccgggaccctgagatggggggaaagccgagaagg1260
aagaaccctcaggaaggcctgtacaatgaactgcagaaagataagatggcggaggcctac1320
agtgagattgggatgaaaggcgagcgccggaggggcaaggggcacgatggcctttaccag1380
ggtctcagtacagccaccaaggacacctacgacgcccttcacatgcaggccctgccccct1440
cgctaa1446
<210>20
<211>481
<212>prt
<213>人工序列
<400>20
metglupheglyleusertrpleupheleuvalalaileleulysgly
151015
valglncysglnilevalleuthrglnserproalailemetserala
202530
serproglyglulysvalthrmetthrcysseralaserserserval
354045
iletyrmethistrptyrglnglnlysserglythrserserlysarg
505560
trpiletyraspthrserlysleualaserglyvalproalaargphe
65707580
serglyseralaserglyalasertyrserleuserilesersermet
859095
glualagluaspalaalathrtyrtyrcysglnglntrpgluileasn
100105110
proleuthrpheglyalaglythrlysleugluleulysglyglygly
115120125
glyserglyglyglyglyserglyglyglyglyserglnvalglnleu
130135140
glngluserglyproglyleuvalvalhisserglnserleuserile
145150155160
thrcysthrvalserglypheserleuileglytyrglyvalasntrp
165170175
valargglnproproglylysserleuglutrpleuglymetiletrp
180185190
cysaspglyargthrasptyrasnserglyleulysserargleuser
195200205
ileserlysaspasnserlysserglnvalpheleulysmetasnser
210215220
leuglnthraspaspthralaargtyrtyrcysalaargglytyrgly
225230235240
asnhisalatrpleualatyrtrpcysglnglythrleuvalserval
245250255
seralathrthrthrproalaproargproprothrproalaprothr
260265270
ilealaserglnproleuserleuargproglualacysargproala
275280285
alaglyglyalavalhisthrargglyleuaspphealacysaspile
290295300
tyriletrpalaproleualaglythrcysglyvalleuleuleuser
305310315320
leuvalilethrleutyrcyslysargglyarglyslysleuleutyr
325330335
ilephelysglnprophemetargprovalglnthrthrglngluglu
340345350
aspglycyssercysargpheproglugluglugluglyglycysglu
355360365
leuargvallyspheserargseralaaspalaproalatyrglngln
370375380
glyglnasnglnleutyrasngluleuasnleuglyargarggluglu
385390395400
tyraspvalleuasplysargargglyargaspproglumetglygly
405410415
lysproargarglysasnproglngluglyleutyrasngluleugln
420425430
lysasplysmetalaglualatyrsergluileglymetlysglyglu
435440445
argargargglylysglyhisaspglyleutyrglnglyleuserthr
450455460
alathrlysaspthrtyraspalaleuhismetglnalaleupropro
465470475480
arg
- 还没有人留言评论。精彩留言会获得点赞!