一种带有标记的棉花雄性不育系的创制方法与流程
本发明属于植物基因工程技术领域,具体涉及一种带有标记的棉花雄性不育系的创制方法及其应用。本发明提供利用crispr-cas9系统创造带有新型标记的棉花雄性不育系创制方法。选取特异针对ghems1基因的靶位点sgrna1、sgrna2,与cas9蛋白在棉花内稳定表达,获得带有新型标记的棉花雄性不育系。转基因再生植株的花药表面带有黑斑表型可稳定遗传至下一代,且棉花雄性不育性稳定。本发明的优点在于克服了传统人工去雄方法做杂交费时费力的缺陷,并且不育株带有黑斑标记,便于早期确定不育株。
背景技术:
杂种优势可以大幅度提高农作物产量,棉花作为重要的经济作物,也具有明显的杂交优势,陆地棉品种间杂交种的强优势组合可增产20%~30%。在20世纪90年代取得突破,培育出一大批抗虫优质杂交棉品种,在生产上大面积推广应用。例如,中国农业科学院棉花研究所的培育的“中棉所”系列,华中农业大学培育的的“华杂棉”系列,山东省棉花研究中心培育的“鲁棉研”系列,湖南省棉花研究所培育的“湘杂棉”系列等等。在棉花雄性不育利用方面,四川省农业科学院于1972年发现“洞a型”细胞核雄性不育系后,随即开展棉花细胞核雄性不育系及杂交种选育,培育出“川杂”系列核不育杂交种。国内多个单位利用引进“洞a型”核不育两用系或转育出新的核不育系,也培育一系列杂交种。中国农业科学院棉花研究所引进ms5ms5ms6ms6双隐性核不育系,通过杂交和回交改良,培育出“中棉所38”等多个杂交棉品种。河北省邯郸市农业科学院从海陆杂种后代中筛选出104-7a胞质不育系,育成了“邯杂98-1”等多个国家审定或省审定的三系杂交棉品种。现有的人工去雄授粉制种方法费工费时,近年来随着农村劳动力短缺,杂交棉种子成本逐年高涨,杂交棉应用面积大幅度减少,因此创造出育性稳定可靠的雄性不育系是我国杂交棉生产必须解决的技术问题。
crispr/cas9系统直接作用于dna,可对几乎任意基因组区域进行编辑,影响相应基因的功能,方法简单、精确、高效,成为近年植物基因组学研究的热点。在棉花上,wang等(wangetal.2018)针对1-脱氧木酮糖-5-磷酸合成酶(cloroplastoalterados,cla)的a、d亚基因组设计了两个sgrna,以指导cas9介导的异源四倍体棉花基因组编辑。测序结果表明,两个靶位点分别发生了80%以上的突变频率,所有样品均未检测到脱靶的情况,说明crispr/cas9系统可以高效率和高特异性在异源四倍体棉花基因组上产生dna水平突变。
基因编辑在植物不育系的创制上得到有效应用,水稻tms5是中国应用广泛的温敏核雄性不育系的主效基因,zhou等在tms5的编码区设计了10个靶位点,使用crispr/cas9系统进行定向诱变,创造了新的不含转基因元件的温敏不育系。建立的tms5ab二元构建体,可用于籼、粳两个亚种的温敏不育系快速培育(zhouetal.2016)。ms45是玉米中发现的一个雄性不育基因,该基因编码一种花粉发育必需的类胡萝卜素合成酶。singh等(singhetal.2018)为了研究ms45基因在小麦中的作用,使用crispr/cas9技术,对小麦3个同源染色a、b和d的ms45同源基因进行了编辑,获得突变体。三个同源位点的同时突变,造成小麦的雄性不育,该不育系可被非同时突变的材料恢复育性。chen等通过基因编辑技术,对玉米的育性相关基因ms8进行了编辑,得到新的玉米雄性不育系(chenetal.2018)。通过基因编辑技术创造棉花雄性不育系应是一个便捷有效的手段。
技术实现要素:
本发明的目的在于克服棉花现有杂交育种中雄性不育系不完全败育、无明显标记的不足,提供一种带有标记快速创制棉花雄性不育系的创制方法及其应用。本发明包括prgeb32-ghu6.7-nptⅱ载体构建(wangetal.2018),针对ghems1基因构建基因编辑载体来转化棉花,创造出带有特定标记的棉花雄性不育系。
本发明的技术方案如下所述:
一种同时被敲除了三个基因的且花药表面带有黑斑标记的棉花雄性不育系的创制方法,包括下列步骤:
(1)在陆地棉中筛选得到可以作为标记的ghems1基因的三个dna片段,所述的dna片段分别如seqidno:1(命名为ghir_a08g010860),seqidno:2(命名为ghir_d08g010810)和seqidno:3所示(命名为ghir_a09g018830),以三个dna片段的保守区域设计共有的靶位点sgrna1和sgrna2;
其中sgrna1的核苷酸序列如下所示:
sgrna1:aacctgaccgctctttacat;
sgrna2的核苷酸序列分别如下所示:
sgrna2:cagtgaaattgttggagtca;
(2)利用重叠延伸的pcr方法将sgrna1-guidrna和sgrna2-guidrna串连,然后通过一步克隆法构建到crispr/cas9载体prgeb32-ghu6.7-nptⅱ(wangetal.2018),得到高效转化载体ghu6.7::sgrna1-sgrna2。
(3)利用农杆菌介导的方法转化棉花(棉花品种如陆地棉品种豫668,但是实施本发明不限于该棉种和品种),得到带有标记的棉花雄性不育系。
(4)通过t0代单株测序,page(polyacrylamidegelelectrophoresis)胶和高通量测序验证ghems1基因产生编辑并无脱靶现象。
(5)通过对t1代单株观察,测序鉴定本发明的基因编辑方法的可遗传性。
(6)通过半薄切片和tunel染色实验方法(minetal.2013),得到一种突变体ghems1,该突变体无法形成中间层和绒毡层,因而小孢子母细胞由于得不到营养进而降解。
(7)将t1代完全不育材料与野生型(即非转基因)陆地棉品种杂交获得杂交f1代,其杂交后代表现该不育系雌性发育正常。
本发明克隆的的sgrna1、sgrna2可在培育棉花雄性不育系中应用。
本发明的有益效果如下:
(1)本发明发现了利用构建的ghu6.7::sgrna1-sgrna2载体同时突变棉花中ghir_a08g010860基因片段、ghir_d08g010810基因片段、和ghir_a09g018830三个基因片段可以创造出棉花雄性不育植株。
(2)本发明鉴定的sgrna1和sgrna2序列具有较高特异性核编辑效率。
(3)本发明中的雄性不育系带有黑斑标记,容易识别,便于早期确定不育株。且雄性不育系黑斑表型可稳定遗传至下一代,不育性稳定。
(4)本发明的雄性不育系败育率为100%。
附图说明
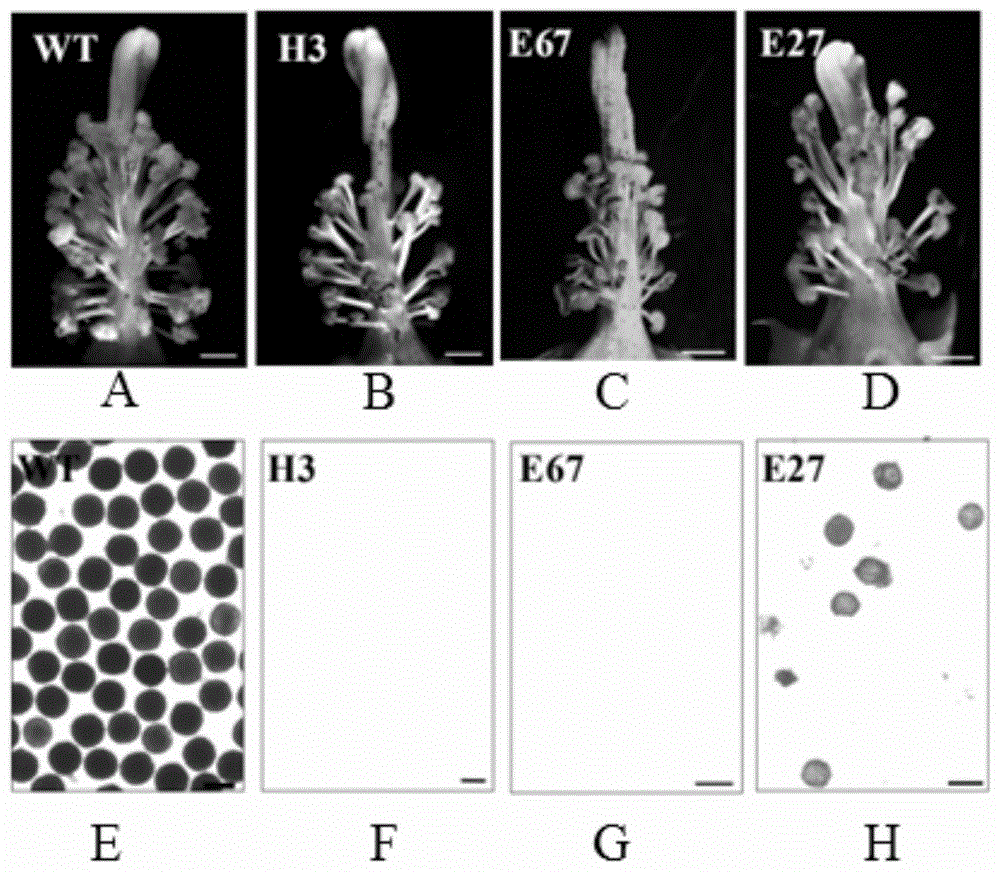
图1:是本发明的t0代单株表型图。附图标记说明:图1中的a图:wt(wildtype)野生型单株花药,图1中的b图,图1中的c图,图1中的d图分别为h3、e67、e27突变体单株花药;图1中的e、图1中的f、图1中的g、图1中的h分别为wt、h3、e67、e27花粉活力ttc染色。
图2:是本发明的h3单株序列编辑图。附图标记说明:sgrna靶位点和pam区域分别以绿色和红色背景突出显示。长度在右侧显示。省略的核苷酸由虚线表示,并且其长度在上方标记。
图3:是本发明的t0、f1代植株编辑page(polyacrylamidegelelectrophoresis)检测。附图标记说明:图3中的a图:ghir_a08g010860、ghir_d08g010810基因的sgrna1和sgrna2侧翼片段,图3中的b图:ghir_a09g018830基因的sgrna1和sgrna2侧翼片段。
图4:是本发明的ghems1编辑f1代不同时期表型变化。与野生型相比,f1代植物的花药在第7阶段出现黄点,在第10阶段和第13阶段逐渐加深,最终形成坏死样的黑点。附图标记说明:图4中的a图、图4中的b图、图4中的c图分别为野生型在第7、10、13时期的花药,图4中的d图、图4中的e图、图4中的f图分别为野生型在第7、10、13时期的单个花药;图4中的g图、图4中的h图、图4中的i图分别为突变体h3-3在第7、10、13时期的花药,图4中的j图、图4中的k图和图4中的l图分别为突变体h3-3在第7、10、13时期的单个花药。比例尺:200μm。
图5:是本发明的h3-3f1代与野生型花药组织切片。附图标记说明:图5的上半部是wt,图5的下半部是h3-3。其中:图5中的a图、图5中的b图、图5中的c图、图5中的d图、图5中的e图、图5中的f图分别为野生型在2~3mm、5~6mm、6~7mm、7~8mm、9~10mm、>24mm时期的花药切片,图5中的g图、图5中的h图、图5中的i图、图5中的j图、图5中的k图、图5中的l图分别为突变体h3-3在2~3mm、5~6mm、6~7mm、7~8mm、9~10mm、>24mm时期的花药切片。比例尺:100μm
图6:是本发明的h3-3f1代细胞凋亡tunel染色。附图标记说明:图6中的a图、图6中的b图分别为野生型在7~8mm、9~10mm时期的花药tunel染色,c、d分别为突变体h3-3在7~8mm、9~10mm时期的花药tunel染色,。比例尺:100μm。
dms:退化的小孢子;e,表皮;en,内皮;msc,小孢子母细胞;mp,成熟花粉;ml,中间层;msp,小孢子;t,绒毡层;tds,四分体;wt,野生型。
具体实施方式
对序列表的说明:
序列表seqidno:1是本发明的基因片段(ghir_a08g010860基因片段)的核苷酸序列。长度为3828bp。
序列表seqidno:2是本发明的基因片段(ghir_d08g010810基因片段)的核苷酸序列。长度为3828bp。
序列表seqidno:3是本发明的基因片段(ghir_a09g018830基因片段)的核苷酸序列。长度为3753bp。
序列表seqidno:4是ghems1基因三个保守区域共同靶位点sgrna1的核苷酸序列。
序列表seqidno:5是ghems1基因三个保守区域共同靶位点sgrna2的核苷酸序列。
实施例1:sgrna的设计
1.ghems1基因的sgrna设计
选陆地棉ghems1(ghir_a08g010860,ghir_d08g010810和ghir_a09g018830)的保守区域设计共有的靶位点sgrna1(其序列如序列表seqidno:4所示)和sgrna2(其序列如序列表seqidno:5)。
表1ghems1基因的sgrna序列
表1说明:加粗处的碱基为pam(前间区序列邻近基序,protospaceradjacentmotifs)。
实施例2:crispr/cas9载体构建
利用重叠延伸的pcr方法将两个sgrna与trna、grna进行串连,然后用一步克隆法构建到crispr/cas9载体上。
设计如下所述的引物序列:
正向引物p1:aagcatcagatgggcaaacaaagcaccagtggtctag,
反向引物p2:atgtaaagagcggtcaggtttgcaccagccgggaat;
正向引物p3:aacctgaccgctctttacatgttttagagctagaaata,
反向引物p4:tgactccaacaatttcactgtgcaccagccgggaat;
正向引物p5:aagcatcagatgggcaaacaaa,
反向引物p6:ttctagctctaaaactgactccaacaatttcactg;
具体操作步骤:
(1)第一次pcr扩增出两个小片段,将其中第一个片段用引物p1、p2连接成trna、sgrna1,第二个片段用引物p3、p4连接sgrna1、grna、trna、sgrna2。pcr反应体系如表2所示。
表2pcr反应体系
pcr条件:95℃5min,95℃30sec,55℃30sec,72℃20sec,共3个循环;然后95℃30sec,60℃30sec,72℃20sec,共27个循环,72℃5min,15℃,保存。
(2)利用重叠延伸pcr将片段1和2拼接,产生片段3pcr的反应体系如表3所示。
表3pcr反应体系
pcr条件:95℃5min,95℃30sec,59℃30sec,72℃20sec,共28个循环,72℃5min,15℃保存。
(3)载体酶切(bsai购自北京neb公司,货号:r3733l),酶切体系如表4所示。
表4酶切体系
37℃下孵育5h。
(4)连接(clonexpressentrystepcloningkit购自南京诺唯赞生物科技有限公司)
37℃孵育30min,冰上静置5min。
(5)转化至gv3101农杆菌感受态,卡那霉素筛选,pcr阳性鉴定(为本领域的常规方法),u6-7f作正向引物测序(gcrich)。
u6-7f:tgtgccactccaaagacatcag
实施例3:利用crispr-cas9系统转化棉花
将上述构建好的载体通过农杆菌介导的转化方法导入棉花宿主细胞。具体转化步骤如下:
(1)挑选饱满、健康的陆地棉豫668(由河南省农业科学院选育,惠赠,但是实施本发明不限于该品种)种子,剥去种皮后,用0.1%的hgcl2浸泡10min,再用无菌水洗涤3次,将灭菌后的棉种,接种于无菌苗萌发培养基上,在黑暗条件下,置于28℃恒温箱中培养4-6d;
(2)取无菌苗下胚轴,切成0.5-0.8cm小段接种于0.5od值的用农杆菌活化培养基悬浮的农杆菌菌液中,侵染10mim后,用无菌滤纸吸干下胚轴表面的菌液;
(3)将下胚轴接种于共培养培养基中,在21℃下共培养48h;再转到选择培养基中,每1个月继代培养1次,直到获得胚性愈伤组织;将胚性愈伤组织转入分化培养基,获得大量胚状体;
(4)将获得的胚状体转移到胚萌发及成苗培养基,直到获得转基因植株幼苗。
转化用的培养基成分及配制方法:
无菌苗萌发培养基:1/2ms大量元素,15g/l葡萄糖,2.5g/l的phytagel;ph=6.1-6.2。
愈伤组织诱导培养基:msb,24-d0.1mg/l,kt0.1mg/l,3%glucose,0.3%phytagel;ph=5.85-5.95。
农杆菌活化培养基:胰化蛋白胨5g/l,nacl5g/l,mgso4.7h2o0.1g/l,kh2po4,0.25g/l,甘露醇5g/l,甘氨酸1.0g/l;ph=5.85-5.95。
共培养培养基:msb+2,4-d0.1mg/l,kt0.1mg/l,50mg/las,3%glucose,0.25%phytagel,ph=5.8。
选择培养基:msb,2,4-d0.1mg/l,kt0.1mg/l,3%glucose,0.3%phytagel,卡那霉素50mg/l,头孢霉素400mg/l;ph:5.85-5.95。
分化培养基:在msb培养基中去掉nh4no3,将kno3用量加倍,gln1.0g/l,asn0.5g/l,iba0.5mg/l,kt0.15mg/l,3%glucose,0.25%phytagel;ph=6.1-6.2。
生根培养基:1/2ms无机盐+b5有机物,15g/l葡萄糖,2.5g/l的phytagel;ph=5.90-5.95;
msb:ms培养基+b5维生素。
实施例4:检测crispr-cas9对ghems1基因的编辑效果
设计引物同时扩增两个sgrna的侧翼序列,产物采用桑格测序。扩增用的相关引物序列如下所示:正向引物a08d08-f:cctggctaacaatttccttt,反向引物a08d08-r:atatcaactggcagagtccc;正向引物a09-f:cttcagtctttaacctccct,反向引物a09-r:aactggcaacgtaccttcta。统计结果如表6所示。在三个单株中三个基因编辑效率较高。图2展示在h3单株中三个基因的编辑情况,碱基缺失在3~27碱基范围内。图3利用page对h3(t0代)、f1代检测sgrna编辑情况,扩增用的相关引物序列如下所示:正向引物a08d08-1-f:tatcccgcctgaaattggtgag,反向引物a08d08-1-r:aatttcaggtggtattttccctgta;正向引物a08d08-2-f:agagtatctttcagagcttcca,反向引物a08d08-2-rcatcaaactatttgaactccat;正向引物a09-1-f:catcccgccagatgttggtgaa,反向引物a09-1-r:gaggtggcaactgcccactg;正向引物a09-2-f:cgagtatcttgctgagcttccg,反向引物a09-2-r:tccatcaaagtttctgaactccac。结果表明f1代与t0代具有相同的片段,但是由于wt授粉,产生了一些新的编辑类型,例如在h3-3中,四个sgrna都产生了新的编辑类型。
表6不同株系编辑效率统计
实施例5:表型及突变遗传验证
图1为再生植株的不育表型图,从图1中可以看出:h3与e67完全无花粉,e27花粉畸形且不育,而野生型ttc染色后活力较好。图4是本发明的ghems1编辑h3×wt的f1代不同时期表型变化。与野生型相比,f1代植株的花药在第7时期出现黄点,在第10和第13时期颜色逐渐加深,最终形成坏死的黑点。与t0代h3表型一致。由图5所示,对野生型和h3-3单株的6个时期进行花药切片观察,发现h3-3无法形成中间层和绒毡层,在>24mm时期,小孢子母细胞降解,最终导致h3-3无花粉形成导致败育。图6中tunel染色结果显示野生型在7~8mm、9~10mm绒毡层和花粉具有细胞胞凋亡信号,而突变体在9~10mm时期出现小孢子母细胞降解信号。
ttc花粉活力染色观察具体步骤:
取8克ttc(2,3,5-三苯基氯化四氮唑,分子量334.8)溶于1l磷酸缓冲液(三水合磷酸氢二钾26.6g,磷酸氢二钾10.2g)配制成2%的工作液,4℃避光保存。取用时分装于1.5ml离心管中,将花药塞入,37℃培养箱中反应30min后取出在显微镜下观察。
半薄切片具体步骤如下:
(1)取新鲜的样放在2ml的离心管里,加入2mlfaa固定液,将取到的材料浸泡在固定液中,放在4℃冰箱中2-4h,尔后将装有材料的离心管抽真空处理1h,分4次,每次15min。
(2)样本脱水,将样本分别浸入梯度浓度的乙醇洗液(50%,70%,80%,90%),室温下每步放置15min。
(3)样本染色,将样品侵入含有伊红的95%浓度的乙醇溶液里,4℃放置过夜。
(4)样本洗涤,将样品浸入无水乙醇里,清洗三四遍,每次清洗需要换新的管子。
(5)样本脱水,将样品放入100%乙醇(含无水cuso4)中放置15min,此步操作重复3次。
(6)样本洗涤,将样品浸入无水乙醇里,清洗三四遍
(7)将经脱水后的组织块放入200ul离心管中,加入100ul的预渗透液先配制预渗透液(v无水乙醇:v渗透剂(常规试剂)=1:1),预渗透12h。
(8)预渗透完成后,然后加入100ul的渗透液,渗透24个小时。
(9)取1.2ml渗透液加入到2ml离心管中,然后加入harderⅱ,在震荡器上将二者充分混匀。将混匀后的包埋剂分装到十个200ul离心管中,每管120ul,加完后看一下管底,要将产生的气泡去掉。之后,每管中放入一个组织块。并不时用解剖针沾取包埋剂,待包埋剂可以粘稠到可以撤出丝状时,用解剖针调整组织块的位置,使其竖直地站在包埋块中,经多次调整后,确保组织块的位置不再改变,将管盖合上。将样品放在37℃恒温箱中处理24h,通风橱中吹12h.
(10)用单面刀片在装有包埋块的离心管管壁上沿长轴方向划几道划痕,划痕要深至触到包埋块。再将管底切除,注意不要切到包埋块。然后用牙签从底部将包埋块从离心管中捅出,修块。
(11)修完块,用切片仪切片,将切好的切片放在有多聚赖氨酸的载玻片上,烘干机上展片,烘干。
(12)将烘干好的切片放入甲苯胺蓝中染色,染色时间90s,之后放在通风橱里吹干。
(13)切片吹干之后,用移液器取中性树胶滴加在烘干的切片上,加盖coverslip,放置37℃,12h以上,树胶凝固之后就可以观察了。
tunel染色具体步骤如下:
(1)组织切片(附着于显微镜载玻片上的),将载玻片浸入装有新鲜二甲苯的染色缸,室温放置5分钟。重复一次,总共用二甲苯洗两次。
(2)样本洗涤,将载玻片浸入装有100%乙醇的染色缸,室温放置5分钟。
(3)样本顺序复水,将样本浸入梯度浓度的乙醇洗液(100%,95%,85%,70%,50%),室温下每步放置3分钟。
(4)样本洗涤,将载玻片浸入0.85%nacl,室温放置5分钟。
(5)样本洗涤,将载玻片浸入pbs,室温放置5分钟。
(6)组织切片固定,将载玻片浸入4%配制于pbs中的不含甲醇的多聚甲醛溶液,室温放置15分钟。
(7)样本洗涤,将载玻片浸入pbs,室温放置5分钟。重复一次,总共用pbs洗两次。
(8)去除组织上的液体,并将载玻片放置于一个平面。用pbs以1:500稀释蛋白酶k储液(10mg/ml),配成20μg/ml的蛋白酶k溶液。在每片载玻片上加上100μl20μg/ml的蛋白酶k以覆盖组织切片。室温孵育载玻片8-10分钟。
(9)样本洗涤,将载玻片浸入装有pbs的染色缸,室温放置5分钟。
(10)组织切片洗涤后固定,将载玻片浸入4%配制于pbs中的不含甲醇的甲醛溶液,室温放置5分钟。
(11)样本洗涤,将载玻片浸入pbs,室温放置5分钟。
(12)轻叩载玻片以去掉多余的液体。用100μl平衡缓冲液覆盖细胞。在室温下放置5-10分钟平衡。
(13)在平衡细胞的同时,在冰上解冻核苷混合物,并且依照表1,准备足够量的用于所有实验的和可选阳性对照反应的rtdt孵育缓冲液。对于面积小于5cm的一个标准反应,其体积是50μl,用50μl乘上实验和阳性对照反应的数目来确定所需rtdt孵育缓冲液的总体积。对于表面积更大的样本,可成比例的增大试剂体积。在平衡后的区域周围用吸水纸吸掉100μl平衡缓冲液中的大部分,然后在5cm面积的细胞上加上50μlrtdt孵育缓冲液。
(14)把塑料盖玻片盖在细胞上以保证试剂的平均分布。在湿盒的底部放上用水浸湿的纸巾。将载玻片置于湿盒内,在37℃孵育60分钟发生加尾反应。将湿盒用铝箔纸包裹以避免光照。
(15)用去离子水1:10稀释20xssc,加入足量配好的2xssc装满一个标准的染色缸(40ml)。移除塑料盖玻片,将载玻片浸入染色缸中的2xssc,室温放置15min以终止反应。确保20xssc的全部盐类在稀释之前都是溶解的(第9步)。
(16)洗涤样本,将载玻片浸入新鲜的pbs中,室温放置5min。重复两次,总共洗三次以去除未掺入的荧光素-12-脱氧三磷酸尿苷。
(17)样本在染色缸中染色,在黑暗中将载玻片浸入装有40ml碘化丙啶溶液的染色缸,室温放置15分钟,此处碘化丙啶溶液是用pbs新配稀释到1μg/ml的。
(18)洗涤样本,将载玻片浸入去离子水中,室温放置5min。重复两次,总共洗三次。
(19)滴干载玻片上多余的水并且用吸水纸擦拭细胞周边的区域。
(20)如第19步所述马上分析样本。(21)用橡胶胶水或透明指甲油封边,并晾干5-10min。
(22)在荧光显微镜下分析样本,用标准的荧光过滤装置在520±20nm的荧光下观察绿色荧光;在>620nm下观察碘化丙啶的红色荧光,以及在460nm观察蓝色的dapi。如有必要,可将载玻片在4℃黑暗条件下存放过夜。
主要参考文献:
1.chen,r.,q.xu,y.liu,j.zhang,d.ren,g.wang&y.liu(2018)generationoftransgene-freemaizemalesterilelinesusingthecrispr/cas9system.frontplantsci,9,1180.
2.min,l.,l.zhu,l.tu,f.deng,d.yuan&x.zhang(2013)cottonghckidisruptsnormalmalereproductionbydelayingtapetumprogrammedcelldeathviainactivatingstarchsynthase.plantj,75,823-35.
3.singh,m.,m.kumar,m.c.albertsen,j.k.young&a.m.cigan(2018)concurrentmodificationsinthethreehomeologsofms45genewithcrispr-cas9leadtorapidgenerationofmalesterilebreadwheat(triticumaestivuml.).plantmolecularbiology,97,371-383.
4.wang,p.,j.zhang,l.sun,y.ma,j.xu,s.liang,j.deng,j.tan,q.zhang,l.tu,h.daniell,s.jin&x.zhang(2018)highefficientmultisitesgenomeeditinginallotetraploidcotton(gossypiumhirsutum)usingcrispr/cas9system.plantbiotechnologyjournal,16,137-150.
5.zhou,h.,m.he,j.li,l.chen,z.huang,s.zheng,l.zhu,e.ni,d.jiang,b.zhao&c.zhuang(2016)developmentofcommercialthermo-sensitivegenicmalesterilericeaccelerateshybridricebreedingusingthecrispr/cas9-mediatedtms5editingsystem.scirep,6,37395。
序列表
<110>华中农业大学
<120>一种带有标记的棉花雄性不育系的创制方法
<141>2019-12-13
<160>5
<170>siposequencelisting1.0
<210>1
<211>3828
<212>dna
<213>棉花(gossypiumhirsutum)
<220>
<221>gene
<222>(1)..(3828)
<400>1
atggagtttaagcttttgtttcacttcctctgtttccttcatttgttgttatcgatatca60
ggggcaacgatagagcaagcggacggaaacccagacagggaagctctggtttccttcaaa120
agagggcttcaaaaccctcattttctatcatcatggaaccaaagaattcatttctgcaag180
tgggatggtgtcacttgtcggctcggccgagtcacctcgctttctcttccatctcggtcc240
ttgacaggttctctctctccgtccctttcgtccctctcaaacctcaccctcctagacctg300
tcctctaactccttctttggccaaattccgactgagttggccgagttgactctcctcaag360
accctcaaactcgggtctaactctttcaccggaaacatcccttccgagctgggcagcctg420
aatgcacttcggacgctagaactttccaccaatgcactcactggcacagtgccggctaag480
cttggacagttaactcagctacagttcttggacctggctaacaatttcctttcaggttct540
ttcccttcaacactctttgaaaaccttcagtctttaacctctctggatatttccaacaat600
tctttctctggtaatatcccgcctgaaattggtgagctaaaaaacctgaccgctctttac660
attggaatcaaccaatttacagggaaaataccacctgaaattggtaaactttcactcctt720
gagaactttttttcaccctcttgttccataacaggacctttgcctaaacaactatccaat780
ctaaaatcactcaccaagctcgacctttcttataacccattgaaatgttccattccaaag840
tctataggaaagttacagaatttgactatattgaacctcgtctacaccgagctcaatggt900
tcaataccagcggagcttgggaactgccggaatttgatgatgttgatgctgtctttcaat960
tcgctgtccgggtccttgcctgaggagctttcaagtttgcctatgctgacgttttctgct1020
gaaacgaaccagctttctgggccactgccaccttggcttgggaaatggacccaggtggag1080
tctttgctactttcaaacaaccacttttcaggtaacatccctcctgaaattgagaattgc1140
tcaaggcttaagcaccttagtttgagcaataacaagttgtcgggttcgataccaagagag1200
ttatgcaatgcaaaatcactctttgaggttgatctcgatggtaataacctttcggttacc1260
attgaggatgtgtttgtgaactgtagaaatcttgctcagttggttttgctcaataatcac1320
attaatggttcaatcccagagtatctttcagaacttccattgatggttattgaccttgac1380
tccaacaatttcactggtaccattcctgtgagtttatggagttcaaatagtttgatggag1440
ttttctgctggaaataatatgctagaggggactctgccagttgatattggaaatgcggtt1500
acattggaaacgcttgttcttagcggtaatcgtttgaagggaaatataccaaaggagatc1560
ggcaatctcacagctctttctgtgctaaacttgaactcgaatttcttggaaggaaatata1620
cctgttgaaattggatactgcaaagcattgacaacgctggatcttggaaacaacaatttc1680
agtggatcaattcccatggaacttgcagaccttgatcagctgcagtgcctggttctttct1740
cacaataatctatccggttccatcccttggaaaccatcttcatatttccatcaggctaat1800
ctgcccgacttgagctttgtgcagcatcatggagtattcgatctgtcacacaataggttg1860
actggtcccatacctgaagaattgggaaactgtgcagtggtggtggatcttcttctcaac1920
aacaacatgcttactggctggattccaggatcactttctcgtttgacaaatcttacaact1980
ttggatttgtctggaaatttgctgacgggtcctattcctgttgaattcggtgactctctc2040
aagcttcagggcttgtatttggggaataaccagcttacaggaaccatctctgaaagctta2100
ggccgtgtgggtagtttggtaaagttaaatttgactggcaataggttatctggtgtagtt2160
cctgcaagttttgggaacttgaaagaacttactcacttggatttaagcaataataagctt2220
gccggtgagctaccttcgtcattgtcccagatgcttaacctggtggggctctatgttcag2280
aagaataggcttgctggtgagatacataacctgttctcaaattccgtgtcttggaagatt2340
gaagacttgaatttgagtaataacattttctatggaagcttgccccaatctttgggcaat2400
ttgtcttatttgatgtacttggatcttcatggaaataaatttagtggtgccattccttca2460
gaaattggtaacttgatgcaactagagtattttgatttatccagaaacagattttccgga2520
cagattccagcggaagtgtgtggtttgttcagtctgttctacttgaatctagcagaaaac2580
cggttgggaggacctgtgcccagaaatggtatttgtcaaaacctgtcaaaaatctttcta2640
gctgggaacaatgatctgtgtggaagaattacaggtttggagtgccagatcagatgttct2700
gaaagatcttctttgttaaatgcctggggccttgcagggattgtggctgcaagtgtcttt2760
atcatttttgcttcagcctttgctgtacgcagatggattttgcggagtggccagctgagt2820
gatccagaagaaattgaggaaagcaaattaaacaacttcctggatcaaaatctctgcttc2880
ttgagcagcagcagcaggtcaaaggagccgctgagcatcaatatagcgacatttgagcag2940
ccactcctgaaactcacactcggtgacatccttgagggaacccatcatttttgcaagacc3000
aacataatcggagatggaggctttggaactgtttacaaagctaagttgcctagtgggaag3060
actgttgcagttaagaagctaagccaggccaaaacacaaggcaatcgagaattcatagct3120
gaaatggaaaccttgggaaaggtgaagcaccaaaatcttgttccattgcttggatactgt3180
tctttgggtgaggagaagctccttgtttacgaatacatgatcaatggaagtttggatctt3240
tggttgagaaatcgcagtggagctcttgatgtccttgattggtccaaacgcttcaaaatc3300
gctgttggtgctgctcggggactagcatttcttcaccatggtttcattccccacatcatc3360
catagagatatcaaagccagcaacatcttactcagtgaagactttgaagcaaaagttgcg3420
gacttcgggctggcaagactgattagtgcttgtgaaactcatgttagtactgatatagca3480
gggacttttggttacattccaccagaatatggacagagcggtaggtccactacaaaggga3540
gatgttttcagttttggtgtgatactgctggaattggtgactgggaaagagccaacaggc3600
cctgaatttaaagagattgaaggagggaatttagttggatgggtgactaagaagataaaa3660
aagggtcaggctgcagatgttttagatcctgtggttatgggtgtggattcaaagcagatg3720
atgcttcaggtgctcagtattgctgcagtttgcctggccgagaatcctgctaacagacct3780
accatgcttcaagtgttgaagctcctcaaagggatccacaaggagtaa3828
<210>2
<211>3828
<212>dna
<213>棉花(gossypiumhirsutum)
<220>
<221>gene
<222>(1)..(3828)
<400>2
atggagtttaagcttttgtttcacttcctctgtttcctctatttgttgttaccgatatca60
ggggcaacgagagagcaagcagacggaaacccagataggggagctctggtttccttcaaa120
agagggcttcaaaaccctcattttctatcatcatggaaccaaaaaatccatttctgcaag180
tgggatggtgtcacatgtcggctcggccgagtcacctcgctttctcttccatctcggtcc240
ttgacaggttctctctctccgtccctttcgtccctcttaaacctcaccctcctagatctg300
tcctctaactccttctttggccaaattccgactgagttggccgagttgactctcctcgag360
accctcaaactcgggtctaactctttcaccggaaatatccctcccgagctgggcagcctg420
aatacacttcgtacgctagaactttccaccaatgcactcactggcacagtgccgactaag480
cttggacagttaactcagctacagttcttggacctggctaacaatttcctttcaggttct540
ctcccttcaacactctttgaaaaccttcagtctttaacctctctggatatttccaacaat600
tctttctctggtaatatcccgcctgaaattggtgagctaaaaaacctgaccgctctttac660
attggaatcaaccaatttacagggaaaataccacctgaaattggtaaactttcactcctt720
gagaactttttttcaccctcttgttccataacaggacctttgcctgaacaactatccaat780
ctaaaatcactcaccaagctcgacctttcttataacccattgaaatgttccattccaaag840
tctataggaaagttacagaatttgactatattgaacctcgtctacaccgagctcaatggt900
tcaataccagcggagcttgggaactgccggaatttgatgatgttgatgctgtctttcaat960
tcgctgtccgggtccttgcctgaggagctttcaagtttgcctatgctgactttttctgct1020
gaaacgaaccagctttctgggccactgcccccttggcttgggaaatggaaccaggtggag1080
tctttgctactttcaaacaaccacttttcaggtaacatccctcctgaaattgagaattgc1140
tcaaggcttaagcaccttagtttgagcaataacaagttgtcgggttcgataccaagagag1200
ttatgcaatgcagaatcactctttgaggttgatctcgatggtaataacctttcgggtacc1260
attgagaatgtgtttgtgaactgtagaaatcttgctcagttggttttgcttaataatcac1320
attaatggttcaatcccagagtatctttcagagcttccattgatggttattgaccttgac1380
tccaacaatttcactggtaccattcctgtgagtttatggagttcaaatagtttgatggag1440
ttttctgctggaaataatatgctagaggggactctgccagttgatattggaaatgcggtt1500
acattggaaactcttgttcttagcggtaatcgtttgaagggaagtataccaaaggagatc1560
ggcaatctcacagctctttctgtgctaaacttgaattcgaatttcttggaaggaaatata1620
cctgttgaaattggagactgcaaagcattgacaacggtggatcttggaaacaacaatttc1680
agtggatcaattcccatggaacttgcggaccttgatcagctgcagtgcctggttctttct1740
cacaataatctatccggttccatcccttggaaaccatcttcatatttccatcaggctaat1800
ctgcccgacttgagctttgtgcagcatcatggagtattcgatctgtcacacaataggttg1860
actggtcccatacctgaagaattgggaaactgtgctgtagtggtggatcttcttctcaac1920
aacaacatgcttactggcaggattccaggatcactttctcgtttgacaaatcttacaact1980
ttggatttgtctggaaatttgctgagaggtcctattcctgttgaattcggtgactctctc2040
aagcttcagggcttgtatttggggaataaccagcttacaggaaccatctctgaaagctta2100
ggtcgtgtgggtagtttggtaaagttaaatttgactggcaataggttatctggtgtagtt2160
cctgcgagttttgggaacttgaaagagcttactcacttggatttaagcaataataagctt2220
gccggtgagctaccttcgtcattgtcccagatgcttaacctggtggggctctatgttcag2280
aagaataggctttctggtgagatacataacctgttctcaaattccgtgtcttggaagatt2340
gaagacttgaatttgagtaataacattttctttggaagcttgcctcaatctttgggcaat2400
ttgtcttatttgatgtacttggatcttcatggaaataaatttactggtgccattccttca2460
gaaattggtaacttgatgcaactagagtattttgatgtatctggaaacagattatccgga2520
cagattccagaggaagtgtgcagtttgttcagtctgttctacttgaatctagcagaaaac2580
cggttgggaggacctgtgcccagaaatggtatttgtcaaaacctgtcaaaaatctttcta2640
gctgggaacaatgatttgtgtggaagaattacaggtttggagtgccagatcagaagttct2700
gaaagatcttctttgttaaatgcctggggccttgcagggattgtggctgcaagtgtcttt2760
atcatttttgcttcagcctttgttgtacgcagatggattatgcggagtggccagctgagt2820
gatccagaagaaattgaggaaagcaaattaaacaacttcctggatcaaaatctctgcttc2880
ttgagcagcagcagcaggtcaaaggagccgctgagcatcaatatagcgacatttgagcag2940
ccactcctgaaactcacactcggtgacatccttgagggaacccatcacttttgcaagacc3000
aacataatcggagatggaggctttggaactgtttacaaagctaagttgcctagtgggaag3060
actgttgcagttaagaagctaagccaggccaaaacacaaggcaatcgagaattcatagct3120
gaaatggaaaccttgggaaaggtgaagcaccaaaatcttgttccattgcttggatactgt3180
tctttgggtgaggagaagctccttgtttacgaatacatgatcaatggaagtttggatctt3240
tggttgagaaatcgcagtggagctcttgatgtccttgattggtccaaacgcttcaaaatc3300
gctgttggtgctgctcggggactagcatttcttcaccatggcttcattccccacatcatc3360
catagagatatcaaagccagcaacatcttactcagtgaagactttgaagcaaaagttgcg3420
gacttcgggctggcaagactgattagtgcttgtgaaactcatgttagtactgatatagca3480
gggacttttggttacattccaccagaatatggacagagcggtaggtccactacaaaggga3540
gatgttttcagttttggtgtgatactgctggaattggtgactgggaaagagccaacaggc3600
cctgaatttaaagagattgaaggagggaatttagttggatgggtgactaagaagataaaa3660
aagggtcaagctgcagatgttttagatcctgtggttatgagtgtggattcaaagcagatg3720
atgcttcaggtgctcagtattgctgcagtttgcctggccgagaaccctgctaacagacct3780
accatgcttcaagtgttgaagctcctcaaagggatccacaaggagtaa3828
<210>3
<211>3753
<212>dna
<213>棉花(gossypiumhirsutum)
<220>
<221>gene
<222>(1)..(3753)
<400>3
atggagtctaagctttcgtttcagttcctctgttttctacatctgttgttggtatttata60
tgggggggcattagagcgcaagaagagccaaacccagaaagggaagccctcatttccttc120
agaacaagacttcaaaatcctcatctcctttcttcatggaaccaacaactccatcactgc180
caatgggatggtgtgatgtgccaactcggtcgagtcaccgttcttgctcttacatctcgc240
tccttgaaaggtcctctctcttcctctctttcctcactctccagcctcacccttctggac300
ttgtcctccaactttttgtttggccaaataccggttgagttggctgagttgactatgctc360
cgcatcctcaaactcgggtccaactttttcacgggaaaaatccctccggaactgggcagc420
ctgaaagcccttcgtacgctagaactttcctccaatggactcagtggcacagtgccggct480
cagctggggcagttgactcacttagagttcttggacctggcaaacaatttcctttcagat540
gttggtgaacttaaaaacctgaccgctctttacattggcattaatcaattcagtgggcag600
ttgccacctcaaattggtaacctttcactgctcgagaacttcttttcaccctcttgttcg660
ataactgggcccttacctgaagaactattcagtctacagctgcttaccaagcttgacctt720
tcatataacccattgaaatgttccattccaaagtctacaggaaagttaaagaatttgagt780
atattgaaccttgtttacactgagctcaatggttcaataccagcagagcttgggaactgc840
cagaacttgatgatgttgatgctgtctttcaattccttatccggctctttgcctgaggag900
ctttccaacttgcctatgttgactttttccgctgaaaagaatcagctctctgggccattg960
ccgccttggcttgggaaatggatcaaaatggagtccttgctgctttcaagcaaccgcttt1020
tcaggtaacatcccacctgagttagggggttgttccatgctgaagcaccttagtttgagc1080
aataacatcttgtcgggtcagataccaagagagctatgtaatgcggaatcacttttggag1140
attgatcttgacggtaataacctttcggggaccattgaaaatgtttttgtcaagtgtaga1200
aatctcaaccagttggttttggttaacaatcacatcaatggttcagtgcccgagtatctt1260
gctgagcttccgttgatggttattgaccttgactccaacaatttcactggcaggatacct1320
gtgagtttgtggagttcagaaactttgatggagttttctgctggaaataatatgctagaa1380
ggtacgttgccagttgaaattggtaatgctgttacattgcagacccttgttcttaatcgt1440
aatcttttgaggggcagaataccgaaggagattggcaatctcacagctctctctgtgctt1500
aacttgaactcgaatttcctagaaggatatataccagttgaaattggagactgcaaagga1560
cttacaacgttggatcttggaaacaacaatttgagtggaccaatcccggtggaacttgct1620
gatctgggactgctgcagtgcctggttctctcgcacaataatctatcaggttcaatccct1680
cggaagccatcattgtatttccatcaggctaatatgcccgatttaagctttgttcagcat1740
catggagtattcgatttgtccaacaatagactgactggtccaatacctgaagaattgggg1800
aactgtgttgtggtggtggatcttctgcttagcaaaaacatgcttaccggcaacattcca1860
ggatcactttctcgattgacaaatcttacaactttggatttgtctgggaatgtgctaaca1920
ggttcaattcctgtagaatttggtgactctctcaaccttcagggcttgtatttggggaat1980
aaccggcttactggaaccatccctggaagcttaggccgtgttggtagtttggtaaagtta2040
aacttgactggcaataagttatctggtgttattcctgcaagttttgggaacttgaaagag2100
cttactcacttggatttaagccgtaatgatcttactggtgagctaccttcttcactgtcc2160
cagatgcttaacctggtggggctttatgttcagcagaataggctttctggtggggtaggt2220
aatctcttctccaatgccctctcttggaagattgaagacatgaatttgagtaataacatt2280
ttcaatggaaacttgccacaatctttgggcaatttgtcttatgcgacttacctggatctt2340
catgggaataagtttactggtgagatcccttcagaagttggtaacttgatgcaacttgaa2400
tattttgatgtatctggaaacagattatctggacagattccagagagagtatgcgggatg2460
ttcaatctattctacttgaatctggcagaaaaccggttggaaggtcctgtaccccgaact2520
ggtatttgccaaaacctgtccaggacatttctctctgggaacagtgatctctgtggcaga2580
attatgggtttagaatgccagatcagaagttttgacagatcttctttgttaagtgcttgg2640
ggccttgctgcggtcacagctgcaagtgtctttatcatttttacttcggcctttgctcta2700
cggagatggattatgagtagtagtcagcaatgtgatccagaagaaattgaggaaagtaaa2760
ctaaaatcctttatagatcaagagctctatttcttgagtagcagcaggtcgaaggagccg2820
ctgagcatcaatatagcgatattcgagcagccactcctgaaactgacactaggcgacatc2880
cttgagggcactagtcatttttgtaagacgaatgtgataggagatggaggctttggaacg2940
gtttacaaagctaagttgcctagtgggaagacagttgcggttaagaagctaagccaggca3000
aaaacgcagggccatcgagaattcatagctgaaatggaaaccttgggtaaggtgaaacac3060
caaaatcttgtcccattgcttggatactgttctttggccgaggagaaactccttgtttat3120
gaatacatgattaatggaagtttggatctgtggttgagaaaccgcaccggagcccttgat3180
gtccttgactggtccaaacgcttcaaaatcgcaataggtgctgcgcggggactggctttc3240
cttcaccatggtttcattcctcatattatccatagagatatcaaagccagcaacatctta3300
ctcagtgaagactttgaaccaaaagttgcggacttcgggctggcaagacttataagtgct3360
tgcgaaactcatgttagtactgacatagcagggacattgggttacatcccaccagaatat3420
ggacagagtggtagatgcactacgaagggagatgtttacagttttggtgtaattctactg3480
gaattggtgactggaaaagagccaacaggccctgattttaaagagattgaaggaggcaat3540
ttggttggttgggtttctaagaagatgagaatgggtgaagctgctgacgttttagatgca3600
atggttttgaatgtggactcaaagcagatgatgctcaaggtgctgagtattgctgaagtt3660
tgcttgtctgaaaatcctgctaatagaccaactatgcttcatgtgctgaagctccttaaa3720
gggatcaaggaggattacctgtctttctactag3753
<210>4
<211>20
<212>dna
<213>棉花(gossypiumhirsutum)
<220>
<221>gene
<222>(1)..(20)
<400>4
aacctgaccgctctttacat20
<210>5
<211>20
<212>dna
<213>棉花(gossypiumhirsutum)
<220>
<221>gene
<222>(1)..(20)
<400>5
cagtgaaattgttggagtca20
- 还没有人留言评论。精彩留言会获得点赞!