基于RNN的基因调控网络构建与动态差异性分析方法与流程
本发明涉及一种基于rnn的基因调控网络构建与动态差异性分析方法。
背景技术:
基因调控网络的建模与演化分析能够很好的挖掘基因表达数据中的深层信息,是当前生物信息学研究的重要领域和关键问题。二十世纪90年代以来,随着基因芯片技术的发展和二代测序技术的兴起,基因调控网络建模的研究取得了巨大进展。
基因调控网络建模主要根据基因表达数据推理网络中的调控关系,并表示为拓扑结构,属于依靠数据挖掘进行的逆向工程研究。构建基因调控网络首先需要确定网络模型,然后根据模型选择合适的建模算法。经典的网络模型包括布尔网络、关联网络、微分方程、贝叶斯网络。
(a)布尔网络。布尔网络对基因状态做了相应简化,用布尔函数代替了微分和导数描述基因间的相互关系。该模型的缺点在于不精确性,仅仅通过使用固定的逻辑规则刻画和反映基因间相互作用,并不能准确描述真实的基因调控网络拓扑结构,而且对基因数据进行离散化时不可避免的会造成很多重要的表达信息丢失。kauffman等人最先提出了布尔网络的分析框架模型,随后akusu等人对布尔网络在推理过程中的最少样本数进行证明。liang等人设计了reveal算法,在原有的离散化模型上尽可能少的减少信息损失。此外,lyla等人提出了一种新的概率布尔网络(pbn),这是对传统布尔网络的拓展,同时量化基因间作用关系和灵敏度从而解决模型选择过程中的不确定性,提高了模型的精确性。
(b)关联网络。关联网络的建模主要通过基因表达数据间的关联度实现。通常使用互信息、皮尔森相关系数等测度计算基因间的相似度,若基因对间的相似度高于某一阈值,则该基因对在网络中直接连通。butte等人首先利用互信息计算所有基因对之间的关联度,然后设置互信息阈值。后来发现,若基因对间具有相同或相近的调控机制,则两个基因的关联度较高,尤其是同一转录因子的靶基因或同一条生物通路上的基因。margolin等人提出arcane方法,通过信息论构建关联网络,该方法的优点是模型的建立简单易操作,但是构建的网络存在很多假阳性的边。为降低所构建网络结构的假阳性率,得到接近真实拓扑的调控网络,一般在计算基因对间的关联度时隔绝其它基因的影响。
(c)贝叶斯网络。贝叶斯网络(bn)通过局部概率的乘积来近似描述整体网络结构的复杂概率分布,属于概率图模型,将节点之间的连边表示为节点间存在的概率依赖关系。动态贝叶斯网络(dbn)是对静态贝叶斯网络模型的扩展,通过引入时间因素形成动态变化网络,更加真实地表示随机系统的动态性。基因调控网络本质上是一个复杂而连续的动态网络系统,所以在具体建模的时候,往往对dbn进行简化从而降低计算复杂度。dbn克服了静态bn有向无环的不足,更好地刻画了基因调控网络的动态特性,提高了模型的预测精度。norbert为了能够从基因扰动型实验数据中学习动态贝叶斯网络,利用离散化方法来对基因表达数据进行预处理,结合基因调控的负反馈与时延因素提出新的数据整合模型,利用并行算法加速构建基因调控网络。
随着2006年hinton教授在《科学》上的一篇文章,深度学习拉开帷幕,并在各个领域表现不俗。同时,学术界和行业都强调了深度学习的洞察力在生物信息学中的应用,例如基于深度学习的蛋白质结构预测、基因调控码学习、基因表达预测、癌症分类预测、复杂疾病分类、多平台癌症数据综合分析等。
guillen等人设计基于多层感知器的深度学习算法捕获基因表达特征进行癌症分类,表明了神经网络可以高效率地对不同的样本进行分类,在最后的预测结果中实现了较高精度。bhat等人通过深度生成学习检测癌症,使用对抗性特征学习过程挖掘数据特征,然后使用常规分类器进行分类。最终试验通过指定适当的超参数,在两个不同数据集上执行得相当好。danaee等人使用堆叠去噪自动编码器(sdae)从高维基因表达谱中提取深度功能特征,通过分析sdae连接矩阵确定了一组高度互动的基因用于癌症生物标志物检测。chira等人使用基因表达值随时间推移的模式开发基于形状的聚类模型,并且进一步结合基因表达水平与输出值之间的相关关系,考虑共同表达模式与测量输出的关系,以指导结果的生物学解释。singh等人提供层叠特征选择与堆叠稀疏自动编码器(ssae)从数据中学习高级特征,每层执行特征选择是一种启发式的,可以在每个阶段获得相关特征,并且在调整过程中减少计算量,该算法在gemler数据库的36个数据集上进行了测试,其中35个数据集的效果超越了gemler基准测试结果。liang等人提出了一种多峰深度信念网络(dbn)的新学习模型,从多平台观测数据对癌症患者进行聚类,并为个性化癌症治疗提供了有效指导。同时应用对比度发散(cd)学习算法,以无监督的方式推断多模态dbn模型参数。xie等人基于多层感知器和堆叠去噪自动编码器(mlp-sae)的深度学习回归模型预测变异基因型的基因表达,其中堆叠去噪自动编码器用于训练回归模型以提取有效特征,并利用多层感知器进行反向传播,同时通过添加dropout防止过拟合。chen等人设计了一种深度学习方法(d-gex),充分捕捉基因表达间的非线性相关关系,利用大约1000个标记基因推断剩余的靶基因表达,旨在降低基因表达谱测定成本。
技术实现要素:
为了克服已有基因调控网络建模及差异性分析方法的精确性较差的不足,本发明提供一种精确性较好的基于rnn的基因调控网络构建与动态差异性分析方法。
本发明解决其技术问题所采用的技术方案是:
一种基于rnn的基因调控网络构建与动态差异性分析方法,包括以下步骤:
第一步、基于deeprnn的基因动态调控网络构建
基因表达数据表示为
第二步、基于亚型内动态调控网络的时序变化演化分析
定义c1亚型在t0时刻的有向加权图拓扑结构表示为
第三步、基于亚型间动态调控网络的网络差异演化分析
不同亚型网络的演化分析包括动力学分析、差异性分析和扰动分析,
所述动力学分析使用差分方程对离散的网络动力学行为进行分析,对于不同亚型的动态调控网络,分析同一时间段关联基因对的节点度值、连边权重、表达变化量相对比率;通过提取不同亚型网络的关联特征,并以此为基础构建多网络协同演化模型;
所述差异性分析对相同时间窗口内不同亚型间的两个基因调控网络作基于节点局部结构特征的减法运算,检测网络结构间存在的差异边,根据差异网络鉴别关键枢纽基因,然后利用go信息和kegg通路功能富集性分析检验所发现基因集的显著性,得到癌症亚型相关控制基因作为进一步生物实验的检验标记;
所述扰动控制分析中,关键枢纽基因节点在细胞生化过程中具有以下特征:同功能中心,即该节点附近的基因属于某类功能的基因集;同驱动中心,即受到该节点表达调控的同距离区间内的基因具有类似的生化功能,对于关键枢纽节点的调控输入一个随机扰动υper,对不同网络在同距离区间内的同功能基因集取交集,得到亚型网络间的动态调控差异节点。
进一步,所述第一步中,基于deeprnn的基因动态调控网络构建包括以下步骤:
1.1预处理,首先,提取亚型网络之间的信息基因,然后,将同一亚型内部的样本按照百分比随机分为训练集80%,验证集10%,测试集10%,进一步,将同一样本的基因表达按照时间序列展开作为输入向量:
1.2激活函数与损失函数,采用relu非饱和激活函数,值域为[0,+∞),公式如下:
其中
deeprnn由一个输入层、一个或多个循环体隐藏层和一个输出层组成,所有隐藏的层都有相同数量的隐藏单元,将上一时刻的状态与当前时刻的输入拼接成一个大的向量作为循环体中神经网络的输入,得到第l层的第j个单元的信号输出
其中h是隐藏单元个数,
其中m'表示训练样本个数,n表示每个训练样本基因个数,ωm(i,j)表示在t时刻样本m中的基因gi对基因gj的作用效果,即连边权重,
1.3dropout方法,在训练过程中,对于每个训练样本的隐藏单元及其边缘将会以概率为p被暂时丢弃;因此前向传播和后向传播将在一个特别“薄”的稀疏网络上进行;对于deeprnn,只在同一时刻的不同层循环体之间使用dropout,即仅在同一时刻t中,从h1到hlast的不同层循环体之间使用dropout;将在区间[0%,25%]之间比较不同程度的正则化效果,寻找最优dropout比率;
1.4加速梯度优化和权重初始化,拟采用动量法进行加速优化,即通过在迭代过程中累积损失函数的梯度方向来代替梯度进行参数更新,对于神经网络参数θ的损失函数l(·),动量计算公式如下:
其中,μ∈[0,1]是动量系数,η是学习率;
隐藏层单位的权重使用均匀分布进行采样,定义如下:
其中ni,no分别表示隐藏单元的扇入扇出个数;
1.5输出,在循环体中的神经网络供给当前时刻的输出后,将会使用另外一个全连接神经网络实现将当前时刻的状态转化为最终的输出。
再进一步,所述第二步中,网络的拓扑属性是描述网络本身及其内部节点或边结构特征的测度,包括:
聚类系数,体现部分节点间存在的密集连接性质,在有向网络中,标准化的聚类系数被定义为:
其中kout表示节点v的出度,n表示所有v所指向的节点彼此存在的边数,
介数表明一个节点在其他节点彼此连接中所起的作用,标准化至[0,1]区间的计算公式如下:
其中σij是节点i到节点j的最短路径条数,σivj表示σij中通过节点v的路径条数;
紧密度是描述一个节点到网络中其他所有节点平均距离的指标,定量衡量节点接近网络“中心”的程度,节点v的紧密度cv计算公式如下:
其中dvj表示节点v到节点j的最短距离(路径中所经过边的权重之和最小)。紧密度越小,节点越接近中心。
基于网络结构的拓扑属性变化在时间序列上对时间窗口δt进行微分展开,得到动态调控网络的时空演化测度γ'(·)的计算公式如下:
其中θ表示函数参数,ωcc、ωb、ωc分别为对应指标的影响权重;
通过分析动态网络在不同时刻的节点指标(ccv、bv、cv),挖掘在不同时间窗口内的关键调控基因节点,解释其在生命活动过程中扮演的重要性。
所述第三步中,所述动力学分析过程中,动力差异计算公式如下:
其中θ表示节点度值、连边权重、表达变化量相对比率三项指标,
所述第三步中,所述差异性分析过程中,检测网络结构间存在的差异边的计算公式如下:
其中
所述第三步中,所述扰动控制分析中,得到亚型网络间的动态调控差异节点,表达式为:
其中
本发明的技术构思为:分析同一癌症亚型和不同癌症亚型的基因调控差异,针对表达数据中癌症基因间的高度非线性相关性,基于深层循环神经网络(deeprecurrentneuralnetwork,deeprnn)对不同癌症亚型在用药后的连续时序变化下的基因表达数据构建调控网络,分析亚型间的表达差异性。
在基因表达数据的癌症关联基因特征提取后,完成癌症亚型的聚类分析,针对不同的亚型聚类结果分别构建对应的基因调控网络分析其差异性。本项目提出基于深层循环神经网络(deeprnn)的基因调控网络建模方法,利用深层循环神经网络的时序处理特性,预测基因动态调控网络的节点度值与连边权重。其次,纵向分析不同时间窗口中相同亚型调控网络的节点与连边变化,挖掘相关基因在癌症演化过程中的调控功能,以及对病症发展的后续阶段进行预测。最终,横向分析不同亚型间的调控网络差异,并对时间序列下的协同演化过程中的差异变化进行生物学意义上的解释,为个性化临床治疗方案提供科学合理的指导。
本发明的有益效果主要表现在:精确性较好。
附图说明
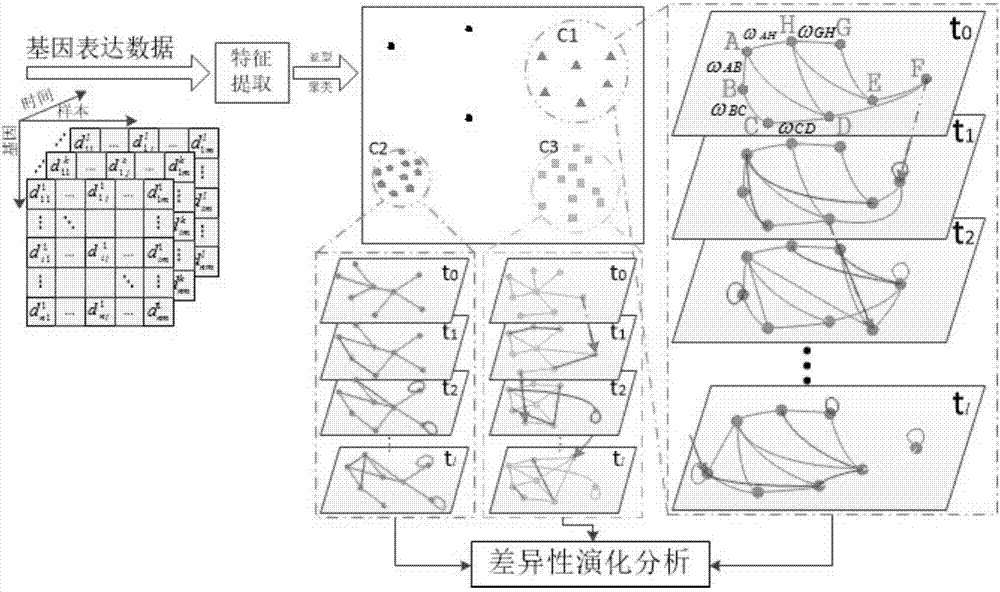
图1是基因表达动态时序网络及差异性演化分析示意图。
图2是基于deeprnn的基因调控时序网络构建框图。
图3是亚型内部时序展开动态调控网络构建示意图。
图4是不同亚型间的基因调控网络渐变演化示意图。
具体实施方式
下面结合附图对本发明作进一步描述。
参照图1~图4,一种基于rnn的基因调控网络构建与动态差异性分析方法,分析同一癌症亚型和不同癌症亚型的基因调控差异,针对表达数据中癌症基因间的高度非线性相关性,基于深层循环神经网络(deeprecurrentneuralnetwork,deeprnn)对不同癌症亚型在用药后的连续时序变化下的基因表达数据构建调控网络,分析亚型间的表达差异性;
如图1所示,首先,在t0时刻的癌症样本被聚类为c1、c2、c3三种亚型,其余三个黑点表示奇异样本。对于c1类簇,基于deeprnn的调控网络构建如蓝色虚线框中的t0时刻网络,显示根据a-h的8个信息基因构建调控网络,并通过真阳率、假阳率、阳性预测率、准确率对网络性能进行定量评价;然后,在后续的数据流输入后,网络的节点度值、连边权重值及节点位置发生迁移,得到了诸如t1、t2…tl的动态演化调控网络,从而设计基于多层次动力系统模型的分析方法揭示基因间调控过程中的逻辑关系;最后,进行不同亚型在网络间的横向分析,对于不同亚型间的两个基因调控网络作基于节点局部结构特征的减法运算,检测网络结构间存在的差异边,得到差异网络进而鉴别关键枢纽基因,同时利用go信息和kegg通路功能富集性分析检验所发现基因集的显著性,最终识别出癌症亚型相关控制基因作为进一步生物实验的检验标记。
为了验证本项目提出的算法在处理真实癌症基因表达数据的实时性、有效性和可靠性,并且获得算法的优化参数,本项目将先对常用基因表达数据库(如geo、tcga、smd、gxd、gent等)中的癌症表达标准数据进行有针对性的分类与分析,验证算法性能。
所述基因调控网络构建与动态差异性分析方法包括以下步骤:
第一步、基于deeprnn的基因动态调控网络构建
基因的表达具有时空性,是基因与外界环境相互作用的结果,会根据当前的表达状况决定未来的表达,所以适合利用deeprnn的历史记忆效应,学习训练隐藏层参数,最终以矩阵形式输出调控网络权值。如图2所示为按时序展开的循环神经网络的构建及调控权重的训练过程。
基因表达数据表示为
1.1预处理。首先,提取亚型网络之间的信息基因,一方面是因为样本的过长输入时间序列间隔会导致优化时的“梯度弥散”问题;另一方面也是因为在某一调控过程中的无关基因相当于噪声,使用强有力的控制基因能够更好的挖掘调控关系。然后,将同一亚型内部的样本按照百分比随机分为训练集80%,验证集10%,测试集10%。进一步,将同一样本的基因表达按照时间序列展开作为输入向量:
1.2激活函数与损失函数。激活函数作为非线性处理单元(如sigmoid、tanh函数),实现的功能是将来自前一层的输入线性组合结果动态范围压缩到特定值域。为了缓解深度神经网络的“梯度弥散”问题,加快训练收敛速度,拟采用relu这类非饱和激活函数(值域为[0,+∞)),公式如下:
其中
deeprnn由一个输入层、一个或多个循环体隐藏层和一个输出层组成。所有隐藏的层都有相同数量的隐藏单元,将上一时刻的状态与当前时刻的输入拼接成一个大的向量作为循环体中神经网络的输入,得到第l层的第j个单元的信号输出
其中h是隐藏单元个数,
其中m'表示训练样本个数,n表示每个训练样本基因个数,ωm(i,j)表示在t时刻样本m中的基因gi对基因gj的作用效果,即连边权重,
1.3dropout方法。dropout是对神经网络进行模型平均和正则化的技术。在训练过程中,对于每个训练样本的隐藏单元及其边缘将会以概率为p被暂时丢弃。因此前向传播和后向传播将在一个特别“薄”的稀疏网络上进行。对于deeprnn,一般只在同一时刻的不同层循环体之间使用dropout,即仅在同一时刻t中,从h1到hlast的不同层循环体之间使用dropout,这样能够使得网络更加健壮。参考相关文献,将在区间[0%,25%]之间比较不同程度的正则化效果,寻找最优dropout比率。
1.4加速梯度优化和权重初始化。拟采用动量法进行加速优化,即通过在迭代过程中累积损失函数的梯度方向来代替梯度进行参数更新。对于神经网络参数θ的损失函数l(·),动量计算公式如下:
其中,μ∈[0,1]是动量系数,η是学习率,在训练过程中随着错误率变化而不断减小,使用动量法在训练深度神经网络时能够提高收敛速度。深度网络的权重使用归一法进行初始化,旨在稳定训练过程中的激活和反向传播梯度的差异。隐藏层单位的权重使用均匀分布进行采样,定义如下:
其中ni,no分别表示隐藏单元的扇入扇出个数。
1.5输出。在循环体中的神经网络供给当前时刻的输出后,将会使用另外一个全连接神经网络实现将当前时刻的状态转化为最终的输出。
第二步、基于亚型内动态调控网络的时序变化演化分析
通过基因表达数据构建动态基因调控网络来体现真实的动态调控过程能够更精准地反映调控机理,理解基因之间的相互作用机制。如图3所示为某一亚型样本内部的信息基因以时间序列展开后的动态调控网络构建示意图。
定义c1亚型在t0时刻的有向加权图拓扑结构表示为
网络的拓扑属性是描述网络本身及其内部节点或边结构特征的测度。主要包括以下几项:
聚类系数。聚类系数体现了部分节点间存在的密集连接性质,在有向网络中,标准化的聚类系数被定义为:
其中kout表示节点v的出度,n表示所有v所指向的节点彼此存在的边数。
介数。介数表明了一个节点在其他节点彼此连接中所起的作用,标准化至[0,1]区间的计算公式如下:
其中σij是节点i到节点j的最短路径条数,σivj表示σij中通过节点v的路径条数。介数越高,意味着节点在保持网络连接紧密性中越重要。
紧密度。紧密度是描述一个节点到网络中其他所有节点平均距离的指标,可以定量衡量节点接近网络“中心”的程度。节点v的紧密度cv计算公式如下:
其中dvj表示节点v到节点j的最短距离(路径中所经过边的权重之和最小)。紧密度越小,节点越接近中心。
为了描述调控网络的动力学性质,基于网络结构的拓扑属性变化在时间序列上对时间窗口δt进行微分展开,得到动态调控网络的时空演化测度γ'(·)的计算公式如下:
其中θ表示函数参数,ωcc、ωb、ωc分别为对应指标的影响权重。
通过分析动态网络在不同时刻的节点指标(ccv、bv、cv),挖掘在不同时间窗口内的关键调控基因节点,解释其在生命活动过程中扮演的重要性。
第三步、基于亚型间动态调控网络的网络差异演化分析
亚型间的网络分析是为了充分挖掘历史表达数据的时空特性、调控的变化规律、以及调控网络中节点和连边的迁移演化,从而提高网络建模算法的准确度和可靠性,并对基因表达表达变化和网络动态演化进行预测。如图4所示为不同亚型间的基因调控网络渐变演化示意图,其中差异网络是由不同亚型间的两个基因调控网络作基于节点局部结构特征的减法运算得到的。
不同亚型网络的演化分析包括动力学分析、差异性分析和扰动分析。
动力学分析。由于基因表达数据采样的时间间隔较长,使用差分方程对离散的网络动力学行为进行分析。对于不同亚型的动态调控网络,分析同一时间段关联基因对的节点度值、连边权重、表达变化量相对比率,动力差异计算公式如下:
其中θ表示节点度值、连边权重、表达变化量相对比率三项指标,
差异性分析。对相同时间窗口内不同亚型间的两个基因调控网络作基于节点局部结构特征的减法运算,检测网络结构间存在的差异边,计算公式如下:
其中
扰动控制分析。关键枢纽基因节点在细胞生化过程中具有以下特征:同功能中心,即该节点附近的基因属于某类功能的基因集;同驱动中心,即受到该节点表达调控的同距离区间内的基因具有类似的生化功能。对于关键枢纽节点的调控输入一个随机扰动υper,对不同网络在同距离区间内的同功能基因集取交集,得到亚型网络间的动态调控差异节点。具体表达式为:
其中
- 还没有人留言评论。精彩留言会获得点赞!