基于全卷积网络的交叠、粘连染色体自动分割方法与流程
本发明涉及交叠、粘连染色体自动分割技术领域,具体涉及一种基于全卷积网络的交叠、粘连染色体自动分割方法。
背景技术:
人工染色体分析是一项非常繁重的工作,医务人员必须把染色体的显微图片用眼睛和手画方式重现出来,再比较、分析、配对、排序、分类等等一系列的工作浪费了大量的人力和物力。目前国内开发的一些染色体自动分析系统,染色体分析系统是通过高清晰的摄像机架设于显微镜上,摄像机从显微镜捕获镜下的影像,通过数据线连接到专用计算机,计算机运行染色体分析软件,影像在软件中显示。虽然可以实现对染色体的自动分割,但是对染色体重叠、粘连图像的分割存在困难,申请号为200810218815.1的对比文件中虽然公开了对染色体重叠、粘连图像的分割方法,但对比文件中仅对x型重叠、粘连的染色体进行分割,不能有效的对y型和多型叠加的染色体进行分割。
技术实现要素:
为解决上述技术问题,本发明提出了一种基于全卷积网络的交叠、粘连染色体自动分割方法,以达到对重叠、粘连的染色体高效的自动分割,覆盖范围广,降低人工分割的工作量和难度的目的。
为达到上述目的,本发明的技术方案如下:一种基于全卷积网络的交叠、粘连染色体自动分割方法,所述方法步骤包括:
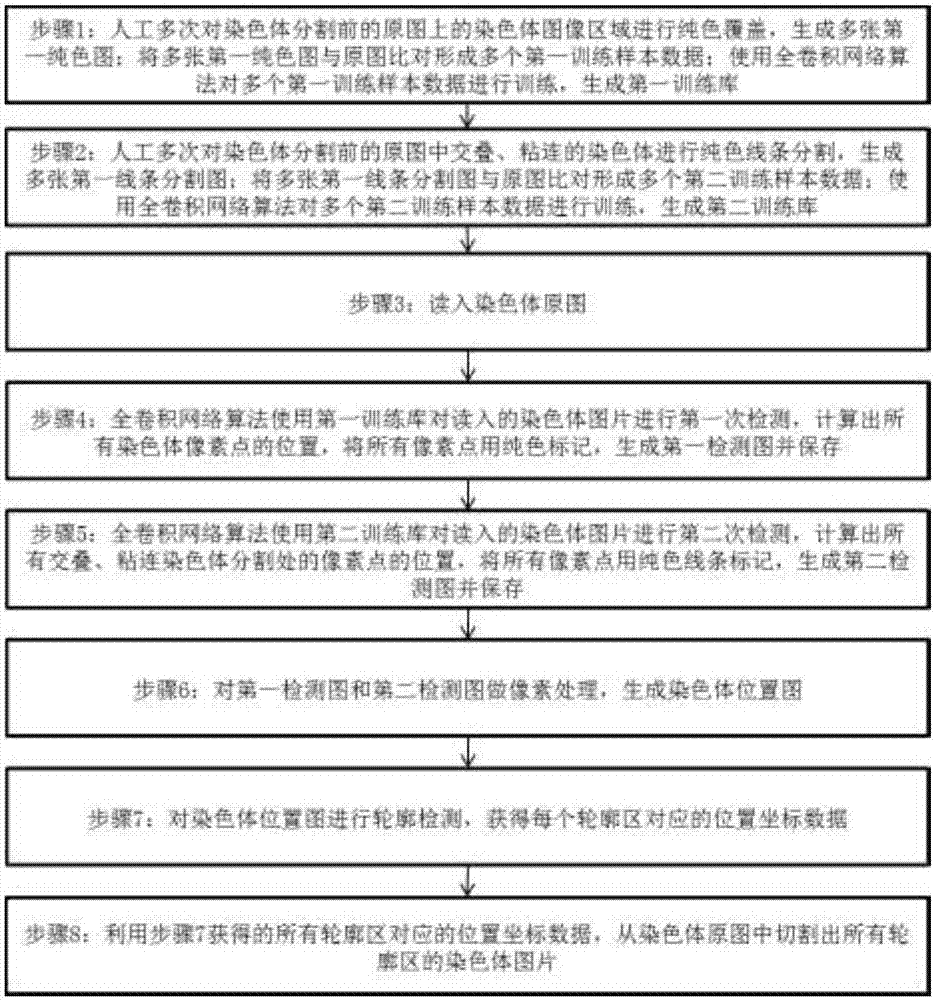
步骤1:人工对多张染色体分割前的原图上的染色体图像区域进行纯色覆盖,生成多张第一纯色图;将多张第一纯色图与第一纯色图自身对应的原图比对形成多个第一训练样本数据;使用全卷积网络算法对多个第一训练样本数据进行训练,生成第一训练库;
步骤2:人工对多张染色体分割前的原图中交叠、粘连的染色体进行纯色线条分割,生成多张第一线条分割图;将多张第一线条分割图与第一线条分割图自身对应的原图比对形成多个第二训练样本数据;使用全卷积网络算法对多个第二训练样本数据进行训练,生成第二训练库;
步骤3:读入染色体原图;
步骤4:全卷积网络算法使用第一训练库对读入的染色体图片进行第一次检测,计算出所有染色体像素点的位置,将所有像素点用纯色标记,生成第一检测图并保存;
步骤5:全卷积网络算法使用第二训练库对读入的染色体图片进行第二次检测,计算出所有交叠、粘连染色体分割处的像素点的位置,将所有像素点用纯色线条标记,生成第二检测图并保存;
步骤6:对第一检测图和第二检测图做像素处理,生成染色体位置图;
步骤7:对染色体位置图进行轮廓检测,获得每个轮廓区对应的位置坐标数据;
步骤8:利用步骤7获得的所有轮廓区对应的位置坐标数据,从染色体原图中切割出所有轮廓区的染色体图片。
作为优选的,其特征在于,所述步骤1、步骤2、步骤4和步骤5中用于标记的纯色均为纯蓝色。
作为优选的,其特征在于,所述步骤1中将多张第一纯色图与原图比对形成多个第一训练样本数据是对比第一纯色图和原图的共有特征,得出染色体模型特征;所述步骤2中将多张第一线条分割图与原图比对形成多个第二训练样本数据是对比第一线条分割图和原图的共有特征,得出分割线模型特征。
作为优选的,其特征在于,所述对第一检测图和第二检测图做像素处理,生成染色体位置图的具体方法为:将第一检测图中的纯色像素点置为纯白色,图上的其余像素点置为纯黑色;对上一步生成的图片进行像素点扫描,将所有位置和第二检测图中纯色像素点位置相同的像素点置为纯黑色得到染色体位置图,提高染色体区域与其他区域颜色上的对比度,便于染色体切割。
作为优选的,其特征在于,所述染色体位置图为染色体位置为纯白色、其余位置为纯黑色的图片。
作为优选的,所述从染色体原图中切割出所有轮廓区的染色体图片后,对所有的染色体图片进行锐化和边缘增强并保存每张处理后的染色体图片。
本发明具有如下优点:
(1).本发明通过全卷积网络对交叠、粘连的染色体进行快速有效的分割,可以覆盖多种形状的染色体,降低了人工分割的难度和工作量,提高分割效率。
附图说明
为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍。
图1为本发明实施例公开的基于全卷积网络的交叠、粘连染色体自动分割方法流程图;
图2为本发明实施例公开的染色体分割前的原图;
图3为本发明实施例公开的第一纯色图;
图4为本发明实施例公开的第一线条分割图;
图5为本发明实施例公开的第一检测图;
图6为本发明实施例公开的第二检测图;
图7为本发明实施例公开的染色体位置图;
图8为本发明实施例公开的染色体图。
具体实施方式
下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述。
本发明提供了一种基于全卷积网络的交叠、粘连染色体自动分割方法,其工作原理是通过全卷积网络对交叠、粘连的染色体进行快速有效的分割,以达到对重叠、粘连的染色体高效的自动分割,覆盖范围广,降低人工分割的工作量和难度的目的。
下面结合实施例和具体实施方式对本发明作进一步详细的说明。
如图1-图8所示,一种基于全卷积网络的交叠、粘连染色体自动分割方法,所述方法步骤包括:
步骤1:人工对多张染色体分割前的原图(如图2所示)上的染色体图像区域进行纯色覆盖,生成多张第一纯色图(如图3所示);将多张第一纯色图与第一纯色图自身对应的原图比对形成多个第一训练样本数据;使用全卷积网络算法对多个第一训练样本数据进行训练,生成第一训练库;
步骤2:人工对多张染色体分割前的原图中交叠、粘连的染色体进行纯色线条分割,生成多张第一线条分割图(如图4所示);将多张第一线条分割图与第一线条分割图自身对应的原图比对形成多个第二训练样本数据;使用全卷积网络算法对多个第二训练样本数据进行训练,生成第二训练库;
步骤3:读入染色体原图;
步骤4:全卷积网络算法使用第一训练库对读入的染色体图片进行第一次检测,计算出所有染色体像素点的位置,将所有像素点用纯色标记,生成第一检测图(如图5所示)并保存;
步骤5:全卷积网络算法使用第二训练库对读入的染色体图片进行第二次检测,计算出所有交叠、粘连染色体分割处的像素点的位置,将所有像素点用纯色线条标记,生成第二检测图(如图6所示)并保存;
步骤6:对第一检测图和第二检测图做像素处理,生成染色体位置图(如图7所示);
步骤7:对染色体位置图进行轮廓检测,获得每个轮廓区对应的位置坐标数据;
步骤8:利用步骤7获得的所有轮廓区对应的位置坐标数据,从染色体原图中切割出所有轮廓区的染色体图片(如图8所示)。
其中,其特征在于,所述步骤1、步骤2、步骤4和步骤5中用于标记的纯色均为纯蓝色。
其中,其特征在于,所述步骤1中将多张第一纯色图与原图比对形成多个第一训练样本数据是对比第一纯色图和原图的共有特征,得出染色体模型特征;所述步骤2中将多张第一线条分割图与原图比对形成多个第二训练样本数据是对比第一线条分割图和原图的共有特征,得出分割线模型特征。
其中,其特征在于,所述对第一检测图和第二检测图做像素处理,生成染色体位置图的具体方法为:将第一检测图中的纯色像素点置为纯白色,图上的其余像素点置为纯黑色;对上一步生成的图片进行像素点扫描,将所有位置和第二检测图中纯色像素点位置相同的像素点置为纯黑色得到染色体位置图,提高染色体区域与其他区域颜色上的对比度,便于染色体切割。
其中,其特征在于,所述染色体位置图为染色体位置为纯白色、其余位置为纯黑色的图片。
其中,所述从染色体原图中切割出所有轮廓区的染色体图片后,对所有的染色体图片进行锐化和边缘增强并保存每张处理后的染色体图片。
在交叠、粘连的染色体分割过程中产生的数据可以再次进行训练,进一步的提高交叠、粘连染色体的切割范围。
以上所述的仅是本发明所公开的一种基于全卷积网络的交叠、粘连染色体自动分割方法的优选实施方式,应当指出,对于本领域的普通技术人员来说,在不脱离本发明创造构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。
- 还没有人留言评论。精彩留言会获得点赞!