面向疾病的基因组匿名化的制作方法
本发明涉及遗传数据的分析。更具体地,本发明涉及关于特定疾病或障碍的遗传数据的分析。
背景技术:
当今,患者的医学和健康记录被收集并用于临床生物信息学研究。除了患者的临床数据、成像数据或生物银行数据之外,还收集其遗传数据,并且分析遗传数据在医学研究以及诊断和既往症中起重要作用。例如,分析患者的遗传数据以发现或改善针对不同疾病的处置。
然而,对其遗传数据的分析可能会对共享他们的遗传数据的患者构成威胁,例如,他们的隐私将受到侵犯。侵害是由于人的基因组包含诸如关于眼睛颜色、肤色的数据的事实。这些遗传数据与嵌入在人的基因组中的其他数据一起,可以通过分析他们遗传数据来识别人。为了保护个人隐私,当为医学生物信息学研究和分析提供遗传数据时,需要对人类基因组的特定部分进行匿名化。
生物信息学研究中用于基因组匿名化的一些现有解决方案试图使整个基因组匿名化,而不考虑要研究的疾病。由于匿名化意味着信息的丢失,这些现有的解决方案还导致关于与要研究的疾病直接相关的基因的信息丢失,这是不希望的。
用于基因组匿名化的其他解决方案考虑取证上下文,其是与本发明所解决的攻击模型不同类型的攻击模型。
此外,鉴于遗传分析被更广泛地采用,患者的同意仅将对他/她的基因组信息收集限于到基因的子集而没有灵活的匿名化解决方案。在研究过程中,这个基因子集可能在以后变得过于有限,并且相关的基因对于分析可能是有用的。即使该人可能已经同意使用该相关基因,因为它仍然与疾病有关,但由于早期的隐私问题,所述基因已经从数据集中缺失。
此外,在隐藏一些患者的遗传信息的同时,匿名化技术还应该能够发现何时需要修改疾病相关的基因的集合,特别是当需要扩大疾病相关的基因的集合时。
us2014/0236833a1公开了一种用于基于个体的遗传身份在个体与第三方之间建立交易的方法,其中,个人允许第三方仅访问和分析要约和建立交易所需的遗传身份的子集。
us2010/0063843a1公开了一种用于掩蔽数据记录访问的基于计算机的方法和系统,其中数据掩码被应用于敏感的个人信息,使得该信息的非掩蔽部分可以用于在选择针对消费者的产品、服务和服务提供商中使用。
技术实现要素:
为了解决上述问题,提出了一种解决方案,其中,基于遗传数据与要研究的疾病相关基因的有多密切地相关,将一个或多个个体的基因组的遗传数据分成不同的层。该关系是基于基因组的途径网络建立的。然后使用不同的匿名化技术来对除了与要研究的疾病直接相关的遗传数据之外的遗传数据层进行匿名化。基于其估计的相关性,为每层遗传数据选择使用的匿名化技术。与要研究的疾病直接相关的遗传数据仍然是非匿名的,并且以可以用于分析。
附图说明
在附图中:
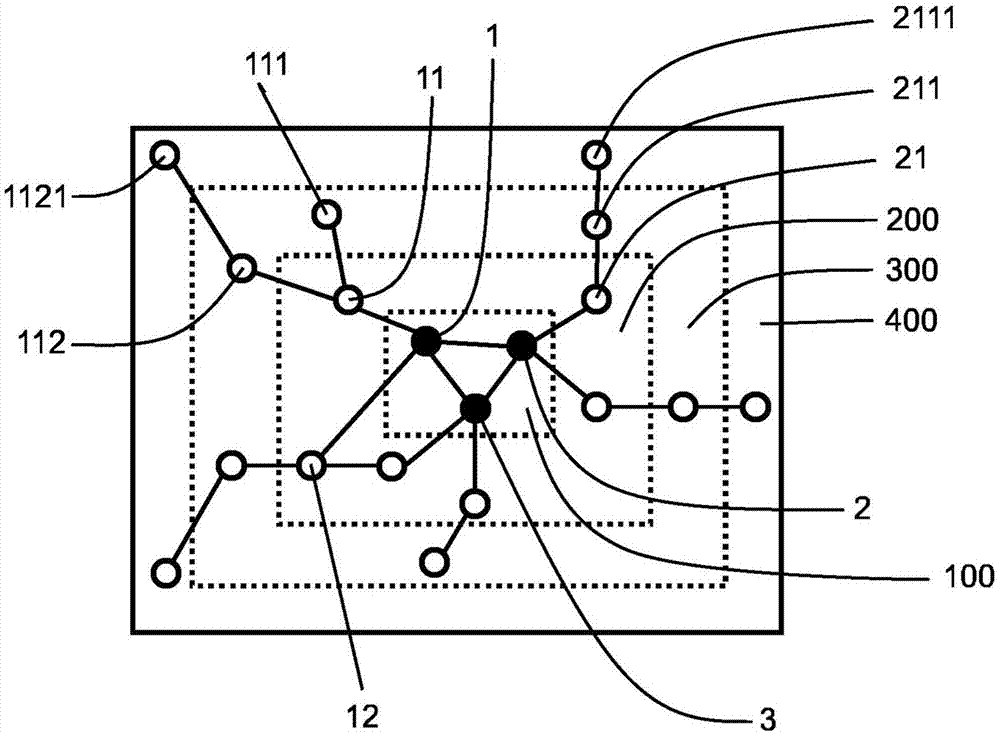
图1表示用于面向疾病的匿名化的分层遗传数据的示意图。
图2表示重新分层遗传数据的示意图。
图3是说明分层的面向疾病的匿名化方法的实施例的步骤的流程图。
图4图示了用于存储用于实现用于匿名化遗传数据的方法的计算机可执行代码的计算机可读介质的示例。
图5图示了被配置用于匿名化遗传数据的系统的实施例。
具体实施方式
在第一方面中,本发明提供了对一种用于遗传数据进行匿名化的方法。
在第二方面中,本发明提供了一种提供对遗传数据进行匿名化的计算机程序产品。
在第三方面中,本发明提供了一种用于对遗传数据进行匿名化的系统。
在第四方面中,本发明提供了所述方法和/或所述计算机程序产品在生物信息学研究和/或诊断中的使用。
以下将关于特定实施例并且参考特定附图来描述本发明,但本发明并不局限于其,而是仅由权利要求来限定。所描述的附图仅是示意性的并且是非限制性的。在附图中,一些要素的尺寸可能是夸张的并且出于说明性目的没有按比例绘制。
根据第一方面,本发明提供了一种用于关于特定疾病来对来自至少一个个体的遗传数据进行匿名化的方法。用于对遗传数据进行匿名化的所述方法包括以下步骤:
提供来自至少一个个体的遗传数据;
选择要研究的疾病;
确定来自所述至少一个个体的所述遗传数据中与所述要研究的疾病直接相关的遗传数据的(一个或多个)子集;
根据所述遗传数据的不与所述要研究的疾病直接相关的子集到到所述要研究的疾病直接相关的所述遗传数据的距离,将所述子集分成不同的层;并且
将不与要研究的疾病直接相关的层或所述层中存在的不与要研究的疾病直接相关的遗传数据匿名化。
在该方法中,使用来自至少一个个体的遗传数据。术语“遗传数据”是指任何种类的遗传信息。术语“遗传数据”包括个体的基因组的核苷酸序列或个体的基因组的一部分的核苷酸序列。“遗传数据”还包括除这样的核苷酸序列之外的遗传信息,例如关于遗传标记的存在或不存在的信息,例如扩增片段长度多态性(aflp),随机扩增多态性dna(rapd),限制性片段长度多态性(rflp),单核苷酸多态性(snp),短串联重复序列(str)和可变数目串联重复序列(vntr)。术语“遗传数据”还包括关于rna和蛋白质的信息。因此,术语“遗传数据”包括关于核酸分子和/或蛋白质的核苷酸序列、氨基酸序列、结构、活性、丰度和/或功能的信息。另外,“遗传数据”包括拷贝数数据,例如关于基因拷贝数或其他核苷酸序列延伸的数据。
术语“个体”是指人类对象。所述人类对象可能会或可能不会受到要研究的疾病的影响。因此,术语“个体”,“人”和“患者”在本公开中同义使用。
表述“提供遗传数据”应被理解为需要获得至少一个个体的遗传数据。然而,至少一个个体的遗传数据不必与该方法直接相关或用于实施该方法。通常,至少一个个体的遗传数据是在先前的点或时间段获得的,并且被电子地存储在合适的电子存储设备和/或数据库中。为了执行该方法,可以从存储设备或数据库中检索并利用遗传数据。
表述“选择要研究的疾病”表示该方法可用于研究或分析任何疾病、病症或医学状况。因此,必须选择或定义特定的疾病、障碍或医学状况,以便随后确定与所述疾病、障碍或医学状况直接相关的遗传数据的子集,以及与所述疾病、障碍或医学状况不直接相关的遗传数据。
关于遗传数据子集与要研究的疾病的关系的术语“直接相关”是指导致所述疾病的遗传基因座和/或基因或与引起这种疾病的遗传基因座和/或基因直线相关的遗传基因座和/或基因。遗传基因座和/或基因包含蛋白质编码区(开放阅读框)以及开放阅读框上游或下游的非蛋白质编码区。所述遗传基因座和/或基因还包括直接参与调节导致所述疾病研究的基因表达的那些基因座和/或基因。因此,“直接相关”包括编码导致要研究的疾病的蛋白质或多肽的那些基因的蛋白质编码区的结构特征,以及直接参与调节编码引起疾病的蛋白质或多肽的基因表达的那些元素的结构特征。
术语“层”是指遗传数据的不与要研究的疾病直接相关的子组。层可包括遗传数据的多个子集。例如,层是与直接疾病相关的核心基因中的任何核心基因具有相同距离的基因的子集,其中,两个不同的层具有两个不同的这种距离。每个层被分配一种匿名方法,其中,可以为多个层分配相同的匿名方法。
在一个实施例中,用于匿名化遗传数据的方法旨在用于通过生物信息学方式研究特定疾病,即通过使用软件工具,使用数学和统计技术对生物学查询进行计算机分析,以分析和解释生物学数据与特定疾病的相关性。该实施例通常需要使用多个个体的遗传信息。
在用于匿名化遗传数据的方法的另一个实施例中,所述方法旨在用于在诊断中使用,其中,分析个体的遗传信息的遗传倾向和/或所述个体的特定疾病或病症的发生。
该方法可以应用于任何疾病、障碍或医学状况。等研究的疾病是有意选择的特定疾病。在一个实施例中,已知要研究的疾病是与特定基因型相关的疾病。这些疾病的实例是癌症、免疫系统疾病、神经系统疾病、心血管疾病、呼吸系统疾病、内分泌和代谢疾病、消化系统疾病、泌尿系统疾病、生殖系统疾病、肌肉骨骼疾病、皮肤病、先天性代谢紊乱等、其他先天性疾病(例如前列腺癌、糖尿病、代谢紊乱或精神疾病)。
在所述方法中,基于遗传数据与要研究的疾病的关系,将所述至少一个个体的遗传数据分组成遗传信息的子集或层。因此,已知与要研究的疾病直接相关的那些遗传数据(核心-疾病基因)被分组为不被匿名的子集。
与要研究的疾病直接相关的“遗传数据”包括与要研究的疾病有联系的基因、标记、rna和蛋白质,优选地在于,所述遗传数据的主题的序列、结构、活性、丰度和/或功能导致疾病被研究或者是要研究的疾病的直接后果。遗传数据可能涉及蛋白质编码区内和/或蛋白质编码区外的一个或多个基因的核苷酸序列。遗传数据也可能涉及调节基因。将与要研究的疾病直接相关的遗传数据放入可称为“核心”的子组中。
不与要研究的疾病直接相关的遗传数据被分组为至少一个子集或层。从理论上讲,层数可以高达x-1,其中,x表示给定基因组中的基因的数目。优选地,基于与一种或多种核心疾病基因的距离程度,将与所研究的疾病不直接相关的遗传数据分组为两层或更多层中的一层,其中,如果遗传数据的子集与不同的核心-疾病基因具有不同的距离,则选择最近的距离。在一个实施例中,子集或层的数量等于或小于10,优选地,子集/层的数量是2、3、4、5、6、7、8、9或10。因此,在其中,层数为1的示例性实施例中,遗传数据被分为直接疾病相关数据,或非直接疾病相关的数据或非疾病相关的数据。在替代实施例中,其中,层数为2或更多,遗传数据被分成直接疾病相关的数据子集和几个不直接疾病相关的数据的子集。
对于确定遗传数据子集与要研究的疾病的关系和/或其与要研究的疾病直接相关的遗传数据子集的相对距离,利用了基因组途径网络。
基因组途径网络通过互联网上的数据库可用并且可访问,并且可以被建立-例如-针对特定疾病,例如前列腺癌(http://www.genome.jp/dbget-bin/www_bget?pathway:map05215),ii型糖尿病(http://www.genome.jp/dbget-bin/www_bget?pathway:map04930)或帕金森病(http://www.genome.jp/dbget-bin/www_bget?pathway:map05012)。
在另外和/或替代实施例中,关于特定病症未建立基因组途径网络。这种更通用的基因组途径网络数据库的示例是reactome开源策划和同行评审途径数据库(www.reactome.org),pathway/genome数据库的biocyc数据库集合(www.biocyc.org),途径公共途径信息数据库(www.pathwaycommons.org)以及基因本体协会数据库(www.geneontology.org)。
在另外和/或替代实施例中,string数据库(https://www.string-db.org)被使用。string是已知和预测的蛋白质-蛋白质相互作用的数据库。相互作用包括直接(物理)和间接(功能)关联;它们源于计算预测,源于生物之间的知识转移,以及源于从其他(主要)数据库聚合的相互作用。string数据库中的相互作用源自基因组背景预测,高通量实验室实验,(保守的)基因共表达,自动文本挖掘和数据库中的先前知识。string数据库在2016年6月底涵盖来自2031个生物体的9643763种蛋白质。string数据库由string协会运营,string协会包括瑞士生物信息学研究所,cpr-nnf蛋白质研究中心和欧洲分子生物学实验室。
与待研究和存在于核心层中的疾病直接相关的遗传数据不是匿名的,并且因此可以无限制地进行分析。
通过使用选自包括统计匿名化、加密和安全多方匿名化和计算的组中的技术,对不与要研究的疾病直接相关的遗传数据和/或遗传数据层进行匿名化。
这些匿名化技术允许对数据进行分析,但由于其属性,此分析受到限制。统计匿名化意味着信息丢失,但将其余信息保持在人类可读的形式中。这允许对数据执行分析,但结果受到从一开始就丢失信息的限制。加密技术不会丢失信息,但此信息不可用。但是,如果有任何迹象表明加密信息对于研究是必要的,则隐私官员能够通过解密该集合来扩展核心疾病信息。存在中间解决方案,其中,像同态加密、多方计算和/或对加密数据的其他操作的现代技术用于将核心疾病集与加密层组合。在这些情况下,隐私敏感信息将保密,而隐私官员可以公开这些操作的结果。这些技术在分析中插入延迟,并且因此限制了可以对数据执行的可能分析。
在一个实施例中,统计匿名化选自包括以下项的组:k-匿名,l-多样性,t-接近度和δ-存在。
k-匿名是l.sweeney创建的形式隐私模型。目标是如果尝试识别数据,则使每个记录与定义的数量(k)的其他记录无法区分。如果对于具有给定属性的集合的任何数据记录,至少存在与这些属性匹配的k-1个其他记录,那么数据的该集合是k-匿名的[j.sedayao,“enhancingcloudsecurityusingdataanonymization”,2012年6月。[在线]。可发现于:http://www.intel.nl/content/dam/www/public/us/en/documents/best-practices/enhancing-cloud-security-using-data-anonymization.pdf。(2015年1月26日存取)],[l.sweeney,"k-anonymity:amodelforprotectingprivacy,"int.j.uncertain.fuzzinessknowl.-basedsyst.,vol.10,no.5,pp.557-570,2002]。k的典型值是3[m.templ,b.meindl,a.kowarik和s.chen的“introductiontostatisticaldisclosurecontrol(sdc)”,2014年8月。[在线]。可发现于:http://www.ihsn.org/home/sites/default/files/resources/ihsn-working-paper-007-oct27.pdf。(2015年1月26日存取)]。
l-多样性改善了匿名化,超出了k-匿名性提供的范围。两者之间的区别在于,虽然k-匿名要求准标识符的每个组合具有k个条目,但是l-多样性要求对于每个准标识符组合存在l个不同的敏感值[j.sedayao,“enhancingcloudsecurityusingdataanonymization”,2012年6月[在线]。可发现于:http://www.intel.nl/content/dam/www/public/us/en/documents/best-practices/enhancing-cloud-security-using-data-anonymization.pdf。[2015年1月26日存取]][4]。
t-接近度要求任何等价类中敏感属性的分布接近整个表中属性的分布(即,两个分布之间的距离应不大于阈值t)[n.li,t.li和s.venkatasubramanian在dataengineering,2007.icde2007.ieee23rdinternationalconferenceon,2007中的“t-closeness:privacybeyondk-anonymityandl-diversity"]。l-多样性要求确保每组中敏感值的“多样性”,但它没有考虑这些值的语义接近程度。这是通过t-接近度来完成的。
δ-存在是用于基于公开己知的数据的泛化来评估在表格中识别个体的风险的度量。δ-存在是数据集的一个很好的度量,其中“知道个体在数据库中构成”隐私风险。[menergiz,m.atzori和c.clifton在proceedingsofthe2007acmsigmodinternationalconferenceonmanagementofdata,beijing,china,2007中的“hidingthepresenceofindividualsfromshareddatabases”。
匿名技术“可搜索加密”,“同态加密”和“安全多方计算”具有以下优点:加密数据的解密实际上不是必需的,但是在加密域中执行数据处理是可行的。这些技术之间的主要区别在于他们所做出的权衡取舍。可搜索加密将处理限制为简单的关键字匹配。完全同态加密可以进行任何类型的处理,但具有极大的密文大小并且计算量非常大。多方计算更好地扩展,但要求非串通计算机一起工作以进行处理。
在另外和/或替代实施例中,遗传数据和/或遗传数据的不与要研究的疾病直接相关的层通过加密进行匿名化,优选地选自包括同态加密、可搜索加密和不可延展加密的组。
与基因去除相比,不可延展的加密具有以下优点:数据不会丢失并且统计学家可以注意到在基因组的特定方向上存在更多数据。此外,当注意到某个基因应该被归类为核心疾病基因时,可以创建新的基因组分层,并根据新的核心疾病基因重新匿名化基因组。
在另外的和/或替代实施例中,匿名化考虑了层内遗传数据与核心的接近度,其中包含更接近核心疾病的遗传数据的层使用涉及丢失较少信息的技术进行匿名化,并且因此仍然允许一定程度的分析。
在另外和/或替代实施例中,不同的层通过不同的技术匿名化,优选地取决于遗传数据的层的子集到遗传数据的与要研究的疾病直接相关的子集的距离。通过不同技术对不同层进行匿名化可以提高数据安全性,因为非故意地解码遗传数据变得更加困难。
通过本文公开的方法匿名化的遗传信息的特性是可检测的,因为至少一个子集-核心层-是人类可读的。遗传数据的子集是统计上匿名的数据是人类可读的。此外,可以通过使用工具来检测统计上匿名的数据,这些工具验证数据是否具有类似2-匿名的属性。在一个实施例中,所述工具选自arx-匿名工具,utd匿名化工具箱,μ-argus,r-packagesdcmicro,康奈尔匿名化工具包,parat,cats去识别平台,irifieldshield,gedis工作室匿名化,safelink,anu数据挖掘组,数据交换工具包,ruby数据匿名工具和可逆日志匿名工具。
arx数据匿名工具(http://arx.deidentifier.org/anonymization-tool/)可以通过比较输出和输入来检查数据是否正确匿名化,如果数据是csv格式则不应该有所不同。
utd匿名化工具箱(http://cs.utdallas.edu/dspl/cgi-bin/toolbox/index.php)涵盖了以下匿名化模型:k-匿名,l-多样性,t-接近度。它可以与arx数据匿名工具相同的方式使用。
μ-argus(anti-re-identificationgeneralutilitysystem)是由荷兰统计局开发的软件包,(http://neon.vb.cbs.nl/casc/software/mumanual4.2.pdf)。该软件包提供风险方法,后随机化(pram),数字微聚合,排名交换。代码可在此处获得:http://neon.vb.cbs.nl/casc/mu.htm。
r-packagesdcmicro是一个r包工具。它可用于生成匿名微数据。该工具可以从以下位置下载:http://cran.r-project.org/web/packages/sdcmicro/.sdcmicro包含几乎所有用于匿名和连续变量匿名化的流行方法。此工具使用gpl许可证。
康奈尔匿名工具包(cat)(http://sourceforge.net/projects/anony-toolkit/)实施两个隐私标准:l-多样性和t-接近度。给定特定隐私标准,有许多匿名化策略来实现这一标准,例如数据泛化,数据交换,数据扰动等。cat目前仅支持数据泛化机制。
parat(http://www.privacyanalytics.ca/software/)是一个集成的去识别和屏蔽软件,其专注于健康数据。它是商业上可获得的。parat可以处理结构化数据和非结构化数据,并使用不同的保护方法:屏蔽,针对不同类型变量的去识别:直接标识符,准标识符。
cats去识别平台(https://www.custodix.com/index.php/cats)cats(custodixanonymisationservices)是一个面向服务的平台,用于数据的去识别。cats支持以通用和可扩展的方式匿名化不同类型的数据(csv,xml,hl7,dicom)。它可以整合到自动数据流中,也可以用于手动去识别。
irifieldshield(http://www.iri.com/solutions/data-masking/de-identification/anonymize)提供去识别、编码、加密、数据屏蔽、随机化和假名化的功能。
gedisstudio匿名化(http://www.gedis-studio.com/anonymization.html)提供数据加密和加扰的匿名化,但也提供数据屏蔽。可以在考虑数据分布的同时完成数据屏蔽。
safelink(https://www.uni-due.de/soziologie/schnell_forschung_safelink_software.php)是隐私保护记录链接程序的规范和实现,它使用加密散列(密钥hmac)。
澳大利亚国立大学数据挖掘小组(http://datamining.anu.edu.au/projects/linkage.html)旨在开发基于单向散列和/或加密的蒙眼记录链接技术。
数据交换工具包可以在这里找到:(http://www.niss.org/sites/default/files/dstk-afk.pdf)。
ruby数据匿名化工具(https://www.ruby-toolbox.com/projects/data-anonymization)使用白名单和黑名单概念来处理直接标识符的去除。代码可以在这里找到:https://github.com/sunitparekh/data-anonymization。
可逆日志匿名工具(http://blog.cassidiancyber-security.com/post/2014/01/reversible-log-anonymization-tool)是一种被设置为在用匿名值替换客户日志中的敏感字段同时生成查找表的工具。
在另外和/或替代实施例中,加密数据的子集允许在密码文本上进行比较并因此揭示可用于分析要研究的疾病的信息。可以通过以下来检测加密数据的分析:
-通过数据库数据检索分析,其中,来自数据库的加密数据被选择并在系统的正在对加密数据执行操作的其他部分中本地使用;和/或
-通过流量分析,其揭示在除本地机器之外的其他机器上执行的多方计算。
由于其灵活的匿名化,所述方法是有利的。该方法允许遗传数据的去匿名化和重新匿名化。基于研究进展,以前匿名化的遗传数据可以通过执行第一次匿名化的相同过程和实体或第三方来恢复和新分类。
在替代和/或另外的实施例中,所述方法还包括分析与要研究的疾病直接相关的遗传数据。通常,关于要研究的疾病的遗传数据的分析必须由与匿名遗传数据的实体不同的实体进行。
参考图1,示出了遗传数据的分层的面向疾病的匿名化。在该实施例中,遗传数据被认为是基因。每个基因用圆圈表示。与要研究的疾病直接相关的基因是核心基因(1、2、3)并存在于核心(100)中。这些核心基因显示为实心圆圈。提供三层(200、300、400)用于携带不与要研究的疾病直接相关的基因。不与要研究的疾病直接相关的基因显示为空心圆圈。基因11和12与核心基因1直接相连,如代表各基因的圆圈之间的实线所示。基因11和12在第1层(200)中分组,其承载与核心基因最接近但不与要研究的疾病直接相关的那些基因。基因111和112与基因11直接相连,但与核心基因1的关系不那么密切。因此,基因111和112被放入第2层,其含有与核心基因直线相关的基因相比与核心基因距离更远地相关的基因。层200、300、400和所述层中包含的基因是匿名的,其中,核心100和核心疾病基因1、2、3不是匿名的。
图2图示了在去匿名化和重新匿名化之后如图1所示的分层的面向疾病的匿名化,其包括基因21作为与要研究的疾病直接相关的核心基因。如图1中所示,基因21最初被认为是与核心基因2直接相连的基因,但与要研究的疾病没有直接关系。如果基因21由于研究和开发的进展而被理解为与要研究的疾病直接相关,则将其包括在核心1中,如图2中所示。另外,基因211与基因21直接相连也将被移入第二接近核心的层,即从层300移动到层200,其中,层200、300、400和所述层中包含的基因是匿名的,但核心100和核心疾病基因1、2、3、21不是匿名的。因此,如果给定基因被确定为核心疾病基因,则任何与所述给定基因直接相连的基因(即由所述基因编码的基因或多肽直接与另一基因或由所述另一基因编码的多肽相互作用),被分类到距核心更近的一层的层。由于所述基因和/或由所述基因编码的多肽的直接相互作用,将所述给定基因与所述给定基因直接相迦的所述另一基因分类到更靠近核心的一层。
图3表示图示用于遗传数据的疾病定向匿名化的方法的实施例的示意流程图,其中,步骤500表示收集和存储一个或多个个体的遗传数据。在步骤510中,选择要研究的疾病。然后在步骤520中确定核心-疾病基因,并且基于基因组途径网络和基因与核心-疾病基因的接近度将基因分成不同的层。在步骤540中,存在于除核心层之外的层中的遗传数据是匿名的。
根据第二方面,本发明提供了一种用于对遗传数据进行匿名化的计算机程序产品,所述计算机程序产品包括指令,所述指令在计算机上执行时使所述计算机执行用于对至少一个个体的遗传数据进行匿名化方法的至少一个步骤,所述方法包括以下步骤:
提供来自至少一个个体的遗传数据;
选择要研究的疾病;
确定所述遗传数据的至少一个子集,遗传数据的所述子集与所述要研究的疾病直接相关;
将不与所述要研究的疾病直接相关的剩余遗传数据分成多个子集,基于这些子集到与要研究的疾病直接相关的遗传数据的接近度将所述子集被分组到多于一个的层中,其中,优选地,所述接近度是基于对应于所述遗传数据的基因组途径网络来建立的;
对包含不与要研究的疾病直接相关的遗传数据的子集的所述多于一个的层进行匿名化。
在一个实施例中,所述计算机程序产品包括指令,所述指令在被执行时对包含遗传数据的不与要研究的疾病直接相关的子集的一个或多个层进行匿名化。通过使用选自统计匿名化,加密和安全多方匿名化和计算的组中的至少一种技术来执行一个或多个层的匿名化,如本文之前关于本发明的第一方面所述。
在另外和/或替代实施例中,计算机程序产品包括指令,所述指令在执行时将不与要研究的疾病直接相关的剩余遗传数据分成一个或多个子集,并基于这些子集中到与要研究的疾病直接相关的遗传数据的接近度分成一个或多个层。
在另外和/或替代实施例中,所述计算机程序产品包含指令,所述指令在执行时确定遗传数据的至少一个子集,遗传数据的所述子集与要研究的疾病直接相关。
在一个实施例中,如图3中所描述的方法可在计算机上被实施为计算机实施的方法,被实施为专用硬件,或者被实施为两者的组合。如在图4还示出的,针对计算机的指令,例如,可执行代码,可以被存储在计算机可读介质470上,例如机器可读物理标记的系列480的形式和/或作为具有不同的电学,例如,磁、或光学性质或值的元素。可执行代码可以以瞬态或者非瞬态的方式被存储。计算机可读介质的范例包括存储器设备,光学存储设备,集成电路,服务器,在线软件等。图4示出了光盘470。
应认识到,本发明适用于适于将本发明付诸实践的计算机程序,尤其是载体上或载体中的计算机程序。所述程序可以是源代码、目标代码、源代码和目标代码中间的代码(例如,以部分编译形式的形式)的形式,或者是适于用于在实施根据本发明的方法中使用的任何其它形式。还应理解,这样的程序可以具有许多不同的架构设计。例如,实施根据本发明的系统和方法的功能的程序代码可以被细分为一个或多个子例程。在这些子例程间分布功能的多种不同方式对本领域的技术人员将是显而易见的。子例程可以一起存储在一个可执行文件中以形成自包含的(self-contained)程序。这样的可执行文件可以包括计算机可执行指令,例如处理器指令和/或解释器指令(例如java解释器指令)。替代地,子例程中的一个或多个或全部可以被存储在至少一个外部库文件中并且与主程序静态或动态地,(例如在运行时)链接。主程序包括对子例程中的至少一个的至少一个调用。子例程也可包括对彼此的功能调用。涉及计算机程序产品的实施例包括与本文阐述的方法中的至少一个的每个处理阶段相对应的计算机可执行指令。这些指令可以被细分成子例程和/或存储在一个或多个可以静态或动态地链接的文件中。关于计算机程序产品的另一实施例包括与本文中提出的至少一种系统和/或产品的每个单元相对应的计算机可执行指令。这些指令可以被细分成子例程和/或存储在一个或多个可以静态或动态地链接的文件中。
计算机程序的载体可以是能够承载所述程序的任何实体或设备。例如,载体可包括数据存储设备,例如是rom(如cd-rom、或半导体rom),或是磁记录介质(例如硬盘)。此外,载体可以是可传输载体,例如电信号或光学信号,其可以经由电缆或光缆或通过无线电或其他手段被传输。当程序被实现在这样的信号中时,载体可以由这样的电缆或其他设备或单元组成。替代地,载体可以是嵌入了程序的集成电路,所述集成电路适于执行相关方法或在相关方法的实施中使用。
根据第三方面,本发明提供了一种用于对遗传数据进行匿名的系统。所述系统包括:
数据接口,其被配置为接收至少一个个体的遗传数据;用户输入接口,其被配置为从用户输入设备接收用户输入命令,用于选择要研究的疾病;以及
处理器,其被配置为:
确定来自所述至少一个个体的所述遗传数据中与所述要研究的疾病直接相关的遗传数据的(一个或多个)子集;
根据子集到与要研究的疾病直接相关的遗传数据的距离,将不与要研究的疾病直接相关的遗传数据的子集分成不同的层,其中,优选地,所述距离是基于对应于所述遗传数据的基因组途径网络来建立的;并且
将不与要研究的疾病直接相关的层或所述层中存在的不与要研究的疾病直接相关的遗传数据匿名化。
图5示出了系统600,其被配置为对遗传数据进行匿名化。系统600包括数据接口620,数据接口620被配置为访问至少一个个体的遗传数据624。数据接口620还与数据库634的基因组途径网络632通信。在图6的示例中,数据接口620被示出为连接到外部存储库622,例如合适的电子存储设备和/或数据库,其包括至少一个个体的遗传数据624。该数据接口620还连接到基因组途径网络632。替代地,所述至少一个个体的遗传数据624以及可以从系统600的内部数据存储器访问数据库634。通常,数据接口620可以采用各种形式,例如到局域网或广域网(例如因特网)的网络接口,到内部或外部数据存储的存储接口等。
此外,系统600被示出为包括被用户输入接口640用户输入接口640被配置为接收来自用户输入设备740的用户输入命令742,以使用户能够提供用户输入,例如选择或定义特定疾病、障碍或医学状况用于随后确定与所述疾病、障碍或医学状况直接相关的遗传数据的子集,以及不与所述疾病、障碍或医学状况直接相关的遗传数据,选择或选出与所选遗传数据相对应的基因组途径网络632。用户输入设备740可以采取各种形式,包括但不限于计算机鼠标、触摸屏、键盘等。图5示出了用户输入设备是计算机鼠标740。通常,用户输入接口640可以是与用户输入设备740的类型相对应的类型,即,它可以是与其对应的用户设备接口。
系统600还被示出为包括处理器660,处理器660被配置为确定遗传数据624的至少一个子集100,遗传数据624的子集100与要研究的疾病直接相关;将与要研究的疾病无直接关系的剩余遗传数据分成一个或多个子集并分成一个或多个层(200、300、400),基于这些子集与遗传数据的接近程度,这些数据与要研究的疾病直接相关;并且对包含不与要研究的疾病直接相关的遗传数据子集的一个或多个层进行匿名化。
处理器660被配置为通过利用基因组途径网络632来确定遗传数据子集与要研究的疾病的关系和/或与要研究的疾病直接相关的遗传数据子集的相对距离。
基因组途径网络632可通过以下方式在互联网上访问数据库,并且可以例如针对诸如前列腺癌ii型糖尿病或帕金森病的特定疾病来建立。
在一个示例中,基于所接收的用户输入命令742,处理器660可以将所述至少一个个体的遗传数据624经由数据接口620发送到所选择的基因组途径网络632。作为返回,处理器660可以从基因组途径网络632接收结果,所述结果指示指示遗传数据子集与要研究的疾病的关系和/或其至遗传数据与要研究的疾病直接相关的子集的相对距离。随后,处理器660可以进一步基于接收到指示遗传数据与要研究的疾病的关系的结果来将所述至少一个个体的遗传数据分组成遗传信息的子集或层。因此,已知与要研究的疾病直接相关的那些遗传数据(核心-疾病基因)由处理器660分组为子集100。不与要研究的疾病直接相关的遗传数据和/或遗传数据的层(200、300、400)随后根据其与遗传数据的与要研究的疾病直接相关的子集的相对距离而被分组。在这里,两个基因之间的“距离”由某些类型的相互作用决定。这样相互作用可以是共表达,蛋白质-蛋白质相互作用,共同发表等,或其任何组合。例如,string数据库列出了相互作用的一些可能(http://www.string-db.org/help/getting_started/#evidence)。处理器600还被配置为通过从包括统计匿名化、加密和安全多方匿名化和计算的一组算法中选择一个或多个算法来匿名化遗传数据和/或不与要研究的疾病直接相关的遗传数据的层(200、300、400)。该算法组被存储在存储器670中(图5中未示出)。
在优选的示例中,数据库634可以被包括在系统600中。因此,基于所接收的用户输入命令742,处理器660可以从外部储存库622接收至少一个个体的遗传数据624。处理器660还可以确定与数据库634相关联的遗传数据的子集。随后,处理器可以根据子集到与要研究的疾病直接相关的遗传数据的距离来将遗传数据的不与要研究的疾病直接相关的子集分成不同的层;稍后,处理器660可以对不与要研究的疾病直接相关的层或所述层中存在的不与要研究的疾病直接相关的遗传数据进行匿名化。下面可以找到说明如何对遗传数据的子集进行分类和匿名化的详细的示例。
处理器600还被配置为将匿名遗传数据662生成到输出设备760,例如显示器。替代地,显示器760可以是系统600的内部部分。
替代地,处理器600可以被配置为自动选择或定义特定的疾病,病症、障碍或医学状况,用于随后确定与所述疾病、障碍或医学状况直接相关的遗传数据的子集,以及不与所述疾病、障碍和医学状况直接相关的遗传数据,并且自动选择或选出对应于所选遗传数据的基因组途径网络632。
根据第四方面,本发明涉及该方法和/或计算机程序产品在生物信息学研究和/或诊断中的使用。
在一个实施例中,所述方法和/或计算机程序产品在生物信息学研究中使用。所述方法和/或计算机程序产品在生物信息学研究中的使用包括采集多个个体的遗传数据。第四方面所涵盖的所述方法和/或计算机程序产品在生物信息学中的研究领域中可以使用的生物信息学研究领域是且基因组学、遗传学、转录组学、蛋白质组学和系统生物学。
在替代实施例中,所述方法和/或计算机程序产品在诊断中被使用,其中,利用个体的遗传数据来分析个体是否受特定疾病的影响或是否有患上所述疾病或受所述疾病影响的风险。
本发明可应用于诊断领域和基因组学领域,其中,个体的遗传数据以分层结构组织,具有可立即用于进一步分析的核心数据集,以及敏感性增加的层,其可以在加密数据计算中揭示或使用。本发明改进了个体以及数据所有者的个体同意收集过程。个人确信他们的遗传数据被正确地匿名化,同时允许由研究进展引发的重新匿名化。因此,通过允许访问“与要对疾病进行研究相关的遗传数据进行分析或研究”,更容易定义个体的同意。
当提及单数名词时使用例如“一”、“一个”、“所述”的词语时,这包括该名词的复数,除非另有特别说明。此外,说明书和权利要求中的术语第一、第二、第三等用于区分相似元件,而不一定用于描述顺序或时间顺序。应理解,如此使用的术语在适当的情况下是可互换的,并且本文描述的本发明的实施方案能够用不同于本文描述或说明的其他顺序操作。此外,说明书和权利要求中的术语顶部,底部,上方,下方,之外等用于描述目的,而不一定用于描述相对位置。应理解,如此使用的术语在适当的情况下是可互换的,并且本文描述的本发明的实施方案能够用不同于本文描述或说明的其他顺序操作。应注意,在本说明书和权利要求中使用的术语“包括”不应被解释为限于此后列出的装置;其不排除其他元素或步骤。因此,表述“包括单元a和b的设备”的范围不应限于仅由部件a和b组成的设备。这意味着对于本发明,设备的仅相关部件是a和b。
应当注意,上面提及的实施例范例而不是限制本发明,并且本领域技术人员能够设计出许多替代实施例而不脱离所附权利要求的范围。在权利要求中,置于括号中的任何附图标记不应构成对权利要求的限制。本发明可以借助于包括若干不同元件的硬件来实施,以及借助于适当地编程的计算机来实施。在枚举了若干单元的装置型权利要求中,这些单元中的几个可以由同一硬件项来实现。尽管特定措施是在互不相同的从属权利要求中记载的,但是这并不指示不能有利地使用这些措施的集合。
范例
关于前列腺癌的面向疾病的基因组匿名化
在第一步中,针对前列腺癌途径,通过查找kegg途径数据库来检索核心前列腺癌基因列表(http://www.genome.jp/dbget-bin/www_bget?pathway:map05215)。
使用keggorthology检索了总共70个基因作为该途径的一部分,因为该数据库将属于多个物种的所有基因分组为直系同源组,消除了任何冗余。这70个基因都是被认为与前列腺癌直接相关的基因。这70个基因被归为“核心”。所述基因是:
pik3c=磷脂酰肌醇-4,5-二磷酸3-激酶[ec:2.7.1.153];
pten=磷脂酰肌醇-3,4,5-三磷酸3-磷酸酶和双特异性蛋白磷酸酶;klk3=semenogelase[ec:3.4.21.77];ctnnb1=连环蛋白beta1;bad=细胞死亡的bcl-2拮抗剂;bcl2=凋亡调节因子bcl-2;cdk2=细胞周期蛋白依赖性激酶2[ec:2.7.11.22];nfkb1=核因子-kappa-bp105亚基;tcf7=转录因子7;pik3r=磷酸肌醇-3-激酶,调节亚基;hras=gtpasehras;gsk3b=糖原合成酶激酶3beta[ec:2.7.11.26];sos=无七之子;htpg,hsp90a=分子伴侣htpg;egf=表皮生长因子;pdgfa=血小板衍生生长因子亚基a;egfr,erbb1=表皮生长因子受体[ec:2.7.10.1];fgfr1=成纤维细胞生长因子受体1[ec:2.7.10.1];pdgfra=血小板衍生生长因子受体α[ec:2.7.10.1];grb2=生长因子受体结合蛋白2;braf=b-raf原癌基因丝氨酸/苏氨酸蛋白激酶[ec:2.7.11.1];raf1=raf原癌基因丝氨酸/苏氨酸蛋白激酶[ec:2.7.11.1];map2k1,mek1=丝裂原活化蛋白激酶1[ec:2.7.12.2];map2k2,mek2=丝裂原活化蛋白激酶激酶2[ec:2.7.12.2];mapk1_3=丝裂原活化蛋白激酶1/3[ec:2.7.11.24];atf4,creb2=环amp依赖性转录因子atf-4;casp9=半胱天冬酶9[ec:3.4.22.62];tp53,p53=肿瘤蛋白p53;akt=rac丝氨酸/苏氨酸-蛋白激酶[ec:2.7.11.1];ikbka,ikka,chuk=核因子kappa-b激酶亚基抑制剂alpha[ec:2.7.11.10];tcf7l1=转录因子7样1;tcf7l2=转录因子7样2;lef1=淋巴增强子结合因子1;ep300,crebbp,kat3=e1a/creb结合蛋白[ec:2.3.1.48];ccnd1=细胞周期蛋白d1;ins=胰岛素;nfkbia=nf-kappa-b抑制剂alpha;rela=转录因子p65;erbb2/her2=受体酪氨酸蛋白激酶erbb-2[ec:2.7.10.1];insrr=胰岛素受体相关受体[ec:2.7.10.1];igf1r=胰岛素样生长因子1受体[ec:2.7.10.1];pdgfrb=血小板衍生生长因子受体β[ec:2.7.10.1];fgfr2=成纤维细胞生长因子受体2[ec:2.7.10.1];pdgfc_d=血小板衍生生长因子c/d;igf1=胰岛素样生长因子1;creb1=环amp反应元件结合蛋白1;pdpk1=3-磷酸肌醇依赖性蛋白激酶-1[ec:2.7.11.1];rb1=视网膜母细胞瘤相关蛋白;e2f3=转录因子e2f3;cdkn1b,p27,kip1=细胞周期蛋白依赖性激酶抑制剂1b;cdkn1a,p21,cip1=细胞周期蛋白依赖性激酶抑制剂1a;ccne=细胞周期蛋白e;mdm2=e3泛素-蛋白连接酶mdm2[ec:2.3.2.27];foxo1=叉头盒蛋白o1;mtor,frap,tor=丝氨酸/苏氨酸蛋白激酶mtor[ec:2.7.11.1];ikbkb,ikkb=核因子kappa-b激酶亚基抑制剂beta[ec:2.7.11.10];ikbkg,ikkg,nemo=核因子kappa-b激酶亚基抑制剂gamma;kras,kras2=gtpasekras;nras=gtpasenras;nr3c4,ar=雄激素受体;tgfa=转化生长因子,alpha;araf,araf1=a-raf原癌基因丝氨酸/苏氨酸蛋白激酶[ec:2.7.11.1];creb5,crebpa=环amp反应元件结合蛋白5;creb3;=环状amp响应元件结合蛋白3;nkx3-1=同源框蛋白nkx-3.1;e2f2=转录因子e2f2;hsp90b,tra1=热激蛋白90kdabeta;srd5a2=3-氧代-5-α-甾体4-脱氢酶2[ec:1.3.1.22];pdgfb=血小板衍生生长因子亚基b;以及e2f1=转录因子e2f1。
在随后的步骤中,创建核心前列腺癌网络,将核心前列腺癌基因列表复制粘贴到string数据库搜索页面(http://string-db.org/cgi/input.pl?input_page_active_form=multiple_identifiers)来创建网络:
http://bit.ly/28xp7ht(71个基因,选项‘最低要求的互动得分’:低置信度(0.150),选项‘禁用网络气泡内的结构预览’开启)
此后,创建了前列腺癌网络的第一层。
要创建第一层,“数据设置”并在‘2ndshell’字段输入:“选择了不超过20个相互作用者”。已添加的基因成为第一层的一部分(91个基因-71个基因=20个基因)。
在下一步骤中,创建了前列腺癌网络的第二层和外层。
要创建第二层,将这些基因输入string数据库搜索页面,并再次针对‘2ndshell’选项选择:‘不超过50个相互作用者'。所有新基因弹出,变为第二层的一部分(50个基因)。
在该示例中,第三层(或者,在这种情况下,外层)包括人类基因组中不属于核心或第一层的所有基因。
在随后的步骤中,基因组数据被匿名化。对于匿名化,使用具有针对100个个体的完整基因组(20457个基因,根据string数据库)的基因组数据(例如表达数据)的数据集。
71个基因的核心未被匿名化,因为需要这些前列腺癌相关基因的所有信息。
第一层20个基因通过统计匿名化进行匿名化,因为来自这些基因的信息可能很重要。更确切地说,这是通过泛化或抑制这些基因的值来实现的,以便针对所选择的k(例如k=2)和l(例如l=3)实现k-匿名性和l-多样性属性。
使用同态加密对第二层50个基因进行匿名化,因为来自这些基因的信息可能也是重要的。当该层具有更多数量的基因(例如,大于或等于50)时,所述方法可以更方便地应用。
20316个基因的外层是通过不可延展加密来匿名的,因为来自这些基因的信息对于我们对前列腺癌的特定研究并不重要。
- 还没有人留言评论。精彩留言会获得点赞!