一种肿瘤突变分析方法、系统、终端及可读存储介质与流程
本发明属于肿瘤基因检测技术领域,尤其涉及一种基于二代测序的肿瘤体细胞突变检测的生物信息分析方法、系统、终端及计算机可读存储介质。
背景技术:
我国是肿瘤高发国家,肿瘤基因检测对于我国肿瘤防治具有重要意义。随着二代测序(ngs)的快速发展,基因检测已经越来越多的指导临床肿瘤治疗实践,以分子检测的结果指导肿瘤临床治疗可以带来更多获益。肿瘤相关的突变有snv、indel、cnv、融合等,目前一些药物如靶向药物具有特异性针对某种肿瘤基因突变达到精准杀伤的效果,不同肿瘤患者肿瘤驱动基因突变存在差异,可能会导致不同患者对同一药物产生不同、甚至截然相反的治疗效果,因此通过基因检测,了解患者发生了那种基因突变,适合应用哪种药物,也就达到了“量体裁衣”的效果,做到了“精准医疗”。然而,现有的生物信息分析流程复杂,参数繁多,需要经验丰富的专业生物信息分析人员才能完成;并且一般只能检测一种或者几种突变类型,检测的不够全面。因此,一种能够全面快速地检测肿瘤类型的分析方法成为实际需求。
技术实现要素:
本发明的目的在于提供一种基于二代测序的肿瘤体细胞突变检测的生物信息分析方法、系统、终端及计算机可读存储介质的技术问题。
为了解决上述技术问题,本发明提供了一种基于二代测序的肿瘤突变分析方法,其特征在于,该方法包括:
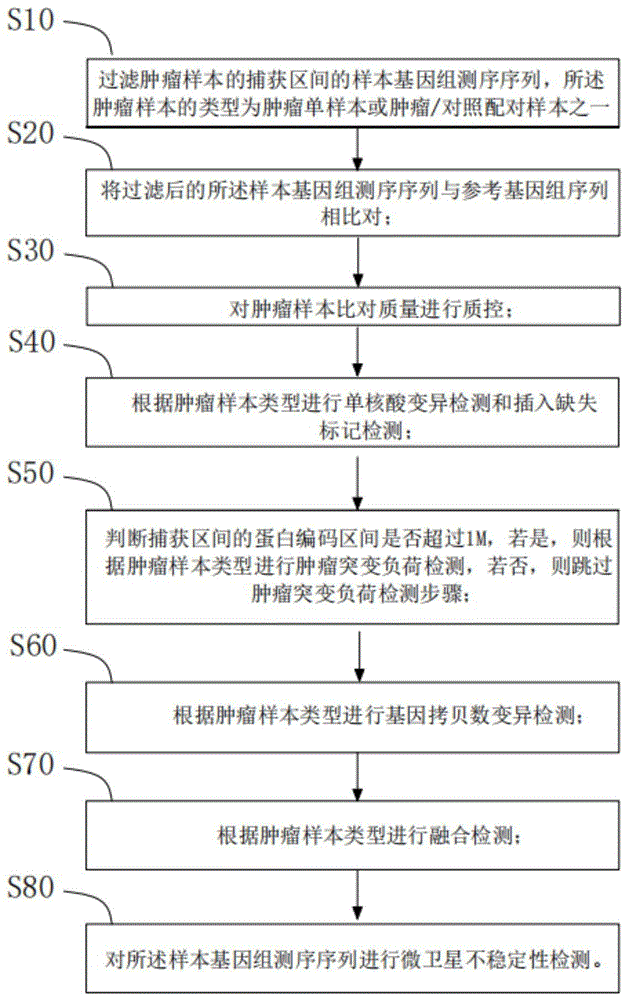
过滤肿瘤样本的捕获区间的样本基因组测序序列,所述肿瘤样本的类型为肿瘤单样本或肿瘤/对照配对样本之一;
将过滤后的所述样本基因组测序序列与参考基因组序列相比对;
对肿瘤样本比对质量进行质控,
根据肿瘤样本类型进行单核酸变异检测和插入缺失标记检测
判断捕获区间的蛋白编码区间是否超过1m,若是,则根据肿瘤样本类型进行肿瘤突变负荷检测,若否,则跳过肿瘤突变负荷检测步骤;
根据肿瘤样本类型进行基因拷贝数变异检测;
根据肿瘤样本类型进行融合检测;
对所述样本基因组测序序列进行微卫星不稳定性检测。
具体地,所述根据肿瘤样本类型进行单核酸变异检测和插入缺失标记检测的步骤中:
当所述肿瘤样本的类型为肿瘤单样本时,对所述肿瘤单样本进行单核酸变异检测和插入缺失标记检测,检测参数为突变丰度阈值不小于0.01,突变最低质量值为20,最低深度为10;
当所述肿瘤样本的类型为肿瘤/对照配对样本时,对所述肿瘤/对照配对样本进行单核酸变异检测和插入缺失标记检测,过滤参数为突变预测p值不大于0.05,突变丰度阈值不小于0.01,变异支持序列数不小于2条,对照突变支持率不大于0.01。
具体地,所述根据肿瘤样本类型进行肿瘤突变负荷检测的步骤中:
当所述肿瘤样本的类型为肿瘤单样本时,过滤掉所述肿瘤单样本中丰度低于5%的单核酸变异突变、插入缺失标记突变、已知的胚系突变、预测的胚系突变和已知的肿瘤驱动突变,保留肿瘤蛋白编码区间中的所有突变/肿瘤蛋白编码区间长度即为肿瘤突变负荷tmb值;
当所述肿瘤样本的类型为肿瘤/对照配对样本时,依次过滤掉所述肿瘤单样本中丰度低于5%的单核酸变异突变和插入缺失标记突变,保留肿瘤蛋白编码区间中的所有突变/肿瘤蛋白编码区间长度即为肿瘤突变负荷tmb值;
具体地,所述根据肿瘤样本类型进行基因拷贝数变异检测的步骤中:
当所述肿瘤样本的类型为肿瘤单样本时,对所述肿瘤单样本建立基线,再根据cbs算法对所述肿瘤单样本进行基因拷贝数变异检测;
当所述肿瘤样本的类型为肿瘤/对照配对样本时,根据cbs算法对所述肿瘤/对照配对样本进行基因拷贝数变异检测。
具体地,所述根据肿瘤样本类型进行融合检测的步骤中:
当所述肿瘤样本的类型为肿瘤单样本时,通过manta单样本模式进行融合检测;
当所述肿瘤样本的类型为肿瘤/对照配对样本时,通过manta配对样本模式进行融合检测。
具体地,所述对样本基因组测序序列进行微卫星不稳定性检测的步骤中:
获取所述捕获区间基因组序列的微卫星位点;
建立上述微卫星位点正常人群样本基线;
进行微卫星不稳定性检测。
具体地,所述获取所述探针捕获区间的微卫星位点;建立参考样本的基线;通过msings工具进行微卫星不稳定性检测的步骤具体包括:
扫描参考基因组序列获取所有的微卫星位点;
根据基因组位置获取所述探针富集芯片捕获区间的微卫星位点;
生成所述捕获区间的微卫星位点的基线。
基于所述生成的微卫星位点的基线进行微卫星不稳定性分析。微卫星不稳定性分析判定条件为不小于20%以上的微卫星位点不稳定为msi-high,10%-20%的微卫星位点不稳定为msi-low,<10%的微卫星位点不稳定为mass。
一种基于二代测序的肿瘤突变分析系统,所述系统包括:
过滤单元,过滤样本基因组测序序列;
肿瘤样本比对与质量质控生成单元,将过滤后的样本基因组测序序列与参考基因组序列相比对,比对质量质控生成肿瘤样本,所述肿瘤样本的类型为肿瘤单样本或肿瘤/对照配对样本之一;
单核酸变异检测和插入缺失标记检测单元,根据肿瘤样本类型进行单核酸变异检测和插入缺失标记检测;
肿瘤突变负荷检测单元,判断芯片捕获区间的蛋白编码区间是否超过1m,若是则根据肿瘤样本类型进行肿瘤突变负荷检测,若否则跳过肿瘤突变负荷检测步骤。
基因拷贝数变异检测单元,根据肿瘤样本类型进行基因拷贝数变异检测;
融合检测单元,根据肿瘤样本类型进行融合检测;
微卫星不稳定性检测单元,对样本进行微卫星不稳定性检测。
肿瘤样本生成单元分别与过滤单元、单核酸变异检测和插入缺失标记检测单元、肿瘤突变负荷检测单元、基因拷贝数变异检测单元、融合检测单元和微卫星不稳定性检测单元相连接。
一种终端,包括存储器、处理器以及存储在所述存储器中并可在所述处理器上运行的计算机程序,所述处理器执行所述计算机程序时实现所述系统的功能。
一种计算机可读存储介质,所述计算机可读存储介质存储有计算机程序,所述计算机程序被处理器执行时实现所述系统的功能。
本发明提供一整套自动化肿瘤突变生物信息分析流程,能够快速自动、全面的检测snv(单核酸变异)、indel(插入缺失标记)、融合、cnv(基因拷贝数变异)、tmb(肿瘤突变负荷)、msi等突变及标志物,能够挖掘更多肿瘤精准治疗靶点信息,为患者选择潜在获益的靶向药物提供更多帮助。本发明同时支持肿瘤单样本和肿瘤/对照配对样本模式,对于缺乏正常配对样本的肿瘤样本也能快速准确的检测出各种突变,能够适应不同的需求。
附图说明
图1是本发明实施例一的整体方法流程图;
图2是本发明实施例二的系统结构框图;
过滤单元1,肿瘤样本生成单元2,单核酸变异检测和插入缺失标记检测单元3,肿瘤突变负荷检测单元4,基因拷贝数变异检测单元5,融合检测单元6,微卫星不稳定性检测单元7
具体实施方式
下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明的一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有作出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
精准医学寻求利用基因组数据,帮助正确的患者在正确的时间提供正确的治疗。我国是肿瘤高发国家,肿瘤基因检测对于我国肿瘤防治具有重要意义。随着二代测序(ngs)的快速发展,基因检测已经越来越多的指导临床肿瘤治疗实践,以分子检测的结果指导肿瘤临床治疗可以带来更多获益。肿瘤相关的突变有snv、indel、cnv、融合等,目前一些药物如靶向药物具有特异性针对某种肿瘤基因突变达到精准杀伤的效果,不同肿瘤患者肿瘤驱动基因突变存在差异,可能会导致不同患者对同一药物产生不同、甚至截然相反的治疗效果,因此通过基因检测,了解患者发生了那种基因突变,适合应用哪种药物,也就达到了“量体裁衣”的效果,做到了“精准医疗”。因此对肿瘤突变进行全面的检测,能够挖掘更多肿瘤精准治疗靶点信息,为患者选择潜在获益的靶向药物提供更多帮助。近年来,肿瘤免疫治疗的研究突飞猛进,正在成为肿瘤治疗的一个重要方向。肿瘤突变负荷(tmb)、微卫星不稳定(msi)等免疫治疗标志物为临床医生选择免疫治疗药物提供更多指导,使得肿瘤患者从免疫治疗中得到更多获益。因此,全面检测各种类型的突变以及免疫治疗标志物将是基于ngs肿瘤基因检测的一个发展趋势。现有分析系统主要存在以下几个缺点:①生物信息分析流程复杂,参数繁多,需要经验丰富的专业生物信息分析人员才能完成;②目前的生信分析系统一般只能检测一种或者几种突变类型,检测的不够全面。针对上述不足,本发明进行以下创新:①将复杂生物信息分析整合成一套自动化分析流程,并对分析参数进行了大量优化,按照指定格式准备输入文件和数据,可以全自动完成分析,拿到最终结果。②本发明的生物信息分析流程能够快速准确、全面的检测snv、indel、融合、cnv、tmb、msi等突变及标志物,能够挖掘更多肿瘤精准治疗靶点信息,为患者选择潜在获益的靶向药物提供更多帮助。③本发明同时支持肿瘤单样本和肿瘤/对照配对样本模式,对于缺乏正常配对样本的肿瘤样本也能快速准确的检测出各种突变,能够适应不同的需求。
实施例一
参见图1,本实施例提供的一种基于二代测序的肿瘤突变分析方法,该方法包括:
步骤s10:过滤肿瘤样本的捕获区间的样本基因组测序序列,所述肿瘤样本的类型为肿瘤单样本或肿瘤/对照配对样本之一;
具体地,利用fastp自动识别并去除序列中含有的接头序列;去除测序质量差或者n含量高的序列,具体过滤参数为-q15-u50-n10。统计序列的数据量,q20,q30质量,gc含量等相关信息。
步骤s20:将过滤后的样本基因组测序序列与参考基因组序列相比对;
具体地,①通过bwamem算法将过滤后的序列比对到参考基因组(hg19)
②对①的结果利用picard里面的markduplicates去除由于pcr引入的重复序列
③对②的结果利用pisces-hygea对indel附近的区域进行重新比对
④对③步骤产生的bam文件使用gatk-baserecalibrator计算出了所有需要进行碱基重校正的read和特征值,然后使用gatk-applybqsr重新调整原来bam文件中的碱基质量值,并使用这个新的质量值重新输出一份新的bam文件。
步骤s30:对肿瘤样本比对质量进行质控;
具体地,①使用bamdst软件统计比对率、平均深度、捕获效率、pcr重复率、覆盖度等信息。
②质控标准为比对率不小于99%,平均深度不小于500x,并且500x以上的覆盖度不小于80%。
步骤s40:根据肿瘤样本类型进行单核酸变异检测和插入缺失标记检测;
单核酸变异检测(snv)和插入缺失标记检测(indel)分为两种情况,一是只有肿瘤单样本的情况下使用pisces工具进行检测,二是如果有肿瘤/对照配对样本,则使用varscan进行突变检测。
具体地,肿瘤单样本snv、indel检测:
当所述肿瘤样本的类型为肿瘤单样本时,对所述肿瘤单样本进行单核酸变异检测和插入缺失标记检测,检测参数为突变丰度阈值不小于0.01,突变最低质量值为20,最低深度为10;
进一步地,①使用pisces工具(piscesvariantqualityrecalibration工具)对步骤s30最终生成的质量矫正后的bam进行snv、indel检测。检测参数为突变丰度阈值不小于0.01,突变最低质量值为20,最低深度为10。
进一步地,②使用pisces工具对①生成的vcf结果进行质量矫正。
具体地,肿瘤/对照配对样本snv、indel检测:
当所述肿瘤样本的类型为肿瘤/对照配对样本时,对所述肿瘤/对照配对样本进行单核酸变异检测和插入缺失标记检测,过滤参数为突变预测p值不大于0.05,突变丰度阈值不小于0.01,变异支持序列数不小于2条,对照突变支持率不大于0.01。
进一步地,①对步骤s3生成的肿瘤/对照样本使用samtoolsmpileup生成varscan所需的mpileup文件,参数为:-b-q1-c50-d20000。
进一步地,②使用varscansomatic工具配对样本模式进行突变检测,过滤参数为突变预测p值不大于0.05,突变丰度阈值不小于0.01,变异支持序列数不小于2条,对照突变支持率不大于0.01。
步骤s50:判断捕获区间的蛋白编码区间是否超过1m,若是,则根据肿瘤样本类型进行肿瘤突变负荷检测,若否,则跳过肿瘤突变负荷检测步骤。
具体地,如果panelcds区间超过1m,则进行tmb计算,否则将不进行计算。tmb计算也分为两种情况,即肿瘤单样本模式和肿瘤/对照配对样本模式。
具体地,肿瘤单样本tmb计算:
当所述肿瘤样本的类型为肿瘤单样本时,依次过滤掉所述肿瘤单样本中丰度低于5%的单核酸变异突变、插入缺失标记突变、已知的胚系突变、预测的胚系突变和已知的肿瘤驱动突变,保留肿瘤蛋白编码区间中的所有突变/肿瘤蛋白编码区间长度即为肿瘤突变负荷tmb值;
进一步地,肿瘤单样本tmb计算将按照下面步骤依次进行:
①过滤掉丰度低于5%的snv、indel突变。
②过滤掉dbsnp、exac、1000g、esp6500数据库中已知的胚系突变。
通过somatic-germline/zygosity(sgz)algorithm算法过滤掉预测的胚系突变。
③过滤掉intogen数据库中已知的肿瘤驱动突变
④保留的panelcds区间中的所有突变/panelcds区间长度(m)即为tmb值。
具体地,肿瘤/对照配对样本tmb计算
当所述肿瘤样本的类型为肿瘤/对照配对样本时,过滤掉所述肿瘤单样本中丰度低于5%的单核酸变异突变和插入缺失标记突变,保留肿瘤蛋白编码区间中的所有突变/肿瘤蛋白编码区间长度即为肿瘤突变负荷tmb值。
进一步地,肿瘤/对照配对样本tmb计算将按照下面步骤依次进行:
①过滤掉丰度低于5%的snv、indel突变。
②保留的panelcds区间中的所有突变/panelcds区间长度(m)即为tmb值。
步骤s60:根据肿瘤样本类型进行基因拷贝数变异检测;
具体地,cnv分为两种情况,如果是肿瘤单样本,则需要先使用cnvkitreference建立基线,再使用cnvkitcbs算法进行cnv检测。如果是双样本,则使用cnvkit配对模式进行体细胞cnv检测。
具体地,肿瘤单样本cnv检测:
具体地,当所述肿瘤样本的类型为肿瘤单样本时,对所述肿瘤单样本建立基线,再使用cbs算法对所述肿瘤单样本进行基因拷贝数变异检测;
进一步地,肿瘤单样本cnv检测将按照下面步骤依次进行:
①肿瘤单样本情况下需要先建立基线,基线样本要求为血液白细胞样本或者癌旁组织样本,样本数量不小于10,样本深度不小于500x。
②基线建立方法为:使用cnvkitcoverage生成所有样本对应检测panel的深度覆盖文件(sample.targetcoverage.cnn和sam-ple.antitargetcoverage.cnn);使用cnvkitreference工具基于上述所有深度覆盖文件生成基线。
③基于②生成的基线使用cnvkit对肿瘤样本进行cnv检测,检测算法为cbs算法。
具体地,肿瘤/对照配对cnv检测:
当所述肿瘤样本的类型为肿瘤/对照配对样本时,根据cbs算法对所述肿瘤/对照配对样本进行基因拷贝数变异检测。
进一步地,肿瘤/对照配对cnv检测将按照下面步骤依次进行:
①使用cnvkit肿瘤/对照双样本模式进行cnv检测,检测算法为cbs算法。
步骤s70:根据肿瘤样本类型进行融合检测;
具体地:当所述肿瘤样本的类型为肿瘤单样本时,通过manta单样本模式进行融合检测;
当所述肿瘤样本的类型为肿瘤/对照配对样本时,通过manta配对样本模式进行融合检测。
进一步地,如果是单样本,则使用manta单样本模式进行融合检测,如果是配对样本则使用manta配对样本模式进行检测。过滤条件为支持突变reads比例不小于0.01,splitreads不小于3,pairreads不小于3。
步骤s80:对样本基因组测序序列进行微卫星不稳定性检测。
具体地,msi检测先需要获得panel的微卫星位点,然后建立正常样本的基线,使用msings进行msi检测。
具体地,获取所述样本基因组测序序列的肿瘤蛋白编码区间的微卫星不稳定性位点;
进一步地,扫描参考基因组序列获取所有的微卫星性位点;根据基因组位置获取所述微卫星位点中在捕获区间的微卫星位点;
具体地,建立上述微卫星位点的正常人群基线;
进一步地,生成所述捕获区间的微卫星位点的基线。
具体地,进行微卫星不稳定性检测。
进一步地,基于所述生成捕获区间微卫星位点的基线进行微卫星不稳定性分析。微卫星不稳定性分析判定条件为不小于20%以上的微卫星位点不稳定为msi-high,10%-20%的微卫星位点不稳定为msi-low,<10%的微卫星位点不稳定为mass。
具体地,具体方法如下:
①使用msisensorscan工具扫描整个参考基因组(hg19)获得所有的微卫星位点
②根据基因组位置获得①中在panel捕获区间的微卫星位点
③使用msings生成②中微卫星位点基线。基线样本要求为正常人血液白细胞样本,测序深度不小于500x,样本数量不小于20。
④使用msings基于③生成的基线进行msi分析。msi判定条件为不小于20%以上的微卫星位点不稳定为msi-high,10%-20%的微卫星位点不稳定为msi-low,<10%的微卫星位点不稳定为mass。①-③步可以在肿瘤样本分析之前准备好,后续分析可以使用③生成的文件作为流程分析配置文件,直接进行样本msi分析。
实施例二
本实施例提供一种基于二代测序的肿瘤突变分析系统,参见图2,所述系统包括:
过滤单元,过滤样本基因组测序序列;
肿瘤样本比对及质量控制单元,将过滤后的样本基因组测序序列与参考基因组序列相比对,比对质量质控生成肿瘤样本,所述肿瘤样本的类型为肿瘤单样本或肿瘤/对照配对样本之一;
单核酸变异检测和插入缺失标记检测单元,根据肿瘤样本类型进行单核酸变异检测和插入缺失标记检测;
肿瘤突变负荷检测单元,判断panel捕获区间的蛋白编码区间是否超过1m,若是则根据肿瘤样本类型进行肿瘤突变负荷检测,若否则跳过肿瘤突变负荷检测步骤。
基因拷贝数变异检测单元,根据肿瘤样本类型进行基因拷贝数变异检测;
融合检测单元,根据肿瘤样本类型进行融合检测;
微卫星不稳定性检测单元,对样本基因组进行微卫星不稳定性检测。
肿瘤样本比对与质控单元分别与过滤单元、单核酸变异检测和插入缺失标记检测单元、肿瘤突变负荷检测单元、基因拷贝数变异检测单元、融合检测单元和微卫星不稳定性检测单元相连接。
具体地,所述过滤单元利用fastp自动识别并去除序列中含有的接头序列;去除测序质量差或者n含量高的序列,具体过滤参数为-q15-u50-n10。统计序列的数据量,q20,q30质量,gc含量等相关信息。
具体地,肿瘤样本比对单元通过bwamem算法将过滤后的序列比对到参考基因组(hg19),利用picard里面的markduplicates去除由于pcr引入的重复序列,利用pisces-hygea对indel附近的区域进行重新比对,产生的bam文件使用gatk-baserecalibrator计算出了所有需要进行碱基重校正的read和特征值,然后使用gatk-applybqsr重新调整原来bam文件中的碱基质量值,并使用这个新的质量值重新输出一份新的bam文件。
具体地,肿瘤样本质控生成单元使用bamdst软件统计比对率、平均深度、捕获效率、pcr重复率、覆盖度等信息,质控标准为比对率不小于99%,平均深度不小于500x,并且500x以上的覆盖度不小于80%。
具体地,肿瘤单样本snv、indel检测时:
单核酸变异检测和插入缺失标记检测单元使用pisces工具(piscesva-riantqualityrecalibration工具)对步骤s30最终生成的质量矫正后的bam进行snv、indel检测。检测参数为突变丰度阈值不小于0.01,突变最低质量值为20,最低深度为10。使用pisces工具对①生成的vcf结果进行质量矫正。
具体地,肿瘤/对照配对样本snv、indel检测时:
单核酸变异检测和插入缺失标记检测单元对步骤s3生成的肿瘤/对照样本使用samtoolsmpileup生成varscan所需的mpileup文件,参数为:-b-q1-c50-d20000。使用varscansomatic工具配对样本模式进行突变检测,过滤参数为突变预测p值不大于0.05,突变丰度阈值不小于0.01,变异支持序列数不小于2条,对照突变支持率不大于0.01。
具体地,肿瘤突变负荷检测单元判断panel捕获区间蛋白编码区间是否超过1m,若是则根据肿瘤样本类型进行肿瘤突变负荷检测,若否则跳过肿瘤突变负荷检测步骤。
具体地,肿瘤单样本tmb计算:
当所述肿瘤样本的类型为肿瘤单样本时,肿瘤突变负荷检测单元依次过滤掉丰度低于5%的snv、indel突变;过滤掉dbsnp、exac、1000g、esp6500数据库中已知的胚系突变。通过somatic-germline/zygosity(sgz)algorithm算法过滤掉预测的胚系突变;过滤掉intogen数据库中已知的肿瘤驱动突变;保留的panelcds区间中的所有突变/panelcds区间长度(m)即为tmb值。
具体地,肿瘤/对照配对样本tmb计算
肿瘤突变负荷检测单元过滤掉丰度低于5%的snv、indel突变。保留的panelcds区间中的所有突变/panelcds区间长度(m)即为tmb值。
具体地,如果是肿瘤单样本,基因拷贝数变异检测单元则需要先使用cnvkitreference建立基线,再使用cnvkitcbs算法进行cnv检测。如果是双样本,则使用cnvkit配对模式进行体细胞cnv检测。
具体地,肿瘤单样本cnv检测:
具体地,基因拷贝数变异检测单元先建立基线,基线样本要求为血液白细胞样本或者癌旁组织样本,样本数量不小于10,样本深度不小于500x。基线建立方法为:使用cnvkitcoverage生成所有样本对应检测panel的深度覆盖文件(sample.targetcoverage.cnn和sample.antitargetcoverage.cnn);使用cnvkitreference工具基于上述所有深度覆盖文件生成基线。基于生成的基线使用cnvkit对肿瘤样本进行cnv检测,检测算法为cbs算法。
具体地,肿瘤/对照配对cnv检测:
具体地,基因拷贝数变异检测单元使用cnvkit肿瘤/对照双样本模式进行cnv检测,检测算法为cbs算法。
具体地,如果是单样本,则融合检测单元使用manta单样本模式进行融合检测,如果是配对样本则使用manta配对样本模式进行检测。过滤条件为支持突变reads比例不小于0.01,splitreads不小于3,pairreads不小于3。
具体地,微卫星不稳定性检测单元,使用msisensorscan工具扫描整个参考基因组(hg19)获得所有的微卫星位点。根据基因组位置获得在panel捕获区间的微卫星位点。使用msings生成的微卫星位点基线。基线样本要求为正常人血液白细胞样本,测序深度不小于500x,样本数量不小于20。使用msings基于生成的基线进行msi分析。msi判定条件为不小于20%以上的微卫星位点不稳定为msi-high,10%-20%的微卫星位点不稳定为msi-low,<10%的微卫星位点不稳定为mass。上述可以在肿瘤样本分析之前准备好,后续分析可以使用生成的文件作为流程分析配置文件,直接进行样本msi分析。
实施例三
本实施例提供一种控制终端,以及应用于该终端的计算机可读存储介质,所述计算机可读存储介质存储有计算机程序,所述计算机程序被处理器执行时实现上述实施例二所述系统的功能。
所述终端包括存储器、处理器以及存储在所述存储器中并可在所述处理器上运行的计算机程序,
示例性的,计算机程序可以被分割成一个或多个模块,一个或者多个模块被存储在存储器中,并由处理器执行,以完成本发明。一个或多个模块可以是能够完成特定功能的一系列计算机程序指令段,该指令段用于描述计算机程序在用户终端中的执行过程。
本领域技术人员可以理解,上述中控单元的描述仅仅是示例,并不构成对中控单元的限定,可以包括比上述描述更多或更少的部件,或者组合某些部件,或者不同的部件,例如可以包括输入输出设备、网络接入设备、总线等。
具体地,所述处理器可以是中央处理单元(centralprocessingunit,cpu),还可以是其他通用处理器、数字信号处理器(digitalsignalproces-sor,dsp)、专用集成电路(applicationspecificintegratedcircuit,asic)、现成可编程门阵列fieldprogrammablegatearray,fpga)或者其他可编程逻辑器件、分立门或者晶体管逻辑器件、分立硬件组件等。
进一步地,通用处理器可以是微处理器或者该处理器也可以是任何常规的处理器等,所述处理器是所述中控单元的控制中心,利用各种接口和线路连接整个中控单元的各个部分。
具体地,所述存储器可用于存储所述计算机程序和/或模块,所述处理器通过运行或执行存储在所述存储器内的计算机程序和/或模块,以及调用存储在存储器内的数据,实现所述中控单元的各种功能。
进一步地,所述存储器可主要包括存储程序区和存储数据区。
其中,存储程序区可存储操作系统、至少一个功能所需的应用程序(比如声音播放功能、图像播放功能等)等;存储数据区可存储根据终端的使用所创建的数据(比如音频数据、电话本等)等。此外,存储器可以包括高速随机存取存储器,还可以包括非易失性存储器,例如硬盘、内存、插接式硬盘,智能存储卡(smartmediacard,smc),安全数字(securedigital,sd)卡,闪存卡(flashcard)、至少一个磁盘存储器件、闪存器件、或其他易失性固态存储器件。
具体地,所述中控单元集成的模块/单元如果以软件功能单元的形式实现并作为独立的产品销售或使用时,可以存储在一个计算机可读取存储介质中。
基于这样的理解,本发明实现上述实施例方法中的全部或部分流程,也可以通过计算机程序来指令相关的硬件来完成,所述的计算机程序可存储于一计算机可读存储介质中,该计算机程序在被处理器执行时,可实现上述方法实施例的步骤。
其中,所述计算机程序包括计算机程序代码,所述计算机程序代码可以为源代码形式、对象代码形式、可执行文件或某些中间形式等。所述计算机可读介质可以包括:能够携带所述计算机程序代码的任何实体或装置、记录介质、u盘、移动硬盘、磁碟、光盘、计算机存储器、只读存储器(rom,read-onlymemory)、随机存取存储器(ram,randomaccessmemory)、电载波信号、电信信号以及软件分发介质等。
藉此,本发明提供一整套自动化肿瘤突变生物信息分析流程,能够快速自动、全面的检测snv(单核酸变异)、indel(插入缺失标记)、融合、cnv(基因拷贝数变异)、tmb(肿瘤突变负荷)、msi等突变及标志物,能够挖掘更多肿瘤精准治疗靶点信息,为患者选择潜在获益的靶向药物提供更多帮助。本发明同时支持肿瘤单样本和肿瘤/对照配对样本模式,对于缺乏正常配对样本的肿瘤样本也能快速准确的检测出各种突变,能够适应不同的需求。
以上所述仅为本发明的较佳实施例而已,并不用以限制本发明,凡在本发明的精神和原则之内所作的任何修改、等同替换和改进等,均应包含在本发明的保护范围之内。
- 还没有人留言评论。精彩留言会获得点赞!