基因变异位点筛选方法及系统与流程
本申请涉及基因变异位点筛选方法及系统,属于生物医学技术领域。
背景技术:
随着人类基因组计划的顺利完成,开启了人类健康与生命科学研究的新时代。生物样本库的不断发展及技术的日趋成熟,更是为人类疾病尤其是重大慢性疾病的研究提供了丰富的样本资源及临床数据支撑。采用基因芯片技术对样本进行基因分型,通过队列基因数据的生物信息学分析去寻找特定的生物标志物,成为人类攻克一系列复杂疾病的强有力的技术手段。通过基因芯片技术获取基因分型数据,其宝贵价值也日益得到人们的理解与重视,世界各国政府及科研单位更是投入大量资源针对特定国家及地区的特定人群队列进行了诸多人群队列的基因分型工作。
由于不同国家和地区的人群在基因型上有很大区别,所以在对样本进行基因分型时,所使用的基因芯片是有针对性的,其针对的是特定的国家和人群。现有技术中,并没有针对亚洲人群的基因芯片。若想制备针对亚洲人群的基因芯片,需要筛选出针对亚洲人的基因变异位点。现有技术中,使用affymetrix软件从数据集中筛选基因变异位点,但是affymetrix软件筛选的过程中,是从大量的基因变异位点中利用特定的方法筛选出有代表性的基因变异位点,而其他变异位点,虽然并未满足筛选条件,但是其仍然有部分基因变异位点包含有效信息,所以affymetrix软件所筛选出的基因变异位点覆盖并不全面。
技术实现要素:
本发明的目的在于,提供一种基因变异位点筛选方法,以从经过affymetrix软件筛选后的数据集中获取更多的基因变异位点,使得所制备的基因芯片中包含更为全面的基因变异位点。
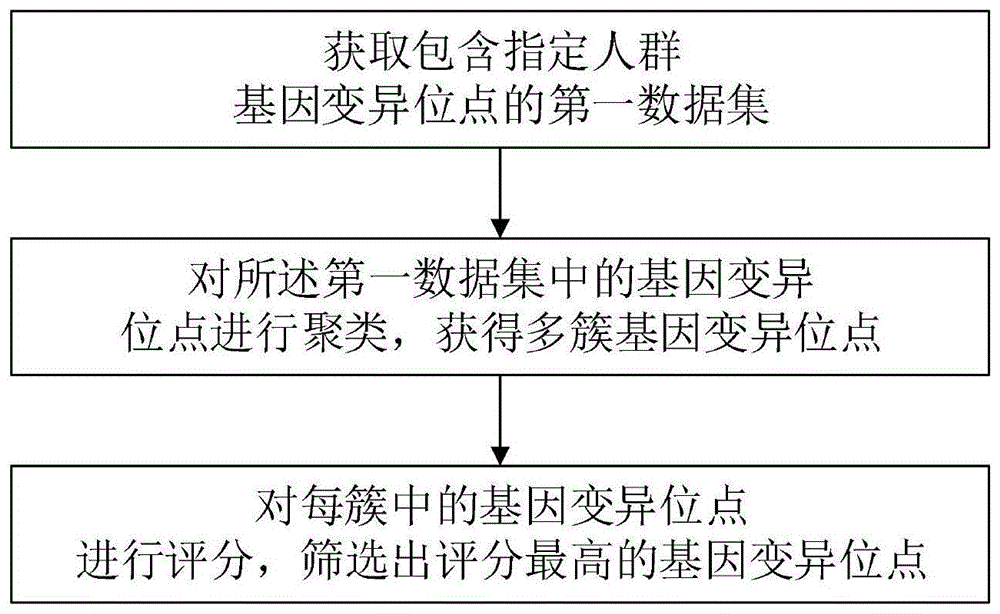
本发明提供了一种基因变异位点筛选方法,包括:
获取包含指定人群基因变异位点第一数据集;
对所述第一数据集中的基因变异位点进行聚类,获得多簇基因变异位点;
对每簇中的基因变异位点进行评分,筛选出评分大于预设阈值的基因变异位点。
优选地,获取包含指定人群基因变异位点的第一数据集,具体为:
获取指定人群的基因测序数据,提取基因测序数据中的基因变异位点,得到原始数据集;
利用预设筛选条件对所述原始数据集进行筛选,将筛选获得的基因变异位点记为第一基因变异位点;
将所述原始数据集中所述第一基因变异位点之外的基因变异位点作为所述第一数据集。
进一步地,所述指定人群为中国人。指定人群的基因测序数据为中国人的30倍测序深度的全基因组测序数据。
优选地,对所述第一数据集中的基因变异位点进行聚类,获得多簇基因变异位点,具体为:
获取所述第一数据集中基因变异位点的连锁不平衡值;
基于所述连锁不平衡值对所述第一数据集中的基因变异位点进行聚类,获得多簇基因变异位点。
进一步地,获取所述第一数据集中基因变异位点的连锁不平衡值,具体为:
获取所述第一数据集中次等位基因频率大于等于3%的基因变异位点,组成聚类数据集;
获取所述聚类数据集中基因变异位点的连锁不平衡值。
进一步地,所述聚类数据集中的基因变异位点的次等位基因频率大于等于5%。
进一步地,获取所述聚类数据集中基因变异位点的连锁不平衡值,具体为:
获取所述聚类数据集中,每个基因变异位点与所述聚类数据集中的其他基因变异位点之间的皮尔逊相关系数rij,其中,0<i,j≤n,n为所述聚类数据集中基因变异位点的数量;
根据所述皮尔逊相关系数rij确定连锁不平衡值
优选地,所述对每簇中的基因变异位点进行评分,具体为:
获取经过湿测试的验证数据集;
判断每簇中的每个基因变异位点是否包含于验证数据集中,如果是,则基于验证数据集中的基因变异位点所使用的探针数量对簇中基因变异位点进行评分,筛选出评分大于预设阈值的基因变异位点。评分过程中,所使用探针数量最少的基因变异位点评分最高。
本发明还提供了一种基于上述基因变异位点筛选方法的计算机系统,所述计算机系统被编程以执行上述基因变异位点筛选方法的步骤。
本发明的基因变异位点筛选方法及系统,相较于现有技术,具有如下有益效果:
本发明的基因变异位点筛选方法,利用评分制对经过聚类的变异位点进行筛选,使得筛选出的基因变异位点具有代表性,保证了筛选的质量。
本发明的基因变异位点筛选方法,更加合理、充分的利用了第一数据集中的基因变异位点,避免了有效基因变异位点的遗漏,使得制备的基因芯片中,包含了更加全面的基因变异位点。
本发明使用全基因组测序数据作为基础数据集,可以获得整个基因组的数据,避免基因不全影响后续制备的基因芯片的精确性,同时,由于全基因组测序数据为高分辨率数据,便于从中获取大型、小型全面的变异位点。
附图说明
图1为本发明一种基因变异位点筛选方法的流程图。
具体实施方式
本发明的基因变异位点筛选方法的流程图参见图1,其具体实施过程为:
本实施例是以包含2641个中国人的30倍测序深度的全基因组测序数据为基本数据集。使用全基因组测序数据作为基本数据集,可以获得整个基因组的数据,避免基因不全影响后续制备的基因芯片的精确性,同时,由于全基因组测序数据为高分辨率数据,便于从中获取大型、小型全面的变异位点。本实施例使用中国人的全基因组测序数据,以便利用中国人的基因变异位点,制备针对中国人的基因芯片。
首先,利用gatk工具从基础数据集中提取基因变异位点,得到原始数据集,然后利用affymetrix软件从原始数据集中筛选出第一基因变异位点,本实施例中共筛选出514221个基因变异位点。由于原始数据集中包含大量的基因变异位点,而affymetrix软件利用设定的条件所筛选出的基因变异位点具有局限性,所筛选出的基因变异位点覆盖并不全面,所以将affymetrix软件筛选后的剩余基因变异作为第一数据集,对该数据集进行进一步的筛选,以筛选出更为全面的基因变异位点。
对所述第一数据集进一步筛选的步骤为:
获取所述第一数据集中次等位基因频率大于等于3%的基因变异位点,组成聚类数据集;优选的,所选取的基因变异位点的次等位基因频率为5%以上。限定基因变异位点的次等位基因的目的在于,位于限定范围内的次等位基因,其包含的信息量更多,更利于制备基因芯片。如不限定次等位基因频率,则会导致数据集较大,增加处理时间及处理繁琐度。然后,获取所述聚类数据集中基因变异位点的连锁不平衡值,计算连锁不平衡值的过程为:
获取所述聚类数据集中,每个基因变异位点与所述聚类数据集中的其他基因变异位点之间的皮尔逊相关系数rij,其中,0<i,j≤n,n为所述聚类数据集中基因变异位点的数量;然后根据所述皮尔逊相关系数rij确定连锁不平衡值
基于所获取连锁不平衡值,以
进一步地,判断每簇中的每个基因变异位点是否包含于验证数据集中;如果簇中基因变异位点包含于验证数据集中,则基于验证数据集中的基因变异位点所使用的探针数量对簇中基因变异位点进行评分,所使用探针数量最少的基因变异位点评分最高,本实施例筛选出的基因变异位点为评分最高的基因变异位点。当然,也可以预设阈值,从而在每个簇中筛选出评分大于阈值的多个基因变异位点。本实施例中验证数据集为affymetrix提供的经过湿测试的基因变异位点数据集。该数据集中包含了很多的在基因芯片上表现较好的基因变异位点。利用本发明的方法,经过上述步骤共获得了104866个基因变异位点。
进一步地,利用affymetrix软件筛选出的514221个基因变异位点和利用本发明的方法筛选出的104866个基因变异位点制备基因芯片,获得的基因芯片上包含的基因更加全面。
本发明的基因变异位点筛选方法,利用评分制对经过聚类的变异位点进行筛选,使得筛选出的基因变异位点具有代表性,保证了筛选的质量。
本发明的基因变异位点筛选方法,更加合理、充分的利用了数据集中的基因变异位点,避免了有效基因变异位点的遗漏,使得制备的基因芯片中,包含了更加全面的基因变异位点。
本发明使用全基因组测序数据作为基础数据集,可以获得整个基因组的数据,避免基因不全影响后续制备的基因芯片的精确性,同时,由于全基因组测序数据为高分辨率数据,便于从中获取大型、小型全面的变异位点。
以上所述,仅是本申请的实施例,并非对本申请做任何形式的限制,虽然本申请以较佳实施例揭示如上,然而并非用以限制本申请,任何熟悉本专业的技术人员,在不脱离本申请技术方案的范围内,利用上述揭示的技术内容做出些许的变动或修饰均等同于等效实施案例,均属于技术方案范围内。
- 还没有人留言评论。精彩留言会获得点赞!