海岛棉GbNAC1在抗黄萎病中的应用的制作方法
本发明属于基因工程技术领域,具体涉及海岛棉GbNAC1基因在抗黄萎病中的应用专利申请。
背景技术:
在植物的生长发育过程中,经常会遭受到各种病原菌或者干旱、高盐、低温等一系列生物与非生物胁迫。这些胁迫通常会导致植物的损伤进而影响到植物的生长发育及产量。转录因子是一类调节基因表达的重要调控基因,在植物抵御逆境胁迫的过程中起着非常重要的作用,通过与目的基因启动子中的特定DNA序列结合以激活或抑制目标基因的转录表达。在植物中存在着大量的编码不同类别转录因子的基因,其中AP2/ERF、bZIP、WRKY、MYB等转录因子与植物的抗逆胁迫反应有关。
棉花是一类非常重要的经济作物,在食品、纺织等领域应用相当广泛。相较于玉米和小麦等其他农作物,棉花在耐盐碱和干旱方面的能力比较强,但是却非常容易感染黄萎病。棉花黄萎病(在主要的棉花种植区域中,大丽轮枝菌是棉花黄萎病的主要致病菌)既是一种通过土壤传播的真菌类维管束病害,又是一种传染性病害。棉花被黄萎病菌入侵后,病株由下部叶片开始发生,逐渐向上发展,病叶边缘稍向上卷曲,叶脉间出现不规则的斑块,甚至整个叶片枯焦,脱落成光杆,茎部维管束褐化,花和果实减少。初步统计表明,在棉花产区黄萎病呈逐年加重趋势,但目前仍然缺乏较为有效的防范手段。
近年来在陆生植物中发现了系列NAC转录因子,构成了庞大的转录因子家族。NAC转录因子家族的命名来源于矮牵牛的NAM、拟南芥的ATAF1、ATAF2及CUC2的首字母缩写。目前已在拟南芥中确认了117种NAC基因,在水稻、葡萄、杨树、大豆中分别确认了151、79、163种NAC基因,NAC转录因子N端含有高度保守的约150个氨基酸结构域,在植物生长发育、逆境胁迫中起着重要作用。
NAC域中包含了5个子域,分别为A、B、C、D、E。A、C、D子域高度保守,且C和D子域含有核定位信号且带正电荷,与DNA结合有关;A可能参与功能二聚体的形成;B和E子域多变,可能是NAC基因功能多样性的原因之一。NAC转录因子的C端为转录调控区,具有激活或抑制靶基因转录的活性。NAC转录因子的C端也存在13个相对保守的基序,分布于不同的NAC亚组中,同一亚组中如C端转录调控区含有丝氨酸、苏氨酸、脯氨酸、谷氨酸等重复出现频率较高的简单氨基酸,这一特性使得同一亚组的NAC转录因子可能具有相似的生物学功能。
研究棉花NAC转录因子在抗黄萎病中的作用,有利于棉花品质改良和抗胁迫性研究工作的进一步开展,对其与黄萎病间的作用机制也有必要加以研究,从而为黄萎病的预防和控制提供一定的借鉴和参考。
技术实现要素:
本发明目的在于提供海岛棉基因GbNAC1在抗黄萎病中的新应用,利用该新用途,可培育新的抗黄萎病的棉花新品种,从而为棉花黄萎病的预防和控制提供新的途径。
本发明所采取的技术方案详述如下。
海岛棉GbNAC1基因在抗黄萎病中的应用,所述GbNAC1基因,其ORF序列含543bp,GenBank ID: KP317496;该基因在细胞中的亚细胞定位在细胞核内;在棉花的根、茎、真叶、子叶、茎尖等组织中均有表达,但在真叶中的表达量最高,而在茎尖中的表达量最低;
该基因与植物黄萎病抗性相关;进一步而言,在棉花受到黄萎病侵染时,GbNAC1基因的表达受到抑制,表达量呈现下降。
进一步转基因实验表明,在将CbNAC1基因沉默后,植物的黄萎病抗性有所降低,而将该基因超表达后,植物对黄萎病的抗性增强,所述植物具体为棉花或者拟南芥等。
本发明首先克隆获得了海岛棉GbNAC1基因,并对该基因结构及相关特性进行了深入研究。在对该基因与植物的黄萎病相关性研究时,发明人发现在植物受到黄萎病菌侵染时,该基因表达量出现了表达量降低的明显变化,进一步将该基因沉默后,植物的抗黄萎病抗性降低,而该基因超表达后,植物对黄萎病抗性增加。基于上述特性,通过培育或者筛选GbNAC1基因超表达的植株,可为获得黄萎病抗性较好植物新品种提供借鉴和参考,因而在植物新品种培育方面具有较好地应用价值。
附图说明
图1为GbNAC1基因的克隆和菌液PCR检测的电泳图,其中A:GbNAC1基因的克隆其中M表示Marker, 1-2表示2个PCR重复;;B:菌液PCR的检测结果,其中M表示Marker,1-5表示5个单菌落;
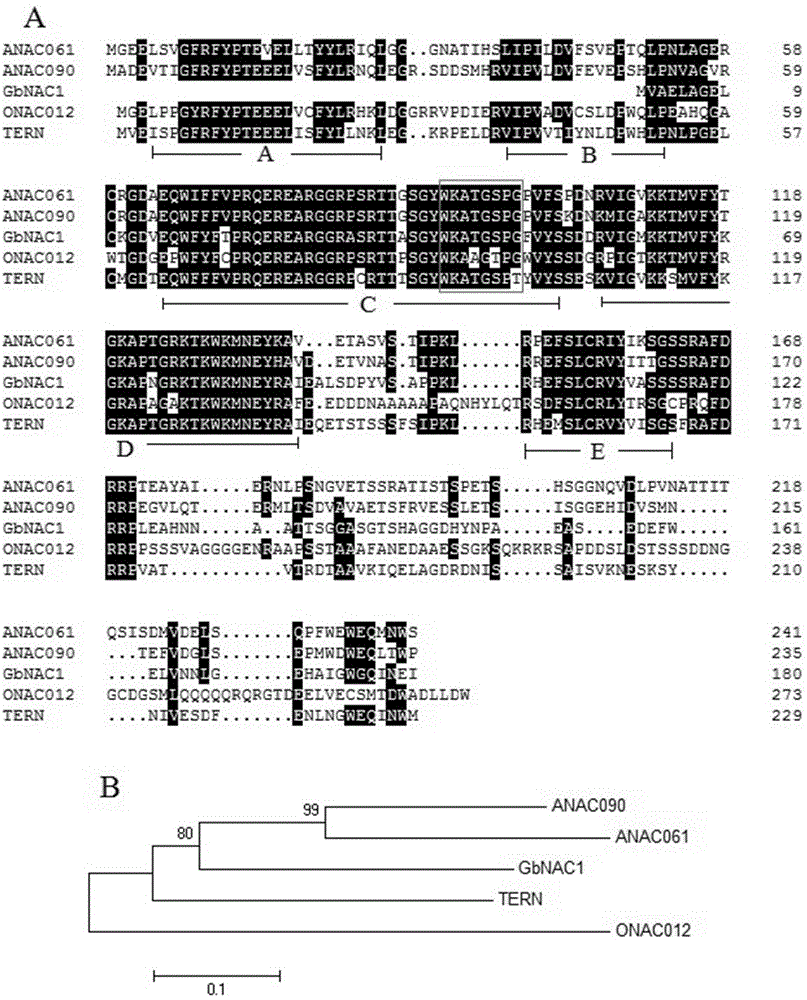
图2为GbNAC1的同源性序列比对和进化树分析,图A为同源性序列比对结果,其中A-E为5个保守结构域,方框表示与WRKY转录因子家族相似且保守DNA结合结构域;图B为进化树分析结果,其中ANAC061 (NP_001118771.1),ANAC090 (NP_197630.1),TERN (XP_009759500.1),ONAC012 (NP_001055672.1);
图3为GbNAC1与雷蒙得氏棉中的NAC转录因子的进化关系和保守结构域分析,其中黑框和箭头指向GbNAC1;
图4为续图3;
图5 为GbNAC1基因在本氏烟表皮细胞的定位;
图6为GbNAC1在转基因拟南芥中的GUS染色分析;
图7为黄萎病处理后GbNAC1基因在组织表达中的定量分析;
图8为GbNAC1基因在海岛棉新海15中的组织表达模式分析;
图9为VIGS干涉植株对黄萎病的抗性分析;其中A:棉花基因cla通过VIGS沉默后的表型;B:TRV:GbNAC1干涉植株的黄萎病抗性;C:发病后茎部解剖图;D:半定量检测基因沉默水平;E:黄萎病菌恢复实验;F:病情指数分析;
图10为GbNAC1超表达拟南芥植株的抗黄萎病菌分析。
具体实施方式
下面结合实施例对本申请做进一步的解释说明,在介绍具体实施例前,首先对下述实施例中所涉及部分生物材料及实验试剂等情况简要介绍如下。
生物材料:
棉花材料包括:海岛棉抗黄萎病品种新海15,16,由中国农业科学院棉花研究所杜雄明研究员提供;
黄萎病菌株:棉花黄萎病强致病力菌系V991,由中国农业科学院植物保护研究所简桂良研究员提供;
拟南芥材料:野生型拟南芥种子(Columbia-0);由河南大学植物逆境实验室提供(亦可由其他公开渠道获得),培养条件为21℃,16 h光照/8 h黑暗;
烟草材料:本氏烟种子(Nicotiana benthamiana);由河南大学植物逆境实验室提供(亦可由其他公开渠道获得),培养条件为25℃,14 h光照/10 h黑暗;
其他生物材料包括:大肠杆菌菌种DH5α、农杆菌菌种GV3101;
植物超表达载体 super- pCAMBIA1300;
BP反应入门载体pDONOR221;
基因亚细胞定位载体PK7FWG2.0;
启动子分析载体 pKGWFS7.0;
VIGS干涉技术载体pTRV1和pTRV2;
上述载体和菌种由河南大学植物逆境实验室提供(亦可由其他公开渠道获得)。
实验试剂:
反转录试剂盒、pMD18-T Vector、T4-DNA连接酶、限制性内切酶EcoR I,Kpn I、Ex Taq酶、荧光定量PCR试剂盒等均为TaKaRa公司产品;
质粒提取试剂盒和胶回收试剂盒购自天根公司;
荧光定量PCR专用板为Labwares公司产品;
RNA提取试剂盒购自Aidlab Biotech(Beijing,China)公司;
所用培养基及溶液有:LB液体(固体)培养基、YEP液体(固体)培养基、0.6 MS培养基、察氏(Czapek)培养基、PDA培养基等,均按本领域常规制备方法制备即可;
其他未描述的抗生素、激素类等试剂均为本领域常用试剂,不再赘述。
实施例1
海岛棉转录因子GbNAC1是本发明的基础,因而首先就该基因的克隆与获得过程简要介绍如下。
一、电子克隆
首先,根据本实验室前期转录组测序,筛选得到一条长1159bp,被注释为NAC家族成员的基因片段(unigene25035_kv-1),在其基础上进行电子克隆,得到一条序列。
二、设计引物,进行PCR扩增
首先,提取RNA,并反转录获得cDNA,具体为:
在棉花长出子叶后,提取棉花的RNA(用液氮研磨法提取RNA,具体步骤参考EASYspin Plus植物RNA快速提取试剂盒说明书),并反转录为cDNA;
cDNA模板制备体系具体设计为:
oligo(dT),2 μL;
RNA,3 μL;
RNA free H2O,8μL;
70℃水浴10min;
5×buffer,5μL;
dNTP,2μL;
M-MLV,1μL;
RNA inhibiter,0.5μL;
RNA free H2O,3.5μL;
42℃、90min,72℃,10min;
其次,设计引物序列,并进行序列扩增,具体为:
设计引物序列,并以上述所制备的cDNA为模板进行PCR扩增,以获得GbNAC1基因序列;
引物序列具体设计为:
GbNAC1F: 5’-ATGGTTGCAGAGCTTGCTGG-3’,
GbNAC1R: 5’-TTATATTTCATTAATTTGTC-3’;
PCR扩增时,20μL扩增体系设计为:
Template cDNA,0.4 μL;
GbNAC1F,0.4 μL;
GbNAC1R,0.4 μL;
5×TransStart FastPfμ Fly Bμffer,4 μL;
2.5 mM dNTP Mixture,2 μL;
TransStart FastPfμ DNA Polymerase,0.4 μL;
ddH2O,12.4μL;
PCR扩增程序, 94℃变性3 min;94℃变性30 s,56℃退火30 s,72℃延伸1 min,35个循环;72℃延伸10 min。
三、将所获得的GbNAC1基因片段与T载体连接
将上步中的PCR扩增产物,进行纯化并与pMD18-T载体连接,转化DH5α并进行菌液PCR检测,对阳性克隆进行测序验证(测序公司为华大)。
GbNAC1基因的PCR扩增如图1A所示,结果表明GbNAC1基因的ORF大小为543bp,菌液 PCR检测结果如图1B所示,得到5个阳性克隆。根据测序结果,我们向NCBI提交GbNAC1基因序列,获得GenBank ID: KP317496。
对GbNAC1基因的cDNA序列进一步进行相关的序列分析。利用ORF Finder(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)预测ORF的序列,利用DNAMAN软件进行了序列相似性分析,最后利用MEGA软件进行了序列的进化分析(采用Neighbor-Joining的计算方法构建了进化树)。
结果表明,GbNAC1的ORF含543bp,编码180个氨基酸。
根据查阅的文献资料,并利用DNAMAN软件,选取几个TERN类别的基因(ANAC061, ANAC090, TERN, ONAC012)进行序列对比,结果表明发现该基因含有C、D和E三个典型的结构域,并且含有与WRKY转录因子家族相似且保守DNA结合结构域(图2A)。
利用MEGA软件对上述基因序列构建进化树(Neighbor-Joining),结果发现GbNAC1与拟南芥中的ANAC061和ANAC090的同源性较高(图2B)。
利用HMMER软件,我们在雷蒙得氏棉里面搜到129个NAC家族的转录因子,发现GbNAC1与棉花D基因组的NAC基因相似度最高,并且含有C、D和E三个典型的NAC结构域(图3、图4)。
实施例2
为了进一步了解GbNAC1基因的相关特性,发明人利用特定载体对GbNAC1基因在细胞内的亚细胞定位、及利用GUS基因标记法对GbNAC1基因在植物组织中的表达情况进行了分析,相关实验过程简述如下。
GbNAC1基因在细胞内的亚细胞定位
利用Gateway技术将GbNAC1基因重组到亚细胞定位载体pK7FWG2.0,然后转化本氏烟(Nicotiana benthamiana),通过激光共聚焦显微镜观察GbNAC1基因在细胞内的定位表达情况。具体实验过程为:
首先,设计引物序列,以从棉花新海16叶片所提取的基因组gDNA作为模板,进行GbNAC1序列的扩增,电泳并回收扩增产物,引物序列如下:
nlsGbNAC1F:5’-AAAAAGCAGGCTATGGTTGCAGAGCTTGCTGG-3’,
nlsGbNAC1R:5’-AGAAAGCTGGGTCTATTTCATTAATTTGTC-3’。
其次,利用Gateway技术将GbNAC1基因重组到亚细胞定位载体pK7FWG2.0上;其中BP反应体系设计如下:
BP酶,0.5μL;
所回收PCR产物(所扩增GbNAC1序列),0.5μL;
pDONR221,0.5μL;
TE 8.0,1μL;
25℃保温一小时。
第三,保温结束后,将重组质粒转化大肠杆菌并涂板进行筛选,待平板上长出单菌落后,挑取单菌落进行扩大培养,并进行菌液PCR鉴定;对鉴定正确的阳性菌株进行测序验证;对测序验证正确的重组质粒再通过同源重组方式连接到目的载体pK7FWG2.0上,即LR反应,具体反应体系为:
LR酶,0.5μL;
重组质粒,0.5μL;
pK7FWG2.0,0.5μL;
TE 8.0,1μL;
25℃保温一小时。
第四,保温结束后,将重组质粒转化大肠杆菌并涂板进行筛选,待平板上长出单菌落后,挑取单菌落进行测序验证,测序验证正确的重组质粒即为重组的含有GbNAC1基因的亚细胞定位重组载体pK7FWG2.0,然后将该重组载体转化农杆菌GV3101备用,以便进一步浸染转化本氏烟(Nicotiana benthamiana)。
第五,将本氏烟(Nicotiana benthamiana)种子点播于湿润营养土表面,在表面覆盖一层塑料薄膜以保持湿润,种子萌发后,将萌发的烟草幼苗在温室中(25℃,14 h光照/10 h黑暗)培养约三周备用。
第六,将“第四步”中所获得的转化后农杆菌菌株接种于100 mL液体LB培养基中(50 mg/mL壮观霉素,50 mg/mL庆大霉素,100 mmol/L乙酰丁香酮),28℃摇瓶培养过夜,待OD600=0.6左右时,离心收集菌体,然后再将菌体重悬于MS悬浮液中(含有100 mmol/L乙酰丁香酮);
选择“第五步”中叶片大小适中的烟草叶片,将上述重悬后菌液注射于其下表皮,直到菌液充满整个叶片,在光周期16 h/8 h、25℃的条件下培养2 d;
随后注射菌液的烟叶的下表皮撕下来一小块,在激光共聚焦显微镜下进行观察与拍照。
结果显示,GbNAC1在细胞核内表达;为进一步验证,对其进行DAPI染色,结果显示绿色荧光的部位发出蓝光,证明该基因确实是在细胞核中表达(图5)。
染色法对GbNAC1基因在植物组织中的表达分析
首先,构建含有GUS基因的重组表达载体,具体为,在phytozome网站上查找GbNAC1基因的启动子序列,然后设计如下引物序列:
pGbNAC1F:5’- AAAAAGCAGGCTGACTTGTAAACTGGTGCCTAT -3’,
pGbNAC1R:5’- AGAAAGCTGGGTGTAGCTGATCTATACGTGTTGT -3’;
通过Gateway技术将上述引物扩增的GbNAC1基因的启动子序列构建到GUS载体pKGWFS7.0上(具体操作参考上述GbNAC1基因在细胞内的亚细胞定位过程中相关操作);将筛选获得的构建正确的重组质粒转化农杆菌菌株GV3101备用;
其次,将拟南芥种子用0.1%氯化汞消毒5 min,无菌水清洗5次,点种在0.6% MS培养基上,4℃春化2~3天,然后放入材料室内,待其长出幼苗后移入土中;
第三,利用拟南芥花絮侵染法,将上述含有含有重组质粒的农杆菌菌株浸染拟南芥花絮,并筛选获得转基因植株;
第四,收集“第三”步中转基因植株种子,于含卡那霉素(50mg/mL)的MS培养基上进一步筛选,并培养获得阳性种子;收集阳性种子并播种,待其植株长大后,将整个实验植株剪下,用清水清洗干净后整个浸入染色液中,37℃暗处理过夜,次日分别用90%和70%的酒精脱色数小时,直到植株呈透明状,干燥后于显微镜下观察变蓝的部位并拍照。
结果表明,GbNAC1基因在根、茎、叶中均有表达,但是主要集中在维管束(图6)。
实施例3
本实施例主要介绍一下GbNAC1基因在棉花组织中的分布情况及其在黄萎病菌诱导下基因表达的变化情况,相关实验过程简述如下。
GbNAC1基因在棉花组织中的分布情况
为了对GbNAC1基因的功能有初步了解,因而有必要就GbNAC1基因在不同组织中的分布情况有初步了解,相关实验过程主要为:
将海岛棉新海15种子播种后,待其长出两片真叶时,分别取样根、茎、真叶、子叶、茎尖(样品处理前-80℃保存),液氮研磨法分别提取其RNA,并反转录为cDNA,作为荧光定量PCR的扩增模板。
荧光定量PCR对不同组织样品中的GbNAC1基因表达模式进行分析,结果如图7所示。
从图7中可以看出,GbNAC1基因在上述各组织中均有表达,与GUS染色结果相一致,但是在真叶中的表达量最高,而在茎尖中的表达量最低。
黄萎病菌诱导模式下棉花中GbNAC1基因表达变化情况
25℃下,将棉花种子在培养箱中浸泡水中8~12h,然后置于培养皿中保湿培养一天,种子萌发后移入营养土中生长(25 ℃,16 h光照/8 h黑暗);棉花幼苗两叶一心后,进行黄萎病病菌接种处理,采用伤根法接种黄萎病病菌,具体为,将棉花幼苗从盆钵中取出,放置于黄萎病菌液(将黄萎病菌v991在察氏液体培养基中振荡培养(25℃,150 rpm),培养四天后用纱布过滤菌液,菌液浓度为1×107个/mL)中浸泡40s,然后再放回盆钵中培养;同时设置对照组作为对比(对照组以蒸馏水替代黄萎病菌感染液,其他操作相同)。
接种病菌后,分别在0 h、1 h、4 h、8 h、12 h、24 h、36 h、48 h和72 h的时间点取样叶片,提取RNA后反转录获得cDNA,以该cDNA为模板,采用qRT-PCR技术对GbNAC1基因的表达情况进行分析。
qRT-PCR时,引物序列设计如下:
qGbNAC1F: 5’- GACCTTGAGCCTTGGGACC-3’,
qGbNAC1R: 5’- CTTCCCTCTCTTGTCTTGGTGTA -3’;
qRT-PCR时,以棉花Ubiquitin7 gene(UBQ7)基因作为内参基因,引物序列设计如下:
UBQ7-F:5’-GAAGGCATTCCACCTGACCAAC-3’,
UBQ7-R:5’-CTTGACCTTCTTCTTCTTGTGCTTG-3’;
利用ABI 7300 Real Time PCR系统(Applied Biosystems,USA),使用SYBR绿色荧光(Bio-Rad,USA)进行定量分析,结果如图8所示。
从图8中可以看出,在浸染黄萎病菌后,除在72 h的时间点以外,每个时间点中,实验组的表达量均低于空白对照组,因为黄萎病的诱导是动态的,所以出现诱导表达不连续的现象,该结果表明黄萎病菌能抑制该基因的表达,初步表明,目的基因在感染黄萎病初期受诱导表达量降低。
实施例4
发明人使用VIGS技术对棉花植株内的GbNAC1基因进行了沉默,并进行黄萎病接种,借此来研究GbNAC1基因与黄萎病的关系,相关实验过程介绍如下。
一、构建含有GbNAC1基因的VIGS载体
首先,设计引物序列,并进行PCR扩增,获得GbNAC1基因序列,引物序列具体为:
vGbNAC1F:5’-CCGGAATTCTCATACTTGTAGACCAAAGGAAC-3’,
vGbNAC1R:5’-CGGGGTACCAGTGATAGCATAGAAAAGCAATA-3’;
其次,将TRV2空载体和PCR扩增获得的GbNAC1基因序列片段分别用EcoR I和Kpn I进行双酶切,50 μL酶切体系设计如下:
PCR扩增产物(或TRV2质粒),43μL;
EcoR I,0.75μL;
Kpn I ,0.75μL;
0.5×K Buffer,5μL;
BSA,0.5μL;
37℃酶切过夜,分别回收并纯化酶切产物;
采用T4 DNA连接酶对上述酶切产物进行连接,以构建重组质粒,10μL连接体系设计如下:
PCR酶切产物,7μL;
pTRV2酶切产物,1μL;
T4 DNA连接酶,1μL;
10×Buffer,1μL;
16℃连接过夜。
将上述连接产物转化DH5α感受态细胞,涂板筛选并进行菌液PCR鉴定,对鉴定正确的阳性克隆质粒进一步进行测序鉴定,获得构建正确的重组质粒载体;将构建正确的重组质粒载体转化农杆菌GV3101,备用。
所述连接产物转化DH5α感受态细胞的具体过程为:将连接产物与大肠杆菌DH5α感受态细胞均匀混合,冰上放置30min,然后42℃水浴热激90s,立即置于冰上2min;然后加入空白液体LB培养液1mL,37℃、180 rpm摇床振荡培养1 h,然后将菌液均匀涂布于含有卡那霉素(50mg/ml)的LB培养皿中,37℃倒置培养12~16 h;
挑取菌落进行培养,然后进行菌液PCR,对鉴定正确的菌液测序,测序正确的质粒即为重组后的质粒载体,将测序正确的克隆提取质粒后保存备用。
所述重组质粒载体转化农杆菌GV3101的具体过程为:将农杆菌GV3101感受态细胞200μL与重组质粒5μL均匀混合后,冰上放置30min;于液氮中速冻70s后迅速放入37℃水浴中5min,再置于冰上2min;加入空白液体LB培养液1mL,28℃、180 rpm振荡培养4h,然后将菌液均匀涂布于含有50mg/mL卡那霉素和50mg/mL庆大霉素的LB培养皿中,28℃倒置培养2~3天;
待平板上长出均匀的单菌落,挑取数个单菌落于液体LB培养液(50mg/mL卡那霉素+50mg/mL庆大霉素)中,28℃、220 rpm振荡培养数小时,待到菌液混浊,挑取混浊的菌液做菌液PCR检测,然后进行菌液PCR鉴定,对鉴定正确的阳性克隆菌液直接转化棉花操作,或者加甘油后-80℃保存备用。
二、转化棉花
取pTRV-GbNAC1、pTRV1、pTRV2(空载体)和pTRV-GhCla1农杆菌分别于含庆大霉素(50mg/mL)、卡那霉素(50mg/mL)的液体LB培养基培养至OD600=0.6~0.8左右时,收集菌体并重悬于浸染液,浸染液成分和各成分终浓度为:MES(1M)、AS(150 mM)、MgCl2(1M)。
将pTRV1分别与pTRV-GbNAC1、pTRV2(空载体)以及pTRV-GhCla1的农杆菌悬浮液以1︰1的比例混合,即:pTRV1+TRV2、pTRV1+ pTRV-GbNAC1,pTRV1+ pTRV-GhCla1;并将重悬液混合均匀后25℃静置1h。
当棉花幼苗的两片子叶展平时,用注射器将菌悬液注射到子叶中,避光培养24 h后,再恢复正常条件(光照16 h/黑暗8 h)培养。
在棉花幼苗生长到两叶一心时,提取RNA并反转录获得cDNA,进行半定量RT-PCR检测,以确认VIGS的干涉效率。
之所以采用GhCla1基因的干涉情况作为VIGS体系的阳性对照,是因为GhCla1基因是一个与叶绿素合成相关的基因,若其被成功干涉掉,则叶片会出现白化症状(图9A),因而易于对干涉效率做出评价,结果表明该VIGS体系是有效的。
半定量RT-PCR检测结果表明, GbNAC1基因被高效干涉掉(图9D)。
在确认GbNAC1基因被沉默掉后,对棉花幼苗进行黄萎病接种处理(具体操作参考实施例3)。
实验结果表明,用黄萎病菌液处理一周左右后,棉花植株开始表现出相应的黄萎病症状,但GbNAC1基因沉默后的棉花植株发病较为明显(图9B),并且病情指数显著升高(9F)。
棉花感染黄萎病菌且发病以后,维管束一般会褐化,所以发明人进一步对发病后棉花进行剖茎处理,根据实验组和对照组的茎剖面,可以发现,实验组茎的褐化现象明显加重,发病更为严重(图9C)。
进一步进行黄萎病菌恢复实验,将黄萎病侵染的实验组和对照组的棉花幼苗的茎分别剪成1 cm长的片段,并用70%的酒精中清洗30 s,然后再用0.1%的HgCl2清洗1 min,最后用无菌水冲洗5次;将上述处理过的茎段放置于PDA培养基上,25℃培养一周后观察。
结果表明实验组干涉植株的发病情况比对照组要严重(9E)。
实施例5
为了更好的研究GbNAC1基因的相关功能,发明人借助拟南芥这一植物中的模式植物,通过在拟南芥中超量表达该基因,以研究GbNAC1基因超表达后植株的黄萎病抗性。相关实验介绍如下。
一、构建含有GbNAC1基因的超表达载体
首先,设计引物序列,并对GbNAC1基因序列进行PCR扩增,引物序列设计为:
oeGbNAC1F:5’-GCTCTAGAATGGTTGCAGAGCTTGCTGG-3’,
oeGbNAC1R:5’-GGACTAGTTTATATTTCATTAATTTGTCCCCA-3’;
其次,将super-pCAMBIA1300载体和上述PCR扩增获得的GbNAC1基因序列片段分别用Xba I和Spe I进行双酶切,回收并纯化酶切产物;
第三,利用T4 DNA连接酶将上述酶切产物,在16℃连接过夜;
第四,将连接产物转化大肠杆菌DH5α感受态细胞,并筛选获得阳性克隆菌株,经测序验证后,对构建正确的重组质粒进行提取;
第五,将构建正确的重组质粒转化农杆菌GV3101,PCR检测鉴定,对鉴定正确的菌液加甘油-80℃保存备用。
二、农杆菌介导拟南芥的遗传转化
采用农杆菌介导的花絮浸染法转化拟南芥,并筛选获得转基因植株,具体构建过程为:
将拟南芥种子用0.1%的氯化汞消毒5 min,再用无菌水清洗5次,点种在0.6% MS培养基(50mg/mL卡那+25μg/mL潮霉素)上,春化2~3天后放到光照培养室,观察苗的生长状况,将未生根的或已生根但叶片呈黄白色生长异常的苗舍弃,已经生根的并且叶片生长正常的苗移栽到土壤中,正常培养;
待拟南芥结出果荚后,将种子回收;
重复上述种植过程,连续进行三代培养以筛选获得纯合的超表达转基因植株。
三、对超表达拟南芥植株的黄萎病侵染
将步骤二中所收获的纯合的转基因拟南芥种子进行播种,待生长30天后用黄萎病菌液进行处理,观察表型变化,并对拟南芥发病株数进行统计。
结果表明,GbNAC1基因超表达后,发病株数明显低于野生型植株。因此可以认为,GbNAC1基因超量表达后,转基因拟南芥对黄萎病菌的抗性明显增强(图10)。
综合VIGS实验和超表达实验,可以得出GbNAC1基因在抗黄萎病过程中起正调控作用。
需要说明的是,上述实施例中所涉及的实验操作,部分实验操作具有一定的通用性,因而未加详细描述,未详细描述部分内容以其他实施例中相关操作为准或者参考现有技术即可,不再赘述。
- 还没有人留言评论。精彩留言会获得点赞!